Forml at forst det eksperimentelle grundlag for moderne



































































- Slides: 97

Formål - at forstå det eksperimentelle grundlag for moderne molekylærbiologisk/genteknologisk arbejde -at kunne bruge dette grundlag til at tolke virkelige molekylærbiologiske data taget fra den videnskabelige litteratur - at kunne bruge dette grundlag til at designe molekylærbiologiske experimenter til belysning af biologiske problemstillinger - at give en dybgående behandling af den makromolekylære syntese og dennes regulering

Eksamensrelevansen af dette er: -At dette ikke er et fag man består ved at kunne lærebogen uden af (tværtimod) -Derimod består man ved at forstå, læs og forstå derfor de originale eksperimenter som lærebogen er rig på i de enkelte afsnit. Forstå hvad de viser og hvorledes samme teknik generelt kan bruges - Sørg for at i forstår problemstillingerne og løsningerne på de opgaver vi gennemgår hver onsdag eftermiddag -Erfaringsmæssigt oplever de studerende problemorienterede opgaver som svære -Fortvivl ikke hvis opgaverne virker svære når i forbereder jer hjemme. Det vigtige er, at forstår dem efter regnetimerne

Restriktions enzymer (restriktions endonukleaser)

Restriktions enzymer genkender og binder en bestemt nukleotid-sekvens og spalter DNAet. Kan deles op i tre typer. Type I binder til en bestemt DNA sekvens, men skærer et tilfældigt sted derfra. Type II binder og skærer samme sekvens. Type III binder og skærer også samme sekvens. Type II enzymer vi bruger i laboratoriet.

Restriktionsenzymer navngives efter den bakterie hvorfra de oprindelig er isoleret Sædvanligvis har de et navn på tre bogstaver, det første fra genus og de næste to fra species navnet. Nogle gange har enzymer et fjerde bogstav som refererer til den specifikke stamme enzymet oprindeligt er fundet i. De første tre bogstaver af enzymnavnene skrives altid med stort. F. eks. Sma. I, Serratia marcescens Eco. RI, Escherichia coli Pst. I, Providencia stuartii

Type II enzymer genkender og spalter som regel en 4, 6 eller 8 nukleotider lang sekvens af dobbeltstrenget DNA. Nogle skærer DNAet lige over (blunt end skæring). F. eks. Sma. I, 5’ CCCGGG 3’ 3’ GGGCCC 5’ CCCOH 3’ 3’ GGGp 5’ 5’ p. GGG 3’ 3’OHCCC 5’

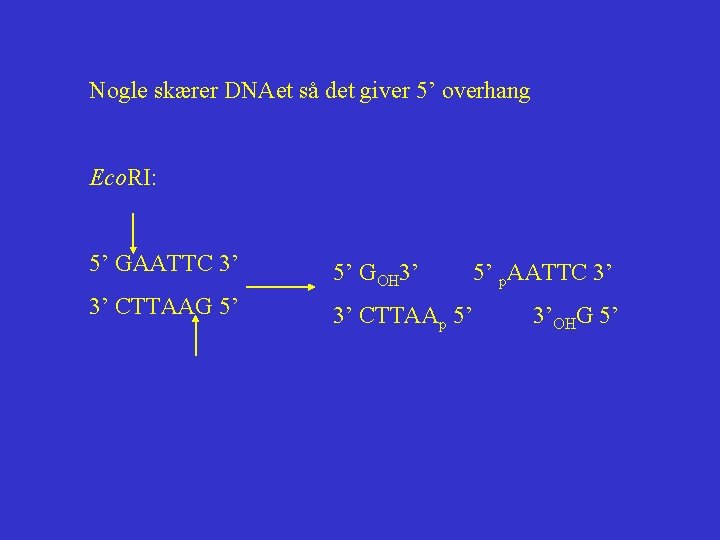
Nogle skærer DNAet så det giver 5’ overhang Eco. RI: 5’ GAATTC 3’ 5’ GOH 3’ 3’ CTTAAG 5’ 3’ CTTAAp 5’ 5’ p. AATTC 3’ 3’OHG 5’

Nogle skærer med 3’ overhang Pst I: 5’ CTGCAG 3’ 5’ CTGCAOH 3’ 3’ GACGTC 5’ 3’ Gp 5’ 5’p G 3’ 3’ OHACGTC 5’

http: //www. neb. com/nebecomm/products/category 1. asp New England Biolabs, den største leverendør af restriktionsenzymer

Hvor skærer restriktionsenzymerne i et givet stykke DNA ? Der findes diverse freeware på nettet bl. a. webcutter 2 http: //rna. lundberg. gu. se/cutter 2/

atgtct aaaggtgaag aattattcac tggtgttgtc ccaattttgg ttgaattaga 121 tggtgatgtt aatggtcaca aattttctgt ctccggtgaaggtg atgctactta 181 cggtaaattg accttaaaat ttatttgtac tactggtaaa ttgccagttc catggccaac 241 cttagtcact actttcggtt atggtgttca atgttttgct agatacccag atcatatgaa 301 acaacatgac tttttcaagt ctgccatgcc agaaggttat gttcaagaaa gaactatttt 361 tttcaaagat gacggtaact acaagaccag agctgaagtc aagtttgaag gtgatacctt 421 agttaataga atcgaattaa aaggtattga ttttaaagaa gatggtaaca ttttaggtca 481 caaattggaa tacaactata actctcacaa tgtttacatc atggctgaca aacaaaagaa 541 tggtatcaaa gttaacttca aaattagaca caacattgaa gatggttctg ttcaattagc 601 tgaccattat caacaaaata ctccaattgg tgatggtcca gtcttgttac cagacaacca 661 ttacttatcc actcaatctg ccttatccaa agatccaaac gaaaagagag accacatggt 721 cttgttagaa tttgttactg ctgctggtat tacccatggt atggatgaat tgtacaaata 781 a

Restriktionsenzymer forekommer sammen med et modifikationssystem for at undgå fordøjelse af bakteriens eget DNA Modifikationsenzymerne er methylaser, som methylerer nukleotider i restriktionssitet Modifikation af den ene DNA streng nok til at forhindre nedbrydning (ellers ville bakterien få problemer under DNA replikationen) Dette har den praktiske betydning, at man skal vælge en E. coli stamme, der er restriktions-negativ, når man ønsker at introducere DNA fra en anden organisme eller anden E. coli stamme for at undgå fordøjelse af DNAet. Type I og III’s nuklease aktivitet sidder i samme protein som methyleringsaktiviteten For Type II enzymer er aktiviteterne adskilt i forskellige proteiner

For at DNA stabilt kan opretholdes i en voksende bakteriepopulation er det essentielt at - det kan replikeres -at det fordeles effektivt mellem moder og dattercelle -stabil opretholdelse af DNA fragmenter fra en anden organisme må derfor sikres ved at DNA fragmentet ”sættes sammen med” DNA som har de to ovennævnte egenskaber -dette DNA benævnes en ”vektor” og er typisk enten et plasmid eller en virus/bakteriofag -plasmider er naturligt forekommende, autonomt replikerende cirkulære stykker DNA som bibringer bakterien en selektiv fordel i forskellige miljøer (f. eks. antibiotika resistens) -in vitro fremstillede varianter af naturligt forekommende plasmider og bakteriofager.




Hvordan får vi DNAet ind i E. coli igen ? Det ligerede DNA introduceres i en E. coli stamme ved transformation (hvis et plasmid) eller ved transduktion (hvis en bakteriofag) For at E. coli kan optage DNA skal cellerne gøres kompetente ved behandling med Ca. Cl 2 eller ved at cellerne udsættes for et kraftigt elektrisk felt i tilstedeværelse af DNA (elektroporation)

Hvordan får man fat på en bestemt genomisk eller c. DNA klon ? Med en klon vil vi forstå en bakterie der indeholder en vektor med et bestemt stykke genomisk/c. DNA Fremstilling af genom og c. DNA biblioteker Screening af genom- og c. DNA biblioteker Ekspressionskloning Komplementation i mikroorganismer Subtraktionskloning Microarray Differential display

Konceptet er at få indsat genomsekvenser eller c. DNA sekvenser i en vektor på den mest effektive måde, så alle sekvenser i genomet/alt m. RNA er repræsenteret i biblioteket. Biblioteker fremstilles næsten altid i E. coli En vigtig faktor er her introduktion af DNAet i E. coli. (plasmider transformeres, bakteriofager transduceres)

Hvor stort skal et bibliotek være for at alle sekvenser er med ? N = ln(1 -P)/ln(1 - 1/n) N = antal kloner i biblioteket P = sandsynligheden for at en N= ln(1 -0. 95) given sekvens er med i ln(1 - 1/2. 6 106 kb/20 kb) biblioteket N = 4. 2 105 n = genomets størrelse divideret med den gennemsnitlige fragmentlængde i biblioteket data er fra fremstiling af et genombibliotek fra mus

Når man skal fremstille et repræsentatitvt bibliotek er det vigtigt at alle parametre er optimerede En af de vigtige parametre er introduktion af det rekombinante DNA ind i E. coli. Her kan man benytte sig af at bakteriofager fra naturens side er selekteret til at inficere deres værtsorganisme yderst effektivt Rekombinante bakteriofager har således en evne til at introducere DNA, med en mellem 10 og 100 gange højere effektivitet end transformation med plasmid DNA

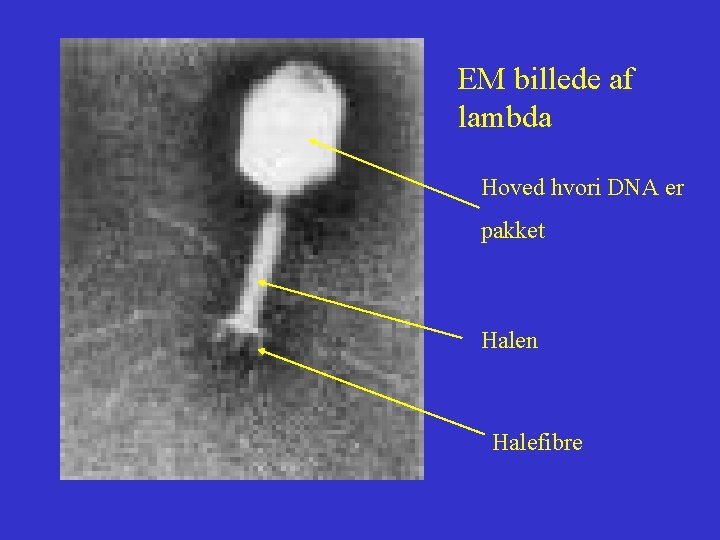
EM billede af lambda Hoved hvori DNA er pakket Halen Halefibre

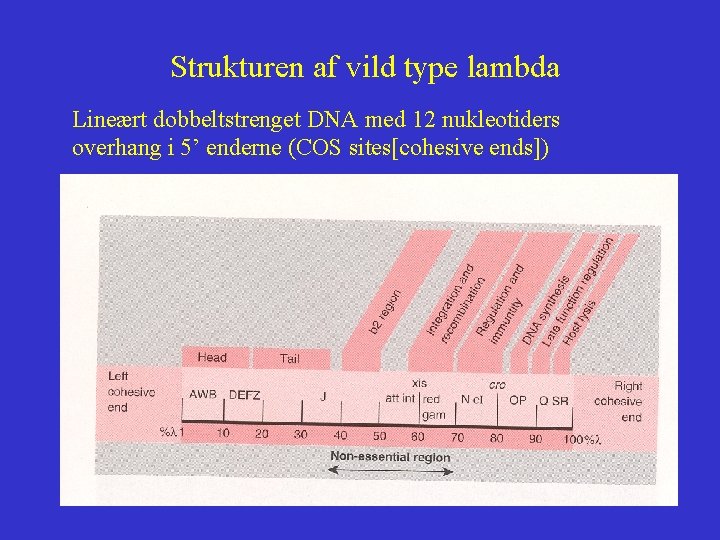
Strukturen af vild type lambda Lineært dobbeltstrenget DNA med 12 nukleotiders overhang i 5’ enderne (COS sites[cohesive ends])

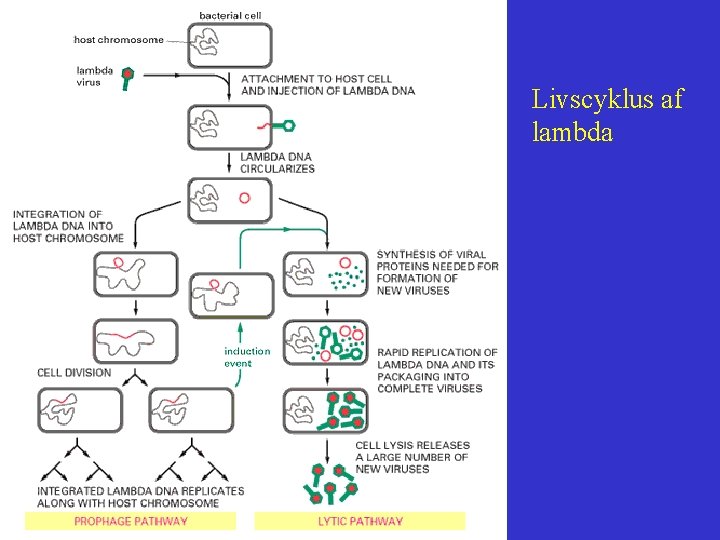
Lambda livscyklus lysogen cyklus lytisk cyklus

Livscyklus af lambda

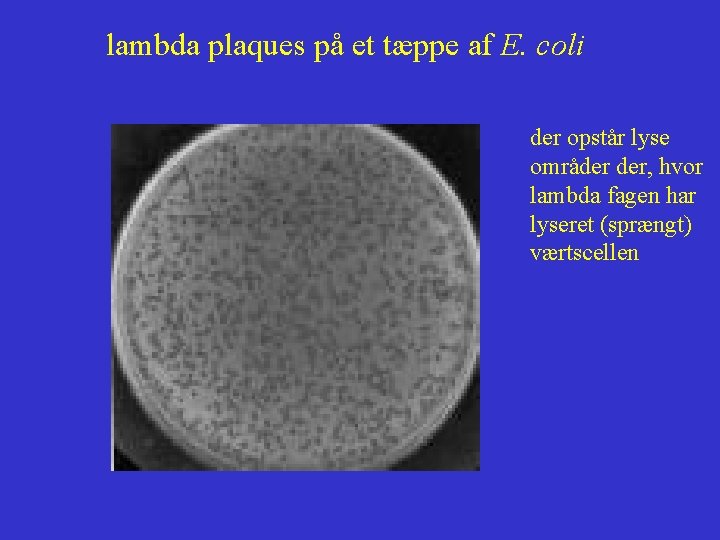
lambda plaques på et tæppe af E. coli der opstår lyse områder der, hvor lambda fagen har lyseret (sprængt) værtscellen



Krav til størrelsen af lambda for at den kan pakkes i faghoveder. fra 75% til 105% af vildtypens størrelse

En vild type lambda kan ikke gro på en E. coli stamme der er lysogen for bakteriofagen P 2 En lambda der mangler rekombinationsgenerne red, gam kan derimod godt gro på en P 2 lysogen stamme Erstattes red gam regionen med ”fremmed” DNA kan den rekombinante fag gro på en P 2 lysogen stamme Spi fænotypen Spi = sensitive to P 2 inhibition red, gam

To typer lambda vektorer Replacement vektorer Insertion vektorer

Vigtigt at der findes unikke restriktionssites i lambda til at klone genomisk eller c. DNA. Vigtigt at rekombinante fager nemt kan skilles fra ikke rekombinante, da det ellers er svært at se, hvor stor biblioteket reelt er Nemt at flytte DNA fra lambda fagen til et plasmid Mulighed for in vitro transkription

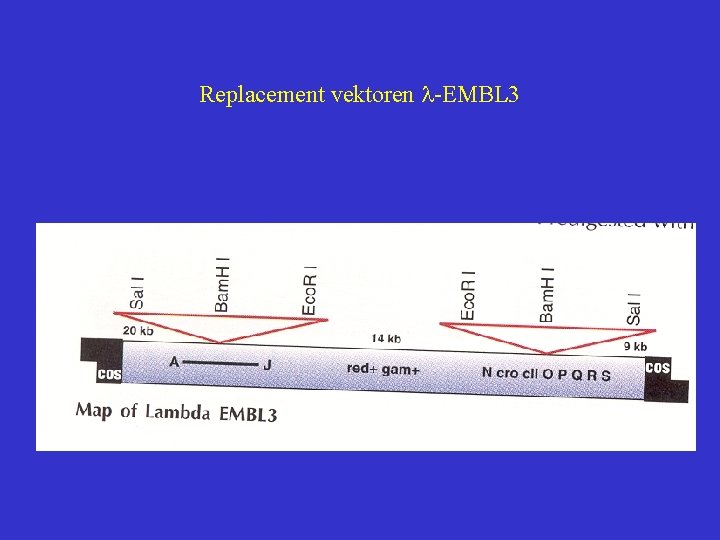
Replacement vektoren l-EMBL 3

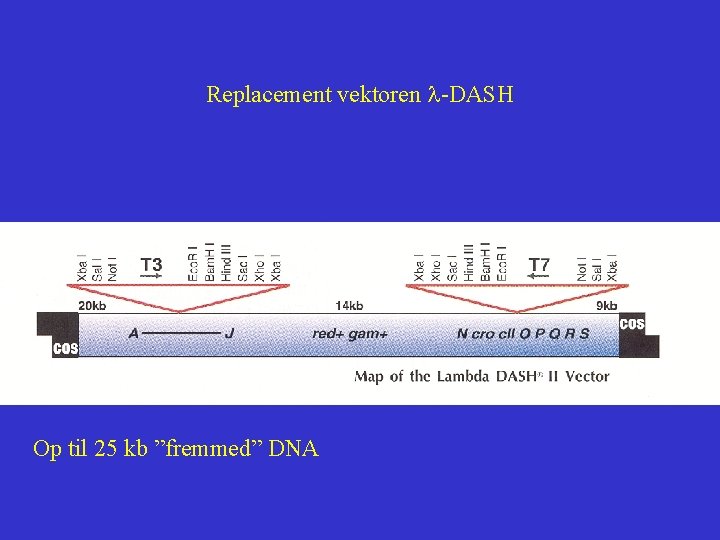
Replacement vektoren l-DASH Op til 25 kb ”fremmed” DNA

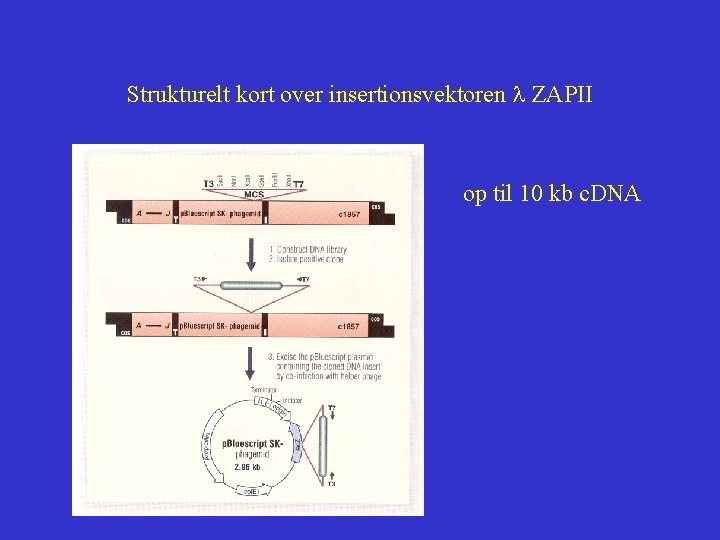
Strukturelt kort over insertionsvektoren l ZAPII op til 10 kb c. DNA

Insertions vektoren l ZAP express

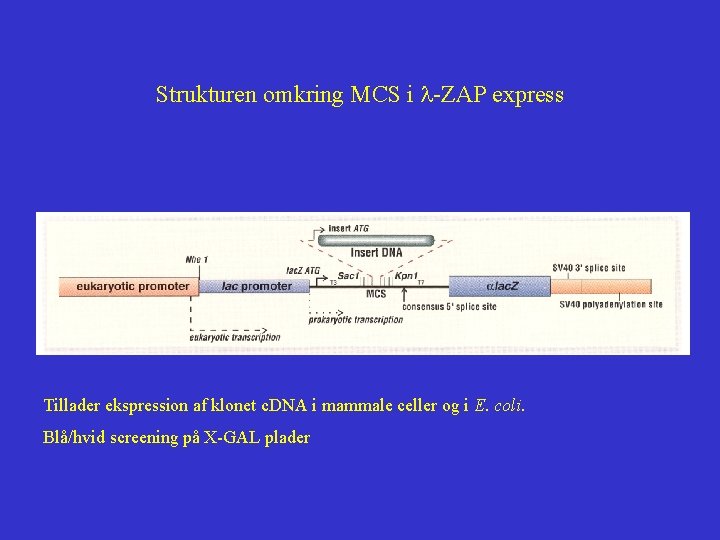
Strukturen omkring MCS i l-ZAP express Tillader ekspression af klonet c. DNA i mammale celler og i E. coli. Blå/hvid screening på X-GAL plader

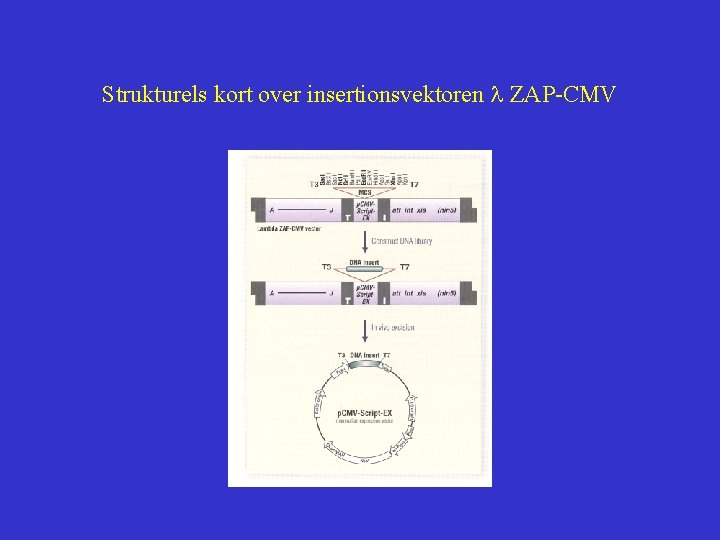
Strukturels kort over insertionsvektoren l ZAP-CMV

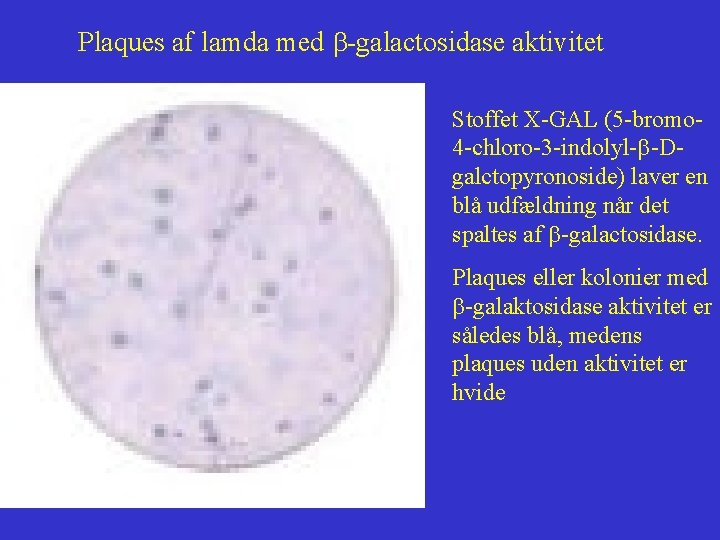
Plaques af lamda med b-galactosidase aktivitet Stoffet X-GAL (5 -bromo 4 -chloro-3 -indolyl-b-Dgalctopyronoside) laver en blå udfældning når det spaltes af b-galactosidase. Plaques eller kolonier med b-galaktosidase aktivitet er således blå, medens plaques uden aktivitet er hvide

Strukturelt kort over Cosmidet Super. Cos Ori, E. coli replikations origin Neo. R, resistens gen mod neomycin (G 418) SV 40 ori, origin fra SV 40 virus Cos, cos sites til pakning i l hoveder T 3/7, T 3/7 promotorer til in vitro transkription op til 50 kb fremmed DNA

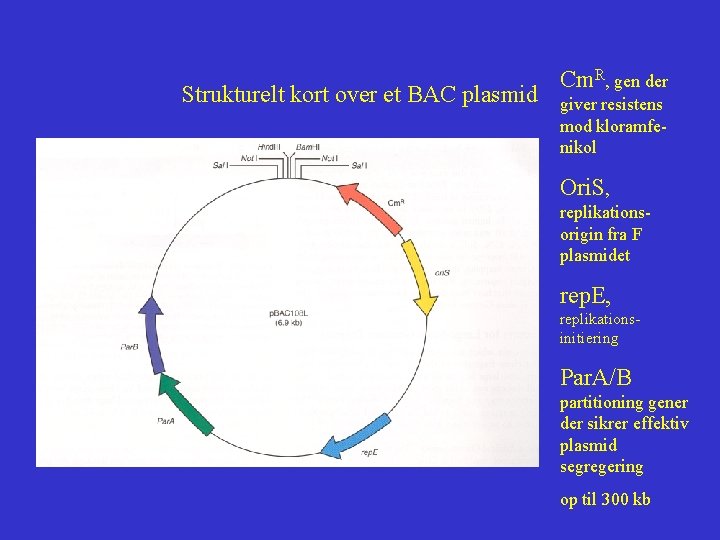
Strukturelt kort over et BAC plasmid Cm. R, gen der giver resistens mod kloramfenikol Ori. S, replikationsorigin fra F plasmidet rep. E, replikationsinitiering Par. A/B partitioning gener der sikrer effektiv plasmid segregering op til 300 kb

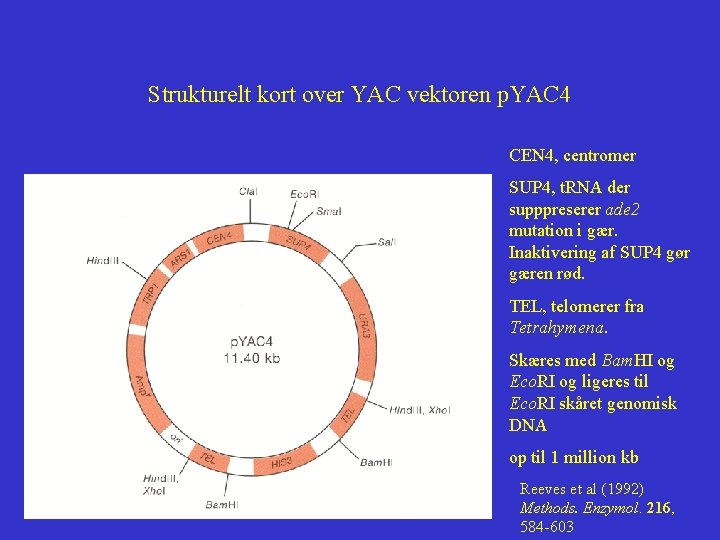
Strukturelt kort over YAC vektoren p. YAC 4 CEN 4, centromer SUP 4, t. RNA der supppreserer ade 2 mutation i gær. Inaktivering af SUP 4 gør gæren rød. TEL, telomerer fra Tetrahymena. Skæres med Bam. HI og Eco. RI og ligeres til Eco. RI skåret genomisk DNA op til 1 million kb Reeves et al (1992) Methods. Enzymol. 216, 584 -603

Hvordan får vi fremskaffet definerede overlappende genomiske DNA fragmenter ? Vi vil gerne have sekvensen af sammenhængende stykke genomisk DNA Løsningen er at skære DNAet partielt (ufuldstændigt) med et enzym der skærer ofte, typisk Sau 3 A (5’ GATC 3’) Sau 3 A genererer ender kan ligeres til Bam. HI skåret vektor DNA

Sau 3 A genkendelsessites Hvis vi skærer DNAet med lidt Sau 3 A i så kort tid, at vi får genereret fragmenter mellem 14 -20 kb vil vi få en række forskellige overlappende fragmenter ud af det. Kan vi bestemme sekvensen af de overlappende fragmenter, kan vi sammenstykke sekvensen af hele området.

c. DNA syntese m. RNA oprenses på en affinitetssøjle med polyd. T eller poly. U Kræver enzymet revers transkriptase der fremstiller en DNA kopi af m. RNA Revers transkriptasen kræver en ”primer” (et 15 – 25 nukleotider langt stykke DNA eller RNA) for at kunne starte c. DNA syntesen Kræver d. NTP (de-oxy-nukleotid trifosfater) og de rigtige fysisk-kemiske forhold Nogle revers transkriptaser har aktivitet ved 55 -60 o. C, hvilket er en fordel, hvis m. RNA har meget sekundær struktur Som primere kan anvendes polyd. T som annealer til poly. A halen på m. RNA eller random hexamerer (en blanding af alle 26 mulige hexamerer) eller en m. RNA specifik primer.

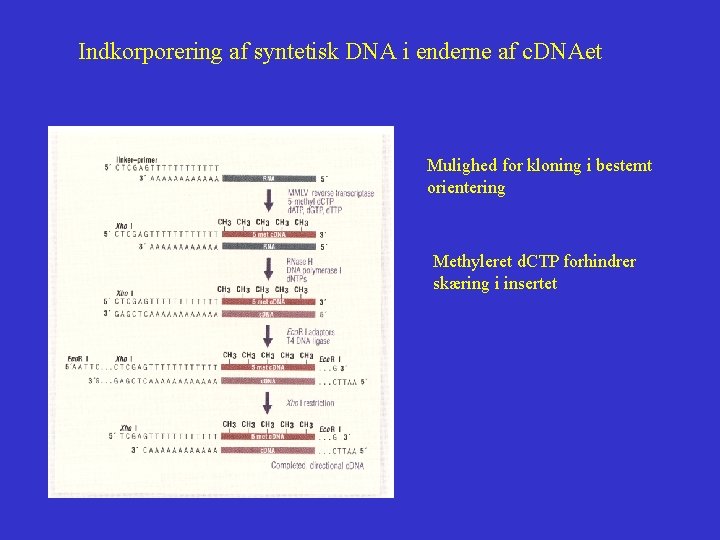
Indkorporering af syntetisk DNA i enderne af c. DNAet Mulighed for kloning i bestemt orientering Methyleret d. CTP forhindrer skæring i insertet

Homopolymer tailing af c. DNA


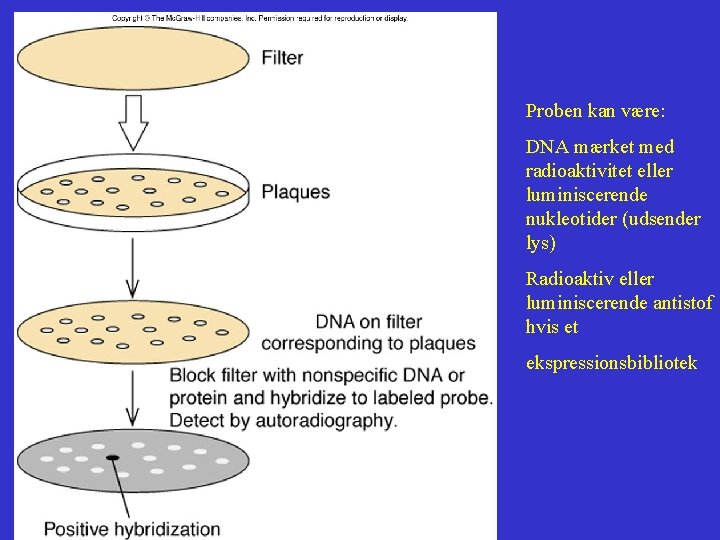
Screening af biblioteket kræver noget information om det gen man ønsker at klone (eller information om en del af det protein man ønsker at klone genet for)

Screening med en probe Screening med et antistof

Hvordan får man fat i proben ? Nogle har klonet et lignende gen Nogle har klonet det samme gen fra en anden specie Nogle har oprenset proteinet og bestemt noget af den primære struktur.

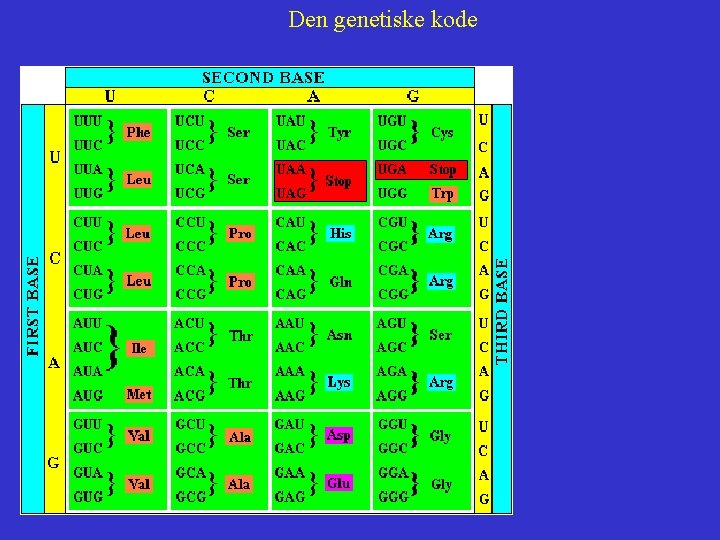
Den genetiske kode

Plaque lift eller eventuelt koloni lift

Proben kan være: DNA mærket med radioaktivitet eller luminiscerende nukleotider (udsender lys) Radioaktiv eller luminiscerende antistof hvis et ekspressionsbibliotek


Polymerase chain reaction (PCR) måde at amplificere DNA på Kræver: to specifikke primere (syntetisk DNA, typisk mellem 18 og 25 nukleotider lange) en termostabil DNA polymerase d. NTP, Mg 2+ og buffer

Kloning af manglende 5’ eller 3’ sekvenser af c. DNA EST (expressed sequence tags) små stumper c. DNA sekvens fra sekventering af c. DNA biblioteker



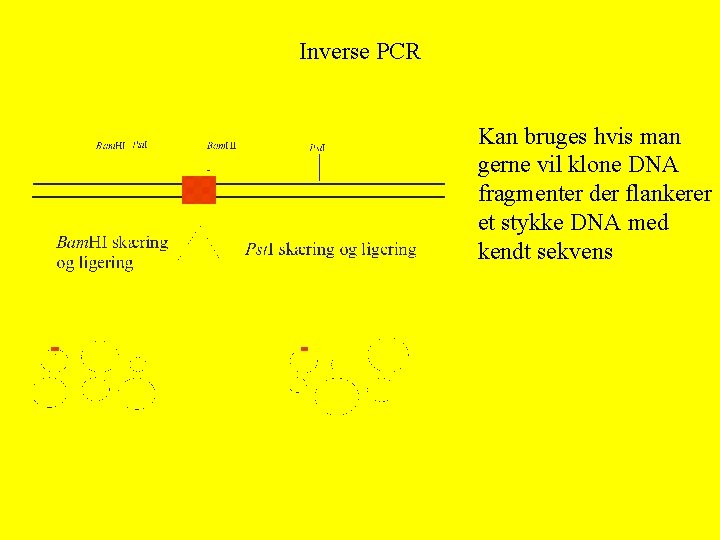
Inverse PCR Kan bruges hvis man gerne vil klone DNA fragmenter der flankerer et stykke DNA med kendt sekvens

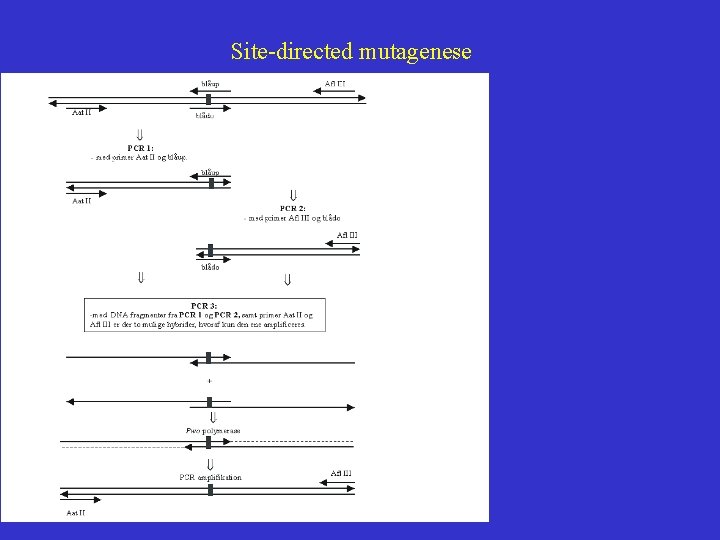
Site-directed mutagenese

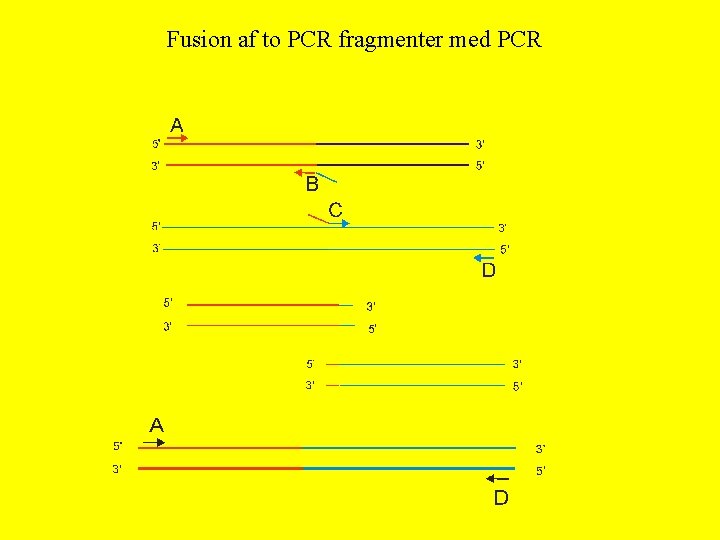
Fusion af to PCR fragmenter med PCR

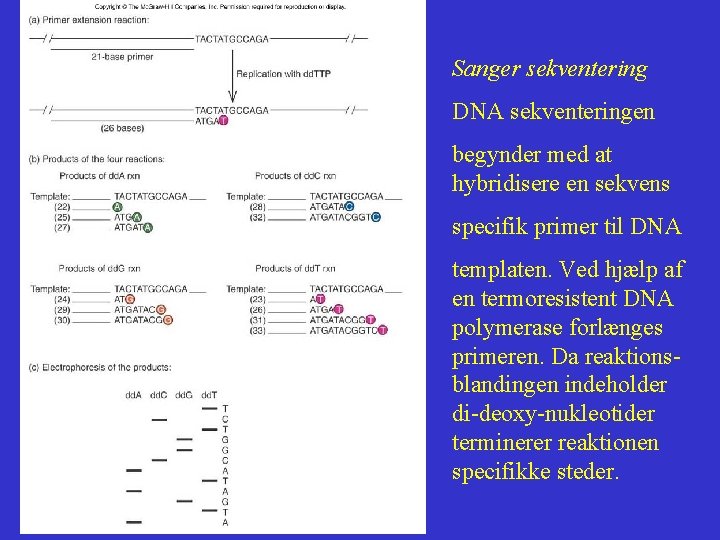
Sanger sekventering DNA sekventeringen begynder med at hybridisere en sekvens specifik primer til DNA templaten. Ved hjælp af en termoresistent DNA polymerase forlænges primeren. Da reaktionsblandingen indeholder di-deoxy-nukleotider terminerer reaktionen specifikke steder.

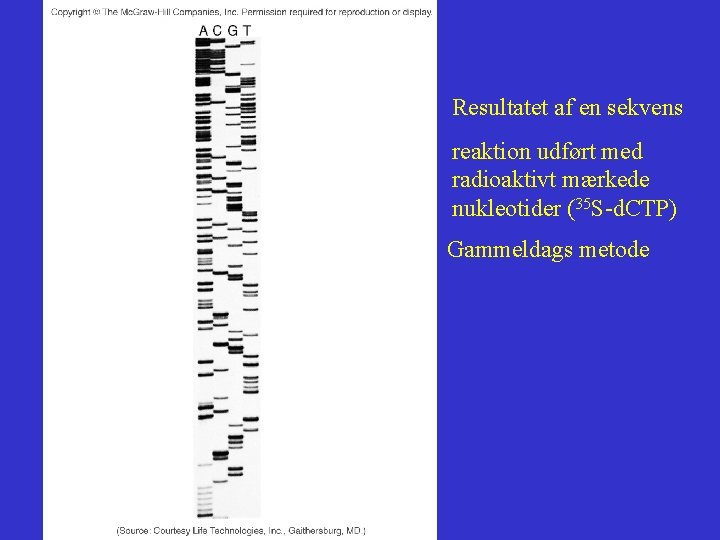
Resultatet af en sekvens reaktion udført med radioaktivt mærkede nukleotider (35 S-d. CTP) Gammeldags metode


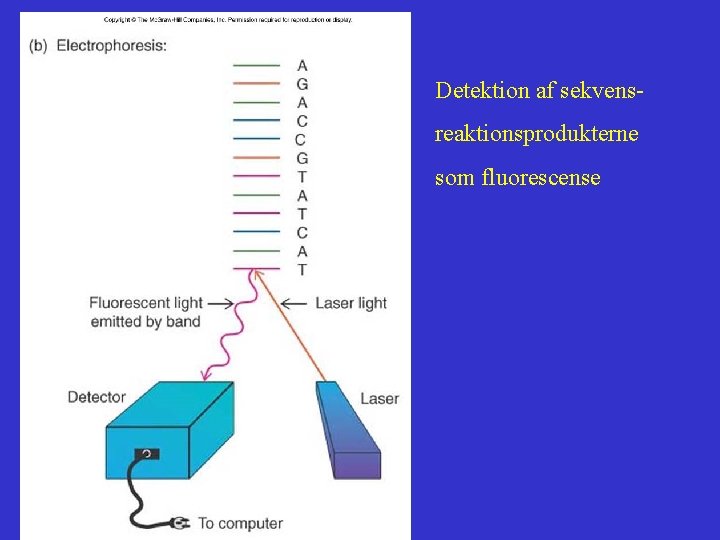
Detektion af sekvensreaktionsprodukterne som fluorescense

http: //www. mwg-biotech. com/html/all/index. php

Ekspressionskloning 1 hybrid analyse (c. DNA der koder for DNA-bindende protein og hvor bindingssitet er kendt) 2 hybrid analyse (c. DNA der koder for et protein der kan binde til et andet protein) screening af ekspressionsbibliotek (c. DNA der koder for en bestemt enzymatisk aktivitet)

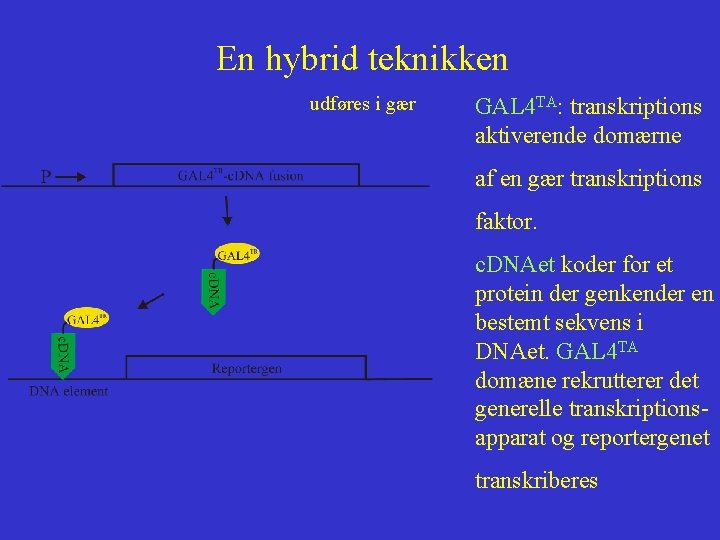
En hybrid teknikken udføres i gær GAL 4 TA: transkriptions aktiverende domærne af en gær transkriptions faktor. c. DNAet koder for et protein der genkender en bestemt sekvens i DNAet. GAL 4 TA domæne rekrutterer det generelle transkriptionsapparat og reportergenet transkriberes

2 -hybrid analyse GAL 4 DNA binder til en bestemt DNA sekvens men kan ikke aktivere transkriptionen. Kun hvis X og Y kan binde til hinanden vil GAL 4 TA kunne rekruttere transkriptionsapparatet og reportergenet transkriberes.

Ekspressionskloning i xenopus oocytter

Den afrikanske klofrø Xenopus laevis

Ovarier fra Xenopus laevis

Udtagning af æg fra oocyttens ovarier

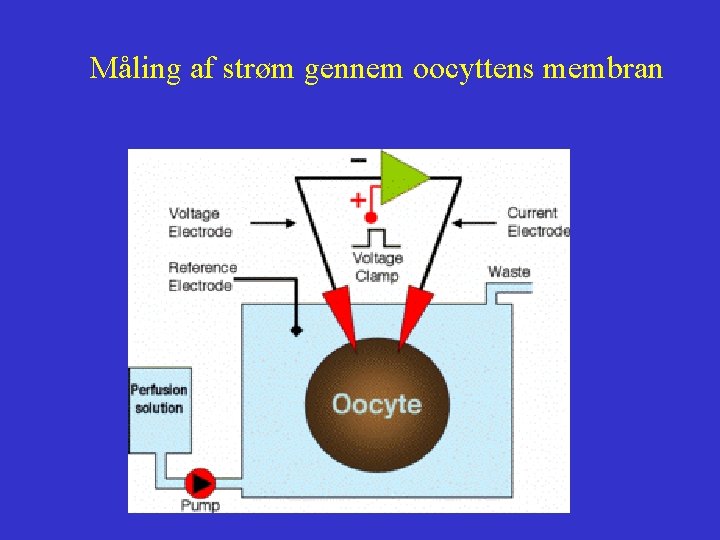
Måling af strøm gennem oocyttens membran

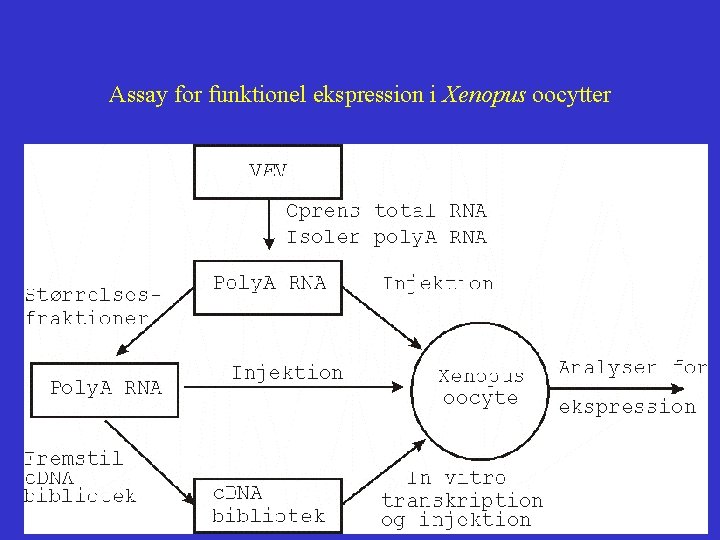
Assay for funktionel ekspression i Xenopus oocytter

Screening af c. DNA bibliotek ved funktionel ekspression i Xenopus oocytter

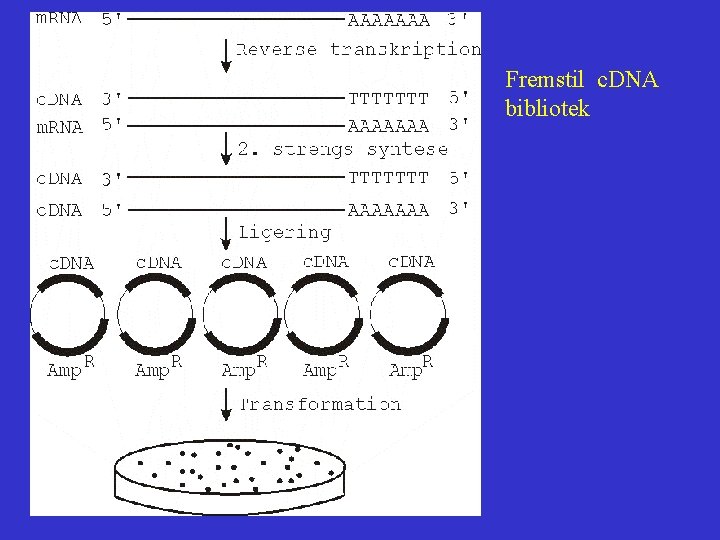
Fremstil c. DNA bibliotek

Kloning af differentielt udtrykte gener (c. DNA) • Formålet er at klone c. DNA, der representerer differentielt udtrykte gener (f. eks. En cancer celle i forhold til en normal celle) • Er hele genomet sekventeret og der findes microarray kan differentielt udtrykte gener identificeres ved microarray forsøg. • Er helet genomet ikke kendt/der ikke findes microarray kan benyttes differential display eller subtraktiv kloning

Differential display • Konceptet er at visualisere alle m. RNA molekylær i en celle under to forskellige fysiologiske omstændigheder for at identificere hvilke m. RNA molekyler, der er differentielt udtrykt • Og få fat i stykke c. DNA for hvert differentielt udtrykt gen. • c. DNA stumpen bruges efterfølgende til at klone fuldlængde c. DNA.

Generering af tilfældige c. DNA fragmenter • m. RNA kan deles op i fire grupper efter hvilket nukleotid, der sidder før det første A i poly. A halen 5’ A AAAAAAA 3’ 5’ G AAAAAAA 3’ 5’ C AAAAAAA 3’ 5’ T AAAAAAA 3’ Revers transkription med polyd. TC, polyd. TG eller polyd. TA primer vil generere c. DNA fra ca. 1/3 af cellens m. RNA (Hvis der er et eller flere A’er før poly. A halen kommer der på et tidspunkt et G, C eller T)

3’ OH c. DNA CTTTTT 5’ p GAAAA 3’ OH m. RNA Tilsæt en blanding af 40 tilfældige 13 nukleotider lange primere ( ) og polyd. TC primeren ( 3’ OH CTTTTTTTTT 5’ p 3’ OH CTTTTTTTTT 5’ p 3’ OH CTTTTTTTTT 5’ p PCR a-[32 P]-GTP )

For at visualisere alle de genererede c. DNA fragmenter separeres de i en denaturerende poly-akrylamid-gel (indeholder urinstof) De separerede bånd visualiseres ved Phospho. Image analyse Kontrol Prøve Differentielt udtrykt m. RNA

c. DNA, der repræsenterer differentielt udtrykt m. RNA, kan ekstraheres fra gelen og PCR amplificeres med polyd. TC primeren og de 40 tilfældige 13 merer. Dernæst sekventeres PCR fragmentet Sekvensen kan bruges til at screene et c. DNA bibliotek eller til at designe primere til 5’ og 3’ RACE.

Subtraktiv kloning (at finde m. RNA der er differentielt udtrykt) Vi fremstiller et c. DNA bibliotek fra det væv, vi er interesseret i (kunne være cancervæv) Vi skal så have skaffet prober til at identificere, hvilke m. RNA’er, der findes i cancervæv men ikke i rask væv Her kan vi benytte os af, at m. RNAet i cancervævet kan opddeles i to klasser. De, som er specifikke for cancervæv og de, som findes i begge typer væv.

1. strengs syntese med radioaktive nukleotider på RNA fra det væv, hvorfra vi vil finde specifikt udtrykt m. RNA fra kontrolvævet. Nogle findes i begge typer væv nogle eventuelt kun i kontrolvævet

Hybridiserer vi nu vores radioaktivt mærkede 1. strengs syntese til overskud af m. RNA fra kontrolcellerne, vil noget af det enkeltstrengede c. DNA hybridisere til m. RNA andet vil forblive enkeltstrenget Bruger vi nu denne blanding til at screene vores c. DNA bibliotek, vil det kun være det enkeltstrengede radioaktivt mærkede c. DNA der kan hybridisere til de plaques, der udgør vores c. DNA bibliotek. På denne måde får vi kun identificeret plaques, der indeholder c. DNA repræsenterende m. RNA, der er udtrykt i vores celler i forhold til kontrolcellerne Vi vil så isolere de interessante plaques og bestemme DNA sekvensen af det indsatte DNA.

Kvantifikation af m. RNA mængden -northern blot (analog til Southern blot, blot anvendes RNA). Til bestemmelse af få transkripter. -RNase protection. Til bestemmelse af få transkripter. -real time PCR (quantitative PCR, q. PCR). Til bestemmelse af få transkripter. -microarray (gene chips) (for bestemmelse af transcriptomet) -SAGE (serial analysis of gene expression) (for bestemmelse af transcriptomet)

http: //biocompare. com/pcr/tutorial/qpcr/flash_go. html

MX 4000 fra Stratagene

Fluorescens detektion i real time PCR maskinen

Southern blotting måde at analysere bestemte DNA fragmenter i en blanding af DNA

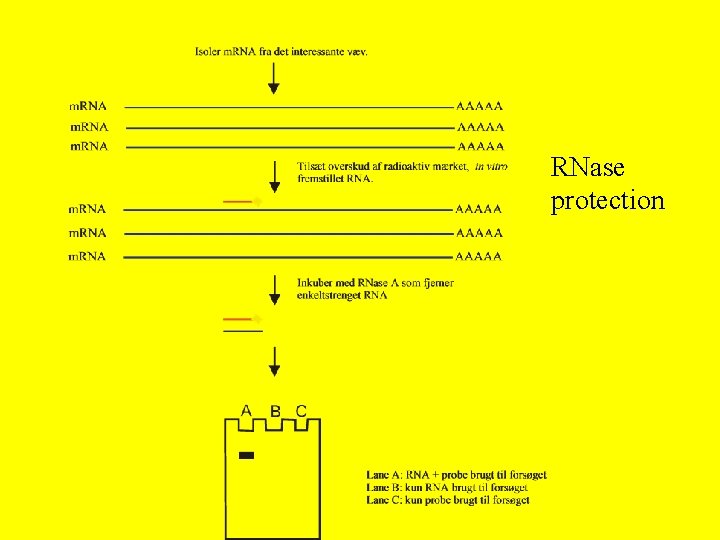
RNase protection

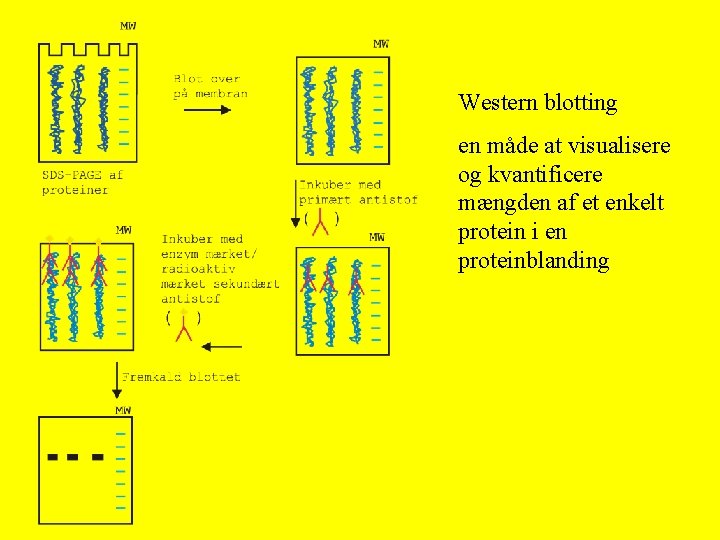
Western blotting en måde at visualisere og kvantificere mængden af et enkelt protein i en proteinblanding

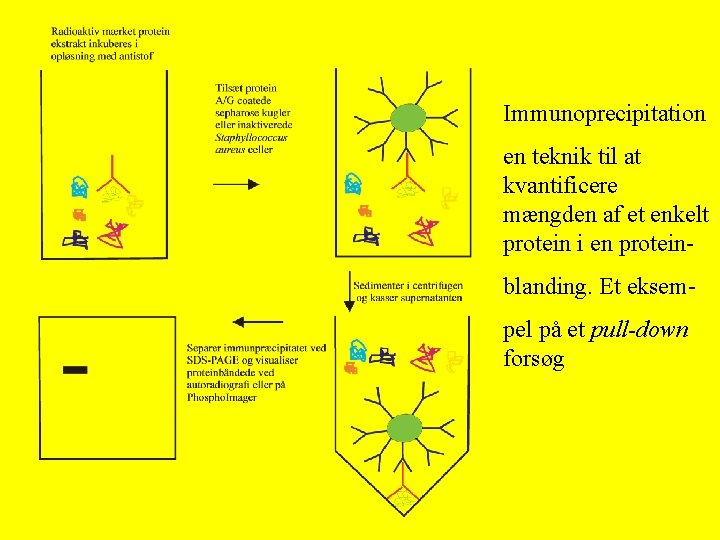
Immunoprecipitation en teknik til at kvantificere mængden af et enkelt protein i en proteinblanding. Et eksempel på et pull-down forsøg

Pulse-chase forsøg til undersøgelse af makromolekylers halveringstid, syntese hastighed og modifikationer - konceptet er at mærke en population af molekyler i ganske kort tid (pulsen )med radioaktivitet og dernæst tilsættekæmpe overskud af kolde molekyler (chasen)

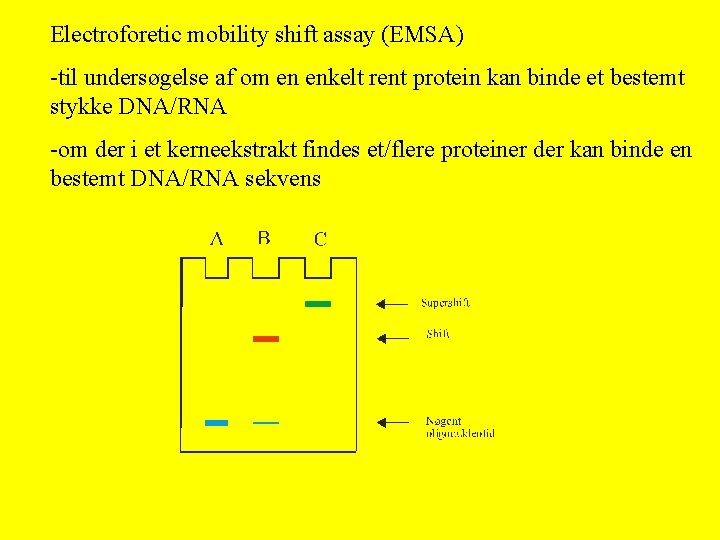
Electroforetic mobility shift assay (EMSA) -til undersøgelse af om en enkelt rent protein kan binde et bestemt stykke DNA/RNA -om der i et kerneekstrakt findes et/flere proteiner der kan binde en bestemt DNA/RNA sekvens
 Yttrandefrihetslagen
Yttrandefrihetslagen Milestone planning tool
Milestone planning tool Eksperimentelle design
Eksperimentelle design Orubbliga rättigheter
Orubbliga rättigheter Aktiv exspektans
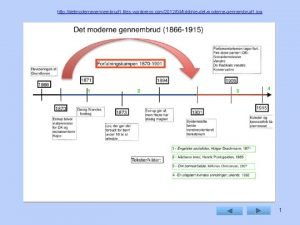
Aktiv exspektans Skagensmalerne det moderne gennembrud
Skagensmalerne det moderne gennembrud Forleggersystemet
Forleggersystemet Amalie skram det moderne gennembrud
Amalie skram det moderne gennembrud Georg brandes
Georg brandes Realismen og naturalismen
Realismen og naturalismen Amalie skram det moderne gennembrud
Amalie skram det moderne gennembrud Naturalisme og realisme
Naturalisme og realisme Det moderne gennembrud årstal
Det moderne gennembrud årstal Det moderne gennembrud kønsroller
Det moderne gennembrud kønsroller Dr forst maths
Dr forst maths At forst
At forst Bostad först grundprinciper
Bostad först grundprinciper Libor forst
Libor forst Dr frost maths
Dr frost maths Det beskrivende og det komplekse kulturbegreb
Det beskrivende og det komplekse kulturbegreb Vem leder bönen i mosken
Vem leder bönen i mosken Varför kallas perioden 1918-1939 för mellankrigstiden
Varför kallas perioden 1918-1939 för mellankrigstiden Egg för emanuel
Egg för emanuel Borstål, egenskaper
Borstål, egenskaper Verktyg för automatisering av utbetalningar
Verktyg för automatisering av utbetalningar Bris för vuxna
Bris för vuxna Bo bergman jag fryser om dina händer
Bo bergman jag fryser om dina händer Trög för kemist
Trög för kemist Stig kerman
Stig kerman Var 1721 för stormaktssverige
Var 1721 för stormaktssverige A gastrica
A gastrica Tobinskatten för och nackdelar
Tobinskatten för och nackdelar Typiska novell drag
Typiska novell drag Tack för att ni har lyssnat
Tack för att ni har lyssnat Cks
Cks Gumman cirkel sång
Gumman cirkel sång Treserva lathund
Treserva lathund Fuktmätningar i betong enlig rbk
Fuktmätningar i betong enlig rbk Påbyggnader för flakfordon
Påbyggnader för flakfordon Avrunda decimaltal
Avrunda decimaltal En lathund för arbete med kontinuitetshantering
En lathund för arbete med kontinuitetshantering Elektronik för barn
Elektronik för barn Tack för att ni har lyssnat
Tack för att ni har lyssnat Tack för att ni har lyssnat
Tack för att ni har lyssnat Rutin för avvikelsehantering
Rutin för avvikelsehantering Bamse för de yngsta
Bamse för de yngsta Presentera för publik crossboss
Presentera för publik crossboss Sju principer för tillitsbaserad styrning
Sju principer för tillitsbaserad styrning Debatt mall
Debatt mall Indikation för kejsarsnitt på moderns önskan
Indikation för kejsarsnitt på moderns önskan Blomman för dagen drog
Blomman för dagen drog Nationell inriktning för artificiell intelligens
Nationell inriktning för artificiell intelligens Vad är referatmarkeringar
Vad är referatmarkeringar Verifikationsplan
Verifikationsplan Shivaismen
Shivaismen Fimbrietratt
Fimbrietratt Etik och ledarskap etisk kod för chefer
Etik och ledarskap etisk kod för chefer Tryck formel
Tryck formel Fredsgudinna
Fredsgudinna Adressändring ideell förening
Adressändring ideell förening Förklara densitet för barn
Förklara densitet för barn Steg för steg rita
Steg för steg rita Borra hål för knoppar
Borra hål för knoppar Smärtskolan kunskap för livet
Smärtskolan kunskap för livet Läkarutlåtande för livränta
Läkarutlåtande för livränta Teckenspråk minoritetsspråk argument
Teckenspråk minoritetsspråk argument Bunden eller fri form
Bunden eller fri form För och nackdelar med firo
För och nackdelar med firo Sju för caesar
Sju för caesar Handledning reflektionsmodellen
Handledning reflektionsmodellen Ekologiskt fotavtryck
Ekologiskt fotavtryck Redogör för vad psykologi är
Redogör för vad psykologi är Bra mat för unga idrottare
Bra mat för unga idrottare Biologiska arvet
Biologiska arvet Myndigheten för delaktighet
Myndigheten för delaktighet Svenskt ramverk för digital samverkan
Svenskt ramverk för digital samverkan Strategi för svensk viltförvaltning
Strategi för svensk viltförvaltning Personlig tidbok fylla i
Personlig tidbok fylla i Datorkunskap för nybörjare
Datorkunskap för nybörjare Vad är verksamhetsanalys
Vad är verksamhetsanalys Kontinuitetshantering i praktiken
Kontinuitetshantering i praktiken Formel för standardavvikelse
Formel för standardavvikelse Ministerstyre för och nackdelar
Ministerstyre för och nackdelar Karttecken brunn
Karttecken brunn Plats för toran ark
Plats för toran ark Claes martinsson
Claes martinsson Inköpsprocessen steg för steg
Inköpsprocessen steg för steg Nyckelkompetenser för livslångt lärande
Nyckelkompetenser för livslångt lärande Tack för att ni lyssnade
Tack för att ni lyssnade Rezultatet e te nxenit
Rezultatet e te nxenit 7 mrekullite e botes
7 mrekullite e botes Odlike srpske moderne
Odlike srpske moderne Stredná odborná škola strojnícka skalica
Stredná odborná škola strojnícka skalica Statele moderne
Statele moderne Tecniche pittoriche moderne
Tecniche pittoriche moderne Moderne ungdomslitteratur
Moderne ungdomslitteratur Massasamenleving 1900
Massasamenleving 1900 Avvisi ammissione unipd
Avvisi ammissione unipd