Cromatografia Gasosa de Lipdios como Ferramenta para Estudos



































































































- Slides: 124

Cromatografia Gasosa de Lipídios como Ferramenta para Estudos de Ecologia Microbiana Marcelo Ferreira Fernandes

Resumo • Parte I. Ecologia Microbiana • Parte II. Diversidade Genética, Estrutural e da Composição Química de Lipídios em Microrganismos • Parte III. Lipídios como Ferramenta para Estudos de Ecologia Microbiana

PARTE I Ecologia Microbiana

Ecologia Microbiana • Objetivos: – Entender a biodiversidade de microrganismos na natureza e como diferentes guildas interagem em comunidades microbianas – Medir a atividade dos microrganismos na natureza e monitorar seus efeitos sobre o ecossistema (componentes bióticos e abióticos)

Interações entre Microrganismos e Impactos Ambientais e Econômicos Derivados de suas Atividades

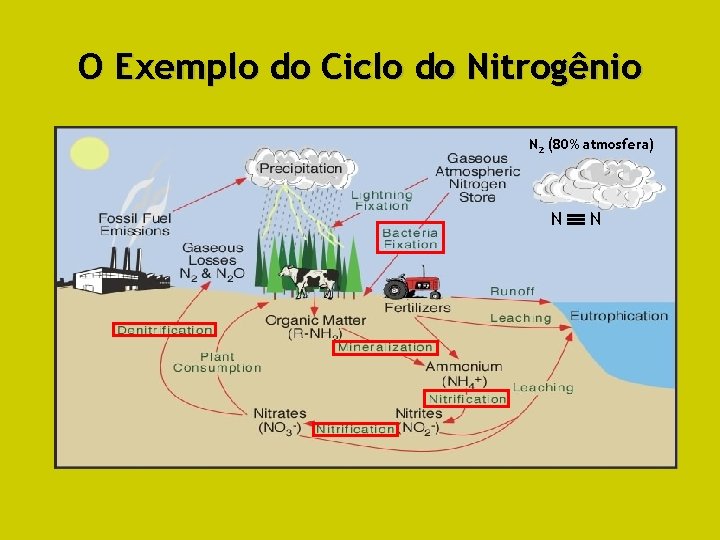
O Exemplo do Ciclo do Nitrogênio N 2 (80% atmosfera) N N

Outros Exemplos de Impactos Ambientais e Econômicos Derivados da Atividade de Microrganismos

• Controle da disponibilidade de nutrientes minerais para as plantas – Ciclagem de nutrientes, associações simbióticas (FBN, micorrizas), perdas de nutrientes • Seqüestro de carbono e balanço de gases de efeito estufa • Melhoria da qualidade física e química do solo – Formação de húmus, formação e estabilização de agregados de solo • Controle biológico de pragas e doenças vegetais e animais • Processos infecciosos (doenças) em plantas e animais • Decomposição de alimentos e outros bens • Biolixiviação de minerais de importância econômica (cobre, ouro, urânio. . . ) • Biorremediação de xenobióticos (pesticidas, petróleo, explosivos, nylon. . . ) • Tratamento de águas; eutroficação de águas e poluição de lençóis freáticos • Corrosão de metais, entupimento de tubulações de água e óleo • Produção de enzimas de importância industrial, fármacos, combustíveis, polímeros para plásticos biodegradáveis etc. . . •

Fatores que Controlam Estes Processos e Impactos • Bióticos – Presença e atividade de uma guilda específica – Interações entre guildas • Abióticas

A Atividade do “Fator Biótico” Visa à Sobrevivência • Obtenção de energia ou de compostos para síntese celular – Ciclos biogeoquímicos (C, N, P, S, Fe, Mn, Hg. . . ) – Associações simbióticas com plantas e animais – Patógenos, predadores e parasitas • Transformação de compostos tóxicos ao crescimento microbiano – Ciclos biogeoquímicos • Proteção contra fatores ambientais adversos • . . .

Diversidade Metabólica Microbiana • Vasta diversidade de “modos de vida” ou de conseguir recursos para sobrevivência – Entre espécies de microrganismos – Dentro da mesma espécie

Obtenção de Energia (Aerobiose) – Corg → CO 2 (Fungos, bactérias, protozoários. . . ) • Decomposição de restos animais e vegetais • Formação do solo (CO 2 + H 2 O H 2 CO 3), formação de humus, ciclagem de nutrientes – NH 4+ → N 2 O → NO 3 - (Nitrosomonas, Nitrobacter. . . ) • Contaminação de águas com NO 3 • Gases de efeito estufa – Fe 2+ → Fe 3+ (Thiobacillus ferroxidans) • Biolixiviação de metais (cobre, ouro, urânio. . . ) – CH 4 + 2 O 2 → CO 2 + 2 H 2 O • Importante dreno de metano – So + H 2 O + 1 ½ O 2 → SO 42 - + 2 H+ (Thiobacillus, Beggiatoa. . . ) • Solubilização de fosfatos de rocha pela acidificação do solo, corrosão de estruturas metálicas. . . – 2 H 2 + O 2 → 2 H 2 O (Alcaligenes, Ralstonia. . . ) – CO + ½ O 2 → CO 2 (carboxidotróficas: Pseudomonas carboxydovorans) • Principal dreno de monóxido de carbono da Terra

Obtenção de Energia (Anaerobiose: Aceptores de Elétrons) – CO 2 → CH 4 (Arqueas metanogênicas: Methanobacterium, Methanococcus) • Gás de efeito estufa e combustível – NO 3 - → NO → N 2 (Alcaligenes, Pseudomonas. . . ) • Perda de fertilizantes • Gases de efeito estufa – Fe 3+ → Fe 2+ (Geobacter, Shewanella, Geospirillum, Thiobacillus ferroxidans, Sulfolobus. . . ) • Degradação de compostos aromáticos (benzoato, toleno) utilizados como fonte de energia (doadores de elétrons) • Biolixiviação de metais de sulfetos metálicos. – SO 42 - + acetato ou H 2 → H 2 S (Desulfovibrio, Desulfobacter. . . ) • Toxidez à vida aquática • Desclorinação redutiva de pesticidas clorados

Obtenção de Compostos para Síntese e Metabolismo Celulares • Fotoautotrofia (algas, cianobactérias, Euglena) – Fixação de CO 2, via luz • Quimoautotrofia (muitas bactérias e arqueas) – Fixação de CO 2, via oxidação de compostos minerais • Heterotrofia (fungos, protozoários, bactérias, arqueas) – C orgânico (saprofitismo, simbiose, parasitismo ou predação) • Fixação Biológica do N 2 (algumas bactérias) – N 2 • . . . Dinitrogenase NH 4+

Interações entre Guildas Microbianas • Rizóbios (fixadores simbióticos de N 2) vs. actinomicetos (produtores de antibióticos) → controle do processo da FBN em soja • Fermentadores (diversos mcgs) vs. metanogênicos (arqueas anaeróbicas) vs. metanotróficos (bactérias anaeróbicas): balanço de metano entre solo e atmosfera → controle do processo de efeito estufa H 2 CO 2 CH 4

Fatores Abióticos de Controle da Atividade, Biomassa e Composição de Comunidades Microbianas

Atividade, Biomassa e Composição Microbianas são Afetadas por: – Disponibilidade de oxigênio • De modo geral, bactérias e arqueas mais versáteis que eucariotos – Disponibilidade de água • Fungos e actinomicetos mais resistentes a seca • Holoarqueas = extremófilos Temperatura • Extremófilos = poucas bactérias; ultra-extremófilos = muitas arqueas – Disponibilidade de nutrientes • Oligotróficos vs. copiotróficos – Disponibilidade de energia – p. H • Fungos = ácido; bactérias = neutro (Extremos em Arquea e Bacterias) – Toxidez por produtos naturais e artificiais – Interações biológicas. . .

Impactos Antropogênicos sobre a Microbiota e Processos Associados • Preparo do solo: – aeração, temperatura, umidade, p. H • Pesticidas e outros compostos químicos: – toxidez seletiva ou geral • Culturas, sistemas de culturas, mudanças de uso da terra – quantidade e qualidade de substrato, relações hospedeiro e microrganismos, temperatura e umidade • Adubações e calagem: – disponibilidade de nutrientes, p. H. . . • Irrigação e drenagem: – disponibilidade de água, temperatura e oxigênio. . . • Pecuária: – disponibilidade de nutrientes, oxigênio. . . • Deposições atmosféricas industriais: – disponibilidade de nutrientes, p. H. . .

Exemplos de Impactos de Atividades Humanas sobre a Atividade, Biomassa e Composição das Comunidades Microbianas

Mudança do Uso da Terra • Substituição de florestas por pastagens: – Pisoteio animal → compactação do solo → reduz fluxo de oxigênio para o solo • Ambiente aeróbico → anaeróbico • Favorecimento de metanogênicos em detrimento de metanotróficos • Aumento da emissão de CH 4 para a atmosfera – Remoção da cobertura vegetal → maior incidência solar → dessecamento da superfície do solo • Limitação de água na superfície, mas não no subsolo • Menor atividade de metanotróficos • Aumento da emissão de CH 4 para a atmosfera


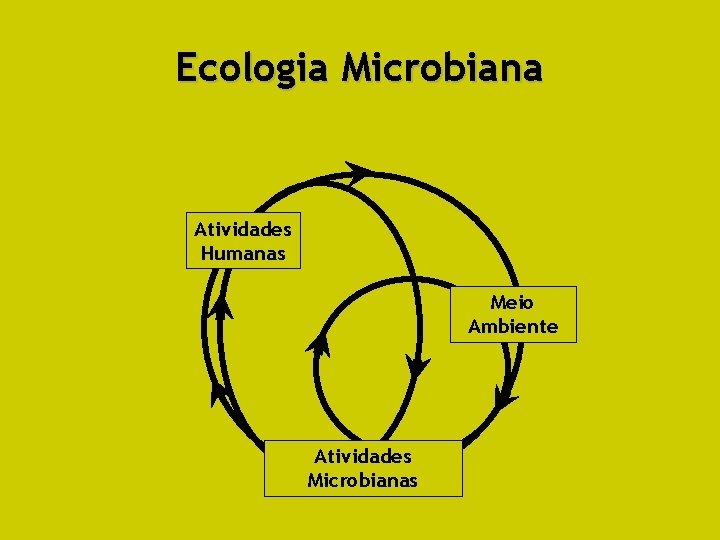
Ecologia Microbiana Atividades Humanas Meio Ambiente Atividades Microbianas

PARTE II Diversidade Genética, Estrutural e da Composição Química de Lipídios em Microrganismos

De Onde Vem a Capacidade dos Microrganismos de Realizar Tantas Funções e de se Adaptar a Tantos Ambientes Distintos?

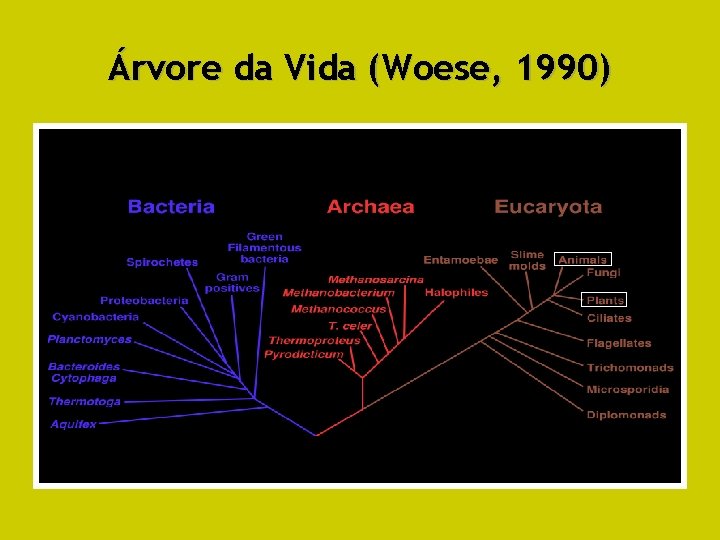
Diversidade Genética • “Alta diversidade de genes” – Diversidade metabólica potencial determinada pelos genes de cada espécie. – Evolução cria novos genes a partir de outros existentes – Acúmulo de genes diferentes do ancestral: nova sps. • Taxonomia molecular baseada em similaridade nas seqüências de genes – Seqüências r. RNA (Woese, 1990) – Três domínios muito distintos • Bacteria • Archea • Eukarya

Árvore da Vida (Woese, 1990)

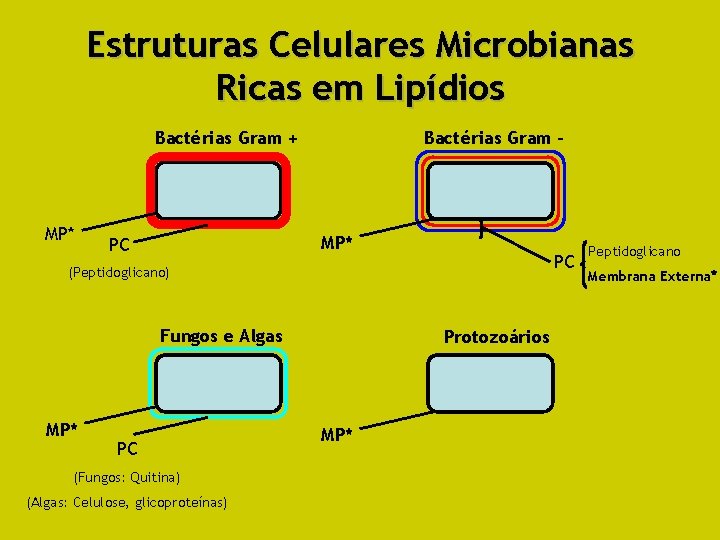
Evolução, estruturas celulares e composições de macromoléculas • Além de gerar diversidade metabólica, a evolução genética resultou em diferenças marcantes em estruturas celulares e composições de macromoléculas entre grupos taxonômicos de microganismos: – Ex: parede celular e estrutura química dos lipídios

Estruturas Celulares Microbianas Ricas em Lipídios Bactérias Gram + MP* Bactérias Gram - MP* PC PC (Peptidoglicano) Fungos e Algas MP* PC (Fungos: Quitina) (Algas: Celulose, glicoproteínas) Protozoários MP* Peptidoglicano Membrana Externa*

Estruturas Celulares Microbianas Ricas em Lipídios (cont. ) Achaea MP* PC (Pseudpeptidoglicano ou glicoproteínas ou polissacarídeos)

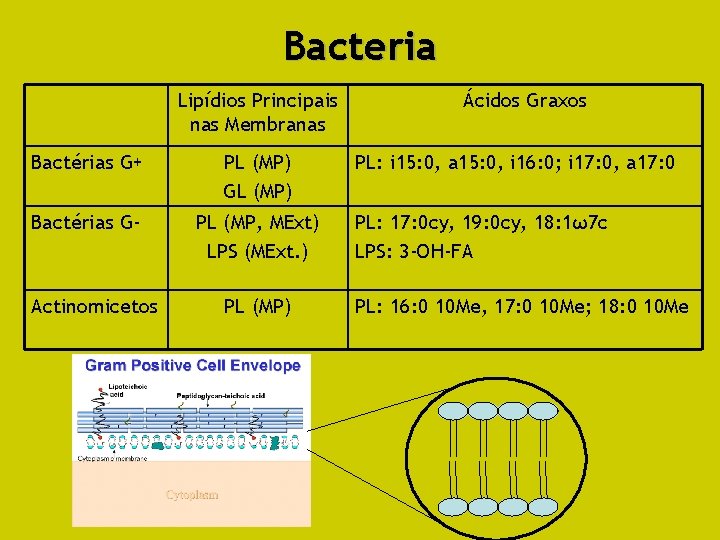
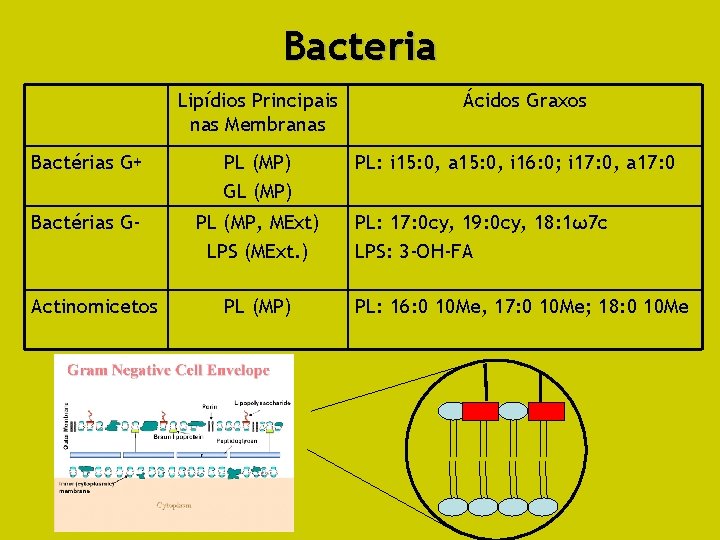
Bacteria Lipídios Principais nas Membranas Bactérias G+ PL (MP) GL (MP) Bactérias G- PL (MP, MExt) LPS (MExt. ) Actinomicetos PL (MP) Ácidos Graxos PL: i 15: 0, a 15: 0, i 16: 0; i 17: 0, a 17: 0 PL: 17: 0 cy, 19: 0 cy, 18: 1ω7 c LPS: 3 -OH-FA PL: 16: 0 10 Me, 17: 0 10 Me; 18: 0 10 Me

Bacteria Lipídios Principais nas Membranas Bactérias G+ PL (MP) GL (MP) Bactérias G- PL (MP, MExt) LPS (MExt. ) Actinomicetos PL (MP) Ácidos Graxos PL: i 15: 0, a 15: 0, i 16: 0; i 17: 0, a 17: 0 PL: 17: 0 cy, 19: 0 cy, 18: 1ω7 c LPS: 3 -OH-FA PL: 16: 0 10 Me, 17: 0 10 Me; 18: 0 10 Me

Archaea

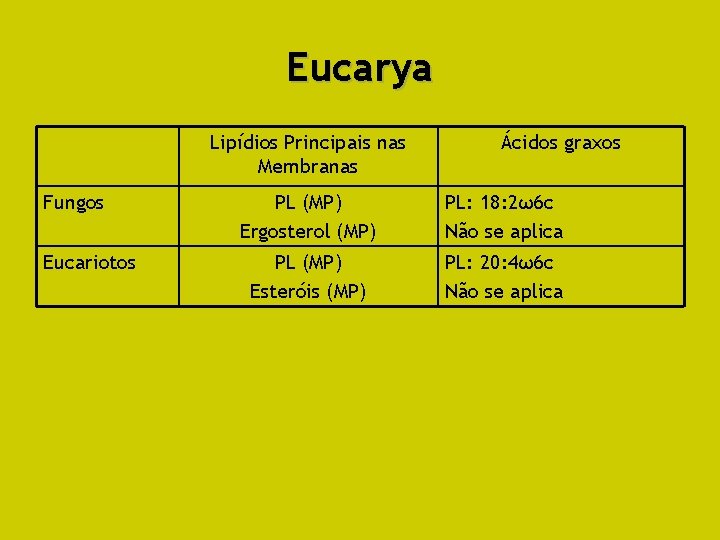
Eucarya Lipídios Principais nas Membranas Fungos Eucariotos Ácidos graxos PL (MP) Ergosterol (MP) PL: 18: 2ω6 c Não se aplica PL (MP) Esteróis (MP) PL: 20: 4ω6 c Não se aplica

PARTE III Lipídios como Ferramenta para Estudos de Ecologia Microbiana

Ecologia Microbiana Aspectos de Interesse • Biomassa microbiana • Atividade microbiana • Composição ou estrutura da comunidade

Métodos para Investigação da Estrutura da Comunidade Microbiana • Dependentes de cultivo – Meios seletivos para organismos de interesse – Plaqueamento da amostra em meio de cultura sólido – Enumeração e identificação de microrganismos

Organismos Cultiváveis Fungos Actinomicetos Protozoários, Bactéria Archaea Algas


Organismos Viáveis, Não-Cultiváveis • Torsvik, V. et al. High Diversity in DNA of Soil Bacteria. Applied and Environmental Microbiology, 56(3): 782 -787, 1990. • Apenas 1 a 10% dos microrganismos na natureza são cultiváveis em laboratório; • Embora não cultiváveis, esses organismos são ativos na natureza; • Exemplos de impacto: – Morris et al. (Nature, 2002): • SAR 11, ser vivo mais abundante do planeta, não-cultivável. – Nitrificação: • Bactérias Nitrosomonas e Nitrobacter (Winogradsky, 1888) • Wuchter et al. (PNAS, 2006): – Archaea (Crenarchaeota): 100 -1000 x mais abundante que bactérias nos oceanos • Leininger et al. (Nature, 2006): – Archaea (Crenarchaeota): ~3000 x mais abundante que bactérias no solo

Métodos de Investigação da Comunidade Microbiana Independentes de Cultivo

Métodos Independentes de Cultivo – DNA • taxons e funções mais específicos • Primers específicos para grupos taxonômicos microbianos (r. RNA) ou genes para enzimas relacionadas a uma determinada função (ex: bactérias nitrificantes = gene amo. . . ) – Lipídios • em geral, compara grandes grupos taxonômicos de microrganismos • poucos marcadores para grupos funcionais específicos • em alguns casos, atividade de guilda com uma função específica pode ser investigada em associação com técnicas isotópicas • Marcador fenotípico

Métodos Baseados em DNA (opcional) • Extração do DNA da amostra ambiental • Amplificação de regiões específicas do DNA (PCR e primers específicos) – Taxonomia (r. RNA) – Funções (genes específicos) • Padrão de bandas em gel de eletroforese – DGGE – LH-PCR – T-RFLP. . . • Clonagem e seqüenciamento de fragmentos amplificados ou recuperados das bandas do gel • Identificação microbiana utilizando-se ferramentas de comparação de similaridades entre seqüências obtidas com as disponíveis em bancos de dados de seqüências de DNA (NCBI)

Métodos Baseados em Lipídios • Princípio de avaliação da estrutura das comunidades microbianas: – Diversidade Estrutural de Ácidos Graxos – Associação entre estrutura e grupos microbianos (biomarcadores microbianos) – Inferências sobre mudanças na estrutura da comunidade a partir de mudanças no perfil de ácidos graxos

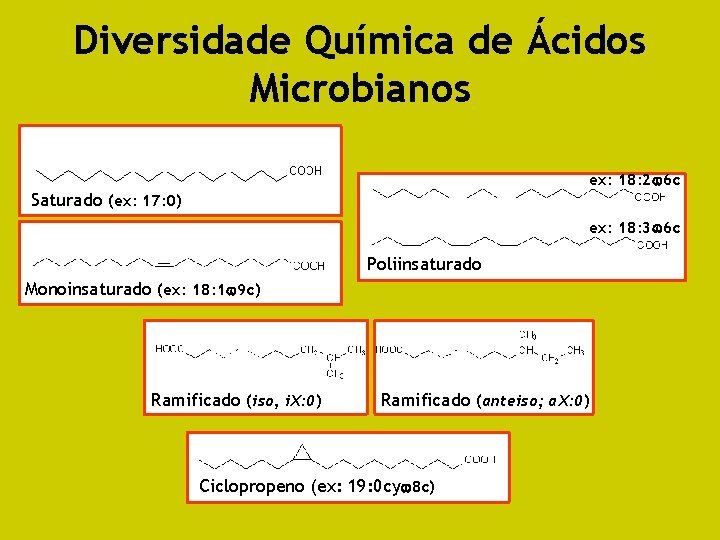
Diversidade Química de Ácidos Microbianos ex: 18: 2 6 c Saturado (ex: 17: 0) ex: 18: 3 6 c Poliinsaturado Monoinsaturado (ex: 18: 1 9 c) Ramificado (iso, i. X: 0) Ramificado (anteiso; a. X: 0) Ciclopropeno (ex: 19: 0 cy 8 c)

Nomenclatura dos Ácidos Graxos • X: Y – X = no de carbonos da molécula – Y = no de insaturações (duplas ligações) Ex: 16: 0 (ácido hexadecanóico) – Se Y>0 (insaturados): • X: Y Zc, onde Z é a posição da primeira insaturação à partir do C distal, e “c” indica a conformação “cis”, e t, a “trans” Ex: 16: 1 5 c; 18: 1 7 t; 18: 2 6 c • i. X: Y, a. X: Y, X: Y 10 -Me – Radical metil lateral na posição 2, 3 ou 10, a partir do C distal Ex: i 15: 0; a 15: 0; i 17: 0; 18: 0 10 -Me • X: Ycy Zc – cy representa presença de anel ciclopropeno Ex: 19: 0 cy 8 c

Ácidos Graxos Biomarcadores • Grupos Taxonômicos • Poucos de Grupos Funcionais Específicos

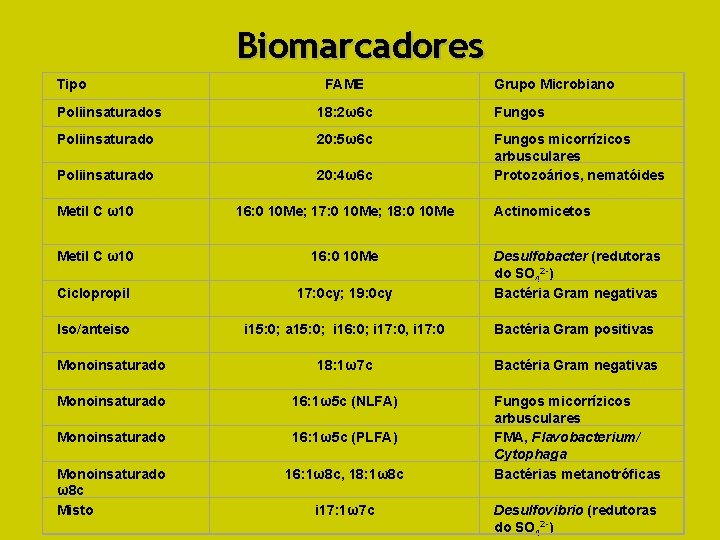
Biomarcadores Tipo FAME Grupo Microbiano Poliinsaturados 18: 2ω6 c Fungos Poliinsaturado 20: 5ω6 c Poliinsaturado 20: 4ω6 c Fungos micorrízicos arbusculares Protozoários, nematóides Metil C ω10 16: 0 10 Me; 17: 0 10 Me; 18: 0 10 Me Metil C ω10 Desulfobacter (redutoras do SO 42 -) Bactéria Gram negativas Monoinsaturado 16: 0 10 Me 17: 0 cy; 19: 0 cy i 15: 0; a 15: 0; i 16: 0; i 17: 0, i 17: 0 18: 1ω7 c Monoinsaturado 16: 1ω5 c (NLFA) Monoinsaturado 16: 1ω5 c (PLFA) Monoinsaturado ω8 c Misto 16: 1ω8 c, 18: 1ω8 c Fungos micorrízicos arbusculares FMA, Flavobacterium/ Cytophaga Bactérias metanotróficas i 17: 1ω7 c Desulfovibrio (redutoras do SO 42 -) Ciclopropil Iso/anteiso Actinomicetos Bactéria Gram positivas Bactéria Gram negativas

Ácidos Graxos em Diferentes Classes de Lipídios Complexos • Ácidos graxos raramente ocorrem livres em células • Normalmente, eles integram lipídios estruturalmente mais complexos • Diferentes tipos de ligações químicas unem os ácidos graxos ao restante da molécula dos lipídios complexos

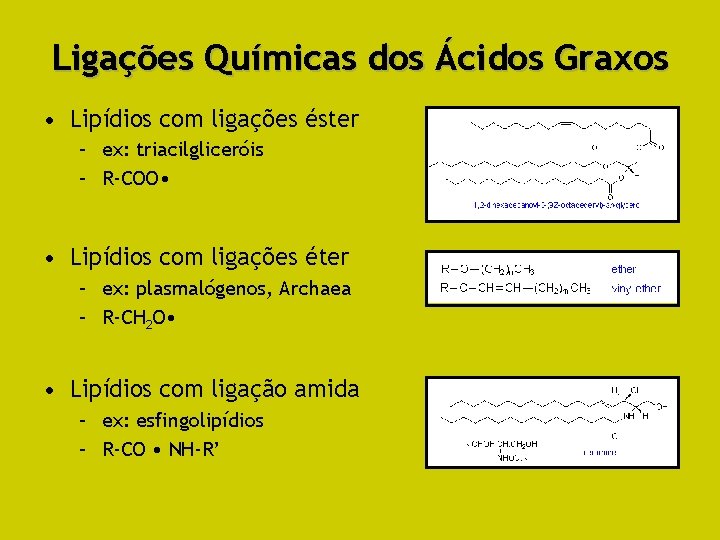
Ligações Químicas dos Ácidos Graxos • Lipídios com ligações éster – ex: triacilgliceróis – R-COO • • Lipídios com ligações éter – ex: plasmalógenos, Archaea – R-CH 2 O • • Lipídios com ligação amida – ex: esfingolipídios – R-CO • NH-R’

Éster Metílico de Ácido Graxo “Fatty Acid Methyl Ester”(FAME) Ácido Graxo COOHR Metanólise Alcalina Branda (PLFA e EL-FAME) ou Saponificação – Metanólise Ácida (MIDI) FAME COOCH 3 Altamente volátil

Saponificação/Metilação • Duas etapas: – Saponificação: Ácido graxo sabão – Metilação: substituição do Na por metil FAME • Condições de reação: – Saponificação: altas temperaturas, 3, 8 M Na. OH em Me. OH: H 2 O (100 o. C, 30’) – Metilação: presença de água, 6 M HCl: Me. OH (85 o. C, 10’) • Ligações-alvo: – Éster, éter, amina, ácidos graxos livres • Altamente danosas à estrutura de alguns ácidos graxos: – ciclopropanos

Metanólise Alcalina Branda • Uma só etapa (transesterificação) – Separação de ácidos graxos das moléculas complexas e esterificação com ·CH 3 • Condições de reação: – 0, 2 M KOH em Me. OH (37 o. C, 1 h) – Ausência de água • Ligações-alvo – Ésteres

Principais Lipídios Complexos Contendo Ácidos Graxos

Fosfolipídios • Universal em membranas celulares, proporções aproximadamente constantes por massa celular – Exceto em Archaea, ligações são principalmente éster • Rápida hidrólise do fosfato após morte microbiana (Fosfolipídio -> Lipídio Neutro) • Microbiota viável

Lipídios Neutros • Presentes em: • Células mortas • Reserva em eucariotos • Reservas de LN em procariotos são raras

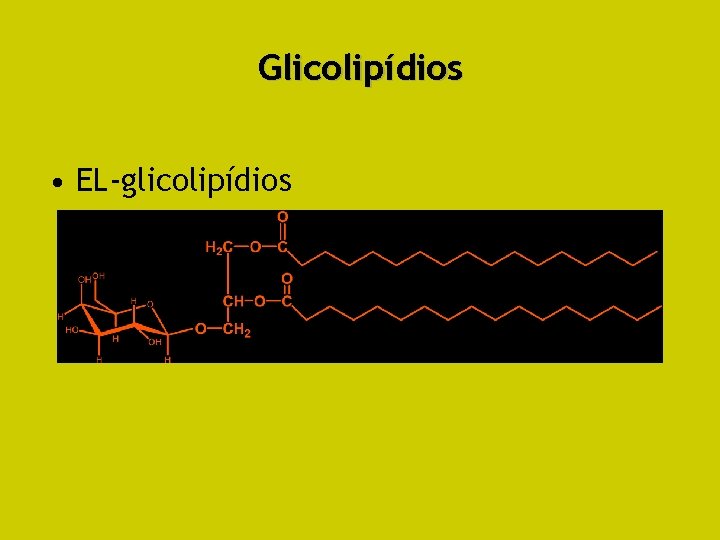
Glicolipídios • EL-glicolipídios

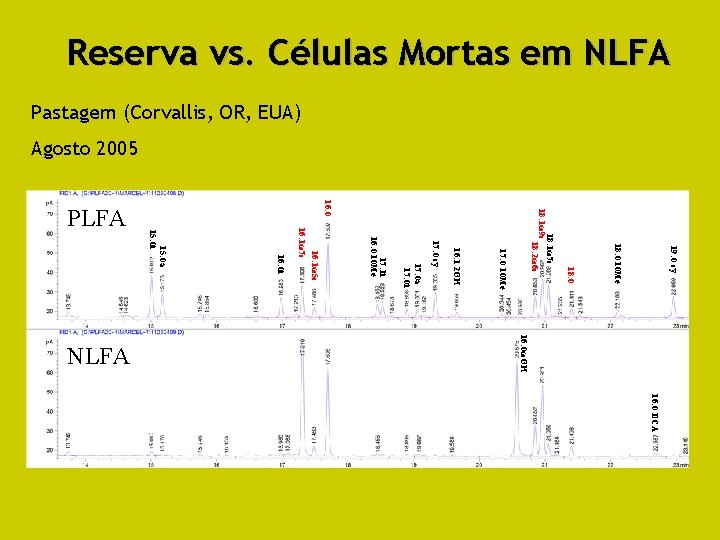
Reserva vs. Células Mortas em NLFA Pastagem (Corvallis, OR, EUA) Agosto 2005 19: 0 cy 18: 0 10 Me 18: 0 17: 1 i 16: 0 10 Me 17: 0 a 17: 0 i 17: 0 cy 16: 1 2 OH 17: 0 10 Me 18: 2ω6 c 18: 1ω7 c 18: 1ω9 c 16: 0 16: 1ω5 c 16: 1ω7 c 16: 0 i 16: 0ωOH NLFA 15: 0 a 15: 0 i PLFA 16: 0 DCA

Protocolos para Extração de FAMEs do Solo

Principais Protocolos de Extração de FAMEs • PLFA (Phospholipid Fatty Acids) – White e Ringelberg (1998) • MIDI (Microbial Identification™) - Sasser (1990) • EL-FAME (Ester-linked FAME) – Direto do solo (Schutter e Dick, 2000) – Extrato lipídico (Drjiber et al. , 2000)

Método: PLFA • Método mais utilizado (padrão) • Mais caro e trabalhoso – Coluna de SPE-Si: ~R$ 10, 00 (no Brasil) – 40 -50 ml de clorofórmio, acetona e metanol amostra-1 – 24 amostras em 2 dias • Extrai apenas ácidos graxos esterificados em fosfolipídios

Protocolo PLFA

Fase 1. Extração de Lipídios Totais do Solo • Bligh e Dyer (1959) modificado – Mistura monofásica metanol: clorofórmio: tampão fosfato 50 m. M (2: 1: 0, 8) – Solo: clorofórmio (1: 1, m/v) – Volumes de água e tampão fosfato 100 m. M – Ex: 4 g solo úmido com 25% umidade (b. u. ) • • 3 g solo seco + 1 g água 6 ml de metanol, 3 ml de clorofórmio, 2, 4 ml TF 50 m. M (combinação de água e TF 100 m. M) 1 ml água do solo + 0, 2 ml água adicionada + 1, 2 ml TF 100 m. M

1. 1. Solo e mistura de solventes em tubo de centrífuga com tampa com revestimento interno de teflon; 1. 2. Agitar por 2 h, decantar “overnight” – Variações: • • sonicação (2 min) tempo de agitação, omissão da decantação; 1. 3. Ressuspender solo e centrifugar (10 min e 2000 x g); 1. 4. Filtrar sobrenadante papel Whatman #1; 1. 5. Adicionar metanol: clorofórmio ao solo remanescente no tubo e repetir passos 1. 3 e 1. 4 mais duas vezes;

1. 6. Partição do filtrado em duas fases: adicionar solução Na. Cl 2 M (solução: clorofórmio, 1: 1); 1. 7. Esperar até que a desaparecimento da turbidez na fase superior (aquosa); 1. 8. Transferir a fase inferior (orgânica) para um tubo de ensaio e secá-la sob atmosfera de N 2 ultra-puro, a 37 o. C. Armazenar a -20 o. C.


Fase 2. Fracionamento das Classes de Lipídios (Cromatografia de Afinidade) • Extração em fase sólida (ácido silícico) • Partição de lipídios entre radicais silanóis ativos nos grânulos de ácido silícico e solventes de polaridade crescente (clorofórmio, acetona e metanol)



2. 1. Colocar coluna de ácido silícico (3 ml, 500 mg) em aparato de vácuo com tubo de ensaio diretamente abaixo da mesma; 2. 2. Adicionar 2 ml de clorofórmio à coluna, sem deixar drenar; 2. 3. Ressuspender extrato de lipídios em volume mínimo (100 -200 μL) de clorofórmio e transferir para coluna de ácido silícico. Repetir 3 vezes para assegurar transferência quantitativa; 2. 4. Acionar vácuo e adicionar 3 x 2 ml de clorofórmio, e coletar esses volumes no tubo de ensaio colocado abaixo; 2. 5. Trocar tubo de ensaio por um novo, adicionar 3 x 2 ml de acetona, e coletar volumes no tubo de ensaio abaixo; 2. 6. Trocar tubo de ensaio por um novo, adicionar 3 x 2 ml de metanol, e coletar volumes no tubo de ensaio abaixo; 2. 7. Secar frações sob atmosfera de N 2 ultrapuro , a 37 o. C, armazenar a -20 o. C.

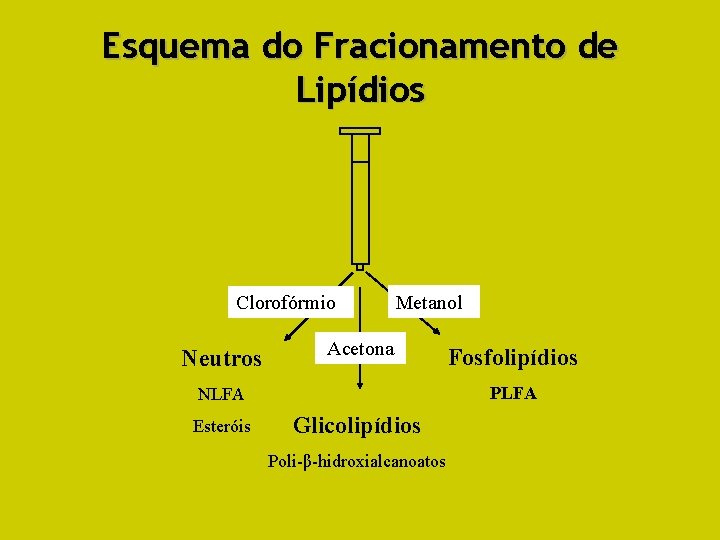
Esquema do Fracionamento de Lipídios Clorofórmio Neutros Metanol Acetona PLFA NLFA Esteróis Fosfolipídios Glicolipídios Poli-β-hidroxialcanoatos


Fase 3. Metanólise Alcalina para Produção dos FAMES de PL • Geração dos FAMEs a partir de fosfolipídios esterificados; • Remoção dos ácidos graxos do restante da molécula lipídica e substituição das ligações com glicerol por grupos metil > aumenta a volatilidade dos compostos • Para PLFA, proceder reação apenas na fração contendo fosfolipídios; • Nota: Dependendo do objetivo do estudo, a fração neutra e glicolipídica também poderão ser tratadas posteriormente para análise cromatográfica de compostos de interesse.

3. 1. Redissolver os lipídios fracionados secos em 1 ml de tolueno: metanol (1: 1, v/v) e 1 ml de KOH metanólico (0, 2 N); 3. 2. Agitar tubo em vortex, rapidamente, e colocá-los em banho-maria a 37 o. C por 15 min. ; 3. 3. Após amostras esfriarem até a temperatura ambiente, adicionar 2 ml de hexano: clorofórmio (4: 1, v/v), e misturar amostra; 3. 4. Neutralize p. H (6 a 7) da mistura com adição de 0, 2 ml de ácido acético 1 N. Verificar p. H final em papel de tornassol; 3. 5. Adicione 2 ml de água destilada para separar fases e misture amostra em vortex por 30 s. Esperar separação de fases se completar (~10 -15 min. ); 3. 6. Transferir fase orgânica (superior) para um tubo de ensaio novo, 3. 7. Reextrair fase inferior: adicionar 2 ml de clorofórmio: hexano (4: 1 v/v), agitar em vortex 30 s. , esperar separação de fases, transferir para tubo novo (repetir 2 x) 3. 8. Evaporar solvente sob N 2 ultrapuro, a 37 o. C; armazenar a -20 o. C, sob N 2.

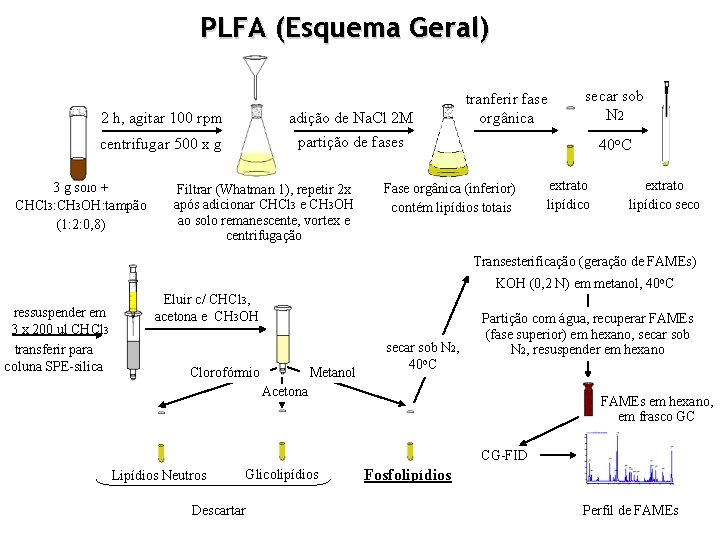
PLFA (Esquema Geral) 2 h, agitar 100 rpm adição de Na. Cl 2 M centrifugar 500 x g partição de fases 3 g solo + CHCl 3: CH 3 OH: tampão (1: 2: 0, 8) Filtrar (Whatman 1), repetir 2 x após adicionar CHCl 3 e CH 3 OH ao solo remanescente, vortex e centrifugação tranferir fase orgânica secar sob N 2 40 o. C Fase orgânica (inferior) contém lipídios totais extrato lipídico seco Transesterificação (geração de FAMEs) KOH (0, 2 N) em metanol, 40 o. C ressuspender em 3 x 200 ul CHCl 3 transferir para coluna SPE-silica Eluir c/ CHCl 3, acetona e CH 3 OH Clorofórmio Metanol secar sob N 2, 40 o. C Partição com água, recuperar FAMEs (fase superior) em hexano, secar sob N 2, resuspender em hexano Acetona FAMEs em hexano, em frasco GC CG-FID Lipídios Neutros Glicolipídios Descartar Fosfolipídios Perfil de FAMEs

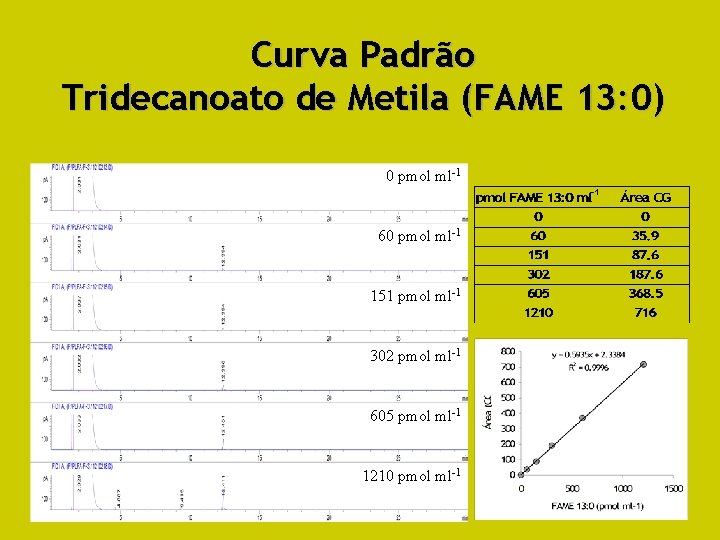
Fase 4. Preparo de Amostras, Separação em CG, Quantificação e Identificação de Compostos 4. 1. PLFA secos são ressuspensos em hexano, agitados em vortex, e transferidos para frasco GC com “insert” de 200 μL; 4. 2. Injeção em CG-FID, equipado com coluna não-polar (ex: 5% diphenyl, 95% methylpolysiloxane fase estacionária; ex: Ultra-2, DB-5, HP-5), com 25 m, 0, 2 mm diam. int. ; 0, 33 m espessura filme); rampa de temperatura: 170 -280 o. C a 4 o. C min-1; mais 5 min. a 280 o. C para limpeza da coluna. 4. 3. Curva-padrão: FAME 13: 0 (“methyl tridecanoic acid”): relação entre área de pico cromatográfico e quantidades conhecidas do FAME. Alternativamente: adição de padrão interno: usualmente FAME 19: 0. Resultados em mol de FAMEs individuais por g de solo (pmol FAME g-1 solo seco). 4. 4. Misturas padrões de FAMEs microbianos (BAME e FAME-37): comparar TR com amostras 4. 5. ECL vs. TR (Biblioteca de ECLs. Ex: MIDI) - Relação linear entre TR e número de C de série homóloga de ácidos graxos saturados de cadeia linear 4. 5. CG-EM (confirmação de compostos): importante, mas, em geral, não realizada.


Curva Padrão Tridecanoato de Metila (FAME 13: 0) 0 pmol ml-1 60 pmol ml-1 151 pmol ml-1 302 pmol ml-1 605 pmol ml-1 1210 pmol ml-1

Brancos - Verificar contaminações das amostras durante procedimento de extração - Todo o procedimento é realizado de modo idêntico às amostras, porém sem solo PLFA Br NLFA Br

Efeito do Desmatamento sobre a Estrutura da Comunidade Microbiana

Floresta Adulta (>150 a) vs. Floresta Recem-Cortada (10 a) 18: 2 6 c fungo >150 anos 16: 0 comum ~ 10 anos

Método: MIDI • Adaptado de método comercial para identificação de bactérias em cultura pura, de acordo com perfil de lipídios celulares totais • Mais rápido (? ) e barato que PLFA • Requer menor quantidades de solo (? ? ) • Mais perigoso (base e ácido concentrados, alta temperatura) • Extração direta, direta sob condições extremas de p. H e temperatura (saponificação/metilação ácida) • Extrai FAMEs de potencialmente todos os lipídios celulares (éster, éter, amida. . . ) e não-celulares (reserva, células mortas, fontes nãomicrobianas, matéria orgânica do solo. . . ), incluindo ácidos graxos livres • Forte potencial de alterar a estrutura química de muitos ácidos graxos, especialmente os com aneis ciclopropeno e os insaturados

Protocolo MIDI

Saponificação • Pesar 3 g de solo em tubo de de vidro (20 ml) com tampa de rosca revestida com Teflon; • Adicionar 3 ml de solução de Na. OH (3, 8 N) em água: metanol (1: 1 v/v); • Agitar em vortex e ferver por 30 min. • Produto: sais de sódio de cadeia longa (sabão)

Metilação Ácida • Adicionar 6 ml de mistura HCl (6 M): metanol (1: 0, 85 v/v); • Aquecer em banho-maria 85 o. C, 10 min. • Produto: FAMEs

Recuperação dos FAMEs • Adicionar 3 ml de hexano (partição de fases); • Transferir todo conteúdo do frasco para tubos de centrífuga de 35 ml, com rosca revestida por Teflon; • Centrifugar a 480 x g por 10 min. para separar MOS da fase orgânica; • Transferir fase orgânica (superior) para tubos de 13 x 100 mm com tampa revestida de Teflon;

Lavagem da Fase Orgânica • Adicionar 3 ml de base fraca (0, 3 M Na. OH) para lavar residual de reagentes ácidos • Agitar tubo lentamente por 5 min • Transferência da fase superior para tubos CG (2 ml)

Análise cromatográfica, quantificação e identificação dos compostos • Idem PLFA

Método: EL-FAME • Mais rápido (!), fácil (!) e barato que PLFA • Extrai ácidos graxos esterificados em diversas classes de lipídios (neutros, glicolipídios e fosfolipídios) • Contribuição de lípídios de reserva e de células mortas (LN), além de glicolipídios esterificados, para o perfil de FAMEs • Duas variações de extração: – direta do solo (Schutter e Dick, 2000); – a partir do extrato de lipídios totais (Drijber et al. , 2000).

Protocolo EL-FAME

Metanólise Alcalina Branda • Pesar 3 g de solo em tubo centrífuga de 35 ml com tampa revestida de Teflon; • Adicionar 15 ml de KOH (0, 2 N) em metanol; • Agitar em vortex e incubar a 37 o. C por 1 h (transesterificação, idêntica à do PLFA) • Agitar tubos em vortex a cada 10 min. durante o tempo de incubação. • Adicionar 3 ml de ácido acético 1, 0 M para neutralizar p. H do conteúdo do frasco

Recuperação dos FAMEs • Partir fases adicionando 10 ml de hexano; • Centrifugar a 480 x g, 10 min. ; • Transferir fase orgânica para tubo de ensaio e secar sob N 2 ultrapuro; • Ressuspender extrato seco em 3 x ~70 µL de hexano, agitando em vortex após cada adição do solvente, e transferir para frasco CG, com insert de 200 µL.

Análise cromatográfica, quantificação e identificação dos compostos • Idem PLFA

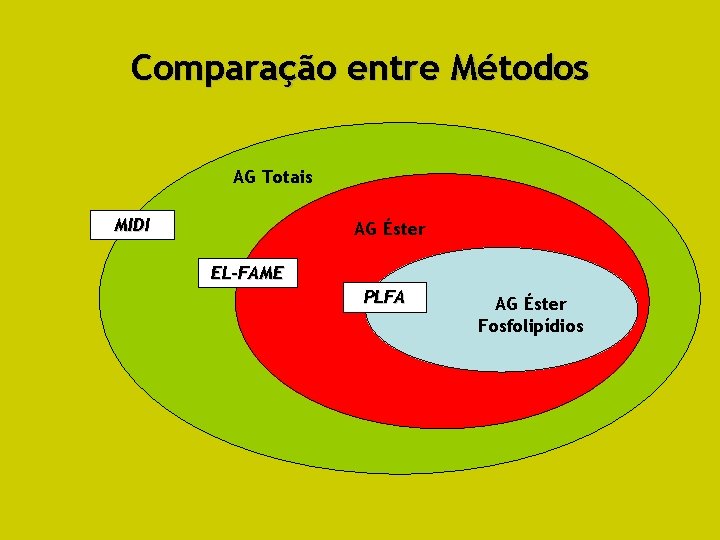
Comparação entre Métodos AG Totais MIDI AG Éster EL-FAME PLFA AG Éster Fosfolipídios

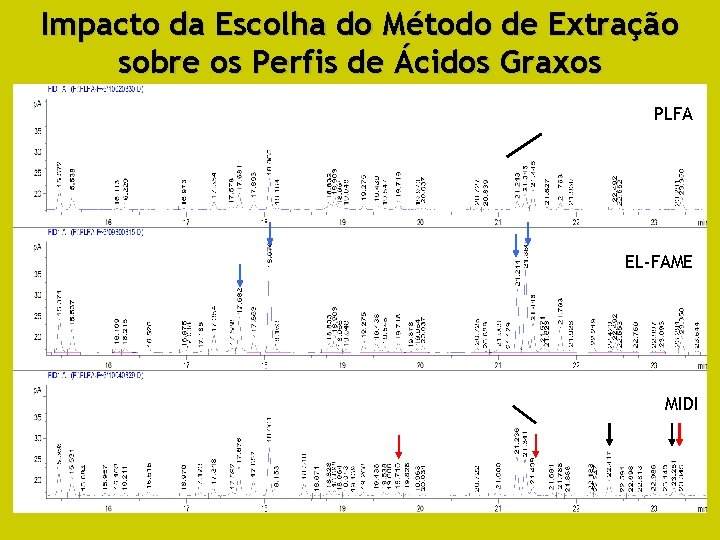
Impacto da Escolha do Método de Extração sobre os Perfis de Ácidos Graxos PLFA EL-FAME MIDI

Efeito da Escolha do Protocolo de Extração sobre a Interpretação de Resultados de Estrutura da Comunidade Microbiana

Amostras de Solo • 29 amostras de solo através do Estado do Oregon, EUA (0 -10 cm depth) • Ecossistemas diversos – Sem vegetação na data de amostragem – Herbáceas (culturas anuais, pastagens e pomares) – Florestas (gimnospermas com >150 a; clareiras de 10 a; mistas) • Ampla faixa de propriedades do solo – COT, MOP, MO-n. P, p. H, textura • Amostras de solo peneiradas (< 2 mm) • Extração de FAMEs pelos 3 métodos

Análise de FAMEs • Cromatografia gasosa • Identificação inicial por comparação dos TR entre amostras e padrões de FAMEs microbianos • Confirmação de compostos abundantes por EM • Dados de composição de AG analisados em mol%

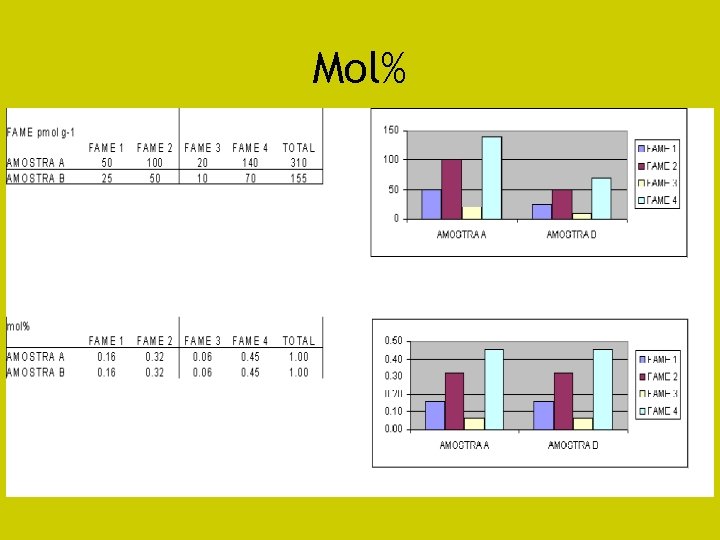
Mol%

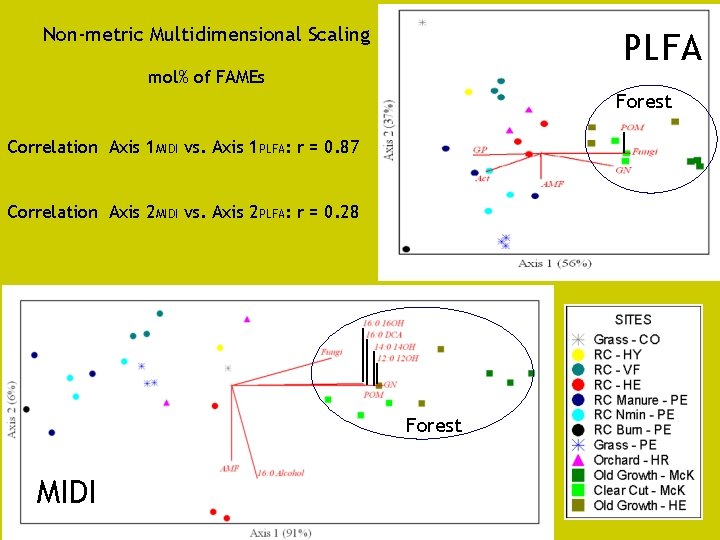
MIDI versus PLFA • Análise estatística: ordenação multivariada NMS – Distância de Sorensen – PC-ORD 4, modo autopiloto “lento e abrangente” – Correlações com variáveis de solo e caracterização da mudanças no perfil de FAMEs utilizando coeficiente de Pearson (“joint plots”)

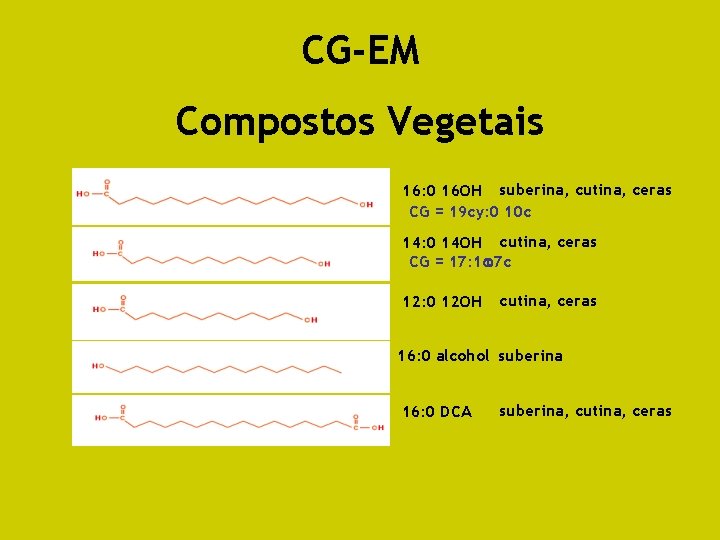
MIDI versus PLFA - Resultados • CG-MS: alguns biomarcadores de substâncias vegetais (cutina, suberina, e ceras) presentes em grandes quantidades no extrato de MIDI, mas não em PLFA

CG-EM Compostos Vegetais 16: 0 16 OH suberina, cutina, ceras CG = 19 cy: 0 10 c 14: 0 14 OH cutina, ceras CG = 17: 1 7 c 12: 0 12 OH cutina, ceras 16: 0 alcohol suberina 16: 0 DCA suberina, cutina, ceras

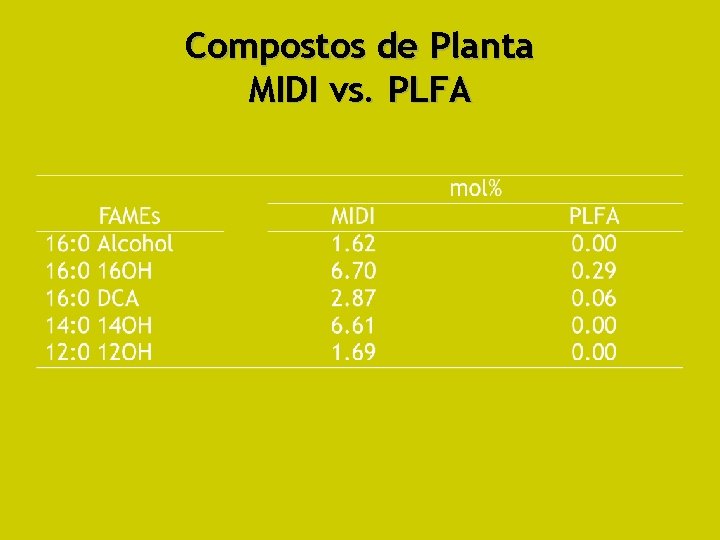
Compostos de Planta MIDI vs. PLFA

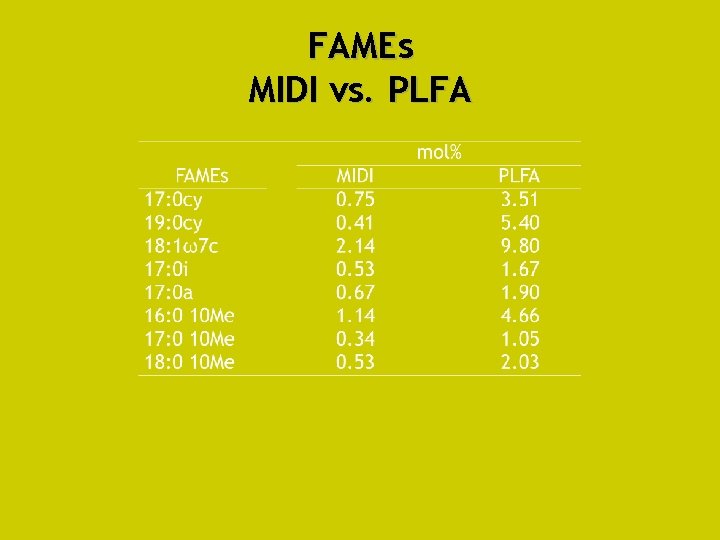
FAMEs MIDI vs. PLFA

Non-metric Multidimensional Scaling PLFA mol% of FAMEs Forest Correlation Axis 1 MIDI vs. Axis 1 PLFA: r = 0. 87 Correlation Axis 2 MIDI vs. Axis 2 PLFA: r = 0. 28 MIDI Forest MIDI

EL-FAME versus PLFA • Mesmo método de produção de FAMEs (metanólise alcalina branda) • Diferentes reservatórios de lipídios alvos de produção de FAMEs PLFA vs. PLFA + NLFA + GLFA

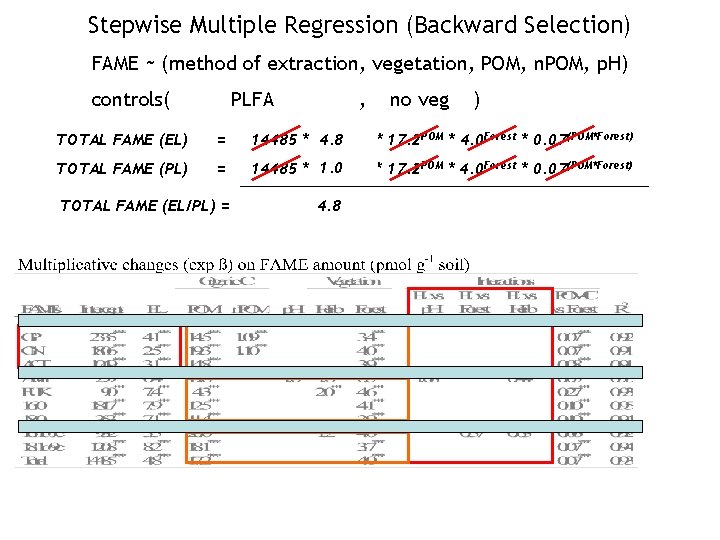
EL-FAME versus PLFA Objetivo: Avaliar a interferência de NL e GL no que se refere à comparação da interpretação de dados de comunidade microbiana entre o EL-FAME e o PLFA (padrão) Amostras de solo: Idem estudo de comparação entre PLFA vs. MIDI Análises estatísticas: Modelos de regrassão múltipla (stepwise) Ordenação das amostras com base na relação entre EL/PL com respeito às concentrações molares dos FAMEs (mol%) nos respectivos extratos

Stepwise Multiple Regression (Backward Selection) FAME ~ (method of extraction, vegetation, POM, n. POM, p. H) controls( PLFA , no veg ) TOTAL FAME (EL) = 14485 * 4. 8(EL=1) * 17. 2 POM * 4. 0 Forest * 0. 07(POM*Forest) TOTAL FAME (PL) = 1. 0(EL=0) * 17. 2 POM * 4. 0 Forest * 0. 07(POM*Forest) 14485 * 4. 8 TOTAL FAME (EL/PL) = 4. 8

Stepwise Multiple Regression (Backward Selection) FAME ~ (lipid source, vegetation, POM, n. POM, p. H) Vegetation: non-vegetation at sampling (control), herbaceous, and forest Lipid source: PLFA (control), NLFA or GLFA Multiplicative changes (exp ß) on FAME amount (pmol g-1 soil) associated with lipid sources (neutral lipids vs. phospholipids; and glycolipids vs. phospholipids

NMS with EL-FAME: PLFA mol% ratios EL-FAME (mol%) 15: 0 i 18: 2 6 c … 20: 4 6 c CO 0. 05 0. 26 0. 20 PMa 0. 09 0. 21 0. 23 Forest No vegetation … OGc 0. 01 0. 34 0. 12 PLFA (mol%) 15: 0 i 18: 2 6 c CO 0. 12 0. 15 0. 11 PMa 0. 16 0. 12 0. 16 OGc 0. 06 0. 22 ELFA/PLFA 15: 0 i 18: 2 6 c CO 0. 41 1. 73 1. 82 PMa 0. 56 1. 75 1. 44 0. 17 1. 53 0. 55 … 20: 4 6 c … OGc Herbaceous

Outros Lipídios Importantes • Ergosterol (esterol): – exclusivo de fungos (biomassa de fungos) – altamente correlacionado com PLFA 18: 2 6 c • Poli-hidroxialcanoatos (PHAs) – reservas em bactérias – em SPE-Si é eluído com acetona

Outros Usos dos Métodos Baseados em Lipídios na Investigação da Microbiota • Diversidade e estrutura da comunidade microbiana • Biomassa microbiana – Total – Grupos microbianos específicos • Estado nutricional de fungos do solo • Respostas de bactérias a estresses ambientais

Biomassa Microbiana Viável Método 1. Soma dos PLFAs BM total: soma de todos os PLFAs 3, 0 x 104 células/pmol PLFA (bactéria) 1, 2 x 104 células/pmol PLFA (algas) BM grupo-específica: soma de PLFAs do grupo Dados quantitativos obtidos na análise de PLFA Método 2. Quantificação do P na fase orgânica Princípio: P na fase orgânica do extrato é derivado de fosfolipídios. Vantagem: simples, dispensa CG

Efeito da Substituição de Floresta por Agricultura (Trigo ~26 anos) sobre a BM

Método BM 1 (Soma dos PLFAs) Floresta vs. Agricultura (Trigo ~26 anos) Floresta Agricultura

Método BM 2. Quantificação do P na Fase Orgânica do Extrato de Solo • Protocolo: White et al. (1979) (resumo) 1. 2. 3. 4. 5. 6. 7. Extração de lipídios totais, por 2 h (Bligh e Dyer, 1959); Partição com Na. Cl 2 N; Recuperação da fase orgânica (inferior); Secagem sob N 2 ultrapuro; Digestão com ácido perclórico, 180 o. C (3 -4 h); Adicionar solução molibdênio (White et al. , 1979); Após 10 min, adicionar solução de ANSA (1 -amino-2 -naftol-4ácido sulfônico); Ferver por 10 min. em banho-maria; Ler absorvância a 830 nm. 8. 9.

Comparação com Outros Métodos de Deteminação da BM (Frostegard et al. , 1991) • P-lipídio vs. SIR (respiração induzida por substrato) SIR ( g CO 2/g solo) = 17, 6 + 0, 13 P-lipídio (nmol P/g solo) R 2 = 0, 86 • P-lipídio vs. conteúdo celular de ATP ( g ATP/g solo) = 0, 92 + 0, 0046 P-lipídio (nmol P/g solo) R 2 = 0, 89

Estado Nutricional de Fungos do Solo • Bååth, 2003 – Princípio: sob excesso de C e deficiência de N ou P, a comunidade fúngica não cresce, mas acumula lipídios. – Experimento e Resultados: • Glucose apenas: NL aumenta e PL permanece constante • Glucose + N + P: NL constante e PL aumenta – Bååth, E. Microbial Ecology, 45: 373 -383, 2003

Protocolo: Estado Nutricional de Fungos do Solo • Bååth et al. , 2003 • Idem procedimento para estrutura da comunidade, exceto: – – Não descartar a fração LN Fração de LN e PL são transesterificada Relação NL: PL do FAME 18: 2 6 c (fungo) Quanto maior a relação NL: PL maior é a deficiência nutricional (N ou P) da comunidade fúngica

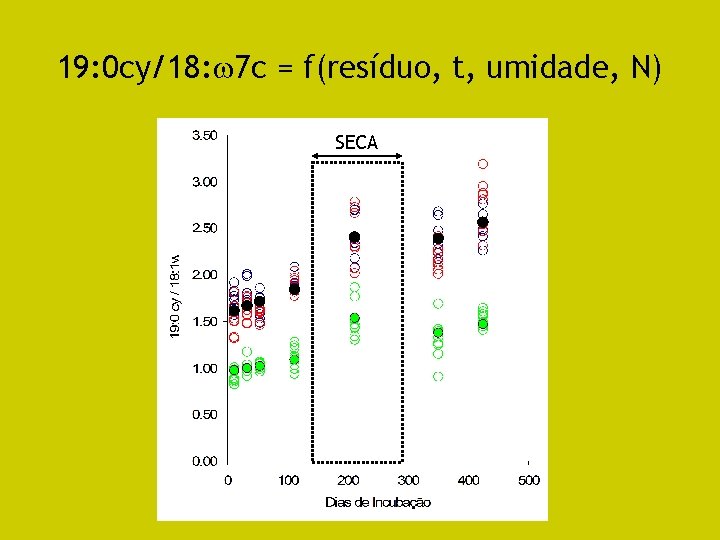
Respostas de Bactérias a Estresses Ambientais • Sob estresses (dessecamento do meio e restrição nutricional) – Aumento da relação entre FAMEs cíclicos e precursores • 19: 0 cy / 18: 1 7 c ou 17: 0 cy /16: 1 7 c – Kieft et al. , 1994, AEM, 60: 3292 -3299, 1994 – Custo de manutenção para mitigar estresse: aumenta gasto de energia, menor eficiência de conversão de carbono em biomassa microbiana: impacto na conservação de MOS.

Experimento (Fernandes, 2007) • Incubação por 425 d, microcosmos, solo de Tabuleiros Costeiros, SE (Argissolo amarelo) • 3 resíduos x 2 N x 7 datas, 4 repetições – Resíduos: controle, palha de milho, casca de coco – Doses N: equivalente 150 kg N ha-1 – Datas: 12, 33, 54, 112, 212, 350 e 425 dias – 130 aos 280 d sob seca (-1, 5 MPa) • Variável resposta: 19: 0 cy/18: 1 7 c • Estatística: Regressão múltipla – stepwise, forward – Modelo inicial: grande média – Modelo cheio: todas as interações entre todos os fatores – F para incluir termos no modelo < 4, 0

Regressão Múltipla (stepwise forward) • Call: lm(formula = ST ~ MILHO + TIME + UMIDO + TIME: MILHO, data = STRESS. 19. INCUB, na. action = na. exclude) • • • Residuals: Min 1 Q Median 3 Q Max -0. 4682 -0. 1053 -0. 006328 0. 111 0. 6178 • • Coefficients: • • • Residual standard error: 0. 1823 on 163 degrees of freedom Multiple R-Squared: 0. 8908 F-statistic: 332. 3 on 4 and 163 df, the p-value is 0 (Intercept) MILHO TIME UMIDO TIME: MILHO Value Std. Error t value Pr(>|t|) 1. 9131 0. 0448 42. 7342 0. 0000 -0. 6297 0. 0451 -13. 9688 0. 0000 0. 0023 0. 0001 20. 2161 0. 0000 -0. 3261 0. 0404 -8. 0648 0. 0000 -0. 0011 0. 0002 -5. 6939 0. 0000

19: 0 cy/18: 7 c = f(resíduo, t, umidade, N) SECA

• FIM

Obrigado pela atenção Marcelo F. Fernandes marcelo@cpatc. embrapa. br 4009 1360
 Camada gasosa
Camada gasosa Ouvidoria como ferramenta de gestão
Ouvidoria como ferramenta de gestão Cunha ferramenta de corte
Cunha ferramenta de corte Linhas de exus
Linhas de exus Ferramenta solver
Ferramenta solver Lipdeos
Lipdeos Lipdios
Lipdios Molecula de lipidio
Molecula de lipidio Lipdios
Lipdios A grande jornada coaching pdf
A grande jornada coaching pdf Lipídios derivados
Lipídios derivados Lipdios
Lipdios Observe as especies a seguir 16o8 23na11
Observe as especies a seguir 16o8 23na11 Periodo composto
Periodo composto O que são estudos de coorte
O que são estudos de coorte Tipos de estudos epidemiológicos
Tipos de estudos epidemiológicos Estudos europeus
Estudos europeus Mites
Mites Economia internacional e estudos europeus
Economia internacional e estudos europeus Ortopatia
Ortopatia Estudo longitudinal
Estudo longitudinal Semana de estudos intensivos
Semana de estudos intensivos Ipeak roteiro de estudos
Ipeak roteiro de estudos Tipos de estudo epidemiologico
Tipos de estudo epidemiologico Tipos de estudo epidemiológico
Tipos de estudo epidemiológico Estudos transversais epidemiologia
Estudos transversais epidemiologia Bons estudos
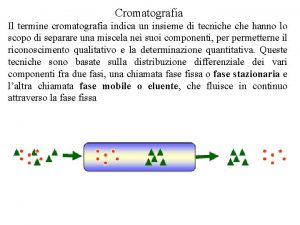
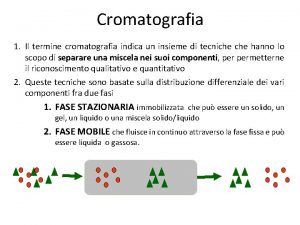
Bons estudos Cromatografia
Cromatografia Afinidade
Afinidade Estrazione clorofilla esperimento
Estrazione clorofilla esperimento Cromatografía líquido-líquido
Cromatografía líquido-líquido Hplc fase inversa
Hplc fase inversa Placa de cromatografia em camada delgada
Placa de cromatografia em camada delgada Cromatografía introducción
Cromatografía introducción Imac cromatografia
Imac cromatografia Objetivos de la cromatografia
Objetivos de la cromatografia Elucion isocratica
Elucion isocratica Corso gascromatografia napoli
Corso gascromatografia napoli Cromatografia a scambio ionico
Cromatografia a scambio ionico Cromatografia spiegazione semplice
Cromatografia spiegazione semplice Objetivos de la cromatografia
Objetivos de la cromatografia Hplc fase normal
Hplc fase normal Mikhail tswett cromatografia
Mikhail tswett cromatografia Cromatografia planare
Cromatografia planare Rumus waktu retensi
Rumus waktu retensi Factor de selectividad cromatografia
Factor de selectividad cromatografia Cromatografia
Cromatografia Pigmentos espinaca cromatografia
Pigmentos espinaca cromatografia Cromatografia
Cromatografia Cromatografia en columna
Cromatografia en columna Cromatografia della clorofilla
Cromatografia della clorofilla Cromatografia de lichide de înaltă performanţă (hplc)
Cromatografia de lichide de înaltă performanţă (hplc) Tu nombre
Tu nombre Cómo cómo dijo
Cómo cómo dijo Si no vives como piensas acabarás pensando como vives
Si no vives como piensas acabarás pensando como vives Vivir como hijos de dios
Vivir como hijos de dios Nadie ha visto a dios
Nadie ha visto a dios Como ser como cristo
Como ser como cristo Virtudes de una persona
Virtudes de una persona Proyecto de vida quien soy
Proyecto de vida quien soy Varianza ejemplos
Varianza ejemplos Mediana datos no agrupados formula
Mediana datos no agrupados formula Obedecer es mejor que los sacrificios
Obedecer es mejor que los sacrificios Bibliografía ejemplo
Bibliografía ejemplo El diario como instrumento para transformar la práctica
El diario como instrumento para transformar la práctica Acatadora
Acatadora Como hacer la introduccion de un informe
Como hacer la introduccion de un informe Ejemplo de tesis discutible
Ejemplo de tesis discutible Carta informal
Carta informal Como hacer una excusa para el colegio
Como hacer una excusa para el colegio Biografia de laura esquivel como agua para chocolate
Biografia de laura esquivel como agua para chocolate Humor da tirinha decorre
Humor da tirinha decorre Estilo directo
Estilo directo He es un texto porque
He es un texto porque Calcule:image10
Calcule:image10 Fiesta de la virgen de juquila
Fiesta de la virgen de juquila Porque tuvo dios que mostrarse para que sepamos como es
Porque tuvo dios que mostrarse para que sepamos como es Como hacer para qu
Como hacer para qu Como hacer para qu
Como hacer para qu Que hacer para qu
Que hacer para qu Como hacer para qu
Como hacer para qu Como colocar a girafa na geladeira
Como colocar a girafa na geladeira Porque edipo mando a llamar a tiresias
Porque edipo mando a llamar a tiresias Cómo preparar un discurso
Cómo preparar un discurso Para la cena como _____.
Para la cena como _____. Notas periodisticas cortas
Notas periodisticas cortas Honra a tu padre y madre
Honra a tu padre y madre Como hacer para qu
Como hacer para qu Como faço para tirar leite materno
Como faço para tirar leite materno Te escribo para saludarte
Te escribo para saludarte Como tomar los medicamentos para el helicobacter pylori
Como tomar los medicamentos para el helicobacter pylori Magic realism in como agua para chocolate
Magic realism in como agua para chocolate Ejemplo de discurso expositivo
Ejemplo de discurso expositivo Esquema de ensayo
Esquema de ensayo Como hacer para qu
Como hacer para qu Macroambiente
Macroambiente Who is the narrator of like water for chocolate
Who is the narrator of like water for chocolate Quien invento el antecesor del ebook
Quien invento el antecesor del ebook Nacemos para ser felices
Nacemos para ser felices Unesp em uma obra para permitir
Unesp em uma obra para permitir Consumir para viver ou viver para consumir
Consumir para viver ou viver para consumir Subtrair para si ou para outrem coisa alheia móvel
Subtrair para si ou para outrem coisa alheia móvel Trabaja para vivir no vivas para trabajar
Trabaja para vivir no vivas para trabajar Reglas para clases virtuales para niños camara encendida
Reglas para clases virtuales para niños camara encendida Reglas para clases virtuales para niños camara encendida
Reglas para clases virtuales para niños camara encendida Todo para todos nada para nosotros
Todo para todos nada para nosotros Modelo bidimensional para evaluar el riesgo para la salud
Modelo bidimensional para evaluar el riesgo para la salud Yo soy de mi amado en hebreo
Yo soy de mi amado en hebreo Injecao ventro glútea
Injecao ventro glútea La leyenda de los temblores
La leyenda de los temblores Frases para cuidar el agua para niños
Frases para cuidar el agua para niños Microcilios
Microcilios Dia del educador
Dia del educador Fuentes sonoras
Fuentes sonoras Como fazer uma webquest
Como fazer uma webquest Como calcular el volumen de un prisma hexagonal
Como calcular el volumen de un prisma hexagonal Correos formales
Correos formales Como se calcula la prima de antiguedad
Como se calcula la prima de antiguedad Como se trabsmite el sida
Como se trabsmite el sida Sintomas vih
Sintomas vih Vigila tus pensamientos porque se convierten en palabras
Vigila tus pensamientos porque se convierten en palabras Las 14 estaciones de jesus
Las 14 estaciones de jesus Qual a diferença entre verbos transitivos e intransitivos
Qual a diferença entre verbos transitivos e intransitivos Trabalenguas nadie silba como silvia silva
Trabalenguas nadie silba como silvia silva Diagrama de bloques de un velador
Diagrama de bloques de un velador