PROGRAMA DE PSGRADUAO EM MEDICINA VETERINRIA DEPTO DE













































- Slides: 62

PROGRAMA DE PÓS-GRADUAÇÃO EM MEDICINA VETERINÁRIA DEPTO DE MEDICINA VETERINÁRIA PREVENTIVA BIOLOGIA MOLECULAR Aula 7

REPLICAÇÃO DO DNA



DNA polimerases: enzimas que sintetizam DNA - Replicação do genoma: REPLICASES - Outras: REPARO e funções auxiliares na replicação - Procariotas: 3 DNA polimerases (DNA pol I, III) -Eucariotas: 5 polimerases ( , ß, , , ) - Mas, apenas UMA é a replicase

PROPRIEDADES das DNA pol - Sintetizam DNA a partir de moldes DNA - Necessitam de PRIMER - Mecanismo de síntese é similar - Síntese na direção 5’ > 3’ - Alta fidelidade - Atividade proofreading

Polimerização

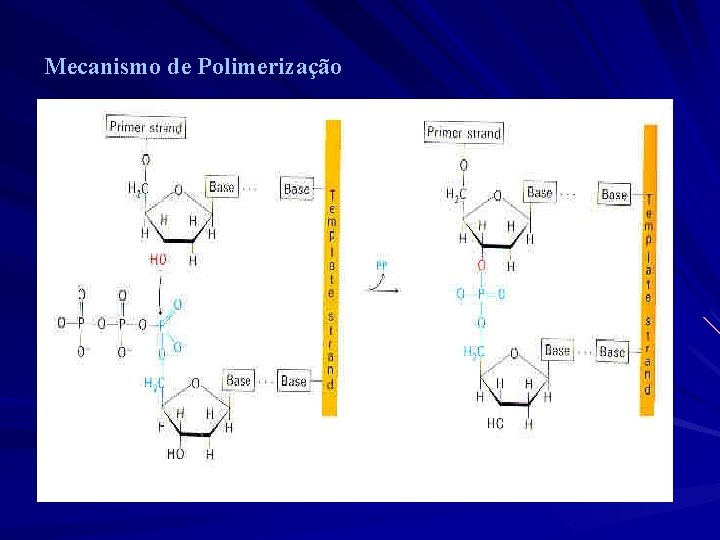
Mecanismo de Polimerização

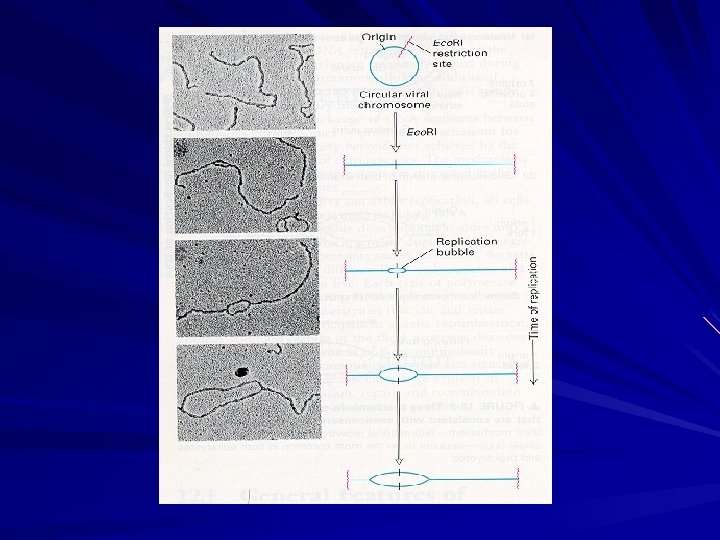
A REPLICAÇÃO É SEMI-CONSERVATIVA


A REPLICAÇÃO É BIDIRECIONAL






A REPLICAÇÃO É SEMI-DESCONTÍNUA

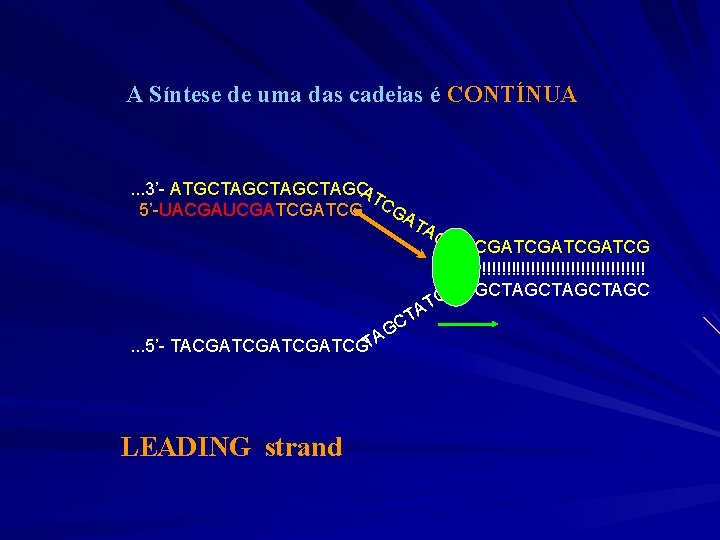
A Síntese de uma das cadeias é CONTÍNUA . . . 3’- ATGCTAGCTAGC AT 5’-UACGAUCGATCG CGA TA . . . 5’- TACGATCGATCGTA LEADING strand TA C G G T ATCGATCG !!!!!!!!!!!!!!!!!!!! C TAGCTAGC

E A SÍNTESE DA OUTRA CADEIA? ? ? ?

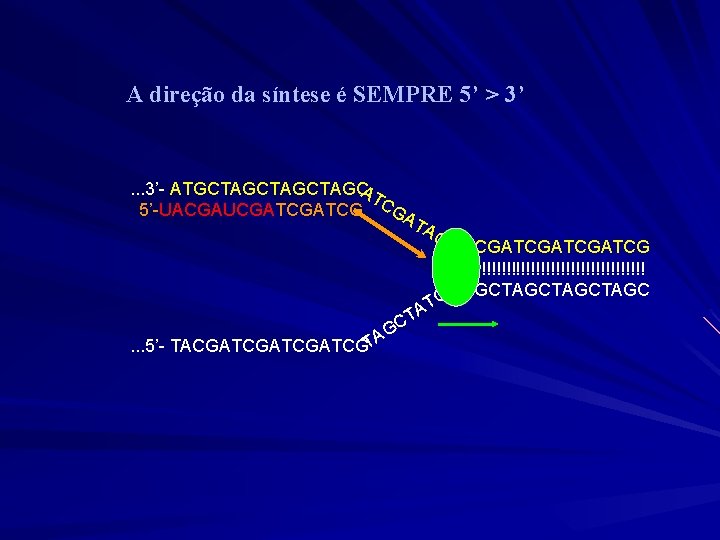
A direção da síntese é SEMPRE 5’ > 3’ . . . 3’- ATGCTAGCTAGC AT 5’-UACGAUCGATCG CGA TA . . . 5’- TACGATCGATCGTA TA C G G T ATCGATCG !!!!!!!!!!!!!!!!!!!! C TAGCTAGC

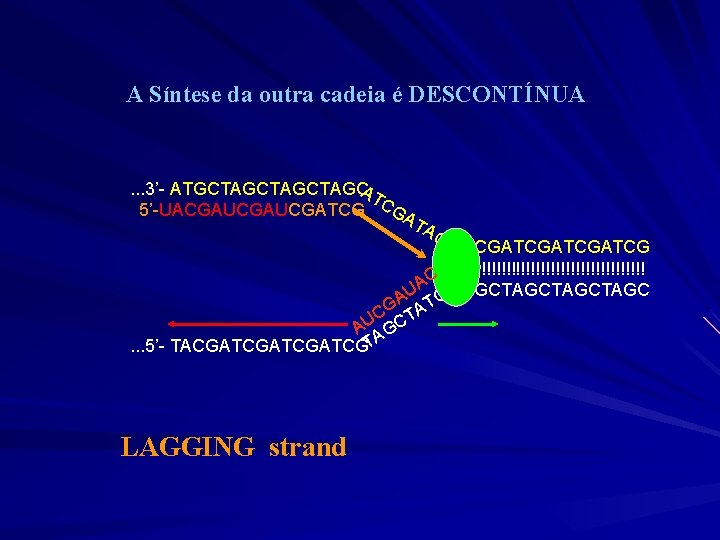
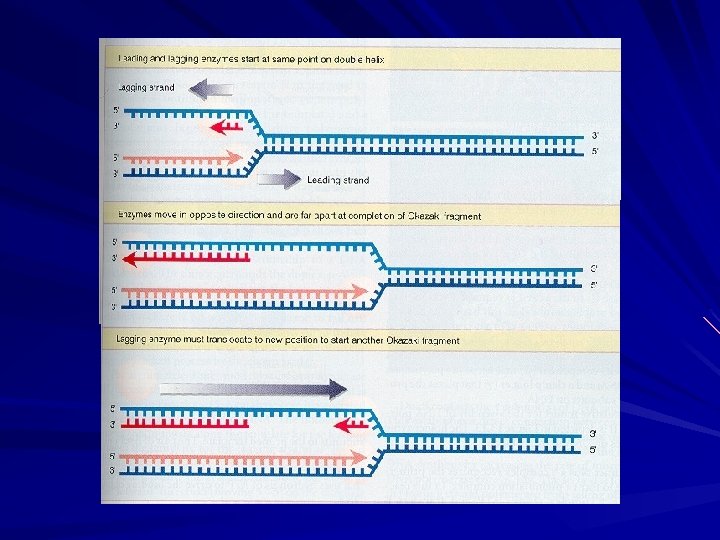
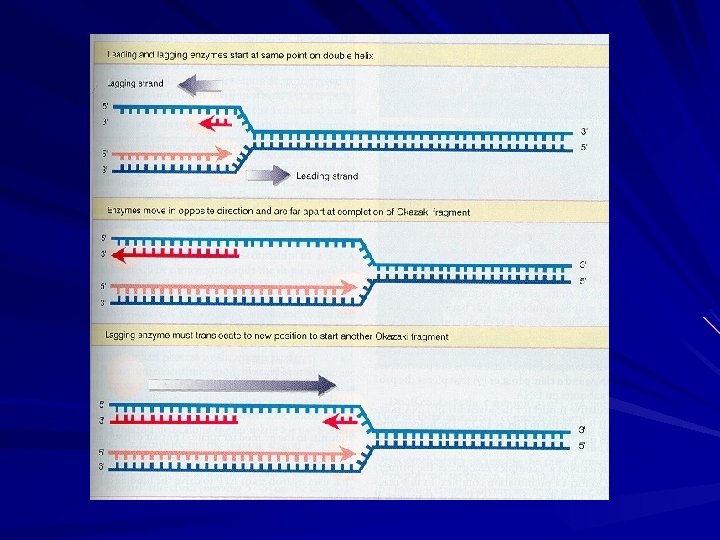
A Síntese da outra cadeia é DESCONTÍNUA . . . 3’- ATGCTAGCTAGC AT 5’-UACGAUCGATCG CGA TA G ATCGATCG !!!!!!!!!!!!!!!!!!!! C A TAGCTAGC U A TC G C TA U A AGC. . . 5’- TACGATCGATCGT LAGGING strand

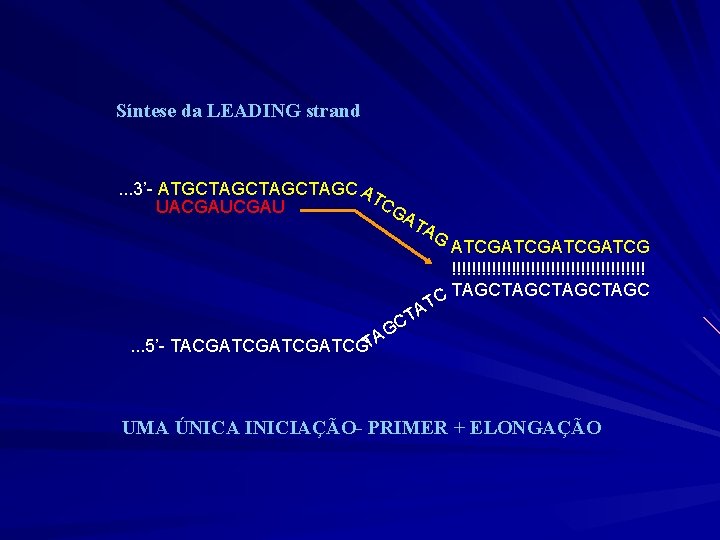
Síntese da LEADING strand . . . 3’- ATGCTAGCTAGC AT CG UACGAU AT . . . 5’- TACGATCGATCGTA AG TA C G ATCGATCG !!!!!!!!!!!!!!!!!!!! C TAGCTAGC T UMA ÚNICA INICIAÇÃO- PRIMER + ELONGAÇÃO

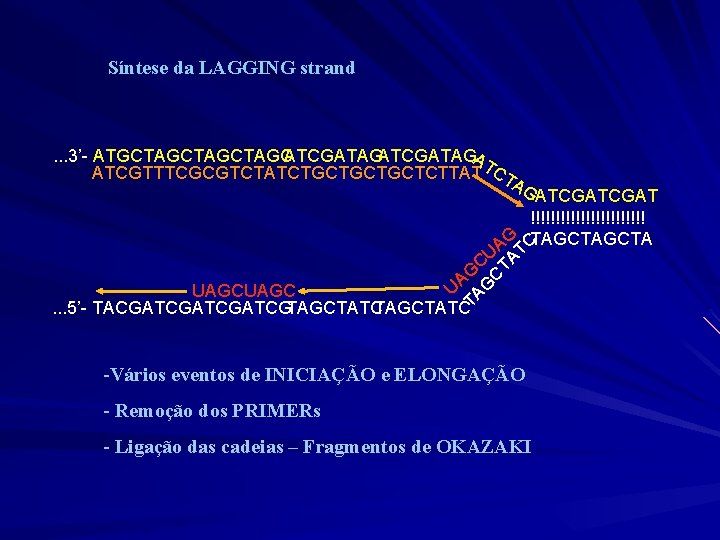
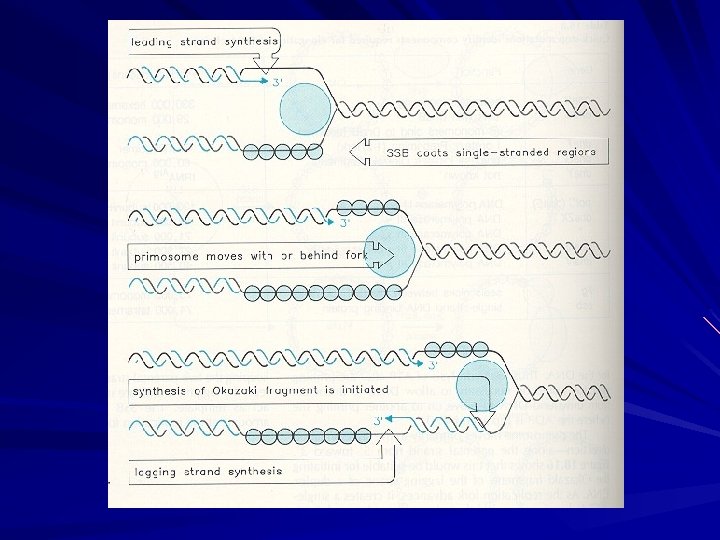
Síntese da LAGGING strand . . . 3’- ATGCTAGCTAGCATCGATAGAT ATCGTTTCGCGTCTATCTGCTGCTGCTCTTAT CT AG C TA G G A U UAGC. . . 5’- TACGATCGATCGTAGCTATC TA TC ATCGAT !!!!!!!!!!!! G TAGCTA A CU -Vários eventos de INICIAÇÃO e ELONGAÇÃO - Remoção dos PRIMERs - Ligação das cadeias – Fragmentos de OKAZAKI





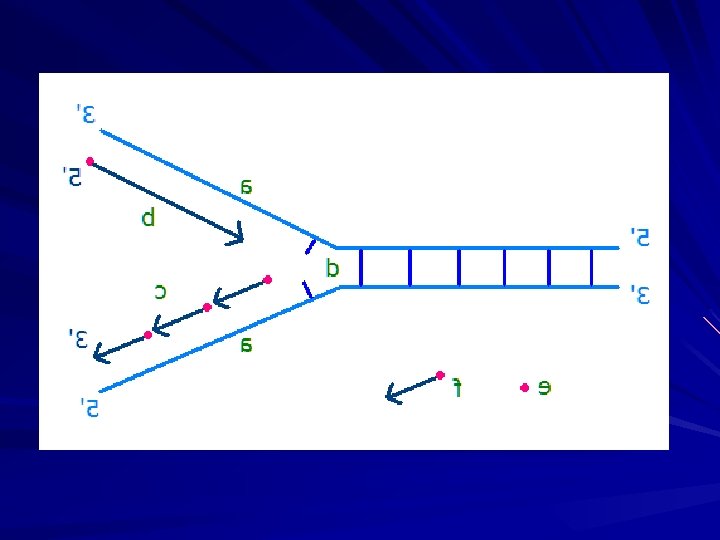
REPLICAÇÃO SEMIDESCONTÍNUA (O filme)


REPLICAÇÃO EM PROCARIOTAS

DNA POLIMERASES em E. coli - DNA pol I – REPARO (tbém auxiliar na Replicação) - DNA pol II - REPARO - DNA pol III – É A REPLICASE!!!! - Apenas a I e III são ESSENCIAIS

PROPRIEDADES DAS DNA pol de E. coli I II III -Polimerização 5’>3’ + + + -Atividade exonuclease -3’>5’ + + + -5’>3’ + - - -Taxa de síntese por seg 600 , 30. 000 -Moléculas por célula 400 , 10 -20

“PROOFREADING” (3’ > 5’ EXONUCLEASE)


DNA pol I (E. coli) Primeira DNA pol caracterizada Polipeptídeo único: 104 k. Da Clivagem gera dois produtos: 68 k. Da e 35 k. Da Klenow fragment (68 k. Da): polimerase, exo 3’ > 5’ Fragmento de 35 k. Da: atividade 5’ > 3’ exonuclease Capaz de iniciar a síntese em “NICKs”(única) Funções – -Reparo (Nick translation) -Termina a síntese da “lagging strand”

NICK TRANSLATION


DNA pol III (Replicase de E. coli) Complexo enzimático (várias subunidades) Atividade polimerase e 3’ > 5’ exonuclease Síntese da leading e lagging strands Fidelidade: 10 -8 - 10 -10 (1 erro/genoma/1000 ciclos) Proofreading- aumenta fidelidade 10 a 200 vezes Síntese: 800 a 1000 nt/s

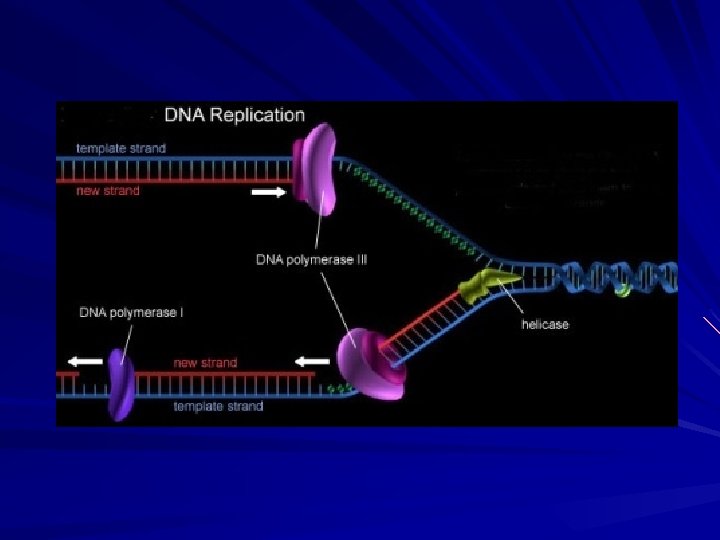
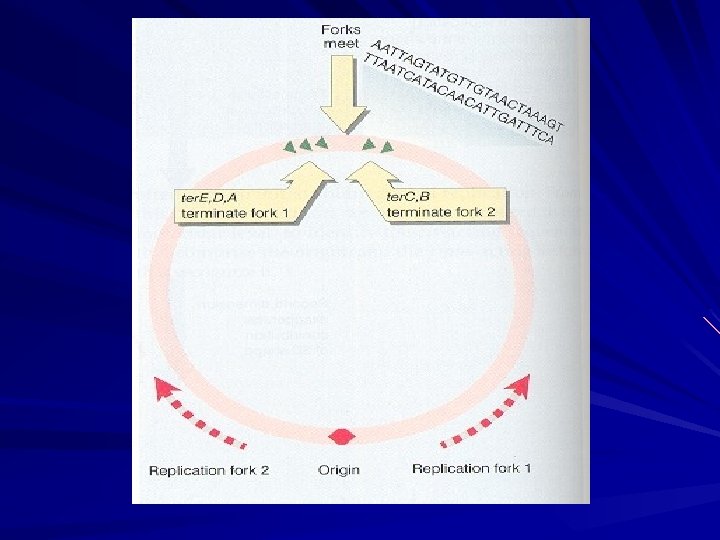
ETAPAS da REPLICAÇÃO 1. INICIAÇÃO Primossoma 2. ELONGAÇÃO Replissoma 3. TERMINAÇÃO/SEGREGAÇÃO

REPLICON: A UNIDADE DE REPLICAÇÃO Inicia na ORIGEM - termina no TERminador BACTÉRIAS: um ÚNICO REPLICON (4. 000 kb) REPLICAÇÃO: 46’ ORI: 245 bp Contém 3 (13 bp repeats), 4 (9 bp repeats) Sítios p/ ligação da proteína Dna. A


ORIgem em E. coli -245 bp -Três 13 mer repeats - Quatro 9 mer repeats

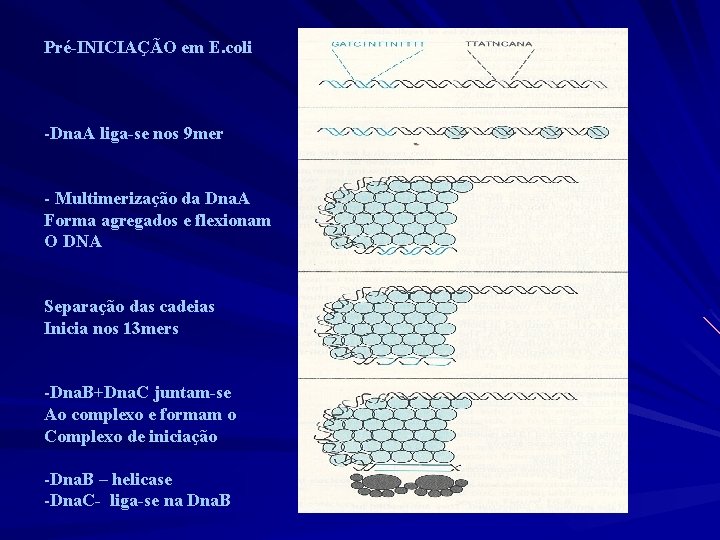
Pré-INICIAÇÃO em E. coli -Dna. A liga-se nos 9 mer - Multimerização da Dna. A Forma agregados e flexionam O DNA Separação das cadeias Inicia nos 13 mers -Dna. B+Dna. C juntam-se Ao complexo e formam o Complexo de iniciação -Dna. B – helicase -Dna. C- liga-se na Dna. B


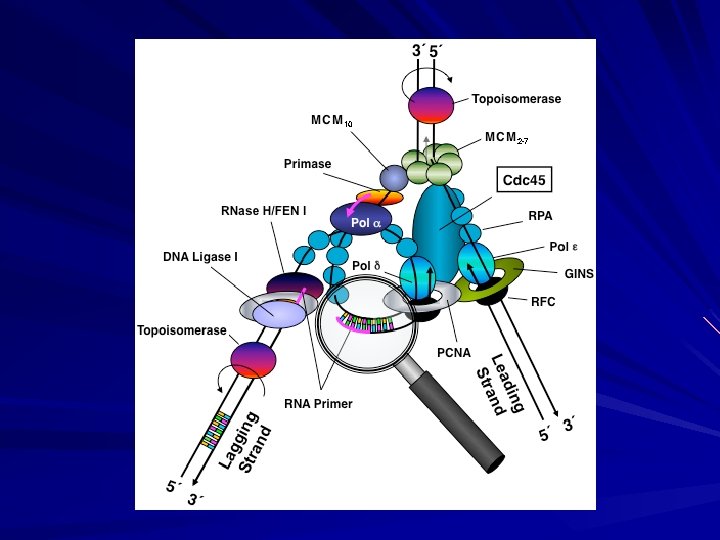
FUNÇÕES NO REPLICATION FORK Função E. coli Helicase Dna. B Primase Dna. G Clamp subunit Catálise Pol III Remoção do Primer E substituição por DNA Pol I Ligação dos Okazaki Ligase

Helicase Primase DNApol III SSB DNApol I



REPLICAÇÃO EM EUCARIOTAS

EUCARIOTAS Múltiplos REPLICONS YEAST (40 kb), mamíferos (100 kb) ORI? Terminador? Iniciam na fase S Síntese (2000 bp/min x 50. 000/min em E. coli) Síntese não é simultânea Replicação completa em 6 horas


DNA pol de EUCARIOTAS Pol - Primase (síntese dos primers RNA) Ld & Lg Pol - Reparo Pol - Replicase (Ld & Lg) Pol - Replicação do DNA mitocondrial Pol - Reparo

PROPRIEDADES DAS DNA POL de EUCARIOTAS -Polimerização 5’>3’ + + + -Atividade exo 3’>5’ - - + + + -Atividade PRIMASE + - - - Localização N M N N N


FUNÇÕES NO REPLICATION FORK Função E. coli Hela cells Helicase Dna. B ? Primase Dna. G Pol alfa/primase Clamp subunit PCNA Catálise Pol III Pol Remoção do Primer Pol I MF 1 Ligação dos Okazaki Ligase I



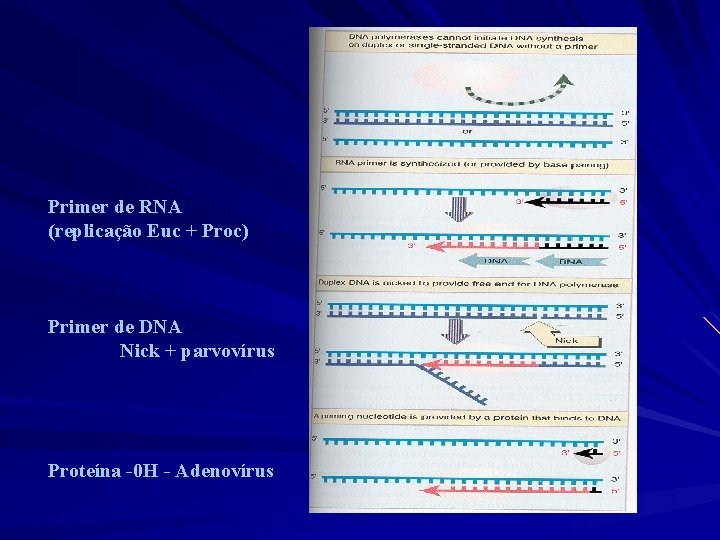
As DNA pol PRECISAM de Primer!!!! PRIMERS utilizados pelas DNA Pol Primer de RNA (replicação procariotas/eucariotas) O próprio DNA-OH (reparo, alguns vírus) Proteína (alguns vírus)

Primer de RNA (replicação Euc + Proc) Primer de DNA Nick + parvovírus Proteína -0 H - Adenovírus

MUTAÇÕES (definição) Origem: 1. Erros da Polimerase durante a REPLICAÇÃO 2. Erros da Polimerase durante o REPARO 3. Lesões (cross-link) durante a interfase Mutações em ponto, deleções e inserções Podem ter efeitos diversos, dep. do tipo, da célula e do local Em células somáticas: podem levar a neoplasias!

MUTAÇÕES EM PONTO: 1. De acordo com a troca de base: Transições ou Transversões 2. De acordo com o efeito: - Silenciosa (mesmo a. a) - “Missense” (a. a. diferente) - “Nonsense” (stop codon) Não altera a fase de leitura 2. Inserção 3. Deleção Alteram a fase de leitura (frameshift)

PROGRAMA DE PÓS-GRADUAÇÃO EM MEDICINA VETERINÁRIA DEPTO DE MEDICINA VETERINÁRIA PREVENTIVA BIOLOGIA MOLECULAR Aula 7
 O que é medicina forense
O que é medicina forense Dipartimento medicina perugia
Dipartimento medicina perugia Medicina dello sport coni
Medicina dello sport coni Mir ramirez
Mir ramirez University of medicine and pharmacy timisoara
University of medicine and pharmacy timisoara Algoritmo als farmaci
Algoritmo als farmaci Gastrostomia
Gastrostomia C.a medicina
C.a medicina Medicina universidad pedro de valdivia
Medicina universidad pedro de valdivia Medicinska fizika
Medicinska fizika Meronimo e homonimo
Meronimo e homonimo Icr medicina
Icr medicina Rcu medicina
Rcu medicina Gndi laudo
Gndi laudo Sistema pico medicina
Sistema pico medicina Fevi normal
Fevi normal Soba cie 10
Soba cie 10 Yatrofisica
Yatrofisica Gradna bolka
Gradna bolka Palacio de medicina
Palacio de medicina Facoltà di medicina cagliari
Facoltà di medicina cagliari Que es la medicina social
Que es la medicina social Master medicina trasfusionale
Master medicina trasfusionale “principiis obsta, sero medicina paratur.”
“principiis obsta, sero medicina paratur.” Telescopagem medicina
Telescopagem medicina Infección tracto urinario
Infección tracto urinario Heliotropo medicina
Heliotropo medicina Soap exemplo
Soap exemplo Atos carnais
Atos carnais Alas peruanas medicina
Alas peruanas medicina Società italiana di medicina ambientale
Società italiana di medicina ambientale Dipartimento di medicina clinica e sperimentale pisa
Dipartimento di medicina clinica e sperimentale pisa Studenti e professori uniti per
Studenti e professori uniti per Medicina di laboratorio federici
Medicina di laboratorio federici Ecomapa familiar
Ecomapa familiar Deguello medicina forense
Deguello medicina forense Unidad docente medicina familiar y comunitaria madrid
Unidad docente medicina familiar y comunitaria madrid Diafonía medicina
Diafonía medicina Hvala na pažnji medicina
Hvala na pažnji medicina Nenad roso
Nenad roso Crai ub cataleg
Crai ub cataleg Apergaminamiento lesion
Apergaminamiento lesion Cus (compression ultrasonography)
Cus (compression ultrasonography) Casosclinicosnn
Casosclinicosnn Vmd medicina
Vmd medicina Instrumentos de medicina familiar
Instrumentos de medicina familiar Vocacion medicina
Vocacion medicina Ramsay rass
Ramsay rass Que es ms en medicina
Que es ms en medicina Medicina pericial
Medicina pericial Notas de peso para medicina
Notas de peso para medicina Socijalna medicina
Socijalna medicina Germanska nova medicina
Germanska nova medicina Tat in laboratorio
Tat in laboratorio Medicina urgenza universitaria pisa
Medicina urgenza universitaria pisa Universidade federal do amazonas medicina
Universidade federal do amazonas medicina Codigo hsi yuan lu
Codigo hsi yuan lu Foca medicinski fakultet
Foca medicinski fakultet Codici classroom unife medicina corsi a scelta
Codici classroom unife medicina corsi a scelta Saag medicina
Saag medicina Medicina espe
Medicina espe Evaluacion preanestesica
Evaluacion preanestesica Casi clinici medicina interna
Casi clinici medicina interna






















































































