Le Malattie genetiche Derivano da mutazioni del patrimonio



























































































- Slides: 123

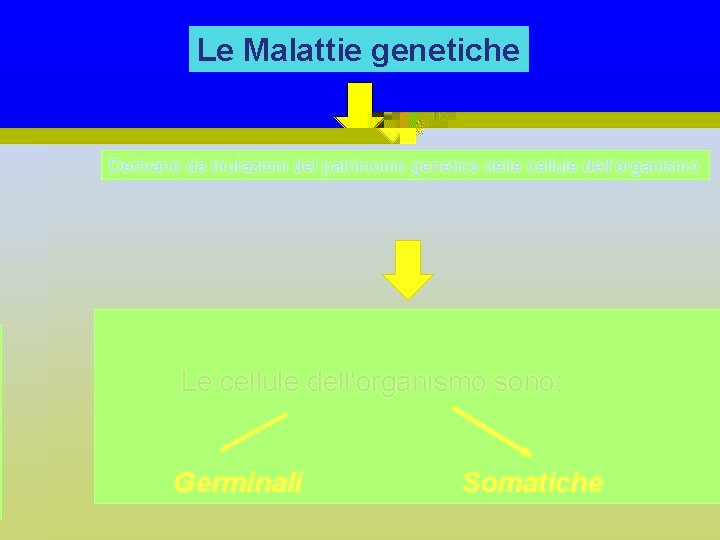
Le Malattie genetiche Derivano da mutazioni del patrimonio genetico delle cellule dell’organismo Le cellule dell'organismo sono: Germinali Somatiche

Mutazioni nelle cellule germinali trasmissione alla prole malattie ereditarie Mutazioni nelle cellule somatiche la malattia non viene trasmessa alla prole Alterazioni in cellule somatiche sono presenti nei tumori

Classificazione delle malattie genetiche Monogeniche o Mendeliane Cromosomiche Multifattoriali dovute all’alterazione di un singolo gene causate da alterazioni del numero o della struttura dei cromosomi dovute all'interazione fra geni diversi ed ambiente

Modalità di trasmissione delle malattie mendeliane Sono autosomiche se trasmesse dai cromosomi autosomici (dal cromosoma 1 al cromosoma 22) X-linked (legate al cromosoma X) se trasmesse dal cromosoma X Mutazioni dominanti: condizioni in un individuo eterozigote, in cui l’allele mutato si esprime fenotipicamente. Mutazioni recessive: condizioni in un individuo eterozigote in cui l’allele mutato non si manifesta fenotipicamente. Le mutazioni recessive devono coinvolgere i due alleli per manifestarsi clinicamente

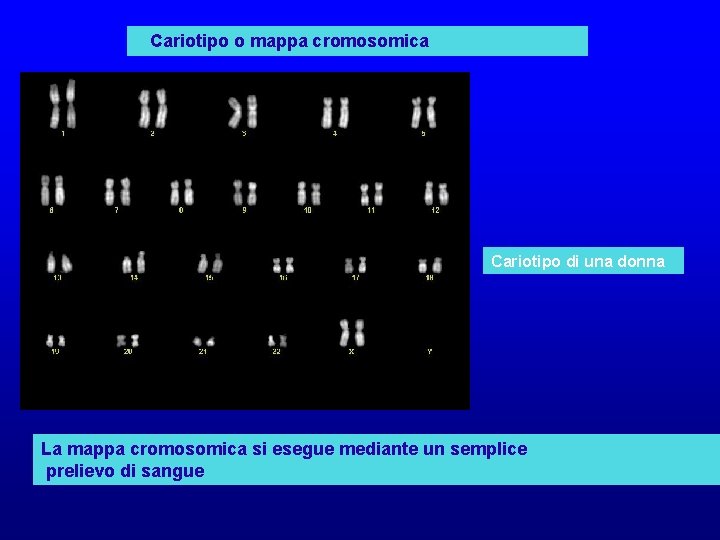
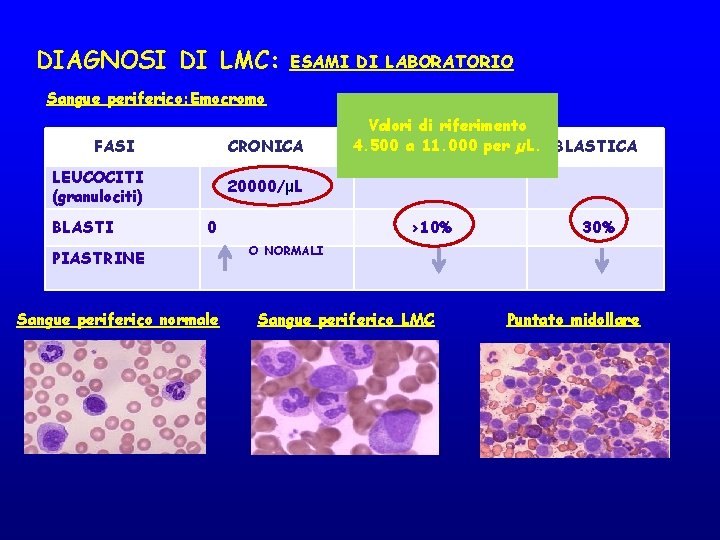
Malattie cromosomiche Causate da anomalie cromosomiche possono essere numeriche strutturali Le più frequenti anomalie numeriche sono le trisomie e le monosomie es. sindrome di Down trisomia del cromosoma 21 Le anomalie cromosomiche vengono evidenziate mediante ANALISI DEL CARIOTIPO

Cariotipo o mappa cromosomica Cariotipo di una donna La mappa cromosomica si esegue mediante un semplice prelievo di sangue

Malattie multifattoriali Sono determinate dall’interazione tra fattori genetici e fattori ambientali Sono trasmesse secondo modalità diverse da quelle mendeliane classiche ( fattori predisponenti all’insorgenza della malattia) Si manifestano con notevole variabilità nelle popolazioni Es. Diabete mellito, malattia celiaca. . etc

Da che cosa sono causate le mutazioni? . Agenti fisici (ad es raggi X o UV) Agenti mutageni chimici I mutageni chimici sono molecole che si combinano con il DNA oppure causano cambiamenti chimici nelle basi nucleotidiche oppure sono simili alle basi nucleotidiche e vengono incorporate al loro posto causando però errori di appaiamento

I diversi tipi di mutazioni Mutazioni geniche (o puntiformi) Mutazioni cromosomiche: §Mutazioni nel numero dei cromosomi § Mutazioni nella struttura

Mutazioni geniche • Le mutazioni geniche o puntiformi sono dovute in gran parte alla sostituzione di una singola base nucleotidica del DNA con un’altra • Altri tipi di mutazione si originano in seguito alla perdita (delezione) o alla inserzione di una base nel filamento del DNA • Sostituzione • Delezione • Inserzione

Sostituzione di una base nucleotidica con un’altra La sostituzione di una base può avere conseguenze più o meno grandi sul prodotto finale (la proteina specificata da quel gene). In base alle conseguenze se ne distinguono tre tipi • mutazioni silenti • mutazioni di senso • mutazioni non senso

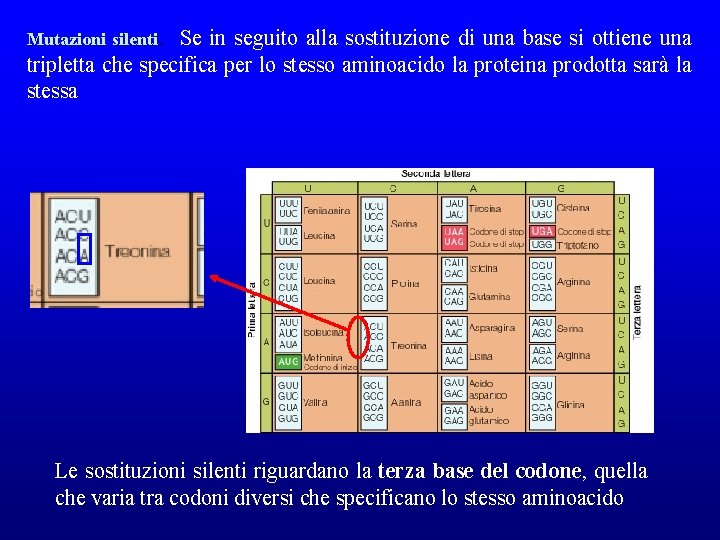
Mutazioni silenti. Se in seguito alla sostituzione di una base si ottiene una tripletta che specifica per lo stesso aminoacido la proteina prodotta sarà la stessa Le sostituzioni silenti riguardano la terza base del codone, quella che varia tra codoni diversi che specificano lo stesso aminoacido

Mutazioni di senso. Nella maggior parte dei casi la nuova tripletta codifica per un diverso aminoacido CCU Prolina GCU Alanina La proteina avrà quindi lo stesso numero di aminoacidi ma una sequenza che differisce per un aminoacido

La gravità degli effetti di una sostituzione dipenderà dalla somiglianza tra l’aminoacido sostituito e il nuovo e dalla posizione della sostituzione Esempio 1 La sequenza dell’emoglobina dell’uomo, del maiale, del cane o del cavallo differisce per parecchi aminoacidi e pur tuttavia tutte queste emoglobine svolgono la funzione in modo efficiente. Alcune regioni della proteina sono molto simili (ben conservate) anche tra specie filogeneticamente lontane (ad esempio nell’emogolobina la regione dell’eme). Esempio 2 D’altra parte una singola sostituzione (la valina al posto di glutammato in posizione 6) che si ha nell’anemia falciforme porta alla completa non funzionalità e alla morte del soggetto

Mutazioni nonsenso. Se il nuovo codone che si forma dalla sostituzione codifica per il segnale di stop avremo una proteina più corta della precedente AAG Lisina UAG Codone di stop

Delezione o inserzione di una base (mutazioni per spostamento della griglia di lettura) Talvolta l’errore consiste nell’inserire una base in più nella sequenza del DNA. Altre volte durante la replicazione o durante la riparazione del DNA si ha la perdita di una base In entrambi i casi la lettura di tutta la sequenza che segue viene completamente alterata A (inserzione) A U G A G G A C U C C C G G A U U A Met Arg Thr Pro Gly Leu A U G A G G A A C U C C Asp Ser C G G A U U A Arg Iso

Meccanismi patogenetici delle mutazioni • Alterazione strutturale di una proteina con formazione di un prodotto anomalo. - Carenza del prodotto normale; - Prodotto alterato che interferisce con quello normale; - Prodotto alterato con funzione diversa. • Alterazione quantitativa (riduzione o abolizione di una proteina WT), per mutazioni che riguardano sequenze di regolazione dell’espressione genica

Mutazioni nella struttura dei cromosomi Esistono 4 tipi principali di mutazione della struttura dei cromosomi • delezione • duplicazione • inversione • traslocazione


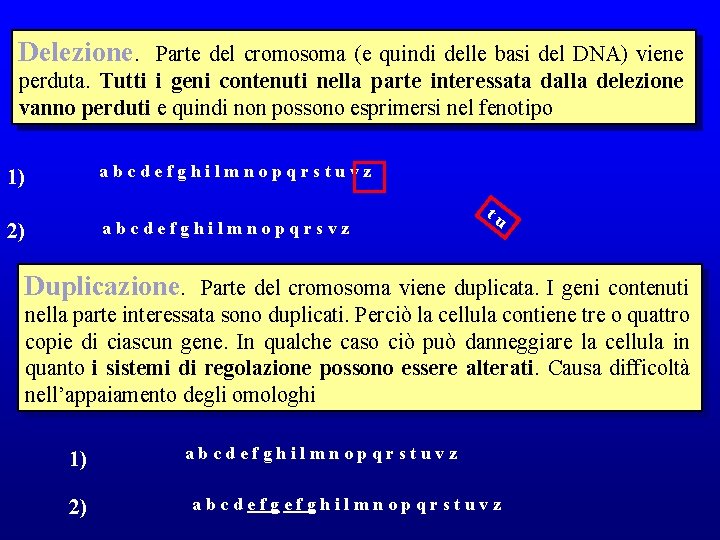
Delezione. Parte del cromosoma (e quindi delle basi del DNA) viene perduta. Tutti i geni contenuti nella parte interessata dalla delezione vanno perduti e quindi non possono esprimersi nel fenotipo abcdefghilmnopqrstuvz 1) abcdefghilmnopqrsvz 2) tu Duplicazione. Parte del cromosoma viene duplicata. I geni contenuti nella parte interessata sono duplicati. Perciò la cellula contiene tre o quattro copie di ciascun gene. In qualche caso ciò può danneggiare la cellula in quanto i sistemi di regolazione possono essere alterati. Causa difficoltà nell’appaiamento degli omologhi 1) 2) abcdefghilmnopqrstuvz abcdefgefghilmnopqrstuvz

Inversione. In seguito ad una rottura, una parte del cromosoma viene riunita in posizione invertita. A parte i geni che si possono trovare nel punto di rottura, non ci sono gravi conseguenze per la cellula. Tuttavia non può avvenire crossing over in questa regione in quanto essa non si appaia con quella del cromosoma omologo 1) abcdefghilmnopqrstuvz 2) abcdefghilmno p q r stuvz 3) abcdefghilmnorqpstuvz

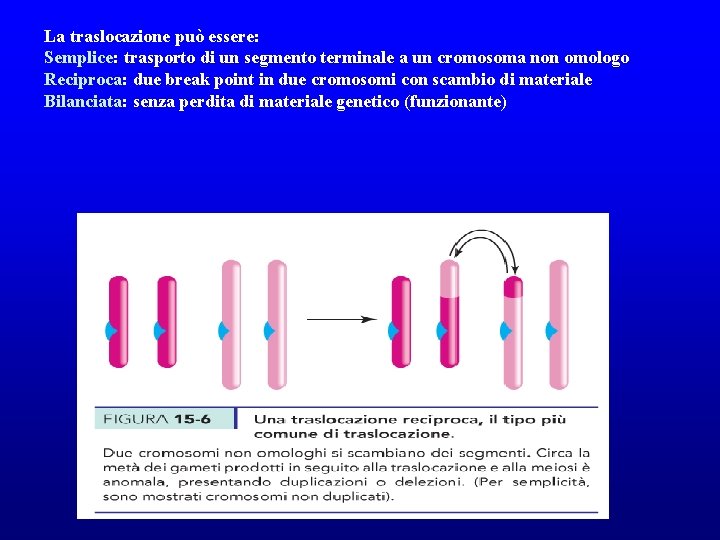
Traslocazione. Mutazione cromosomica che modifica la posizione di un segmento cromosomico e delle sequenze geniche in esso contenute. In seguito ad una rottura, una parte del cromosoma (es n° 6) si stacca e viene inserito in un cromosoma diverso (es n° 12). abcdefghilmnopqrstuvz 1) ABCDEFGHILMNOPQRSTUVZ abcdefghilmnopqrs 2) ABCDEFGHILMNOPQRSTUVZ tuvz

La traslocazione può essere: Semplice: trasporto di un segmento terminale a un cromosoma non omologo Reciproca: due break point in due cromosomi con scambio di materiale Bilanciata: senza perdita di materiale genetico (funzionante)

Variazioni nel numero dei cromosomi aneuploidia: perdita o aggiunta di 1 o più cromosomi rispetto all’assetto cromosomico normale Nullisomia: perdita di una coppia di cromosomi omologhi monosomia: perdita di un singolo cromosoma trisomia: aggiunta di un cromosoma ad una coppia di omologhi: es. trisomia 21 sindrome di Down tetrasomia: aggiunta di una coppia di cromosomi al normale assetto

Il cancro è una malattia genetica • • Deriva da alterazioni della sequenza del DNA Mutazioni somatiche Mutazioni che non vengono trasmesse alla generazione successiva e che non possono essere perciò ereditate Tali mutazioni vengono trasmesse alle cellule figlie dopo la divisione cellulare

Il cancro è una patologia monoclonale Tutte le cellule neoplastiche di un individuo originano da una singola cellula progenitrice La cellula clonale tumorale esposta ad un agente mutageno subisce un danno irreversibile al DNA Lo sviluppo del tumore è un processo che avviene in diverse fasi

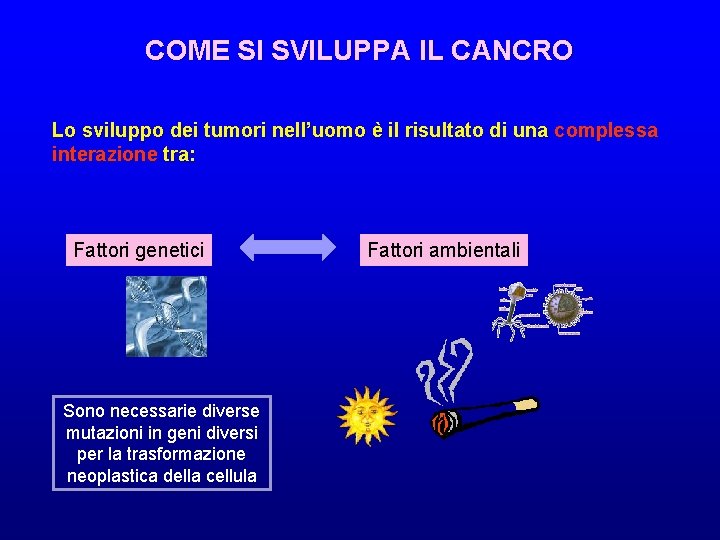
COME SI SVILUPPA IL CANCRO Lo sviluppo dei tumori nell’uomo è il risultato di una complessa interazione tra: Fattori genetici Sono necessarie diverse mutazioni in geni diversi per la trasformazione neoplastica della cellula Fattori ambientali

Vie cellulari alterate dal cancro • Tre sono i processi importanti che regolano il numero globale delle cellule di un individuo: • La divisione cellulare La morte cellulare Il differenziamento • •

Tre tipi di geni • Esistono tre tipi principali di geni mutati che contribuiscono allo sviluppo di un tumore • Oncogeni Geni oncosoppressori Geni coinvolti nel riparo del DNA • •

Oncogeni: geni che controllano positivamente la proliferazione cellulare • Scoperti nei virus trasformanti ad RNA • Il nome deriva dal virus in cui sono stati identificati: e. g. ras da Rous Sarcoma Virus • Controparti cellulari normali: proto-oncogeni che stimolano crescita e divisioni cellulari Proto-oncogeni Oncogeni

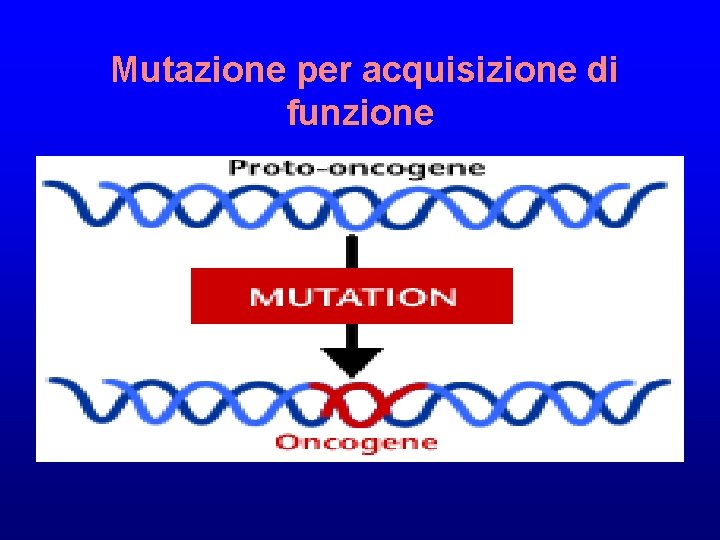
Mutazione per acquisizione di funzione

Ruolo degli oncogeni • • Ruolo normale: stimolazione crescita e proliferazione cellulare Mutazione > Aumento della funzione > trasformazione, invasività

Funzioni dei protoncogeni: stimolazione della crescita • • Fattori di crescita secreti Recettori di superficie cellulare Fattori intracellulari di trasduzione del segnale Proteine nucleari che legano il DNA

Mutazioni • Mutazioni puntiformi • Traslocazioni cromosomiche

Mutazione puntiforme • • • Oncogene ras (carcinoma della vescica) sul cromosoma 12 Mutazione puntiforme: Gly 12 Val Blocco della conversione di ras dalla forma attiva, legata al GTP, alla forma inattiva, legata al GDP, che deregola la crescita cellulare

Ras signaling pathway

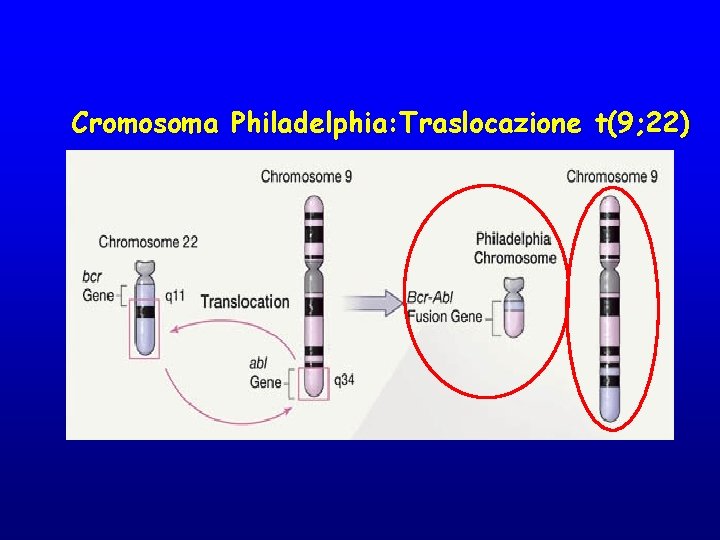
Traslocazione cromosomica: CML t(9; 22)(q 34; q 11)

t(9; 22)(q 34; q 11) • • • 9 q 34: ABL (abelson virus) protooncogene 22 q 11: BCR (breakpoint cluster region) Fusione BCR-ABL sul cromosoma 22 (cromosoma Philadelphia)

Oncogeni • • Mutazioni negli oncogeni sono di solito acquisite Mutazioni dominanti: una sola copia genica mutata è sufficiente a produrre il cancro per acquisizione di funzione “gain of function”

Tumor-suppressor genes Oncosoppressori • Controllo negativo della proliferazione cellulare Prevenzione di crescita cellulare abnorme – Normalmente inibiscono la crescita e la divisione cellulare – PRb: protoncogene presente su 13 q 14 –

Ereditarietà degli oncosoppressori • • La maggior parte dei tumori ereditari sono dovuti all’ereditarietà di un gene oncosoppressore mutato (first hit) Ma è necessario un secondo colpo (second hit) per lo sviluppo del tumore, pertanto il gene agisce in modo recessivo a livello cellulare • L’ereditarietà degli oncogeni è di tipo dominante in quanto basta che sia mutata una copia del gene affinchè compaia il tumore. • Nel caso degli oncosoppressori è necessario che entrambe le copie siano mutate.

Geni coinvolti nel DNA repair • Riparano i danni al DNA • sono considerati un sottogruppo di Geni Oncosoppressori • La perdita della loro funzione determina l’accumulo di mutazioni in altri geni cruciali

Il gene p 53 • • • La più comune modificazione genetica associata a cancro oncosoppressore, “guardiano del genoma” Espresso in grande quantità in cellule con danni al DNA Coinvolto nella fase G 1/S del ciclo cellulare (blocca la replicazione di cellule danneggiate) Coinvolto nell’apoptosi

BRCA 1 e BRCA 2 • Proteine la cui espressione si modifica in caso di “risposta al danno del DNA” • hanno attività di oncosoppressori • Responsabili dell’insorgenza del cancro della mammella

PROTEINA APC (Adenomatous Polyposis Coli) • E’ un gene oncosoppressore coinvolto nel controllo del processo di divisione cellulare • Mutazioni della proteina APC determinano aumento della divisione cellulare • Tumor suppressor gene nel cancro del colon retto

TEST GENETICI: DEFINIZIONE Insieme di analisi che vengono effettuate sul DNA, RNA, cromosomi, proteine metaboliti che consentono di evidenziare alterazioni dovute ad una patologia genetica, ereditabile o meno. Essi si distinguono in test clinici e di ricerca

TEST GENETICI: CLINICI Forniscono al paziente ed alla famiglia una risposta utile per l’inquadramento diagnostico, la prevenzione o un trattamento terapeutico I laboratori che effettuano test clinici devono seguire delle procedure approvate e standardizzate

TEST GENETICI: CLINICI • Test diagnostici: per confermare o escludere una malattia genetica nota o sospetta in un individuo sintomatico • Test predittivi: per individui asintomatici in una famiglia con una storia di una patologia genetica. Tali test sono indicati nel caso in cui si possa ridurre la morbidità o mortalità della malattia. • Test di portatore: per identificare individui con una storia familiare o anche a gruppi etnici con un elevato numero di portatori di uno specifico allele patologico • Test prenatali, effettuati in gravidanza per determinare lo stato di salute del feto. Tali test si effettuano solo se si conoscono in anticipo le mutazioni legate alla patologia nella famiglia

TEST GENETICI di ricerca Tali test hanno invece lo scopo principale è la migliore comprensione di un disordine genetico o lo sviluppo di una nuova tecnologia diagnostica

TEST CLINICI • DIRETTI: quando si conosce la mutazione causativa della malattia • INDIRETTI: si conosce solo la localizzazione del locus (sito cromosomico) sul genoma umano a. l’indagine diretta non mutazione causativa ha identificato la

I TEST DIRETTI si eseguono per diagnosticare • Malattie ereditarie: alterazioni qualitative o quantitative del materiale genetico a livello delle cellule germinali • • • trasmissione alla prole • Malattie acquisite: alterazioni qualitative o quantitative del materiale genetico a livello delle cellule somatiche non viene trasmessa alla prole

I TEST DIRETTI si eseguono per diagnosticare • Malattie ereditarie • Malattie acquisite FIBROSI CISTICA TALASSEMIA DISTROFIA DI DUCHENNE/BECKER EMOFILIA ATROFIA MUSCOLO-SPINALE MALATTIE EMOLINFOPROLIFERATIVE MALATTIE INFETTIVE Tumori

MALATTIE EREDITARIE Sono autosomiche se trasmesse dai cromosomi autosomici (dal cromosoma 1 al cromosoma 22) Sono legate al cromosoma X (x linked) se trasmesse dal cromosoma X Mutazioni dominanti: condizioni in un individuo eterozigote, in cui il l’allele mutato si esprime fenotipicamente. Mutazioni recessive: condizioni in un individuo eterozigote in cui l’allele mutato non si manifesta fenotipicamente.

I test INDIRETTI solo per diagnosticare Malattie ereditarie si conosce la localizzazione del locus (sito cromosomico) sul genoma umano Analisi dei polimorfismi del DNA • Polimorfismo genetico: variazione genetica nella popolazione caratterizzata da sostituzioni, delezioni o inserzioni di basi nel DNA • Può riguardare regioni codificanti • Quindi i polimorfismi rappresentano dei marcatori che vengono trasmessi con la lesione genetica

Analisi molecolare di polimorfismi • di sequenza (SNPs-single nucleotide polimorfism, RFLP-restriction fragment lenght polymorphism) • di lunghezza (VNTR Variable number STR-single tandem repeats) tandem repeats,

VALIDITA’ DEI TEST GENETICI La maggior parte dei test genetici non è in grado di identificare tutte le mutazioni patogenetiche dipende da: • eterogeneità genetica: quando mutazioni in loci genetici diversi possono avere lo stesso effetto fenotipico • eterogeneità allelica : quando mutazioni diverse sono a carico dello stesso locus

TEST GENETICI Validità diagnostica + al test Affetti VP - al test FN Non affetti FP VN Sensibilità del test = veri positivi/totale affetti = VP/(VP + FN) Specificità del test = veri negativi/totale sani = VN/(VN +FP)

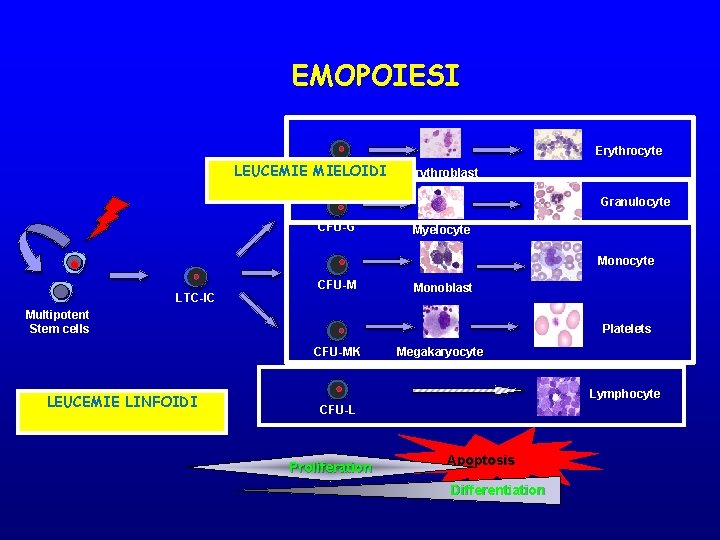
LEUCEMIE • La leucemia è una malattia che deriva dalla proliferazione neoplastica di cellule emopoietiche • Le leucemie sono suddivise in due classi principali in base alla morfologia della linea cellulare da cui originano: Leucemia mieloide – Leucemia linfoide – • Inoltre la leucemia mieloide e linfoide vengono distinte in: croniche caratterizzate da cellule neoplastiche relativamente mature e più simili alle corrispondenti cellule normali. – acute caratterizzate da cellule neoplastiche più immature –

EMOPOIESI Erythrocyte BFU-E LEUCEMIE MIELOIDI Erythroblast Granulocyte CFU-G Myelocyte Monocyte LTC-IC CFU-M Monoblast Multipotent Stem cells Platelets CFU-MK LEUCEMIE LINFOIDI Megakaryocyte Lymphocyte CFU-L Proliferation Apoptosis Differentiation

MECCANISMI DI LESIONE GENICA NEI TUMORI EMOPOIETICI Traslocazioni t(9; 22) in LMC t(15; 17) in LMA t(9; 22) in LLA t(4; 11) in LLA 11 q 23 in LLA LMA Inv(16) in LMA X Delezioni cromosomiali parziali 5 q- in LMA 6, 13, 11 q- in LLC Duplicazioni cromosomiali Trisomia 12 in LLC Mutazioni puntiformi . . AGCTCGG. . . AGTTCGG. . Attivazione RAS in LMA, LLA Internal tandem duplication Duplicazioni in tandem nel gene FLT 3

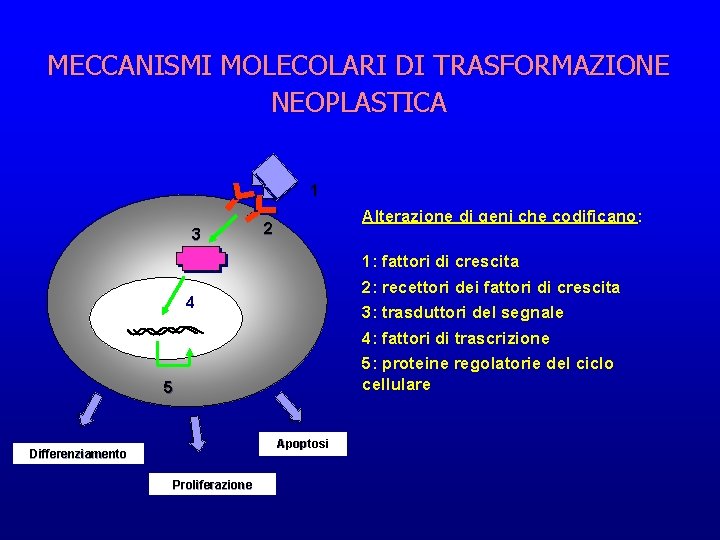
MECCANISMI MOLECOLARI DI TRASFORMAZIONE NEOPLASTICA 1 3 Alterazione di geni che codificano: 2 1: fattori di crescita 2: recettori dei fattori di crescita 3: trasduttori del segnale 4 4: fattori di trascrizione 5: proteine regolatorie del ciclo cellulare 5 Apoptosi Differenziamento Proliferazione

Leucemia Mieloide Cronica (LMC) n Incidenza : 1– 2 per 100. 000 n Mediana dell’età di insorgenza: 53 anni n Aumento di incidenza con l’età (30% dei pazienti ha più di 60 anni n Unica lesione genetica: Cromosoma philadelphia n Alla presentazione 50% diagnosticati attraverso analisi di laboratorio

Decorso Clinico: le fasi della LMC Fase Cronica Durata Mediana 5 - 6 anni Fase Avanzata Fase Accelerata Durata mediana 6– 9 mesi Crisi blastica Sopravvivenza mediana 3– 6 mesi

Cromosoma Philadelphia: Traslocazione t(9; 22)

BCR • • • Breakpoint cluster region gene Locus at 22 q 11 Codifica per la proteina bcr, di 160 -k. Da Ø proteina attivatore di GTPase (GAP)

ABL • • Abelson’s murine leukemia viral oncogene homolog Locus at 9 q 34 Ø è una prioteina con attività chinasica strettamente regolata dall’interazione sterica tra domini di SH 1 con SH 2 e SH 3 e l’ATP

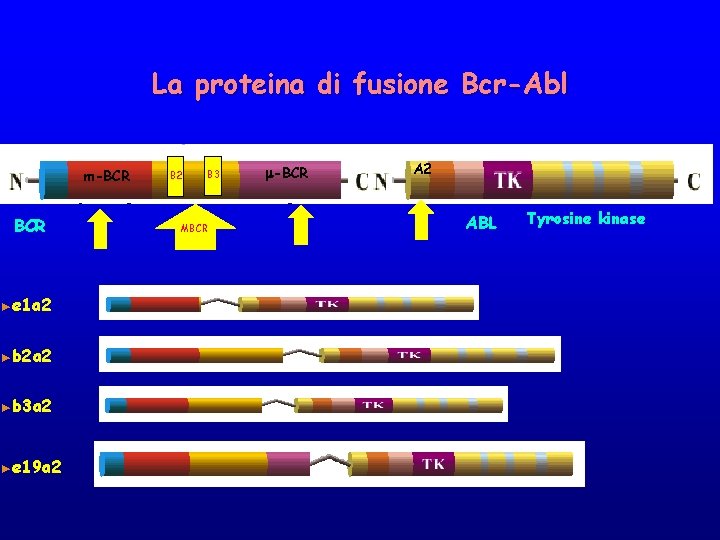
BCR-ABL • In BCR sono state identificate 3 differenti breakpoint cluster regions 1. M-bcr 2. m-bcr 3. μ –bcr m-bcr M-bcr μ-bcr

BCR-ABL Punti di rottura In ABL, i breakpoints di solito cadono in un’ampia regione che parte a monte dell’esone 1 b e finisce a valle dell’esone 1 a. • a 2 • Il contributo di ABL all’m. RNA ibrido è invariabile a causa dello splicing dell’esone 1

La proteina di fusione Bcr-Abl m-BCR ►e 1 a 2 ►b 2 a 2 ►b 3 a 2 ►e 19 a 2 B 3 MBCR m-BCR A 2 ABL Tyrosine kinase

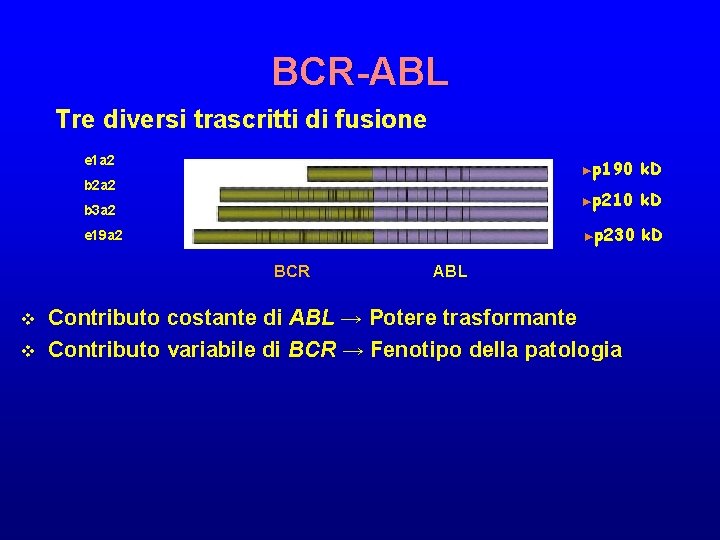
BCR-ABL Tre diversi trascritti di fusione e 1 a 2 b 2 a 2 b 3 a 2 e 19 a 2 BCR v v ►p 190 k. D ►p 210 k. D ►p 230 k. D ABL Contributo costante di ABL → Potere trasformante Contributo variabile di BCR → Fenotipo della patologia

BCR-ABL • BCR incrementa l’attività kinasica di ABL Incremento della proliferazione – Riduzione dell’apoptosi – Riduzione della adesione delle cellule allo stroma midollare –

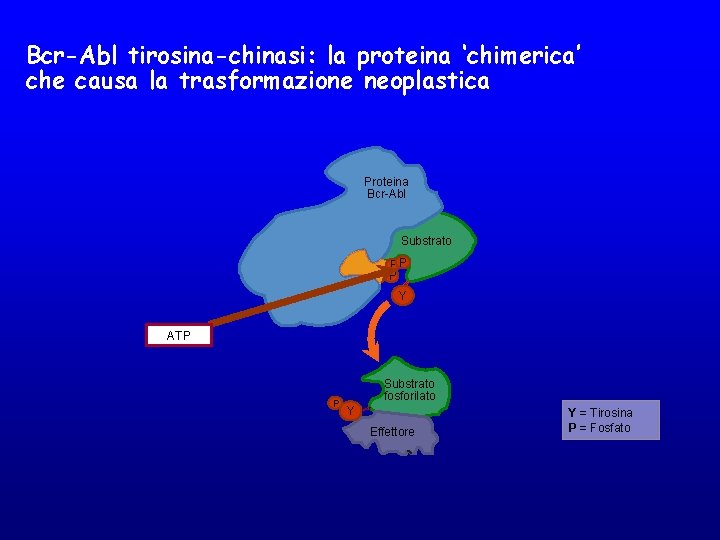
Bcr-Abl tirosina-chinasi: la proteina ‘chimerica’ che causa la trasformazione neoplastica Proteina Bcr-Abl Substrato PP P Y ATP P Substrato fosforilato Y Effettore Y = Tirosina P = Fosfato

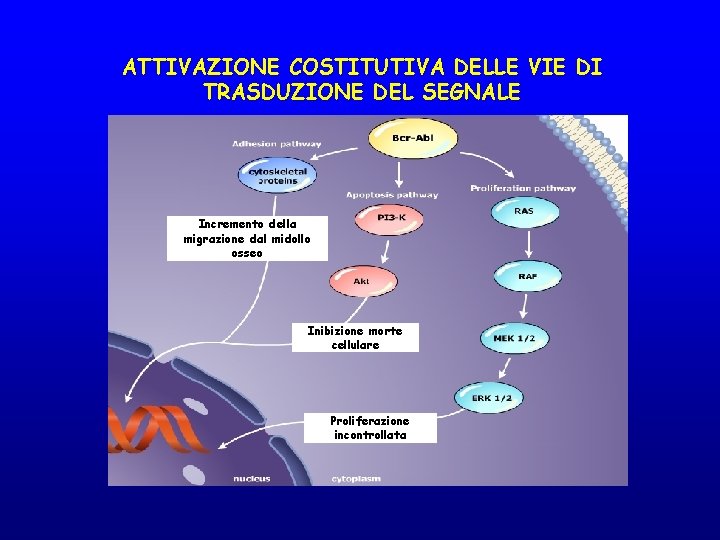
ATTIVAZIONE COSTITUTIVA DELLE VIE DI TRASDUZIONE DEL SEGNALE Incremento della migrazione dal midollo osseo Inibizione morte cellulare Proliferazione incontrollata

APPROCCIO DIAGNOSTICO ALLA LEUCEMIA MIELOIDE CRONICA ESEMPIO: PAZIENTE CON SOSPETTA LEUCEMIA MIELOIDE CRONICA INDAGINI DI I°LIVELLO

LMC in fase cronica: presentazione clinica n Asintomatica nel 50% dei casi n Sintomatologia clinica • Astenia • Febbricola • Perdita di peso/anoressia n Obiettività clinica Splenomegalia (50% dei casi)

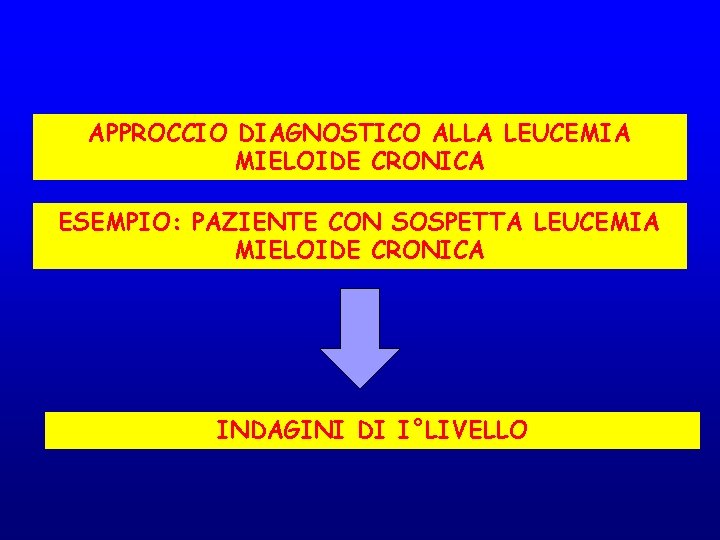
DIAGNOSI DI LMC: ESAMI DI LABORATORIO Sangue periferico: Emocromo FASI CRONICA LEUCOCITI (granulociti) BLASTI Valori di riferimento 4. 500 a 11. 000 per µL. BLASTICA ACCELERATA 20000/µL 0 PIASTRINE Sangue periferico normale >10% 30% O NORMALI Sangue periferico LMC Puntato midollare

Richiesta esami Tipizzazione genetica leucemia mieloide cronica traslocazione t(9; 22)

INDAGINI DI II LIVELLO • Citogenetica Convenzionale • Citogenetica Molecolare – Fluorescence in-situ hybridization (FISH) • Genetica Molecolare – Diagnostica basata su PCR Qualitativa – Monitoraggio basato su PCR Quantitativa

CITOGENETICA CONVENZIONALE La citogenetica convenzionale è una tecnica che permette lo studio del numero e della struttura dei cromosomi (studio del cariotipo) I cromosomi vengono esaminati “bloccati” in metafase. Step: • prelievo del campione (sangue midollare) • allestimento delle colture cellulari • bandeggio dei cromosomi

CITOGENETICA

Citogenetica convenzionale Limiti • • Riarrangiamenti cromosomici complessi Cattiva morfologia cromosomica Basso indice mitotico delle cellule neoplastiche Bassa crescita o fallimento della coltura

INDAGINI DI II LIVELLO • Citogenetica Convenzionale • Citogenetica Molecolare – Fluorescence in-situ hybridization (FISH) • Genetica Molecolare – Diagnostica basata su PCR Qualitativa – Monitoraggio basato su PCR Quantitativa

CITOGENETICA MOLECOLARE (FISH) La Fluorescent In Situ Hybridization (FISH) è una tecnologia che utilizza sonde nucleotidiche marcate (DNA probes) per identificare specifiche regioni di un cromosoma ovvero determinate sequenze del DNA. Step: • denaturazione del DNA • incubazione sonda + DNA denaturato (“annealing”) • rilevazione del segnale della sonda con microscopio a fluorescenza

FISH (fluorescence in situ hybridization)

INDAGINI DI II LIVELLO • Citogenetica Convenzionale • Citogenetica Molecolare – Fluorescence in-situ hybridization (FISH) • Genetica Molecolare – Diagnostica basata su PCR Qualitativa – Monitoraggio basato su PCR Quantitativa

La proteina di fusione Bcr-Abl m-BCR B 2 B 3 MBCR m-BCR A 2 ABL Tyrosine kinase ►e 1 a 2 ►b 2 a 2 ►b 3 a 2 ►e 19 a 2 p 210

METODOLOGIE D’INDAGINE: LA PCR BCR ABL

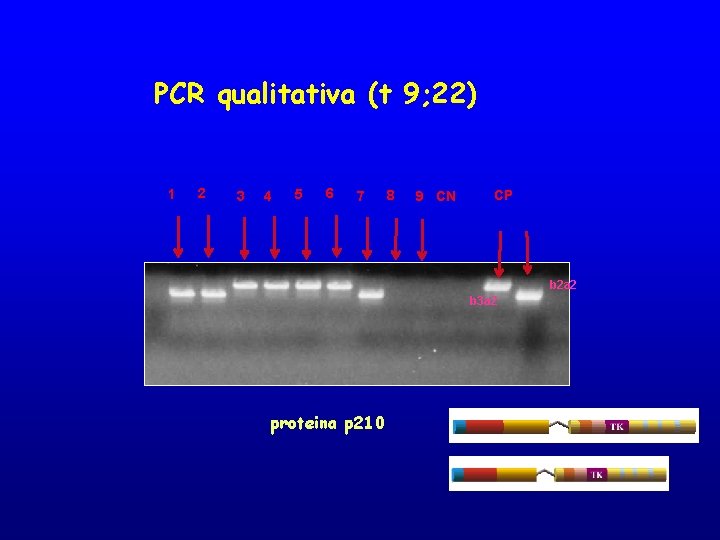
PCR qualitativa (t 9; 22)

PCR qualitativa (t 9; 22) 1 2 3 4 5 6 7 8 9 CN CP b 2 a 2 b 3 a 2 proteina p 210

VARIABILI PRE-ANALITICHE VOLUME SANGUE: DX: 10 ML in EDTA SANGUE PERIFERICO F. UP: 20 m. L in EDTA SANGUE PERIFERICO TEMPI DI CONSEGNA: in giornata/max entro le 24 ore

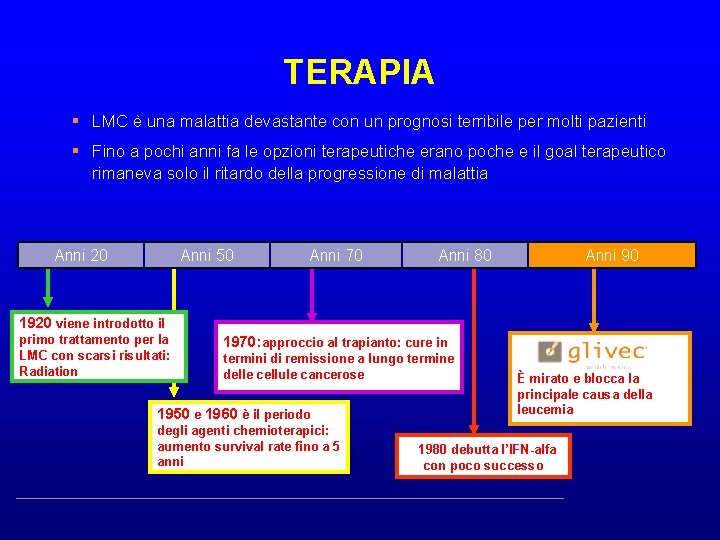
TERAPIA § LMC è una malattia devastante con un prognosi terribile per molti pazienti § Fino a pochi anni fa le opzioni terapeutiche erano poche e il goal terapeutico rimaneva solo il ritardo della progressione di malattia Anni 20 Anni 50 Anni 70 Anni 80 Anni 90 1920 viene introdotto il primo trattamento per la LMC con scarsi risultati: Radiation 1970: approccio al trapianto: cure in termini di remissione a lungo termine delle cellule cancerose 1950 e 1960 è il periodo degli agenti chemioterapici: aumento survival rate fino a 5 anni È mirato e blocca la principale causa della leucemia 11980 debutta l’IFN-alfa con poco successo

Monitoraggio della risposta Risposta ematologica • Conta completa delle cellule ematiche Risposta citogenetica molecolare • Cariotipo • • Ibridizzazione in-situ fluorescente (FISH) • Reazione a catena della polimerasi quantitativa in tempo reale (RQ-PCR) (RT-PCR) qualitativa

Terapia • • • Convenzionale: Interferone Trapianto allogenico di midollo Trapianto autologo di midollo Terapia molecolare STI-571 (Gleevec)

Interferone • • Citochina con effetto antiproliferativo Remissione ematologica in 70 -80% Risposta citogenetica in 40 -60% : Effetti collaterali

Terapia • • Convenzionale: Interferone Trapianto allogenico di midollo Terapia molecolare STI-571 (Gleevec)

Trapianto di midollo osseo – – – Unica terapia curativa Per pazienti in fase cronica Guarigione totale in > 60% entro 1 -2 anni dalla diagnosi per i migliori risultati Compatibilità sistema HLA

Terapia • Convenzionale: – • • • Idrossiurea Interferone Trapianto allogenico di midollo Terapia molecolare STI-571 (Gleevec)

STI-571 • • Signal Transduction Inhibitor STI-571 blocca il legame dell’ATP a BCR-ABL inibendone l’attività kinasica

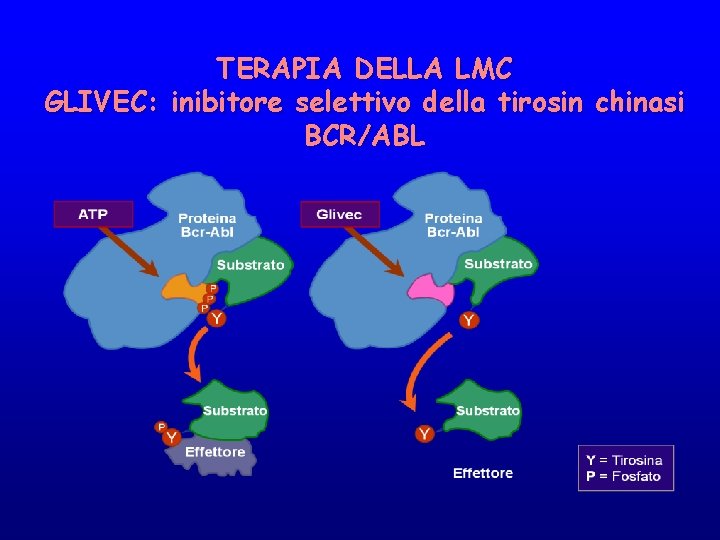
TERAPIA DELLA LMC GLIVEC: inibitore selettivo della tirosin chinasi BCR/ABL

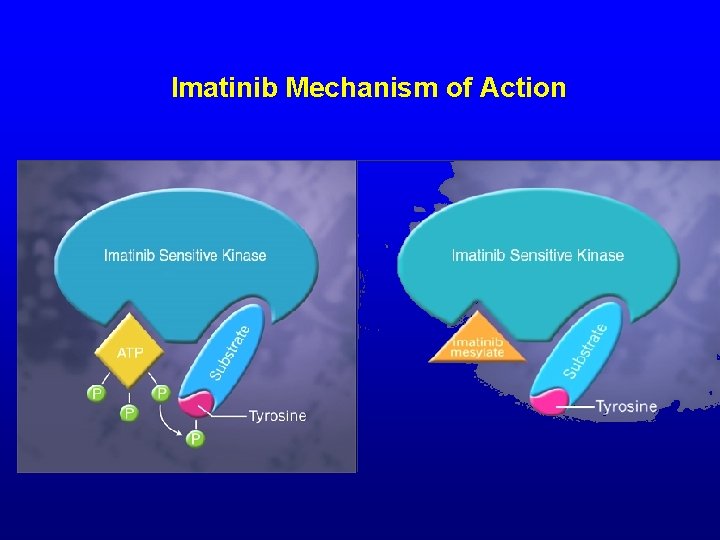
Imatinib Mechanism of Action

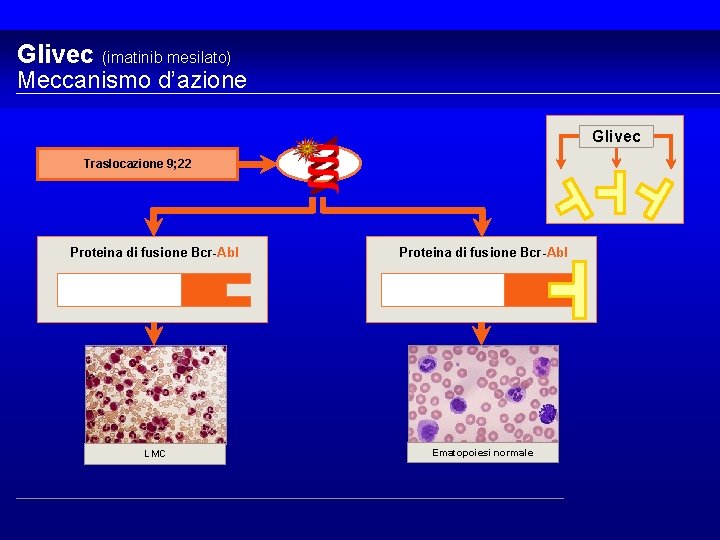
Glivec (imatinib mesilato) Meccanismo d’azione Glivec Traslocazione 9; 22 Proteina di fusione Bcr-Abl LMC Ematopoiesi normale

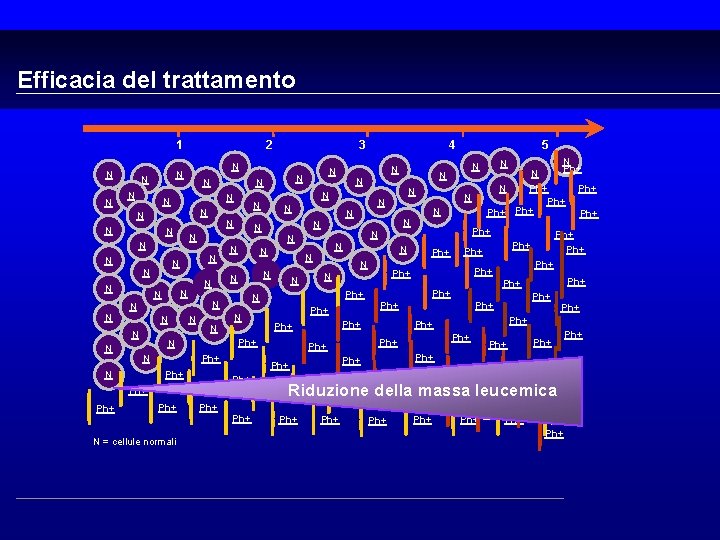
Efficacia del trattamento 1 N N 2 N N N N Ph+ N = cellule normali Ph+ Ph+ Ph+ Ph+ Ph+ Ph+ Ph+ Ph+ Ph+ N Ph+ Ph+ N N Ph+ Ph+ N N Ph+ N N N N N N N N N 5 N N 4 N N N 3 Ph+ Ph+ Ph+ Riduzione della massa leucemica Ph+ Ph+ Ph+ Ph+

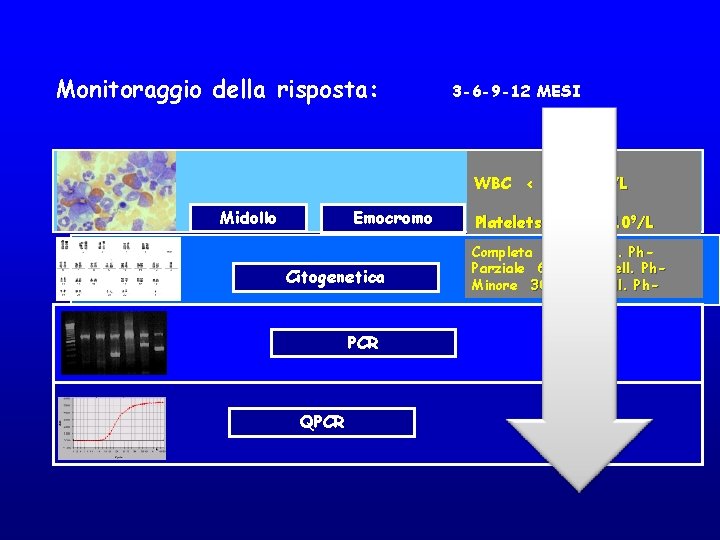
Monitoraggio della risposta: 3 -6 -9 -12 MESI WBC < 10 x 109/L Midollo Emocromo Citogenetica PCR QPCR Platelets <450 x 109/L Completa 100% cell. Ph. Parziale 60%– 90% cell. Ph. Minore 30%– 60% cell. Ph-

Monitoraggio: necessità di quantificare Real Time PCR: applicazioni : - la quantificazione dei livelli di un determinato m. RNA - Efficacia della terapia farmacologica -Valutazione della malattia minima residua

M R D ( Mi n i m a l R e s i d u a l D i s e a s e ) : Quota residua di cellule neoplastiche non eradicate dalla terapia farmacologica ad esempio delle leucemie Tali cellule neoplastiche, presenti ad un livello inferiore alla capacità di rilevazione delle metodiche convenzionali, sono in grado di espandersi e dare o r i g i n e a l l a r e c i d i v a

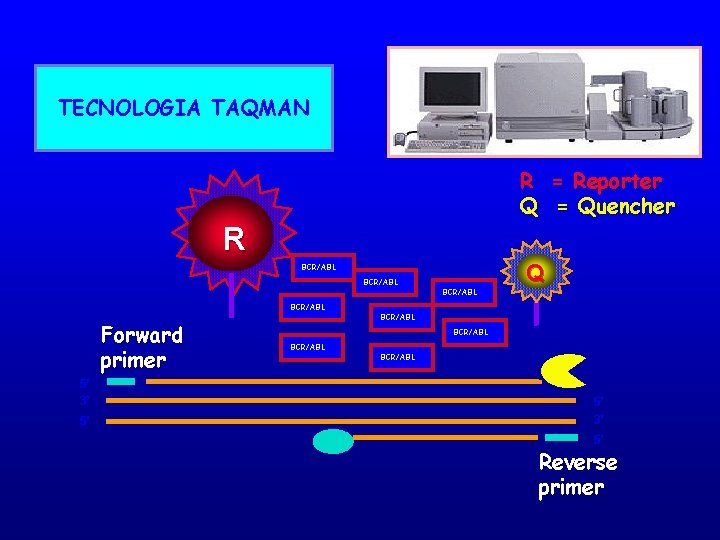
TECNOLOGIA TAQMAN • Sonda oligonucleotidica complementare alla sequenza target, interna ai primers di PCR • La sonda è marcata con un fluoroforo ad una estremità ed un quencher all’altra La distanza tra fluoroforo e quencher è tale da bloccare l’emissione di fluorescenza.

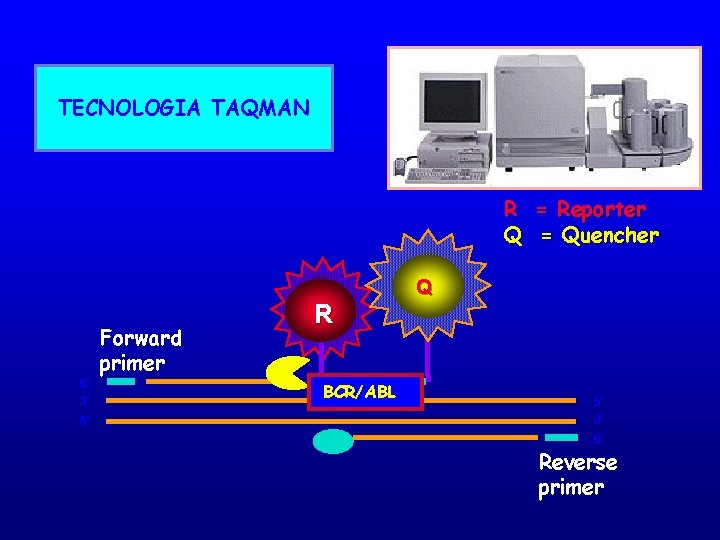
TECNOLOGIA TAQMAN R = Reporter Q = Quencher 5’ 3’ 5’ Forward primer R BCR/ABL Q Q 5’ 3’ 5’ Reverse primer

TECNOLOGIA TAQMAN R = Reporter Q = Quencher R Q Q BCR/ABLL 5’ 3’ 5’ Forward primer BCR/ABLL 5’ 3’ 5’ Reverse primer

TECNOLOGIA TAQMAN R = Reporter Q = Quencher R BCR/ABLL Forward primer 5’ 3’ 5’ BCR/ABLL Q Q BCR/ABLL 5’ 3’ 5’ Reverse primer

Principi della Real-Time • LO STRUMENTO CALCOLA IL PRIMO INCREMENTO SIGNIFICATIVO DELLA QUANTITÀ DI FLUORESCENZA EMESSA DAL PRODOTTO PCR (CT -THRESHOLD CYCLE) PCR QUALITATIVA Ciclo-Threshold (CT ) Fluorescenza di Background

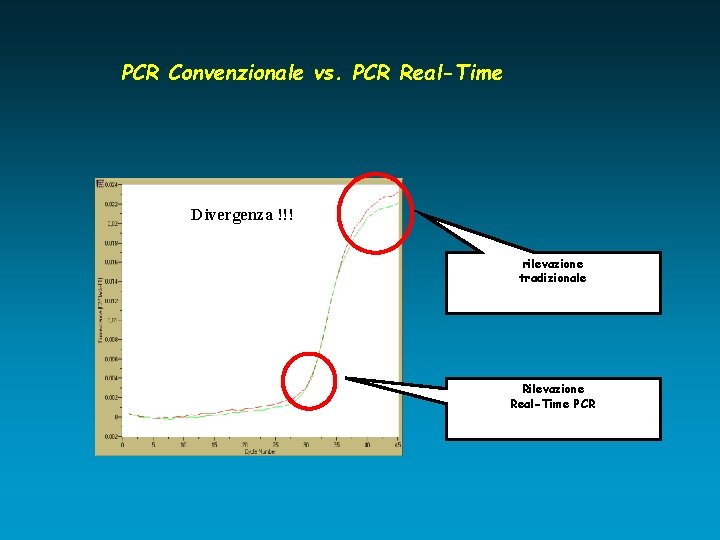
Principio base della Real-Time PCR Si determina QUANDO viene generato il segnale piuttosto che l‘intensità del segnale stesso (analisi all‘end –point) rilevazione tradizionale Sample Ct threshold Ct = threshold cycle: Il numero di cicli al quale il prodotto di PCR supera la soglia di rilevabilità

PCR Convenzionale vs. PCR Real-Time Divergenza !!! rilevazione tradizionale Rilevazione Real-Time PCR

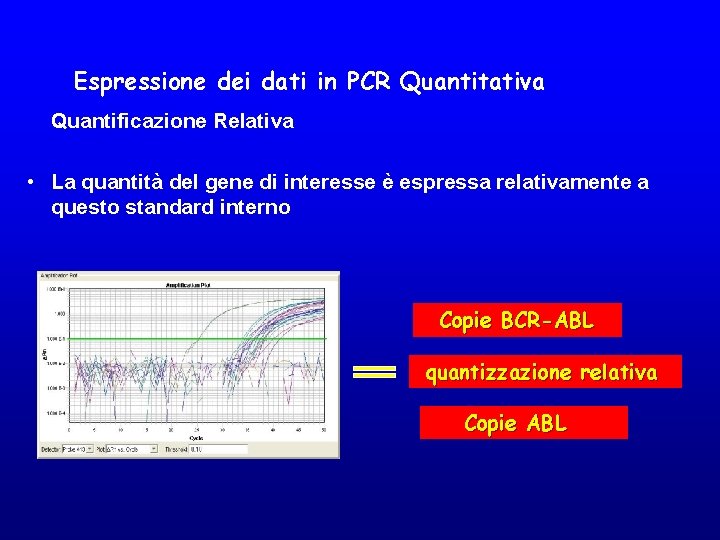
Espressione dei dati in PCR Quantitativa Quantificazione Relativa • La quantità del gene di interesse è espressa relativamente a questo standard interno Copie BCR-ABL quantizzazione relativa Copie ABL

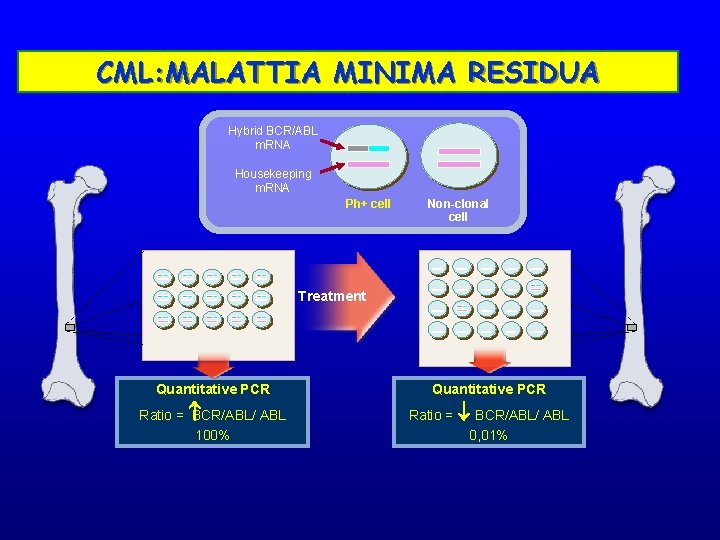
CML: MALATTIA MINIMA RESIDUA Hybrid BCR/ABL m. RNA Housekeeping m. RNA Ph+ cell Non-clonal cell Treatment Quantitative PCR Ratio = BCR/ABL/ ABL 100% Ratio = ¯ BCR/ABL/ ABL 0, 01%

Step analitici del monitoraggio molecolare Peripheral Blood in EDTA Buffy coat within 24 hrs Extract RNA from total leukocytes Reverse transcribe to c. DNA RQ-PCR Ratio BCR-ABL/ABL %

Metodica Buffy-coat Sangue Periferico Il Buffy-coat è costituito dai componenti del sangue ottenuti mediante centrifugazione di un’unità di sangue intero e contenente una notevole quantità di leucociti e piastrine.

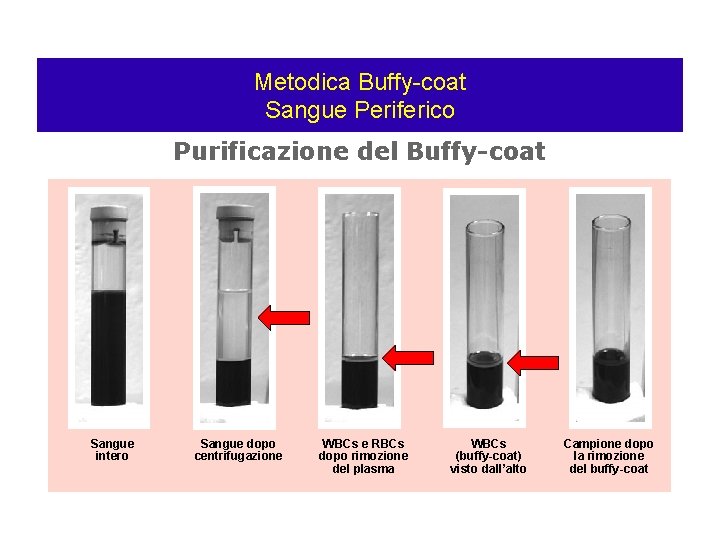
Metodica Buffy-coat Sangue Periferico Purificazione del Buffy-coat Sangue intero Sangue dopo centrifugazione WBCs e RBCs dopo rimozione del plasma WBCs (buffy-coat) visto dall’alto Campione dopo la rimozione del buffy-coat

Quale volume di sangue periferico deve essere prelevato per poter effettuare correttamente l’analisi di RT-PCR e/o Q-PCR? 10 ml alla diagnosi e 20 ml nel f. up Aggiornamento SOP 10 -20 ml • Baccarani M et al. Blood 2006 • Hughes et al. , Blood 2006 • Foroni L. e al. , British Journal of Haematology 2011

– Trasportare i campioni a Temperatura Ambiente (15 -25°C) entro 24 h – Conservare i campioni a Temperatura Ambiente o in alternativa a 4°C per un periodo non superiore alle 48 h – Tali condizioni consentono di preservare la vitalità cellulare e quindi l’integrità degli acidi nucleici – entro 24 h (20%degradazione in 24 h; 50% in 48 h) (Hughes et al, Blood 2006)

Update SOPs 2014

10 1000000 Slope: (-3, 2; -3, 6) Y-intercetta: 41 -42 ± 1 R 2 > 0, 98 Intercetta: limite teorico di determinazione della reazione, o il valore di CT aspettato per il più basso numero di copie del templato bersaglio ad essere amplificato, determinato sull’asse delle X. Slope: indica l’efficienza della reazione ovvero esprime la differenza di Ct che determina il raddoppio di un log del templato


 Ansa sentinella pancreatite
Ansa sentinella pancreatite Liceo scientifico alatri
Liceo scientifico alatri Cromatidio
Cromatidio Nomi derivati con pane
Nomi derivati con pane Malattie del vino
Malattie del vino Mutazioni geniche
Mutazioni geniche Mutazioni puntiformi
Mutazioni puntiformi Mutazioni puntiformi
Mutazioni puntiformi Mutazione missenso
Mutazione missenso Allopoliploidia
Allopoliploidia Poliploidia
Poliploidia Mutazioni
Mutazioni Paradosso di sherman
Paradosso di sherman Malattie cardiovascolari
Malattie cardiovascolari Malattie epifitiche
Malattie epifitiche Malattie
Malattie Malattie dismielinizzanti
Malattie dismielinizzanti Malattie croniche
Malattie croniche Malattie croniche
Malattie croniche Malattie multifattoriali esempi
Malattie multifattoriali esempi Malattie epifitiche
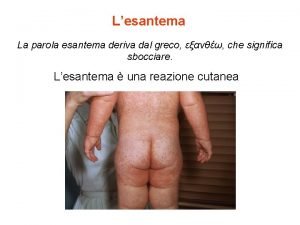
Malattie epifitiche Esantema etimologia
Esantema etimologia Kousmine malattie autoimmuni
Kousmine malattie autoimmuni Patrimonio cultural de la zona sur de chile
Patrimonio cultural de la zona sur de chile Zona norte cultura
Zona norte cultura Oic 17 metodo del patrimonio netto
Oic 17 metodo del patrimonio netto Cuál es el cuál es
Cuál es el cuál es Características del patrimonio natural
Características del patrimonio natural Patrimonio
Patrimonio Que es patrimonio territorial de nicaragua
Que es patrimonio territorial de nicaragua Estado de cambios en el patrimonio neto
Estado de cambios en el patrimonio neto Caracteristicas del patrimonio
Caracteristicas del patrimonio Patrimonio bruto
Patrimonio bruto Niif para pymes seccion 22
Niif para pymes seccion 22 Patrimonio netto isee
Patrimonio netto isee Patrimonio comunitario
Patrimonio comunitario Patrimonio bruto
Patrimonio bruto Podział rzeczy
Podział rzeczy Patrimonio cultural zona austral de chile
Patrimonio cultural zona austral de chile Patrimonio cultural tangible chile
Patrimonio cultural tangible chile Patrimonio netto
Patrimonio netto Cruzadinha sobre patrimonio material e imaterial
Cruzadinha sobre patrimonio material e imaterial Patrimonio bruto
Patrimonio bruto Coordinacion general de patrimonio
Coordinacion general de patrimonio Marco fiscal de una empresa ejemplo
Marco fiscal de una empresa ejemplo Tom werner patrimonio
Tom werner patrimonio Imagenes de patrimonio neto
Imagenes de patrimonio neto Egovia
Egovia Oic patrimonio netto
Oic patrimonio netto Manager itinerari culturali
Manager itinerari culturali Morad patrimonio
Morad patrimonio Imagenes de patrimonio neto
Imagenes de patrimonio neto Project management pilastri
Project management pilastri Alejandra rios patrimonio
Alejandra rios patrimonio Clases de bienes
Clases de bienes Patrimonio natural argentina
Patrimonio natural argentina Una cazuela es patrimonio natural o cultural
Una cazuela es patrimonio natural o cultural Zona norte 2
Zona norte 2 T
T Patrimonio netto
Patrimonio netto Patrimonio netto contabile
Patrimonio netto contabile Capitale circolante negativo
Capitale circolante negativo Ejemplos de patrimonio familiar
Ejemplos de patrimonio familiar El patrimonio empresarial
El patrimonio empresarial Patrimonio de la humanidad en chile
Patrimonio de la humanidad en chile Yo soy del sur ecos del rocio
Yo soy del sur ecos del rocio Diferencias entre la via aerea del adulto y del niño
Diferencias entre la via aerea del adulto y del niño Triangulo de calor
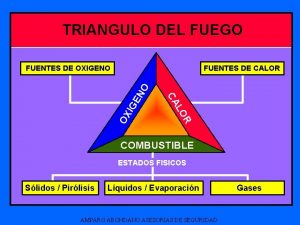
Triangulo de calor Triángulo del fuego
Triángulo del fuego Teatro absurdo caracteristicas
Teatro absurdo caracteristicas Preguntas del tacto
Preguntas del tacto Los colores dela bandera del sena son
Los colores dela bandera del sena son Enlace o relacionante ejemplos
Enlace o relacionante ejemplos Señor abre mis labios en el rosario
Señor abre mis labios en el rosario Sanidad interior: espíritu, alma y cuerpo
Sanidad interior: espíritu, alma y cuerpo Programa usado
Programa usado Pacto de matrimonio en la biblia
Pacto de matrimonio en la biblia Andalucía es mi tierra yo soy del sur
Andalucía es mi tierra yo soy del sur Normas del buen hablante y del buen oyente para niños
Normas del buen hablante y del buen oyente para niños Imagenes del sexting
Imagenes del sexting Organizador grafico del plato del buen comer
Organizador grafico del plato del buen comer Poema caracteristicas
Poema caracteristicas Ejemplo de planteamiento del problema cualitativo
Ejemplo de planteamiento del problema cualitativo Personajes la dama del alba
Personajes la dama del alba Seno transverso cerebro
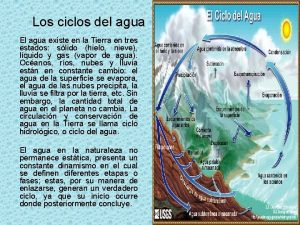
Seno transverso cerebro Objetivos del ciclo del agua
Objetivos del ciclo del agua Símbolos corporativos del sena
Símbolos corporativos del sena Pronador largo
Pronador largo Medida de valores del dinero
Medida de valores del dinero Etapas del ciclo del agua
Etapas del ciclo del agua Linea de tiempo del deporte
Linea de tiempo del deporte Equazione primo e terzo quadrante
Equazione primo e terzo quadrante Kate del castillo kate trillo del castillo
Kate del castillo kate trillo del castillo Edad del agua y del viento inventos
Edad del agua y del viento inventos El canon del nuevo testamento
El canon del nuevo testamento Prospetto reddito e capitale
Prospetto reddito e capitale Organizador grafico del plato del buen comer
Organizador grafico del plato del buen comer Protozoo flagelados
Protozoo flagelados Elementos del sacramento del matrimonio
Elementos del sacramento del matrimonio Poesia al maestro corta
Poesia al maestro corta Tema del cantar del mío cid
Tema del cantar del mío cid El lavacro de bronce
El lavacro de bronce Colorea los círculos de las respuestas correctas
Colorea los círculos de las respuestas correctas Ideazione anancastica
Ideazione anancastica Dimensiones del desarrollo del niño
Dimensiones del desarrollo del niño Estrategias del ciclo de vida del producto ejemplos
Estrategias del ciclo de vida del producto ejemplos Decreto 3222 de 2002 redes de apoyo
Decreto 3222 de 2002 redes de apoyo Area del tronco del cono
Area del tronco del cono Fases del ciclo del nitrógeno
Fases del ciclo del nitrógeno Conde de barcelona mio cid
Conde de barcelona mio cid Cual fue el pecado de salomon
Cual fue el pecado de salomon Resumen de un libro de amor
Resumen de un libro de amor Baloncesto reglas
Baloncesto reglas Definir el alcance de un proyecto
Definir el alcance de un proyecto Ejemplo de tipo de habla asertivo
Ejemplo de tipo de habla asertivo 4 mandamiento significado
4 mandamiento significado Teoría del cargo y del abono
Teoría del cargo y del abono Modelo de comportamiento del consumidor
Modelo de comportamiento del consumidor Placas continentales oceanicas y mixtas
Placas continentales oceanicas y mixtas Solo e pensoso
Solo e pensoso Reyneri introduzione alla sociologia del mercato del lavoro
Reyneri introduzione alla sociologia del mercato del lavoro Romance del rey rodrigo
Romance del rey rodrigo Articulo 184 codigo del trabajo
Articulo 184 codigo del trabajo Grupos del plato del buen comer
Grupos del plato del buen comer