Correlato neural despertar un estudio de resonancia magntica




































- Slides: 74

Correlato neural despertar: un estudio de resonancia magnética funcional ALCAIDE, Santiago Agustín - ROMANO, Alvaro Agustín DIRECTORES: BARTTFELD, PABLO MALDONADO, ANA CAROLINA N OVIEMBRE , 2018


¿Motivación? Clínica Científica

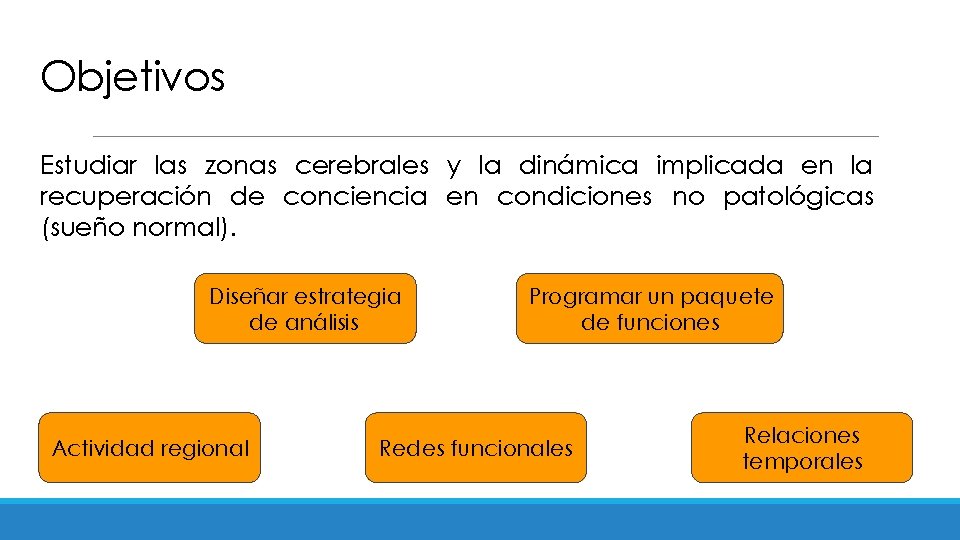
Objetivos Estudiar las zonas cerebrales y la dinámica implicada en la recuperación de conciencia en condiciones no patológicas (sueño normal). Diseñar estrategia de análisis Actividad regional Programar un paquete de funciones Redes funcionales Relaciones temporales

Preguntas ¿Actividad regional? ¿Conectividad? ¿Temporalidad?

¿Qué datos se usan? f(intensidad)

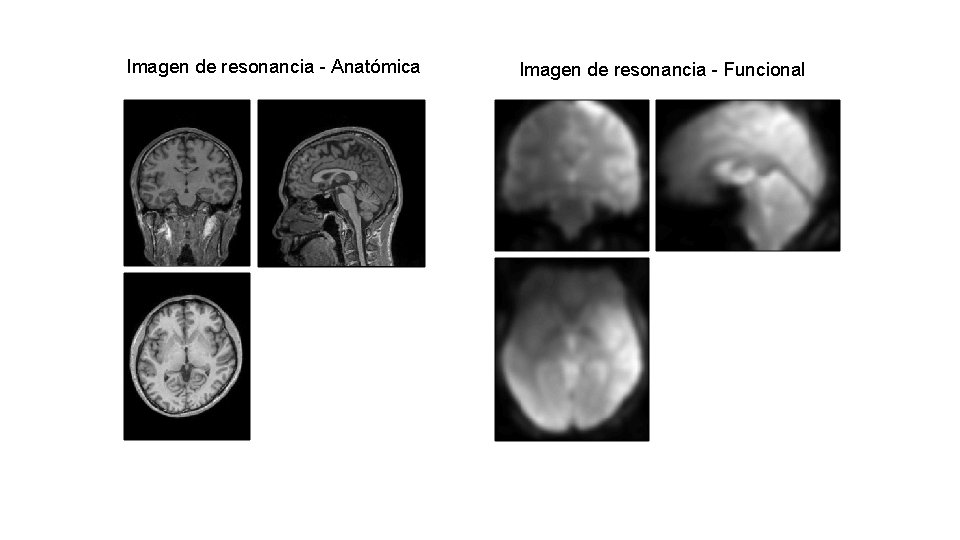
Imagen de resonancia - Anatómica Imagen de resonancia - Funcional

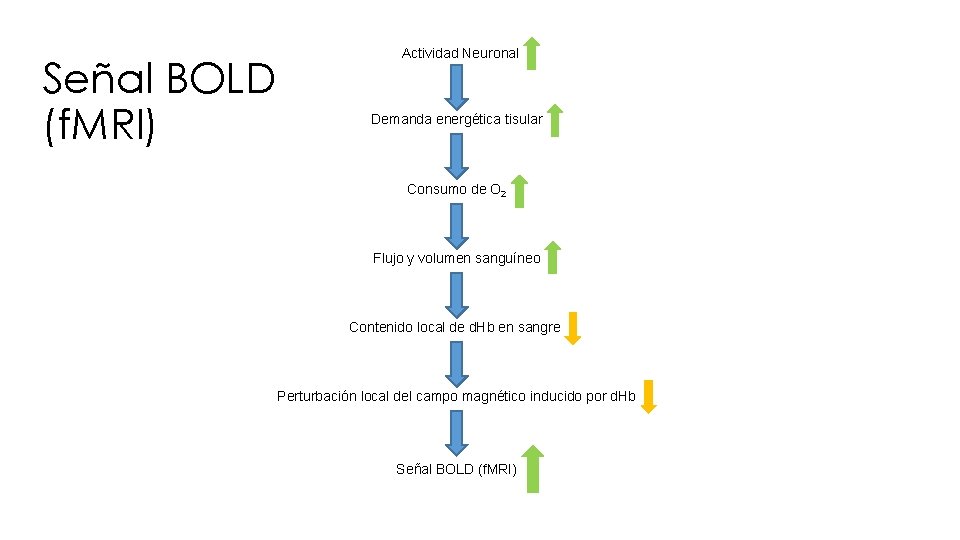
Señal BOLD (f. MRI) Actividad Neuronal Demanda energética tisular Consumo de O 2 Flujo y volumen sanguíneo Contenido local de d. Hb en sangre Perturbación local del campo magnético inducido por d. Hb Señal BOLD (f. MRI)

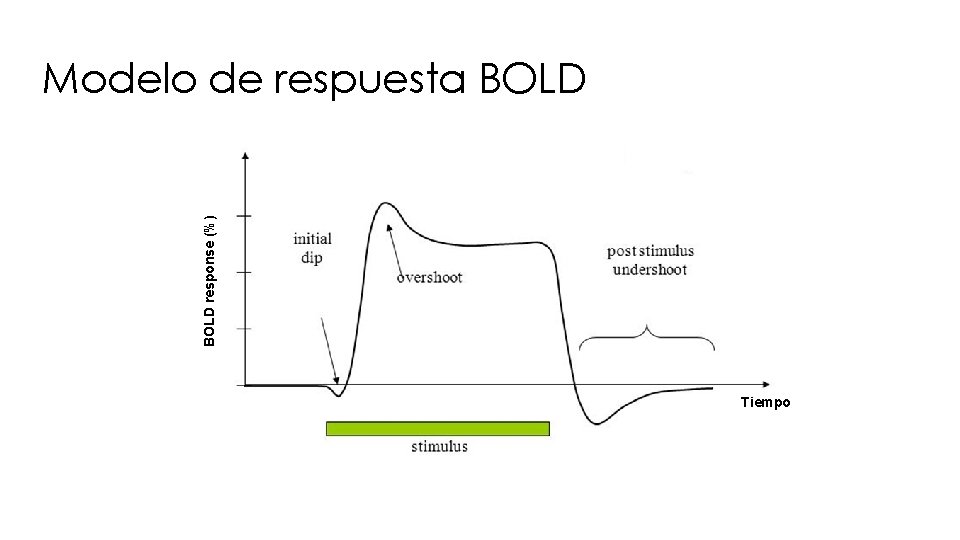
BOLD response (%) Modelo de respuesta BOLD Tiempo

Datos Laboratorio Kamitani (Kioto, Japón)

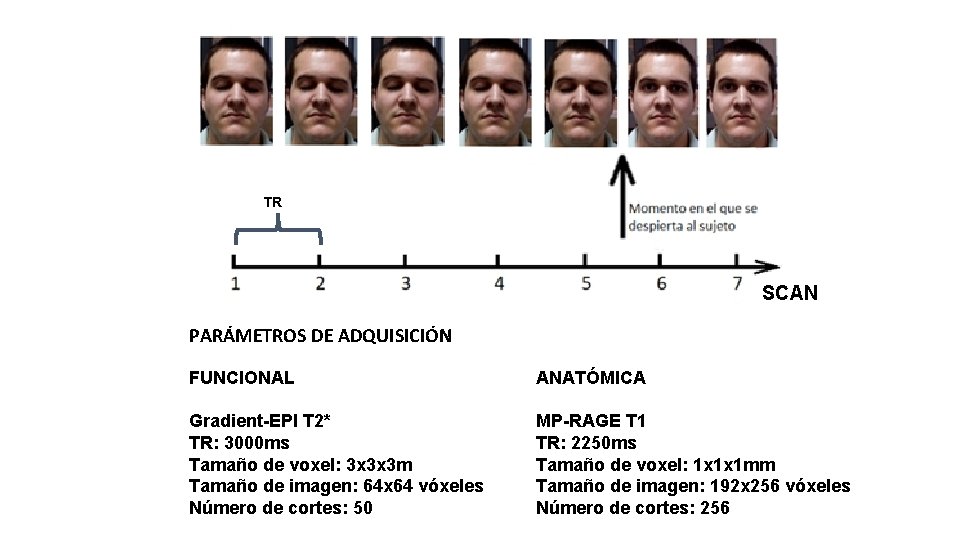
TR SCAN PARÁMETROS DE ADQUISICIÓN FUNCIONAL ANATÓMICA Gradient-EPI T 2* TR: 3000 ms Tamaño de voxel: 3 x 3 x 3 m Tamaño de imagen: 64 x 64 vóxeles Número de cortes: 50 MP-RAGE T 1 TR: 2250 ms Tamaño de voxel: 1 x 1 x 1 mm Tamaño de imagen: 192 x 256 vóxeles Número de cortes: 256

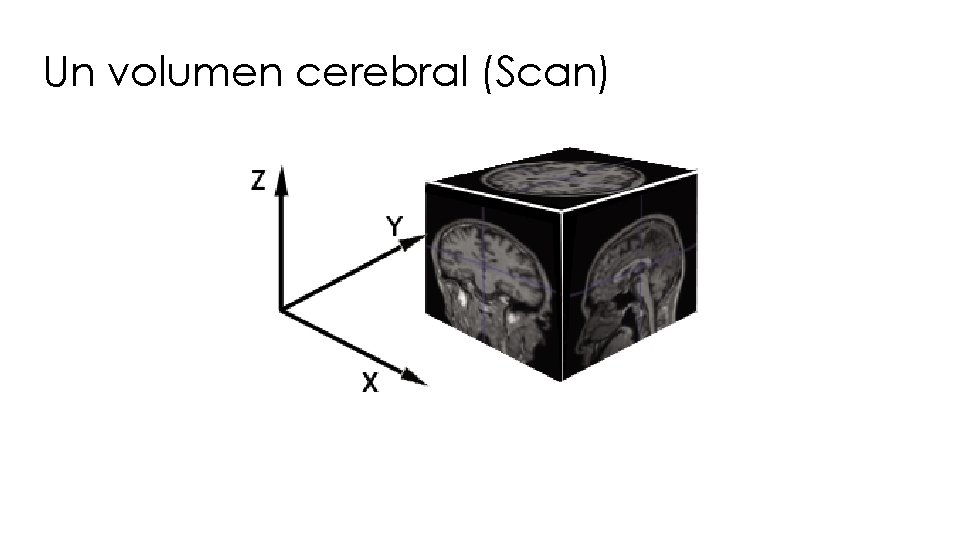
Un volumen cerebral (Scan)

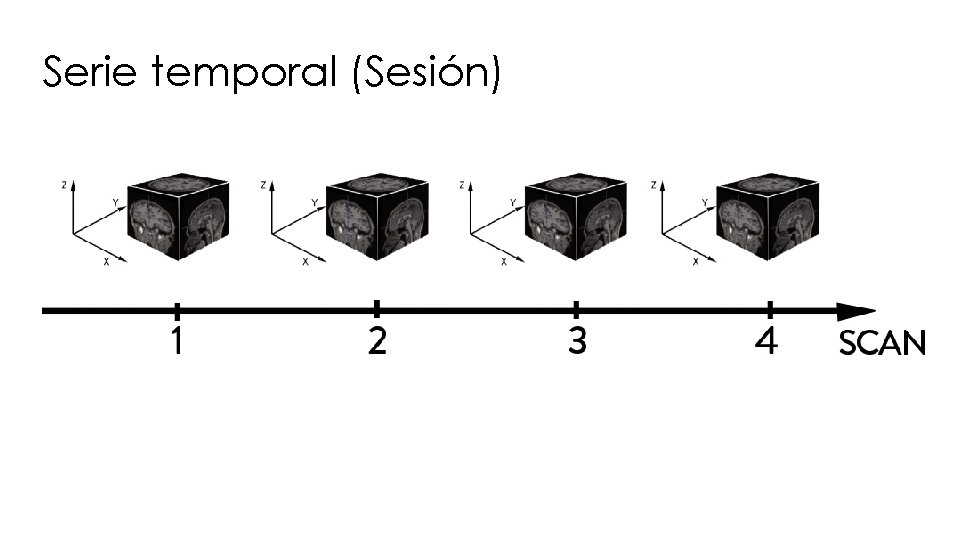
Serie temporal (Sesión)

Sesiones de un mismo sujeto

Todos los sujetos

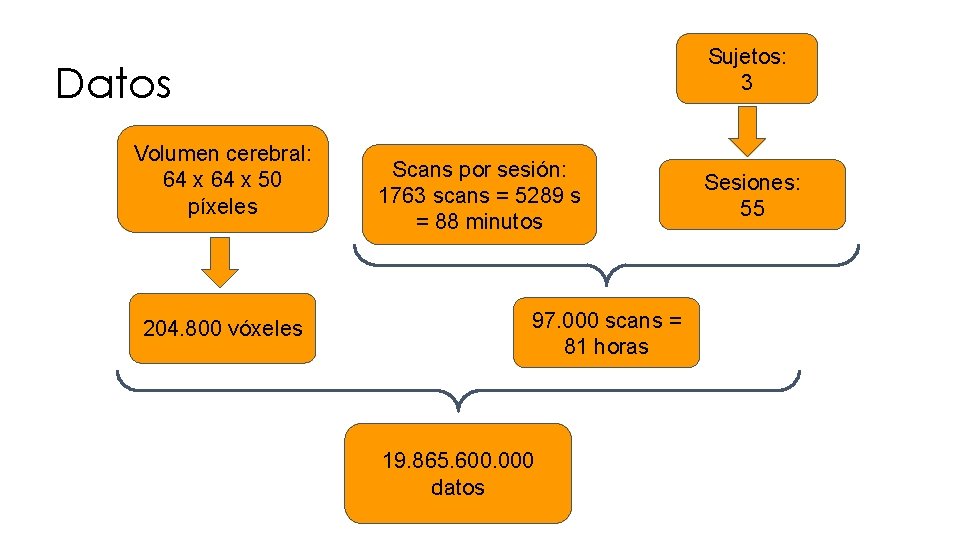
Sujetos: 3 Datos Volumen cerebral: 64 x 50 píxeles 204. 800 vóxeles Scans por sesión: 1763 scans = 5289 s = 88 minutos 97. 000 scans = 81 horas 19. 865. 600. 000 datos Sesiones: 55

Diseño experimental


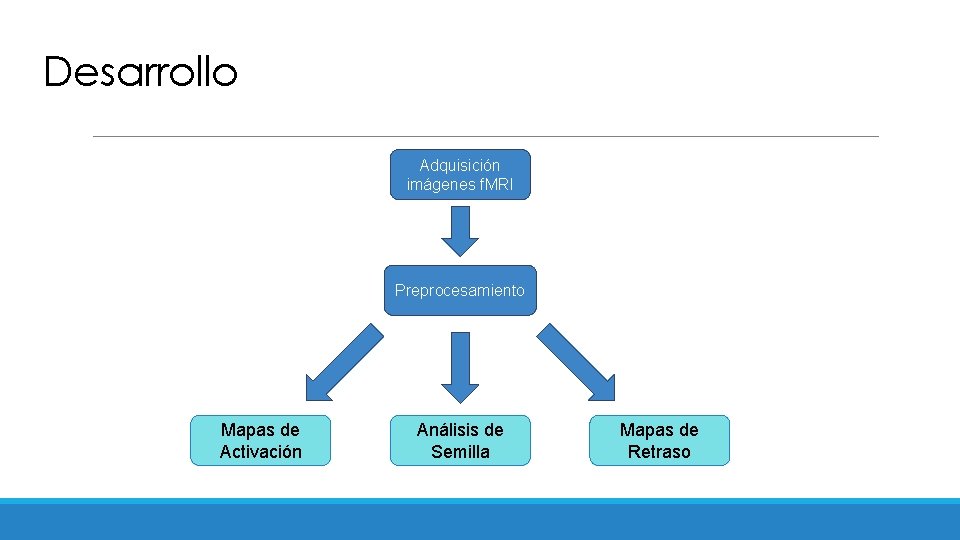
Desarrollo Adquisición imágenes f. MRI Preprocesamiento Mapas de Activación Análisis de Semilla Mapas de Retraso

Preprocesamiento

Etapas Adquisición de imágenes (formato HDF 5) Conversión a formato usado por SPM (NIf. TI) Sincronización temporal de cortes Realineado Suavizado de imágenes funcionales Normalización de imágenes estructurales Corregistro a imágenes estructurales Mapas de Activación Subsampleo Eliminación de componente LCR Mapas de Retrasos Filtro pasa banda (0. 01 – 0. 1 Hz) Análisis de Semilla y Matrices de Conectividad

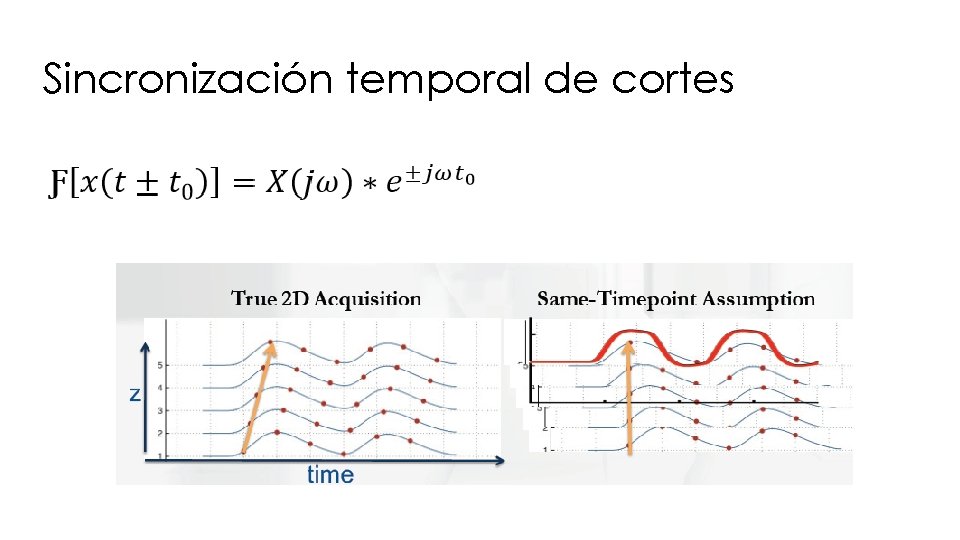
Sincronización temporal de cortes

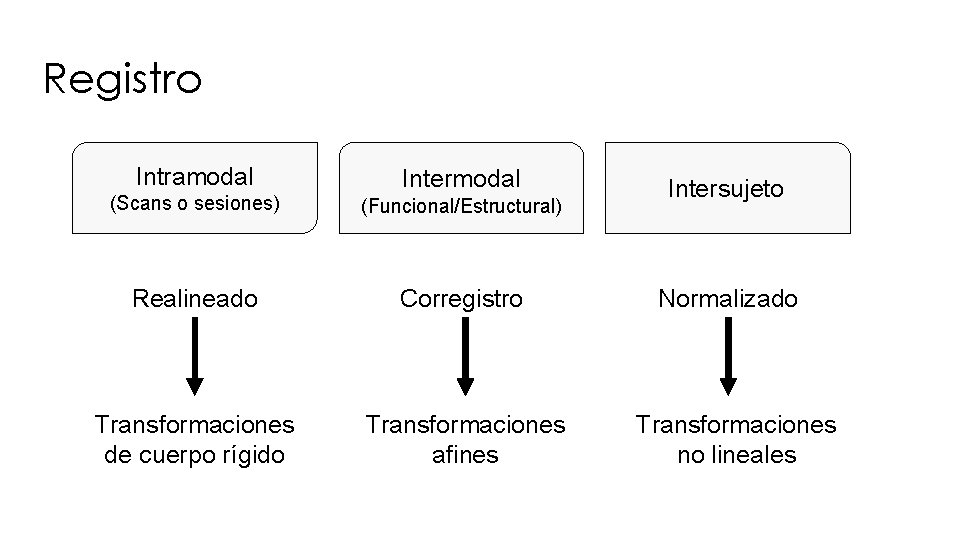
Registro Intramodal Intermodal (Scans o sesiones) (Funcional/Estructural) Realineado Corregistro Transformaciones de cuerpo rígido Transformaciones afines Intersujeto Normalizado Transformaciones no lineales

Realineado

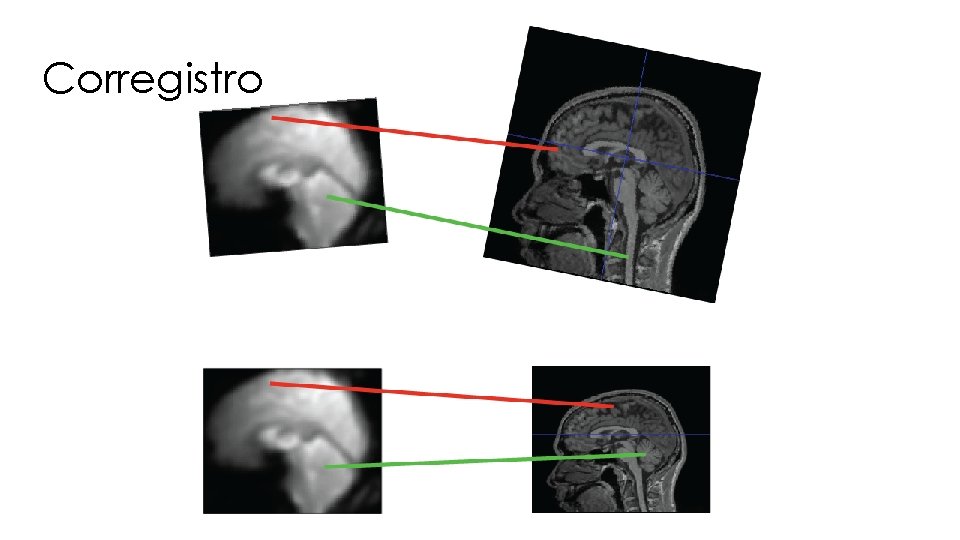
Corregistro

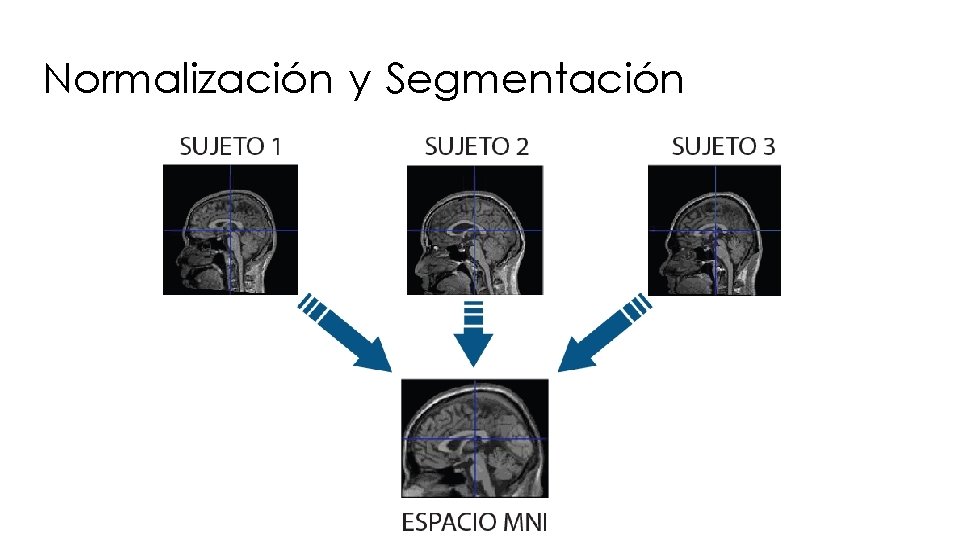
Normalización y Segmentación

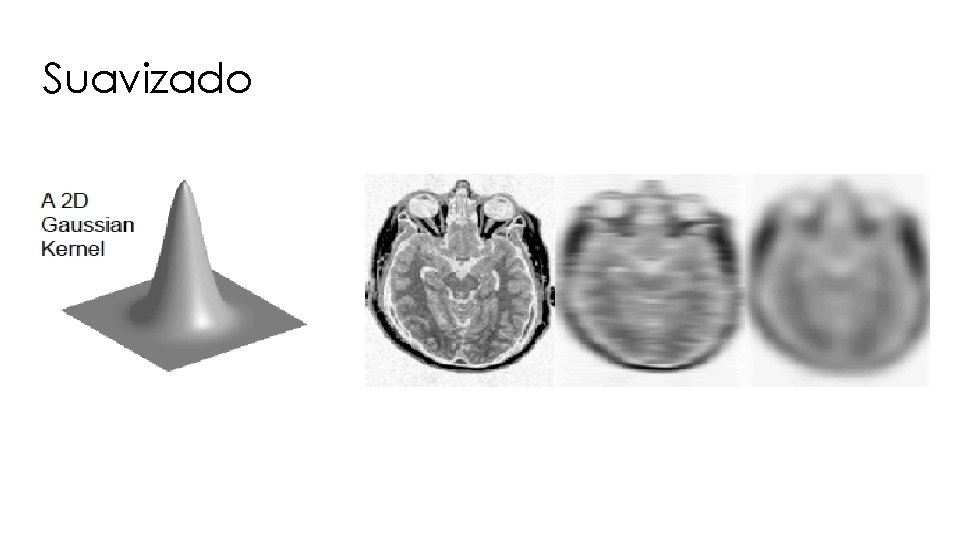
Suavizado

Procesamiento

Líneas de trabajo Mapas de Activación (GLM) Análisis de Semilla (Pearson) Mapas de Retraso (Covarianza) Matrices de Conectividad (Pearson)

Líneas de trabajo Mapas de Activación (GLM) Análisis de Semilla (Pearson) Mapas de Retraso (Covarianza) Matrices de Conectividad (Pearson)

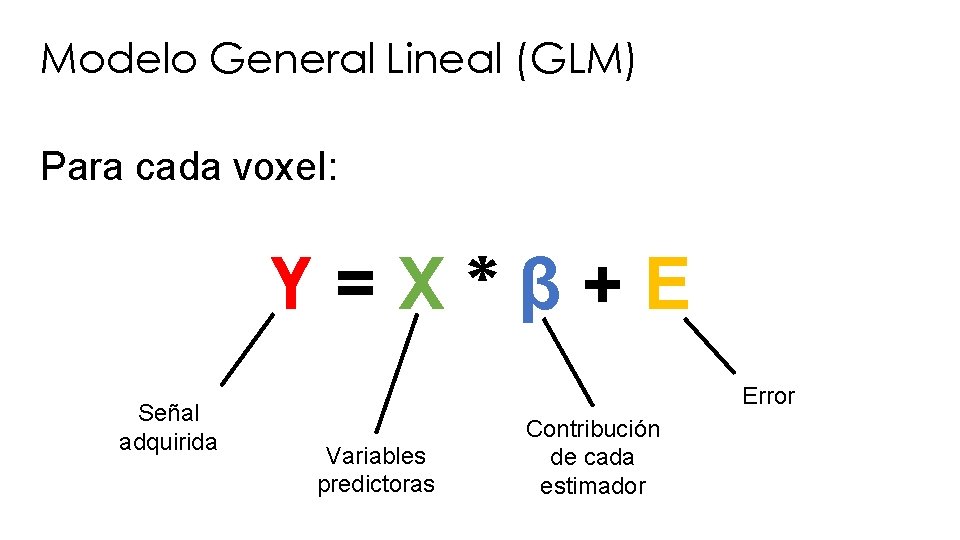
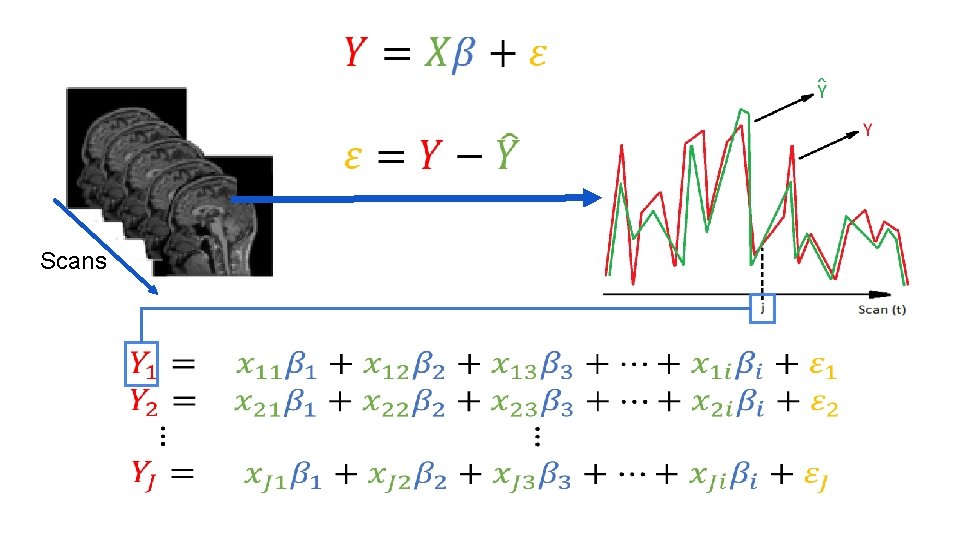
Modelo General Lineal (GLM) Para cada voxel: Y=X*β+E Señal adquirida Error Variables predictoras Contribución de cada estimador


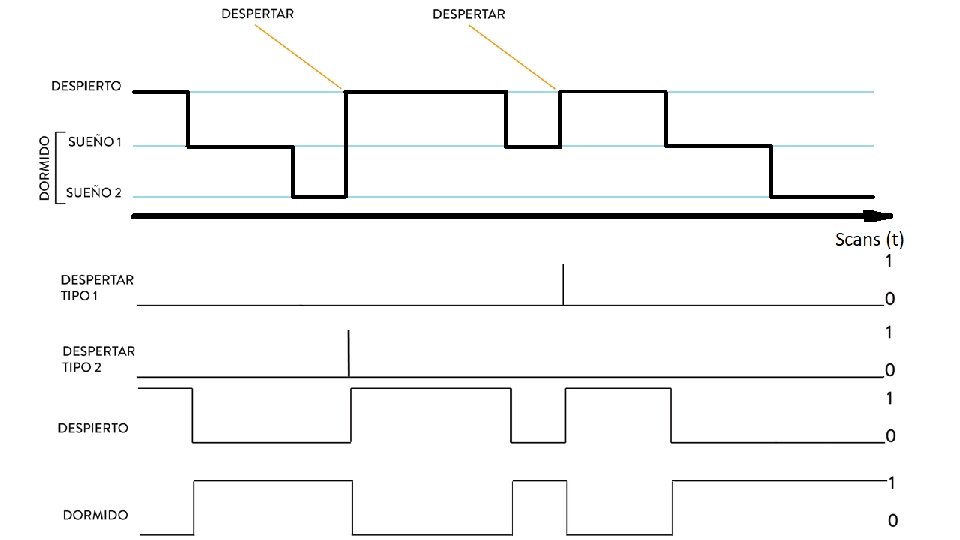
Scans

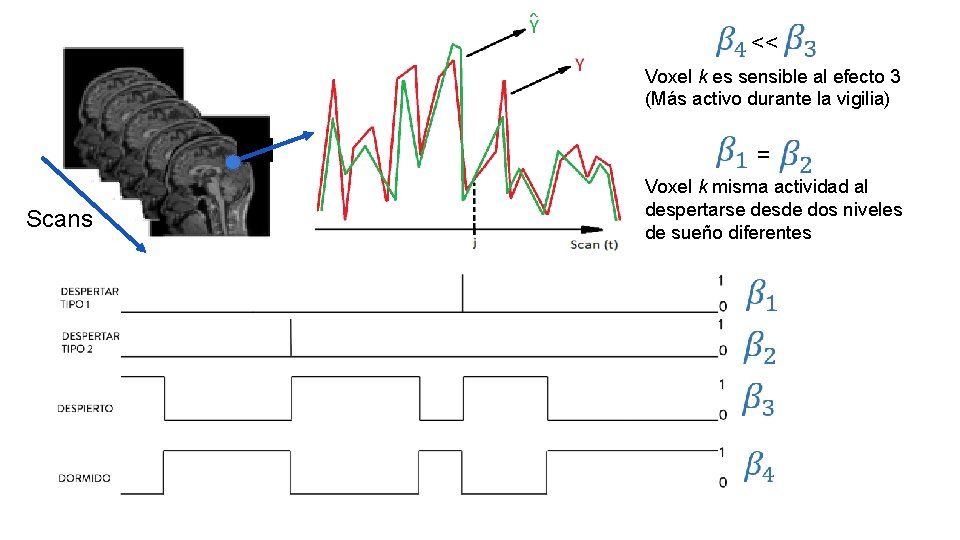
<< Voxel k es sensible al efecto 3 (Más activo durante la vigilia) = Scans Voxel k misma actividad al despertarse desde dos niveles de sueño diferentes


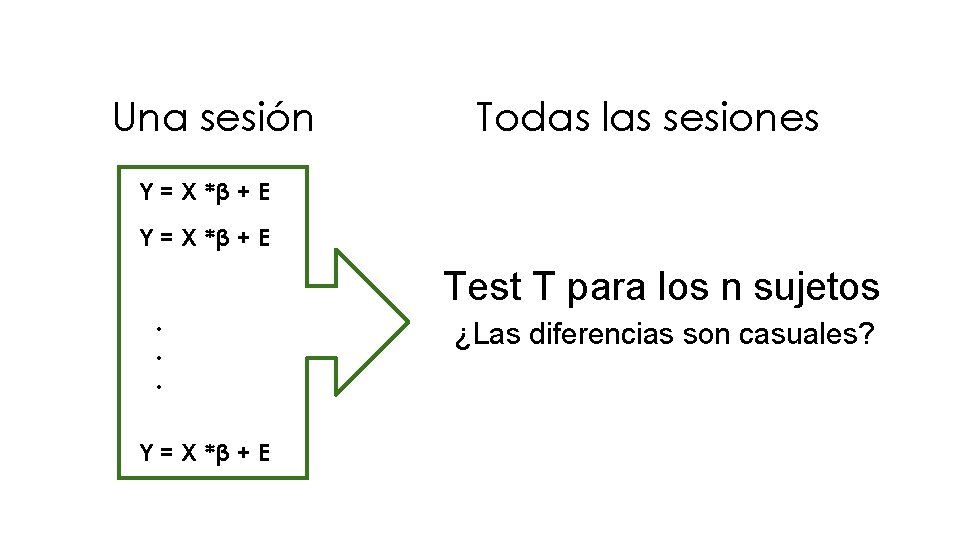
Una sesión Todas las sesiones Y = X *β + E. . . Y = X *β + E Test T para los n sujetos ¿Las diferencias son casuales?


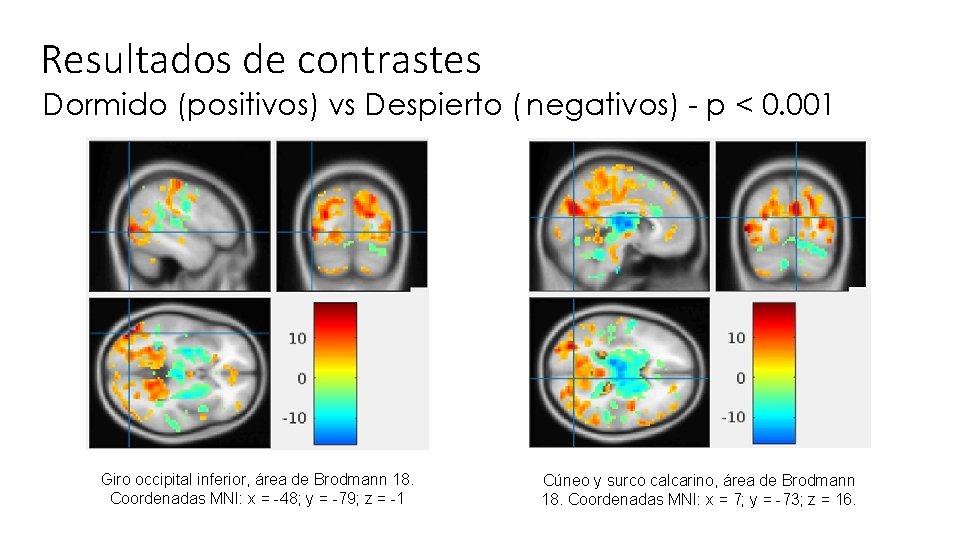
Resultados de contrastes Dormido (positivos) vs Despierto ( negativos) - p < 0. 001 Giro occipital inferior, área de Brodmann 18. Coordenadas MNI: x = -48; y = -79; z = -1 Cúneo y surco calcarino, área de Brodmann 18. Coordenadas MNI: x = 7; y = -73; z = 16.

Resultados de contrastes Despertar tipo 1 (rojo) vs Despertar tipo 2 (celeste) - p < 0. 001 Activación bilateral con un máximo global en la corteza de la Unión Temporoparietal derecha (TPJ). Coordenadas MNI: x = 51; y = -70; z = 8.

Líneas de trabajo Mapas de Activación (GLM) Análisis de Semilla (Pearson) Mapas de Retraso (Covarianza) Matrices de Conectividad (Pearson)

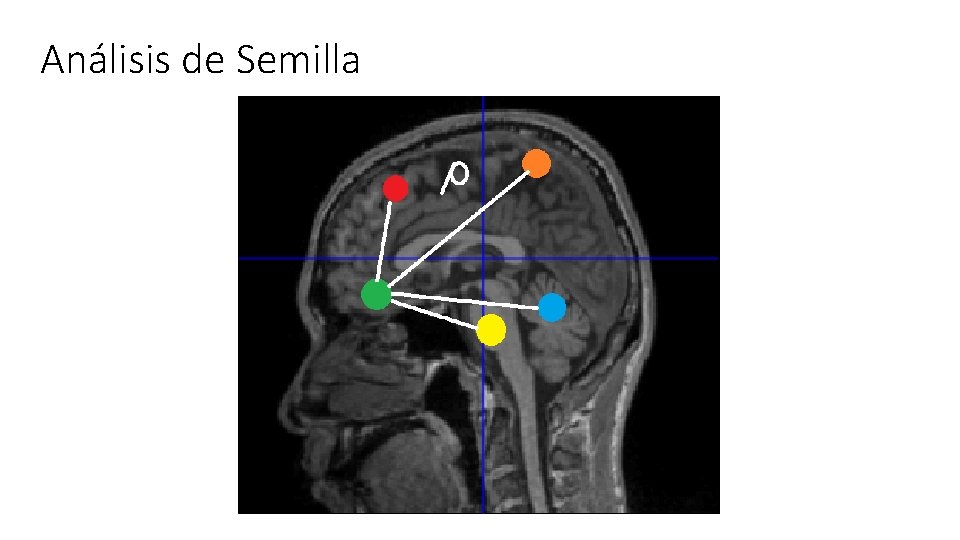
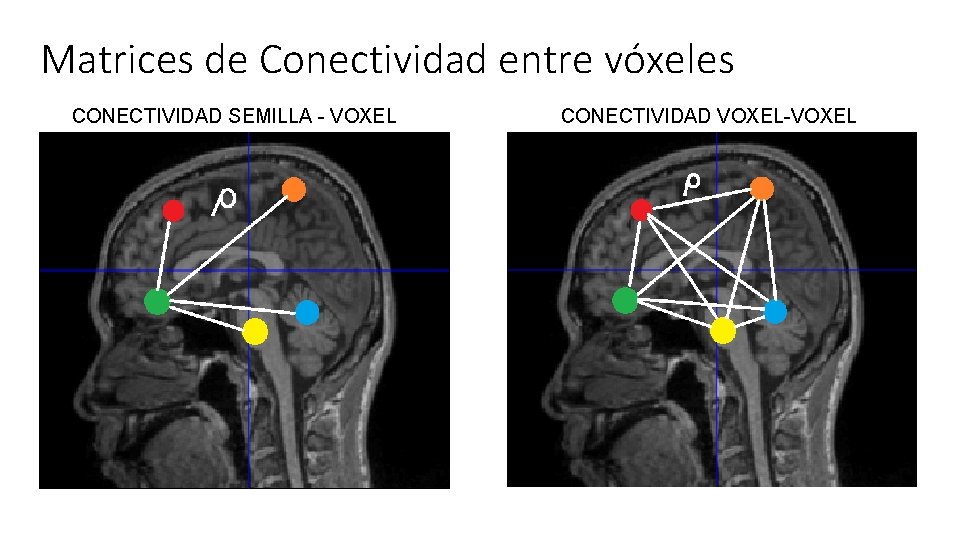
Análisis de Semilla

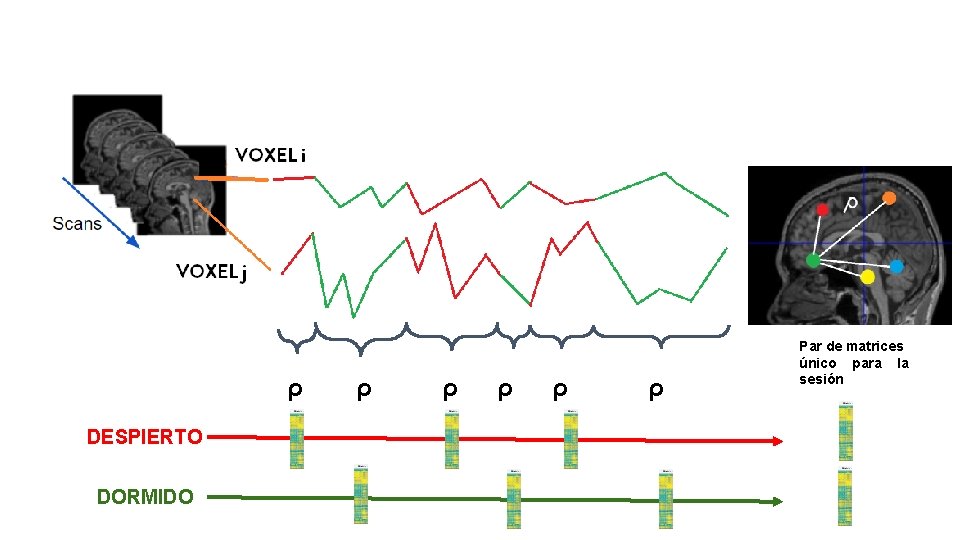
ρ DESPIERTO DORMIDO ρ ρ ρ Par de matrices único para la sesión

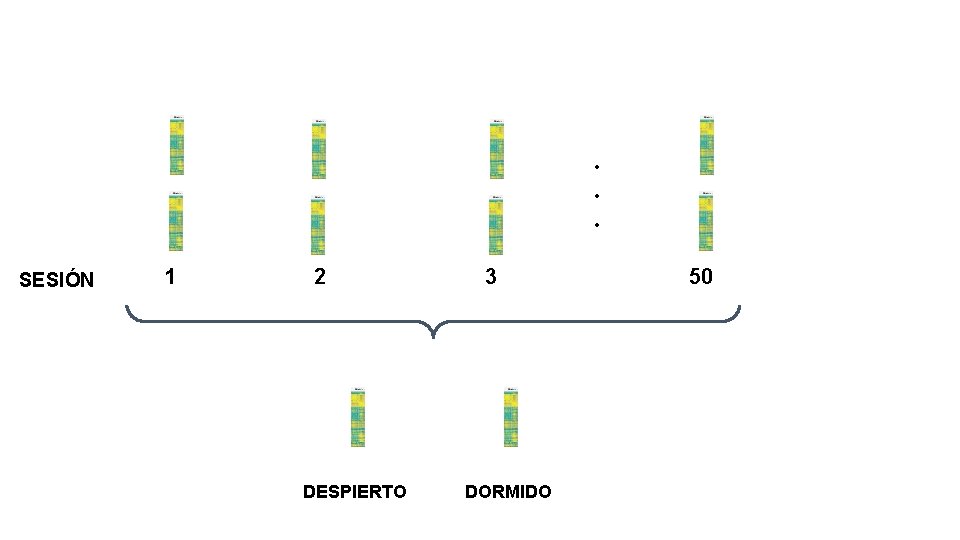
. . . SESIÓN 1 2 DESPIERTO 3 DORMIDO 50

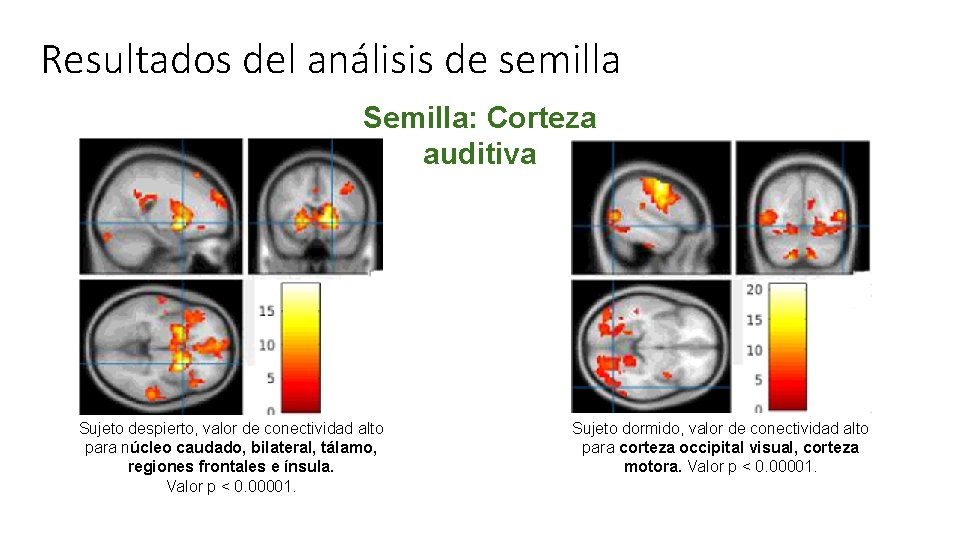
Semillas utilizadas ● INSULA ● PROTUBERANCIA ANULAR ● TÁLAMO ● CORTEZA AUDITIVA


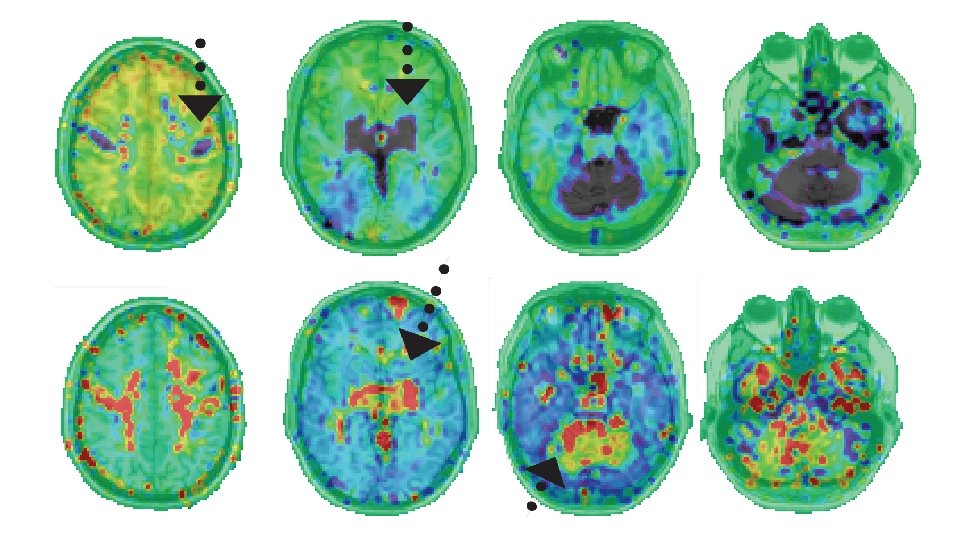
Resultados del análisis de semilla Semilla: Corteza auditiva Sujeto despierto, valor de conectividad alto para núcleo caudado, bilateral, tálamo, regiones frontales e ínsula. Valor p < 0. 00001. Sujeto dormido, valor de conectividad alto para corteza occipital visual, corteza motora. Valor p < 0. 00001.

Líneas de trabajo Mapas de Activación (GLM) Análisis de Semilla (Pearson) Mapas de Retraso (Covarianza) Matrices de Conectividad (Pearson)

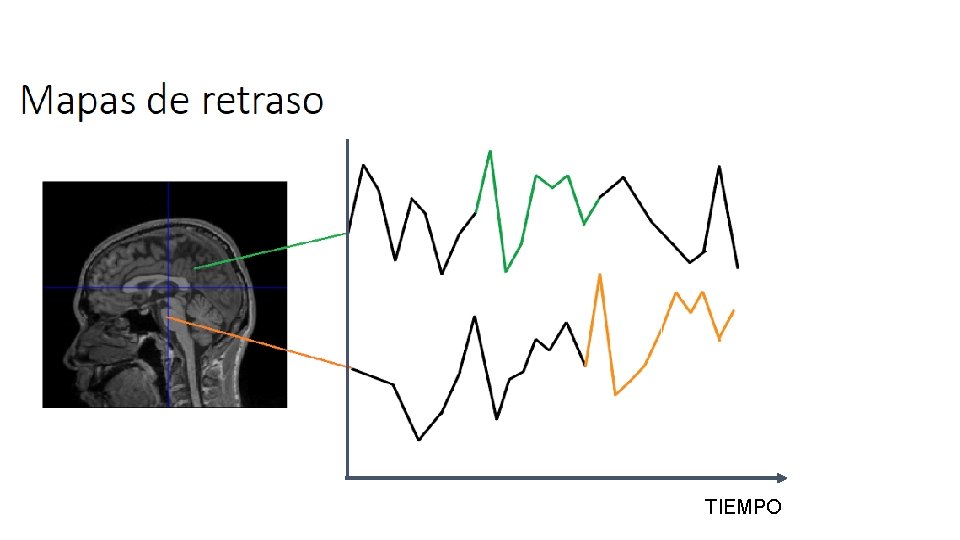
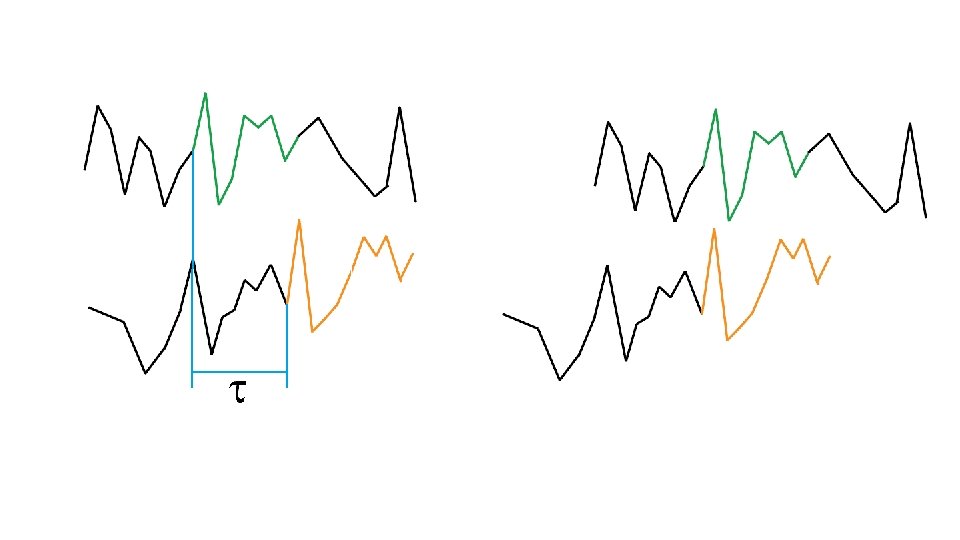
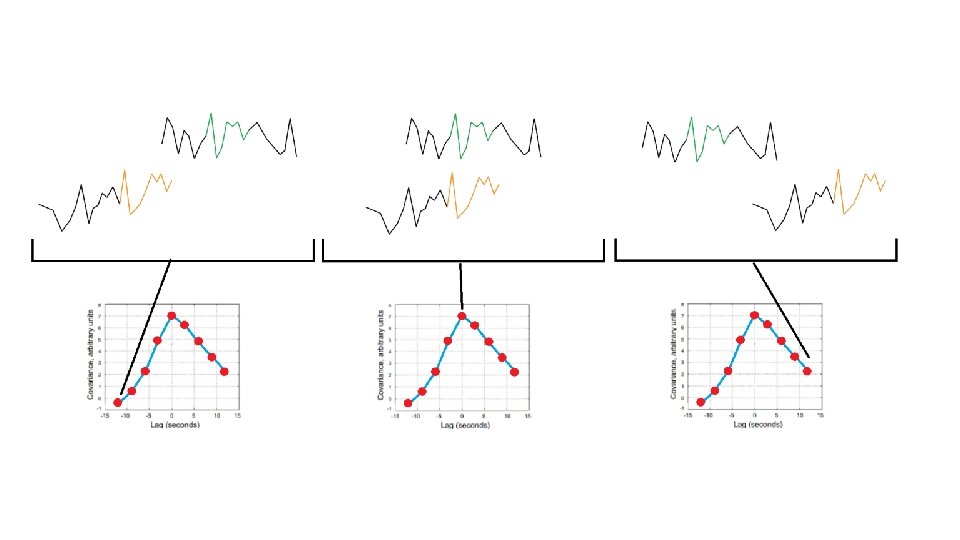
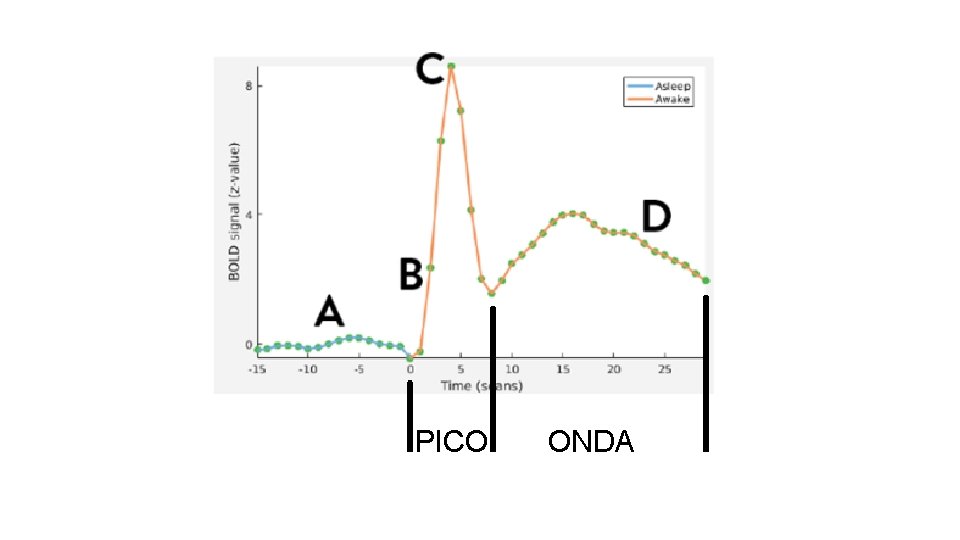
TIEMPO






PICO ONDA

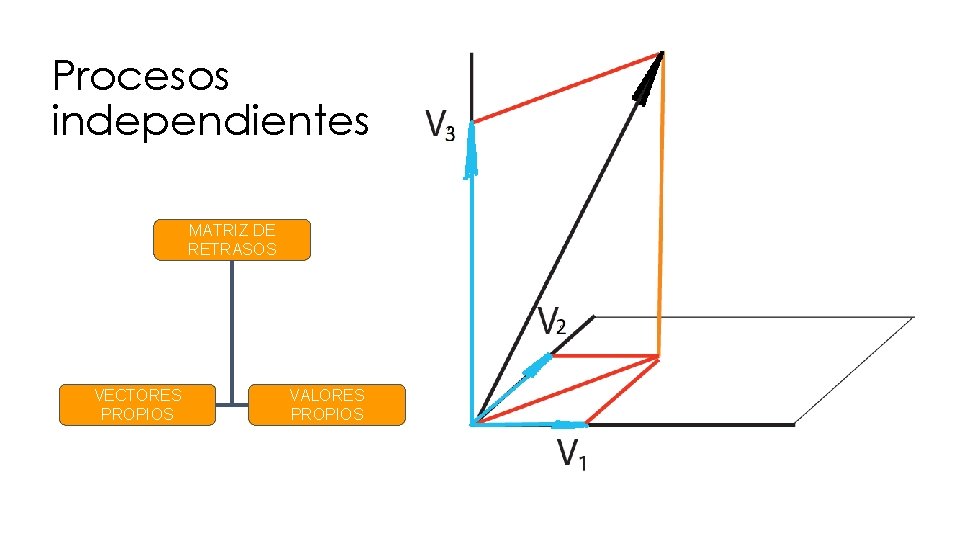
Procesos independientes MATRIZ DE RETRASOS VECTORES PROPIOS VALORES PROPIOS

Hilos - Procesos independientes


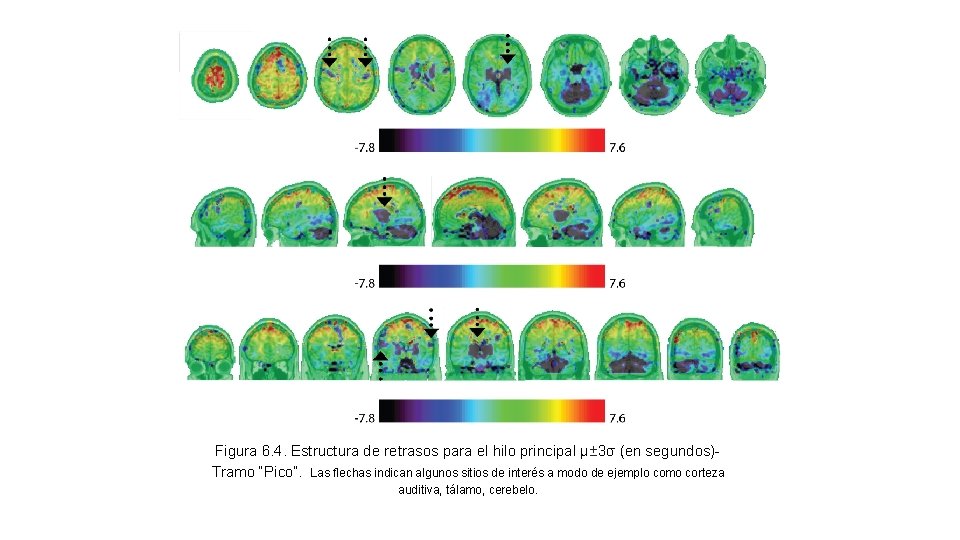
Figura 6. 4. Estructura de retrasos para el hilo principal μ± 3σ (en segundos)Tramo “Pico”. Las flechas indican algunos sitios de interés a modo de ejemplo como corteza auditiva, tálamo, cerebelo.

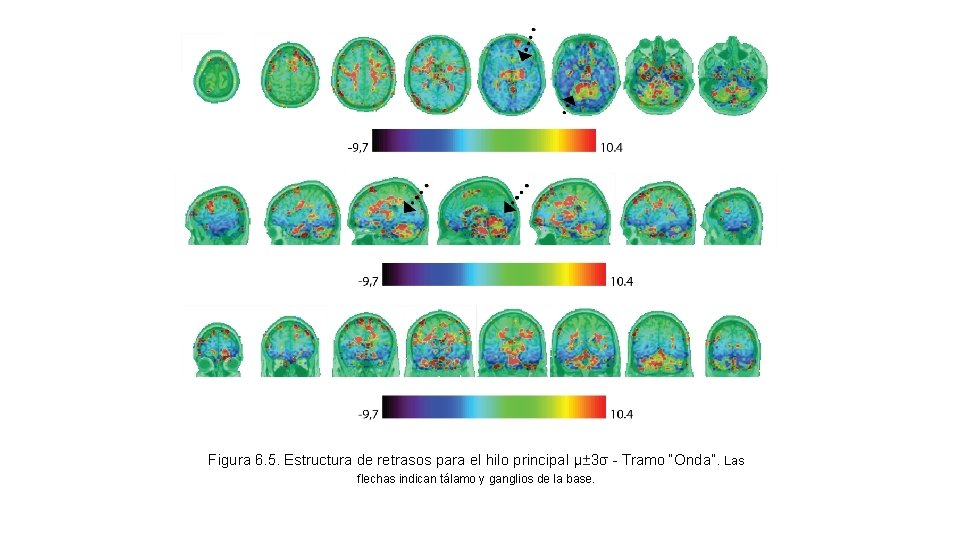
Figura 6. 5. Estructura de retrasos para el hilo principal μ± 3σ - Tramo “Onda”. Las flechas indican tálamo y ganglios de la base.



Líneas de trabajo Mapas de Activación (GLM) Análisis de Semilla (Pearson) Mapas de Retraso (Covarianza) Matrices de Conectividad (Pearson)

Matrices de Conectividad entre vóxeles CONECTIVIDAD SEMILLA - VOXEL CONECTIVIDAD VOXEL-VOXEL

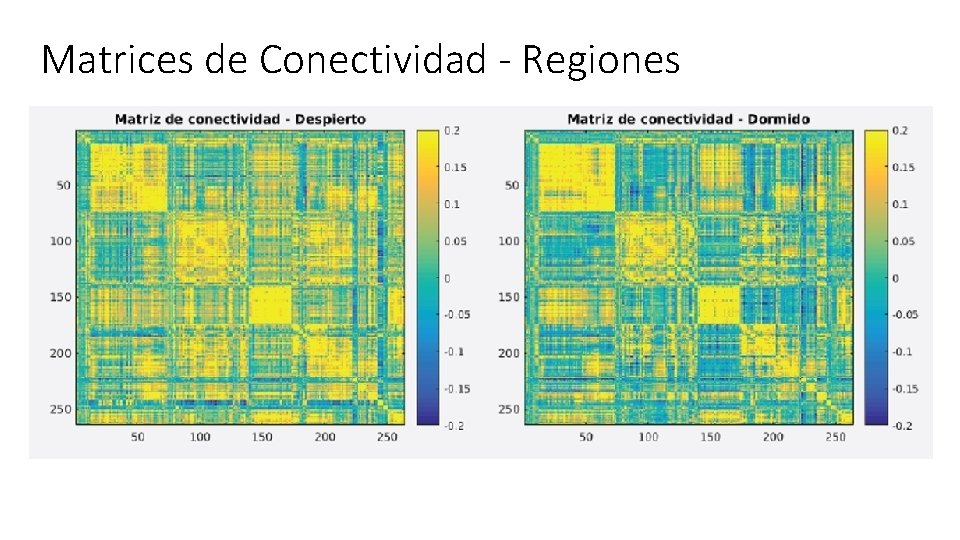
Matrices de Conectividad - Regiones

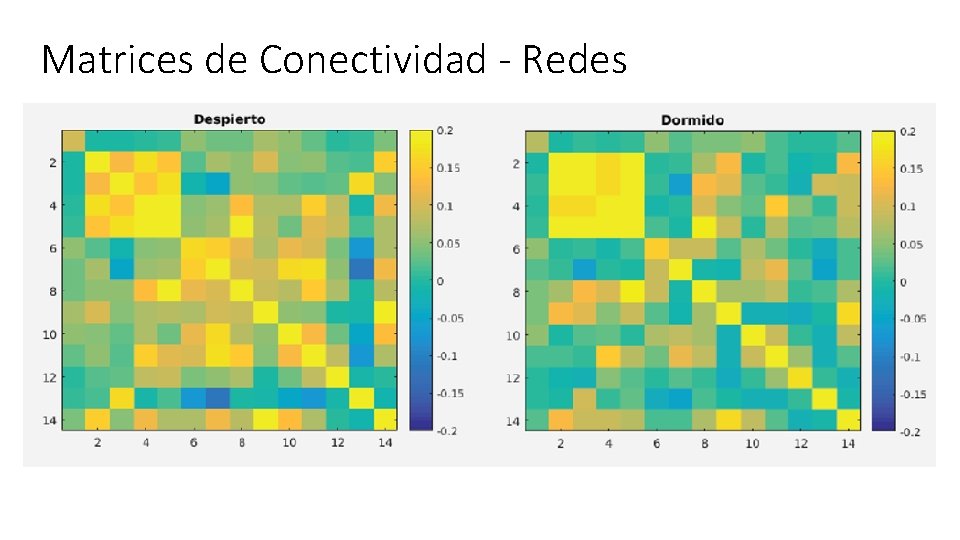
Matrices de Conectividad - Redes

Conclusiones

Conclusiones Mapas de Activación (GLM) Análisis de Semilla (Pearson) Mapas de Retraso (Covarianza) Matrices de Conectividad (Pearson)

Conclusiones Mapas de Activación (GLM) Análisis de Semilla (Pearson) Mapas de Retraso (Covarianza) Matrices de Conectividad (Pearson)

Conclusiones Mapas de Activación (GLM) Análisis de Semilla (Pearson) Mapas de Retraso (Covarianza) Matrices de Conectividad (Pearson)

Conclusiones Mapas de Activación (GLM) Análisis de Semilla (Pearson) Mapas de Retraso (Covarianza) Matrices de Conectividad (Pearson)

Propuestas a futuro

Softwares utilizados ● Matlab o Toolboxes ■ SPM 12 ■ Conn ■ Xj. View ■ REST ■ Mars. Bars ● Anatomist ~20 Scripts elaborados sobre Mat. Lab, C++ y Python

¿Preguntas?

 Esculturas impresionismo
Esculturas impresionismo Oración al despertar (salmo 5)
Oración al despertar (salmo 5) Poesia todas las mañanas sueño al despertar
Poesia todas las mañanas sueño al despertar Gibur runa
Gibur runa Passive and active
Passive and active Los 4 estados de conciencia gnosis
Los 4 estados de conciencia gnosis Os doze sinais do despertar divino
Os doze sinais do despertar divino Frecuencia de resonancia
Frecuencia de resonancia Resonancia schumann en vivo
Resonancia schumann en vivo Curva universal de resonancia
Curva universal de resonancia Difusion y mapa adc
Difusion y mapa adc Birads 4 porcentaje de malignidad
Birads 4 porcentaje de malignidad Grado de insaturacion
Grado de insaturacion Resonancia magnetica de sacroiliacas
Resonancia magnetica de sacroiliacas Resonancia magnetica
Resonancia magnetica Resonancia limbica
Resonancia limbica Emotividad actividad y resonancia
Emotividad actividad y resonancia Resonancia afectiva
Resonancia afectiva Nitrilo grupo funcional
Nitrilo grupo funcional Interferencia de ondas
Interferencia de ondas Friedel craft
Friedel craft Conclusion de resonancia magnetica
Conclusion de resonancia magnetica Eco de gradiente
Eco de gradiente Resonancia del naftaleno
Resonancia del naftaleno Colangio resonancia magnética
Colangio resonancia magnética Arteriopata
Arteriopata Gradiente x y z resonancia
Gradiente x y z resonancia Capacidad normal viable
Capacidad normal viable Tcnicas de estudio
Tcnicas de estudio Estudio solano
Estudio solano Estudio 52
Estudio 52 Velad y orad porque vuestro adversario
Velad y orad porque vuestro adversario Estudio viabilidad residencia tercera edad
Estudio viabilidad residencia tercera edad Estudio organizacional importancia
Estudio organizacional importancia Cuestionario chte
Cuestionario chte Vocabulario cientifico
Vocabulario cientifico Dios castiga a los malos
Dios castiga a los malos Estudio de la santidad
Estudio de la santidad Estudio de caso ejemplo
Estudio de caso ejemplo Estudio tecnico en un proyecto
Estudio tecnico en un proyecto Metodo de deficit acumulado
Metodo de deficit acumulado Estudio de viabilidad
Estudio de viabilidad Estudio independiente mapa mental
Estudio independiente mapa mental Honroso sea el matrimonio
Honroso sea el matrimonio Estudio descriptivo
Estudio descriptivo Jairo sponholz araujo
Jairo sponholz araujo Dios incomparable versiculo
Dios incomparable versiculo Estudio descriptivo y analitico
Estudio descriptivo y analitico Ejercicios de repetibilidad y reproducibilidad
Ejercicios de repetibilidad y reproducibilidad Entrada triunfal de jesus a jerusalen mateo
Entrada triunfal de jesus a jerusalen mateo Tipos de ciencias sociales
Tipos de ciencias sociales Programa de estudio bachillerato técnico en salud
Programa de estudio bachillerato técnico en salud Estudio bíblico los filisteos capturan el arca
Estudio bíblico los filisteos capturan el arca Metodos de estudio del interior terrestre
Metodos de estudio del interior terrestre Estudio administrativo
Estudio administrativo Validez externa e interna
Validez externa e interna 1 samuel 27 estudio biblico
1 samuel 27 estudio biblico El incomparable dios de israel estudio
El incomparable dios de israel estudio Que es un estudio correlacional
Que es un estudio correlacional Nivel de perfil
Nivel de perfil Estudio de valores de allport
Estudio de valores de allport Funcion creciente en todo su dominio
Funcion creciente en todo su dominio Cinco dimensiones de la personalidad
Cinco dimensiones de la personalidad Tipos de estudio descriptivo
Tipos de estudio descriptivo Que son los aspectos éticos
Que son los aspectos éticos Objeto de estudio de la pedagogía social
Objeto de estudio de la pedagogía social No me elegisteis vosotros a mi
No me elegisteis vosotros a mi Primera de reyes 17 8
Primera de reyes 17 8 Tcnicas de estudio
Tcnicas de estudio Estudio del desarrollo de su profesión y su estado actual
Estudio del desarrollo de su profesión y su estado actual Que es el don de ciencia
Que es el don de ciencia Nucleo verbal
Nucleo verbal Preparatoria abierta gratis puebla
Preparatoria abierta gratis puebla Estudio de los valores desde el punto de vista psicológico
Estudio de los valores desde el punto de vista psicológico Estudio inductivo de mateo
Estudio inductivo de mateo