Resonancia magntica nuclear 1 Qu es y para








- Slides: 28

Resonancia magnética nuclear 1. ¿Qué es y para qué sirve? 3. Aplicaciones de RMN en Instrumentación bioquímica 2. Parámetros de un espectro • Cinética y vías metabólicas • Unión de ligandos a sencillo de RMN • • • Intensidad (área bajo la curva) Desplazamiento químico (d). Grupos funcionales en 1 H RMN Desdoblamiento de señales Tiempos de relajación Espectros bidimensionales • • macromoléculas Modificaciones estructurales de proteínas Determinación de estructuras de macromoléculas. 1

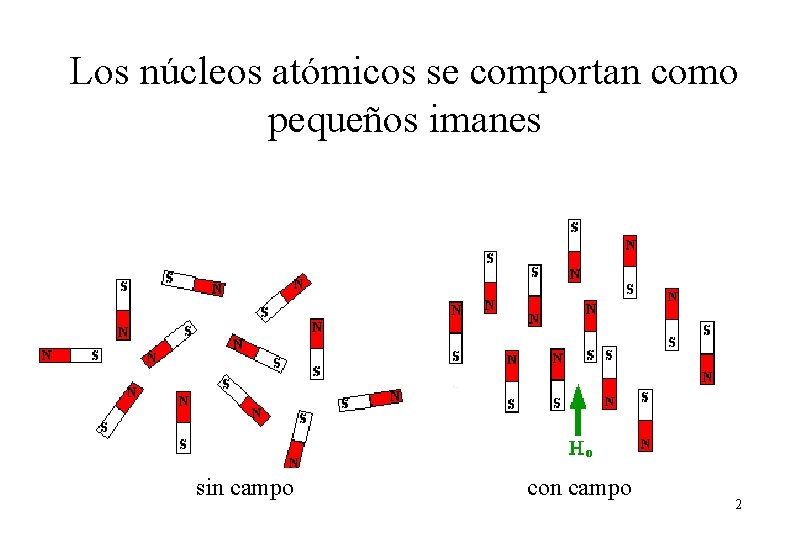
Los núcleos atómicos se comportan como pequeños imanes sin campo con campo 2

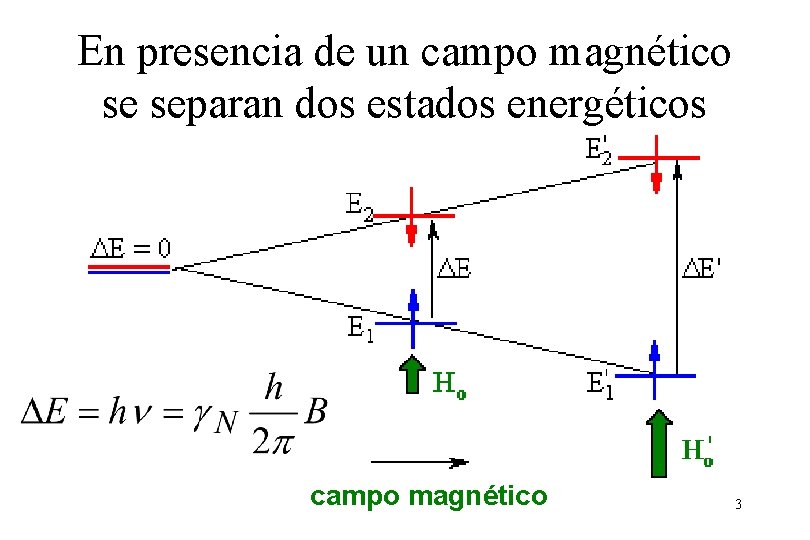
En presencia de un campo magnético se separan dos estados energéticos campo magnético 3

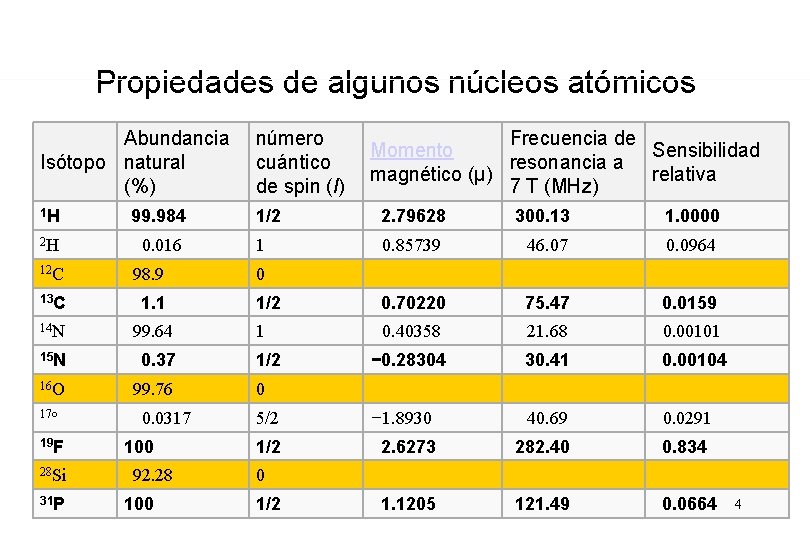
Propiedades de algunos núcleos atómicos Abundancia Isótopo natural (%) número cuántico de spin (I) 1 H 99. 984 1/2 2. 79628 300. 13 1. 0000 2 H 0. 016 1 0. 85739 46. 07 0. 0964 12 C 98. 9 13 C 1. 1 14 N 99. 64 15 N 0. 37 16 O 99. 76 17º 19 F 28 Si 31 P 0. 0317 100 92. 28 100 0 Frecuencia de Momento Sensibilidad resonancia a magnético (μ) relativa 7 T (MHz) 1/2 0. 70220 75. 47 0. 0159 1 0. 40358 21. 68 0. 00101 1/2 − 0. 28304 30. 41 0. 00104 0 5/2 − 1. 8930 40. 69 0. 0291 1/2 2. 6273 282. 40 0. 834 0 1/2 1. 1205 121. 49 0. 0664 4

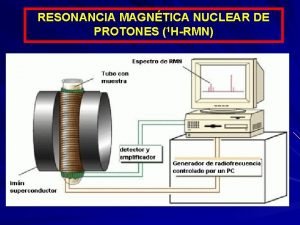
Equipo básico de RMN transmisor de radiofrecuencia Bobinas de barrido receptor de radiofrecuencia Bobinas de barrido Polo del imán Control y registrador Tubo de muestra Generador de barrido 5

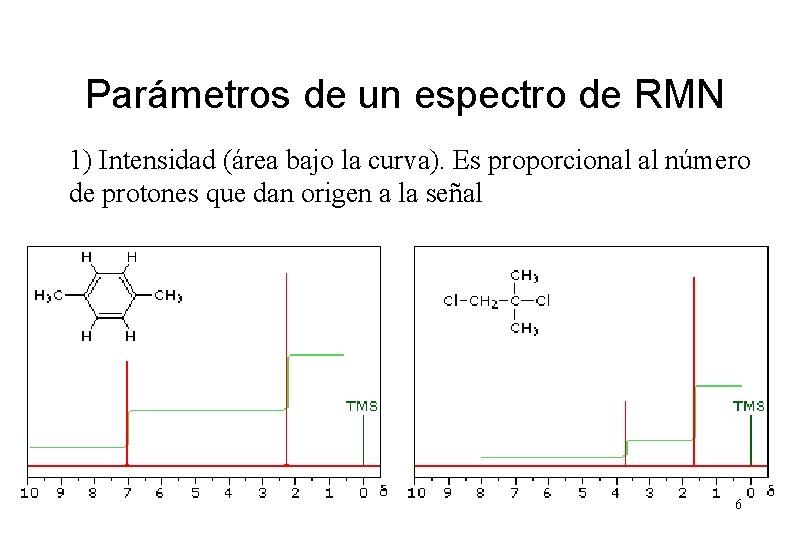
Parámetros de un espectro de RMN 1) Intensidad (área bajo la curva). Es proporcional al número de protones que dan origen a la señal 6

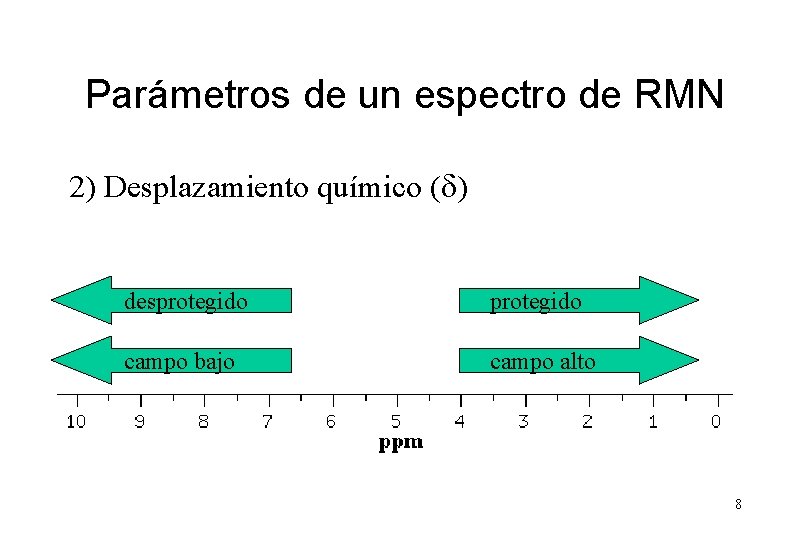
Parámetros de un espectro de RMN 2) Desplazamiento químico (d) Es producto del campo magnético inducido por los electrones vecinos al núcleo 7

Parámetros de un espectro de RMN 2) Desplazamiento químico (d) desprotegido campo bajo campo alto 8

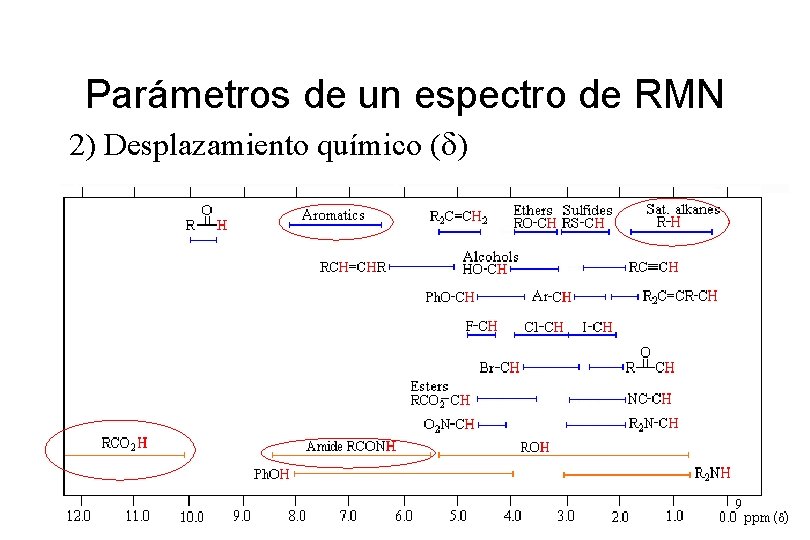
Parámetros de un espectro de RMN 2) Desplazamiento químico (d) 9

Parámetros de un espectro de RMN 2) Desplazamiento químico (d) Observaciones: • Los grupos electronegativos “desprotegen” a los núcleos y tienden a desplazar las señales a “campo bajo” (a mayores valores en ppm) • Los protones unidos a O y N tienen desplazamientos químicos muy variables que son sensibles a la concentración (formación de puentes de hidrógeno) a la temperatura y al disolvente (fenómenos de intercambio químico). • Los sistemas p desprotegen mucho a los protones que tienen unidos y tienden a desplazar las señales a “campo bajo” (a mayores valores en ppm) 10

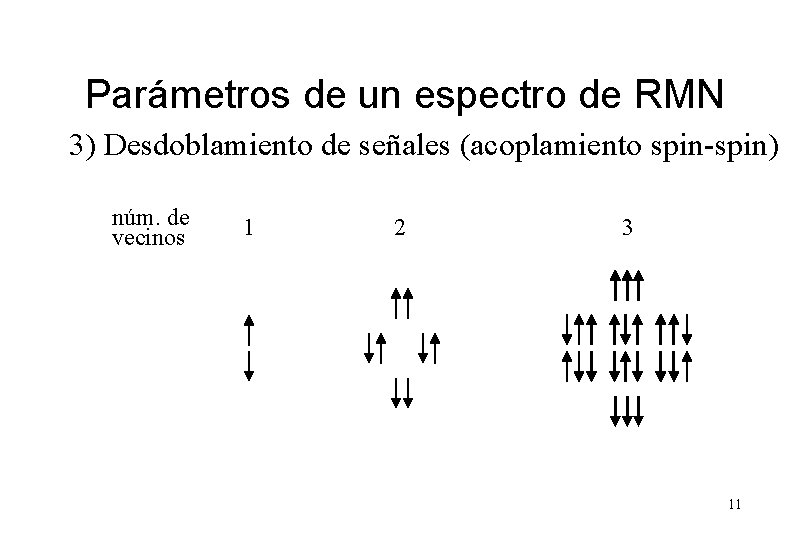
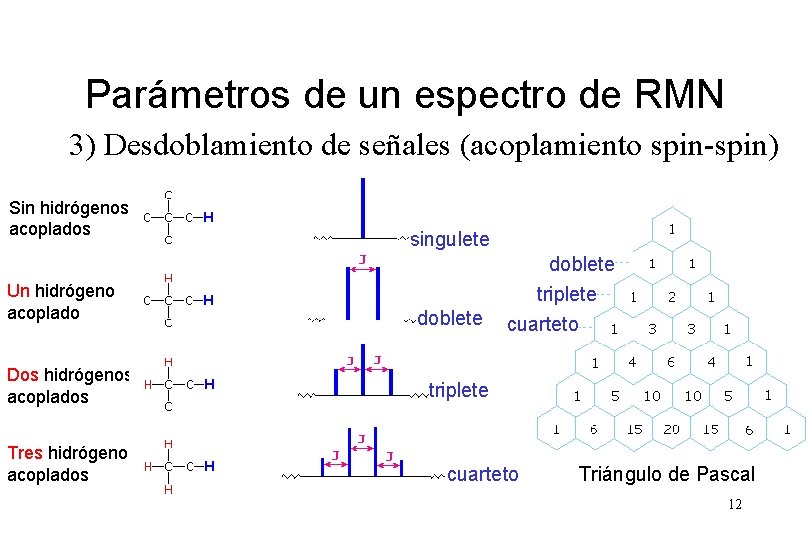
Parámetros de un espectro de RMN 3) Desdoblamiento de señales (acoplamiento spin-spin) núm. de vecinos 1 2 3 11

Parámetros de un espectro de RMN 3) Desdoblamiento de señales (acoplamiento spin-spin) Sin hidrógenos acoplados Un hidrógeno acoplado Dos hidrógenos acoplados Tres hidrógenos acoplados singulete doblete triplete cuarteto Triángulo de Pascal 12

Parámetros de un espectro de RMN 4) Tiempos de relajación El término relajación describe varios procesos por los cuales la magnetización de un estado fuera del equilibrio regresa a la distribución de equilibrio. Es decir, la relajación describe qué tan rápido los spines nucleares “olvidan” la dirección en que estaban orientados. Las velocidades de esta relajación de spines se puede medir y tiene aplicación en espectroscopía y en obtención de imágenes. 13

Interpretación de espectros simples de 1 H RMN Qué se puede obtener del espectro: 1 - ¿Cuántos tipos de H hay? – Indicado por cuántos grupos de señales hay en el espectro 2. - ¿Qué tipos de H ? – Indicados por los desplazamientos químicos de cada grupo 3. - ¿Cuántos H hay de cada tipo? – Indicados por la integración de la señal de cada grupo 4. - ¿Cuál es la conectividad? – A través de los patrones de acoplamiento sabemos qué grupo es vecino de qué grupo. 14

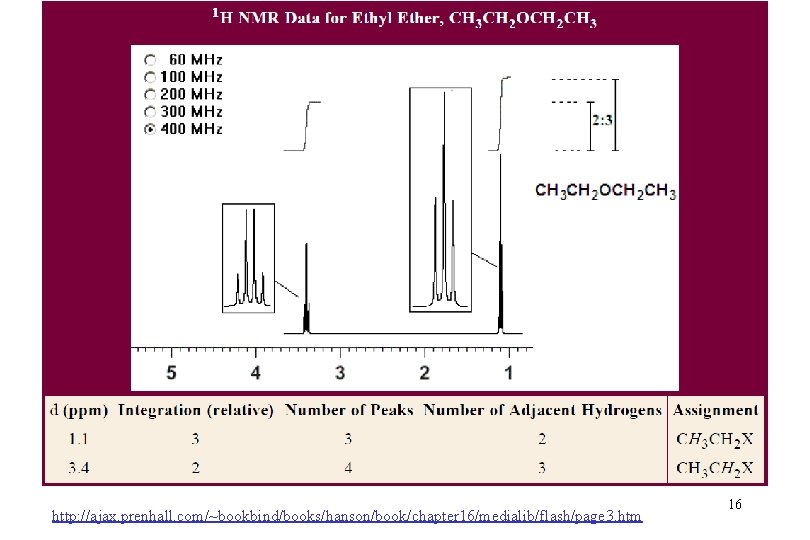
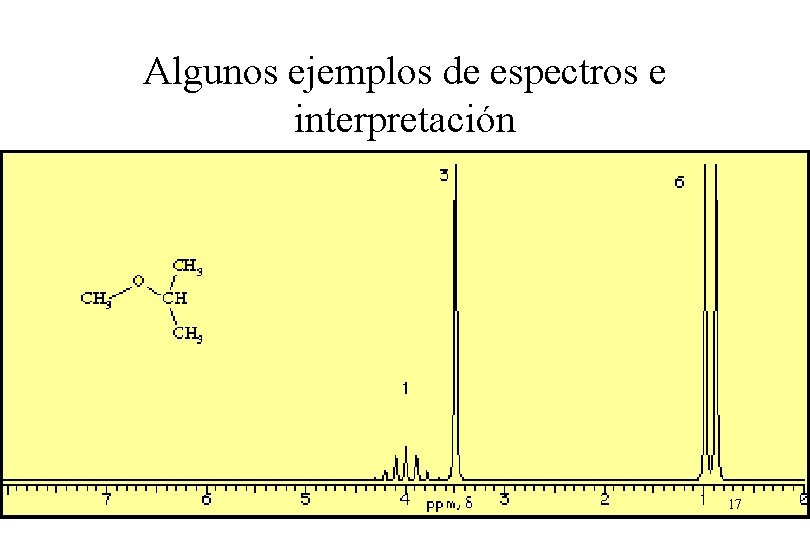
Algunos ejemplos de espectros e interpretación 15

http: //ajax. prenhall. com/~bookbind/books/hanson/book/chapter 16/medialib/flash/page 3. htm 16

Algunos ejemplos de espectros e interpretación 17

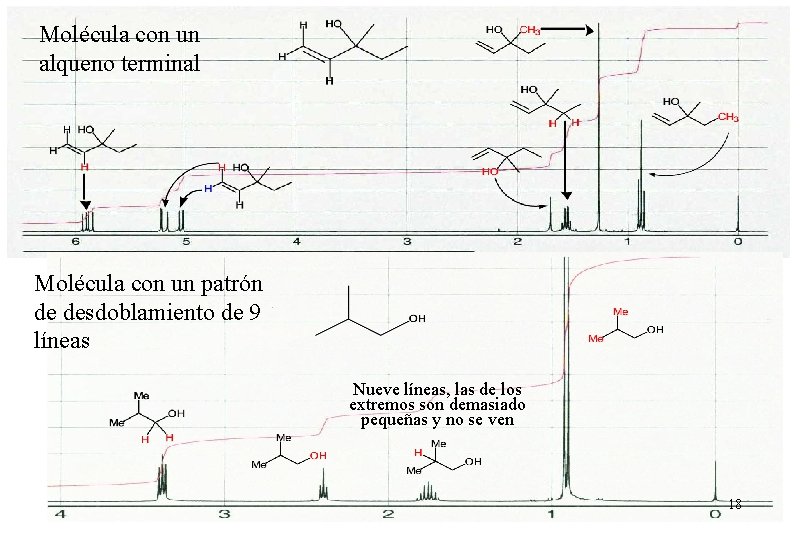
Molécula con un alqueno terminal Molécula con un patrón de desdoblamiento de 9 líneas Nueve líneas, las de los extremos son demasiado pequeñas y no se ven 18

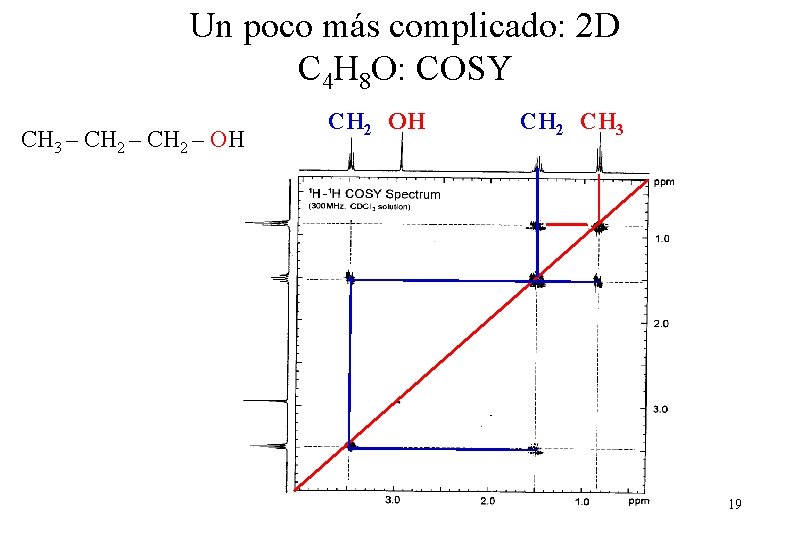
Un poco más complicado: 2 D C 4 H 8 O: COSY CH 3 – CH 2 – OH CH 2 OH CH 2 CH 3 19

Unión de ligando a ADN Robinson, H. and A. H. Wang (1996). Nucleic Acids Res 24(4): 676 -82. Usan 1 H RMN para estudiar las transiciones estructurales en el polinucleótido dúplex A 2 G 15 C 15 T 2 en presencia de la sonda catiónicas Co(NH 3)63+. Las figuras muestran dos secciones del espectro donde se ven algunos de los protones de las bases en presencia de Co(NH 3)63+ en diferentes relaciones de concentración respecto al ADN. 20

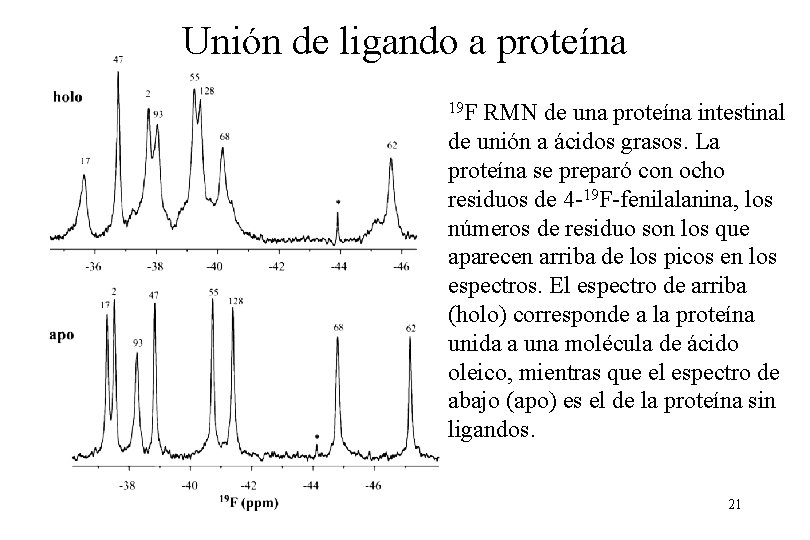
Unión de ligando a proteína 19 F RMN de una proteína intestinal de unión a ácidos grasos. La proteína se preparó con ocho residuos de 4 -19 F-fenilalanina, los números de residuo son los que aparecen arriba de los picos en los espectros. El espectro de arriba (holo) corresponde a la proteína unida a una molécula de ácido oleico, mientras que el espectro de abajo (apo) es el de la proteína sin ligandos. 21

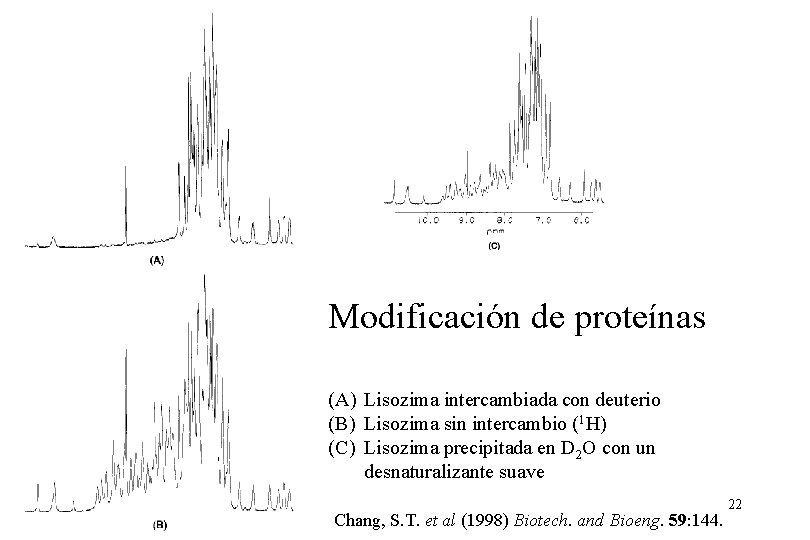
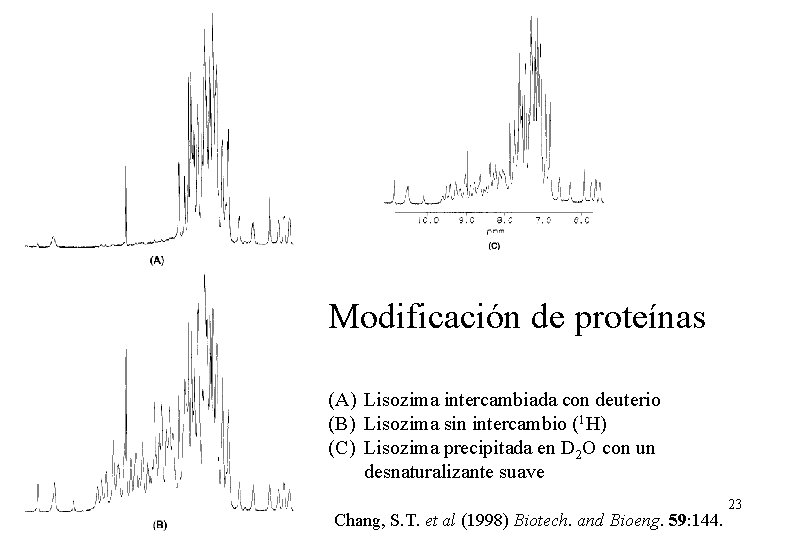
Modificación de proteínas (A) Lisozima intercambiada con deuterio (B) Lisozima sin intercambio (1 H) (C) Lisozima precipitada en D 2 O con un desnaturalizante suave Chang, S. T. et al (1998) Biotech. and Bioeng. 59: 144. 22

Modificación de proteínas (A) Lisozima intercambiada con deuterio (B) Lisozima sin intercambio (1 H) (C) Lisozima precipitada en D 2 O con un desnaturalizante suave Chang, S. T. et al (1998) Biotech. and Bioeng. 59: 144. 23

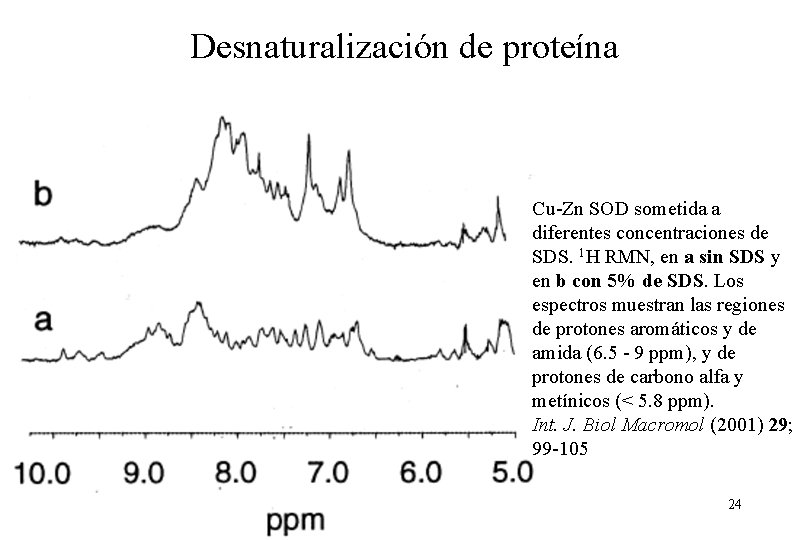
Desnaturalización de proteína Cu-Zn SOD sometida a diferentes concentraciones de SDS. 1 H RMN, en a sin SDS y en b con 5% de SDS. Los espectros muestran las regiones de protones aromáticos y de amida (6. 5 - 9 ppm), y de protones de carbono alfa y metínicos (< 5. 8 ppm). Int. J. Biol Macromol (2001) 29; 99 -105 24

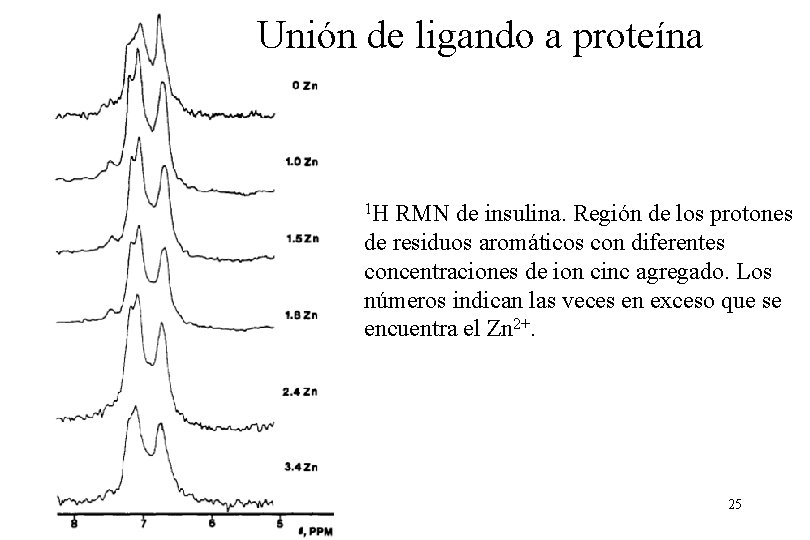
Unión de ligando a proteína 1 H RMN de insulina. Región de los protones de residuos aromáticos con diferentes concentraciones de ion cinc agregado. Los números indican las veces en exceso que se encuentra el Zn 2+. 25

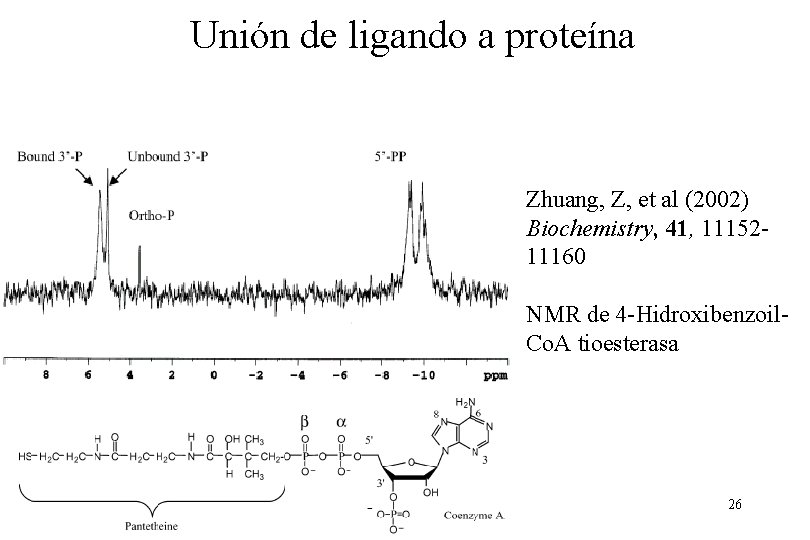
Unión de ligando a proteína Zhuang, Z, et al (2002) Biochemistry, 41, 1115211160 NMR de 4 -Hidroxibenzoil. Co. A tioesterasa 26

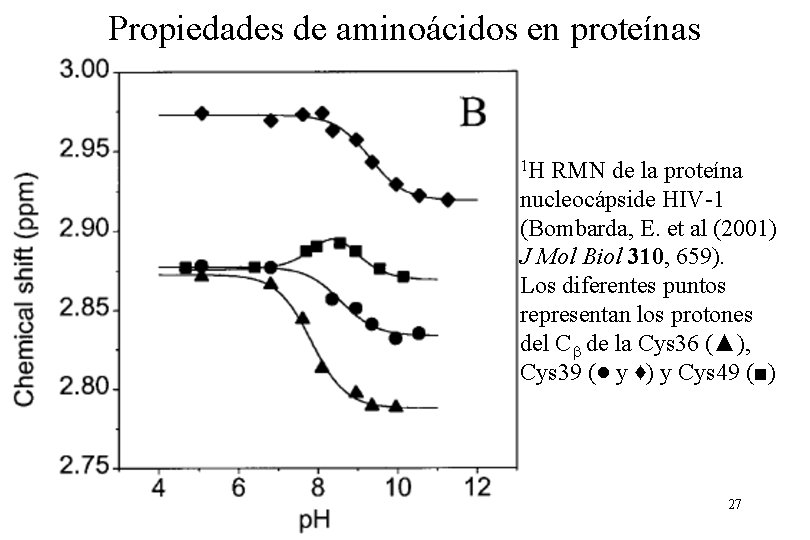
Propiedades de aminoácidos en proteínas 1 H RMN de la proteína nucleocápside HIV-1 (Bombarda, E. et al (2001) J Mol Biol 310, 659). Los diferentes puntos representan los protones del Cb de la Cys 36 (▲), Cys 39 (● y ♦) y Cys 49 (■) 27

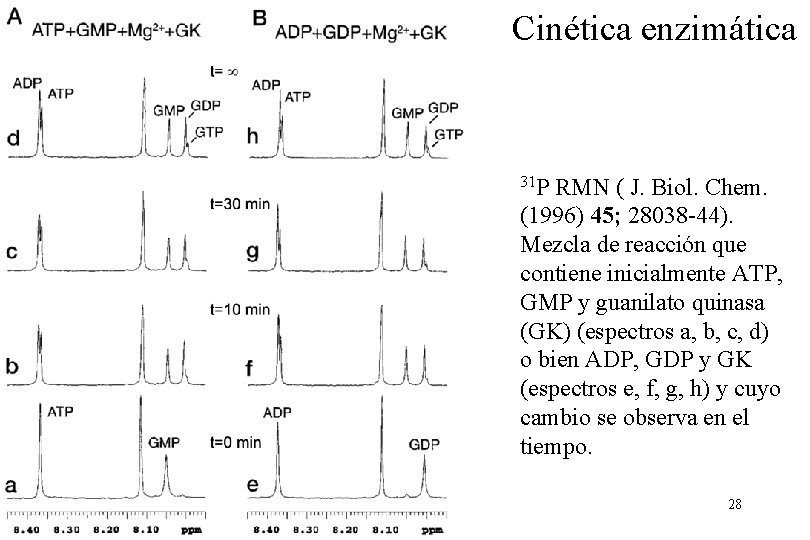
Cinética enzimática 31 P RMN ( J. Biol. Chem. (1996) 45; 28038 -44). Mezcla de reacción que contiene inicialmente ATP, GMP y guanilato quinasa (GK) (espectros a, b, c, d) o bien ADP, GDP y GK (espectros e, f, g, h) y cuyo cambio se observa en el tiempo. 28
 Resonancia magnetica nuclear
Resonancia magnetica nuclear Lesson 15 nuclear quest nuclear reactions
Lesson 15 nuclear quest nuclear reactions Fisión nuclear vs fision nuclear
Fisión nuclear vs fision nuclear Resonancia limbica
Resonancia limbica Friedel craft
Friedel craft Bobinas de gradiente x y z
Bobinas de gradiente x y z Birads 4 porcentaje de malignidad
Birads 4 porcentaje de malignidad Resonancia afectiva
Resonancia afectiva Secuencias spin eco
Secuencias spin eco Resonancia schumann en vivo
Resonancia schumann en vivo Resonancia magnetica de sacroiliacas
Resonancia magnetica de sacroiliacas Acilacion
Acilacion Curva universal de resonancia
Curva universal de resonancia Resonancia magnetica
Resonancia magnetica Interferencia de ondas
Interferencia de ondas Arteriopata
Arteriopata Mapa adc resonancia magnetica
Mapa adc resonancia magnetica Que es el colerico
Que es el colerico Conclusion de resonancia magnetica
Conclusion de resonancia magnetica Q es?
Q es? Nitrilo grupo funcional
Nitrilo grupo funcional Colangio resonancia magnética
Colangio resonancia magnética Núcleo celular
Núcleo celular Intranet venus
Intranet venus Natural transmutation example
Natural transmutation example Concepto de la energia nuclear
Concepto de la energia nuclear Quantum and nuclear physics
Quantum and nuclear physics What are fuels used for
What are fuels used for Spindle fibers
Spindle fibers