Lezione 5 DNA struttura e replicazione Cromosomi 1


























- Slides: 63

Lezione 5 DNA struttura e replicazione Cromosomi 1

Natura del materiale genetico Nei primi decenni del ‘ 900 si raccolgono evidenze che i cromosomi sono costituiti da DNA e proteine. - Qual è la molecola responsabile dell’informazione biologica? - Qual è la sua struttura? Esperimenti di importanza fondamentale (Griffith, Avery et al. , Hershey et al. ) vengono effettuati negli anni ’ 30 -50 su organismi semplici, quali batteri e batteriofagi e dimostrano che il materiale genetico è il DNA 2

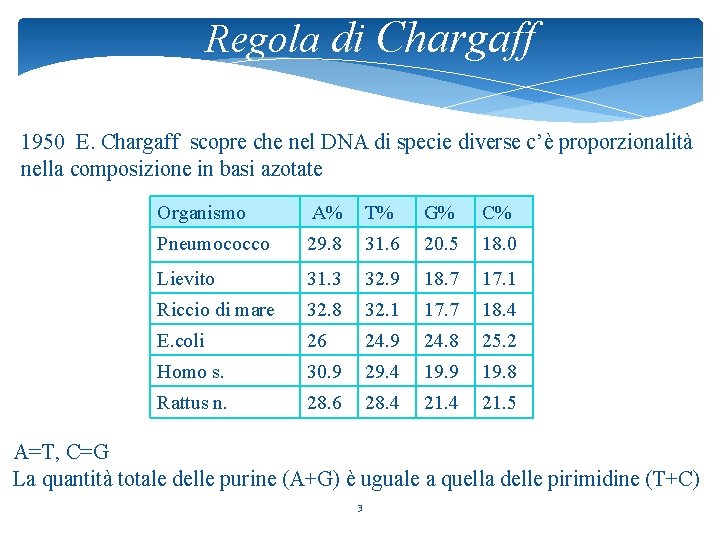
Regola di Chargaff 1950 E. Chargaff scopre che nel DNA di specie diverse c’è proporzionalità nella composizione in basi azotate Organismo A% T% G% C% Pneumococco 29. 8 31. 6 20. 5 18. 0 Lievito 31. 3 32. 9 18. 7 17. 1 Riccio di mare 32. 8 32. 1 17. 7 18. 4 E. coli 26 24. 9 24. 8 25. 2 Homo s. 30. 9 29. 4 19. 9 19. 8 Rattus n. 28. 6 28. 4 21. 5 A=T, C=G La quantità totale delle purine (A+G) è uguale a quella delle pirimidine (T+C) 3

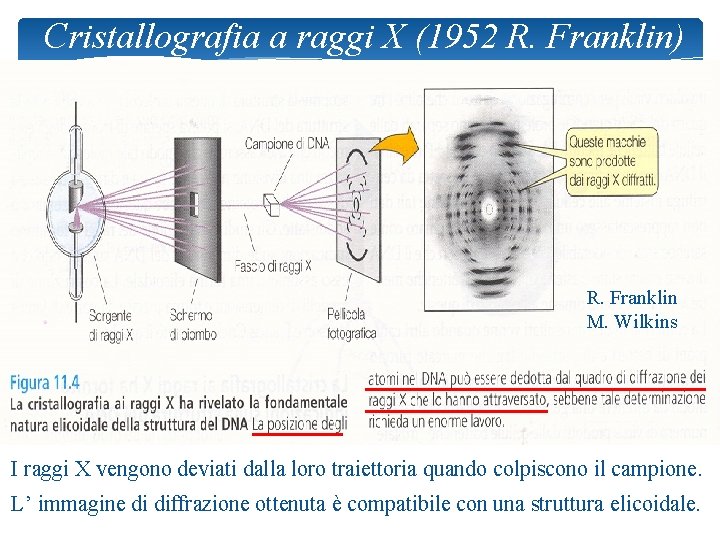
Cristallografia a raggi X (1952 R. Franklin) R. Franklin M. Wilkins I raggi X vengono deviati dalla loro traiettoria quando colpiscono il campione. L’ immagine di diffrazione ottenuta è compatibile con una struttura elicoidale.

Nel 1953 James Watson e Francis Crick, basandosi sulle osservazioni di cristallografia ai raggi X di Rosalind Franklin e sull’osservazione di Erwin Chargaff, propongono il modello a doppia elica per la molecola di DNA 5

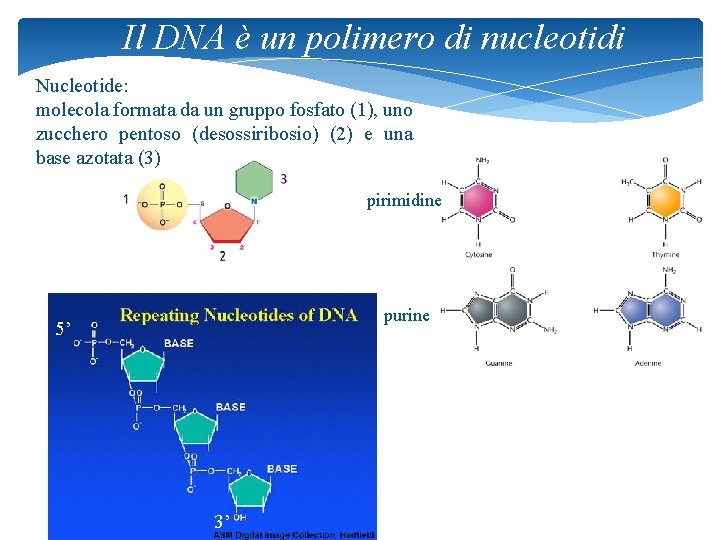
Il DNA è un polimero di nucleotidi Nucleotide: molecola formata da un gruppo fosfato (1), uno zucchero pentoso (desossiribosio) (2) e una base azotata (3) pirimidine purine 5’ 3’

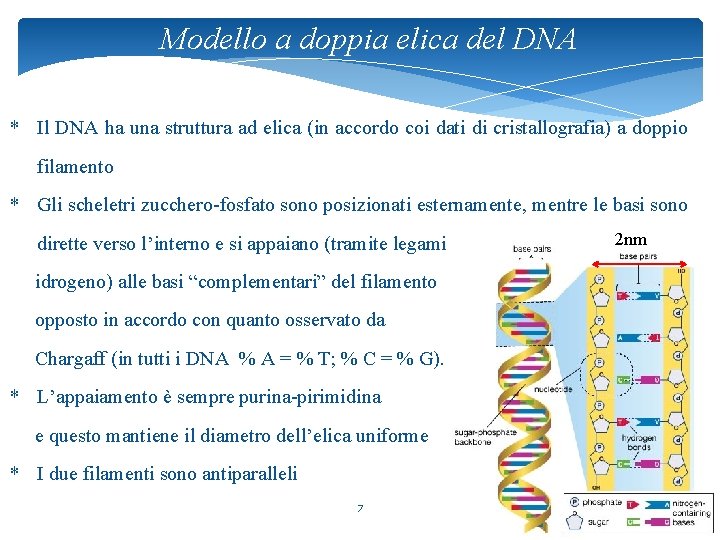
Modello a doppia elica del DNA * Il DNA ha una struttura ad elica (in accordo coi dati di cristallografia) a doppio filamento * Gli scheletri zucchero-fosfato sono posizionati esternamente, mentre le basi sono dirette verso l’interno e si appaiano (tramite legami idrogeno) alle basi “complementari” del filamento opposto in accordo con quanto osservato da Chargaff (in tutti i DNA % A = % T; % C = % G). * L’appaiamento è sempre purina-pirimidina e questo mantiene il diametro dell’elica uniforme * I due filamenti sono antiparalleli 7 2 nm

A quale filamento di DNA si può appaiare la sequenza 5’AAACGCTT 3’ ? 3’TTTGCGAA 5’ a) 5’ TTTGCGAA 3’ NO (orientamento) b) 5’ AAGCGUUU 3’ NO (RNA) c) 5’ UUUGCGAA 3’ NO (RNA e orientamento) d) 5’ AAGCGTTT 3’

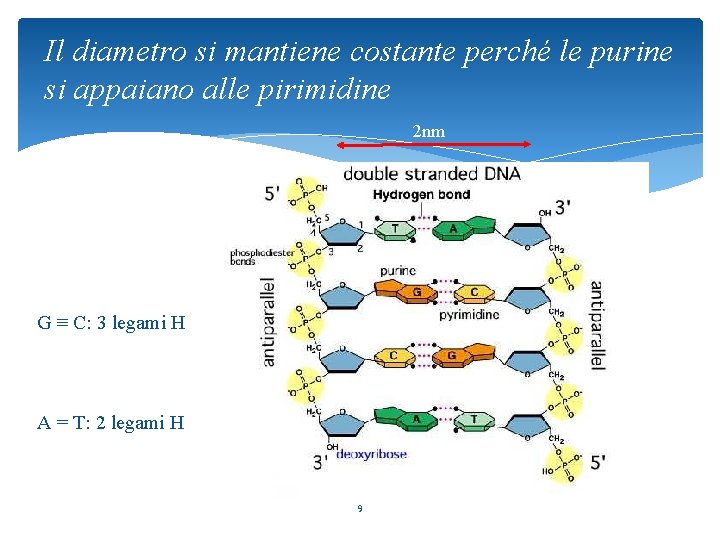
Il diametro si mantiene costante perché le purine si appaiano alle pirimidine 2 nm G ≡ C: 3 legami H A = T: 2 legami H 9

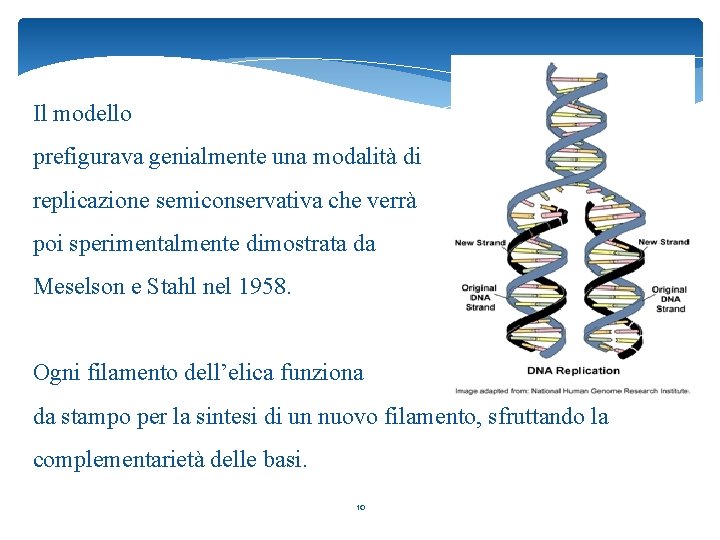
Il modello prefigurava genialmente una modalità di replicazione semiconservativa che verrà poi sperimentalmente dimostrata da Meselson e Stahl nel 1958. Ogni filamento dell’elica funziona da stampo per la sintesi di un nuovo filamento, sfruttando la complementarietà delle basi. 10

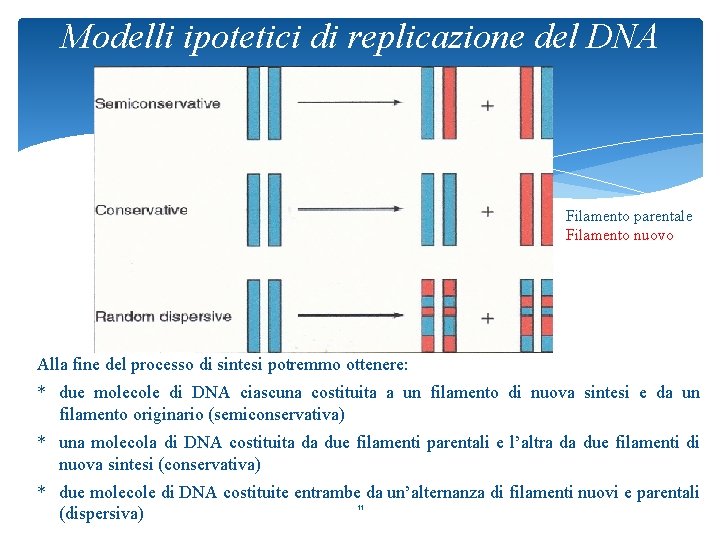
Modelli ipotetici di replicazione del DNA Filamento parentale Filamento nuovo Alla fine del processo di sintesi potremmo ottenere: * due molecole di DNA ciascuna costituita a un filamento di nuova sintesi e da un filamento originario (semiconservativa) * una molecola di DNA costituita da due filamenti parentali e l’altra da due filamenti di nuova sintesi (conservativa) * due molecole di DNA costituite entrambe da un’alternanza di filamenti nuovi e parentali 11 (dispersiva)

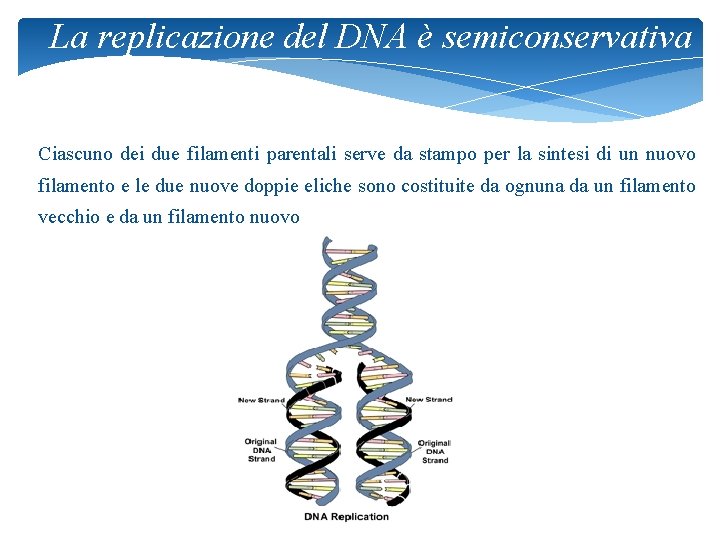
La replicazione del DNA è semiconservativa Ciascuno dei due filamenti parentali serve da stampo per la sintesi di un nuovo filamento e le due nuove doppie eliche sono costituite da ognuna da un filamento vecchio e da un filamento nuovo 12

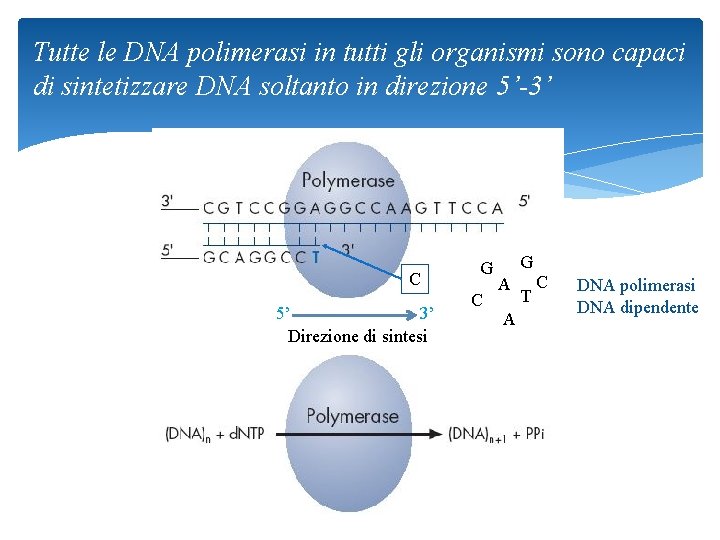
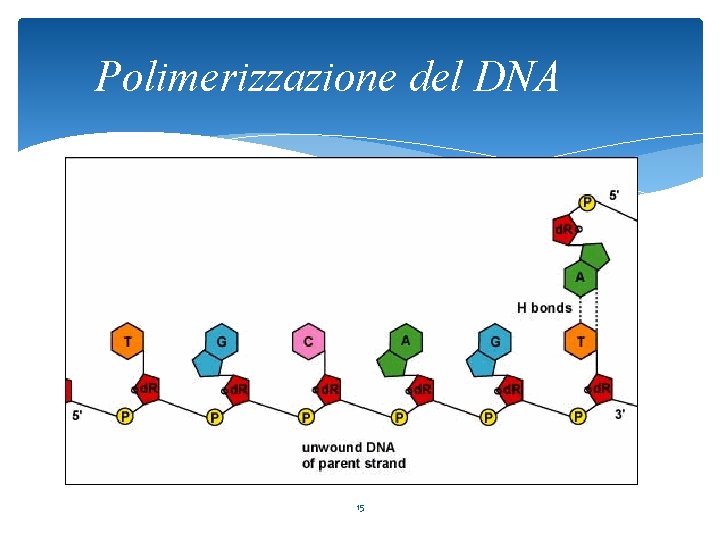
Tutte le DNA polimerasi in tutti gli organismi sono capaci di sintetizzare DNA soltanto in direzione 5’-3’ C 5’ 3’ Direzione di sintesi 13 G C G A A T C DNA polimerasi DNA dipendente

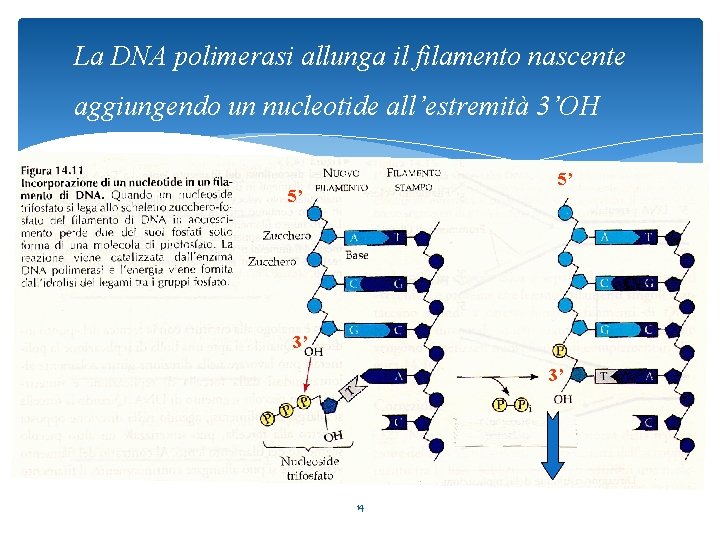
La DNA polimerasi allunga il filamento nascente aggiungendo un nucleotide all’estremità 3’OH 5’ 5’ 3’ 3’ 14

Polimerizzazione del DNA 15

Direzione di sintesi dei due filamenti Poiché estendere le polimerasi solo 5’ 3’ possono l’estremità 3’, mentre entrambi i filamenti servono da stampo per la sintesi di nuovi filamenti, la sintesi deve procedere sui due filamenti stampo in direzione opposta 5’ 3’ 16 5’ 3’

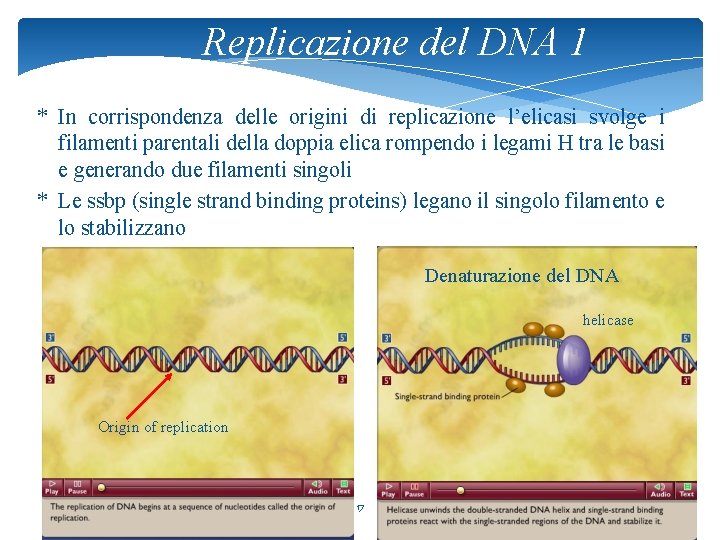
Replicazione del DNA 1 * In corrispondenza delle origini di replicazione l’elicasi svolge i filamenti parentali della doppia elica rompendo i legami H tra le basi e generando due filamenti singoli * Le ssbp (single strand binding proteins) legano il singolo filamento e lo stabilizzano Denaturazione del DNA helicase Origin of replication 17

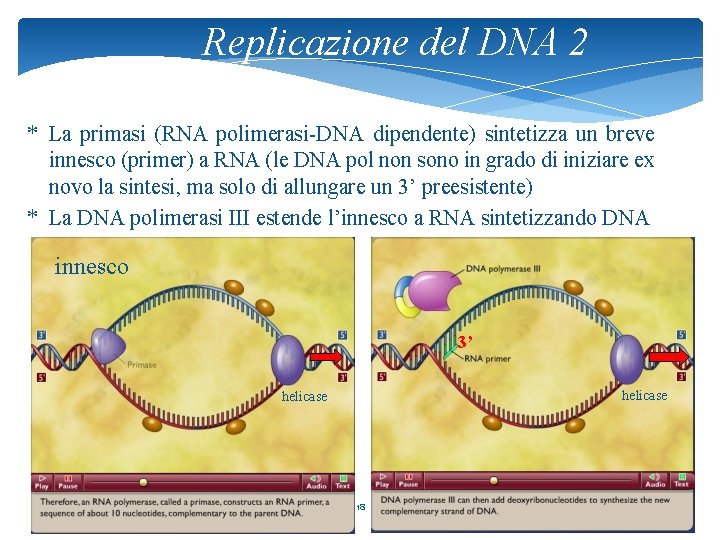
Replicazione del DNA 2 * La primasi (RNA polimerasi-DNA dipendente) sintetizza un breve innesco (primer) a RNA (le DNA pol non sono in grado di iniziare ex novo la sintesi, ma solo di allungare un 3’ preesistente) * La DNA polimerasi III estende l’innesco a RNA sintetizzando DNA innesco 3’ helicase 18

La DNA polimerasi procede, sul filamento guida (leading strand), nella stessa direzione di avanzamento della elicasi e quindi in maniera continua … Ma cosa succede sul filamento complementare? Leading strand helicase 19

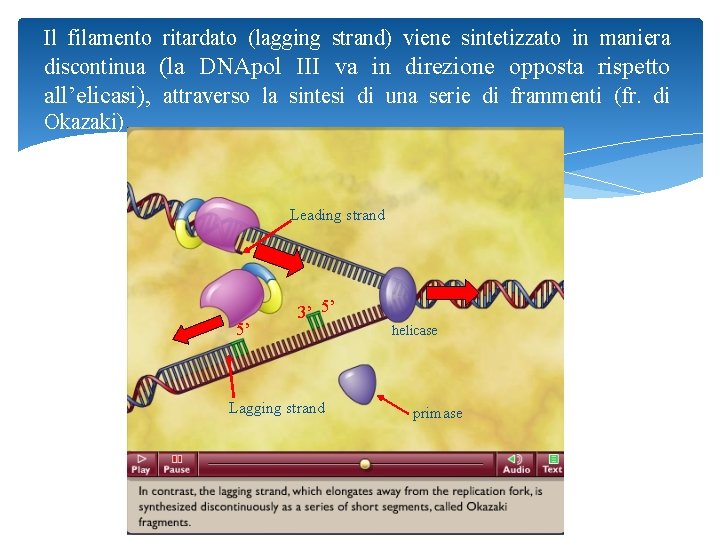
Il filamento ritardato (lagging strand) viene sintetizzato in maniera discontinua (la DNApol III va in direzione opposta rispetto all’elicasi), attraverso la sintesi di una serie di frammenti (fr. di Okazaki) Leading strand 5’ 3’ 5’ helicase Lagging strand primase 20

Replicazione del DNA 3 Rimozione del primer a RNA e “gap filling” da parte della DNA pol I che attacca nucleotidi al 3’ libero del frammento di Okazaki precedente mentre degrada il primer a RNA 5’ helicase 21

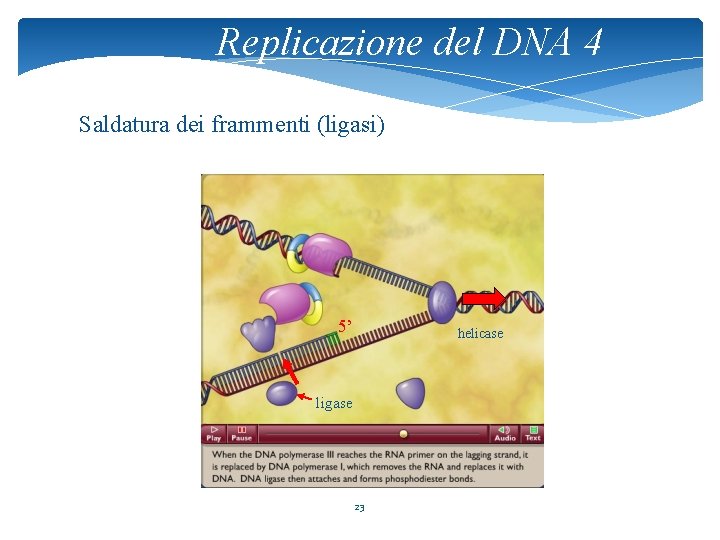
Replicazione del DNA 4 Saldatura dei frammenti (ligasi) 5’ helicase ligase 23

Replicazione del DNA 24

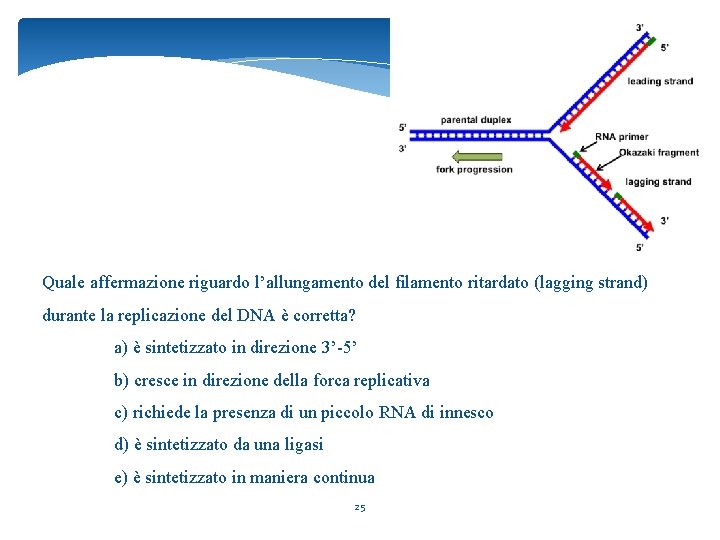
Quale affermazione riguardo l’allungamento del filamento ritardato (lagging strand) durante la replicazione del DNA è corretta? a) è sintetizzato in direzione 3’-5’ b) cresce in direzione della forca replicativa c) richiede la presenza di un piccolo RNA di innesco d) è sintetizzato da una ligasi e) è sintetizzato in maniera continua 25

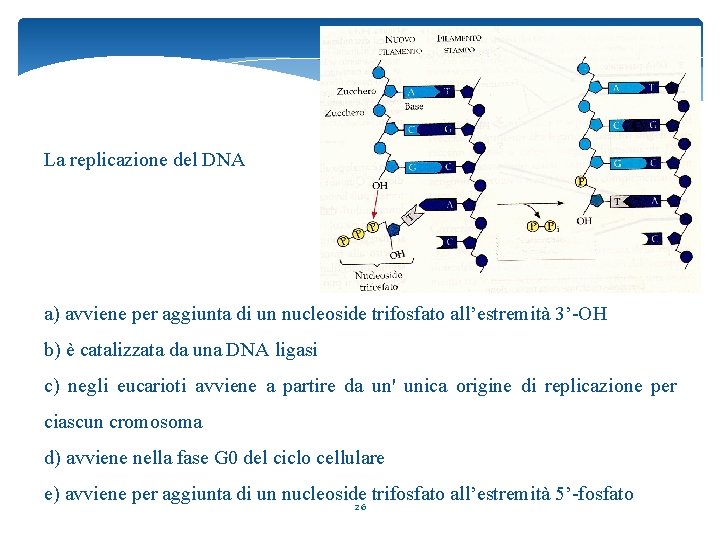
La replicazione del DNA a) avviene per aggiunta di un nucleoside trifosfato all’estremità 3’-OH b) è catalizzata da una DNA ligasi c) negli eucarioti avviene a partire da un' unica origine di replicazione per ciascun cromosoma d) avviene nella fase G 0 del ciclo cellulare e) avviene per aggiunta di un nucleoside trifosfato all’estremità 5’-fosfato 26

La separazione dei filamenti di una struttura elicoidale genera dei superavvolgimenti 27

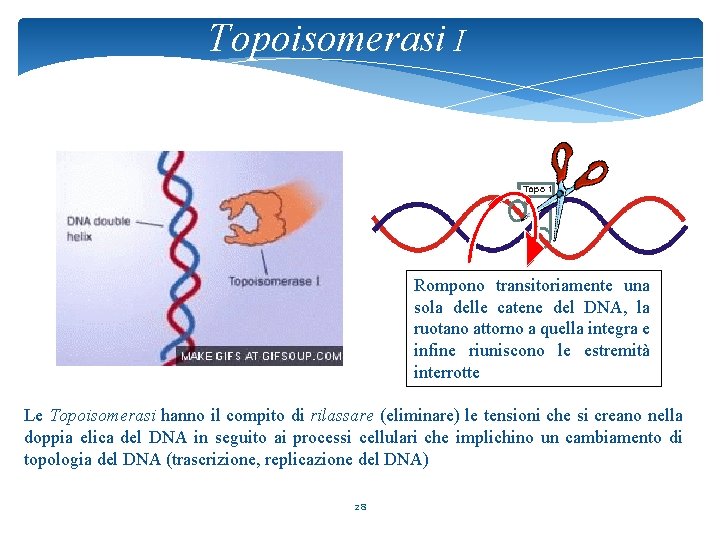
Topoisomerasi I Rompono transitoriamente una sola delle catene del DNA, la ruotano attorno a quella integra e infine riuniscono le estremità interrotte Le Topoisomerasi hanno il compito di rilassare (eliminare) le tensioni che si creano nella doppia elica del DNA in seguito ai processi cellulari che implichino un cambiamento di topologia del DNA (trascrizione, replicazione del DNA) 28

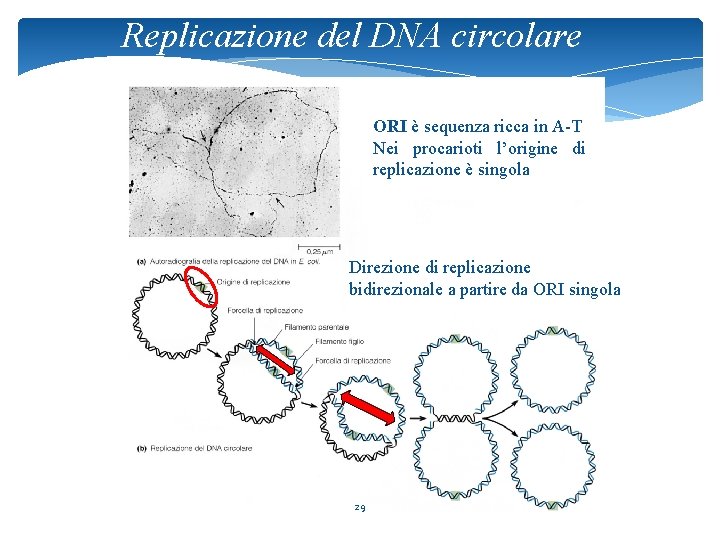
Replicazione del DNA circolare ORI è sequenza ricca in A-T Nei procarioti l’origine di replicazione è singola Direzione di replicazione bidirezionale a partire da ORI singola 29

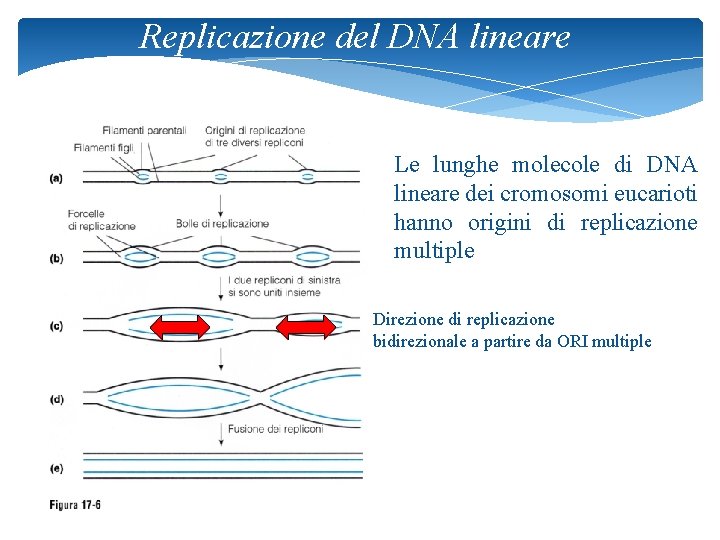
Replicazione del DNA lineare Le lunghe molecole di DNA lineare dei cromosomi eucarioti hanno origini di replicazione multiple Direzione di replicazione bidirezionale a partire da ORI multiple 30

Necessità di ORI multiple negli eucarioti Le lunghe molecole di DNA lineare dei cromosomi eucarioti hanno origini di replicazioni multiple Le DNA polimerasi eucariotiche hanno incorporazione di 75 nucleotidi al secondo una velocità di La lunghezza media di un cromosoma umano è 1. 4 x 108 bp (3. 2 x 109 bp genoma aploide/ 23 cromosomi) Se ci fosse una singola ORI, per duplicare un singolo cromosoma medio occorrerebbero più di 10 giorni In realtà la fase S dura circa 8 ore 31

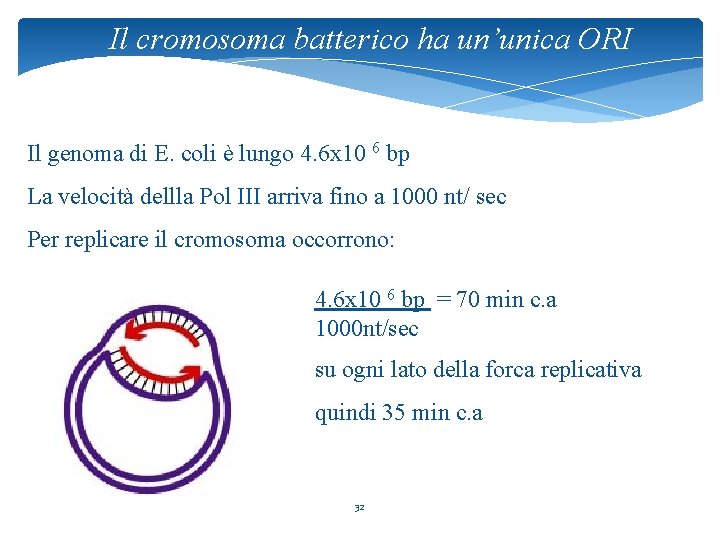
Il cromosoma batterico ha un’unica ORI Il genoma di E. coli è lungo 4. 6 x 10 6 bp La velocità dellla Pol III arriva fino a 1000 nt/ sec Per replicare il cromosoma occorrono: 4. 6 x 10 6 bp = 70 min c. a 1000 nt/sec su ogni lato della forca replicativa quindi 35 min c. a 32

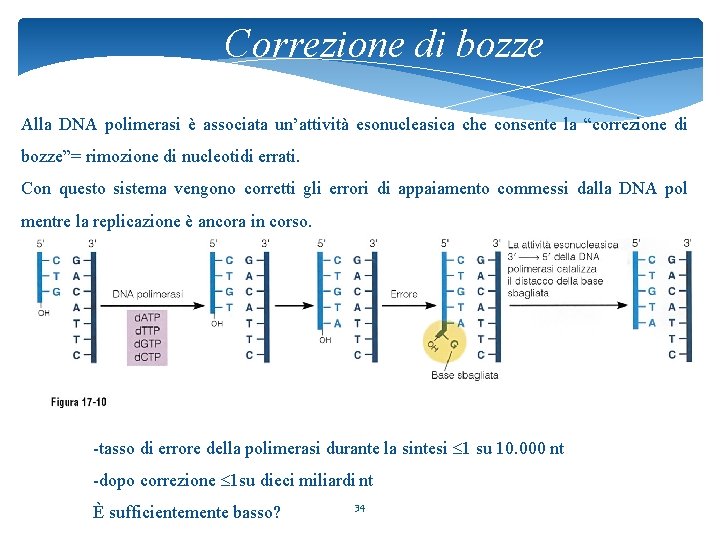
Caratteristiche delle DNA polimerasi * Hanno attività polimerasica SOLO in direzione 5’-3’ * Necessità di innesco a RNA (primer) (non possono iniziare dal nulla la sintesi di una nuova catena) * Attività esonucleasica 3’-5’ correzione di bozze 33

Correzione di bozze Alla DNA polimerasi è associata un’attività esonucleasica che consente la “correzione di bozze”= rimozione di nucleotidi errati. Con questo sistema vengono corretti gli errori di appaiamento commessi dalla DNA pol mentre la replicazione è ancora in corso. -tasso di errore della polimerasi durante la sintesi 1 su 10. 000 nt -dopo correzione 1 su dieci miliardi nt È sufficientemente basso? 34

Tasso di mutazione e correzione degli errori Nel genoma umano (aploide) ci sono c. a. 3 x 109 nt di questi solo 6 x 107 nt (il 2%) sono sequenze codificanti (esoniche) Se non ci fosse correzione di bozze (tasso di errore durante la sintesi 1 x 10 -4 nt): 6000 mutazioni cadrebbero in sequenze codificanti a ogni replicazione del DNA Dopo correzione di bozze (tasso di errore 1 x 10 -10 nt ): 0. 006 mutazioni cadono in sequenze codificanti a ogni replicazione del DNA (1 mutazione ogni 166 replicazioni) Sono comunque maggiori di quelle osservate nella cellula, grazie all’intervento di processi di riparazione del DNA post-replicativi 35

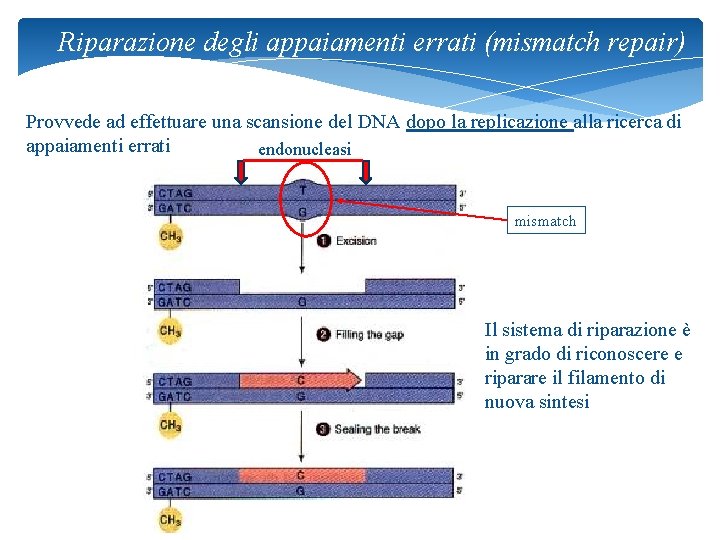
Riparazione degli appaiamenti errati (mismatch repair) Provvede ad effettuare una scansione del DNA dopo la replicazione alla ricerca di appaiamenti errati endonucleasi mismatch Il sistema di riparazione è in grado di riconoscere e riparare il filamento di nuova sintesi 36

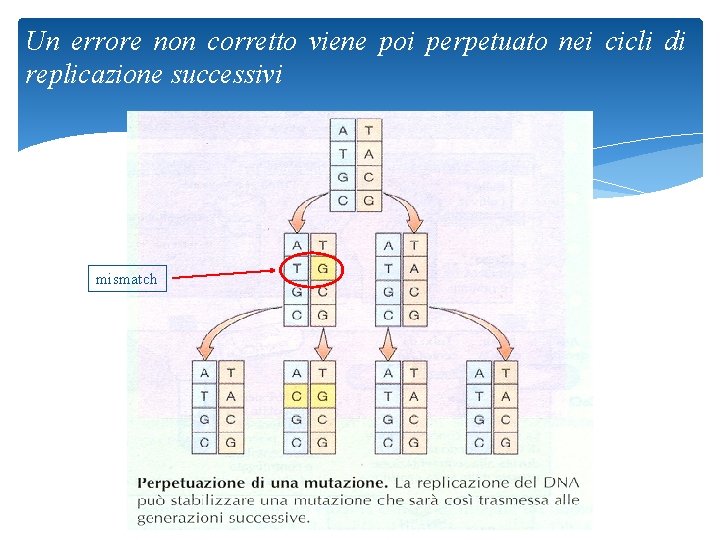
Un errore non corretto viene poi perpetuato nei cicli di replicazione successivi mismatch 37

Cromosomi 38

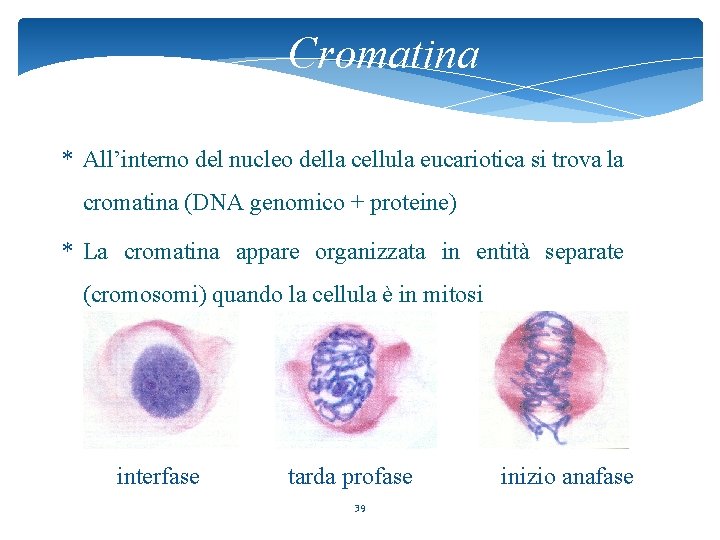
Cromatina * All’interno del nucleo della cellula eucariotica si trova la cromatina (DNA genomico + proteine) * La cromatina appare organizzata in entità separate (cromosomi) quando la cellula è in mitosi interfase tarda profase 39 inizio anafase

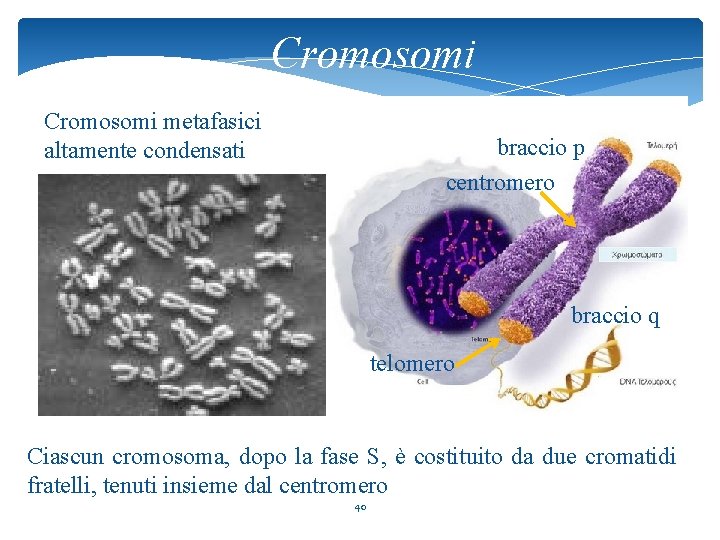
Cromosomi metafasici altamente condensati braccio p centromero braccio q telomero Ciascun cromosoma, dopo la fase S, è costituito da due cromatidi fratelli, tenuti insieme dal centromero 40

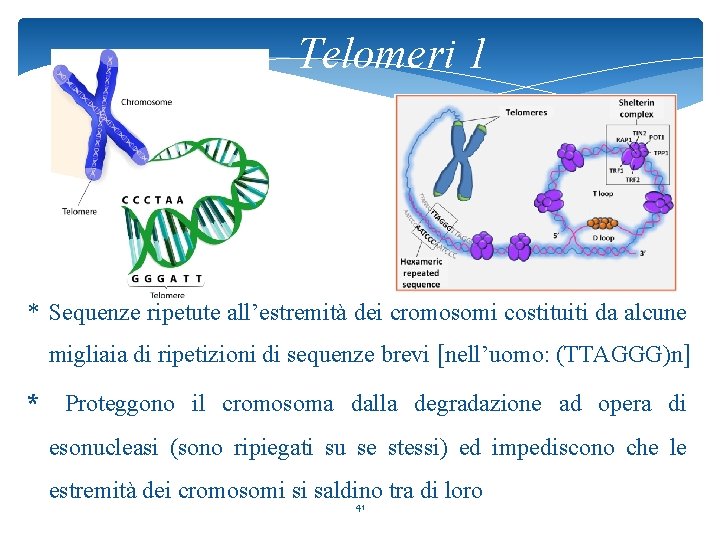
Telomeri 1 * Sequenze ripetute all’estremità dei cromosomi costituiti da alcune migliaia di ripetizioni di sequenze brevi [nell’uomo: (TTAGGG)n] * Proteggono il cromosoma dalla degradazione ad opera di esonucleasi (sono ripiegati su se stessi) ed impediscono che le estremità dei cromosomi si saldino tra di loro 41

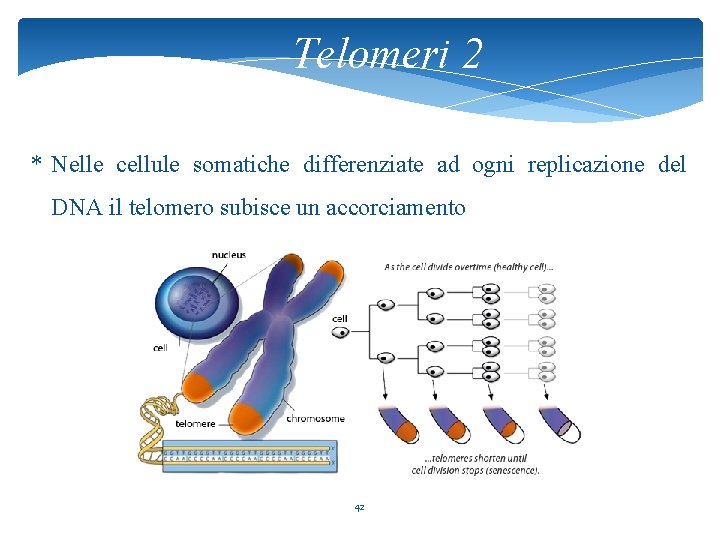
Telomeri 2 * Nelle cellule somatiche differenziate ad ogni replicazione del DNA il telomero subisce un accorciamento 42

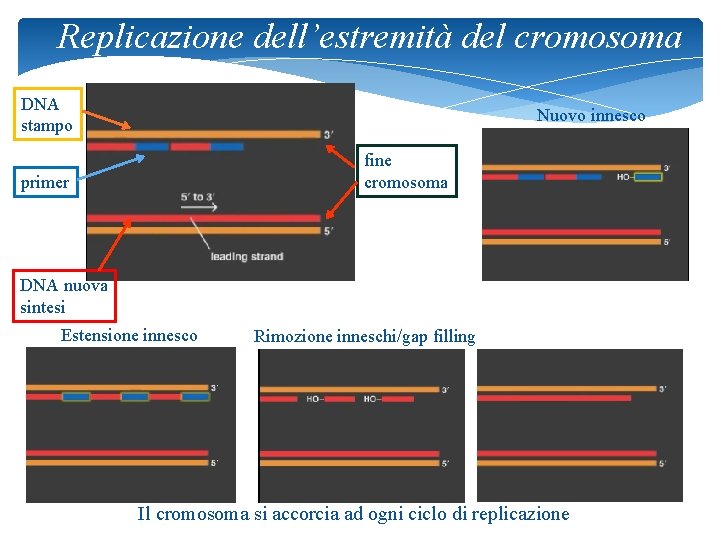
Replicazione dell’estremità del cromosoma DNA stampo Nuovo innesco fine cromosoma primer DNA nuova sintesi Estensione innesco Rimozione inneschi/gap filling Il cromosoma si accorcia ad ogni ciclo di replicazione

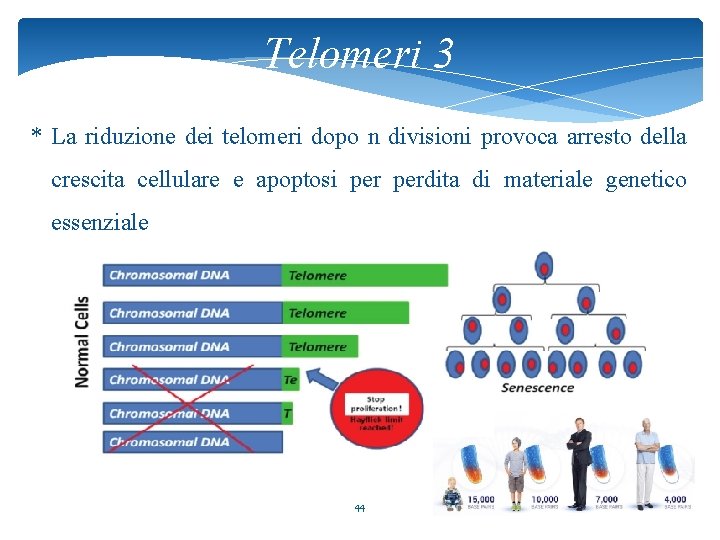
Telomeri 3 * La riduzione dei telomeri dopo n divisioni provoca arresto della crescita cellulare e apoptosi perdita di materiale genetico essenziale 44

Fattori che influenzano la lunghezza dei telomeri Telomeri corti Telomeri lunghi Fumo Alcol Attività fisica Vitamine Stili di vita: Fattori ambientali: Inquinanti atmosferici Pesticidi 45 Arsenico

Telomeri 2 * Nelle cellule germinali, nelle cellule staminali e nelle cellule cancerose la lunghezza dei telomeri rimane costante ad ogni divisione cellulare grazie all’attività dell’enzima telomerasi 46

Centromero * È la regione del cromosoma in cui i cromatidi fratelli sono uniti * Sequenza ripetute * In corrispondenza del centromero ci sono due cinetocori (uno per cromatidio) che servono ad agganciare il cromosoma alle fibre del fuso 47

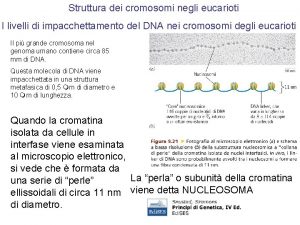
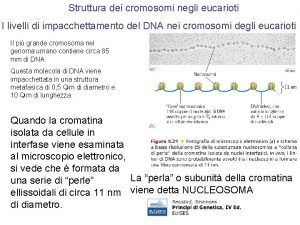
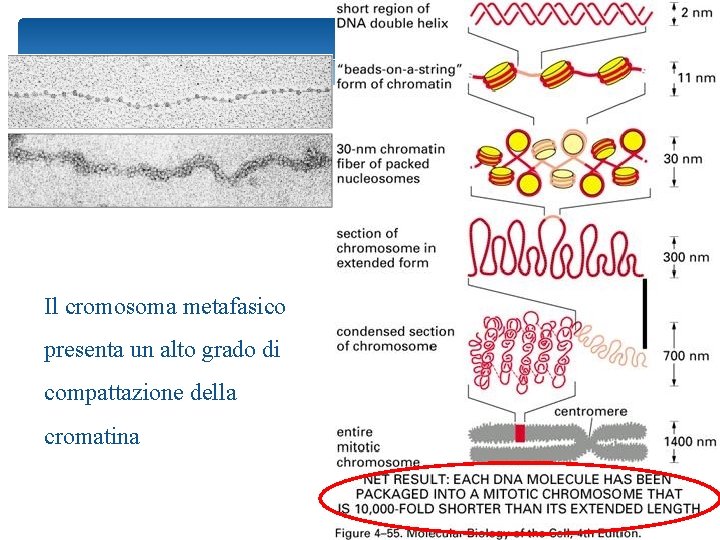
Il DNA genomico degli eucarioti è lunghissimo! * Distanza tra due coppie di basi 0. 34 nm * 3 x 109 bp (numero di basi di un genoma aploide umano) x 0. 34 nm (distanza tra due coppie di basi) = 1 metro (lunghezza di un genoma umano aploide) * Deve essere compattato in un nucleo di pochi m!!! 48

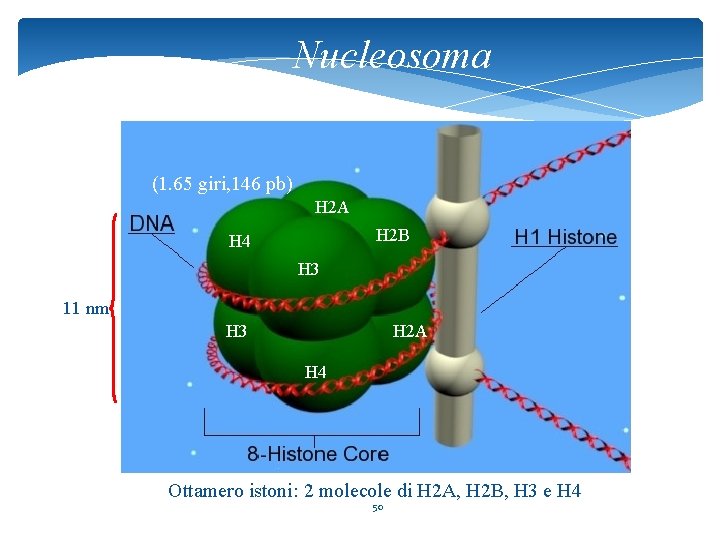
Nucleosoma * L’unità fondamentale della cromatina è il nucleosoma: tratto di DNA (146 bp) avvolto con c. a. due giri attorno a un core proteico formato da un ottamero di proteine istoniche * Istoni: proteine cariche positivamente interagiscono coi gruppi fosfato del DNA 11 nm 2 nm 49 (arg, lys) che

Nucleosoma (1. 65 giri, 146 pb) H 2 A H 2 B H 4 H 3 11 nm H 3 H 2 A H 4 Ottamero istoni: 2 molecole di H 2 A, H 2 B, H 3 e H 4 50

Istone H 1 e fibra da 30 nm * L’istone H 1 (di giunzione) ha la funzione di compattare ulteriormente i nucleosomi * Promuove la formazione della fibra solenoide da 30 nm 11 nm H 1 51

Il cromosoma metafasico presenta un alto grado di compattazione della cromatina 52

Eucromatina-eterocromatina 1 Eterocromatina è più compatta e trascrizionalmente inattiva Eucromatina interfasica trascrizionalmente attiva; si trova allo stato di 53 cromatina distesa ed è accessibile agli enzimi della trascrizione

Eucromatina-eterocromatina 2 Eucromatina: frazione nucleare trascrizionalmente attiva e a bassa densità Eterocromatina: frazione nucleare trascrizionalmente inattiva e densa 54

Cariotipo Il cariotipo di una cellula eucariota è costituito dal numero e dalla morfologia (posizione del centromero e dimensioni) dei suoi cromosomi metafasici Classificazione in base alla posizione del centromero 55

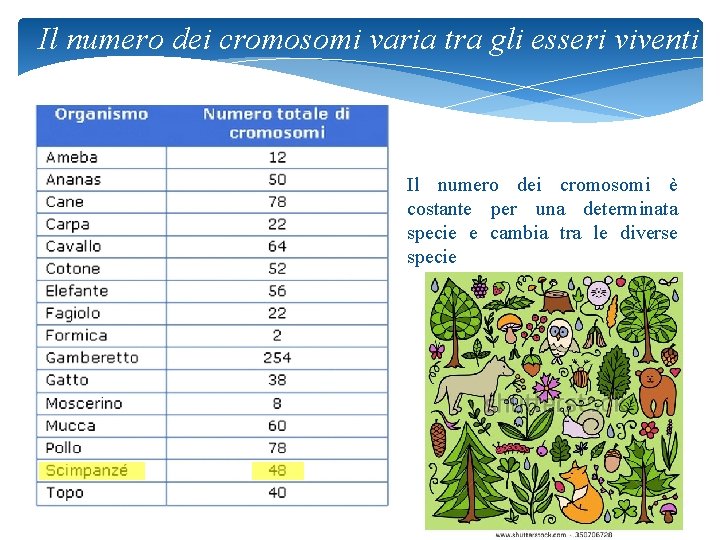
Il numero dei cromosomi varia tra gli esseri viventi Il numero dei cromosomi è costante per una determinata specie e cambia tra le diverse specie

Il cariotipo umano Nel 1955 Tjio e Levan (analizzando centinaia di metafasi in 5 tessuti provenienti da 7 individui) dimostrano che il numero esatto di cromosomi umani è 46

Fusione di cromosomi Tutte le grandi scimmie (bonobo, scimpanzè, gorilla) hanno 48 cromosomi Come mai h. sapiens ne ha 46? C’è stata una riduzione nel numero di cromosomi, dovuto alla fusione di due cromosomi in un nostro antenato 58

Origine evolutiva del cromosoma 2 umano: fusione di cr. 12 e cr. 13 attuali di pan troglodites centromero Sequenze telomeriche centromero * I geni sul cromosoma 2 umano hanno i loro corrispondenti su due piccoli cromosomi presenti nelle grandi scimmie (sono presenti nello stesso ordine). * Nella parte centrale del cromosoma 2 sono presenti sequenze telomeriche. * Il cromosoma 2 ha due centromeri (uno non funzionante) 59

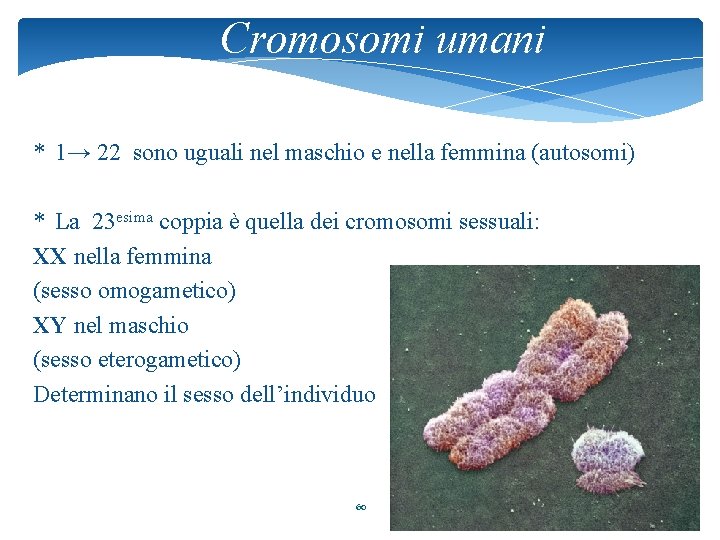
Cromosomi umani * 1→ 22 sono uguali nel maschio e nella femmina (autosomi) * La 23 esima coppia è quella dei cromosomi sessuali: XX nella femmina (sesso omogametico) XY nel maschio (sesso eterogametico) Determinano il sesso dell’individuo 60

Sistema di determinazione del sesso sistema biologico che determina lo sviluppo delle caratteristiche sessuali in un individuo * ambientale es. temperatura (Testudo), CO 2, umidità, fotoperiodo (Gammarus) * genica (asparago: mm Mm ) * Cromosomica (cromosoma X e Y, W e Z negli uccelli) 61

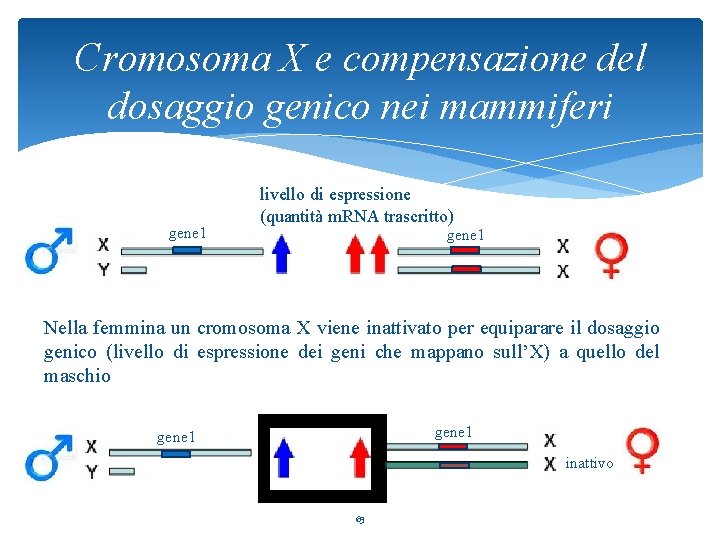
Cromosoma X e cromosoma Y: contenuto genico Cromosoma X 155 Mb c. a. 1100 geni Il cromosoma X contiene molti più geni del cromosoma Y Cromosoma Y 51 Mb c. a. 150 geni 62

Cromosoma X e compensazione del dosaggio genico nei mammiferi gene 1 livello di espressione (quantità m. RNA trascritto) gene 1 Nella femmina un cromosoma X viene inattivato per equiparare il dosaggio genico (livello di espressione dei geni che mappano sull’X) a quello del maschio gene 1 inattivo 63

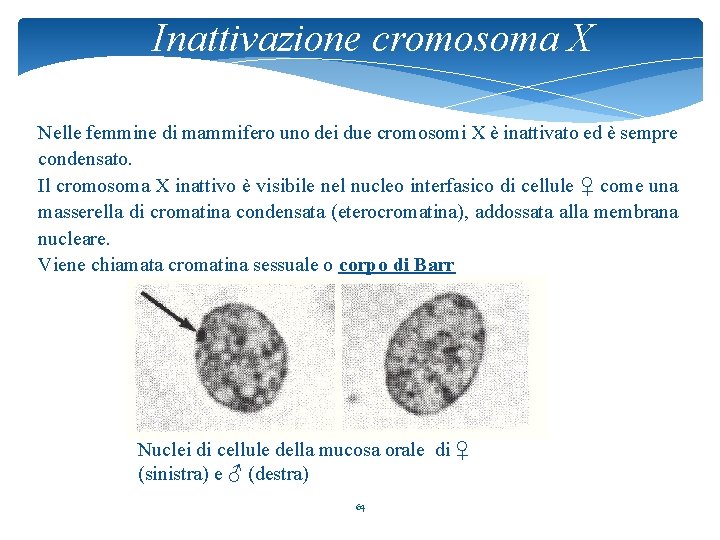
Inattivazione cromosoma X Nelle femmine di mammifero uno dei due cromosomi X è inattivato ed è sempre condensato. Il cromosoma X inattivo è visibile nel nucleo interfasico di cellule ♀ come una masserella di cromatina condensata (eterocromatina), addossata alla membrana nucleare. Viene chiamata cromatina sessuale o corpo di Barr Nuclei di cellule della mucosa orale di ♀ (sinistra) e ♂ (destra) 64
 Replicazione dna
Replicazione dna Sostenibilit
Sostenibilit Alphaherpesvirinae
Alphaherpesvirinae Dna polimerasi teta
Dna polimerasi teta Replicazione a circolo rotante
Replicazione a circolo rotante Legge di mendel occhi
Legge di mendel occhi Cromosomi omologhi
Cromosomi omologhi Politenici
Politenici Cromosomi omologhi
Cromosomi omologhi Cromosomi politenici
Cromosomi politenici Complesso sinaptinemale
Complesso sinaptinemale Insieme di cromosomi
Insieme di cromosomi Cromosoma philadelphia
Cromosoma philadelphia Regole di chargaff zanichelli
Regole di chargaff zanichelli Insieme di cromosomi
Insieme di cromosomi Cromosomi monocromatidici e dicromatidici
Cromosomi monocromatidici e dicromatidici Doppio filamento dna
Doppio filamento dna Struttura del dna
Struttura del dna Bioflix activity dna replication nucleotide pairing
Bioflix activity dna replication nucleotide pairing Coding dna and non coding dna
Coding dna and non coding dna Enzyme involved in dna replication
Enzyme involved in dna replication Function of dna polymerase 3
Function of dna polymerase 3 Dna rna protein synthesis homework #2 dna replication
Dna rna protein synthesis homework #2 dna replication Jigsaw esempio scuola primaria
Jigsaw esempio scuola primaria Esempio di lezione clil diritto
Esempio di lezione clil diritto L'esperienza delle cose moderne e la lezione delle antique
L'esperienza delle cose moderne e la lezione delle antique Italian articles
Italian articles L'esperienza delle cose moderne e la lezione delle antique
L'esperienza delle cose moderne e la lezione delle antique Lezione 7
Lezione 7 Esempio di lezione simulata letteratura italiana
Esempio di lezione simulata letteratura italiana La lezione della farfalla.com
La lezione della farfalla.com Struttura del testo teatrale
Struttura del testo teatrale Formula di struttura alcani
Formula di struttura alcani Schema testo narrativo
Schema testo narrativo Tempo nel testo narrativo
Tempo nel testo narrativo Schema lettera commerciale
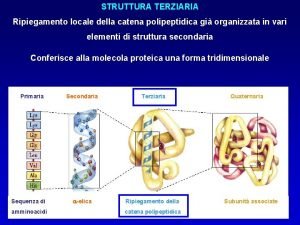
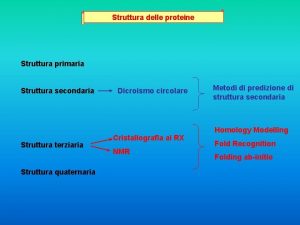
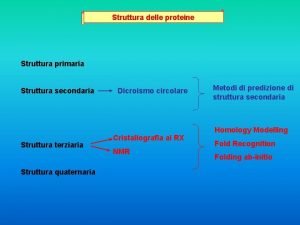
Schema lettera commerciale Struttura terziaria proteine
Struttura terziaria proteine Struttura business plan
Struttura business plan Struttura sociale
Struttura sociale Fondi next generation eu
Fondi next generation eu Dot
Dot Struttura a dittico sofocle
Struttura a dittico sofocle Struttura del computer
Struttura del computer Struttura nativa
Struttura nativa Saggio letterario esempio
Saggio letterario esempio Anacronie
Anacronie Protoxilema
Protoxilema Romanzo struttura
Romanzo struttura Struttura secondaria rna
Struttura secondaria rna Analessi e prolessi
Analessi e prolessi Organigramma radiale
Organigramma radiale La filastrocca è un testo poetico
La filastrocca è un testo poetico Fiaba struttura
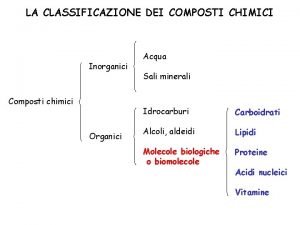
Fiaba struttura Classificazione degli elementi
Classificazione degli elementi Pianta della basilica romana
Pianta della basilica romana Polis struttura
Polis struttura Struttura compositiva
Struttura compositiva Struttura dei carboidrati
Struttura dei carboidrati Differenza tra statuto albertino e costituzione
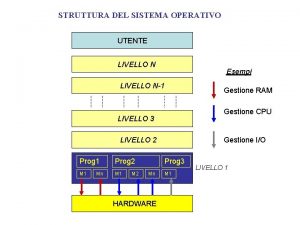
Differenza tra statuto albertino e costituzione Sistema monoprogrammato
Sistema monoprogrammato Come scrivere un tema argomentativo
Come scrivere un tema argomentativo Histoplasma
Histoplasma Giuseppe valitutti
Giuseppe valitutti Struttura divina commedia mappa concettuale
Struttura divina commedia mappa concettuale