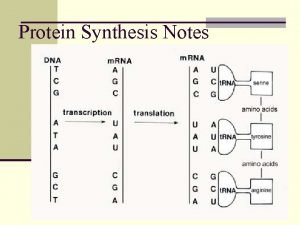
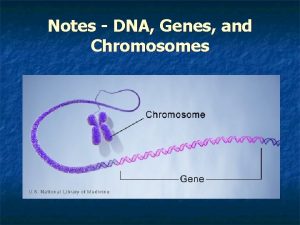
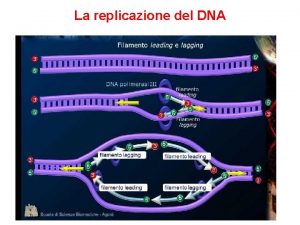
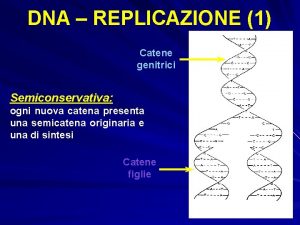
Riparazione del DNA Errori di replicazione del DNA























- Slides: 66

Riparazione del DNA

Errori di replicazione del DNA Meccanismi di riparazione di errori di replicazione Danni al DNA Meccanismi di riparazione del danno al DNA


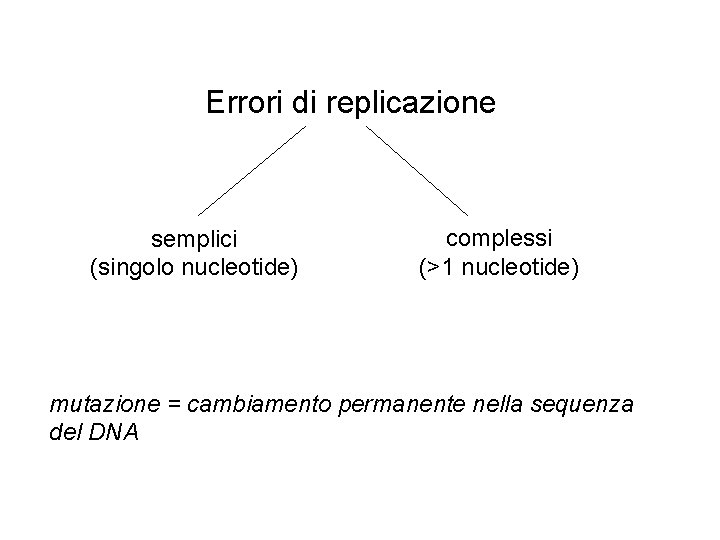
Errori di replicazione semplici (singolo nucleotide) complessi (>1 nucleotide) mutazione = cambiamento permanente nella sequenza del DNA

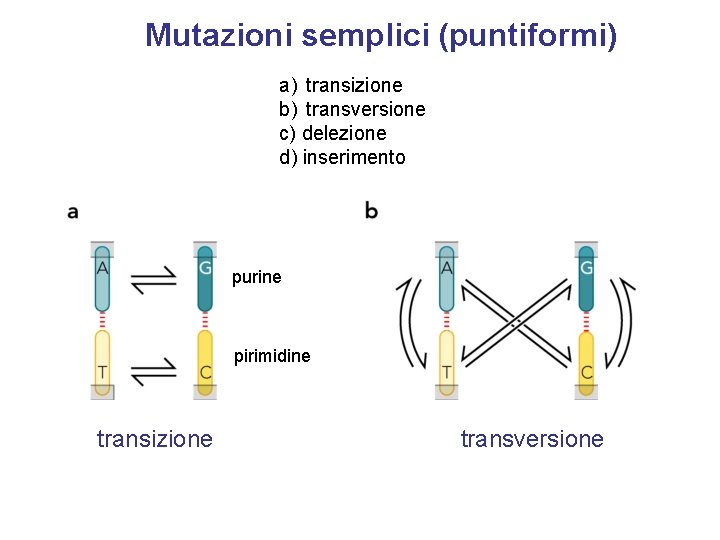
Mutazioni semplici (puntiformi) a) transizione b) transversione c) delezione d) inserimento purine pirimidine transizione transversione

Due linee di difesa contro gli errori di replicazione L’attivita’ di proofreading (correzione delle bozze) aumenta la fedelta’ della replicazione di un fattore di 100 Il sistema di mismatch repair aumenta la fedelta’ della replicazione di > 2 ordini di grandezza Dopo la replicazione un errore, se non riparato subito, puo’fissarsi definitivamente nel DNA

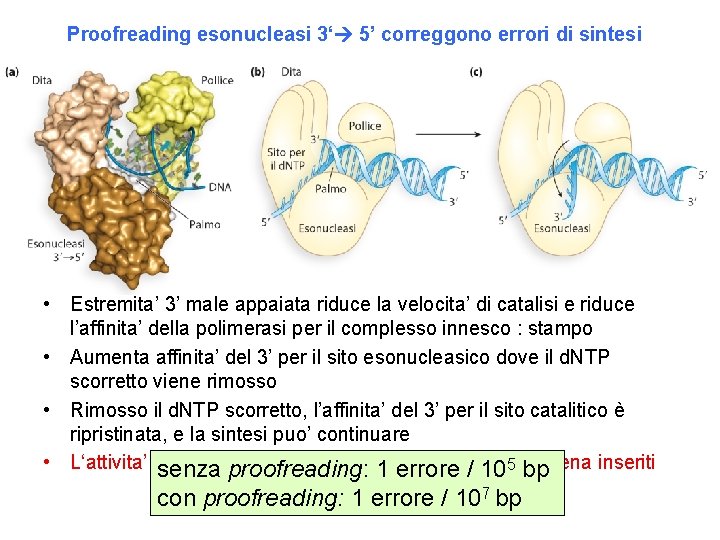
Proofreading esonucleasi 3‘ 5’ correggono errori di sintesi • Estremita’ 3’ male appaiata riduce la velocita’ di catalisi e riduce l’affinita’ della polimerasi per il complesso innesco : stampo • Aumenta affinita’ del 3’ per il sito esonucleasico dove il d. NTP scorretto viene rimosso • Rimosso il d. NTP scorretto, l’affinita’ del 3’ per il sito catalitico è ripristinata, e la sintesi puo’ continuare • L‘attivita’ esonucleasica puo’ solo correggere appena inseriti senza proofreading: 1 errore /errori 105 bp con proofreading: 1 errore / 107 bp

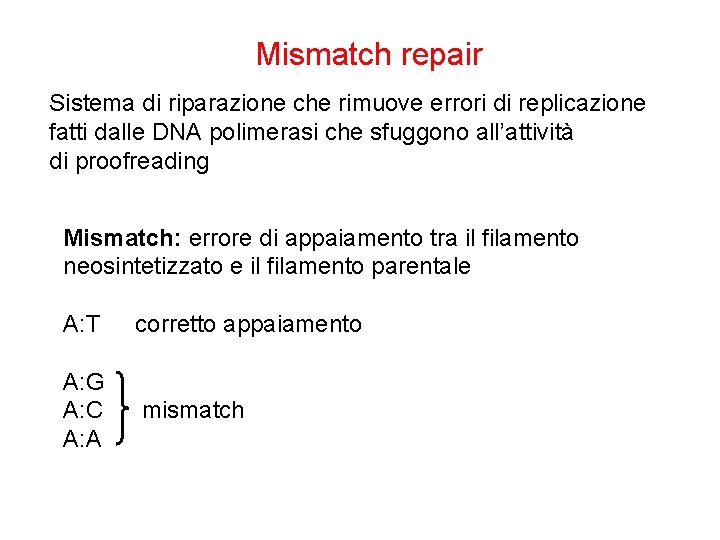
Mismatch repair Sistema di riparazione che rimuove errori di replicazione fatti dalle DNA polimerasi che sfuggono all’attività di proofreading Mismatch: errore di appaiamento tra il filamento neosintetizzato e il filamento parentale A: T corretto appaiamento A: G A: C A: A mismatch

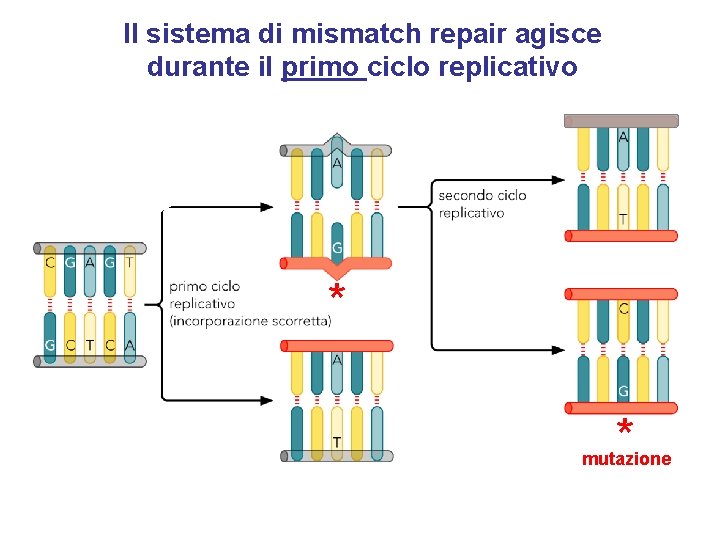
Il sistema di mismatch repair agisce durante il primo ciclo replicativo * * mutazione

Compiti del sistema mismatch repair: -sorvegliare il genoma alla ricerca di errori di appaiamento -trovare gli appaiamenti scorretti -identificare quale filamento e’ da riparare -sostituire il nucleotide scorretto

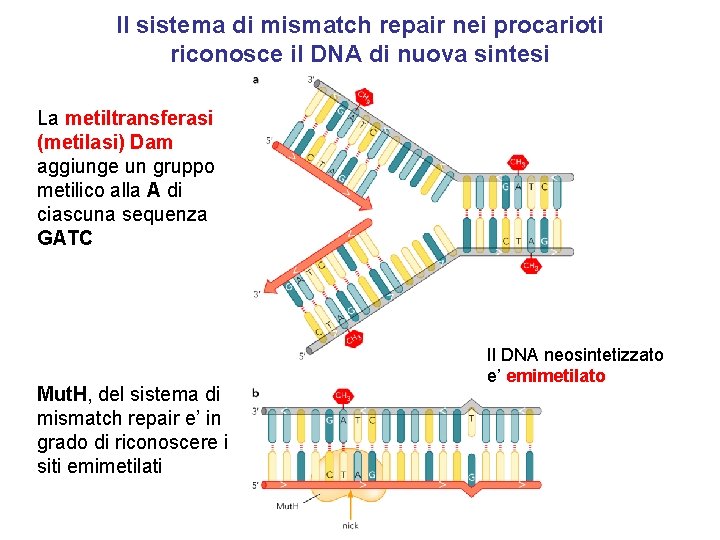
Il sistema di mismatch repair nei procarioti riconosce il DNA di nuova sintesi La metiltransferasi (metilasi) Dam aggiunge un gruppo metilico alla A di ciascuna sequenza GATC Mut. H, del sistema di mismatch repair e’ in grado di riconoscere i siti emimetilati Il DNA neosintetizzato e’ emimetilato

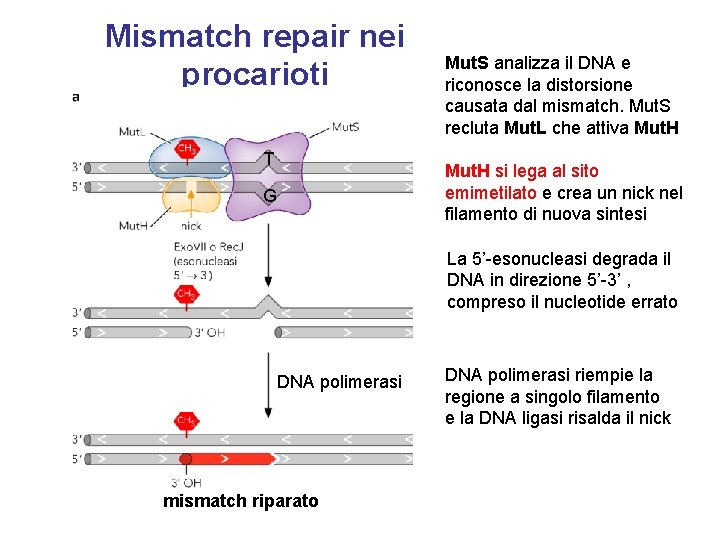
Mismatch repair nei procarioti T Mut. S analizza il DNA e riconosce la distorsione causata dal mismatch. Mut. S recluta Mut. L che attiva Mut. H si lega al sito emimetilato e crea un nick nel filamento di nuova sintesi G La 5’-esonucleasi degrada il DNA in direzione 5’-3’ , compreso il nucleotide errato DNA polimerasi mismatch riparato DNA polimerasi riempie la regione a singolo filamento e la DNA ligasi risalda il nick

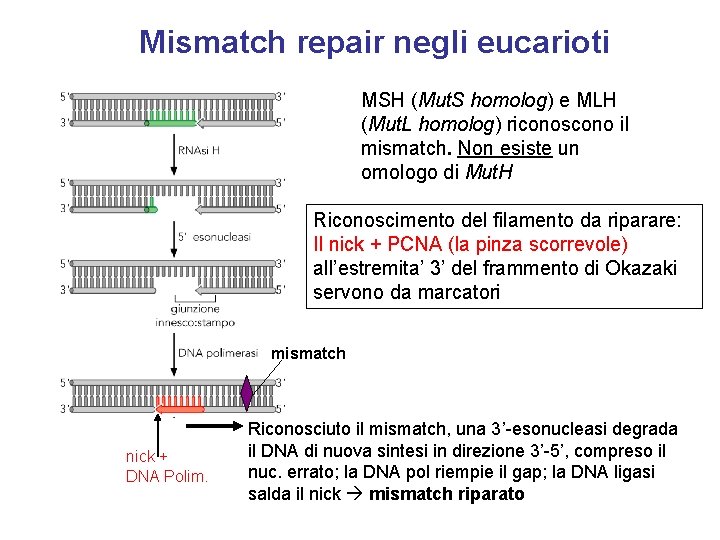
Mismatch repair negli eucarioti MSH (Mut. S homolog) e MLH (Mut. L homolog) riconoscono il mismatch. Non esiste un omologo di Mut. H Riconoscimento del filamento da riparare: Il nick + PCNA (la pinza scorrevole) all’estremita’ 3’ del frammento di Okazaki servono da marcatori mismatch nick + DNA Polim. Riconosciuto il mismatch, una 3’-esonucleasi degrada il DNA di nuova sintesi in direzione 3’-5’, compreso il nuc. errato; la DNA pol riempie il gap; la DNA ligasi salda il nick mismatch riparato

La proofreading corregge errori durante la sintesi del nuovo filamento di DNA Il sistema di mismatch repair corregge errori dopo la sintesi e durante il primo ciclo di replicazione

Mutazioni complesse Grossi riarrangiamenti cromosomici (delezioni, inserzioni, duplicazioni) - presenza di DNA microsatellite (CACA; CCGCCGCCG) - funzionamento aberrante della ricombinazione omologa Lunghe inserzioni: - Elementi mobili, chiamati trasposoni, possono introdurre migliaia di nucleotidi estranei nelle regioni codificanti o regolative di un gene Le mutazioni complesse non possono essere riparate

Errori di replicazione del DNA Meccanismi di riparazione di errori di replicazione Danni al DNA Meccanismi di riparazione del danno al DNA

Possibili cause di danno al DNA • sostanze chimiche ambientali • sostanze fisiche; radiazione (UV) • difetti dei geni che codificano proteine coinvolte nei meccanismi di riparazione

Il danno al DNA puo’ avvenire spontaneamente in seguito a: - idrolisi (depurinazione, deaminazione) - alchilazione - ossidazione - irradiazione

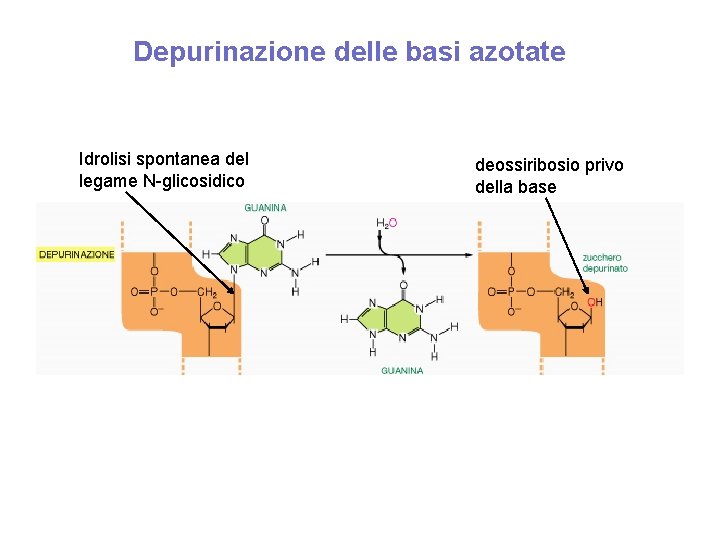
Depurinazione delle basi azotate Idrolisi spontanea del legame N-glicosidico deossiribosio privo della base

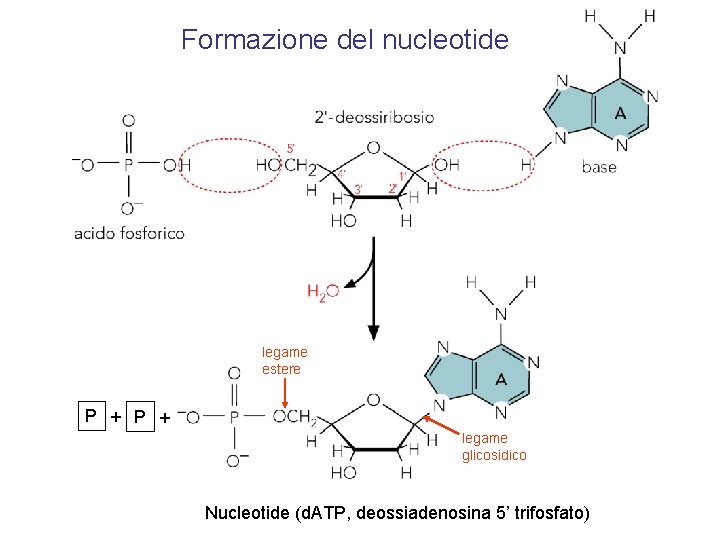
Formazione del nucleotide legame estere P + legame glicosidico Nucleotide (d. ATP, deossiadenosina 5’ trifosfato)

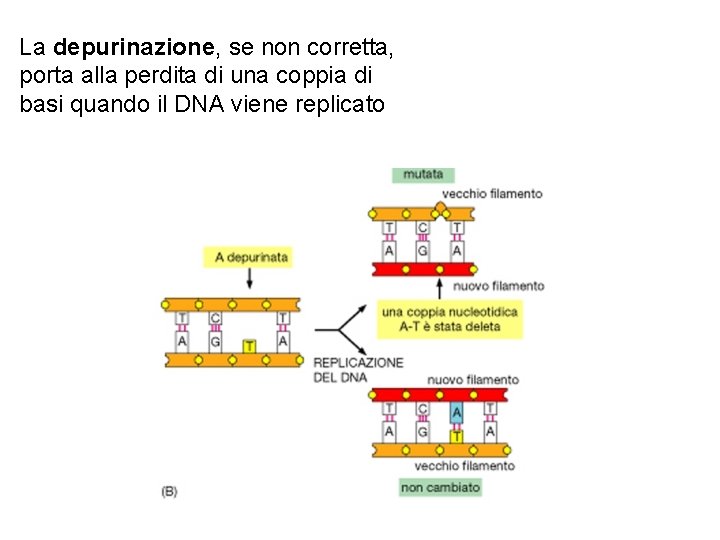
La depurinazione, se non corretta, porta alla perdita di una coppia di basi quando il DNA viene replicato

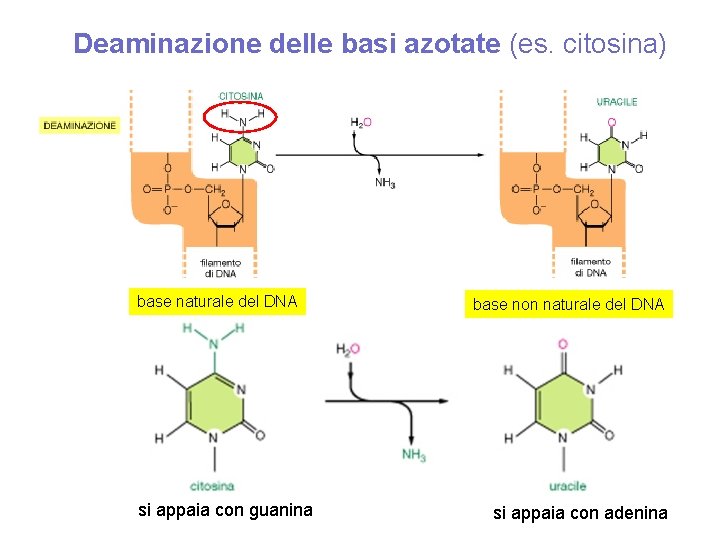
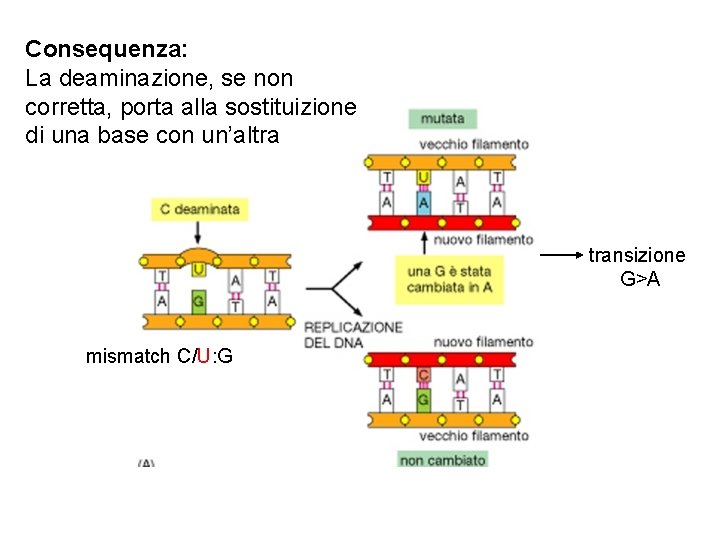
Deaminazione delle basi azotate (es. citosina) base naturale del DNA si appaia con guanina base non naturale del DNA si appaia con adenina

Consequenza: La deaminazione, se non corretta, porta alla sostituizione di una base con un’altra transizione G>A mismatch C/U: G

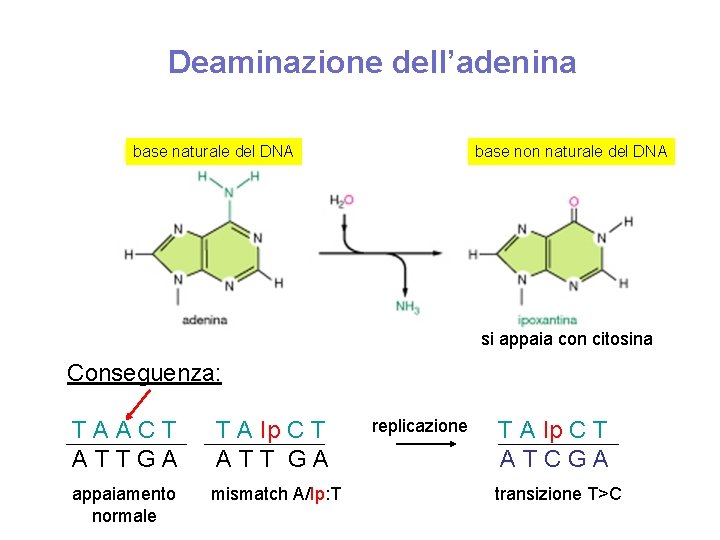
Deaminazione dell’adenina base naturale del DNA base non naturale del DNA si appaia con citosina Conseguenza: TAACT ATTGA T A Ip C T ATT GA appaiamento normale mismatch A/Ip: T replicazione T A Ip C T ATCGA transizione T>C

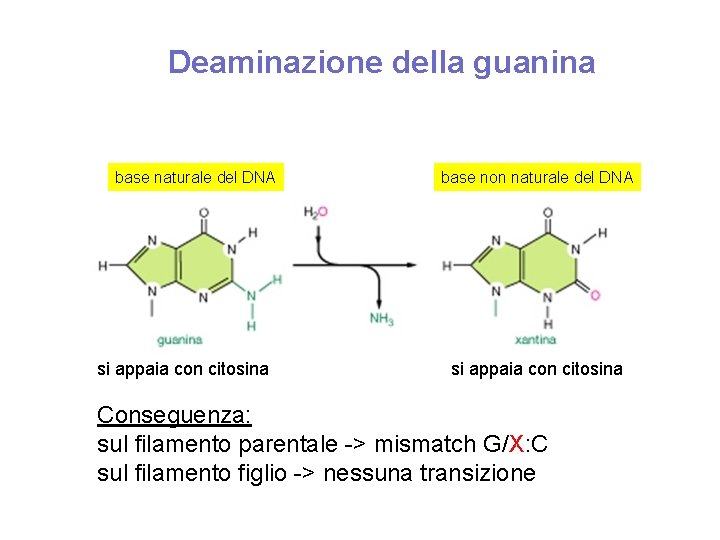
Deaminazione della guanina base naturale del DNA si appaia con citosina base non naturale del DNA si appaia con citosina Conseguenza: sul filamento parentale -> mismatch G/X: C sul filamento figlio -> nessuna transizione

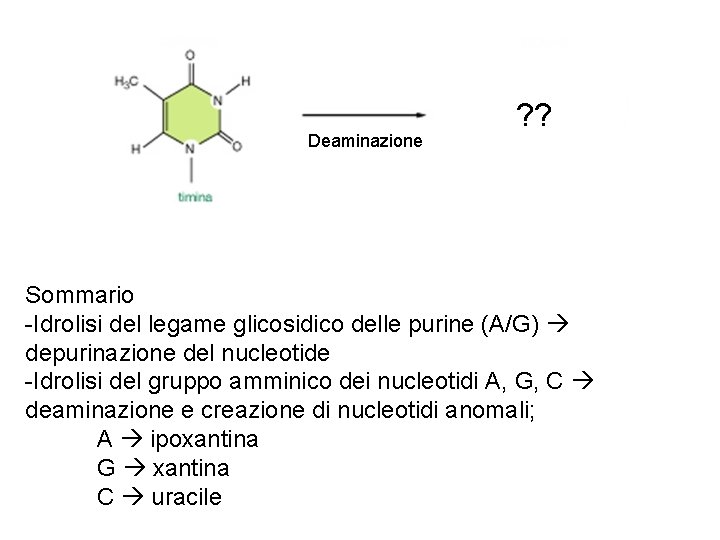
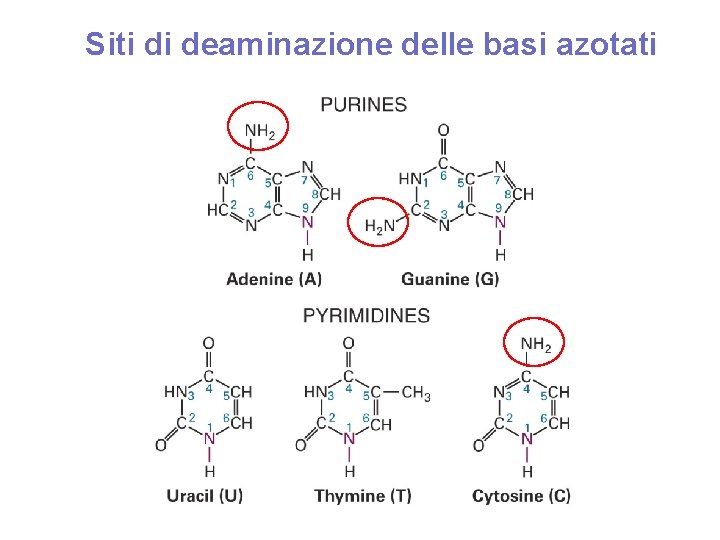
Deaminazione ? ? Sommario -Idrolisi del legame glicosidico delle purine (A/G) depurinazione del nucleotide -Idrolisi del gruppo amminico dei nucleotidi A, G, C deaminazione e creazione di nucleotidi anomali; A ipoxantina G xantina C uracile

Siti di deaminazione delle basi azotati

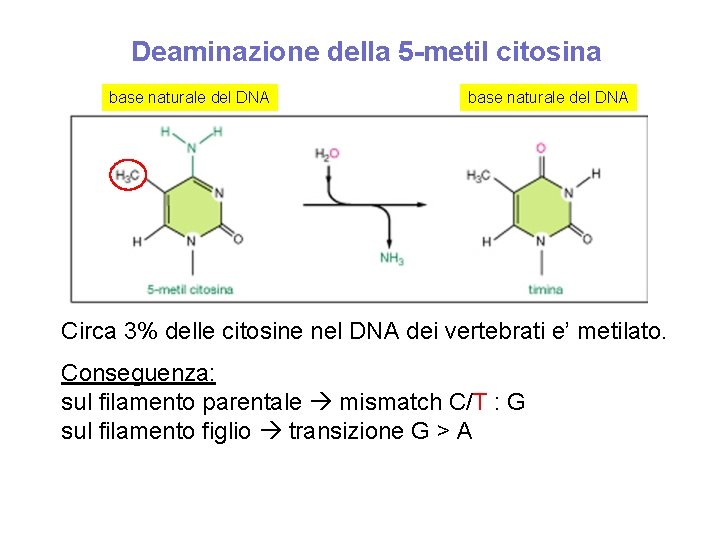
La depurinazione e la deaminazione generano delle basi non naturali del DNA che possono essere riconosciute dai sistemi di riparazione del DNA Se il prodotto della mutazione e’ una base naturale, i sistemi di riparazione non possono riconoscere l’errore Esempio: la deaminazione della 5 -metil citosina La 5 -metil citosina e’ il prodotto dell’attivata’ della DNA metiltransferasi, enzima importante per la regolazione genica

Deaminazione della 5 -metil citosina base naturale del DNA Circa 3% delle citosine nel DNA dei vertebrati e’ metilato. Conseguenza: sul filamento parentale mismatch C/T : G sul filamento figlio transizione G > A

Il danno al DNA puo’ avvenire spontaneamente in seguito a: - idrolisi (depurinazione, deaminazione) - alchilazione - ossidazione - irradiazione

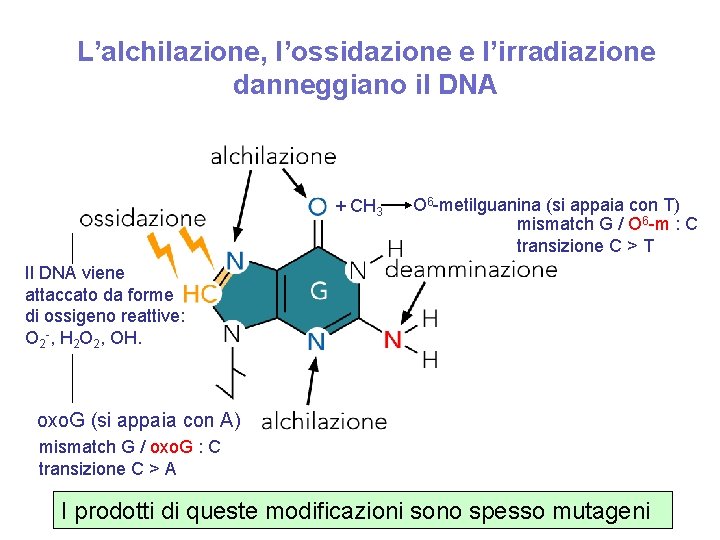
L’alchilazione, l’ossidazione e l’irradiazione danneggiano il DNA + CH 3 O 6 -metilguanina (si appaia con T) mismatch G / O 6 -m : C transizione C > T Il DNA viene attaccato da forme di ossigeno reattive: O 2 -, H 2 O 2, OH. oxo. G (si appaia con A) mismatch G / oxo. G : C transizione C > A I prodotti di queste modificazioni sono spesso mutageni

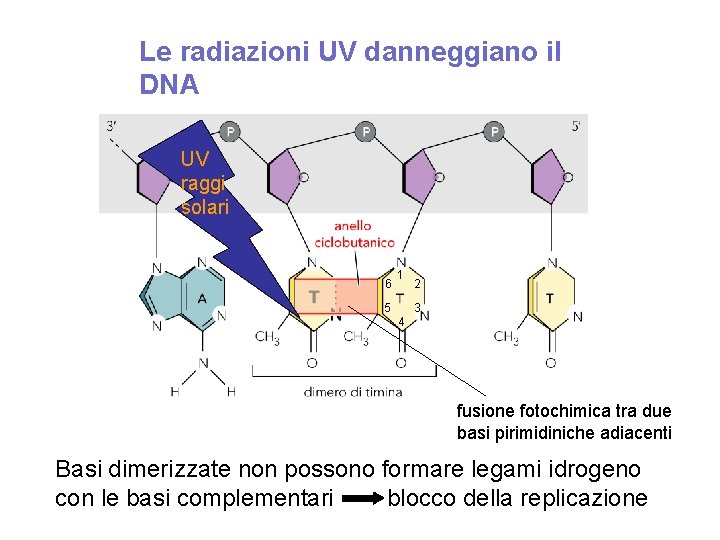
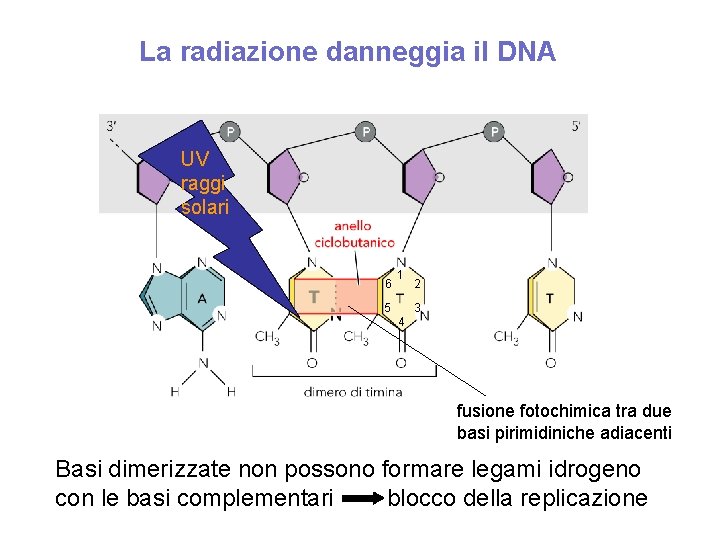
Le radiazioni UV danneggiano il DNA UV raggi solari 6 1 2 3 5 4 fusione fotochimica tra due basi pirimidiniche adiacenti Basi dimerizzate non possono formare legami idrogeno con le basi complementari blocco della replicazione

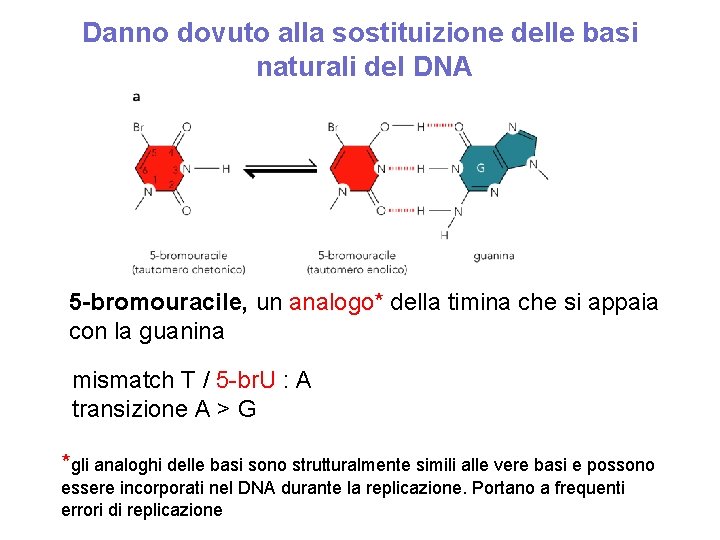
Danno dovuto alla sostituizione delle basi naturali del DNA 5 -bromouracile, un analogo* della timina che si appaia con la guanina mismatch T / 5 -br. U : A transizione A > G *gli analoghi delle basi sono strutturalmente simili alle vere basi e possono essere incorporati nel DNA durante la replicazione. Portano a frequenti errori di replicazione

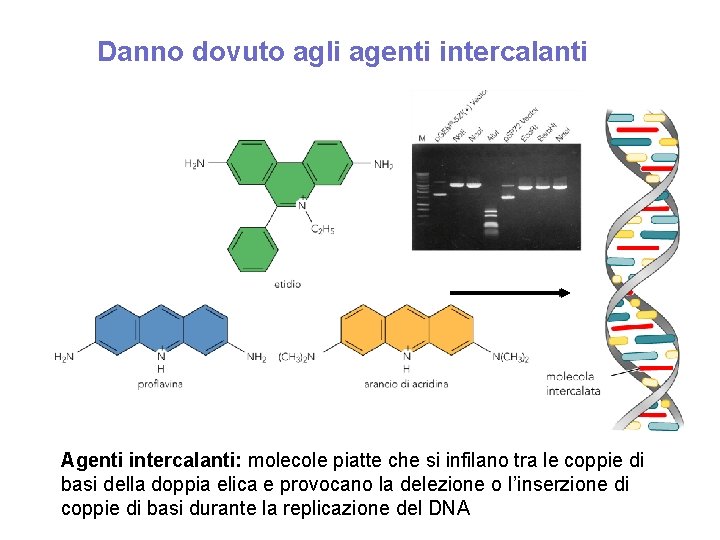
Danno dovuto agli agenti intercalanti Agenti intercalanti: molecole piatte che si infilano tra le coppie di basi della doppia elica e provocano la delezione o l’inserzione di coppie di basi durante la replicazione del DNA

Modificazioni che producono basi non naturali del DNA possono essere riconosciute dai sistemi di riparazione. Modificazioni che producono basi naturali o basi analoghe non vengono riconoscuite e dopo la replicazione del DNA le mutazioni vengono fissate in una permanente alterazione nella sequenza del DNA.

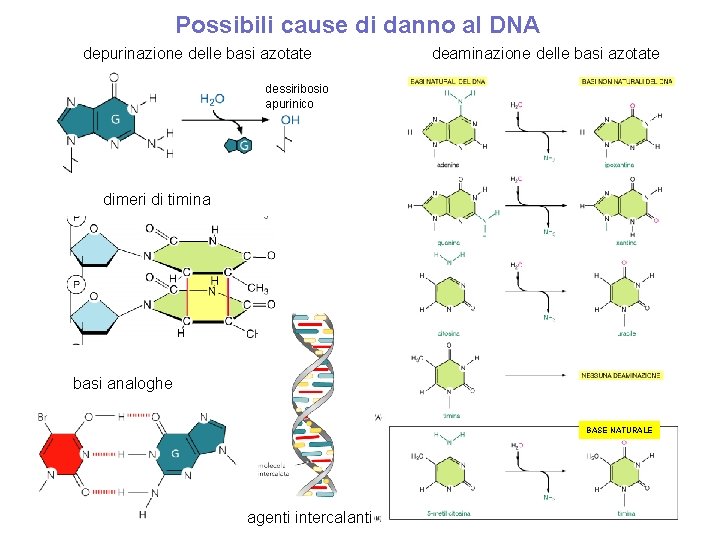
Possibili cause di danno al DNA depurinazione delle basi azotate deaminazione delle basi azotate dessiribosio apurinico dimeri di timina basi analoghe BASE NATURALE agenti intercalanti


Errori di replicazione del DNA Meccanismi di riparazione di errori di replicazione Danni al DNA Meccanismi di riparazione del danno al DNA

Le conseguenze del danno al DNA Mutazioni che bloccano la replicazione o trascrizione -dimeri di pirimidina -incisione (nick) nello scheletro del DNA Conseguenze immediate per la funzionalita’/sopravvivenza della cellula Errori di appaiamento -deaminazione della citosina ad uracile (mismatch C / U : G) -deaminazione della 5 -metil citosina a timina (mismatch C / T : G) Conseguenze per la progenie della cellula colpita dal danno

Processo di riparazione • 1) Riconoscimento del danno (scansione delle basi, ricerca di distorsioni) - Mut. S (E. coli), MSH (umano) • 2) Eliminazione del DNA danneggiato – DNA glicosilasi o endonucleasi • 3) Riparazione – DNA elicasi, Esonuceasi, DNA polimerasi, DNA ligasi

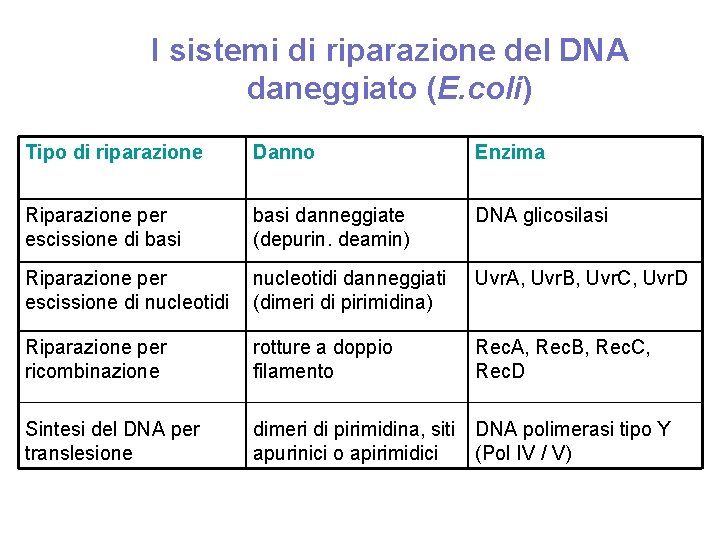
I sistemi di riparazione del DNA daneggiato (E. coli) Tipo di riparazione Danno Enzima Riparazione per escissione di basi danneggiate (depurin. deamin) DNA glicosilasi Riparazione per escissione di nucleotidi danneggiati (dimeri di pirimidina) Uvr. A, Uvr. B, Uvr. C, Uvr. D Riparazione per ricombinazione rotture a doppio filamento Rec. A, Rec. B, Rec. C, Rec. D Sintesi del DNA per translesione dimeri di pirimidina, siti DNA polimerasi tipo Y apurinici o apirimidici (Pol IV / V)

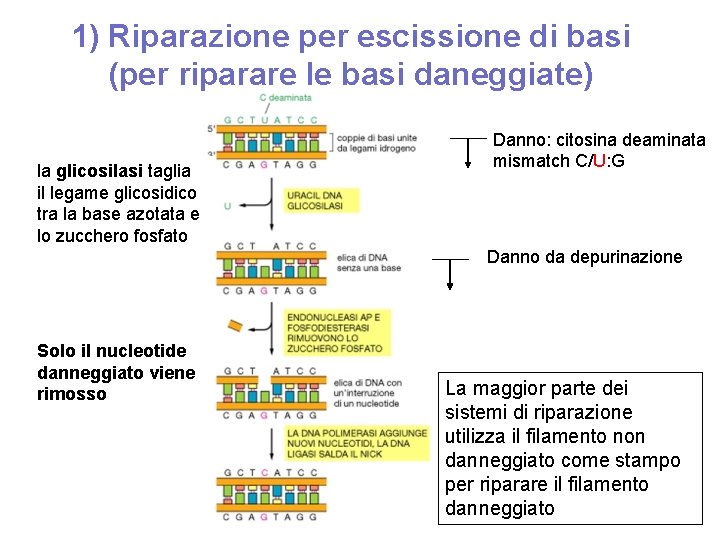
1) Riparazione per escissione di basi (per riparare le basi daneggiate) la glicosilasi taglia il legame glicosidico tra la base azotata e lo zucchero fosfato Danno: citosina deaminata mismatch C/U: G Danno da depurinazione Solo il nucleotide danneggiato viene rimosso La maggior parte dei sistemi di riparazione utilizza il filamento non danneggiato come stampo per riparare il filamento danneggiato

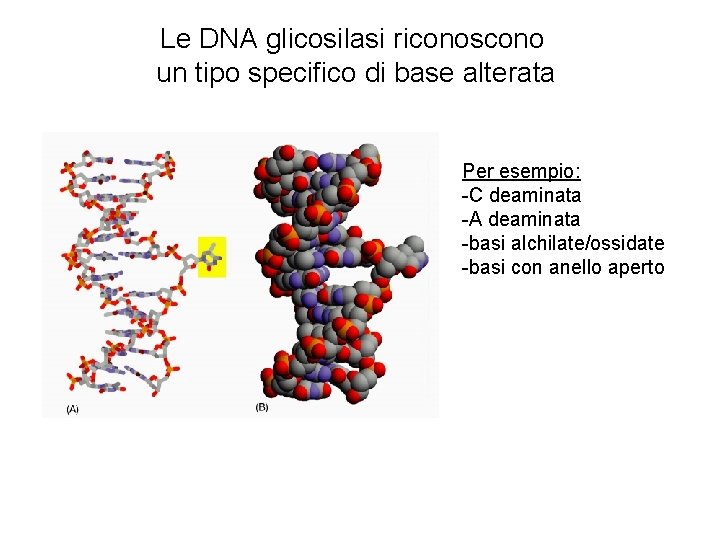
Le DNA glicosilasi riconoscono un tipo specifico di base alterata Per esempio: -C deaminata -A deaminata -basi alchilate/ossidate -basi con anello aperto

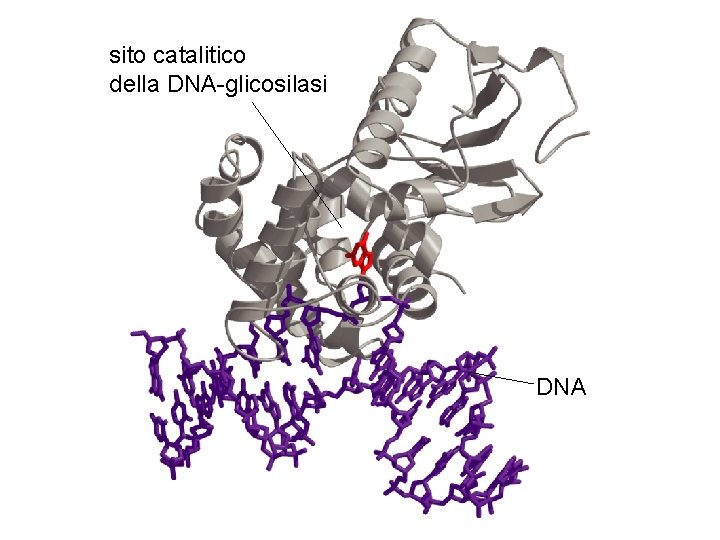
sito catalitico della DNA-glicosilasi DNA

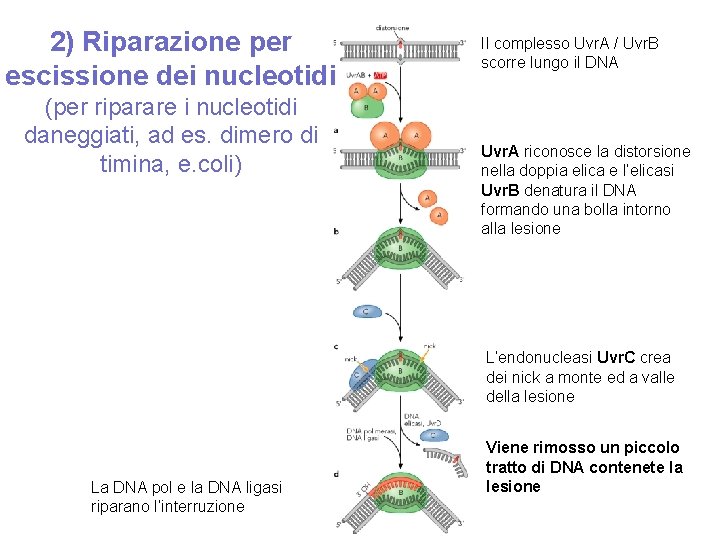
2) Riparazione per escissione dei nucleotidi (per riparare i nucleotidi daneggiati, ad es. dimero di timina, e. coli) Il complesso Uvr. A / Uvr. B scorre lungo il DNA Uvr. A riconosce la distorsione nella doppia elica e l’elicasi Uvr. B denatura il DNA formando una bolla intorno alla lesione L’endonucleasi Uvr. C crea dei nick a monte ed a valle della lesione La DNA pol e la DNA ligasi riparano l’interruzione Viene rimosso un piccolo tratto di DNA contenete la lesione

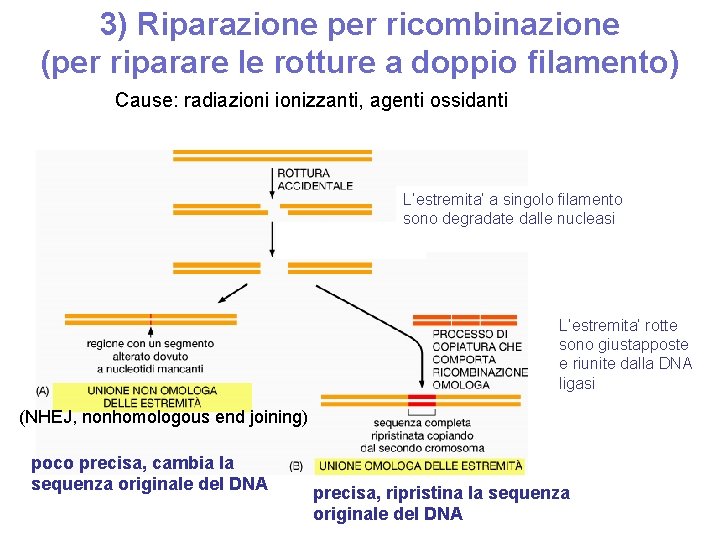
3) Riparazione per ricombinazione (per riparare le rotture a doppio filamento) Cause: radiazionizzanti, agenti ossidanti L’estremita’ a singolo filamento sono degradate dalle nucleasi L’estremita’ rotte sono giustapposte e riunite dalla DNA ligasi (NHEJ, nonhomologous end joining) poco precisa, cambia la sequenza originale del DNA precisa, ripristina la sequenza originale del DNA

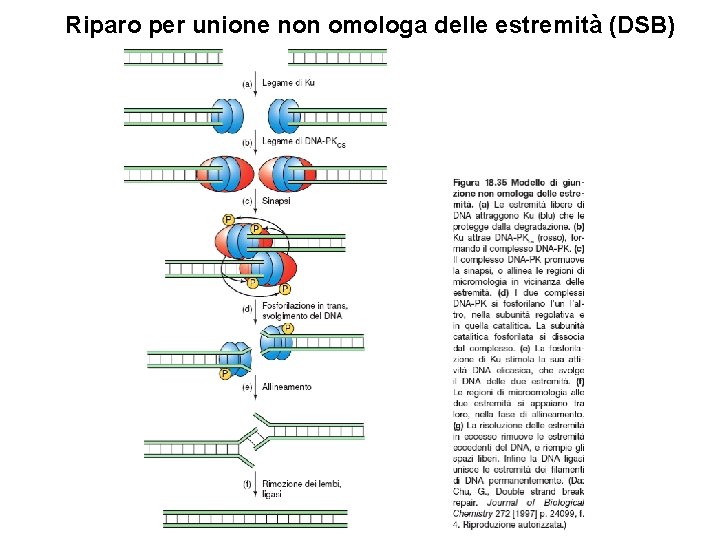
Riparo per unione non omologa delle estremità (DSB)

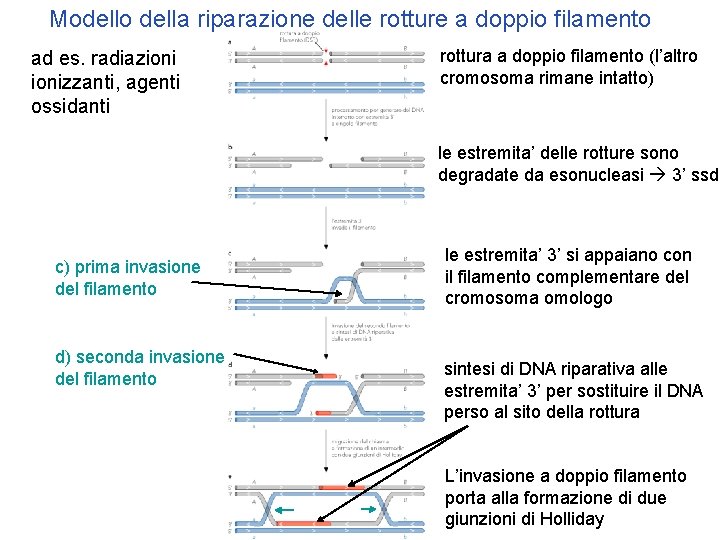
Modello della riparazione delle rotture a doppio filamento ad es. radiazionizzanti, agenti ossidanti rottura a doppio filamento (l’altro cromosoma rimane intatto) le estremita’ delle rotture sono degradate da esonucleasi 3’ ssd c) prima invasione del filamento d) seconda invasione del filamento le estremita’ 3’ si appaiano con il filamento complementare del cromosoma omologo sintesi di DNA riparativa alle estremita’ 3’ per sostituire il DNA perso al sito della rottura L’invasione a doppio filamento porta alla formazione di due giunzioni di Holliday

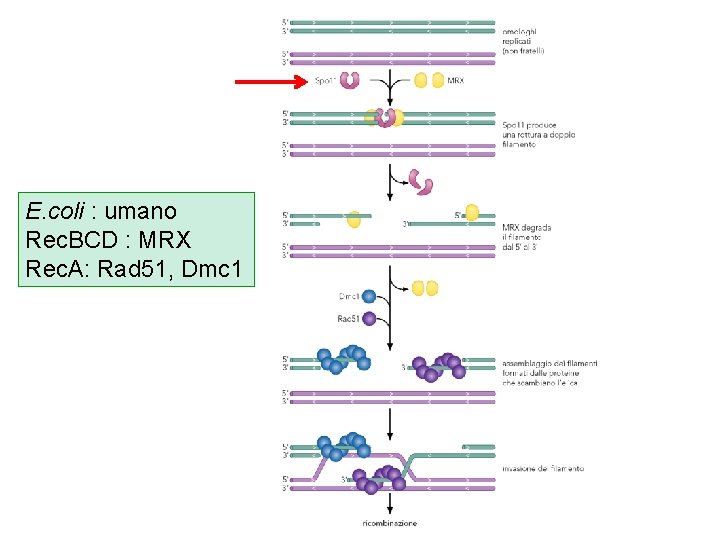
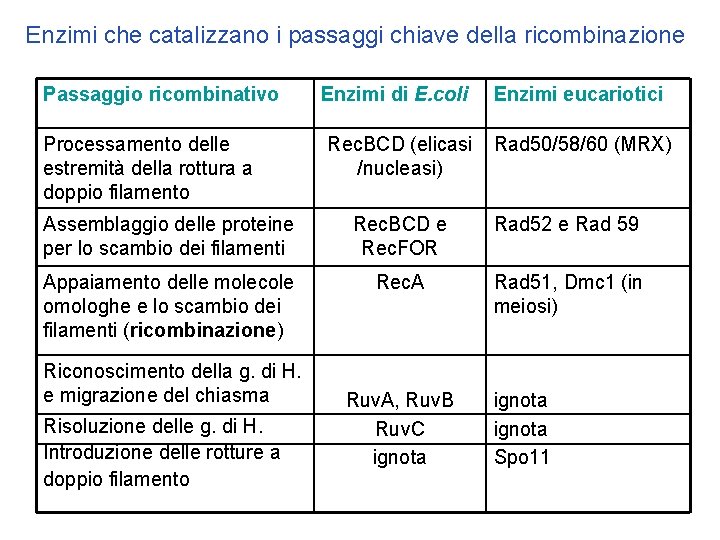
E. coli : umano Rec. BCD : MRX Rec. A: Rad 51, Dmc 1

Enzimi che catalizzano i passaggi chiave della ricombinazione Passaggio ricombinativo Processamento delle estremità della rottura a doppio filamento Enzimi di E. coli Enzimi eucariotici Rec. BCD (elicasi /nucleasi) Rad 50/58/60 (MRX) Assemblaggio delle proteine per lo scambio dei filamenti Rec. BCD e Rec. FOR Rad 52 e Rad 59 Appaiamento delle molecole omologhe e lo scambio dei filamenti (ricombinazione) Rec. A Rad 51, Dmc 1 (in meiosi) Riconoscimento della g. di H. e migrazione del chiasma Risoluzione delle g. di H. Introduzione delle rotture a doppio filamento Ruv. A, Ruv. B Ruv. C ignota Spo 11

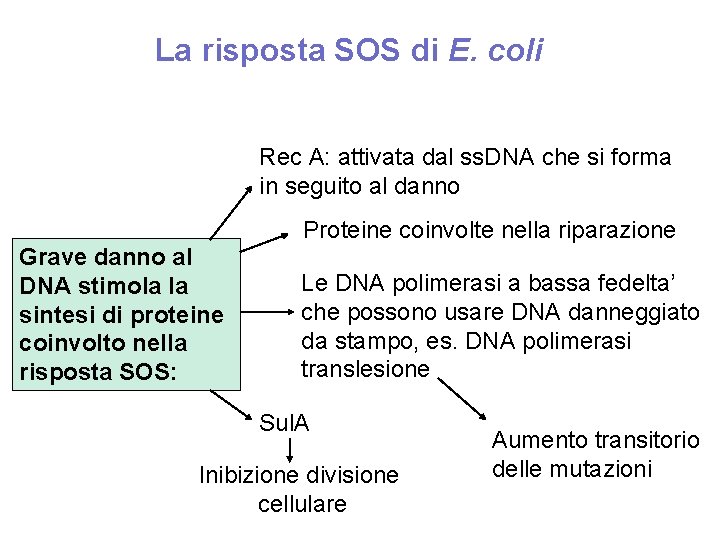
La risposta SOS di E. coli Rec A: attivata dal ss. DNA che si forma in seguito al danno Grave danno al DNA stimola la sintesi di proteine coinvolto nella risposta SOS: Proteine coinvolte nella riparazione Le DNA polimerasi a bassa fedelta’ che possono usare DNA danneggiato da stampo, es. DNA polimerasi translesione Sul. A Inibizione divisione cellulare Aumento transitorio delle mutazioni


Difetti dei meccanismi di riparazione del DNA causano gravi malattie • Mutazioni nelle cellule germinali sono particolarmente pericolose perchè ereditarie • Una mutazione nelle cellule somatiche puo’ dare origine a tumori

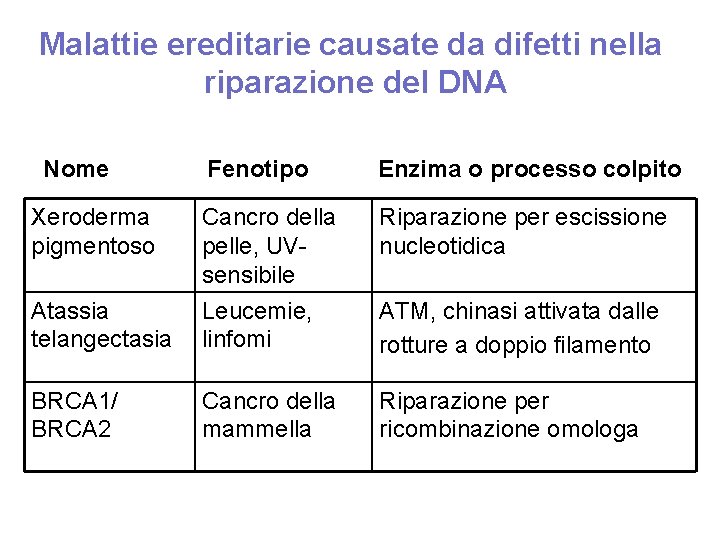
Malattie ereditarie causate da difetti nella riparazione del DNA Nome Fenotipo Enzima o processo colpito Xeroderma pigmentoso Cancro della pelle, UVsensibile Riparazione per escissione nucleotidica Atassia telangectasia Leucemie, linfomi ATM, chinasi attivata dalle rotture a doppio filamento BRCA 1/ BRCA 2 Cancro della mammella Riparazione per ricombinazione omologa

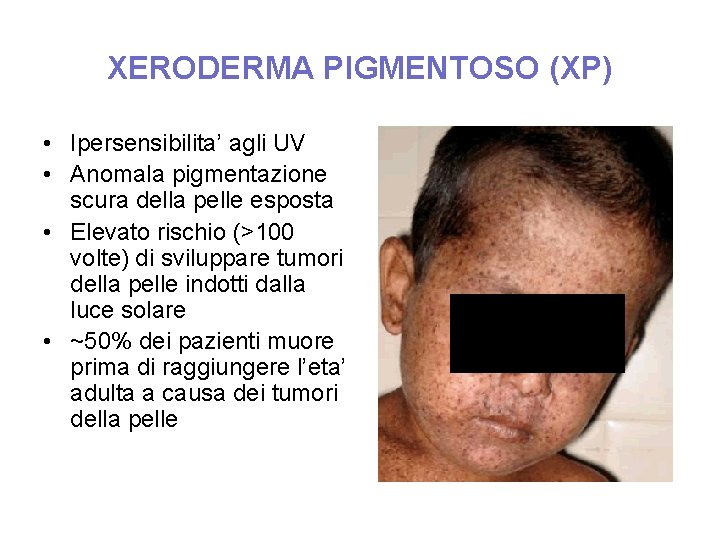
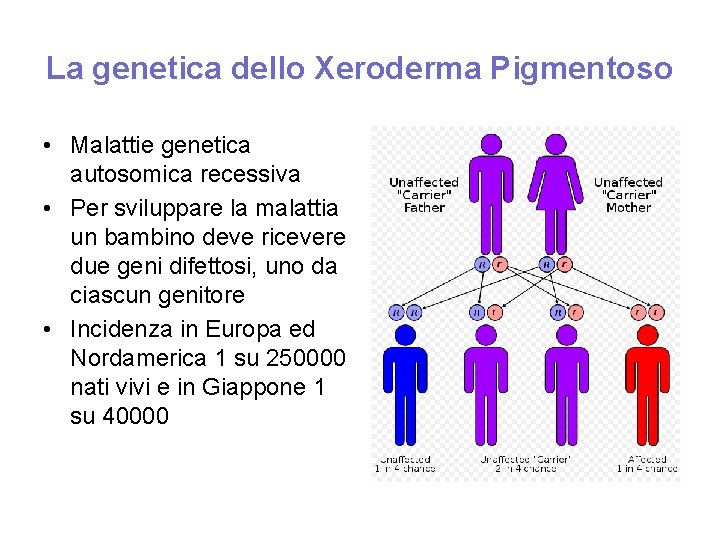
XERODERMA PIGMENTOSO (XP) • Ipersensibilita’ agli UV • Anomala pigmentazione scura della pelle esposta • Elevato rischio (>100 volte) di sviluppare tumori della pelle indotti dalla luce solare • ~50% dei pazienti muore prima di raggiungere l’eta’ adulta a causa dei tumori della pelle

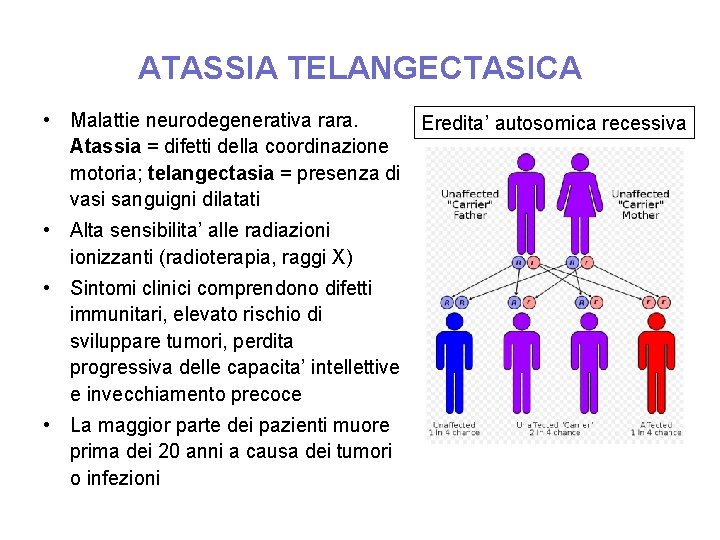
La genetica dello Xeroderma Pigmentoso • Malattie genetica autosomica recessiva • Per sviluppare la malattia un bambino deve ricevere due geni difettosi, uno da ciascun genitore • Incidenza in Europa ed Nordamerica 1 su 250000 nati vivi e in Giappone 1 su 40000

La causa dello XP • La fotosensibilita’ risulta dall’incapacita’ di riparare il danno al DNA indotto dai raggi UV • A causa dei difetti dei geni che codificano per le proteine di riparazione per escissione dei nucleotidi, il danno porta ad un aumento nella frequenza di mutazioni • L’elevata frequenza di mutazioni aumenta il rischio di sviluppare tumori

La radiazione danneggia il DNA UV raggi solari 6 1 2 3 5 4 fusione fotochimica tra due basi pirimidiniche adiacenti Basi dimerizzate non possono formare legami idrogeno con le basi complementari blocco della replicazione

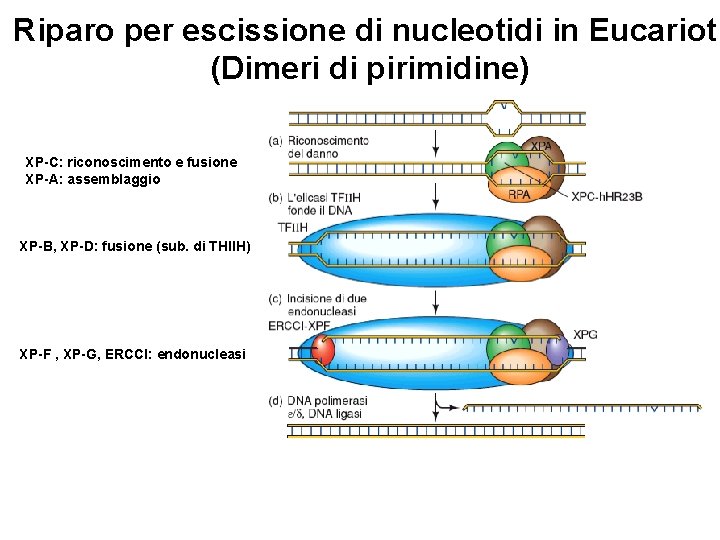
Riparo per escissione di nucleotidi in Eucarioti (Dimeri di pirimidine) XP-C: riconoscimento e fusione XP-A: assemblaggio XP-B, XP-D: fusione (sub. di THIIH) XP-F , XP-G, ERCCI: endonucleasi

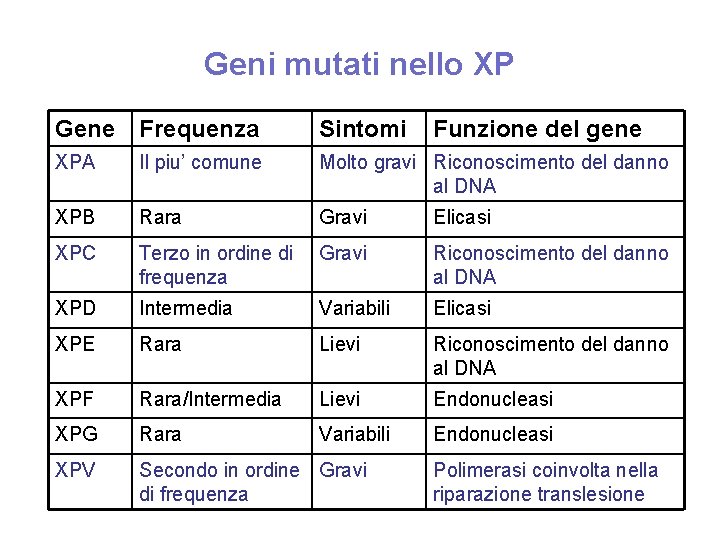
Geni mutati nello XP Gene Frequenza Sintomi Funzione del gene XPA Il piu’ comune Molto gravi Riconoscimento del danno al DNA XPB Rara Gravi Elicasi XPC Terzo in ordine di frequenza Gravi Riconoscimento del danno al DNA XPD Intermedia Variabili Elicasi XPE Rara Lievi Riconoscimento del danno al DNA XPF Rara/Intermedia Lievi Endonucleasi XPG Rara Variabili Endonucleasi XPV Secondo in ordine Gravi di frequenza Polimerasi coinvolta nella riparazione translesione

Il trattamento dello XP • Stile di vita che prevede la protezione totale dalla luce solare • Applicazione di creme solari che proteggano dallo spettro UV • Applicazione topica di creme contenenti enzimi di riparazione incapsulati in liposomi • Terapia genica? --> l’introduzione di un gene di riparazione intatto nelle cellule della pelle permetterebbe alle cellule di sintetizzare una proteina della via di riparazione per l’escissione dei nucleotidi

Malattie ereditarie causate da difetti nella riparazione del DNA Nome Xeroderma pigmentoso Atassia telangectasica Fenotipo Cancro della pelle, UVsensibile Leucemie, linfomi BRCA 1/ BRCA 2 Cancro della mammella Enzima o processo colpito Riparazione per escissione nucleotidica ATM (ataxia telagectasia mutated), chinasi attivata dalle rotture a doppio filamento Riparazione per ricombinazione omologa

ATASSIA TELANGECTASICA • Malattie neurodegenerativa rara. Eredita’ autosomica recessiva Atassia = difetti della coordinazione motoria; telangectasia = presenza di vasi sanguigni dilatati • Alta sensibilita’ alle radiazionizzanti (radioterapia, raggi X) • Sintomi clinici comprendono difetti immunitari, elevato rischio di sviluppare tumori, perdita progressiva delle capacita’ intellettive e invecchiamento precoce • La maggior parte dei pazienti muore prima dei 20 anni a causa dei tumori o infezioni

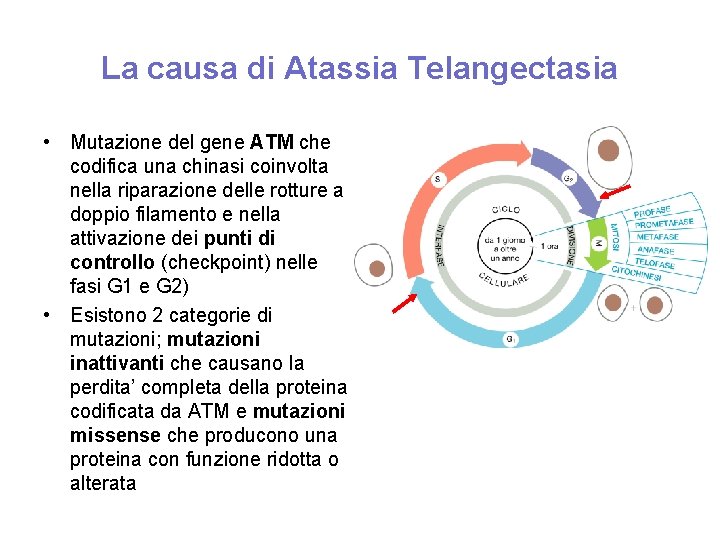
La causa di Atassia Telangectasia • Mutazione del gene ATM che codifica una chinasi coinvolta nella riparazione delle rotture a doppio filamento e nella attivazione dei punti di controllo (checkpoint) nelle fasi G 1 e G 2) • Esistono 2 categorie di mutazioni; mutazioni inattivanti che causano la perdita’ completa della proteina codificata da ATM e mutazioni missense che producono una proteina con funzione ridotta o alterata

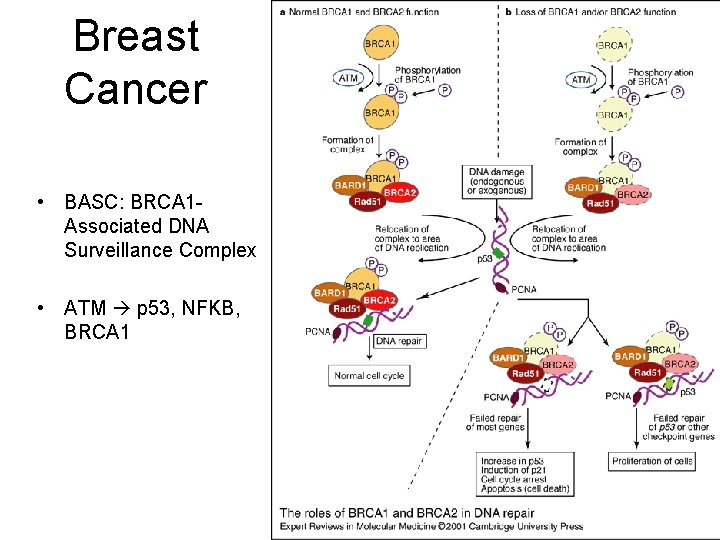
Breast Cancer • BASC: BRCA 1 Associated DNA Surveillance Complex • ATM p 53, NFKB, BRCA 1

SOMMARIO • Gli organismi possono soppravivere solamente se il loro DNA e’ replicato correttamente ed e’ protetto da danni fisici e chimici • L’attivita’ di correzione di bozze della DNA polimerasi e il sistema di mismatch repair sono deputati alla correzione di errori di replicazione • Le cellule possiedono un grande repertorio di enzimi deputati a riparare i danni al DNA, che sarebbero altrimenti letali • Il danno piu’ pericoloso consiste nella rottura della doppia elica (DSB) che viene riparata mediante fusione delle estremità o ricombinazione
 La regolazione degli affetti e la riparazione del sé
La regolazione degli affetti e la riparazione del sé Codice genetico ridondante e degenerato
Codice genetico ridondante e degenerato Replicazione teta
Replicazione teta Replicazione a circolo rotante
Replicazione a circolo rotante Alphaherpesvirinae
Alphaherpesvirinae Legge di propagazione degli errori
Legge di propagazione degli errori Cosa sono gli errori morfologici
Cosa sono gli errori morfologici Turparunt
Turparunt Errori grossolani
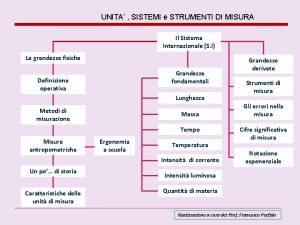
Errori grossolani Mappa concettuale strumenti di misura
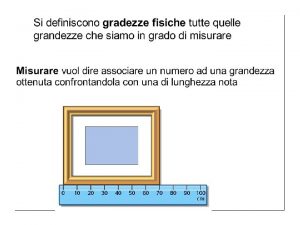
Mappa concettuale strumenti di misura Strumenti di misura analogici
Strumenti di misura analogici Errore di graficismo
Errore di graficismo Errori ingegneria
Errori ingegneria Portata e sensibilità del termometro clinico
Portata e sensibilità del termometro clinico Errori morfologici
Errori morfologici Dislessie
Dislessie Errori accidentali
Errori accidentali Function of dna polymerase 3
Function of dna polymerase 3 Bioflix activity dna replication dna replication diagram
Bioflix activity dna replication dna replication diagram Coding dna and non coding dna
Coding dna and non coding dna What role does dna polymerase play in copying dna?
What role does dna polymerase play in copying dna? Chapter 11 dna and genes
Chapter 11 dna and genes Desnaturalizacion del dna
Desnaturalizacion del dna Surco mayor adn
Surco mayor adn Meiosi
Meiosi Spiralizzazione del dna zanichelli
Spiralizzazione del dna zanichelli Desnaturalizacion del dna
Desnaturalizacion del dna Struttura primaria del dna
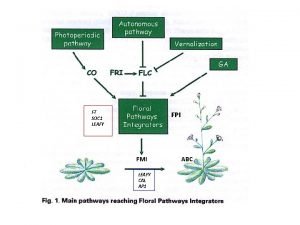
Struttura primaria del dna Fca fpi
Fca fpi Struttura del dna
Struttura del dna Dna yarn
Dna yarn Separating dna
Separating dna Application dna
Application dna Genghis khan dna map
Genghis khan dna map Varning
Varning Dna structure and replication pogil
Dna structure and replication pogil Transcription and translation coloring
Transcription and translation coloring Dna
Dna Dna meaning
Dna meaning Dna replication is semi-conservative
Dna replication is semi-conservative Strawberry dna extraction materials
Strawberry dna extraction materials Dna itsepalvelu
Dna itsepalvelu 5 examples of palindromic dna sequences
5 examples of palindromic dna sequences Translasi dan transkripsi
Translasi dan transkripsi Is botana curus real
Is botana curus real Unlike dna, rna
Unlike dna, rna Virulent
Virulent Characteristic of dna
Characteristic of dna Dna stands for deoxyribonucleic acid
Dna stands for deoxyribonucleic acid Packaging of dna helix
Packaging of dna helix Structer of dna
Structer of dna Adjacent bases dna
Adjacent bases dna Pentose sugar in rna
Pentose sugar in rna Dna bond structure
Dna bond structure Chromosome vs dna vs gene
Chromosome vs dna vs gene Molecular genetics section 1 dna the genetic material
Molecular genetics section 1 dna the genetic material Okazaki fragments
Okazaki fragments Reading frame
Reading frame Dna structure ppt
Dna structure ppt Dna primase
Dna primase Recombinant dna technology applications
Recombinant dna technology applications Model molekula dna
Model molekula dna Humans share 50 of their dna with bananas
Humans share 50 of their dna with bananas Transciption definition
Transciption definition Dna fragments can be separated by
Dna fragments can be separated by Cytoplasm structure
Cytoplasm structure Recombinant dna technology applications
Recombinant dna technology applications