DNA Replication DNA Replication II Three models proposed










- Slides: 46

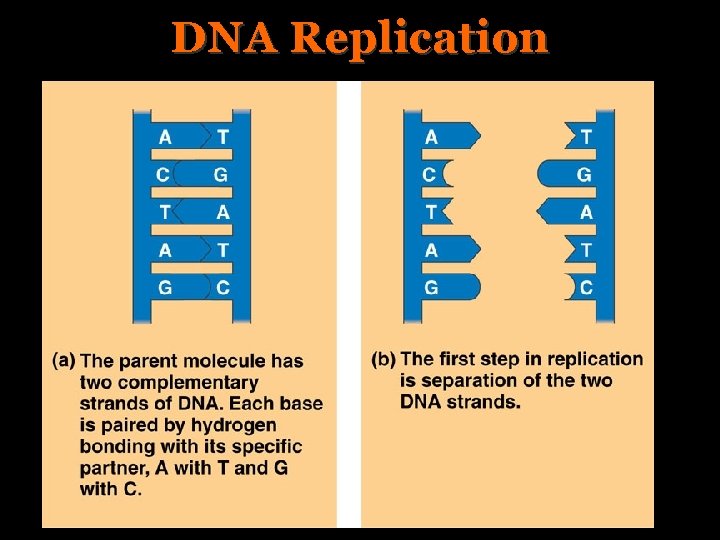
DNA Replication

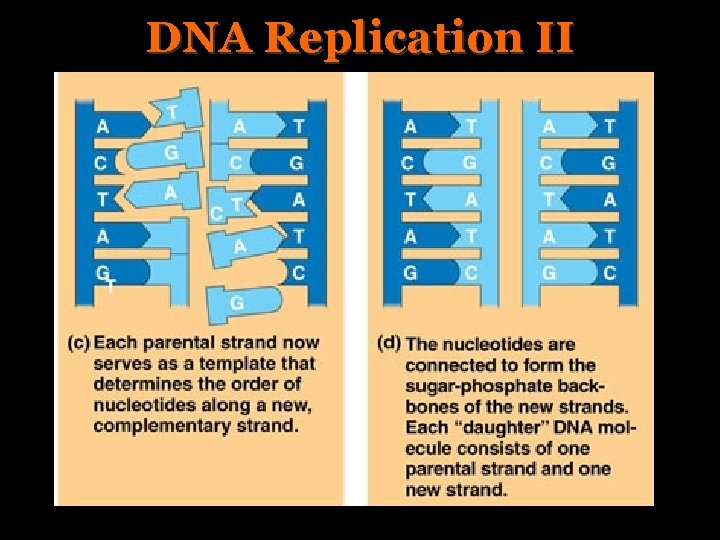
DNA Replication II

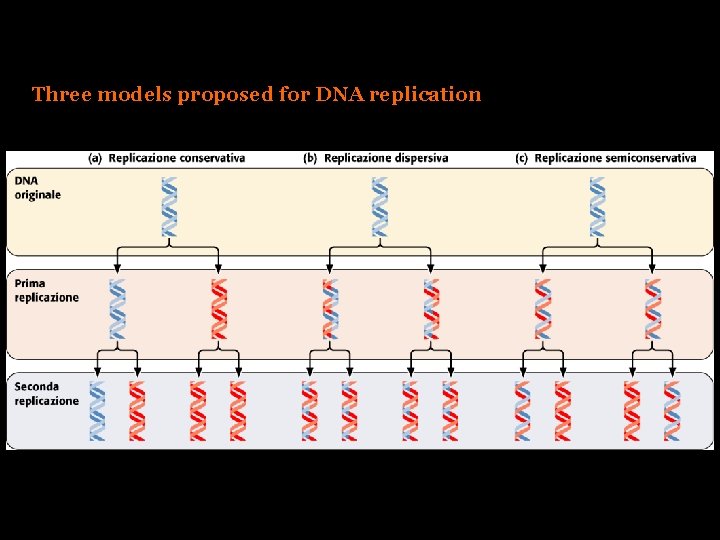
Three models proposed for DNA replication

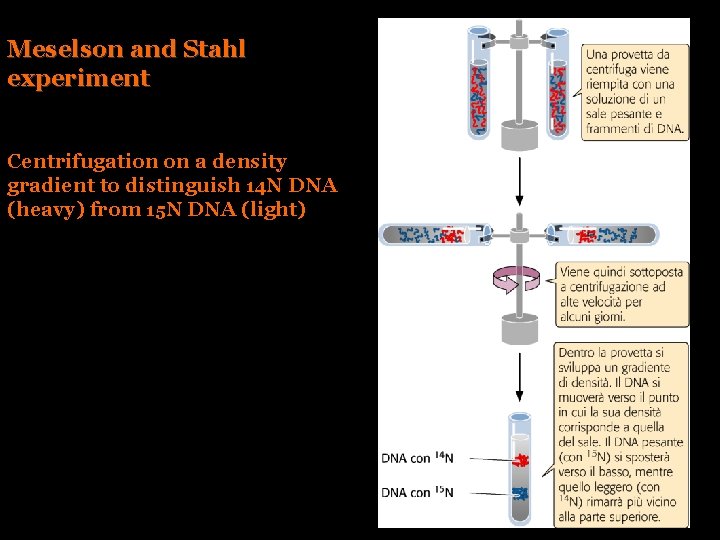
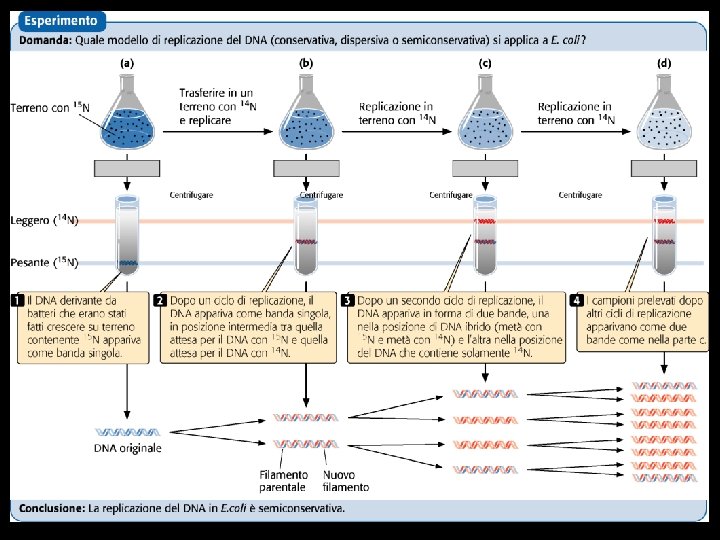
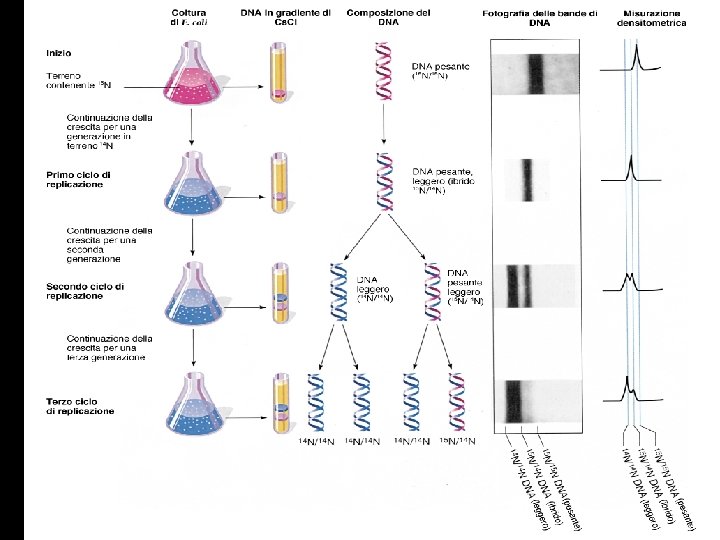
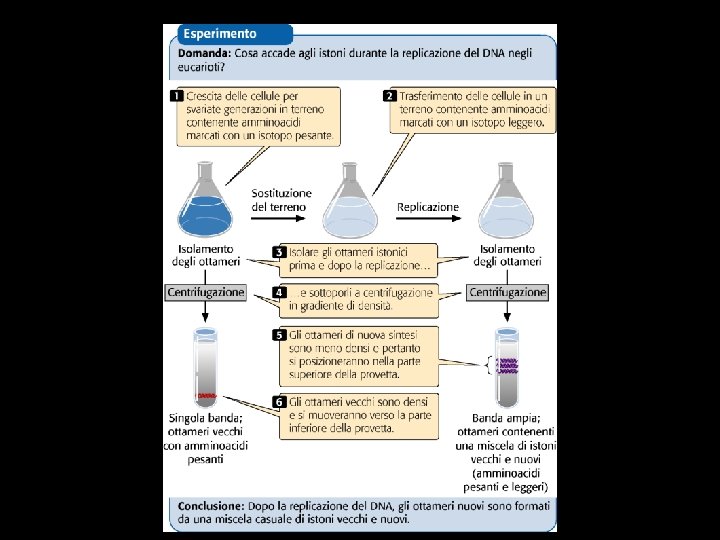
Meselson and Stahl experiment Centrifugation on a density gradient to distinguish 14 N DNA (heavy) from 15 N DNA (light)



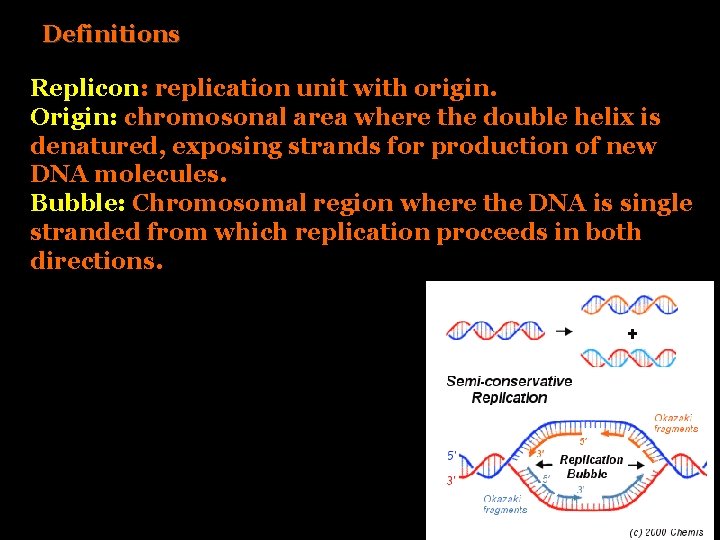
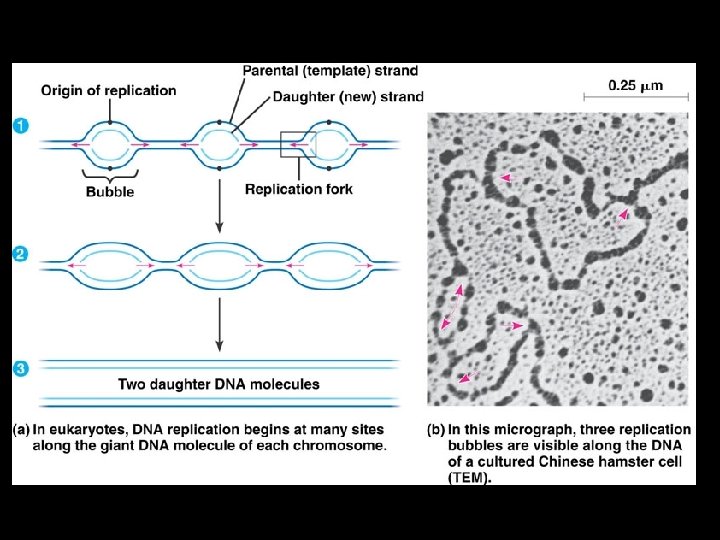
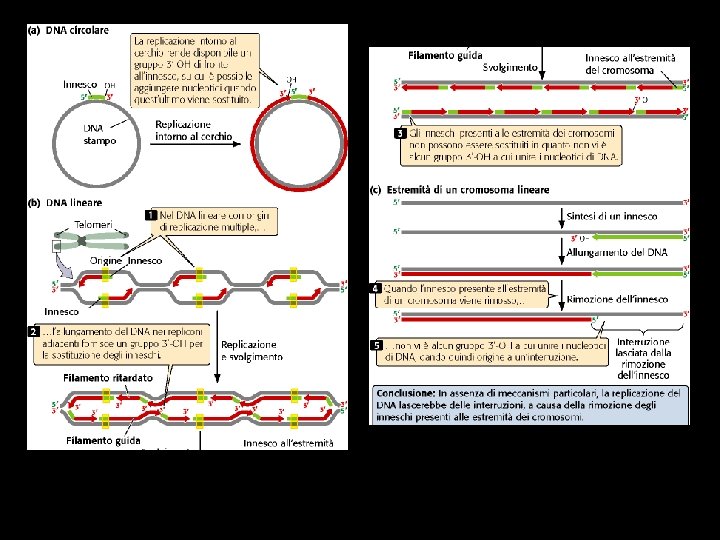
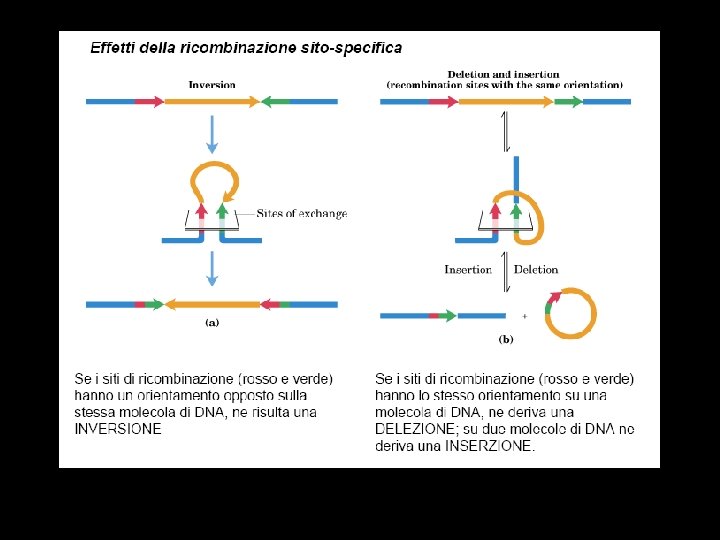
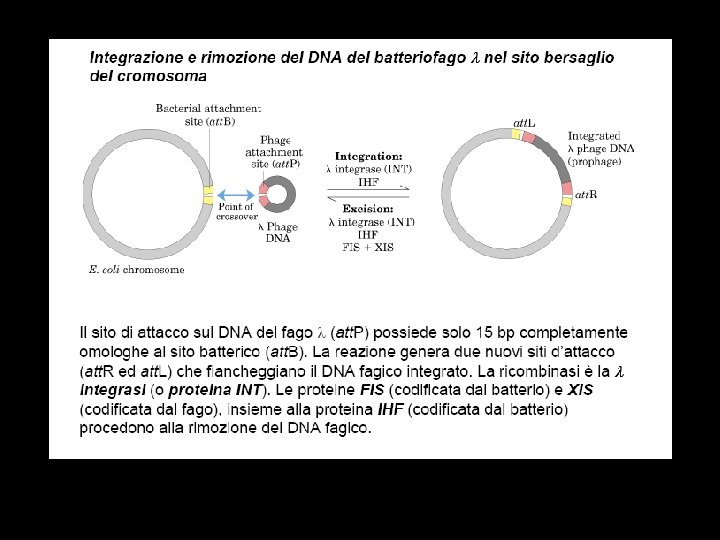
Definitions Replicon: replication unit with origin. Origin: chromosonal area where the double helix is denatured, exposing strands for production of new DNA molecules. Bubble: Chromosomal region where the DNA is single stranded from which replication proceeds in both directions.

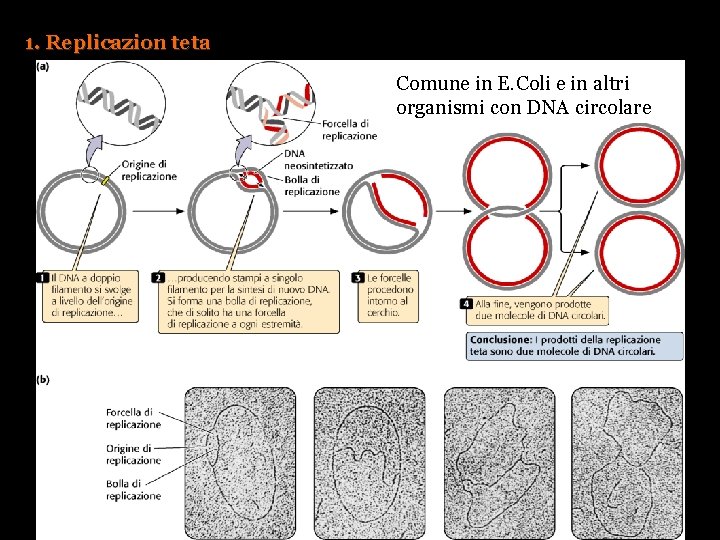
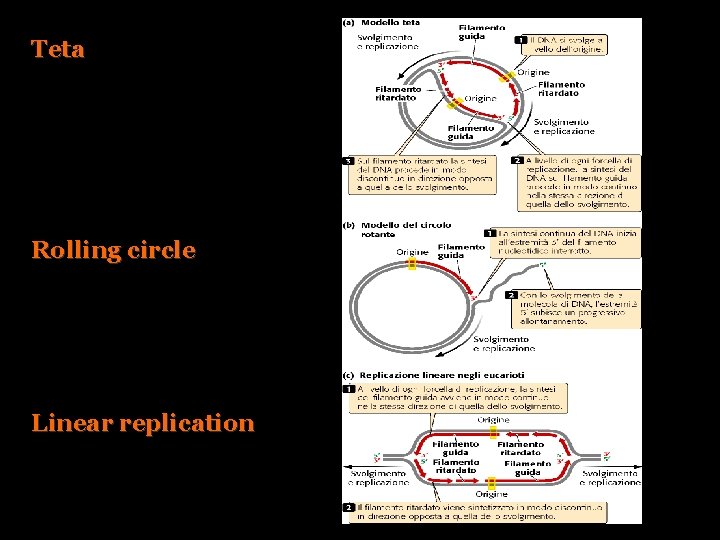
1. Replicazion teta Comune in E. Coli e in altri organismi con DNA circolare

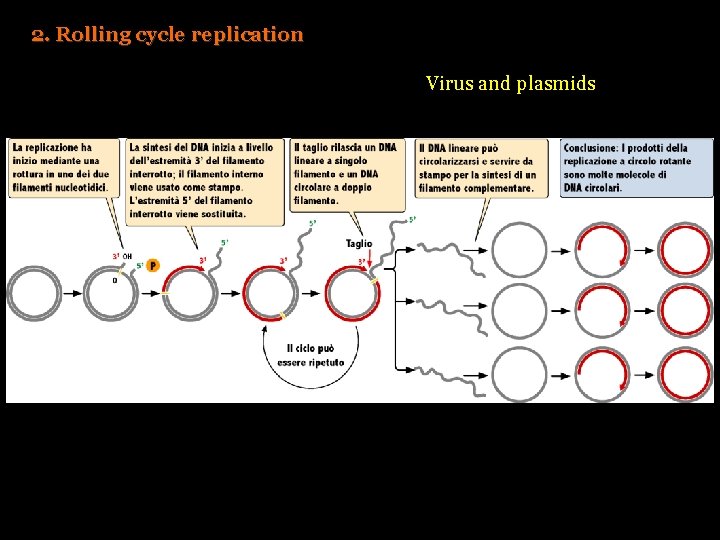
2. Rolling cycle replication Virus and plasmids

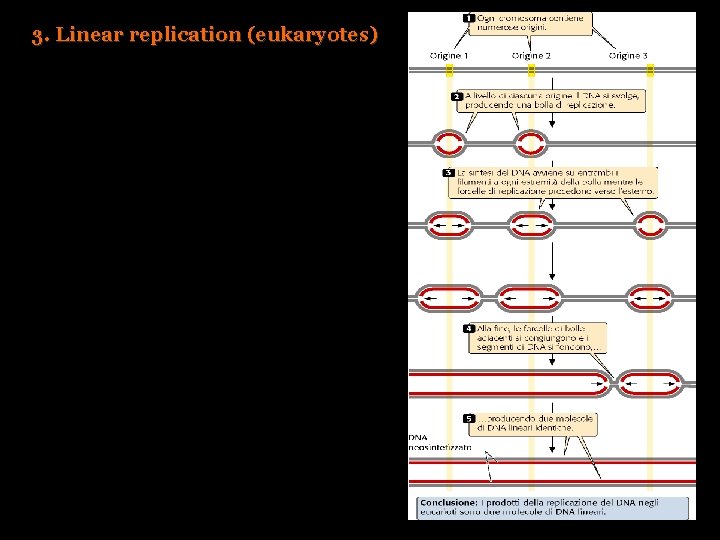
3. Linear replication (eukaryotes)



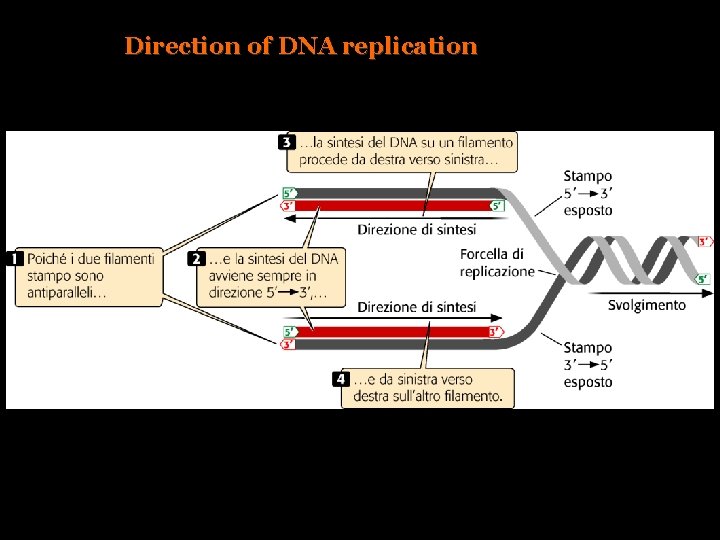
Direction of DNA replication

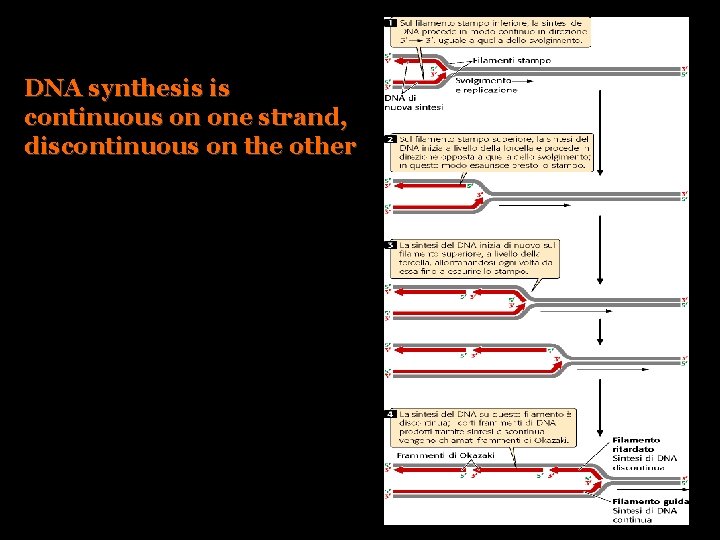
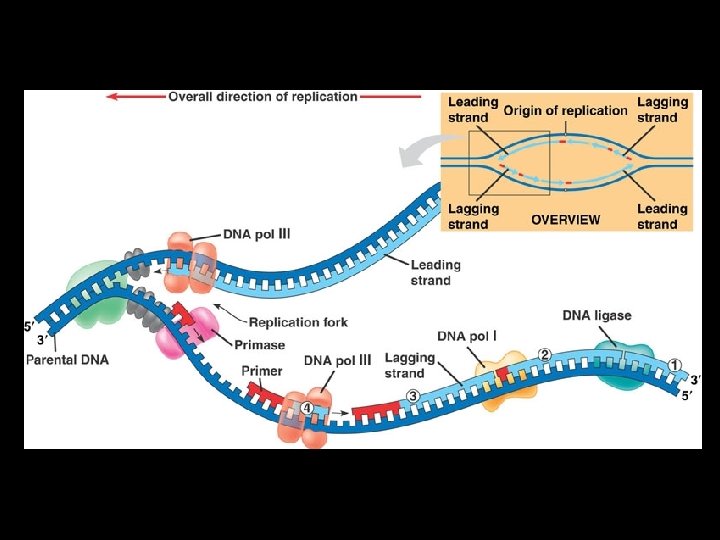
DNA synthesis is continuous on one strand, discontinuous on the other


Teta Rolling circle Linear replication

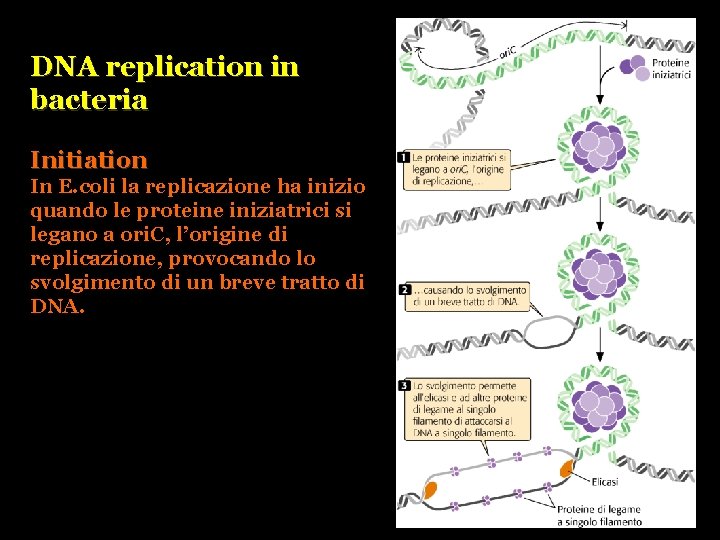
DNA replication in bacteria Initiation In E. coli la replicazione ha inizio quando le proteine iniziatrici si legano a ori. C, l’origine di replicazione, provocando lo svolgimento di un breve tratto di DNA.

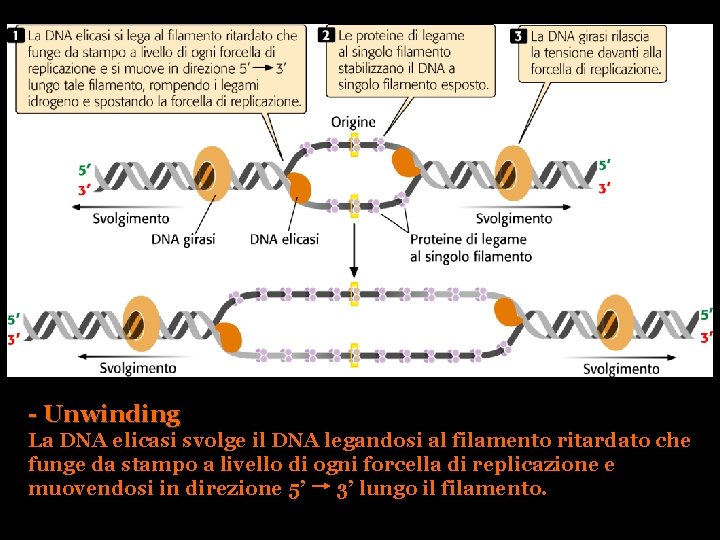
- Unwinding La DNA elicasi svolge il DNA legandosi al filamento ritardato che funge da stampo a livello di ogni forcella di replicazione e muovendosi in direzione 5’ 3’ lungo il filamento.

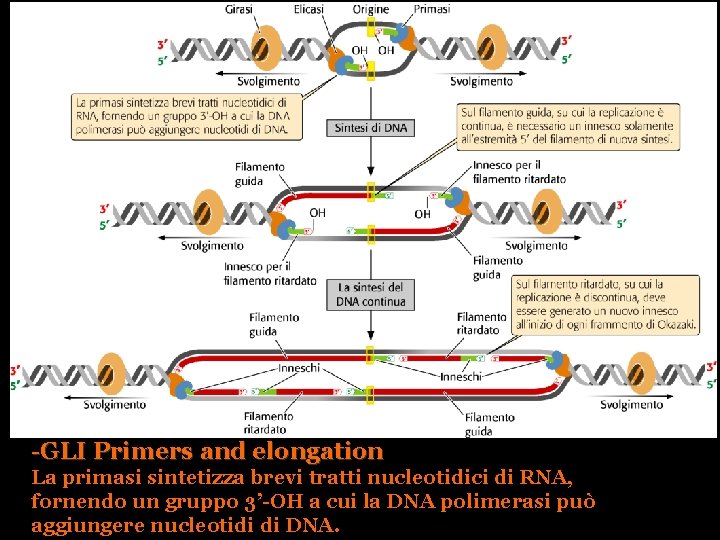
-GLI Primers and elongation La primasi sintetizza brevi tratti nucleotidici di RNA, fornendo un gruppo 3’-OH a cui la DNA polimerasi può aggiungere nucleotidi di DNA.



DNA Polymerase III • • • The "real" polymerase in E. coli At least 10 different subunits "Core" enzyme has three subunits - , , and Alpha subunit is polymerase Epsilon subunit is 3'-exonuclease Theta function is unknown The beta subunit dimer forms a ring around DNA • Enormous processivity - 5 million bases!

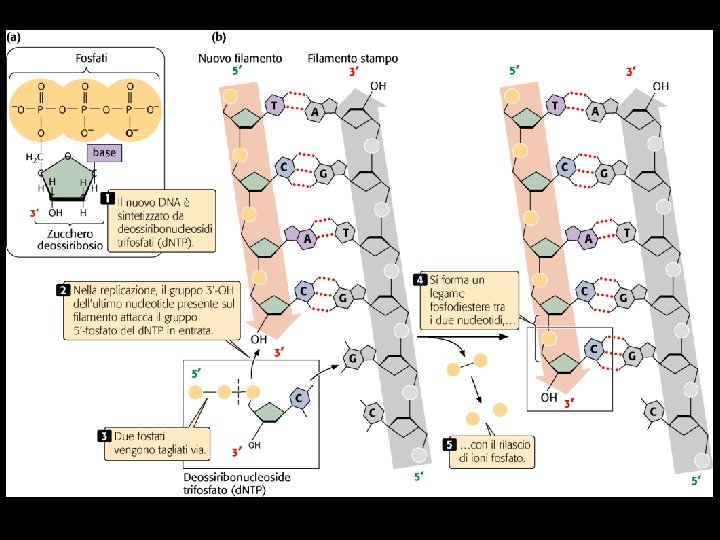
DNA Polymerase I Replication occurs 5' to 3' • Nucleotides are added at the 3'-end of the strand • Pol I catalyzes about 20 cycles of polymerization before the new strand dissociates from template • 20 cycles constitutes moderate "processivity" • Pol I from E. coli is 928 aa (109 k. D) monomer • In addition to 5'-3' polymerase, it also has 3'-5' exonuclease and 5'-3' exonuclease activities

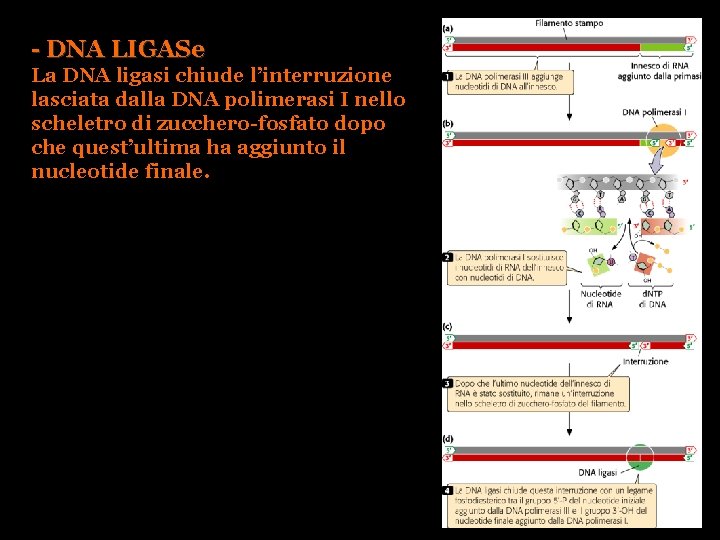
- DNA LIGASe La DNA ligasi chiude l’interruzione lasciata dalla DNA polimerasi I nello scheletro di zucchero-fosfato dopo che quest’ultima ha aggiunto il nucleotide finale.

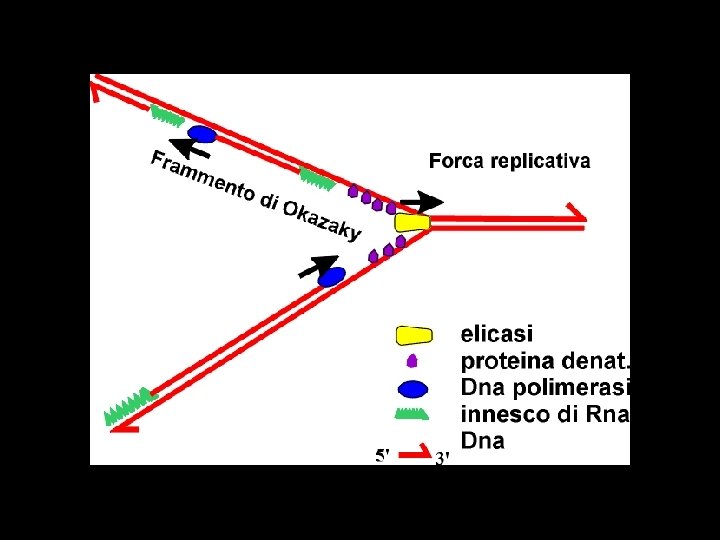
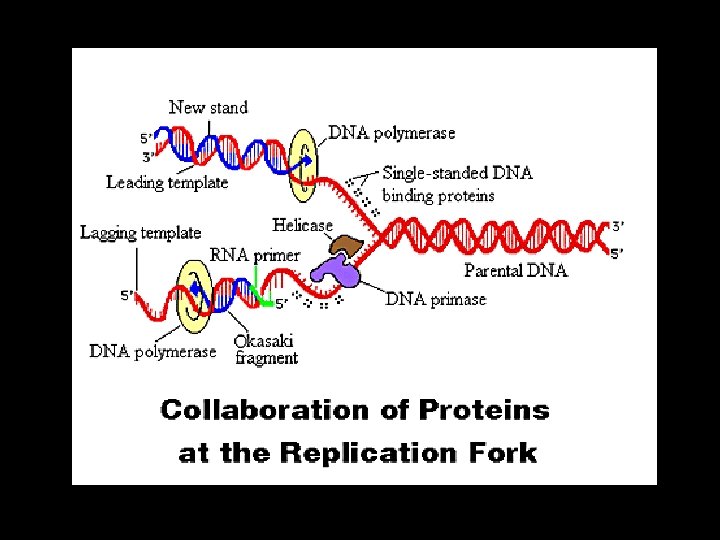
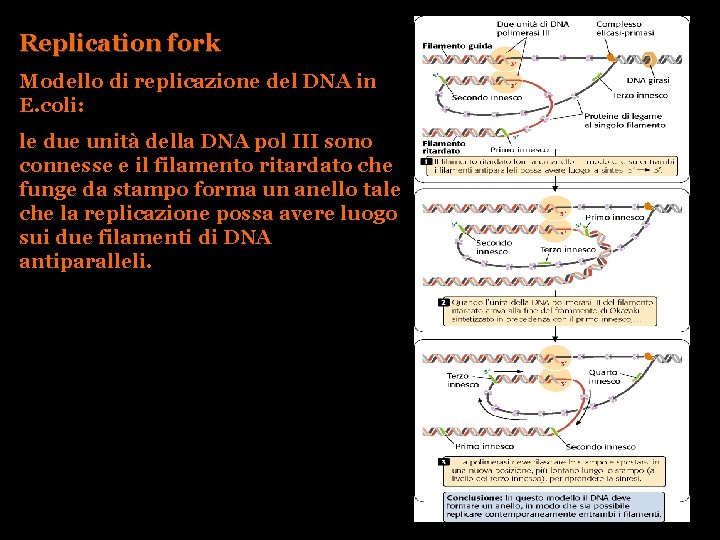
Replication fork Modello di replicazione del DNA in E. coli: le due unità della DNA pol III sono connesse e il filamento ritardato che funge da stampo forma un anello tale che la replicazione possa avere luogo sui due filamenti di DNA antiparalleli.

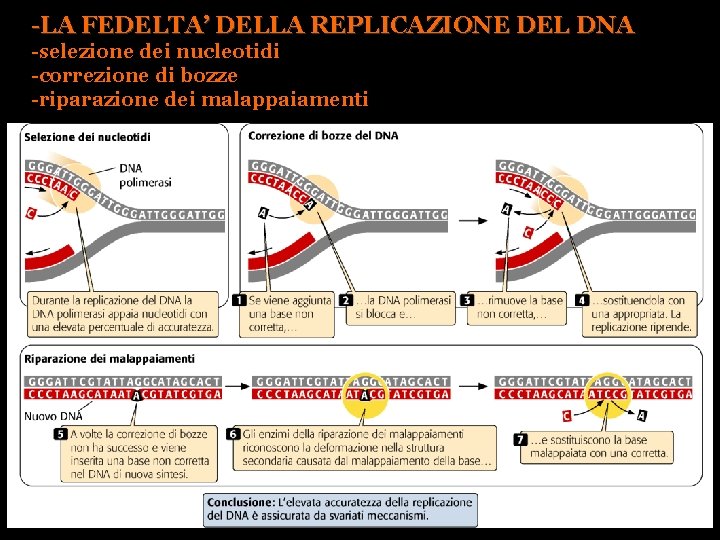
-LA FEDELTA’ DELLA REPLICAZIONE DEL DNA -selezione dei nucleotidi -correzione di bozze -riparazione dei malappaiamenti

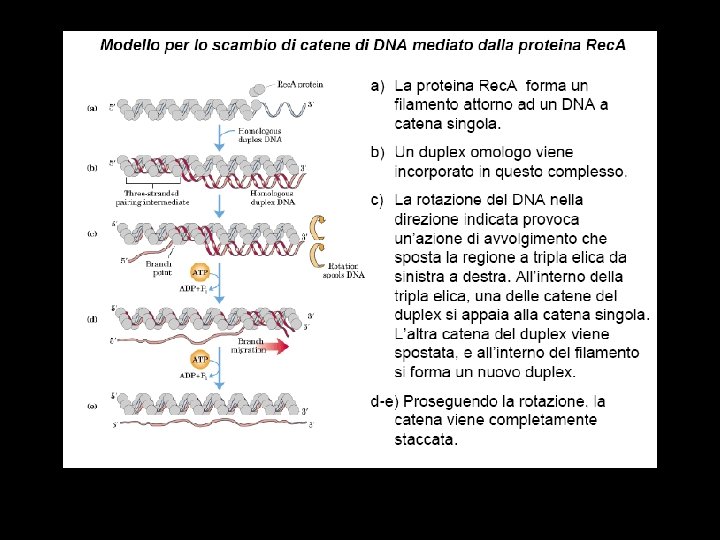
Other Proteins That Assist DNA Replication • Helicase, topoisomerase, single-strand binding protein


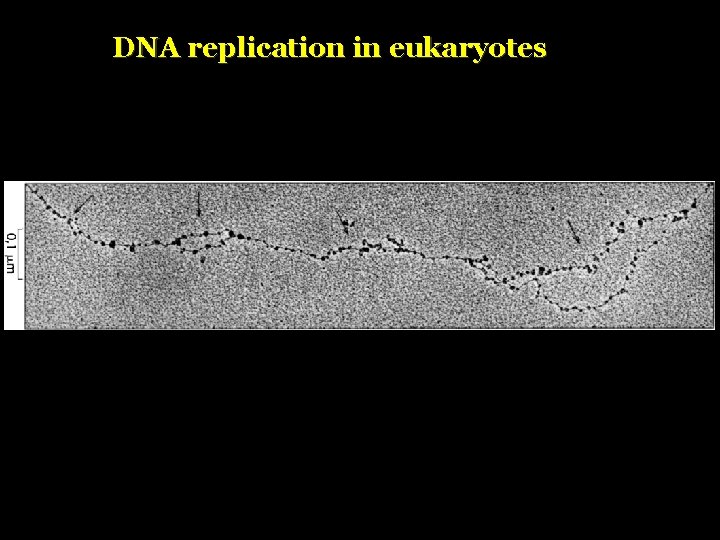
DNA replication in eukaryotes

Polimerasi nei Procarioti I Procarioti possiedono cinque tipi di DNA Polimerasi: • DNA Polimerasi I: implicata nella riparazione del DNA e nella rimozione degli inneschi dei frammenti di Okazaki. Possiede attività esonucleasica sia in direzione 5'->3', sia in direzione 3'->5' • DNA Polimerasi II: indotta da danni al DNA per riparazione incline all'errore (error-prone). Ha attività polimerasica 5'->3' ed esonucleasica 3'->5' • DNA Polimerasi III: l'enzima principale per la replicazione, con attività polimerasica 5'->3' ed esonucleasica (nella correzione di bozze, o proofreading) 3'->5'. Attiva sia nella sintesi del filamento leading, sia in quella dei frammenti di Okazaki. • DNA Polimerasi IV-V: anch'esse coinvolte nella riparazione incline all'errore

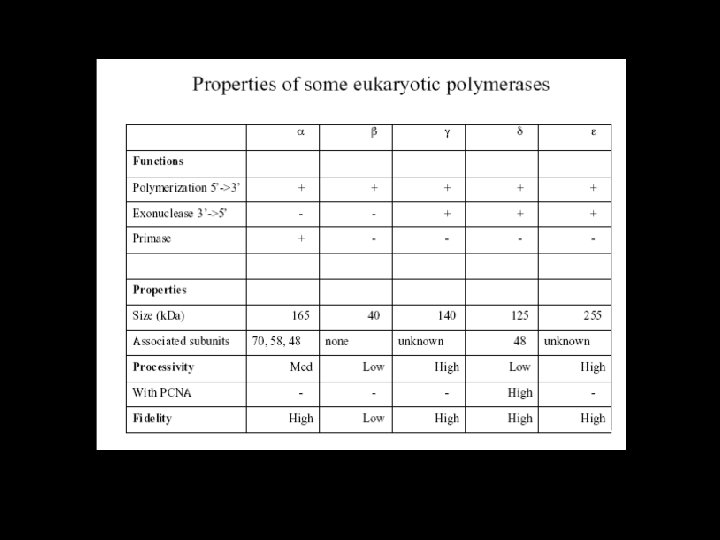
Polimerasi negli Eucarioti Le Polimerasi degli Eucarioti sono costituite invece da: • DNA Polimerasi α: è una primasi (enzima che sintetizza i primer di RNA) e procede inoltre all'allungamento dei primer con alcune centinaia di deossiribonucleotidi. • DNA Polimerasi δ: l'enzima principale della replicazione eucariotica. Sintetizza sia il filamento leading, sia il filamento lagging. Riempie inoltre le interruzioni tra i frammenti di Okazaki. • DNA Polimerasi β: coinvolta nella riparazione del DNA • DNA Polimerasi γ: replica il DNA mitocondriale • DNA Polimerasi ε: stessa funzione della Polimerasi δ.


Prokaryotes 5 polymerases (I, III, IV, V) I--excision repair, removal of RNA primers II-repair III-main enzyme IV, V-repair, special conditions polymerases also exonucleases 1 Origin of replication Okazaki fragments 1000 -2000 nuc. no proteins on DNA Eukaryotes 5 polymerases-- , b, g, d, --polymerization b--repair g--mitochondrial d-main enzyme --unknown not all exonucleases many origins Okazaki fragments 150 -200 nuc. histones



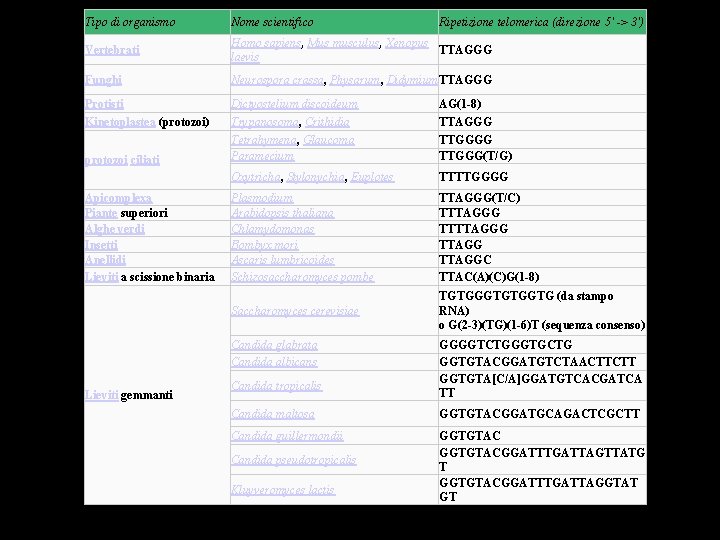
Tipo di organismo Nome scientifico Vertebrati Homo sapiens, Mus musculus, Xenopus TTAGGG laevis Funghi Neurospora crassa, Physarum, Didymium. TTAGGG Protisti Kinetoplastea (protozoi) Dictyostelium discoideum Trypanosoma, Crithidia Tetrahymena, Glaucoma Paramecium AG(1 -8) TTAGGG TTGGG(T/G) Oxytricha, Stylonychia, Euplotes TTTTGGGG Plasmodium Arabidopsis thaliana Chlamydomonas Bombyx mori Ascaris lumbricoides Schizosaccharomyces pombe TTAGGG(T/C) TTTAGGG TTAGGC TTAC(A)(C)G(1 -8) Saccharomyces cerevisiae TGTGGGTGTGGTG (da stampo RNA) o G(2 -3)(TG)(1 -6)T (sequenza consenso) protozoi ciliati Apicomplexa Piante superiori Alghe verdi Insetti Anellidi Lieviti a scissione binaria Candida glabrata Candida albicans Lieviti gemmanti Candida tropicalis Ripetizione telomerica (direzione 5' -> 3') GGGGTCTGGGTGCTG GGTGTACGGATGTCTAACTTCTT GGTGTA[C/A]GGATGTCACGATCA TT Candida maltosa GGTGTACGGATGCAGACTCGCTT Candida guillermondii GGTGTACGGATTTGATTAGTTATG T GGTGTACGGATTTGATTAGGTAT GT Candida pseudotropicalis Kluyveromyces lactis




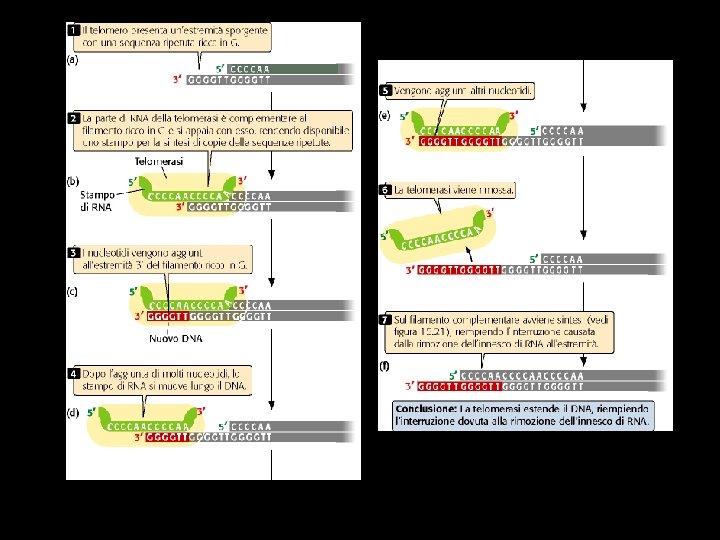
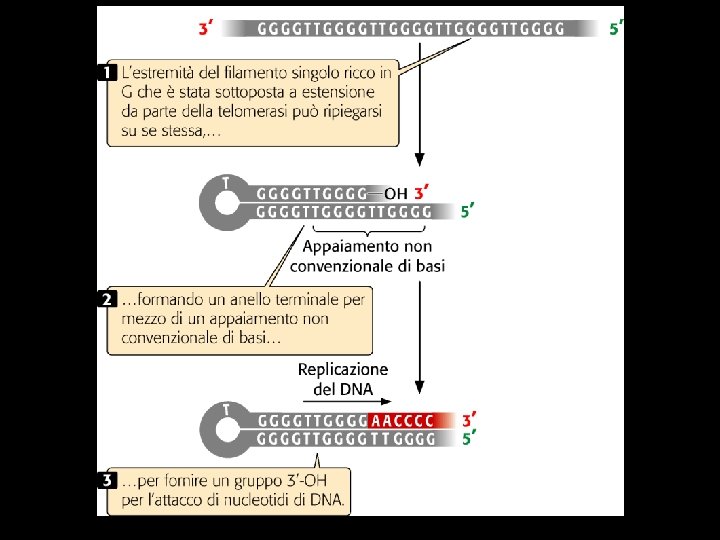
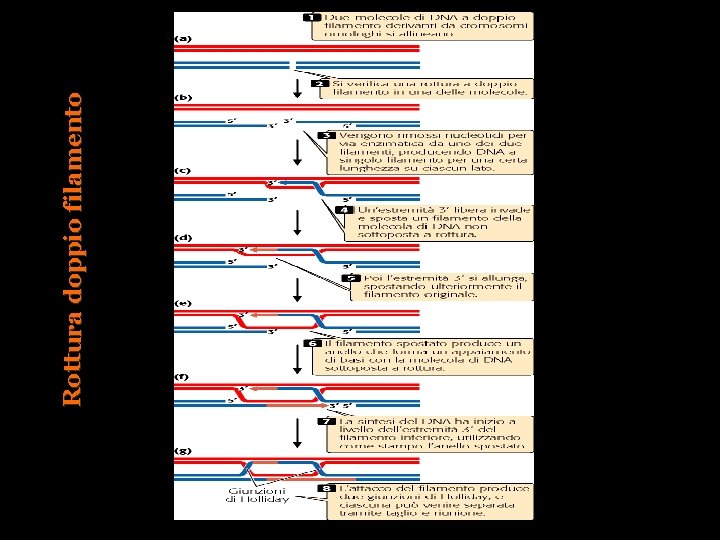
Rottura doppio filamento





