VIRUS A DNA Replicazione nucleare 1 2 DNA













- Slides: 29

VIRUS A DNA Replicazione nucleare 1. 2. DNA lineare DNAss = Parvovirus DNAds = Adenovirus - Herpesvirus DNAds circolare = Poliomavirus e Papillomavirus Replicazione citoplasmatica POXVIRUS Replicazione nucleare e citoplasmatica HEPADNAVIRUS 1

TRASCRIZIONE DEI VIRUS A DNA Nucleare utilizzano la RNA pol II cellulare (ecc. POXVIRUS) Adenovirus POXVIRUS: Utilizzano enzimi presenti nel core del virus. Trascritti senza introni. Assenza di splicing 2

TRASCRIZIONE DEI VIRUS A DNA Proteine virali regolano l’interazione di fattori trascrizionali cellulari con sequenze promoter o enhancer al 5’ dei geni virali (es: HSV-1= VP 16) ØTrascrizione di geni su filamenti diversi di DNA (es: SV 40 = early e late su filamenti opposti) ØPossibilità di m. RNA policistronici con introni Splicing intranucleare Aggiunta di catene di poli A (100 -200 residui di adenina) al 3’ e cap metilato al 5’ 3

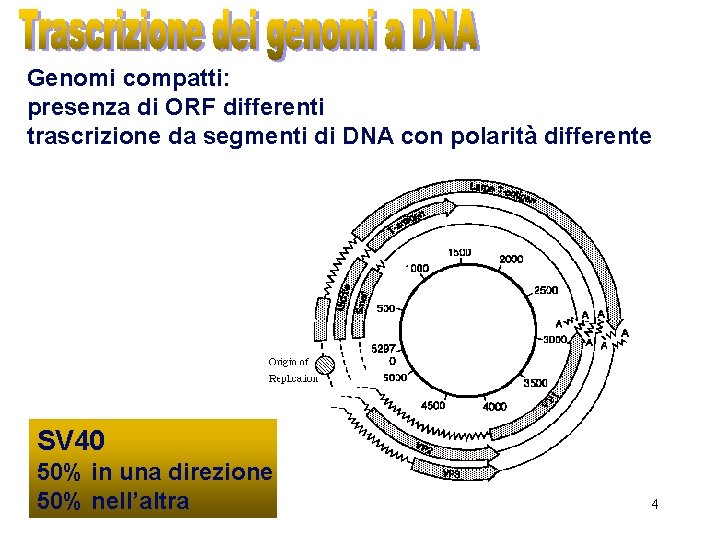
Genomi compatti: presenza di ORF differenti trascrizione da segmenti di DNA con polarità differente Es schema genoma papova SV 40 50% in una direzione 50% nell’altra 4

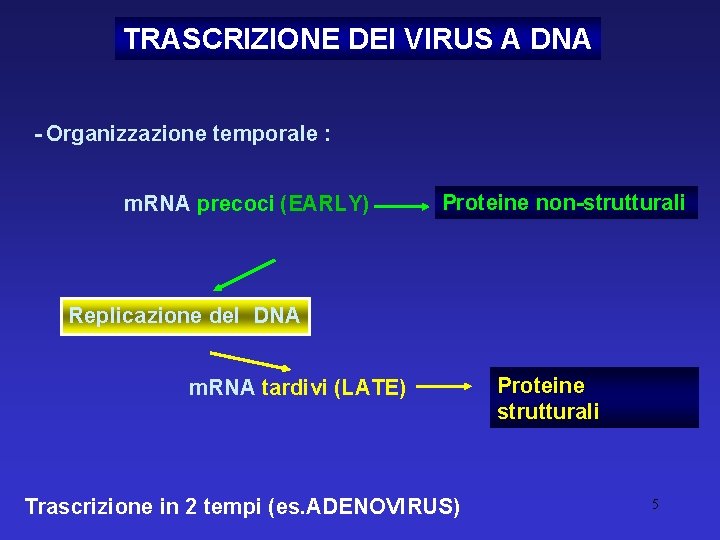
TRASCRIZIONE DEI VIRUS A DNA - Organizzazione temporale : m. RNA precoci (EARLY) Proteine non-strutturali Replicazione del DNA m. RNA tardivi (LATE) Trascrizione in 2 tempi (es. ADENOVIRUS) Proteine strutturali 5

TRASCRIZIONE NEI VIRUS A DNA HERPESVIRUS - Organizzazione temporale (in 3 tempi) : Precoci immediati* (CHX insensibili) m. RNA precoci Precoci ritardati (CHX sensibili) Replicazione del DNA m. RNA tardivi * Utilizzano fattori di trascrizione virali (a-TIF, nel virione) e cellulari (NF-k. B) 6

REPLICAZIONE del DNA VIRALE SEMICONSERVATIVA con vari intermedi di replicazione Parvovirus - Papillomavirus-Poliomavirus: DNA pol cellulare Adenovirus - Herpesvirus: DNA pol virale > velocità > errori Target per antivirali (acyclovir) 7

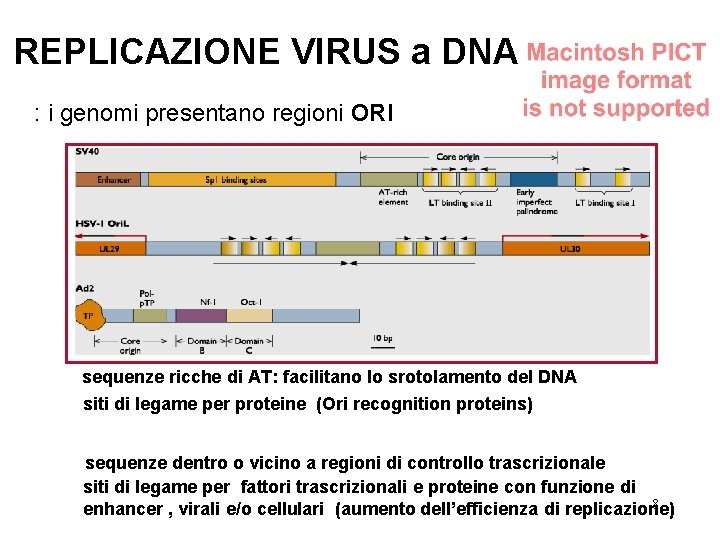
REPLICAZIONE VIRUS a DNA : i genomi presentano regioni ORI sequenze ricche di AT: facilitano lo srotolamento del DNA siti di legame per proteine (Ori recognition proteins) sequenze dentro o vicino a regioni di controllo trascrizionale siti di legame per fattori trascrizionali e proteine con funzione di 8 enhancer , virali e/o cellulari (aumento dell’efficienza di replicazione)

Replicazione del genoma dei virus a DNA : le proteine di riconoscimento della regione ORI Tutti i virus a DNA codificanore almeno una proteina per iniziare la replicazione del genoma lega la regione ORI del genoma virale. il legame della/e proteine tende a distorcere la regione ORI La distorzione della regione ORI facilita il reclutamento di proteine (virali o celluleri) ad attività di elicasi ATP-dipendente che permette lo srotolamento del DNA virale I virus più grandi codificano la DNA polimerasi ed altre proteine necessarie per la replicazione del genoma 9

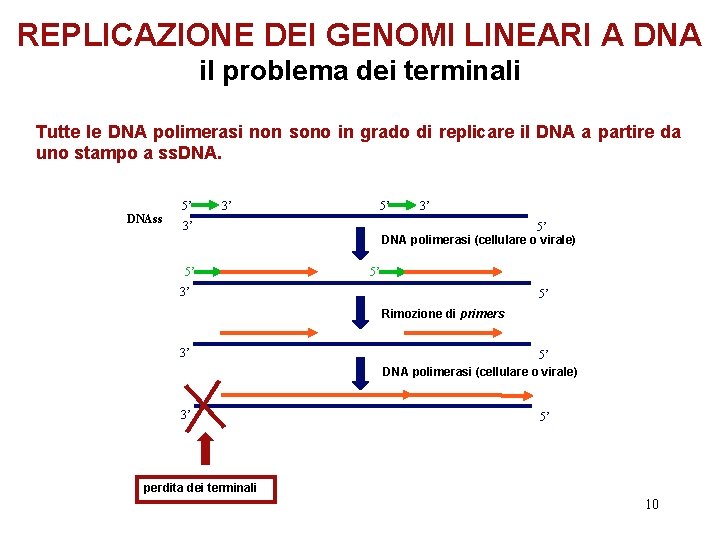
REPLICAZIONE DEI GENOMI LINEARI A DNA il problema dei terminali Tutte le DNA polimerasi non sono in grado di replicare il DNA a partire da uno stampo a ss. DNAss 5’ 5’ 3’ 3’ 5’ DNA polimerasi (cellulare o virale) 5’ 3’ 5’ Rimozione di primers 3’ 3’ 5’ DNA polimerasi (cellulare o virale) 5’ perdita dei terminali 10

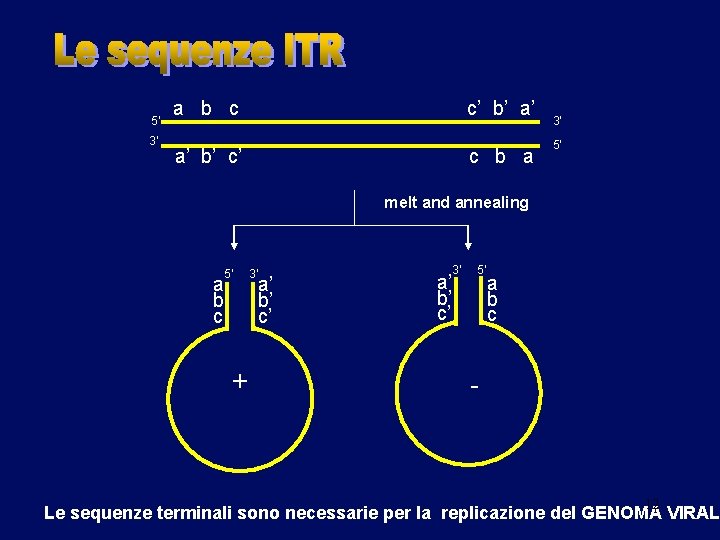
Ridondanza terminale: presenza di “terminal repeats” (TR) (genoma di Adenovirus) Sequenze identiche ……. . . e di “inverted terminal repeats” (ITR) (genoma di Poxvirus, Parvovir ed Herpesvirus). 5’ 3’ a b c c’ b’ a’ a’ b’ c’ c b a 3’ 5’ La lunghezza dei ITR varia da 20 a 150 residui nucleotidici. Nei Poxvirus è di oltre 10. 000 basi (> 5% del genoma) 11

5’ 3’ a b c c’ b’ a’ a’ b’ c’ c b a 3’ 5’ melt and annealing a b c 5’ + 3’ a’ b’ c’ 5’ a b c - 12 Le sequenze terminali sono necessarie per la replicazione del GENOMA VIRAL

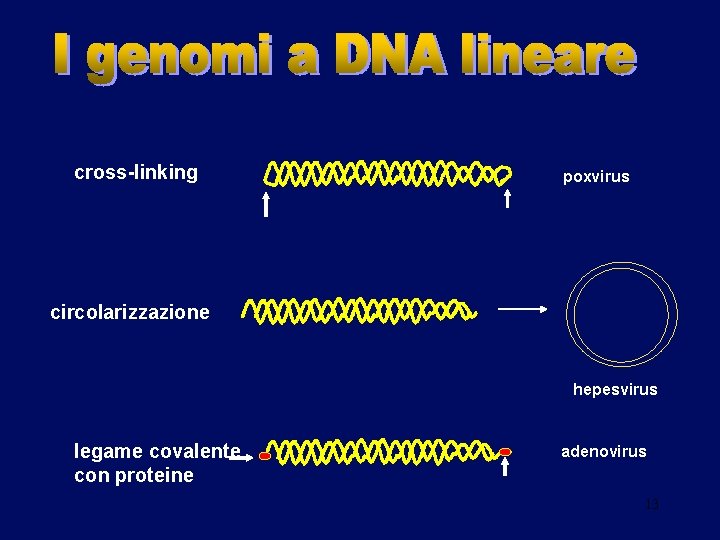
cross-linking poxvirus circolarizzazione hepesvirus legame covalente con proteine adenovirus 13

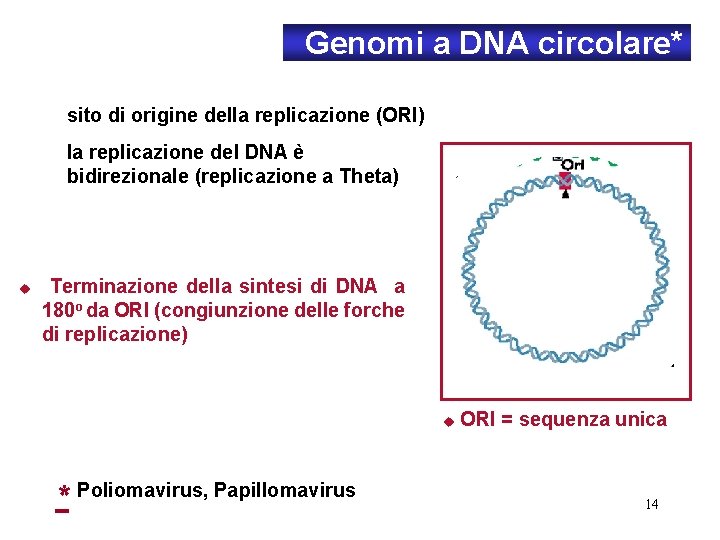
Genomi a DNA circolare* sito di origine della replicazione (ORI) la replicazione del DNA è bidirezionale (replicazione a Theta) u Terminazione della sintesi di DNA a 180 o da ORI (congiunzione delle forche di replicazione) u * Poliomavirus, Papillomavirus ORI = sequenza unica 14

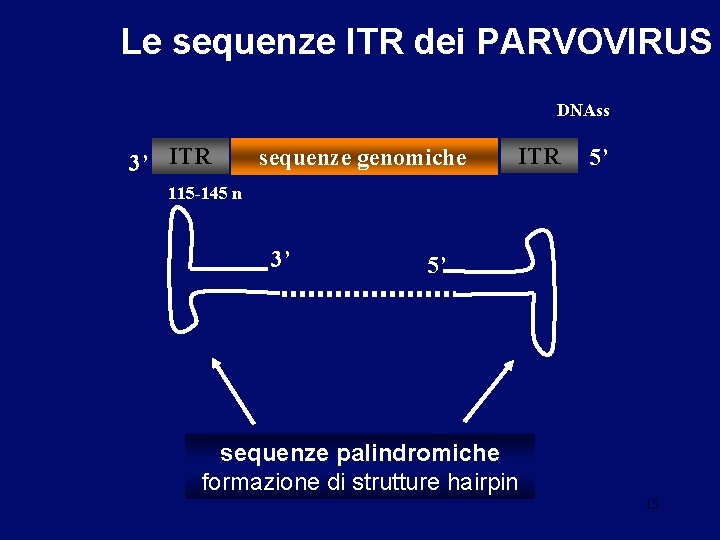
Le sequenze ITR dei PARVOVIRUS DNAss 3’ ITR sequenze genomiche ITR 5’ 115 -145 n 3’ 5’ sequenze palindromiche formazione di strutture hairpin 15

Replicazione Hepadnavirus nel nucleo genoma ds. DNA (circolare incompleto) episomale m RNA sintetizzati dalla pol II cellulare pregenoma a RNA nel citoplasma P (RT/RNasi H) retrotrascrizione RNA: RNA/DNA: DNA/DNA 16

VIRUS A RNA Replicazione citoplasmatica 1. RNA lineare ss -polarità positiva -polarita negativa 2. RNAds = Reovirus Replicazione nucleare ORTHMYXOVIRUS e RETROVIRUS 17

REPLICAZIONE dei virus a RNA polimerasi RNA-dipendenti (Rp. Rd) non utilizzano PRIMERS (la trascrizione e/o replicazione dell’ RNA inizia all’estremità della molecola lineare) > velocità assenza di proof-reading èCaratteristica delle RNA polimerasi RNA dipendenti Resistenti a sostanze che inibiscono le RNA polimerasi DNAdipendenti (Actinomicina D) 18

TRASCRIZIONE di VIRUS a RNA i virus ad RNA non hanno elementi di controllo dell’espressione genica simili a quelli dei virus a DNA meccanismi differenti per regolare l’espressione dei geni virali gli m. RNA virali devono essere organizzati e tradotti come gli m. RNA cellulari i virus a RNA eucariotici devono avere una struttura genomica che genera m. RNA monocistronici 19

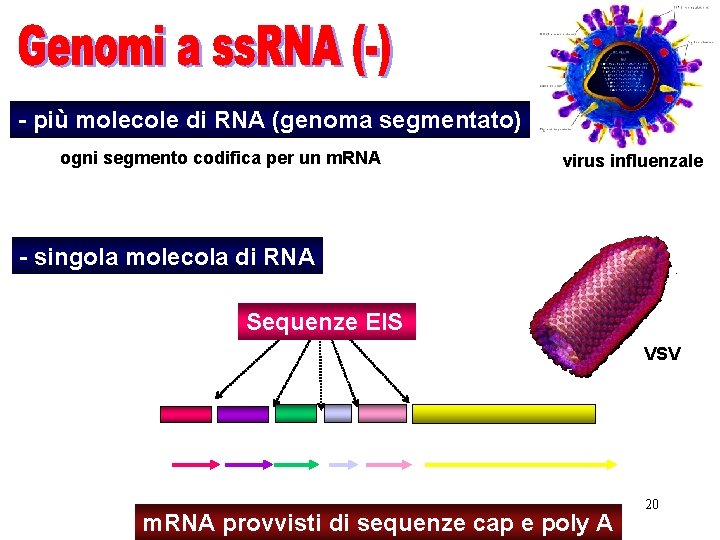
- più molecole di RNA (genoma segmentato) ogni segmento codifica per un m. RNA virus influenzale - singola molecola di RNA Sequenze EIS VSV 3’ m. RNA provvisti di sequenze cap e poly A 5’ 20

Struttura secondaria della sequenza IRES (Internal Ribosome Entry Site) presente al 5’ del RNA genomico. Necessaria per la funzione del genoma come m. RNA. IRES di POLIOVIRUS 21

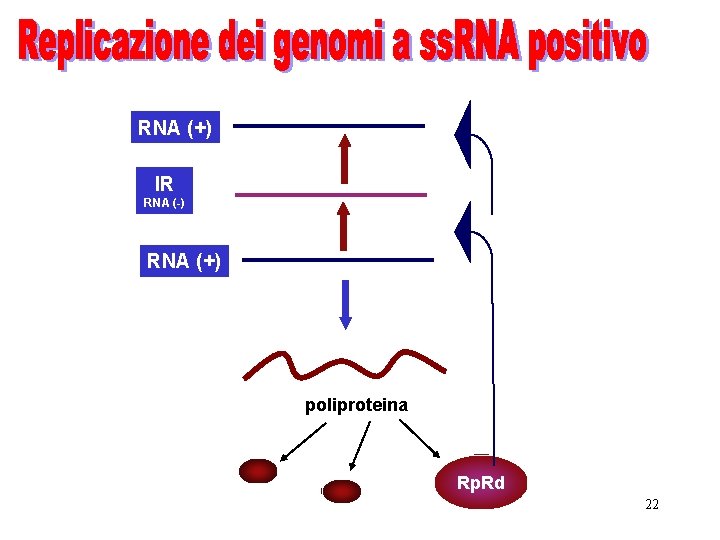
RNA (+) IR RNA (-) RNA (+) poliproteina Rp. Rd 22

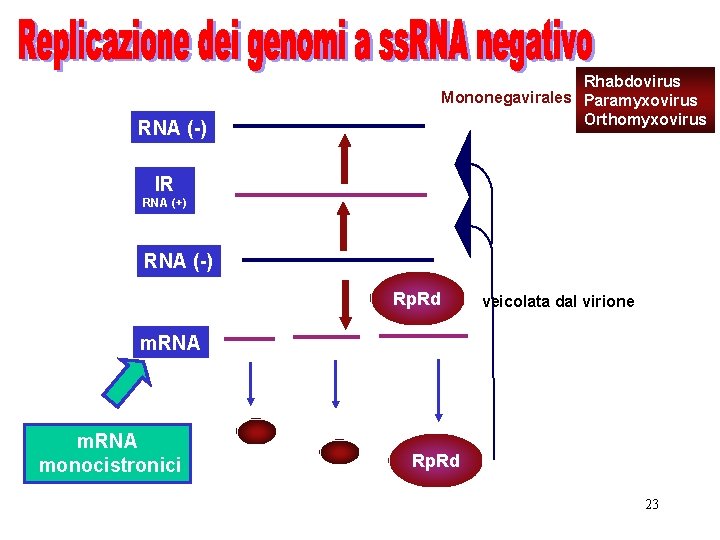
Rhabdovirus Mononegavirales Paramyxovirus Orthomyxovirus RNA (-) IR RNA (+) RNA (-) Rp. Rd veicolata dal virione m. RNA monocistronici Rp. Rd 23

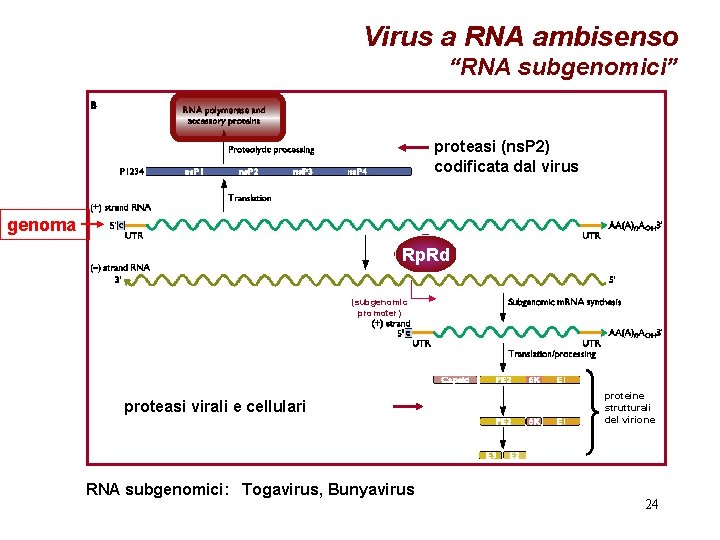
Virus a RNA ambisenso “RNA subgenomici” proteasi (ns. P 2) codificata dal virus genoma Rp. Rd (subgenomic promoter) proteasi virali e cellulari RNA subgenomici: Togavirus, Bunyavirus proteine strutturali del virione 24

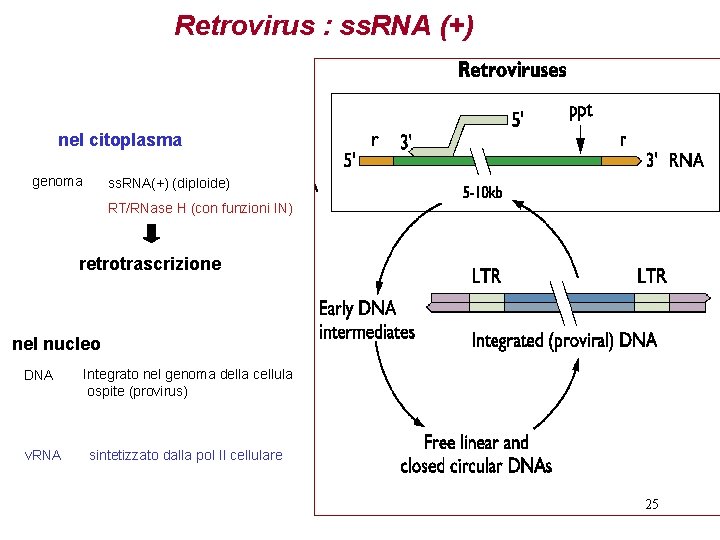
Retrovirus : ss. RNA (+) nel citoplasma genoma ss. RNA(+) (diploide) RT/RNase H (con funzioni IN) retrotrascrizione nel nucleo DNA v. RNA Integrato nel genoma della cellula ospite (provirus) sintetizzato dalla pol II cellulare 25

MATURAZIONE E LIBERAZIONE DEI VIRUS ANIMALI 1. Assemblaggio del capside (procapside) 2. Formazione del nucleocapside Virus a DNA nel NUCLEO (ecc. POXVIRUS - HEPADNAVIRUS) Virus a RNA nel CITOPLASMA (ecc. ORTHOMYXOVIRUS)) LISI 3. LIBERAZIONE GEMMAZIONE ESOCITOSI 26

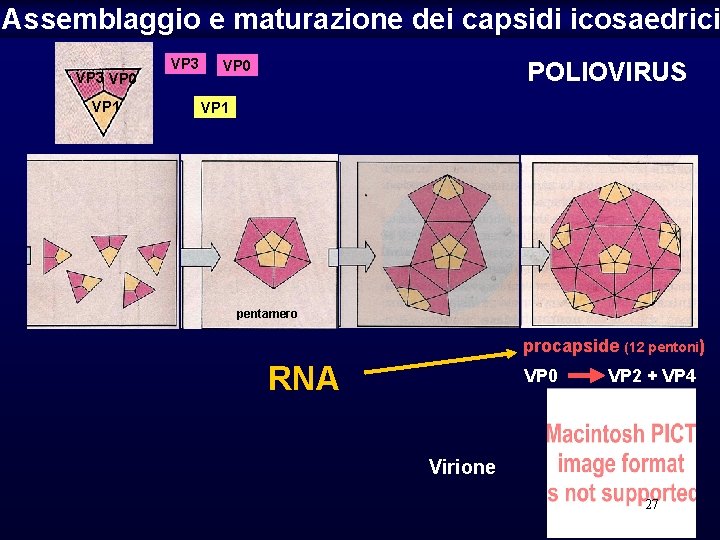
Assemblaggio e maturazione dei capsidi icosaedrici VP 3 VP 0 VP 1 VP 3 VP 0 POLIOVIRUS VP 1 pentamero procapside (12 pentoni) RNA VP 0 VP 2 + VP 4 Virione 27

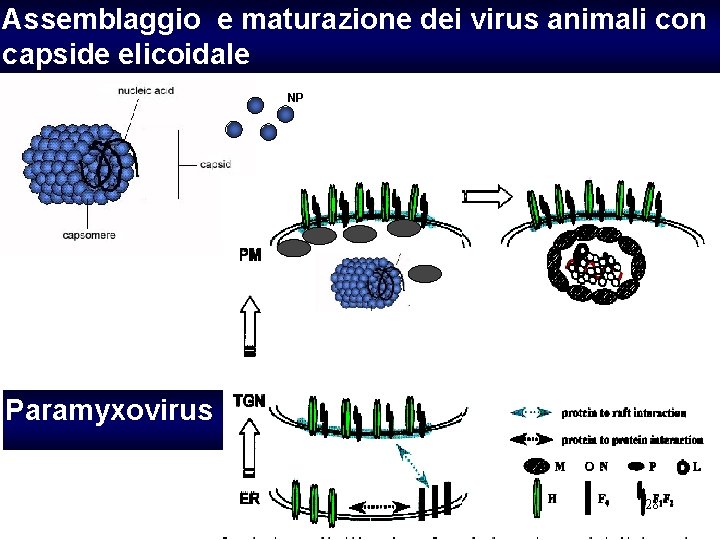
Assemblaggio e maturazione dei virus animali con capside elicoidale NP Paramyxovirus 28

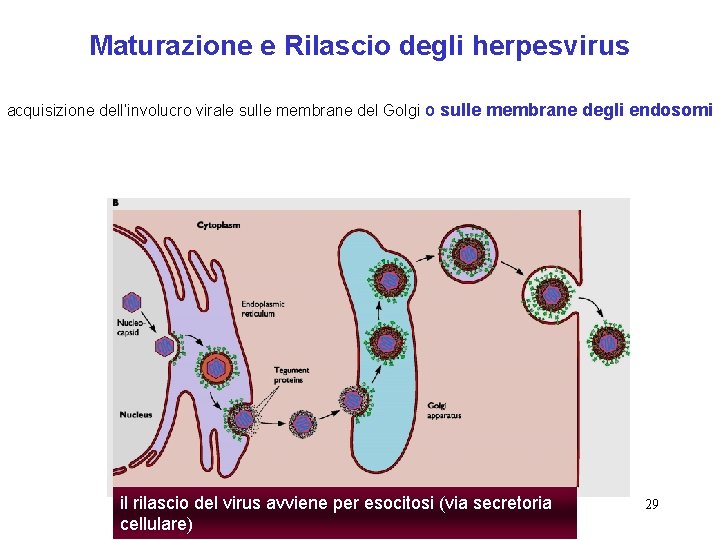
Maturazione e Rilascio degli herpesvirus acquisizione dell’involucro virale sulle membrane del Golgi o sulle membrane degli endosomi ? il rilascio del virus avviene per esocitosi (via secretoria cellulare) 29
 Codice genetico ridondante e degenerato
Codice genetico ridondante e degenerato Dna polimerasi teta
Dna polimerasi teta Replicazione a circolo rotante
Replicazione a circolo rotante Replicazione a cerchio rotante
Replicazione a cerchio rotante Perbedaan replikasi virus dna dan rna
Perbedaan replikasi virus dna dan rna Corona virus dna or rna
Corona virus dna or rna Viral dna
Viral dna Reattore nucleare
Reattore nucleare Cataratta polare anteriore
Cataratta polare anteriore Testo informativo espositivo
Testo informativo espositivo Fermi unità di misura
Fermi unità di misura Fusione nucleare
Fusione nucleare Interazione nucleare debole
Interazione nucleare debole La frase nucleare scuola primaria
La frase nucleare scuola primaria Fissione nucleare
Fissione nucleare Funzionamento centrale nucleare
Funzionamento centrale nucleare Forza debole
Forza debole Fusione nucleare schema
Fusione nucleare schema Interazione nucleare debole
Interazione nucleare debole Funzionamento centrale nucleare
Funzionamento centrale nucleare Detector de radiatii nucleare
Detector de radiatii nucleare Energia nucleare riassunto
Energia nucleare riassunto Marcato pleomorfismo nucleare
Marcato pleomorfismo nucleare Ran gef and ran gap
Ran gef and ran gap Fissione nucleare
Fissione nucleare Interazione nucleare debole
Interazione nucleare debole Interazione nucleare debole
Interazione nucleare debole Esperimento thomson zanichelli
Esperimento thomson zanichelli Fusione nucleare
Fusione nucleare Medicina nucleare spedali civili brescia
Medicina nucleare spedali civili brescia