STRUTTURA TERZIARIA STRUTTURA NATIVA Ripiegamentoconformazione locale della catena










- Slides: 28

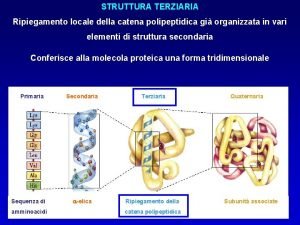
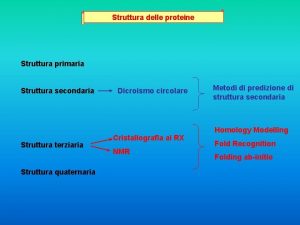
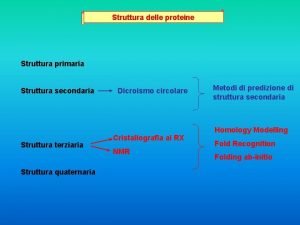
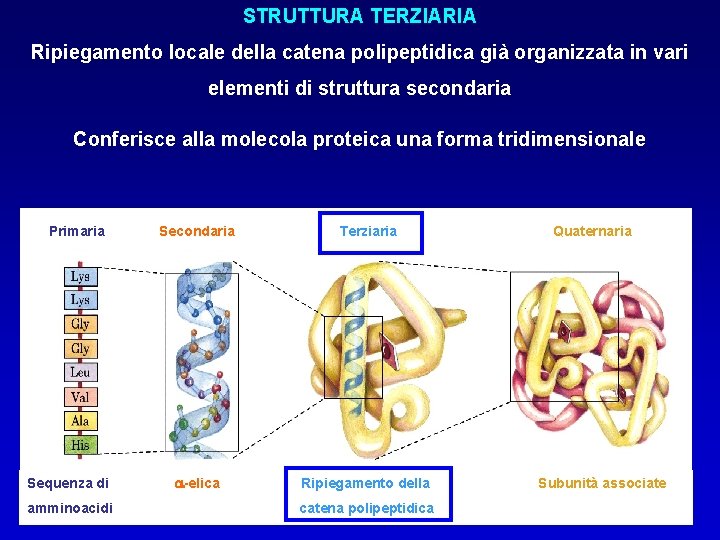
STRUTTURA TERZIARIA STRUTTURA NATIVA Ripiegamentoconformazione locale della catena polipeptidica già organizzata in vari funzionale di una proteina elementi di struttura secondaria CONFORMAZIONE organizzazione spaziale degli una atomi in una proteina Conferisce alla molecola proteica forma tridimensionale La FUNZIONE di una proteina è garantita dalla sua conformazione nativa Primaria Sequenza di amminoacidi Secondaria Terziaria a-elica Ripiegamento della catena polipeptidica Quaternaria Subunità associate

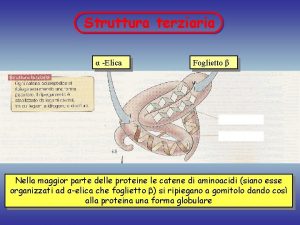
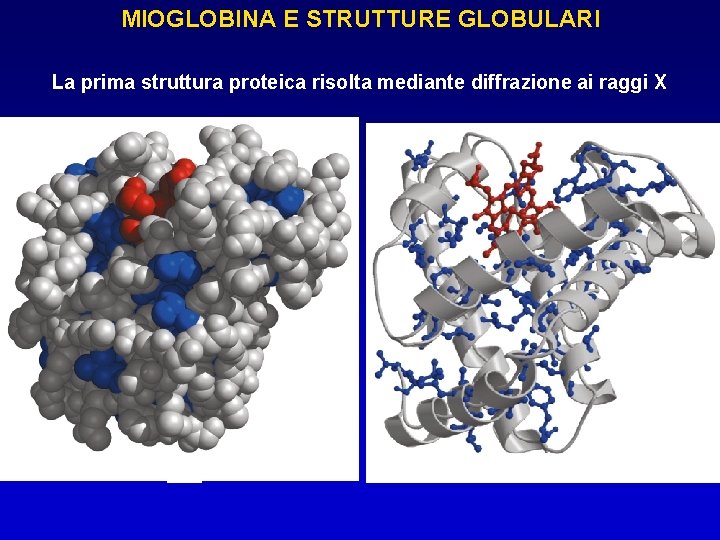
MIOGLOBINA E STRUTTURE GLOBULARI La prima struttura proteica risolta mediante diffrazione ai raggi X Le catene laterali dei residui amminoacidici idrofobici si Complessità e mancanza di dispongono all’interno simmetria La superficie ha un carattere prevalentemente idrofilico

STRUTTURE GLOBULARI Le proteine globulari hanno una struttura compatta Tutte le proteine globulari possiedono una parte esterna ed una interna definite Le catene laterali dei residui amminoacidici idrofobici si dispongono all’interno a formare un denso nucleo idrofobico La superficie delle proteine globulari è idrofilica

Esistono infiniti modi di ripiegamento terziario delle proteine? Esistono motivi strutturali ricorrenti nelle strutture proteiche note STRUTTURE SUPERSECONDARIE O MOTIVI Semplici combinazioni di pochi elementi di struttura secondaria arrangiati secondo una geometria specifica

I diversi motivi strutturali si dispongono a strati in modo da proteggere i residui idrofobici dal solvente acquoso Angolo a-a

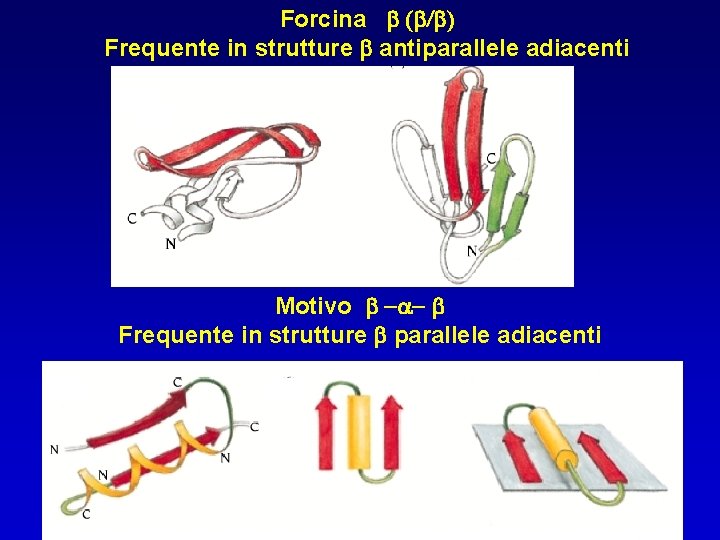
Forcina b (b/b) Frequente in strutture b antiparallele adiacenti Motivo b -a- b Frequente in strutture b parallele adiacenti

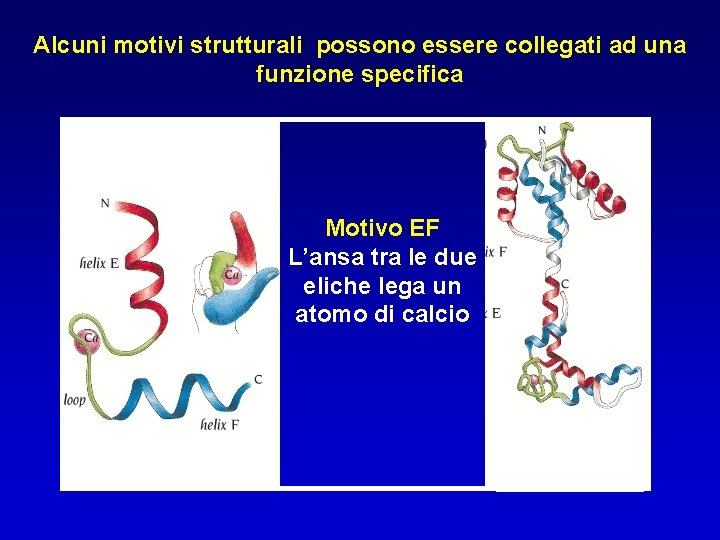
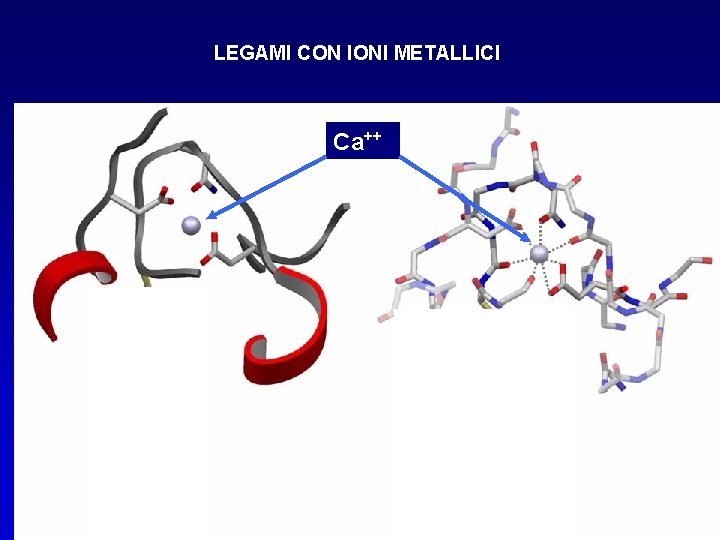
Alcuni motivi strutturali possono essere collegati ad una funzione specifica Motivo EF L’ansa tra le due eliche lega un atomo di calcio

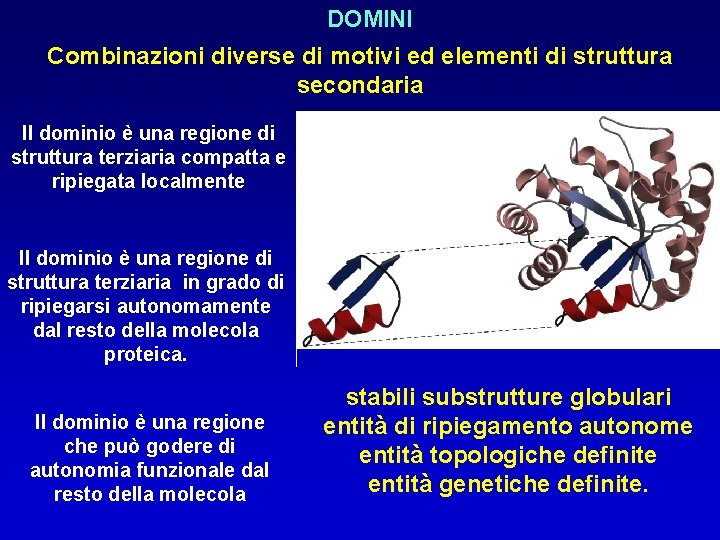
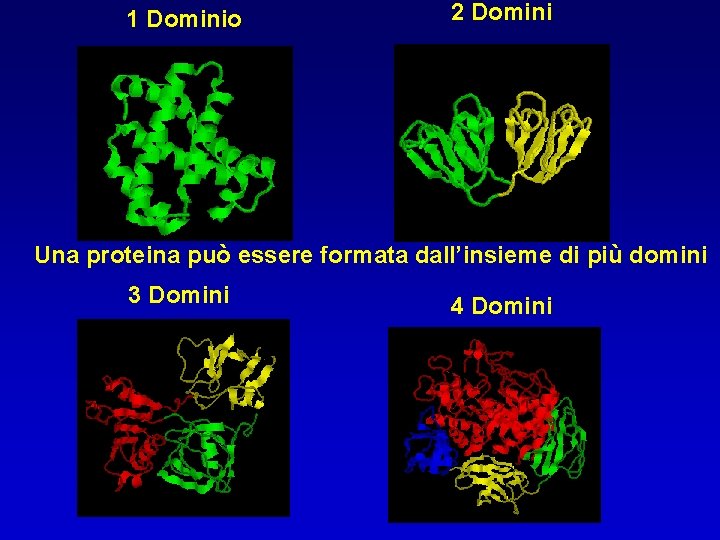
DOMINI Combinazioni diverse di motivi ed elementi di struttura secondaria Il dominio è una regione di struttura terziaria compatta e ripiegata localmente Il dominio è una regione di struttura terziaria in grado di ripiegarsi autonomamente dal resto della molecola proteica. Il dominio è una regione che può godere di autonomia funzionale dal resto della molecola stabili substrutture globulari entità di ripiegamento autonome entità topologiche definite entità genetiche definite.

Alcune combinazioni di motivi strutturali si riscontrano più frequentemente

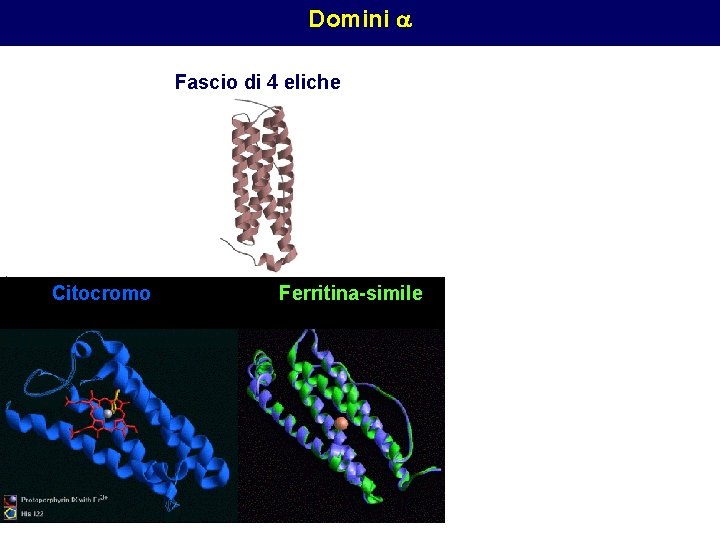
Domini a Fascio di 4 eliche Citocromo Ferritina-simile Fascio di 3 eliche legano il DNA Omeodominio simile

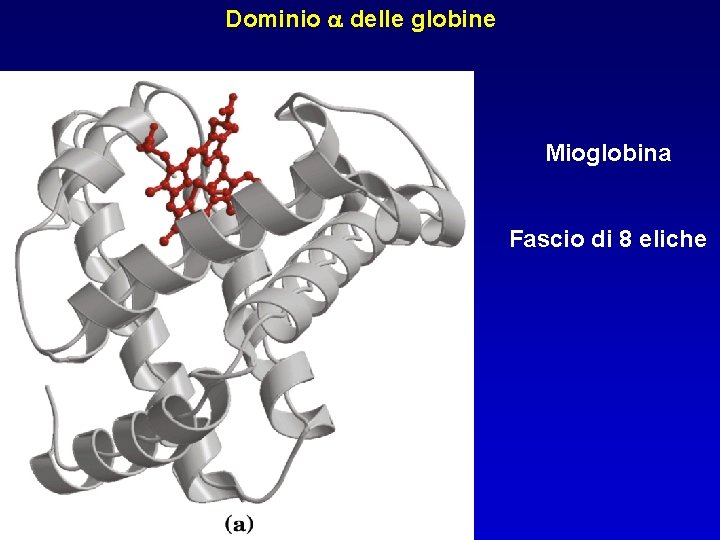
Dominio a delle globine Mioglobina Fascio di 8 eliche

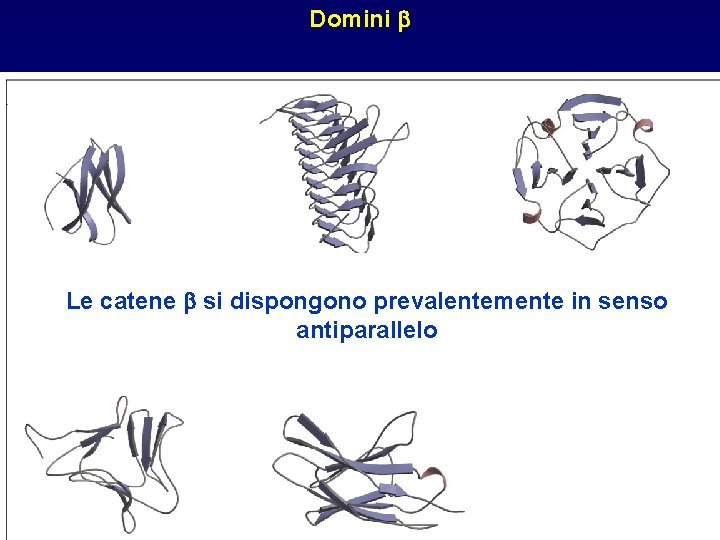
Domini b Le catene b si dispongono prevalentemente in senso antiparallelo

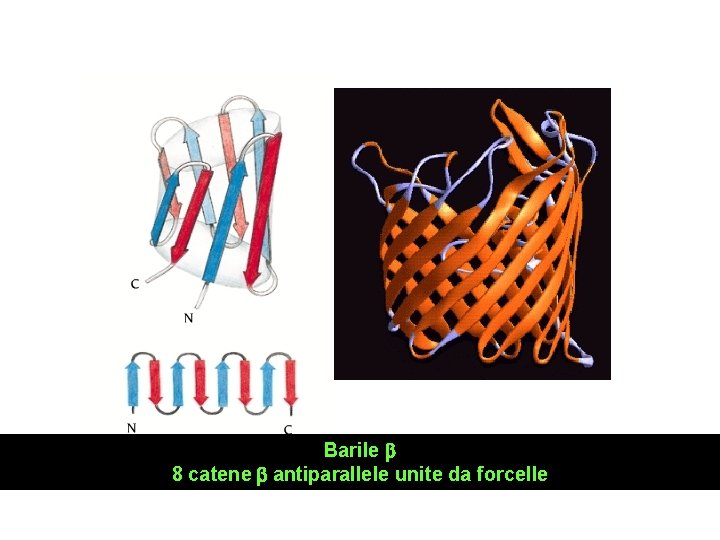
Barile b 8 catene b antiparallele unite da forcelle

Domini a/b Dominio Domini 1 a+b 2 Domini Contengono elementi a e b non combinati secondo i motivi b-a-b Una proteina può essere formata dall’insieme di più domini 3 Domini TIM- barrel Rossman fold 4 Domini Ferro di cavallo

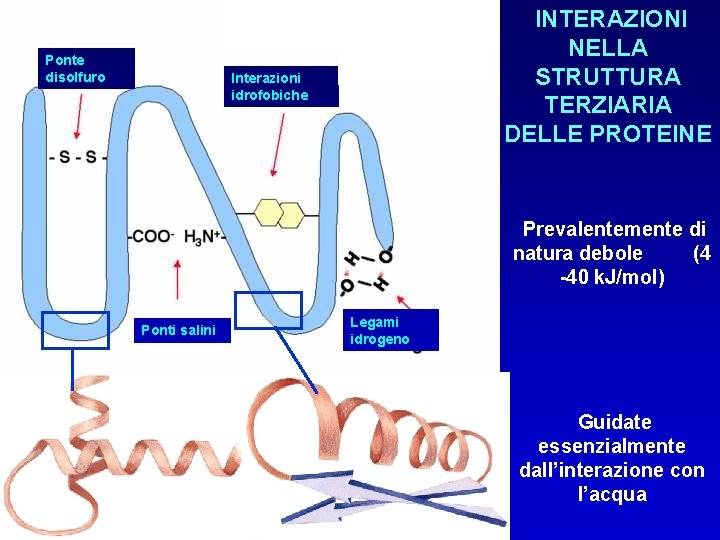
Ponte disolfuro INTERAZIONI NELLA STRUTTURA TERZIARIA DELLE PROTEINE Interazioni idrofobiche Prevalentemente di natura debole (4 -40 k. J/mol) Ponti salini Legami idrogeno Guidate essenzialmente dall’interazione con l’acqua

INTERAZIONI DEBOLI IN AMBIENTE ACQUOSO • Legami idrogeno • Interazioniche H 2 O • Interazioni idrofobiche • Interazioni di Van der Waals Due atomi sufficientemen vicini

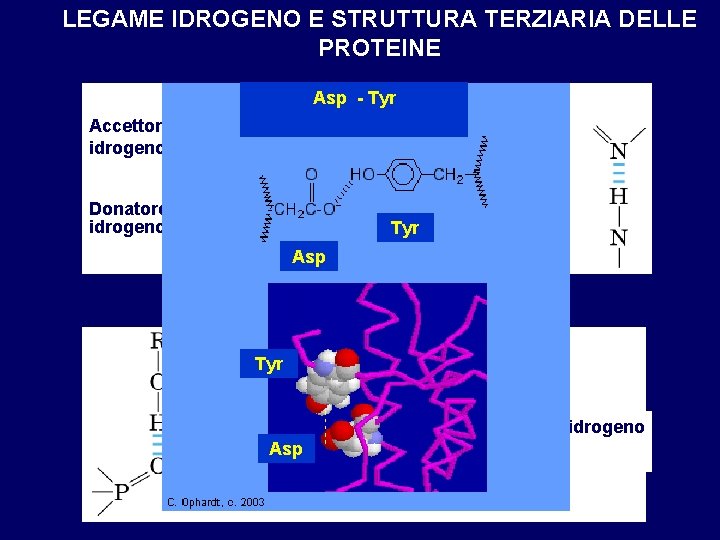
LEGAME IDROGENO E STRUTTURA TERZIARIA DELLE PROTEINE Asp - Tyr Accettore di idrogeno Donatore di idrogeno Tyr Asp Lunghezza di legame: 2, 4 - 3, 5 Å Energia di legame ≈ 4 -21 kj/mol Tyr Legame idrogeno forte Asp Legame idrogeno debole

Lys –carbonile <3. 5 A, 12. 5 -21 k. J/mole LEGAME IDROGENO Ser -NH <3. 5 A, 2 -6 k. J/mole Carbonio Ossigeno Azoto

PONTI SALINI E STRUTTURA TERZIARIA DELLE PROTEINE Arg- Glu - Lys -17 k. J/mol <3. 5 A, 12. 5 Glu Lys

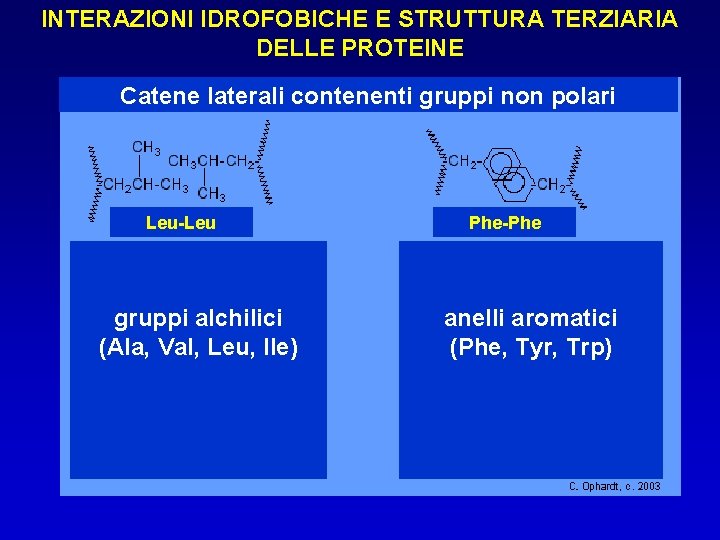
INTERAZIONI IDROFOBICHE E STRUTTURA TERZIARIA DELLE PROTEINE Catene laterali contenenti gruppi non polari Leu-Leu gruppi alchilici (Ala, Val, Leu, Ile) Phe-Phe anelli aromatici (Phe, Tyr, Trp)

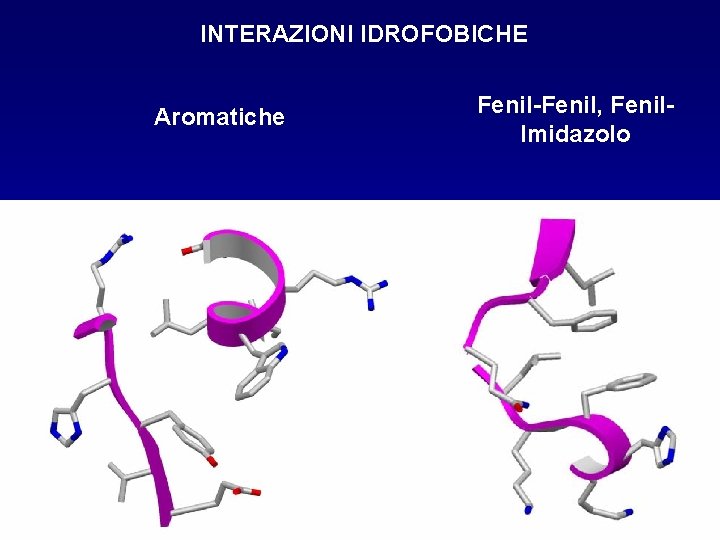
INTERAZIONI IDROFOBICHE Aromatiche Fenil-Fenil, Fenil. Imidazolo

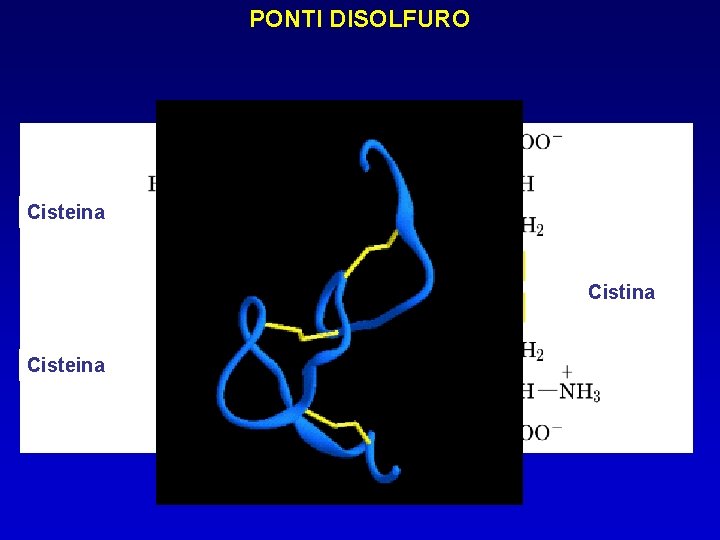
PONTI DISOLFURO Cisteina

LEGAMI CON IONI METALLICI Ca++

Cosa determina queste possibilità di ripiegamento? Dove è scritta la seconda parte del codice genetico? Il paradosso di Levinthal Ribonucleasi = 124 residui; 10100 combinazioni; Tempo per ricerca casuale contatti atomici = 1050 anni Età stimata universo = 1040 anni Tempo osservato = secondi processo spontaneo DG = negativo Il codice di ripiegamento tridimensionale di una proteina è contenuto nella sequenza primaria

STRUTTURE GLOBULARI Le proteine globulari hanno una struttura compatta Tutte le proteine globulari possiedono una parte esterna ed una interna definite Le catene laterali dei residui amminoacidici idrofobici si dispongono all’interno a formare un denso nucleo idrofobico La superficie delle proteine globulari è idrofilica Le interazioni non covalenti tra gli atomi di una proteina sono guidate dalla loro interazione con l’ H 2 O In ambiente acquoso le forze di interazione tra le molecole sono dovute alle proprietà dell’ H 2 O

EFFETTO IDROFOBICO Ogni lipide costringe le molecole di acqua a disporsi in maniera ordinata Riduzione della superficie idrofobica a contatto con l’acqua In ambiente acquoso i residui idrofobici olio tendono ad interagire tra loro H 2 O L’aumentato grado di libertà, cioè l’aumento di entropia del solvente H 2 O è determinante per il acquoso, ripiegamento di una catena polipeptidica Ripiegamento Denaturazione MICELLA Minima superficie idrofobica a contatto con l’acqua MASSIMA ENTROPIA L’effetto idrofobico è la principale forza trainante per il ripiegamento delle proteine Proteina nativa Proteina denaturata

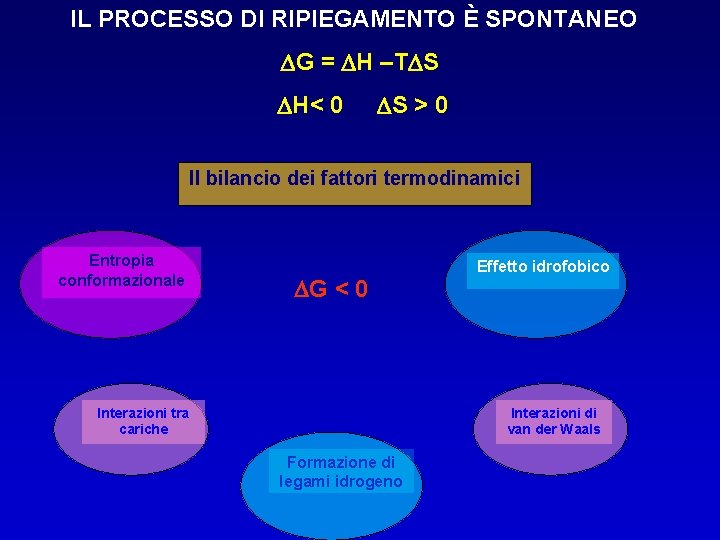
IL PROCESSO DI RIPIEGAMENTO È SPONTANEO DG = DH –TDS DH< 0 DS > 0 Il bilancio dei fattori termodinamici Entropia conformazionale DG < 0 Interazioni tra cariche Effetto idrofobico Interazioni di van der Waals Formazione di legami idrogeno

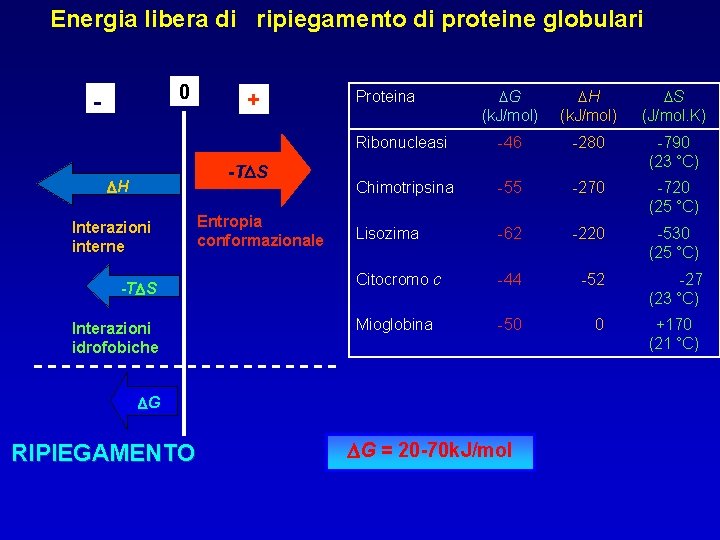
Energia libera di ripiegamento di proteine globulari 0 - + -TDS DH Interazioni interne -TDS Interazioni idrofobiche Entropia conformazionale DG (k. J/mol) DH (k. J/mol) DS (J/mol. K) Ribonucleasi -46 -280 -790 (23 °C) Chimotripsina -55 -270 -720 (25 °C) Lisozima -62 -220 -530 (25 °C) Citocromo c -44 -52 -27 (23 °C) Mioglobina -50 0 +170 (21 °C) Proteina DG RIPIEGAMENTO DG = 20 -70 k. J/mol
 Struttura terziaria proteine
Struttura terziaria proteine Denaturazione proteine
Denaturazione proteine Struttura terziaria proteine
Struttura terziaria proteine Struttura terziaria proteine
Struttura terziaria proteine Struttura terziaria proteine
Struttura terziaria proteine Significado de nativa
Significado de nativa Catena alimentare da disegnare
Catena alimentare da disegnare La piramide ecologica scuola primaria
La piramide ecologica scuola primaria Piramide alimentare savana
Piramide alimentare savana Legame peptidico
Legame peptidico Rna
Rna Punto eutettico ferro carbonio
Punto eutettico ferro carbonio Lentelocale
Lentelocale Polizia locale regione lombardia
Polizia locale regione lombardia Recherche tabou python
Recherche tabou python Locale theory
Locale theory Counterclaim and claim
Counterclaim and claim Pial mission locale
Pial mission locale Statement of the problem and hypothesis in research example
Statement of the problem and hypothesis in research example Sfera locale topografia
Sfera locale topografia Lente locale
Lente locale Mission locale muret
Mission locale muret Polizia locale terre estensi ferrara voghiera masi torello
Polizia locale terre estensi ferrara voghiera masi torello Subduzione
Subduzione Elementi fiaba
Elementi fiaba Forma-sonata schema
Forma-sonata schema Colosso di nerone
Colosso di nerone Regno delle piante
Regno delle piante Cos'è la fabula e cos'è l'intreccio
Cos'è la fabula e cos'è l'intreccio