Gntique des populations empirique Thierry De Mees Interactions









![Génotype et phénotype Bb. Cc=Génotype [B]=Phénotype Le phénotype ne reflète pas nécessairement le génotype Génotype et phénotype Bb. Cc=Génotype [B]=Phénotype Le phénotype ne reflète pas nécessairement le génotype](https://slidetodoc.com/presentation_image/b87bb4c1ab851771c870557e375a1f55/image-32.jpg)













- Slides: 63

Génétique des populations empirique Thierry De Meeûs Interactions hôtes - vecteurs – parasites-environnement dans les maladies tropicales négligées dues aux trypanosomatidae - (INTERTRYP), UMR IRD/CIRAD 177, TA A-17/G, Campus International de Baillarguet, 34398, Montpellier Cedex 5, È +33 (0)4 67 59 37 44 thierry. demeeus@ird. fr http: //t-de-meeus. fr/Tde. Meeus. html

Introduction


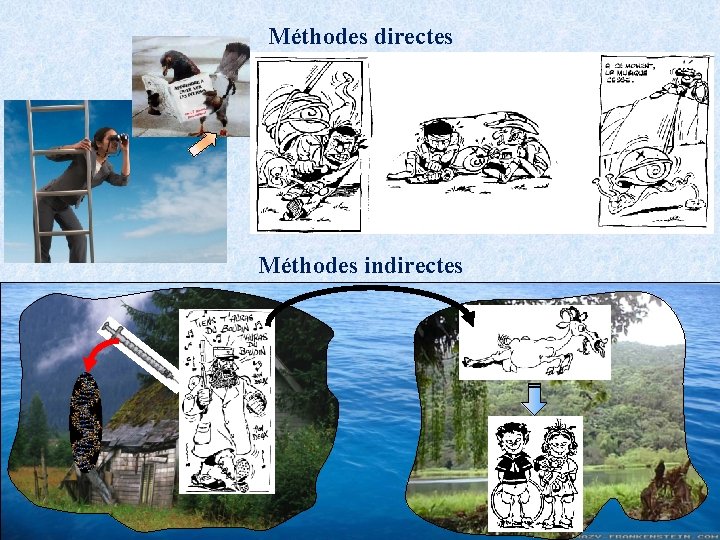
Méthodes directes Méthodes indirectes

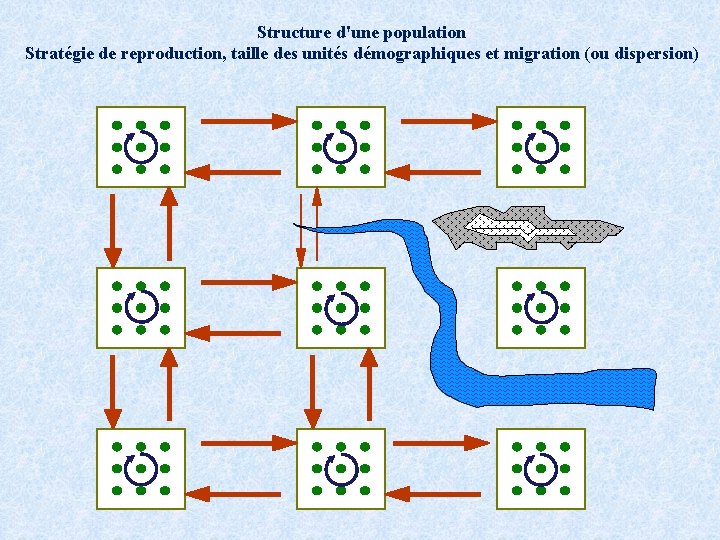
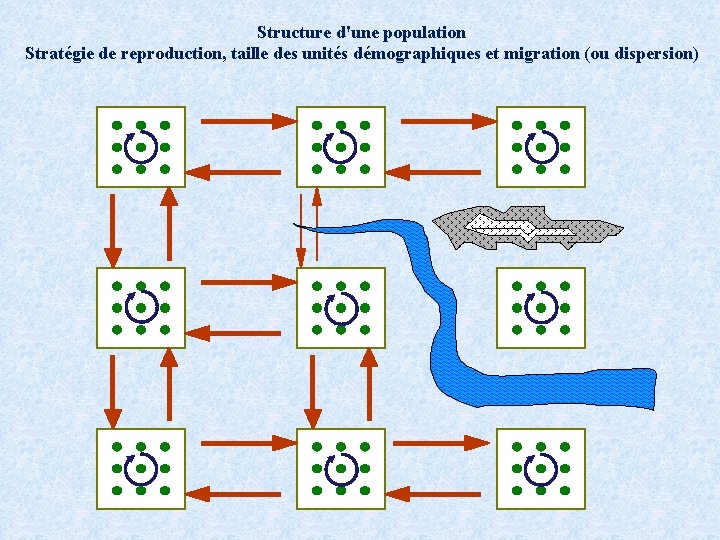
Structure d'une population Stratégie de reproduction, taille des unités démographiques et migration (ou dispersion)

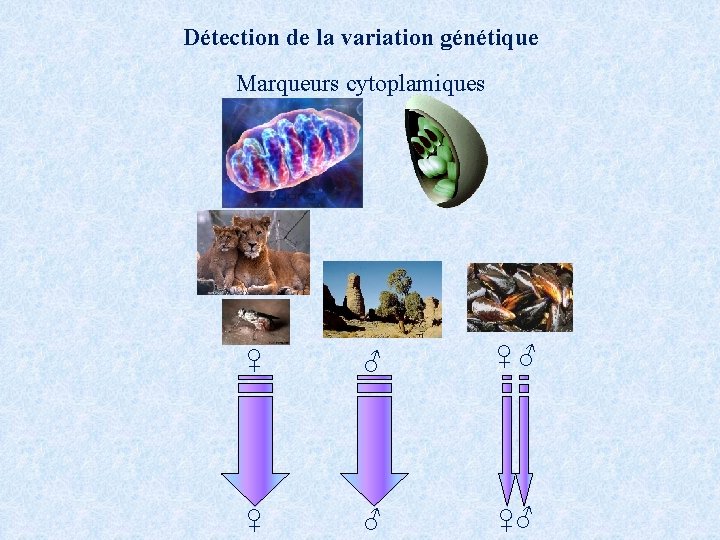
Détection de la variation génétique Marqueurs cytoplamiques ♀ ♂ ♀♂

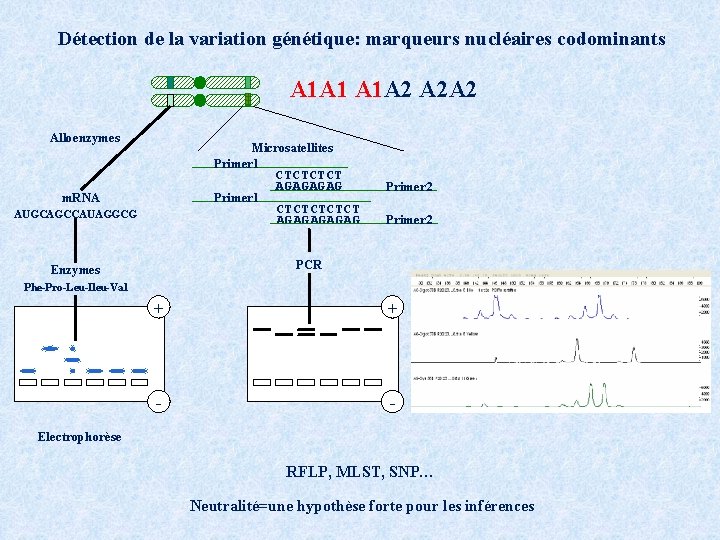
Détection de la variation génétique: marqueurs nucléaires codominants A 1 A 1 A 1 A 2 A 2 A 2 Alloenzymes Microsatellites Primer 1 m. RNA Primer 1 AUGCAGCCAUAGGCG CTCT AGAG Primer 2 CTCTCT AGAGAG Primer 2 PCR Enzymes Phe-Pro-Leu-Ileu-Val + + - - Electrophorèse RFLP, MLST, SNP… Neutralité=une hypothèse forte pour les inférences

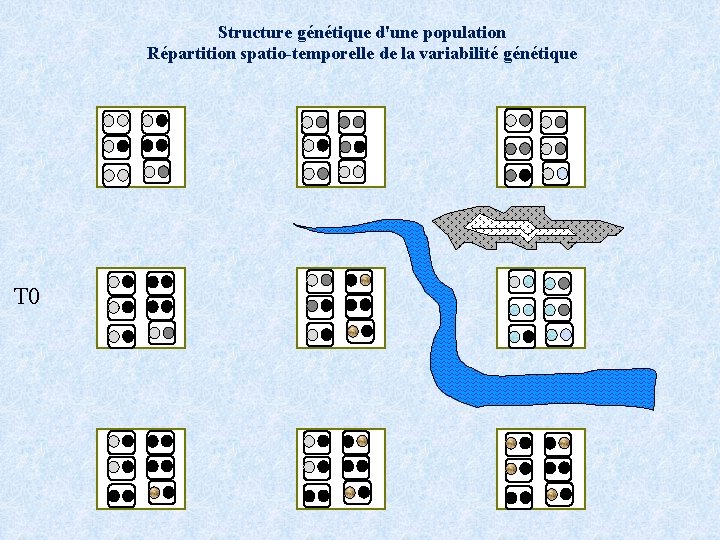
Structure génétique d'une population Répartition spatio-temporelle de la variabilité génétique T 0

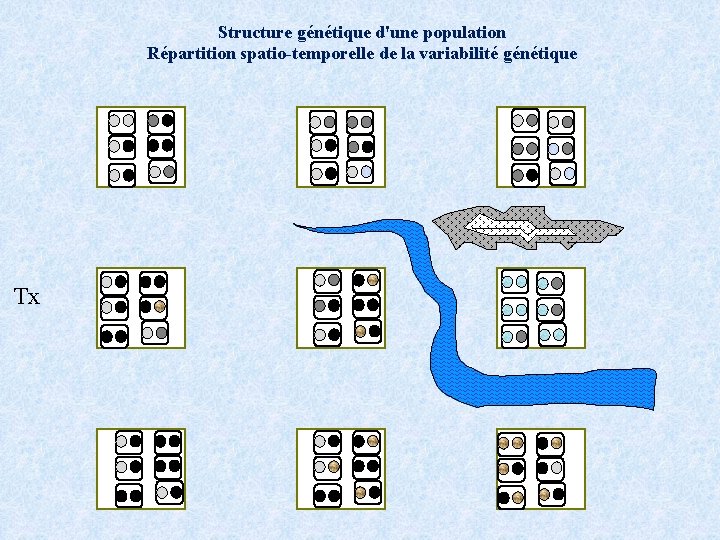
Structure génétique d'une population Répartition spatio-temporelle de la variabilité génétique Tx

Structure d'une population Stratégie de reproduction, taille des unités démographiques et migration (ou dispersion)

I. Notions de biologie moléculaire et de génétique formelle

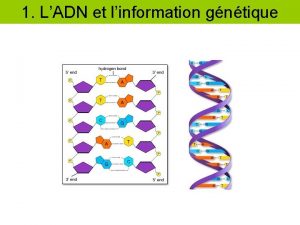
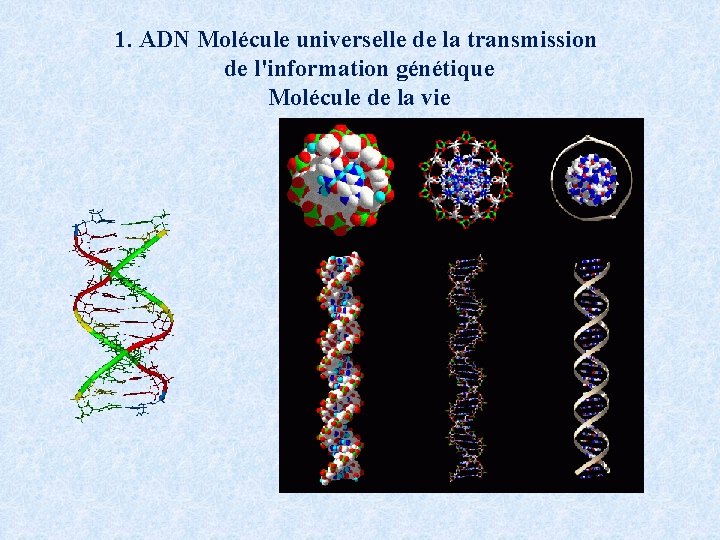
1. ADN Molécule universelle de la transmission de l'information génétique Molécule de la vie

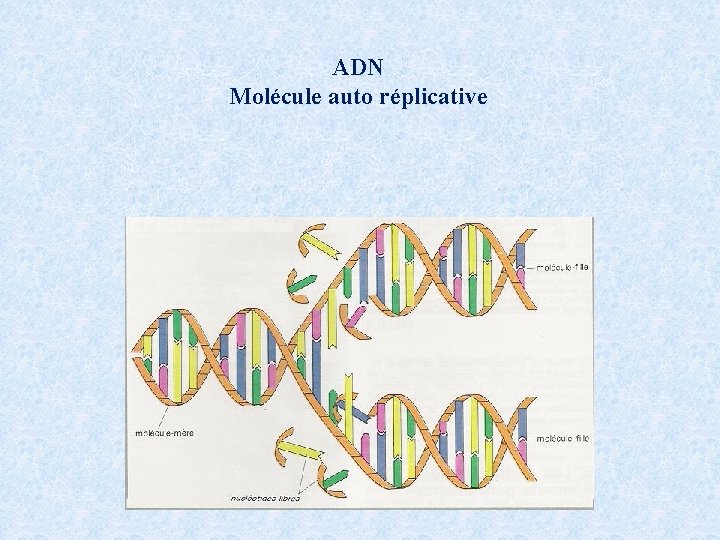
ADN Molécule auto réplicative

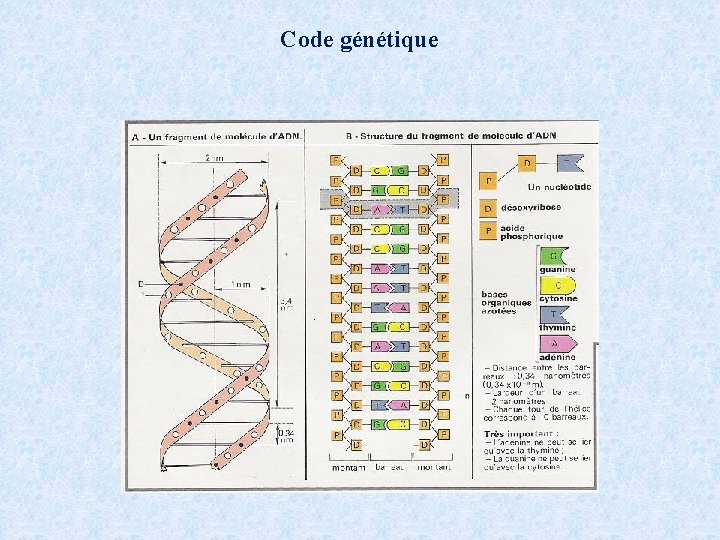
Code génétique

Code génétique: bases puriques et pyrimidiques Purine: Base, constituant essentiel des nucléotides eux même éléments de base des acides nucléiques (ARN et ADN), complémentaires des Pyrimidines. Il en existe deux l'adénine (A) et la guanine (G). Pyrimidines: Base, constituant essentiel des nucléotides eux même élément de base des acides nucléiques (ARN et ADN), complémentaires des purines. Il en existe trois, la thymine (T), l'uracile (U qui prend la place de T dans l'ARN) et la cytosine (C). A est complémentaire de T et U G est complémentaire de C

Code génétique => code génétique "dégénéré"

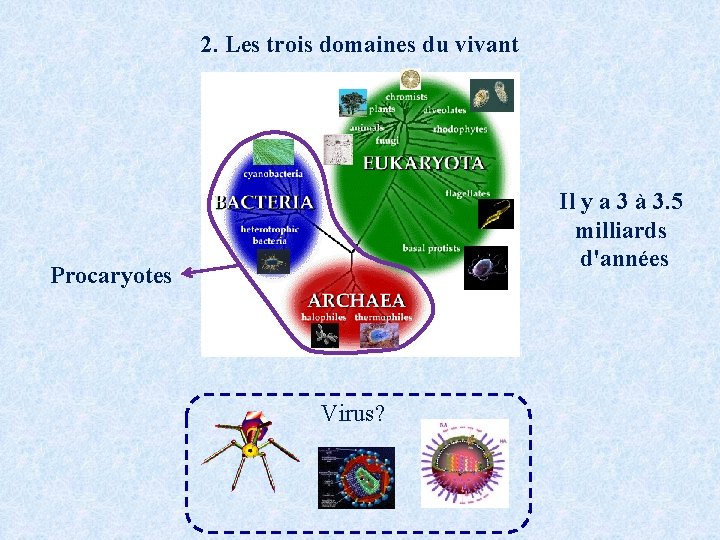
2. Les trois domaines du vivant Il y a 3 à 3. 5 milliards d'années Procaryotes Virus?

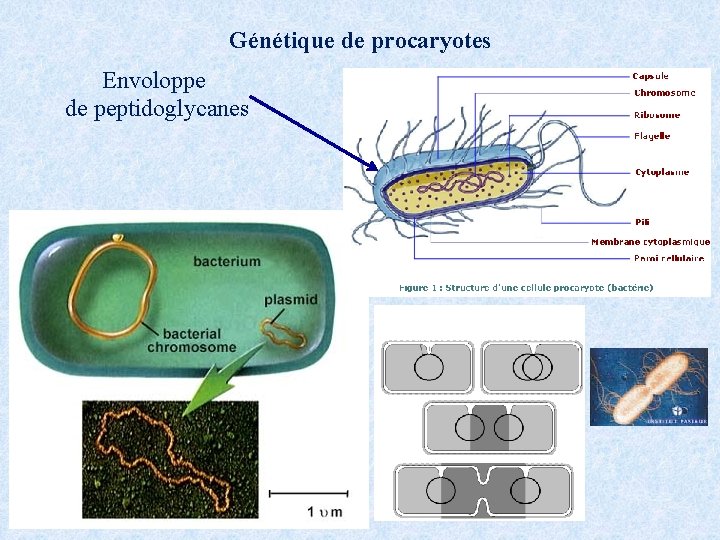
Génétique de procaryotes Envoloppe de peptidoglycanes

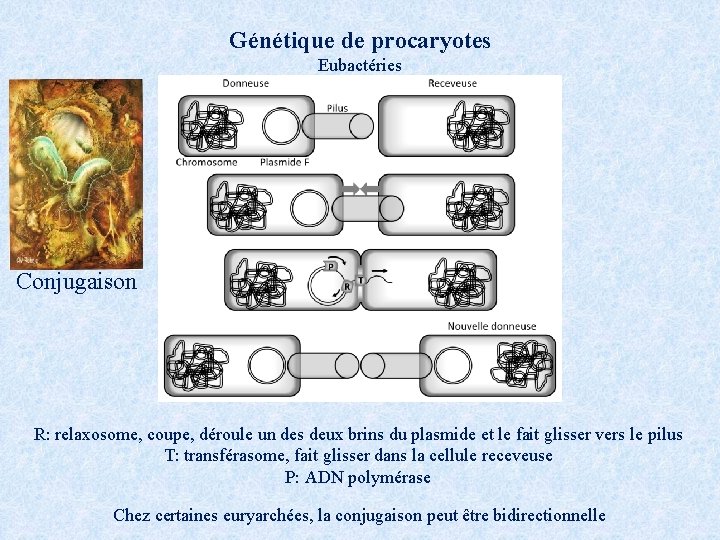
Génétique de procaryotes Eubactéries Conjugaison R: relaxosome, coupe, déroule un des deux brins du plasmide et le fait glisser vers le pilus T: transférasome, fait glisser dans la cellule receveuse P: ADN polymérase Chez certaines euryarchées, la conjugaison peut être bidirectionnelle

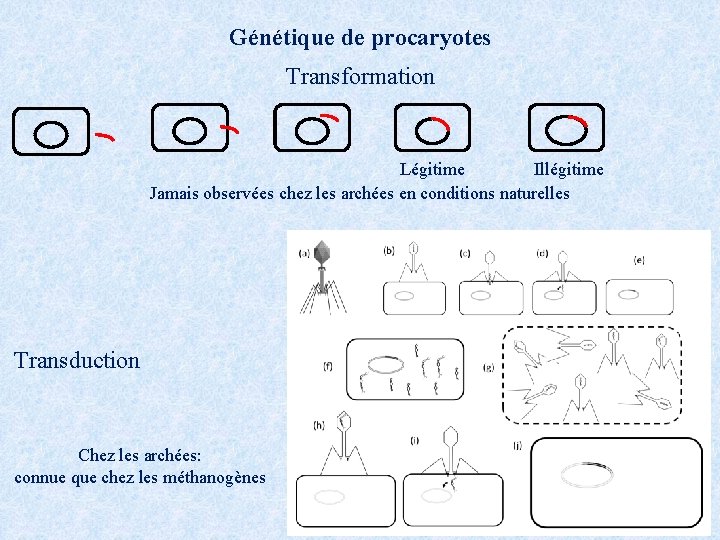
Génétique de procaryotes Transformation Légitime Illégitime Jamais observées chez les archées en conditions naturelles Transduction Chez les archées: connue que chez les méthanogènes

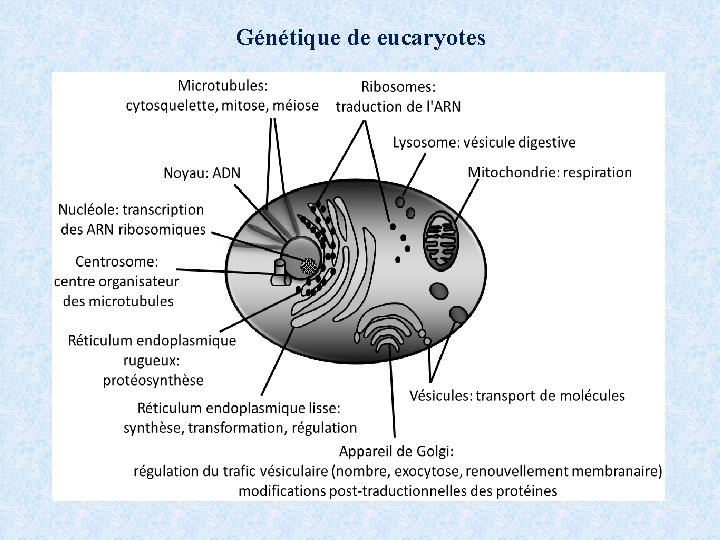
Génétique de eucaryotes

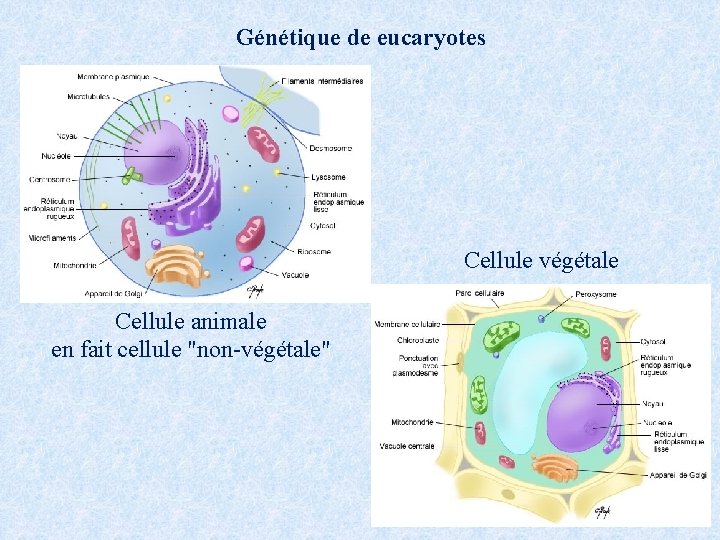
Génétique de eucaryotes Cellule végétale Cellule animale en fait cellule "non-végétale"

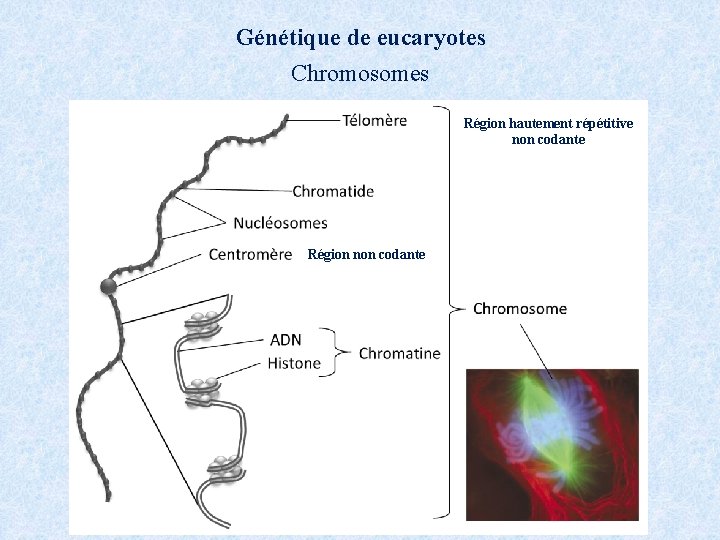
Génétique de eucaryotes Chromosomes Région hautement répétitive non codante Région non codante

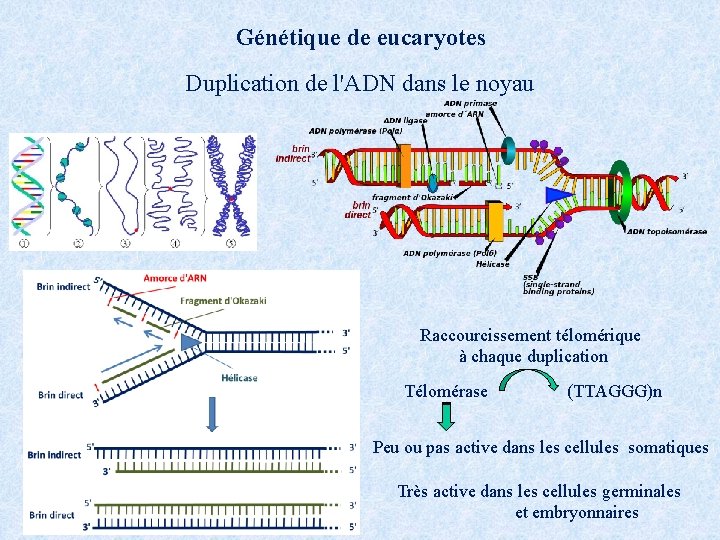
Génétique de eucaryotes Duplication de l'ADN dans le noyau Raccourcissement télomérique à chaque duplication Télomérase (TTAGGG)n Peu ou pas active dans les cellules somatiques Très active dans les cellules germinales et embryonnaires

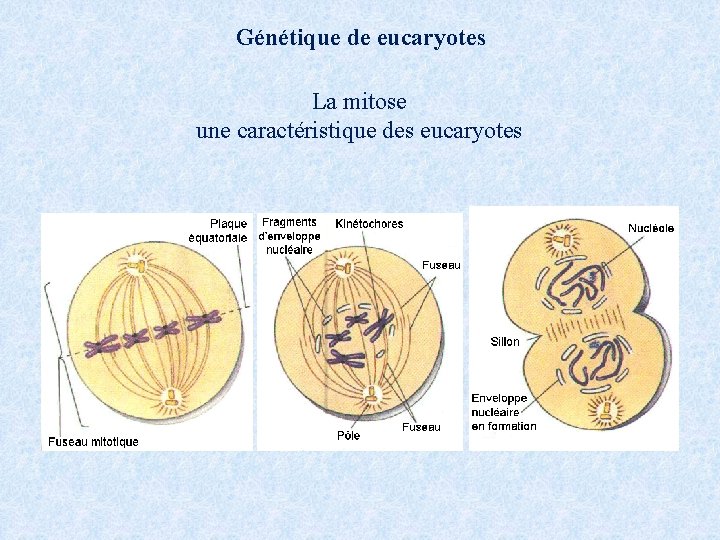
Génétique de eucaryotes La mitose une caractéristique des eucaryotes

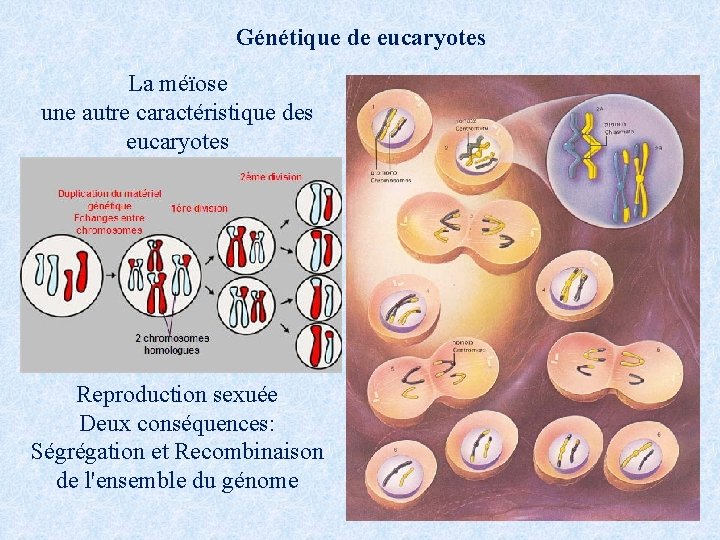
Génétique de eucaryotes La méïose une autre caractéristique des eucaryotes Reproduction sexuée Deux conséquences: Ségrégation et Recombinaison de l'ensemble du génome

3. La mutation, clé de la variation génétique et de l'évolution Mutations ponctuelles Transition: Mutation ponctuelle consistant au remplacement d'une purine par une autre purine (A<=>G) ou d'une pyrimidine par une autre pyrimidine (C<=>T) (antonymique de transversion). Transversion: Mutation ponctuelle consistant au remplacement d'une purine par une pyrimidine ou d'une pyrimidine par une purine (A<=>T, A<=>C, G<=>T) (antonymique de transition), deux fois moins fréquentes que les transitions. Insertions et délétions Éléments transposables (transposons) et rétrovirus Mutations chromosomiques IAM: Infinite Allele Model (si identiques, alors ils le sont par descendance) KAM: K allele model (homoplasie: pas nécessairement identique par descendance) SMM: Stepwise Mutation Model (microsatellites)(-CACA-) TPM: Two Phase Model (SMM+KAM)

Microsatellites et minisatellites Microsatellites: (simple sequence repeats Les minisatellites (aussi VNTR) sont des séquences de 9 à 100 paires de bases (SSR), short tandem repeats (STR) ou répétées en tandem variable number of tandem repeats (VNTR)): répétitions de motifs d'une (poly Taux de mutation u~10 -1 -10 -3 A) à 8 bases comme …CACA… (di -nucléotide), …GAGGAG… Grand nombre d'allèles possibles, K=5 -20 (tri-nucléotide) ou …CATACATA… (tétranucléotide). Taux de mutation u~10 -3 -10 -4 Grand nombre d'allèles possibles, K=10 -50 Primer 1 CTCT AGAG Primer 2 CTCTCT AGAGAG Primer 2 Primer 1 Profile hétérozygote

Single Nucleotide Polymorphic loci (SNPs) ---ATACTTAGTG-----TATGAATCAC-----ATACCTAGTG-----TATGGATCAC--Transitions (A<=>G ou C<=>T) deux fois plus fréquentes que Transversions (A<=>T, A<=>C, G<=>T) Taux de mutation u~10 -9 Nombre possible d'allèles, K=4 en théorie, 2 en pratique Révélation par séquençage

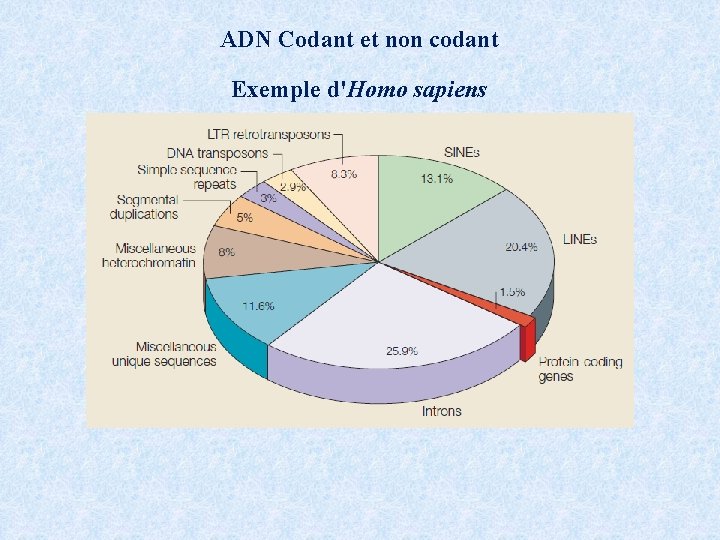
ADN Codant et non codant Exemple d'Homo sapiens

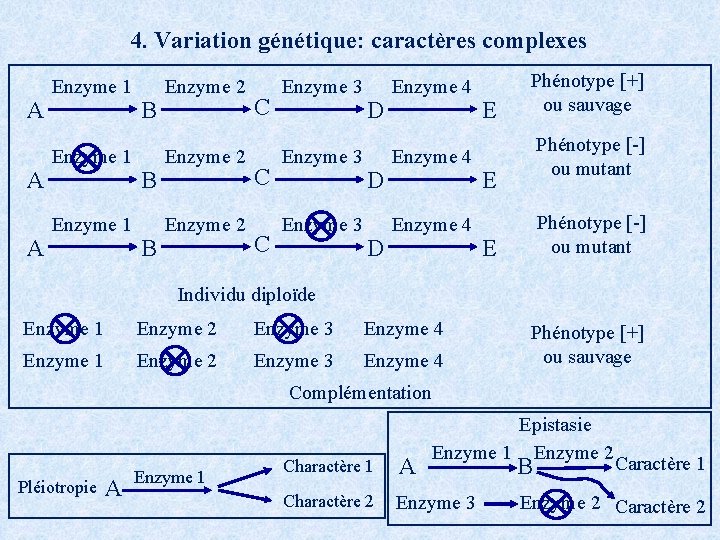
4. Variation génétique: caractères complexes A A A Enzyme 1 B B B Enzyme 2 C C C Enzyme 3 D D D Enzyme 4 E Phénotype [+] ou sauvage Phénotype [-] ou mutant E Individu diploïde Enzyme 1 Enzyme 2 Enzyme 3 Enzyme 4 Phénotype [+] ou sauvage Complémentation Pléiotropie A Enzyme 1 Epistasie Enzyme 1 Enzyme 2 Charactère 1 A Charactère 2 Enzyme 3 B Caractère 1 Enzyme 2 Caractère 2
![Génotype et phénotype Bb CcGénotype BPhénotype Le phénotype ne reflète pas nécessairement le génotype Génotype et phénotype Bb. Cc=Génotype [B]=Phénotype Le phénotype ne reflète pas nécessairement le génotype](https://slidetodoc.com/presentation_image/b87bb4c1ab851771c870557e375a1f55/image-32.jpg)
Génotype et phénotype Bb. Cc=Génotype [B]=Phénotype Le phénotype ne reflète pas nécessairement le génotype Microsatellites Primer 1 CTCT AGAG Primer 2 CTCTCT AGAGAG Primer 2 + -

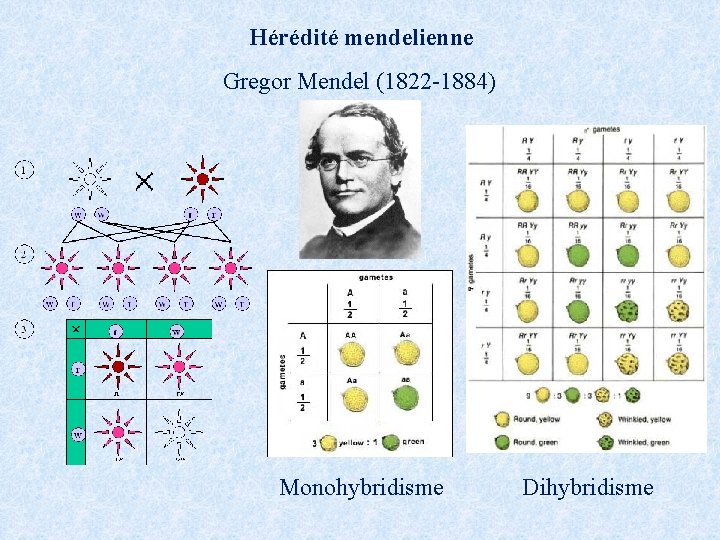
Hérédité mendelienne Gregor Mendel (1822 -1884) Monohybridisme Dihybridisme

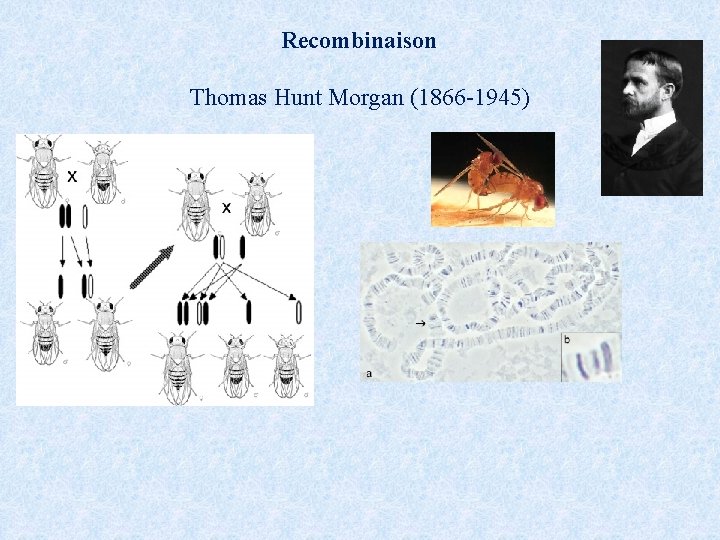
Recombinaison Thomas Hunt Morgan (1866 -1945)

Recombinaison Thomas Hunt Morgan (1866 -1945) Hybridation AABB aabb F 1: 100% Aa. Bb A B a b r Back cross: AABB Aa. Bb (1 -r) /2 AB, r/2 Ab, r/2 a. B, (1 -r)/2 ab AB (1 -r) /2 AABB, r/2 AABb, r/2 Aa. BB, (1 -r)/2 Aa. Bb F 2: Aa. Bb r 100=[f(AABb)+f(Aa. BB)] 100=Distance génétique en centimorgans

II. Bases théoriques de génétique des populations

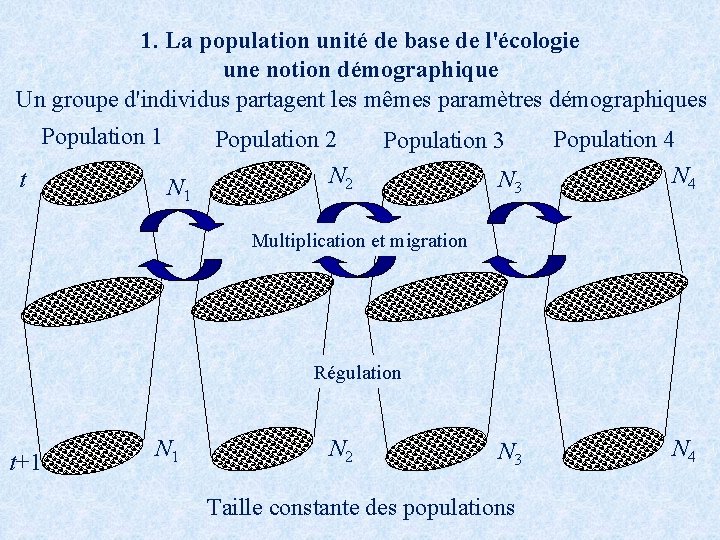
1. La population unité de base de l'écologie une notion démographique Un groupe d'individus partagent les mêmes paramètres démographiques Population 1 t Population 2 N 1 Population 3 N 2 Population 4 N 3 N 4 Multiplication et migration Régulation t+1 N 2 Taille constante des populations

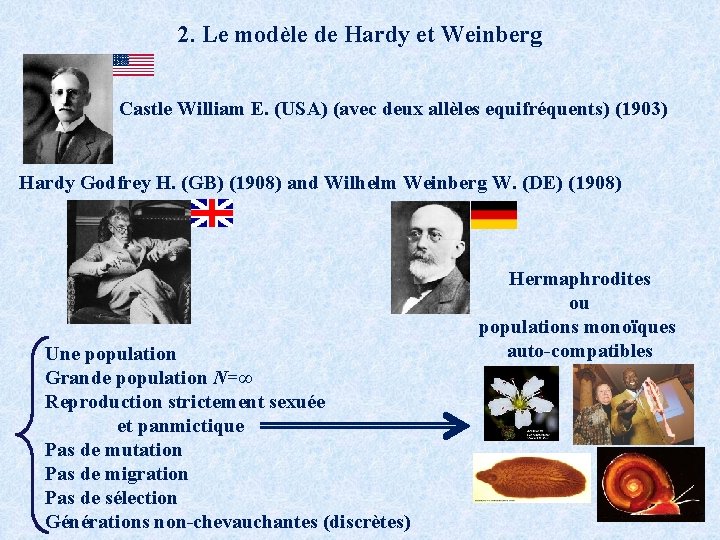
2. Le modèle de Hardy et Weinberg Castle William E. (USA) (avec deux allèles equifréquents) (1903) Hardy Godfrey H. (GB) (1908) and Wilhelm Weinberg W. (DE) (1908) Une population Grande population N=∞ Reproduction strictement sexuée et panmictique Pas de mutation Pas de migration Pas de sélection Générations non-chevauchantes (discrètes) Hermaphrodites ou populations monoïques auto-compatibles

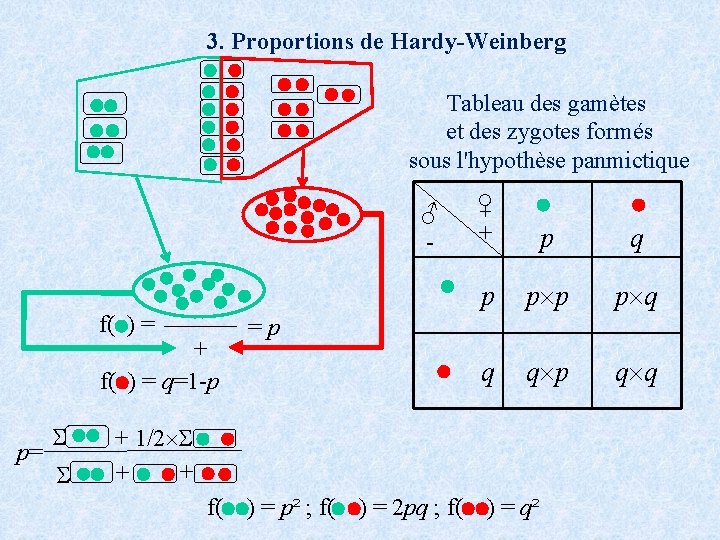
3. Proportions de Hardy-Weinberg Tableau des gamètes et des zygotes formés sous l'hypothèse panmictique ♂ f( ) = + f( ) = q=1 -p p= ♀ + p q p p q q q p q q = p + 1/2 + + f( ) = p² ; f( ) = 2 pq ; f( ) = q²

4. Equilibre de Hardy-Weinberg AA Dt Aa Ht aa Rt ft(A)=pt, ft(a)=qt=1 -pt Panmixie (hermaphrodites) Taille de population N~∞ Migration m=0 Mutation u=0 Pas de sélection Générations discrètes En une génération

Equilibre de Hardy-Weinberg avec trois allèles AA At AB Bt AC Ct BB Dt BC Et ft(A)=pt, ft(B)=qt , ft(C)=rt=1 -pt-qt En une génération CC Jt

Hardy-Weinberg, hétérozygotie observée, hétérozygotie attendue, diversités génétiques et probabilités d'identité A 1, A 2 , . . . Ai, . . . AK p 1, p 2 , . . . pi, . . . p. K

7. Pour plus d’un locus: les déséquilibres de liaison Locus B Locus A Deux loci A et B BB Bb bb AA Aa aa D 2 H 2 R 2 D 1 H 1 R 1 A→p. A B→p. B Gamètes ou haplotypes A_B: p. A_B=p. Ap. B+Dt A_b: p. A_b=p. A(1 -p. B)-Dt a_B: pa_B=(1 -p. A)p. B-Dt a_b: pa_b=(1 -p. A)(1 -p. B)+Dt p. A=p. A_B+p. A_b p. B=p. A_B+ pa_B Dt=p. A_B pa_b-p. A_bpa_B

Deux loci A et B Locus A Locus B AA Aa aa 11 12 22 D 1 H 1 R 1 D 2 H 2 R 2 A→p. A B→p. B Dt=p. A_B pa_b-p. A_bpa_B Gamètes p. A_A=p. Ap. B+Dt p. A_b=p. A(1 -p. B)-Dt pa_B=(1 -p. A)p. B-Dt pa_b=(1 -p. A)(1 -p. B)+Dt Si le taux de recombinaison est r et si la population suit Hardy-Weinberg N grand

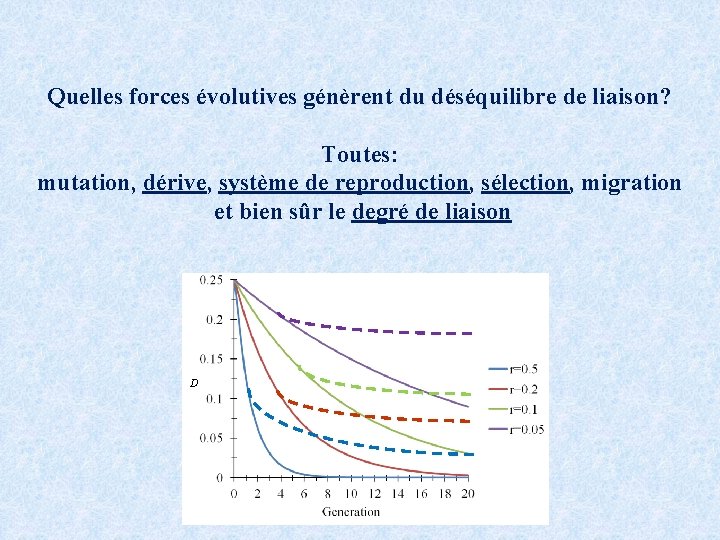
Quelles forces évolutives génèrent du déséquilibre de liaison? Toutes: mutation, dérive, système de reproduction, sélection, migration et bien sûr le degré de liaison D

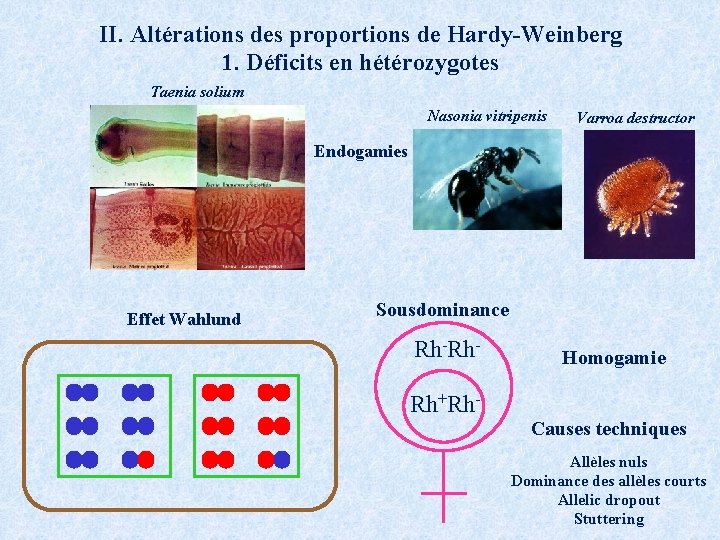
II. Altérations des proportions de Hardy-Weinberg 1. Déficits en hétérozygotes Taenia solium Nasonia vitripenis Varroa destructor Endogamies Effet Wahlund Sousdominance Rh-Rh- Homogamie Rh+Rh. Causes techniques Allèles nuls Dominance des allèles courts Allelic dropout Stuttering

III. Applications à un cas concret: La tique Ixodes ricinus en Europe du Nord

Génétique des populations d'Ixodes ricinus et borréliose de Lyme en Suisse B. valaisiana B. garinii B. afzelii B. burgdorferi B. Spielmanii

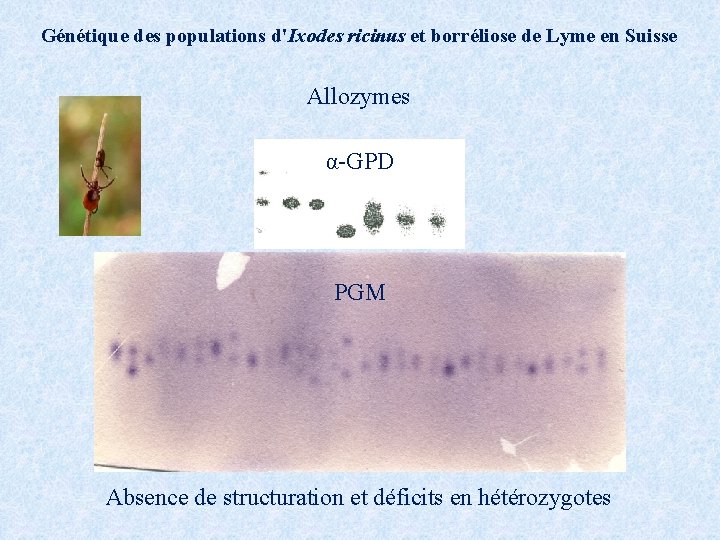
Génétique des populations d'Ixodes ricinus et borréliose de Lyme en Suisse Allozymes α-GPD PGM Absence de structuration et déficits en hétérozygotes

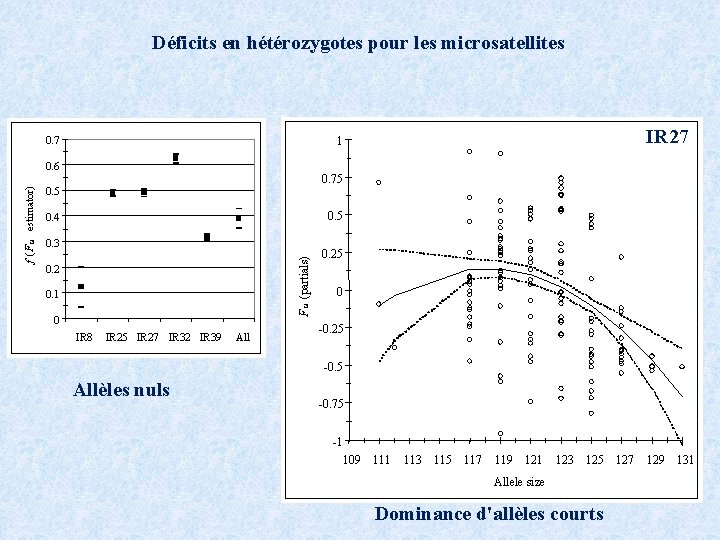
Déficits en hétérozygotes pour les microsatellites 0. 7 IR 27 1 0. 75 0. 4 0. 3 F is (partials) f (F is estimator) 0. 6 0. 2 0. 1 0 IR 8 IR 25 IR 27 IR 32 IR 39 All 0. 25 0 -0. 25 -0. 5 Allèles nuls -0. 75 -1 109 111 113 115 117 119 121 123 125 Allele size Dominance d'allèles courts 127 129 131

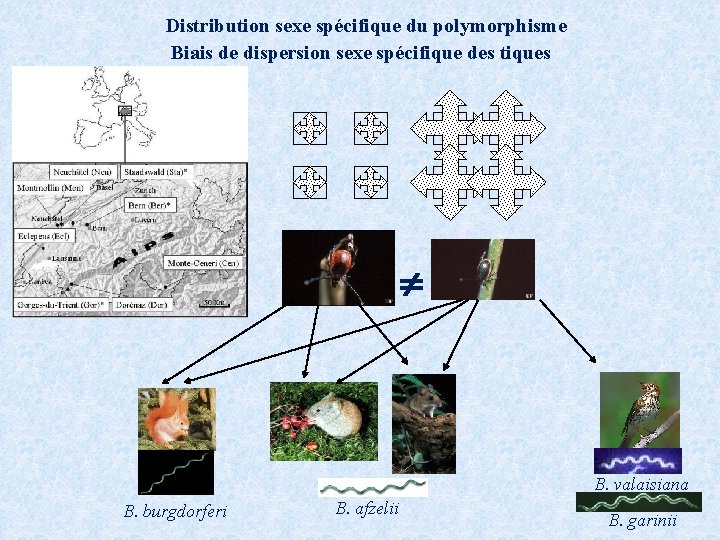
Distribution sexe spécifique du polymorphisme Biais de dispersion sexe spécifique des tiques B. valaisiana B. burgdorferi B. afzelii B. garinii

Détection des Borrelia dans les tiques Pour Borrelia burgdorferi Prévalence of B. burgdorferi ss P =0. 012 0. 08 0. 07 0. 06 0. 05 0. 04 0. 03 0. 02 F M Sex of the tick

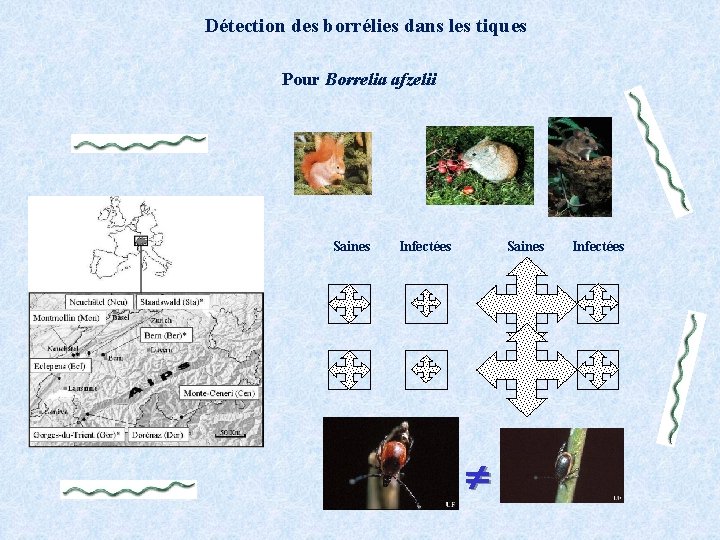
Détection des borrélies dans les tiques Pour Borrelia afzelii Saines Infectées

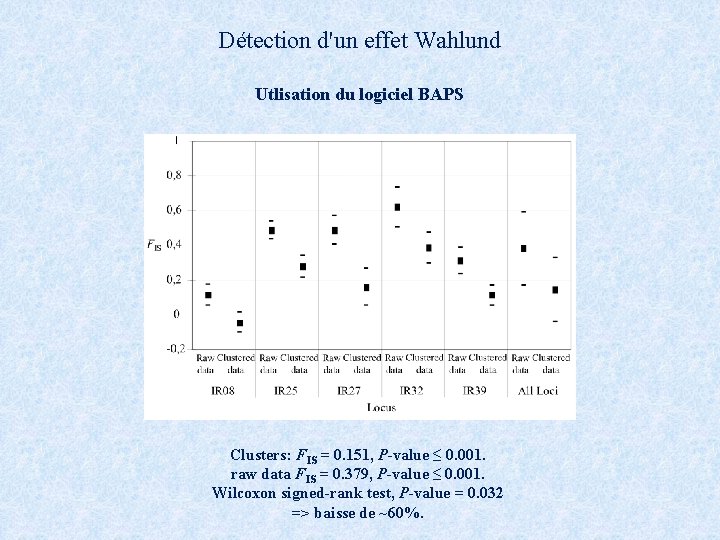
Détection d'un effet Wahlund Utlisation du logiciel BAPS Clusters: FIS = 0. 151, P-value ≤ 0. 001. raw data FIS = 0. 379, P-value ≤ 0. 001. Wilcoxon signed-rank test, P-value = 0. 032 => baisse de ~60%.

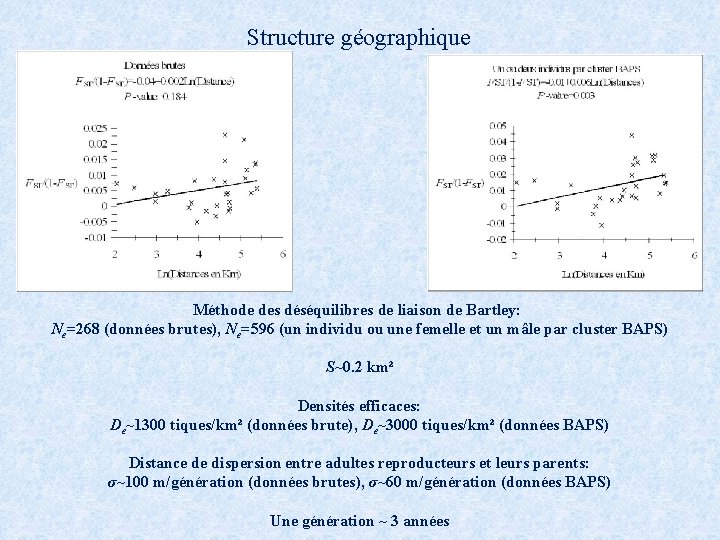
Structure géographique Méthode des déséquilibres de liaison de Bartley: Ne=268 (données brutes), Ne=596 (un individu ou une femelle et un mâle par cluster BAPS) S~0. 2 km² Densités efficaces: De~1300 tiques/km² (données brute), De~3000 tiques/km² (données BAPS) Distance de dispersion entre adultes reproducteurs et leurs parents: σ~100 m/génération (données brutes), σ~60 m/génération (données BAPS) Une génération ~ 3 années

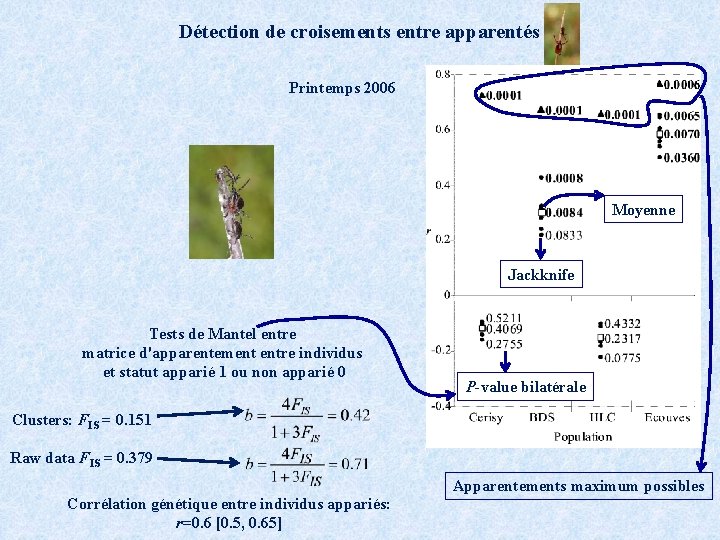
Détection de croisements entre apparentés Printemps 2006 Moyenne Jackknife Tests de Mantel entre matrice d'apparentement entre individus et statut apparié 1 ou non apparié 0 P-value bilatérale Clusters: FIS = 0. 151 Raw data FIS = 0. 379 Apparentements maximum possibles Corrélation génétique entre individus appariés: r=0. 6 [0. 5, 0. 65]

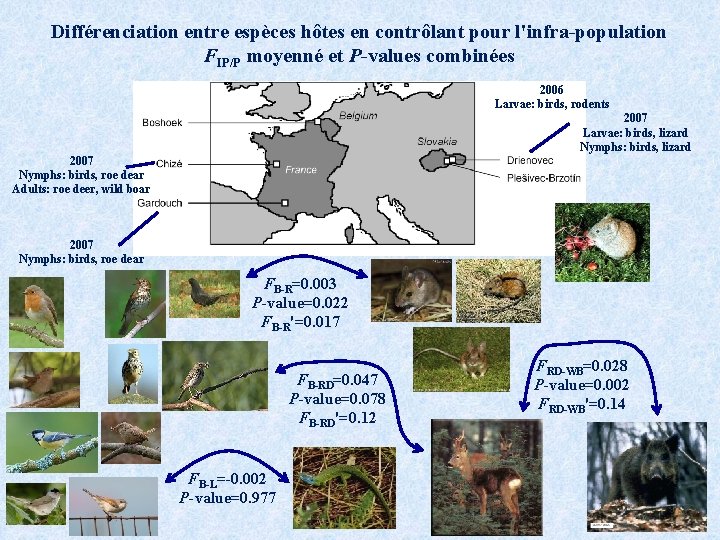
Différenciation entre espèces hôtes en contrôlant pour l'infra-population FIP/P moyenné et P-values combinées 2006 Larvae: birds, rodents 2007 Larvae: birds, lizard Nymphs: birds, lizard 2007 Nymphs: birds, roe dear Adults: roe deer, wild boar 2007 Nymphs: birds, roe dear FB-R=0. 003 P-value=0. 022 FB-R'=0. 017 FB-RD=0. 047 P-value=0. 078 FB-RD'=0. 12 FB-L=-0. 002 P-value=0. 977 FRD-WB=0. 028 P-value=0. 002 FRD-WB'=0. 14

IV. Conclusions et recommandations

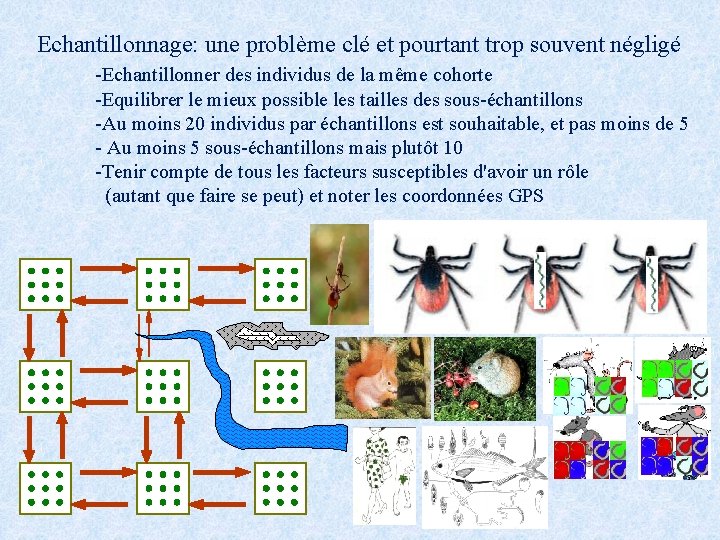
Echantillonnage: une problème clé et pourtant trop souvent négligé -Echantillonner des individus de la même cohorte -Equilibrer le mieux possible les tailles des sous-échantillons -Au moins 20 individus par échantillons est souhaitable, et pas moins de 5 - Au moins 5 sous-échantillons mais plutôt 10 -Tenir compte de tous les facteurs susceptibles d'avoir un rôle (autant que faire se peut) et noter les coordonnées GPS

Le choix des marqueurs est capital - Diploïde (si possible) - Codominants - Autosomiques - Pas moins de 5, 10 -20 c'est mieux - Microsatellites dinucléotidiques (non codants) représentent en principe le meilleur rapport qualité/prix - Vérifier la constance d'un signal d'un locus à l'autre - Ne jamais hésiter à laisser de côté quelques loci outliers

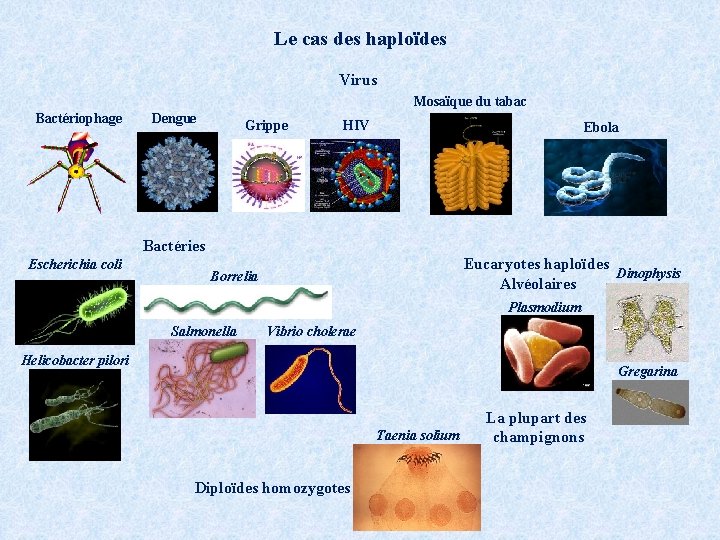
Le cas des haploïdes Virus Mosaïque du tabac Bactériophage Dengue Grippe HIV Ebola Bactéries Escherichia coli Eucaryotes haploïdes Dinophysis Alvéolaires Borrelia Plasmodium Salmonella Vibrio cholerae Helicobacter pilori Gregarina Taenia solium Diploïdes homozygotes La plupart des champignons

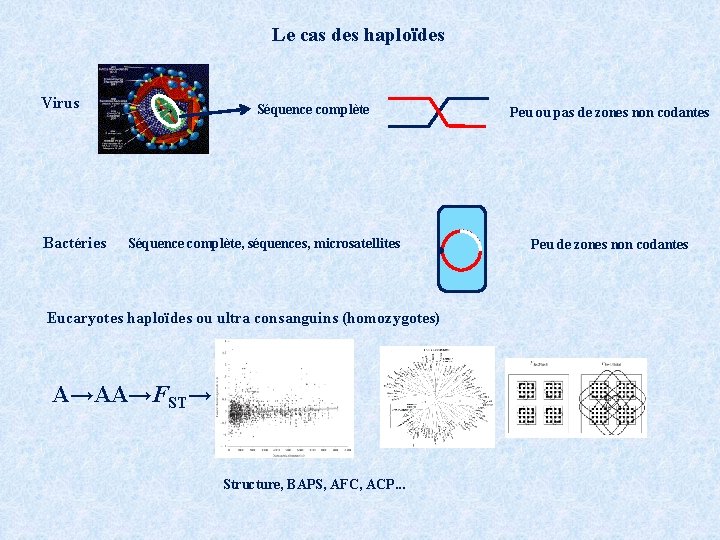
Le cas des haploïdes Virus Bactéries Séquence complète, séquences, microsatellites Eucaryotes haploïdes ou ultra consanguins (homozygotes) A→AA→FST→ Structure, BAPS, AFC, ACP. . . Peu ou pas de zones non codantes Peu de zones non codantes

 Logiciel baps
Logiciel baps Must mees kalasaba taga
Must mees kalasaba taga Vitruviuse mees
Vitruviuse mees Irene pukk
Irene pukk Des des des
Des des des Dynamique des populations
Dynamique des populations Thierry stora
Thierry stora Ensti
Ensti Thierry moreau ucl
Thierry moreau ucl Thierry dostes
Thierry dostes Thierry vansweevelt
Thierry vansweevelt Chateau thierry
Chateau thierry Thierry boidart
Thierry boidart Thierry manciot
Thierry manciot Thierry vanderlinden
Thierry vanderlinden Pacemaker endoveineux
Pacemaker endoveineux Hypoaldostéronisme hyporéninisme
Hypoaldostéronisme hyporéninisme Thierry lenain
Thierry lenain Thierry metais
Thierry metais Thierry davy
Thierry davy Thierry odent
Thierry odent Thierry marchant
Thierry marchant Thierry denolle
Thierry denolle Thierry woller
Thierry woller Thierry duliere
Thierry duliere Alain thierry montpellier
Alain thierry montpellier Multigameserver
Multigameserver Thierry stora
Thierry stora Thierry guedj
Thierry guedj Thierry levoir
Thierry levoir Thierry poirel ceb
Thierry poirel ceb Thierry eeckhout veterinaire
Thierry eeckhout veterinaire Conclusion de milieu de culture
Conclusion de milieu de culture Thierry leblanc
Thierry leblanc Thierry timmermans
Thierry timmermans Thierry michel isoard
Thierry michel isoard Affiche de propagande plan marshall
Affiche de propagande plan marshall Cartographie des flux de valeur
Cartographie des flux de valeur Des attack
Des attack La diffusion des idées des lumières
La diffusion des idées des lumières Diversification des espaces et des acteurs de la production
Diversification des espaces et des acteurs de la production Budget des ventes
Budget des ventes Je t'offrirai des fleurs et des nappes en couleurs
Je t'offrirai des fleurs et des nappes en couleurs Il existe des personnes qui sont des lumières pour tous
Il existe des personnes qui sont des lumières pour tous Volume d un corps
Volume d un corps Passé simple
Passé simple Mesure de niveaux pour liquides et solides
Mesure de niveaux pour liquides et solides Robin des bois des alpes
Robin des bois des alpes Interactions among living things
Interactions among living things Examples of epistasis in humans
Examples of epistasis in humans Modular architecture vs integrated architecture
Modular architecture vs integrated architecture Integumentary interactions with other systems
Integumentary interactions with other systems Grapefruit-drug interactions chart
Grapefruit-drug interactions chart Molecular biology
Molecular biology Congress formal and informal powers
Congress formal and informal powers Interactions
Interactions Interactions between ais and internal and external parties
Interactions between ais and internal and external parties Interactions between atoms occur
Interactions between atoms occur Symbiosis and species interactions keystone webquest
Symbiosis and species interactions keystone webquest Ppi drug interactions
Ppi drug interactions Do all em waves travel at the speed of light
Do all em waves travel at the speed of light Wave
Wave Sertraline interactions
Sertraline interactions Nutrient interactions
Nutrient interactions