TRANSFERENCIA DE MATERIAL GENTICO II AISLAMIENTO DE PLSMIDOS














- Slides: 40

TRANSFERENCIA DE MATERIAL GENÉTICO II: AISLAMIENTO DE PLÁSMIDOS LISIS ALCALINA ENSAYOS DE RESTRICCIÓN

ESQUEMA GENERAL SESIÓN I: TRANSFORMAR cepas de E. coli con un plásmido para que adquieran resistencia a un antibiótico SESIÓN 2: AISLAR el plásmido mediante la técnica de Lisis alcalina SESIÓN 3: FUNCIÓN DE LAS ENZIMAS DE RESTRICCIÓN Cortar el plásmido aislado con enzimas de restricción y visualizarlo en un gel de agarosa teñido con bromuro de etidio

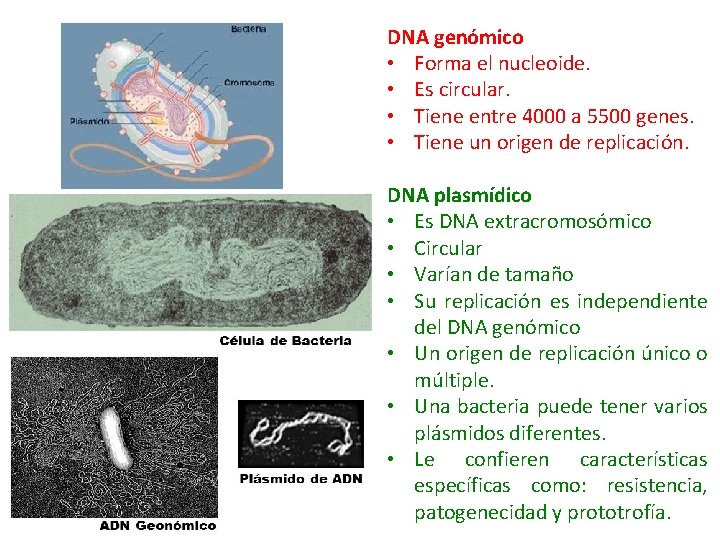
DNA genómico • Forma el nucleoide. • Es circular. • Tiene entre 4000 a 5500 genes. • Tiene un origen de replicación. DNA plasmídico • Es DNA extracromosómico • Circular • Varían de tamaño • Su replicación es independiente del DNA genómico • Un origen de replicación único o múltiple. • Una bacteria puede tener varios plásmidos diferentes. • Le confieren características específicas como: resistencia, patogenecidad y prototrofía.

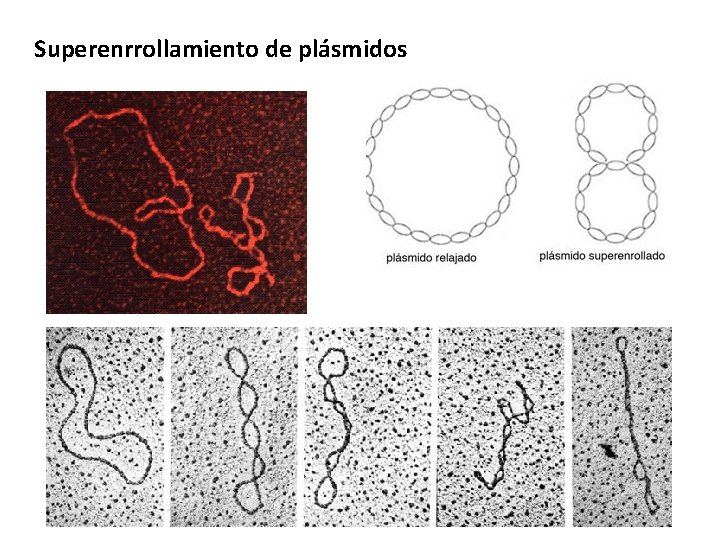
Superenrrollamiento de plásmidos

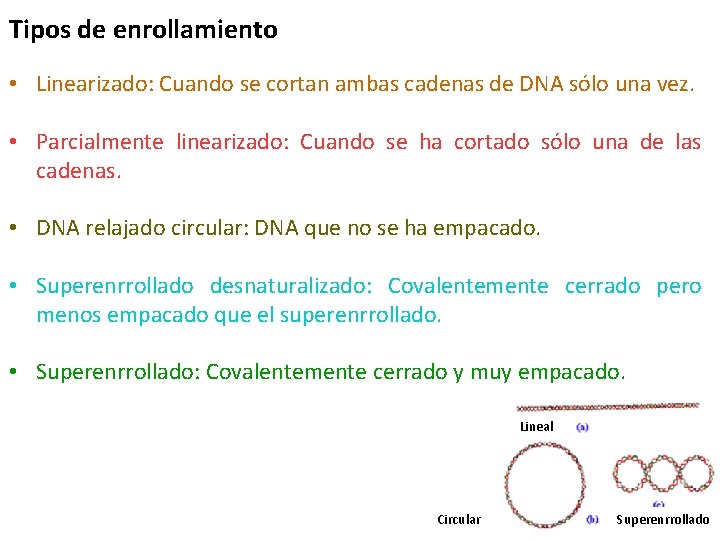
Tipos de enrollamiento • Linearizado: Cuando se cortan ambas cadenas de DNA sólo una vez. • Parcialmente linearizado: Cuando se ha cortado sólo una de las cadenas. • DNA relajado circular: DNA que no se ha empacado. • Superenrrollado desnaturalizado: Covalentemente cerrado pero menos empacado que el superenrrollado. • Superenrrollado: Covalentemente cerrado y muy empacado. Lineal Circular Superenrrollado

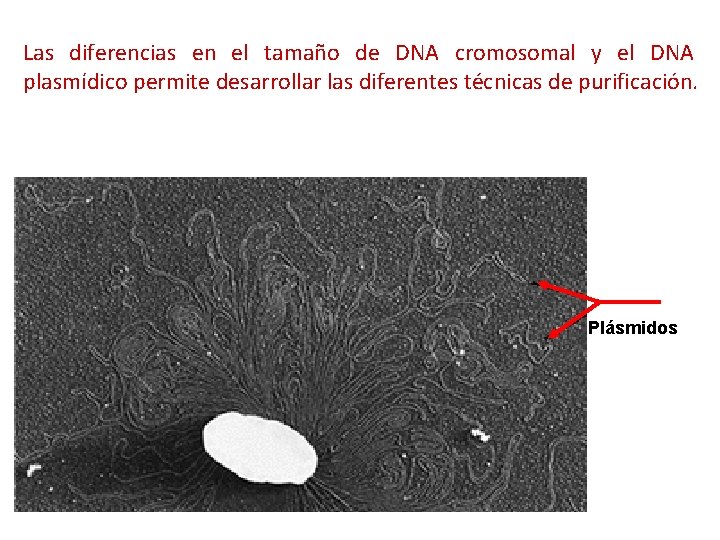
Las diferencias en el tamaño de DNA cromosomal y el DNA plasmídico permite desarrollar las diferentes técnicas de purificación. Plásmidos

Existen distintos procesos para la purificación de DNA plasmídico, pero todos incluyen tres pasos: 1. Crecimiento de bacterias en un medio selectivo de aquellas que llevan el plásmido. 1. Lisis de las bacterias para la liberación del plásmido. 1. Purificación del DNA plasmídico

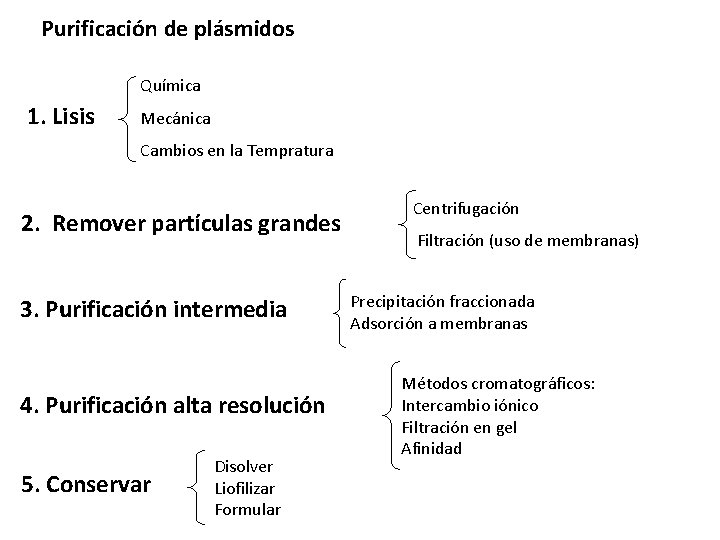
Purificación de plásmidos Química 1. Lisis Mecánica Cambios en la Tempratura 2. Remover partículas grandes 3. Purificación intermedia 4. Purificación alta resolución 5. Conservar Disolver Liofilizar Formular Centrifugación Filtración (uso de membranas) Precipitación fraccionada Adsorción a membranas Métodos cromatográficos: Intercambio iónico Filtración en gel Afinidad

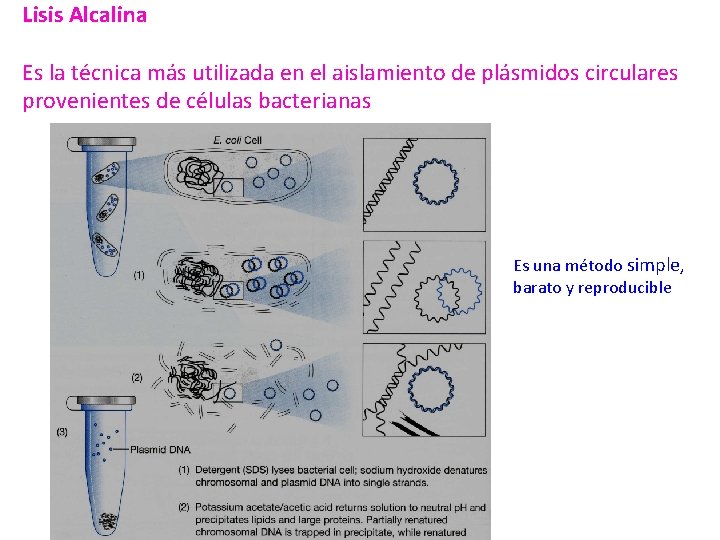
Lisis Alcalina Es la técnica más utilizada en el aislamiento de plásmidos circulares provenientes de células bacterianas Es una método simple, barato y reproducible

Lisis Alcalina Es una técnica que explota las diferencia en las propiedades de desnaturalización y renaturalización del DNA plasmídico (círculos cerrados covalentemente) y el DNA cromosómico (fragmentado). La alcalinización con Na. OH en presencia de un detergente aniónico fuerte (SDS), que provoca la lisis celular, desnaturalización del DNA cromosómico y de las proteínas y la liberación del DNA plasmídico. Los plásmidos se ven poco afectados por su estructura circular y superenrrollada. La presencia de alta sal (acetato de potasio), permite la neutralización del medio y la precipitación de las proteínas y del DNA cromosómico. El DNA cromosómico y los agregados de proteínas insolubles se separan de el DNA plasmídico por centrifugación. El DNA plasmídico queda en el sobrenadante, conservando mayoritariamente su estructura nativa.

Extracción de DNA plasmídico de bacterias de E. coli transformadas. Las bacterias se dejan crecer hasta una fase estacionaria

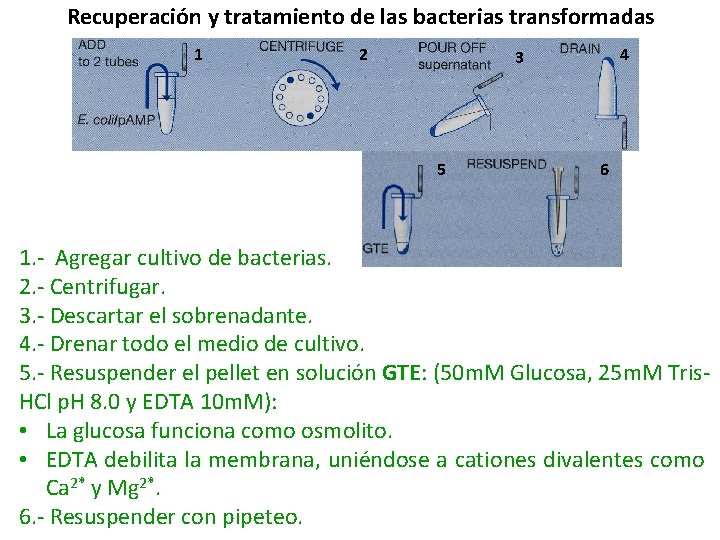
Recuperación y tratamiento de las bacterias transformadas 1 2 4 3 5 6 1. - Agregar cultivo de bacterias. 2. - Centrifugar. 3. - Descartar el sobrenadante. 4. - Drenar todo el medio de cultivo. 5. - Resuspender el pellet en solución GTE: (50 m. M Glucosa, 25 m. M Tris. HCl p. H 8. 0 y EDTA 10 m. M): • La glucosa funciona como osmolito. • EDTA debilita la membrana, uniéndose a cationes divalentes como Ca 2* y Mg 2*. 6. - Resuspender con pipeteo.

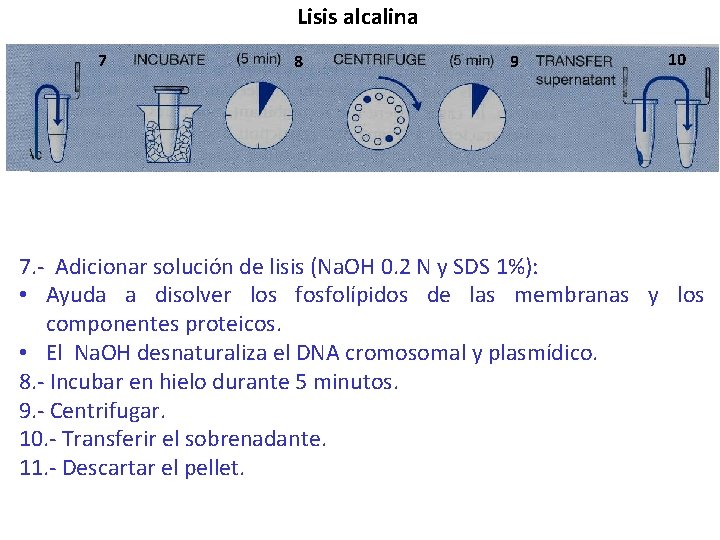
Lisis alcalina 7 8 9 10 7. - Adicionar solución de lisis (Na. OH 0. 2 N y SDS 1%): • Ayuda a disolver los fosfolípidos de las membranas y los componentes proteicos. • El Na. OH desnaturaliza el DNA cromosomal y plasmídico. 8. - Incubar en hielo durante 5 minutos. 9. - Centrifugar. 10. - Transferir el sobrenadante. 11. - Descartar el pellet.

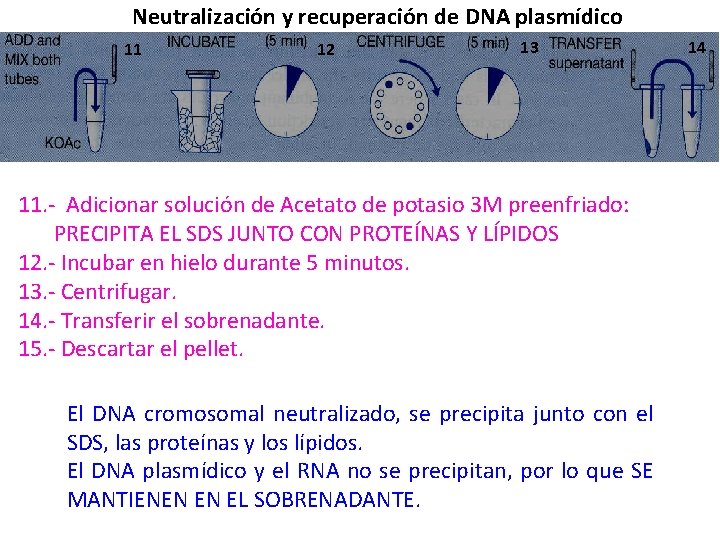
Neutralización y recuperación de DNA plasmídico 11 12 13 11. - Adicionar solución de Acetato de potasio 3 M preenfriado: PRECIPITA EL SDS JUNTO CON PROTEÍNAS Y LÍPIDOS 12. - Incubar en hielo durante 5 minutos. 13. - Centrifugar. 14. - Transferir el sobrenadante. 15. - Descartar el pellet. El DNA cromosomal neutralizado, se precipita junto con el SDS, las proteínas y los lípidos. El DNA plasmídico y el RNA no se precipitan, por lo que SE MANTIENEN EN EL SOBRENADANTE. 14

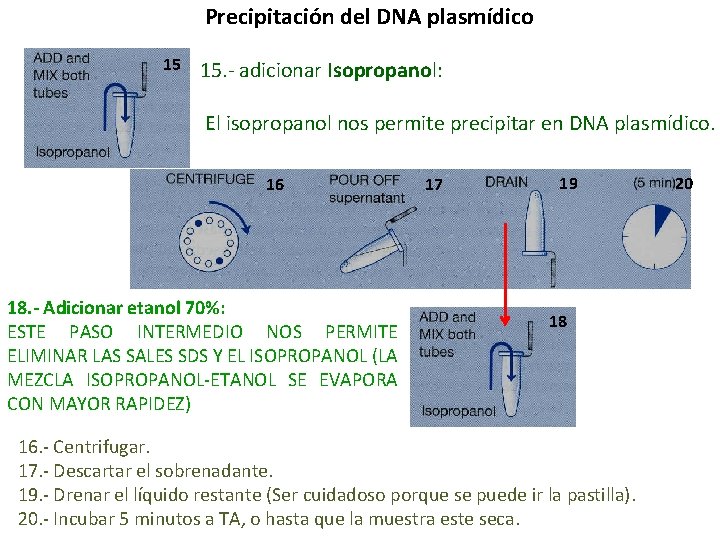
Precipitación del DNA plasmídico 15 15. - adicionar Isopropanol: El isopropanol nos permite precipitar en DNA plasmídico. 16 18. - Adicionar etanol 70%: ESTE PASO INTERMEDIO NOS PERMITE ELIMINAR LAS SALES SDS Y EL ISOPROPANOL (LA MEZCLA ISOPROPANOL-ETANOL SE EVAPORA CON MAYOR RAPIDEZ) 17 19 18 16. - Centrifugar. 17. - Descartar el sobrenadante. 19. - Drenar el líquido restante (Ser cuidadoso porque se puede ir la pastilla). 20. - Incubar 5 minutos a TA, o hasta que la muestra este seca. 20

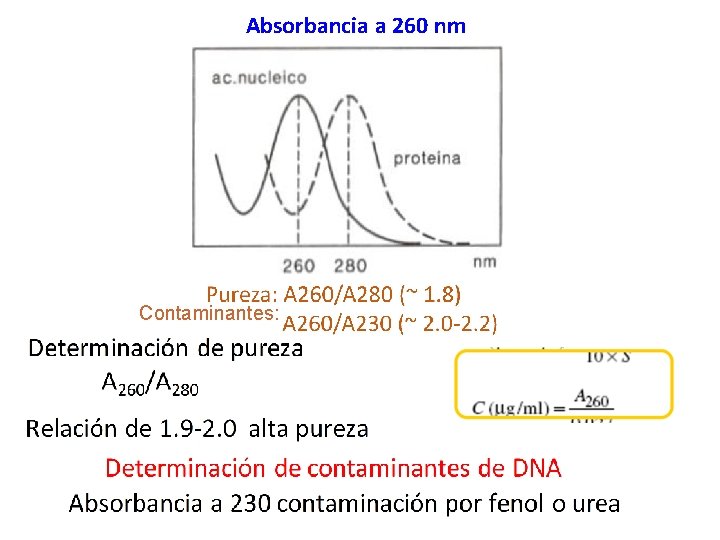
Verificación de la purificación Para monitorear el progreso de la purificación del plásmido, y verificar la correcta purificación, se llevan a cabo dos técnicas principalmente: 1. - Absorbancia a 260 nm 2. - Electroforesis en geles de Agarosa

Absorbancia a 260 nm Contaminantes:

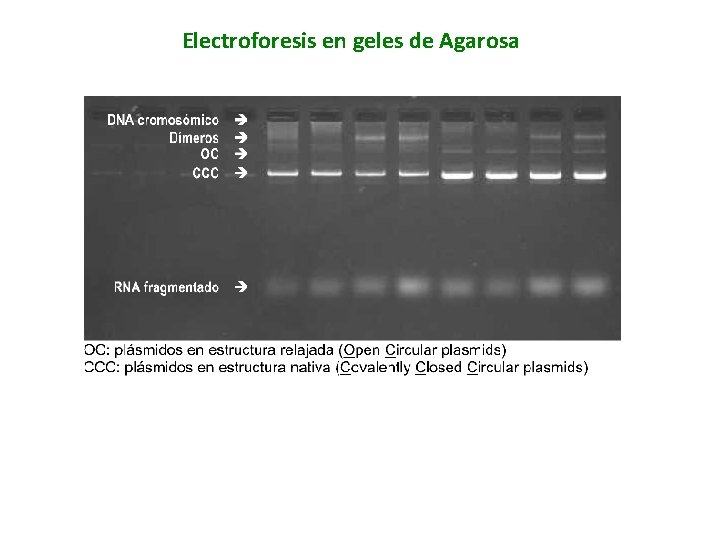
Electroforesis en geles de Agarosa

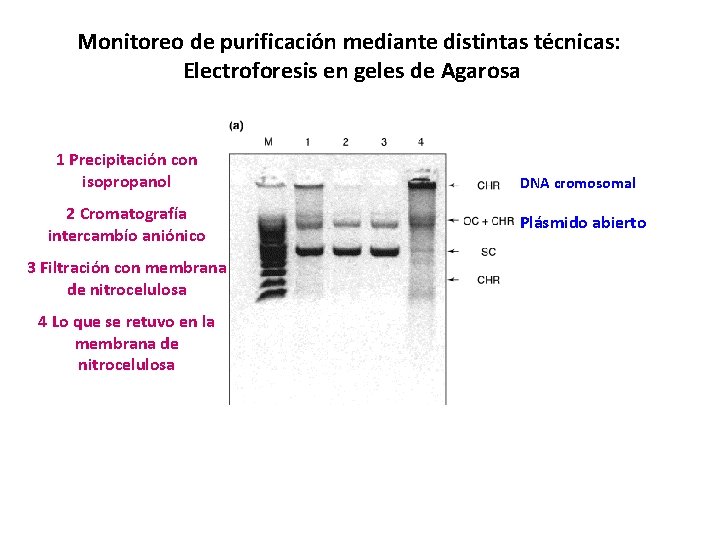
Monitoreo de purificación mediante distintas técnicas: Electroforesis en geles de Agarosa 1 Precipitación con isopropanol 2 Cromatografía intercambío aniónico 3 Filtración con membrana de nitrocelulosa 4 Lo que se retuvo en la membrana de nitrocelulosa DNA cromosomal Plásmido abierto

ESQUEMA GENERAL SESIÓN I: TRANSFORMAR cepas de E. coli con un plásmido para que adquieran resistencia a un antibiótico SESIÓN 2: AISLAR el plásmido mediante la técnica de Lisis alcalina SESIÓN 3: FUNCIóN DE LAS ENZIMAS DE RESTRICCIÓN Cortar el plásmido aislado con enzimas de restricción y visualizarlo en un gel de agarosa teñido con bromuro de etidio

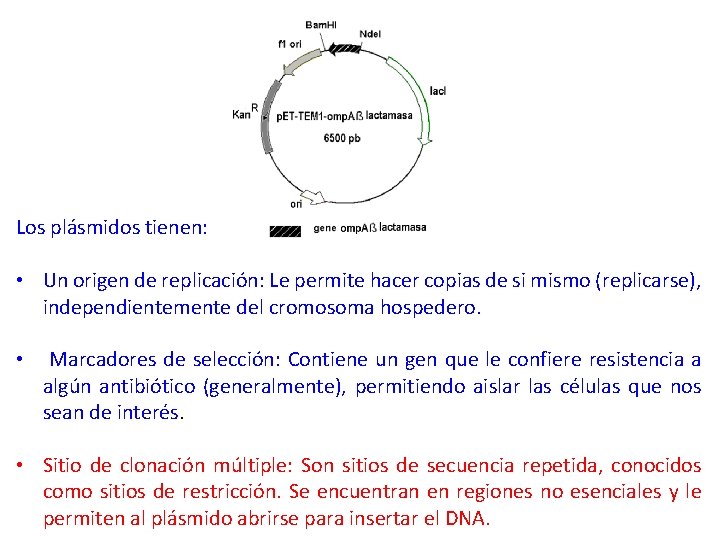
Los plásmidos tienen: • Un origen de replicación: Le permite hacer copias de si mismo (replicarse), independientemente del cromosoma hospedero. • Marcadores de selección: Contiene un gen que le confiere resistencia a algún antibiótico (generalmente), permitiendo aislar las células que nos sean de interés. • Sitio de clonación múltiple: Son sitios de secuencia repetida, conocidos como sitios de restricción. Se encuentran en regiones no esenciales y le permiten al plásmido abrirse para insertar el DNA.

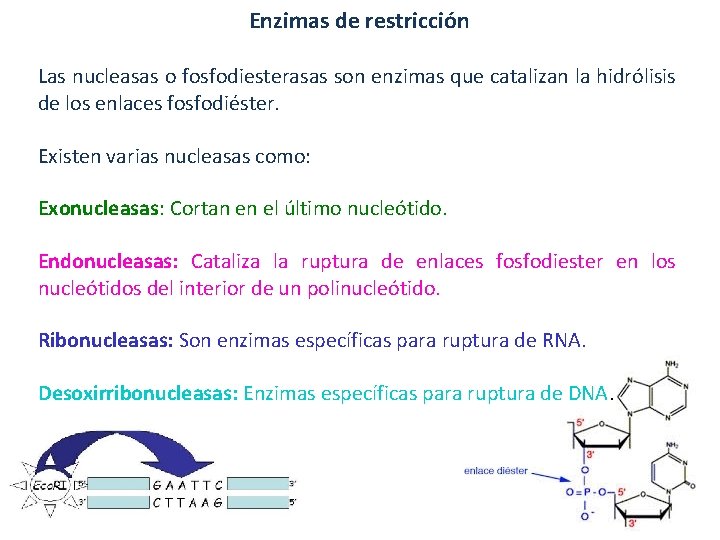
Enzimas de restricción Las nucleasas o fosfodiesterasas son enzimas que catalizan la hidrólisis de los enlaces fosfodiéster. Existen varias nucleasas como: Exonucleasas: Cortan en el último nucleótido. Endonucleasas: Cataliza la ruptura de enlaces fosfodiester en los nucleótidos del interior de un polinucleótido. Ribonucleasas: Son enzimas específicas para ruptura de RNA. Desoxirribonucleasas: Enzimas específicas para ruptura de DNA.

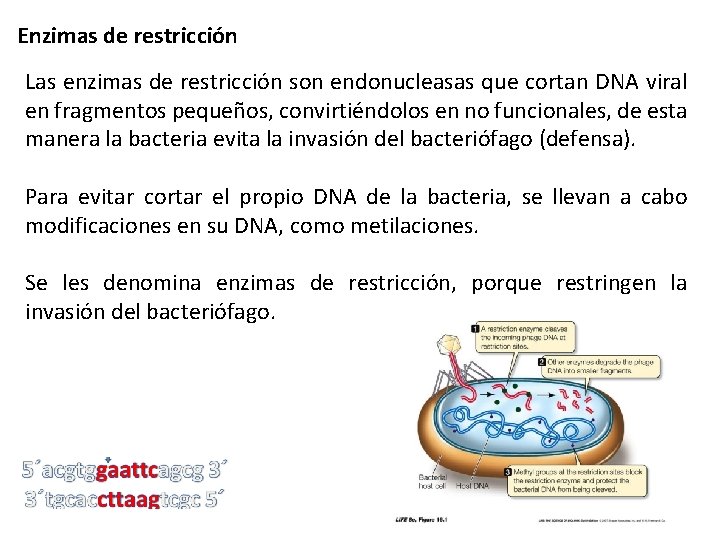
Enzimas de restricción Las enzimas de restricción son endonucleasas que cortan DNA viral en fragmentos pequeños, convirtiéndolos en no funcionales, de esta manera la bacteria evita la invasión del bacteriófago (defensa). Para evitar cortar el propio DNA de la bacteria, se llevan a cabo modificaciones en su DNA, como metilaciones. Se les denomina enzimas de restricción, porque restringen la invasión del bacteriófago.

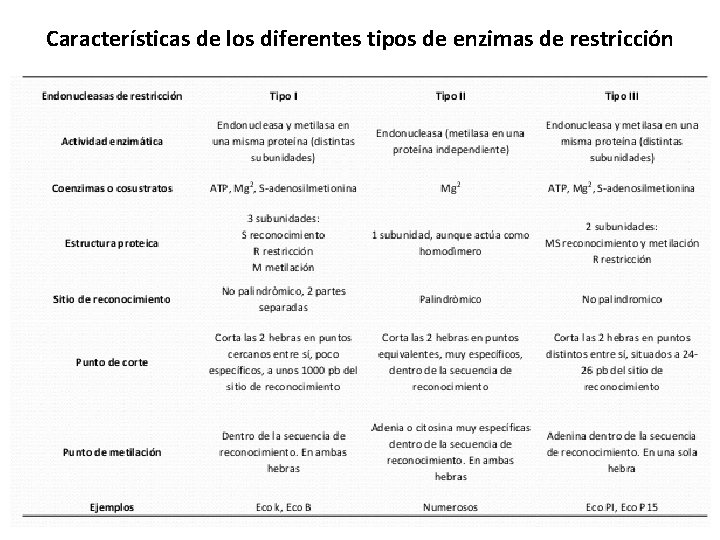
Existen varios tipos de enzimas de restricción. Los tipos I y III: Tienen capacidad de restricción y de metilación. Reconocen regiones grandes y asimétricas. Cortan en un sitio diferente al de reconocimiento: Tipo I: Cortan a una distancia grande de la secuencia de reconocimiento, en sitios al azar, ya sea río arriba o río abajo. La enzima que reconoce el sitio de restricción posee 3 subunidades. Tipo III: Cortan en 5 a 8 pares después del sitio de reconocimiento.

Características de los diferentes tipos de enzimas de restricción

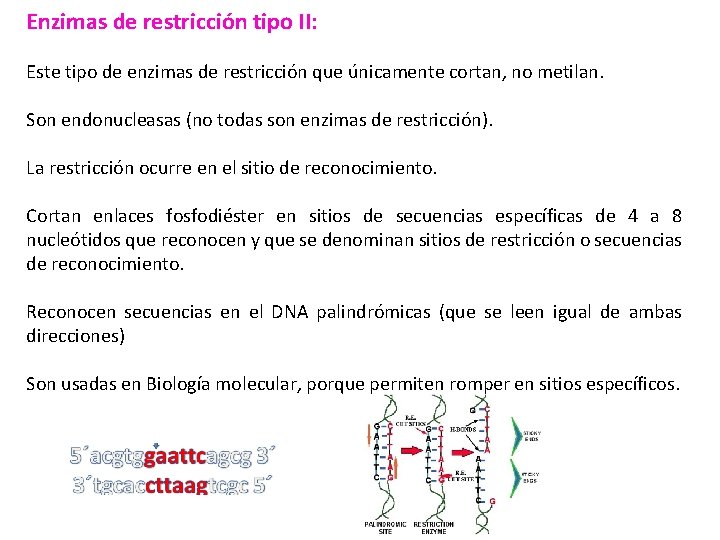
Enzimas de restricción tipo II: Este tipo de enzimas de restricción que únicamente cortan, no metilan. Son endonucleasas (no todas son enzimas de restricción). La restricción ocurre en el sitio de reconocimiento. Cortan enlaces fosfodiéster en sitios de secuencias específicas de 4 a 8 nucleótidos que reconocen y que se denominan sitios de restricción o secuencias de reconocimiento. Reconocen secuencias en el DNA palindrómicas (que se leen igual de ambas direcciones) Son usadas en Biología molecular, porque permiten romper en sitios específicos.

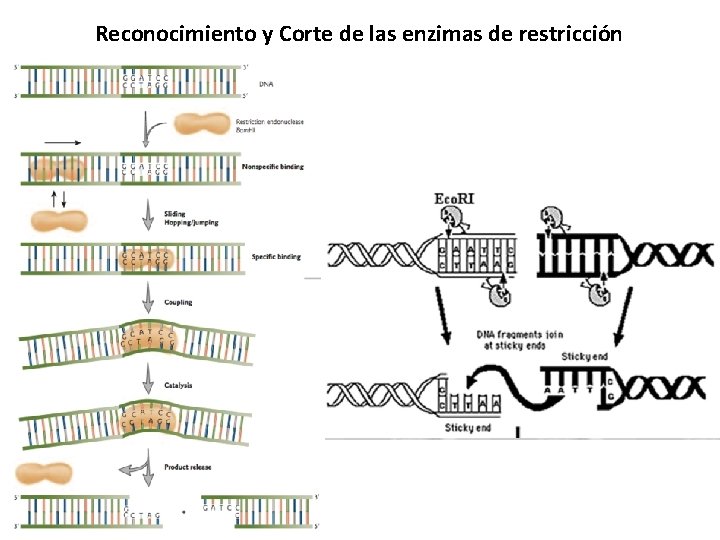
Reconocimiento y Corte de las enzimas de restricción

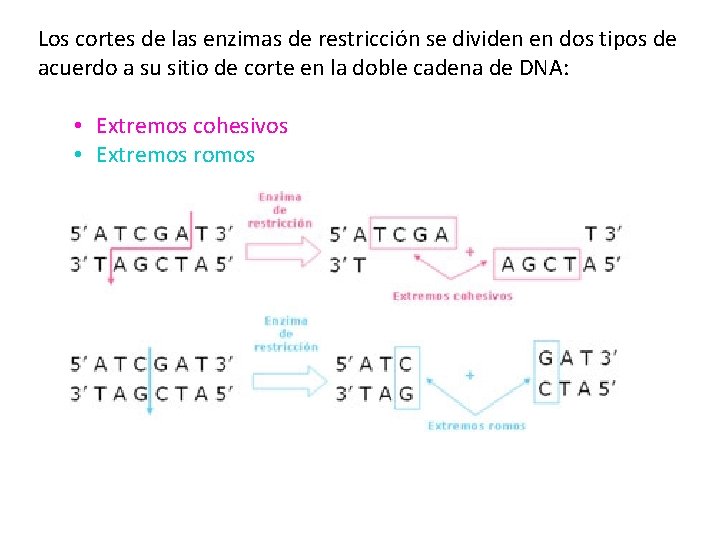
Los cortes de las enzimas de restricción se dividen en dos tipos de acuerdo a su sitio de corte en la doble cadena de DNA: • Extremos cohesivos • Extremos romos

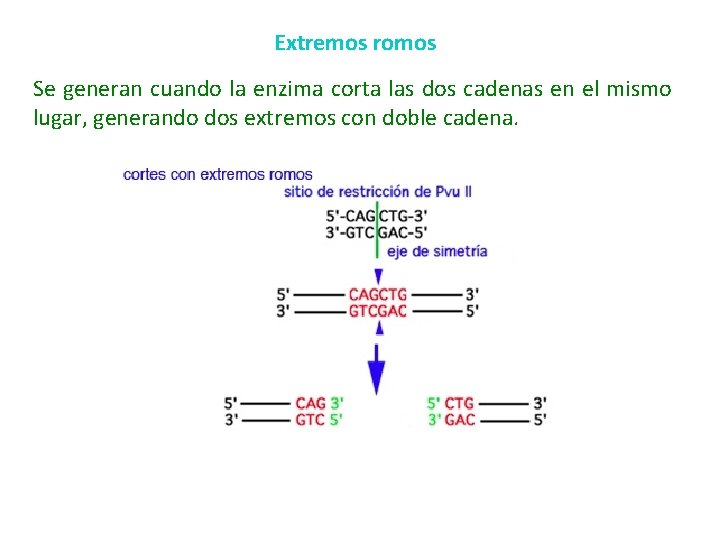
Extremos romos Se generan cuando la enzima corta las dos cadenas en el mismo lugar, generando dos extremos con doble cadena.

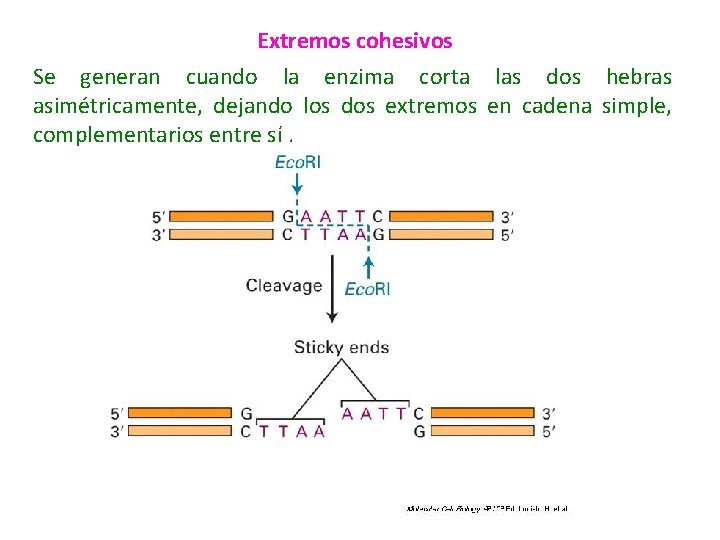
Extremos cohesivos Se generan cuando la enzima corta las dos hebras asimétricamente, dejando los dos extremos en cadena simple, complementarios entre sí.

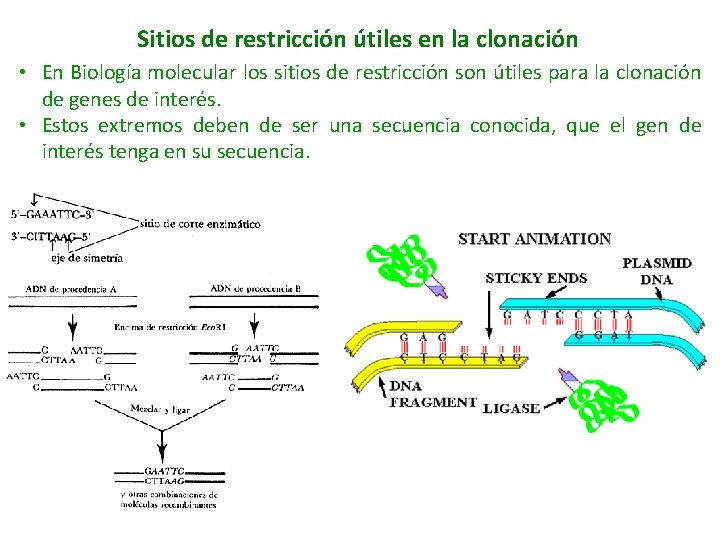
Sitios de restricción útiles en la clonación • En Biología molecular los sitios de restricción son útiles para la clonación de genes de interés. • Estos extremos deben de ser una secuencia conocida, que el gen de interés tenga en su secuencia.

Enzimas de restricción usadas en Biología molecular

Nomenclatura de las enzimas de restricción Las enzimas de restricción se nombran de acuerdo al organismo del cuál provienen y sus características. Nombre: Eco RI E co R I Escherichia coli RY 13 Corresponde a la primera enzima identificada Género Especie Cepa bacteriana Número de identificación


En la práctica: Aislamiento de plásmido mediante Lisis alcalina Objetivos: • Conocer los fundamentos para la purificación del DNA plasmídico y su separación del DNA genómico. • Realizar el aislamiento del DNA plasmídico.

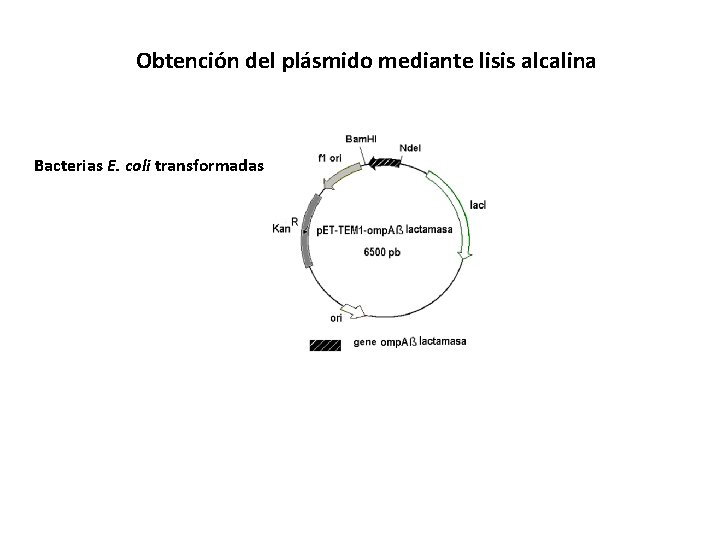
Obtención del plásmido mediante lisis alcalina Bacterias E. coli transformadas


En la práctica: Ensayos de restricción de plásmido Objetivos: • Conocer el principio de separación y detección de ácidos nucleicos en geles de agarosa. • Emplear electroforesis en geles de agarosa para visualizar ácidos nucleicos. • Determinar el tamaño de fragmentos de ácidos nucleicos separados en geles de agarosa. • Conocer la utilidad de las enzimas de restricción en la transformación genética y la biotecnología.

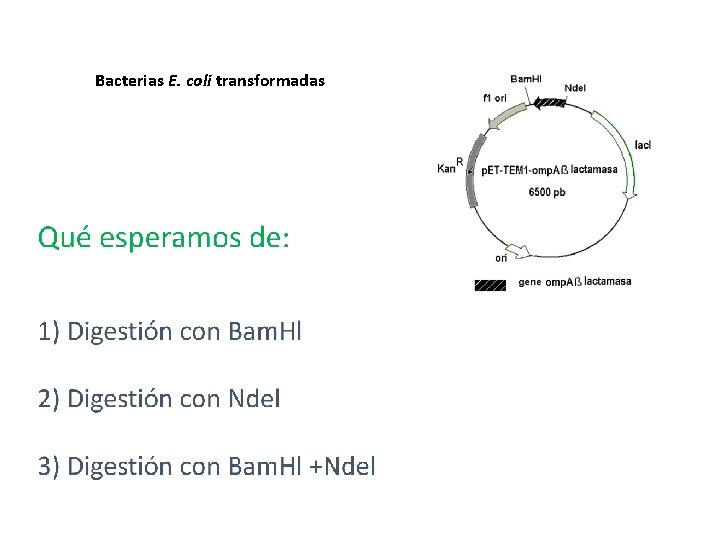
Bacterias E. coli transformadas

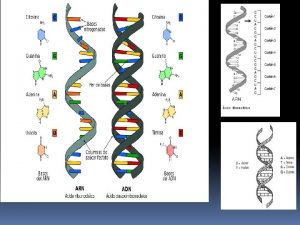
 Codigo gentico
Codigo gentico Plsmidos
Plsmidos Extremos romos y cohesivos
Extremos romos y cohesivos Efecto hipercromico del adn
Efecto hipercromico del adn Aislamiento aereo hospitalario
Aislamiento aereo hospitalario Herramientas y equipos que infrinjan los requisitos de osha
Herramientas y equipos que infrinjan los requisitos de osha Aislamiento parenteral
Aislamiento parenteral Evolucion biologica
Evolucion biologica Visframe
Visframe Transformador de aislamiento
Transformador de aislamiento Aislamiento respiratorio
Aislamiento respiratorio Aislamiento reproductivo
Aislamiento reproductivo Imagenes de aislamiento geografico
Imagenes de aislamiento geografico Cambio filetico
Cambio filetico Time space compression ap human geography
Time space compression ap human geography Refers to the knowledge language values customs
Refers to the knowledge language values customs Example of material culture
Example of material culture Idle time meaning in cost accounting
Idle time meaning in cost accounting Geometric symbol
Geometric symbol Useful and harmful things
Useful and harmful things Transferencia de calor e massa
Transferencia de calor e massa Intercambio de masa
Intercambio de masa Cancer de rion
Cancer de rion Transferencia de registros
Transferencia de registros Mecanismo de transferencia
Mecanismo de transferencia Tasa de transferencia
Tasa de transferencia Ogtdhj hg,hk
Ogtdhj hg,hk Lei de fourier
Lei de fourier Transferencia lejana ejemplos
Transferencia lejana ejemplos Que es el coeficiente global de transferencia de calor
Que es el coeficiente global de transferencia de calor Respuestas
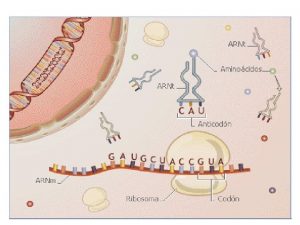
Respuestas Traducción de la información genética
Traducción de la información genética Teorema de norton
Teorema de norton Funcion de transferencia
Funcion de transferencia Calor
Calor Puntos de transferencia
Puntos de transferencia Trs toolbox
Trs toolbox Transferencia documental
Transferencia documental Formula fluxo de calor
Formula fluxo de calor Yunus cengel
Yunus cengel Transferência de calor
Transferência de calor