AISLAMIENTO DE PLSMIDOS James Watson y Francis Crick











- Slides: 17

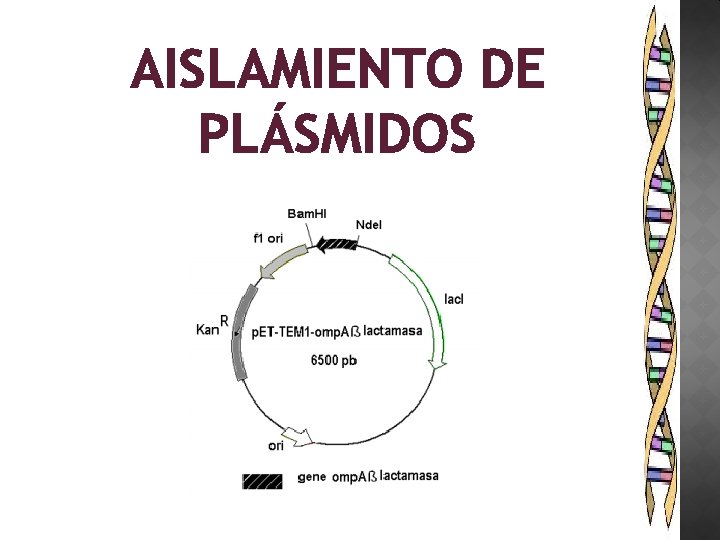
AISLAMIENTO DE PLÁSMIDOS

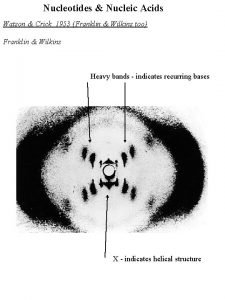
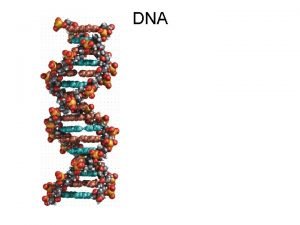
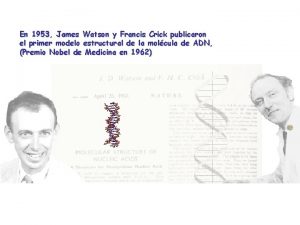
James Watson y Francis Crick en 1953 demostraron que la estructura del ADN consiste en una doble hélice formada por dos cadenas.

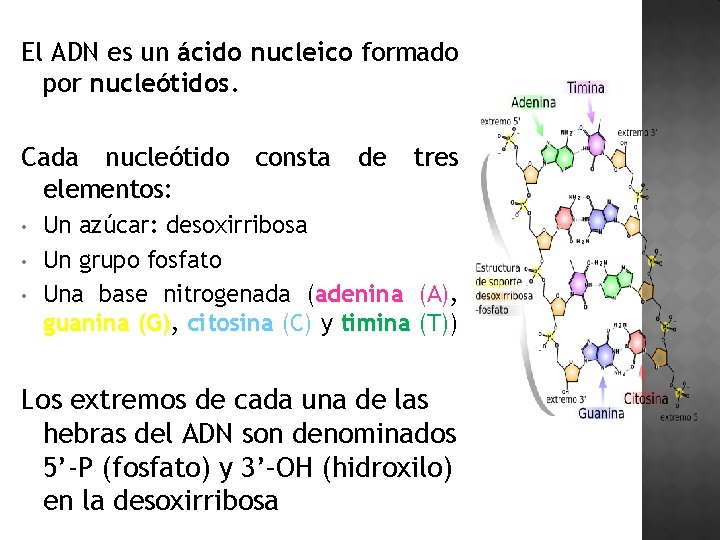
El ADN es un ácido nucleico formado por nucleótidos. Cada nucleótido elementos: • • • consta de tres Un azúcar: desoxirribosa Un grupo fosfato Una base nitrogenada (adenina (A), guanina (G), citosina (C) y timina (T)) Los extremos de cada una de las hebras del ADN son denominados 5’-P (fosfato) y 3’–OH (hidroxilo) en la desoxirribosa

Características de los plásmidos Se replican independientemente del cromosoma del hospedero Portan SOLO genes no esenciales Pueden ser circulares o lineales, de 1 kbp hasta de 1 Mbp Se encuentran superenrrollados, que es la forma más compacta que existe de DNA Se pueden encontrar desde 1 a 3 copias por célula o hasta 100.

Conformaciones de un plásmido Linealizado: Es decir cuando se ha cortado ambas cadenas del DNA sólo una vez. Parcialmente linealizado: Cuando se ha cortado el DNA en una de las cadenas DNA relajado circular. DNA que no se ha empacado. Superenrrolado: Covalentemente cerrado y muy empacado Superenrollado desnaturalizado. Parecido al anterior pero un poco menos empacado

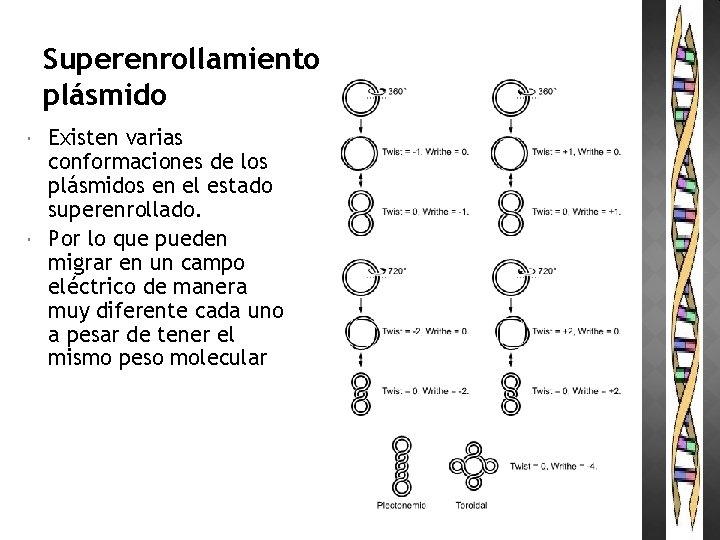
Superenrollamiento plásmido Existen varias conformaciones de los plásmidos en el estado superenrollado. Por lo que pueden migrar en un campo eléctrico de manera muy diferente cada uno a pesar de tener el mismo peso molecular

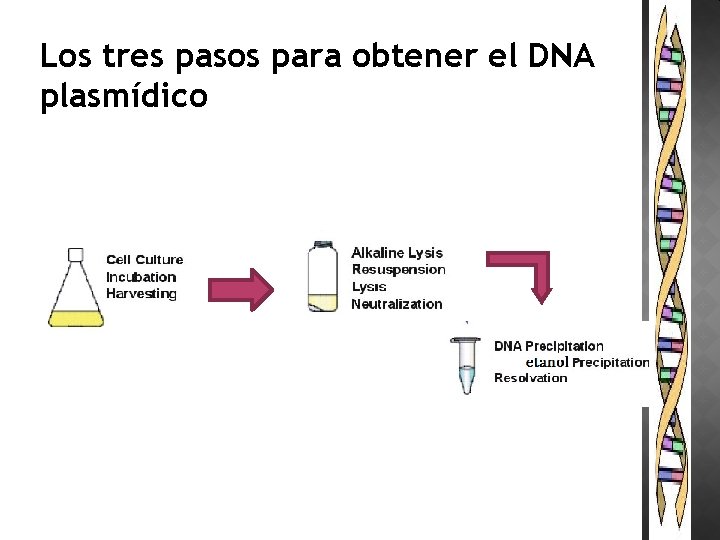
Hay distintos procedimientos para la purificación de DNA plasmídico, aunque todos incluyen los tres pasos siguientes: 1. Crecimiento de las bacterias en un medio selectivo de aquellas que llevan el plásmido. 2. Lisis de las bacterias para la liberación del plásmido 3. Purificación del DNA plasmídico

DESNATURALIZACIÓN ALCALINA O METODO DE LISIS ALCALINA DE BIRNBOIM Y DOLY Este método se basa en la diferente resistencia a valores de p. H elevados del ADN plasmídico y cromosómico. Explota las diferencias en las propiedades de desnaturalización y renaturalización entre el DNA plasmídico y el DNA cromosómico.

Desnaturalización La alcalinización con Na. OH en presencia de un SDS, provoca la lisis celular, la desnaturalización del DNA cromosómico y la liberación de los plásmidos también desnaturalizados.

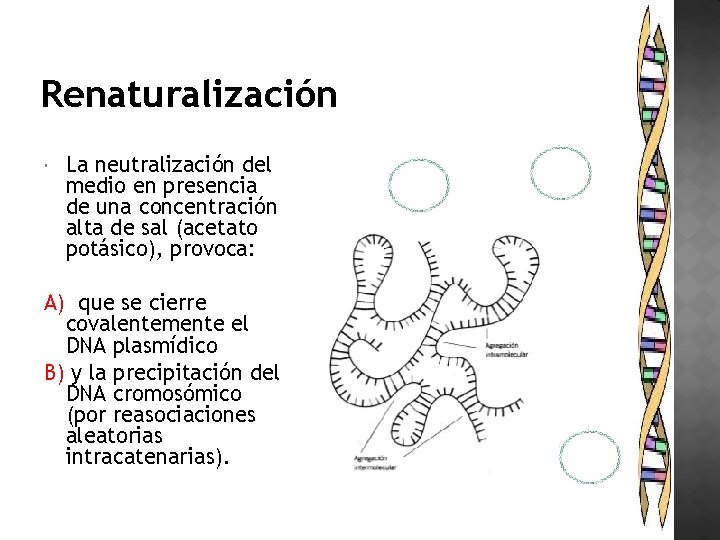
Renaturalización La neutralización del medio en presencia de una concentración alta de sal (acetato potásico), provoca: A) que se cierre covalentemente el DNA plasmídico B) y la precipitación del DNA cromosómico (por reasociaciones aleatorias intracatenarias).

Los agregados insolubles de DNA cromosómico se separan por centrifugación del DNA plasmídico queda en el sobrenadante y conserva mayoritariamente su estructura nativa El ADN plasmídico en el sobrenadante se concentra por precipitación con etanol.

Los tres pasos para obtener el DNA plasmídico

En la práctica: 1. 2. 3. Inocular 5 m. L de célulass transformantes en 5 m. L de medio LB+Kanamicina e incubar a 37◦C durante 12 h aprox. Centrifugar el medio a 10000 rpm por 1 min a 4◦C y descartar el sobrenadante. El paquete celular queda en un microtubo. Resuspender en 300µL de soln GTE fría, agitar en vortex e incubar a Tamb por 5 min.

4. Adicionar 300µL de soln de lisis (que contiene? ) , mezclar por inversión e incubar en hielo por 5 min. 5. Adicionar 300µL de soln fría de acetato de potasio (3 M, p. H=4. 8) y mezcla por inversión e incubar en hielo por 5 min.

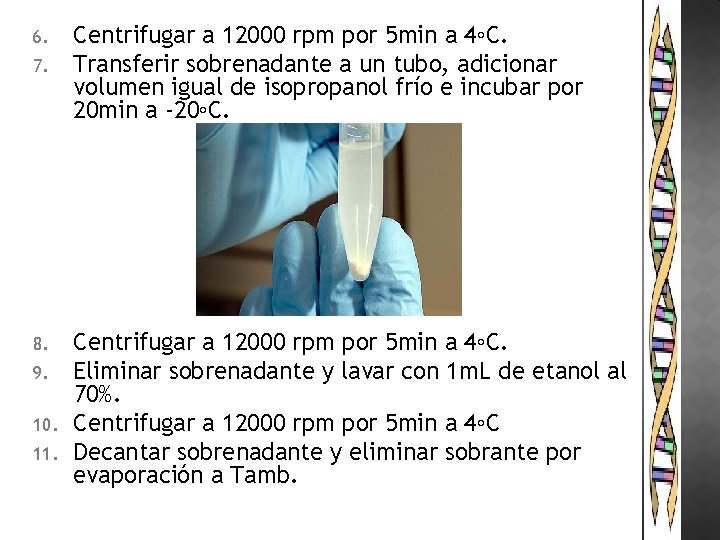
6. 7. 8. 9. 10. 11. Centrifugar a 12000 rpm por 5 min a 4◦C. Transferir sobrenadante a un tubo, adicionar volumen igual de isopropanol frío e incubar por 20 min a -20◦C. Centrifugar a 12000 rpm por 5 min a 4◦C. Eliminar sobrenadante y lavar con 1 m. L de etanol al 70%. Centrifugar a 12000 rpm por 5 min a 4◦C Decantar sobrenadante y eliminar sobrante por evaporación a Tamb.

12. 13. Resuspender con 30µL de agua estéril y guardar a -20◦C. Cuantificar concentración mediante absorbancia a 260 nm (considerar unidad de absorbancia=50µg/m. L).

Cercenado Emilia, Procedimientos en Microbiología Clínica “ Métodos moleculares de tipificación epidemiológica en bacteriología”, SEIMC, http: //www. clinicaelnazareno. org/biolab/sistema/archivos/18. %20 metodos%20 moleculares%20 de%20 tipificacion%20 epi demiologica. pdf
 Watson health francisco 1b
Watson health francisco 1b Francis crick
Francis crick Adenine thymine cytosine and guanine
Adenine thymine cytosine and guanine Francis crick
Francis crick Francis crick nationality
Francis crick nationality Francis crick
Francis crick Francis crick
Francis crick Watson strand crick strand
Watson strand crick strand Watson and crick
Watson and crick Watson and crick thieves
Watson and crick thieves Chargaff rule definition
Chargaff rule definition Modelo de watson y crick
Modelo de watson y crick Phosphoanhydride bond
Phosphoanhydride bond Watson and crick
Watson and crick John b. watson james broadus watson
John b. watson james broadus watson Plsmidos
Plsmidos Extremos romos y cohesivos
Extremos romos y cohesivos Efecto hipercromico del adn
Efecto hipercromico del adn