STRATEGIE REPLIKACJI GENOMW WIRUSOWYCH wirusy RNA wirusy RNA



































- Slides: 46

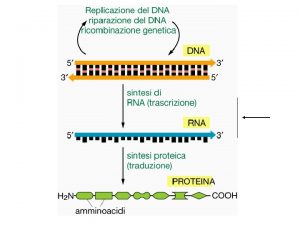
STRATEGIE REPLIKACJI GENOMÓW WIRUSOWYCH

wirusy RNA

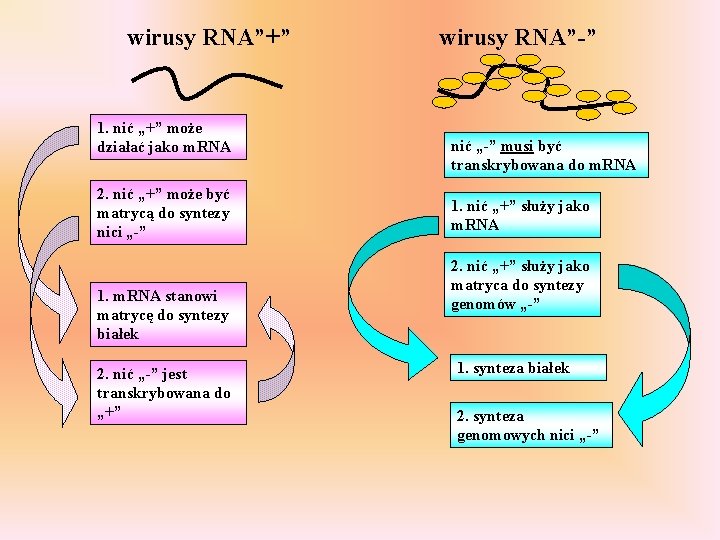
wirusy RNA”+” 1. nić „+” może działać jako m. RNA 2. nić „+” może być matrycą do syntezy nici „-” 1. m. RNA stanowi matrycę do syntezy białek 2. nić „-” jest transkrybowana do „+” wirusy RNA”-” nić „-” musi być transkrybowana do m. RNA 1. nić „+” służy jako m. RNA 2. nić „+” służy jako matryca do syntezy genomów „-” 1. synteza białek 2. synteza genomowych nici „-”

Wirusy RNA”+” Astroviridae Caliciviridae Picornaviridae Coronaviridae Arteriviraidae Flaviviridae Togaviridae

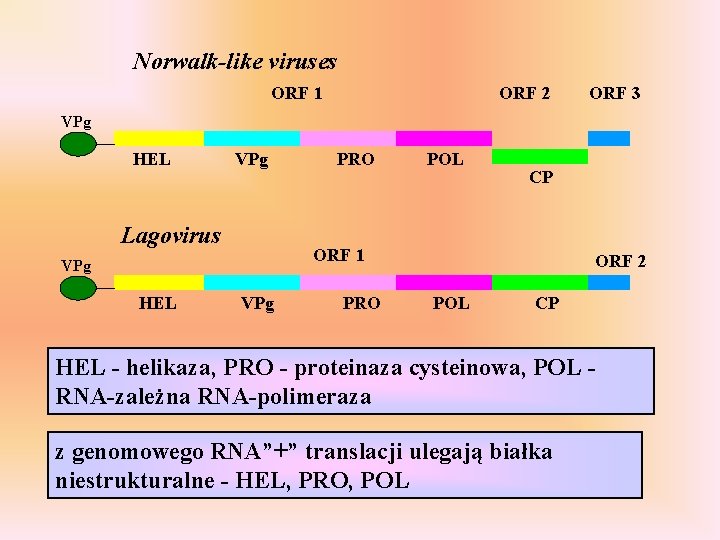
Rodzina: Caliciviridae Rodzaj: Calicivirus „Norwalk-like viruses” „Sapporo-like viruses” Vesivirus nagi, 30 -38 nm Jedna cząsteczka ss. RNA”+” 7. 47. 7 kb, u większości przedstawicieli zawierająca na końcu 5’ białko Vpg Niekiedy dochodzi do enkapsydacji RNA o niepełnej długości - 2. 2 -2. 4 kb

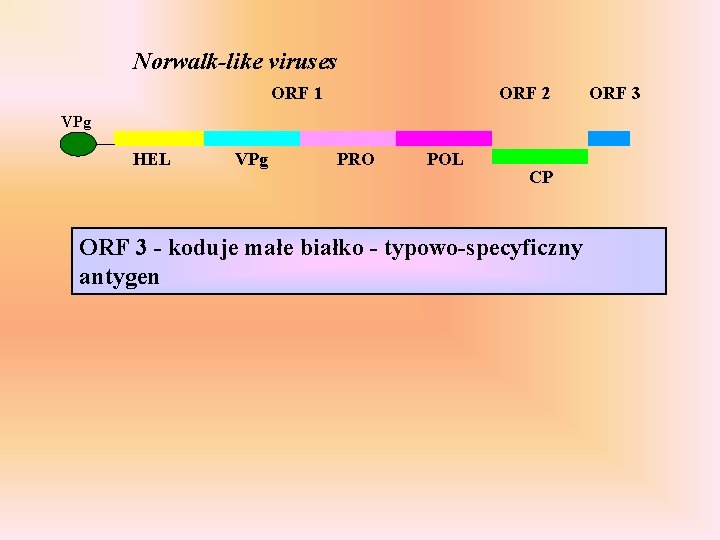
Norwalk-like viruses ORF 1 ORF 2 ORF 3 VPg HEL VPg Lagovirus POL CP ORF 1 VPg HEL PRO VPg PRO ORF 2 POL CP HEL - helikaza, PRO - proteinaza cysteinowa, POL RNA-zależna RNA-polimeraza z genomowego RNA”+” translacji ulegają białka niestrukturalne - HEL, PRO, POL

Norwalk-like viruses ORF 1 ORF 2 VPg HEL VPg PRO POL CP ORF 3 - koduje małe białko - typowo-specyficzny antygen ORF 3

w zakażonych komórkach stwierdza się obecność ds. RNA o długości genomowej i ss. RNA(-); ss. RNA „+” o długości genomu i subgenomowy (-) VPg (+) Obecność ds. RNA o długosci genomowej sugeruje, że replikacja przebiega z wytworzeniem pośredniej formy ss. RNA(-)

Rodzina: Picornaviridae Rodzaj: Enterovirus Rhinovirus Hepatovirus Cardiovirus Aphtovirus Dwudziestościenne, nagie, ok. 30 nm Jedna, zakaźna!!! cząsteczka ss. RNA”+” 7 -8. 5 kb, na końcu 5’ białko Vpg

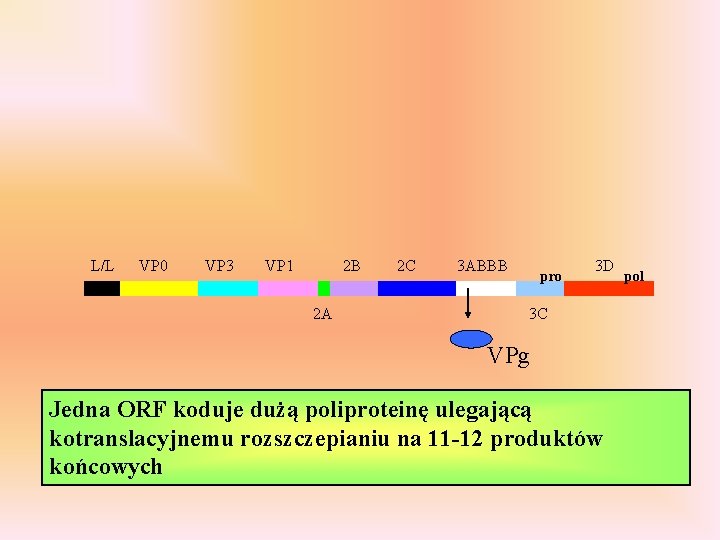
L/L VP 0 VP 3 VP 1 2 B 2 A 2 C 3 ABBB pro 3 D 3 C VPg Jedna ORF koduje dużą poliproteinę ulegającą kotranslacyjnemu rozszczepianiu na 11 -12 produktów końcowych pol

w gładkim reticulum endoplazmatycznym powstają 6 -8 -niciowe formy pośrednie; część nowopowstałych nici „+” kierowana jest do translacji i wytwarzania nowych nici „-”, pozostałe - do enkapsycji Schemat znajdziesz TU

Rodzina: Coronaviridae Rodzaj: Coronavirus Torovirus Otoczkowe: corona - 120 -160 nm, sferyczne, pleomorficzne toro - 120 -140 nm, dyskowate, nerkowate lub pałeczkowate

1 cząsteczka ss. RNA”+” corona ok. 30 kb toro ok. 20 kb Otoczka powstaje przez wypączkowanie wirionów przez błony siateczki endoplazmatycznej i aparatu Golgi’ego; wystające z otoczki cząsteczki glikoproteinowe tworzą „koronę” wokół wirionu Genomowy RNA jest matrycą dla RNA-polimerazy


translacja z genomowego RNA”+” prowadzi do syntezy RNA-zależnej RNA-polimerazy RNA -zależna RNA-polimeraza w pierwszej fazie syntetyzuje nic „antygenomową” („-”) o pełnej długości nić „-” nie występuje w formie wolnej, spotyka się tylko przejściowej postaci dwuniciowej; jej synteza trwa prawdopodobnie przez cały cykl http: //www. rutherford-research. ca/rrx/pharma/images/virus/sars_concept_2. gif na matrycy nici „-” powstaje genomowy RNA „+” oraz 5 -7 m. RNA do translacji pozostałych białek

transkrypcją steruje 5’-sekwencja wiodąca dł. 60 -70 pz; przepisywana jest z końca 3’ nici „-”, oddziela się od niej, ale pozostaje związana z polimerazą w niekodujących regionach międzygenowych leżą sekwencje częściowo komplementarne do wiodącej transkrypty o różnej długości mają taki sam koniec 3’; każdy większy transkrypt ma na końcu 5’ dodatkowy gen translacji ulega tylko jeden gen położony na końcu 5’ w komórce występują też „-” m. RNA! trabskrybowane prawdopodobnie z m. RNA lub powstające wskutek nieciągłej transkrypcji z nici macierzystej „+”

Rodzina: Flaviviridae Rodzaj: Flavivirus Pestivirus Hepacivirus Sferyczne, lipidowa otoczka, 40 -60 nm. Jedna cząsteczka ss. RNA”+”, odpowiednio 10. 7, 12. 5 i 9. 5 kb. Genomowy RNA jest jedynym m. RNA w zakażonej komórce; zawiera pojedyńczą ORF

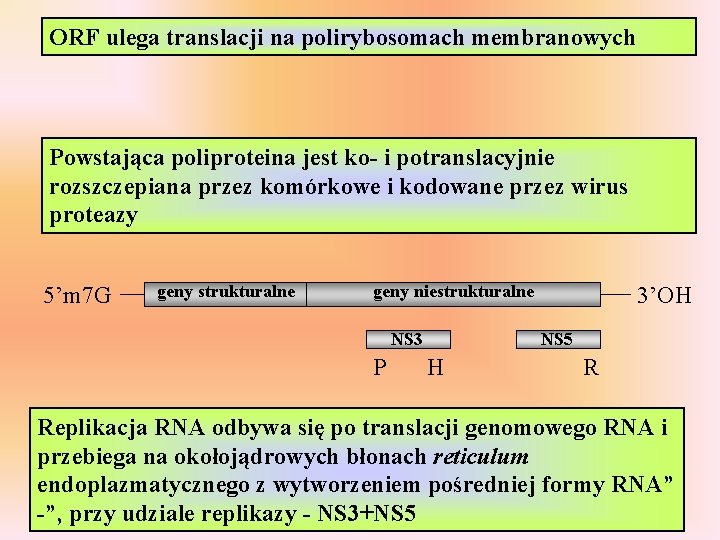
ORF ulega translacji na polirybosomach membranowych Powstająca poliproteina jest ko- i potranslacyjnie rozszczepiana przez komórkowe i kodowane przez wirus proteazy 5’m 7 G geny strukturalne geny niestrukturalne NS 3 P 3’OH NS 5 H R Replikacja RNA odbywa się po translacji genomowego RNA i przebiega na okołojądrowych błonach reticulum endoplazmatycznego z wytworzeniem pośredniej formy RNA” -”, przy udziale replikazy - NS 3+NS 5

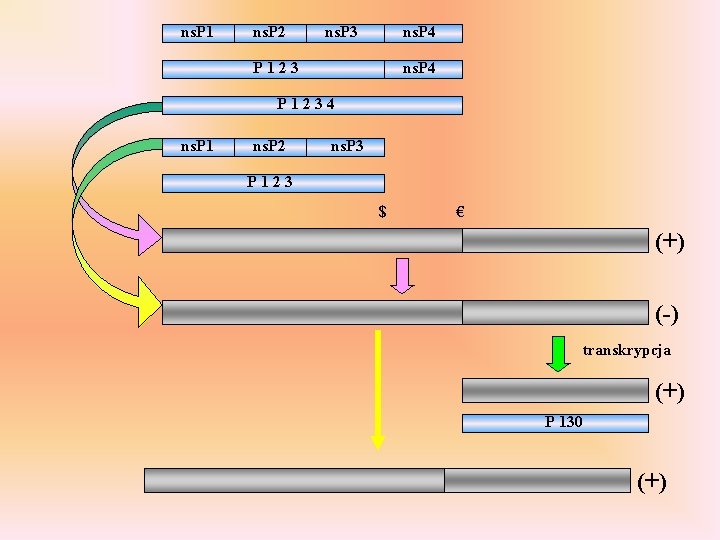
Rodzina: Togaviridae Rodzaj: Alphavirus Rubivirus Sferyczne, lipidowa otoczka, ok. 70 nm. Jedna cząsteczka ss. RNA”+”, 9. 7, 11. 8 kb. Jej część na końcu 5’ służy jako matryca do translacji jednej dużej poliproteiny; dzięki autoproteolitycznej aktywności ulega ona rozpadowi na mniejsze łańcuchy białek niestrukturalnych odpowiedzialnych za replikację genomowego RNA (poprzez formę pośrednią „-”) i wytworzenie krótkiego m. RNA do syntezy białek kapsydu

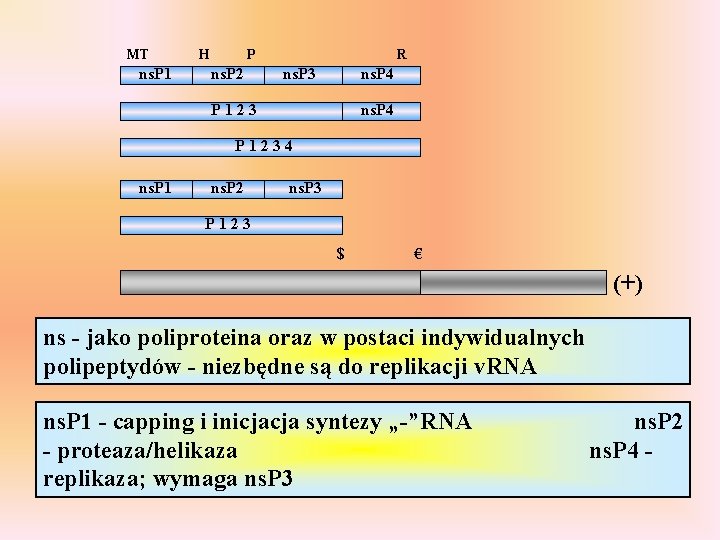
MT ns. P 1 H P ns. P 2 R ns. P 3 ns. P 4 P 1234 ns. P 1 ns. P 2 ns. P 3 P 123 $ € (+) ns - jako poliproteina oraz w postaci indywidualnych polipeptydów - niezbędne są do replikacji v. RNA ns. P 1 - capping i inicjacja syntezy „-”RNA - proteaza/helikaza replikaza; wymaga ns. P 3 ns. P 2 ns. P 4 -

ns. P 1 ns. P 2 ns. P 3 ns. P 4 P 1234 ns. P 1 ns. P 2 ns. P 3 P 123 $ € (+) (-) transkrypcja (+) P 130 (+)

Wirusy RNA”-” Mononegavirales Filoviridae Paramyxoviridae Rhabdoviridae Bornaviridae

Rząd: Mononegavirales Cechy wspólne rzędu: Liniowy niesegmentowany genom ss. RNA „-” 11 -16 kb Helikalny nukleokapsyd Inicjacja transkrypcji pierwotnej przez wirionową RNAzależną RNA polimerazę Podobny układ genów Uzyskiwanie otoczki przez wypączkowywanie

Rodzina: Filoviridae Nitkowate, do 1400 nm x 80 nm, często rozgałęzione ss. RNA 19. 1 kb, z komplementarnymi sekwencjami końcowymi Nukleokapsyd z osią centralną o średnicy ok. 20 nm http: //education. expasy. org/images/Filovirus_virion. jpg

Zakażenie szerzy się przez bliski kontakt, zwłaszcza za pośrednictwem płynów ustrojowych; możliwa jest transmisja kropelkowa Mają tropizm do komórek układu siateczkowośródbłonkowego, fibroblastów i tkanki śródmiąższowej, w szczególności parenchymy wątroby Wirus rozprowadzany jest do wszystkich tkanek, szczególnie dużo stwierdza się go w wątrobie, nerkach, śledzionie i płucach Powoduje gorączkę krwotoczną o bardzo ciężkim przebiegu: Marburg - śmiertelność 30 -35%, Ebola - śmiertelność 50 -88%

L - RNA transkryptaza-polimeraza GP - glikoproteina powierzchniowa NP - nukleoproteina VP 35 - składnik L zawierają sygnały start-stop i konserwatywny pentamer 3’-UAAUU-5’

transkrypcja i replikacja genomu w cytoplazmie, podobna do paramyxo i rhabdo replikacja z wytworzeniem pełnej długości nici „+” w komórkach spotyka się duże ilości nukleokapsydów tworzących cytoplazmatyczne ciałka wtrętowe

Rodzina: Paramyxoviridae Podrodzina: Paramyxovirinae Rodzaj: Respirovirus Rubulavirus Morbilivirus Podrodzina: Pneumovirinae Rodzaj: Pneumovirus Metapneumovirus Ok. 150 nm. lub większe, pleomorficzne

Ok. 150 nm. lub większe, pleomorficzne, często sferyczne ss. RNA „-” od ok. 15. 1 do ok. 15. 9 kb koduje 10 -12 białek, w tym transkryptazę, transferazy, kinazę, neuraminidazę Część wirionów może zawierać ss. RNA „+” 6 -7 elementów transkrypcyjnych koduje 10 -12 białek z których 4 -5 powstaje z 2 -3 ORF zachodzących na siebie u pneumowirusów - 10 białek, 10 ORF

ss. RNA „-” od ok. 15. 1 do ok. 15. 9 kb koduje 10 -12 białek, w tym transkryptazę, transferazy, kinazę, neuraminidazę http: //www. stanford. edu/group/virus/1999/leanna/paramyxo-replication. html Część wirionów może zawierać ss. RNA „+” synteza RNA”+” wymaga ominięcia kodonów normalnie terminujących transkrypcję - replikacyjna aktywność RNA-polimerazy; mechanizm nieznany

Rodzina: Rhabdoviridae Rodzaj: Vesiculovirus Lyssavirus Ephemerovirus Cytorhabdovirus Nucleorhabdovirus 100 -430 x 45 -100 nm ss. RNA „-”, 11 -15 kb

nukleokapsyd aktywny transkrypcyjnie, składnik L zaangażowany w transkrypcję i replikację genomu G - białko odpowiedzialne za przyleganie wirionu do receptorów, endocytozę i fuzję http: //www. mcb. uct. ac. za/cann/335/Rhabdo 3. gif N - składnik nukleokapsydu, bierze udział w transkrypcji i replikacji, generuje odporność NS (P, M 1) - składnik polimerazy m (M 2) - regulator transkrypcji, hamuje syntezy komórkowe

Translacja M, L, N i NS – na polisomach cytoplazmatycznych G - na membranowych N i NS - białka odpowiedzialne za przełączenie działania L z transkrypcyjnego na replikacyjne i umożliwiające wytworzenie pośredniej formy „+”

Wirusy RNA”-” genom segementowany Orthomyxoviridae Bunyaviridae Arenaviridae

Rodzina: Orthomyxoviridae Rodzaj: Influenzavirus A (8) Influenzavirus B (8) Influenzavirus C (7) Thogotovirus (6) wiriony sferyczne lub pleomorficzne 80 -120 nm, ss. RNA”-” segmentowany, 10 -14 kb, segmenty od 900 do 2500 nt

transkrypcja wymaga aktywności enzymatycznej białek wirusowych i komórkowych - RNA-zależnej RNApolimerazy i RNApol. II gospodarza odpowiedzialnej za syntezę starterów (met i cap) – powstaje m. RNA http: //www. mcb. uct. ac. za/cann/335/Orthomyxo 3. gif NP. warunkuje przełączenie działania RNA polimerazy na aktywność replikatywną pierwszy etapem transkrypcji replikatywnej jest wykonanie kopii wszystkich segmentów (bez met i cap, bez poly-A) „+” kopie segementów pozostają w jądrze jako nukleokapsydy (koliste)

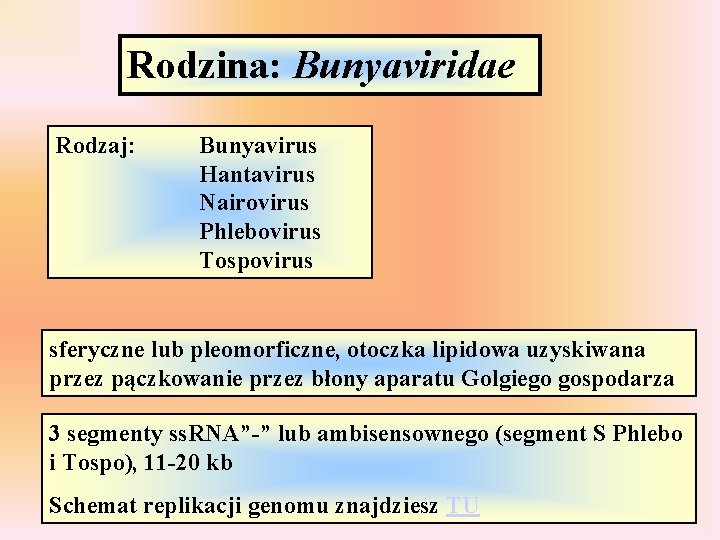
Rodzina: Bunyaviridae Rodzaj: Bunyavirus Hantavirus Nairovirus Phlebovirus Tospovirus sferyczne lub pleomorficzne, otoczka lipidowa uzyskiwana przez pączkowanie przez błony aparatu Golgiego gospodarza 3 segmenty ss. RNA”-” lub ambisensownego (segment S Phlebo i Tospo), 11 -20 kb Schemat replikacji genomu znajdziesz TU

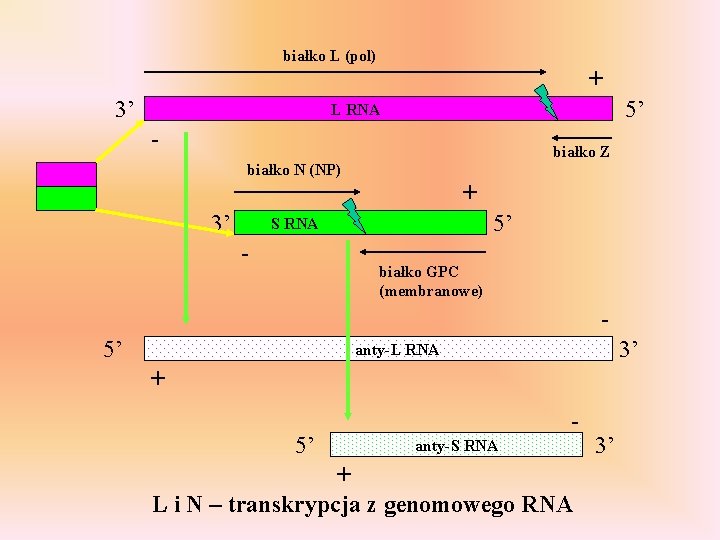
Rodzina: Arenaviridae http: //staff. vbi. vt. edu/pathport/pathi nfo_images/CCHFV/rc. jpe

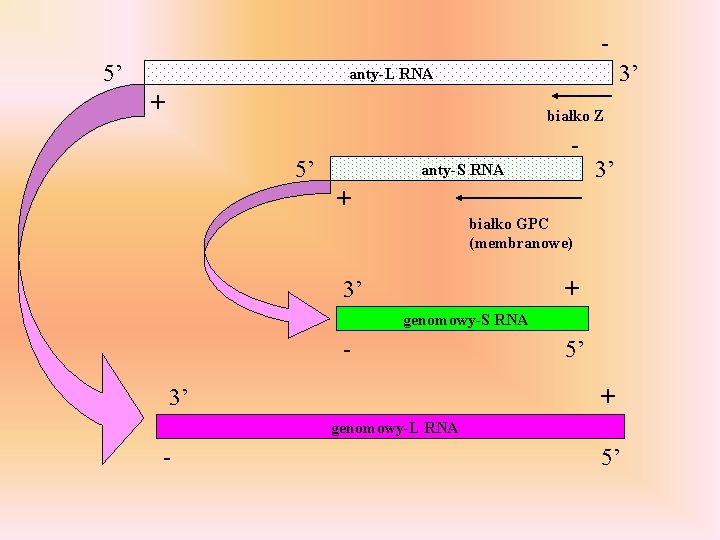
białko L (pol) + 3’ 5’ L RNA - białko Z białko N (NP) + 3’ 5’ S RNA - białko GPC (membranowe) 5’ 3’ anty-L RNA + 5’ anty-S RNA + L i N – transkrypcja z genomowego RNA 3’

5’ 3’ anty-L RNA + białko Z - 5’ anty-S RNA 3’ + białko GPC (membranowe) 3’ + genomowy-S RNA 3’ 5’ + genomowy-L RNA - 5’

Wirusy ds. RNA genom segementowany Reoviridae Birnaviridae

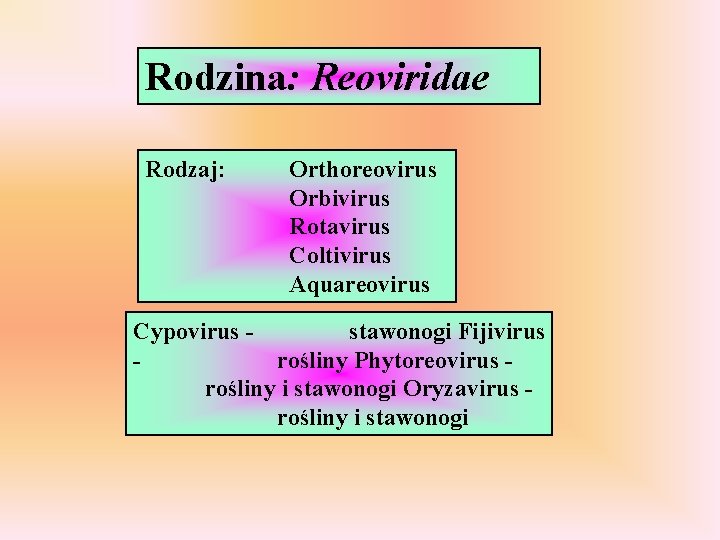
Rodzina: Reoviridae Rodzaj: Orthoreovirus Orbivirus Rotavirus Coltivirus Aquareovirus Cypovirus stawonogi Fijivirus rośliny Phytoreovirus rośliny i stawonogi Oryzavirus rośliny i stawonogi

genom złożony z 10 -12 segmentów ds. RNA transkrypcja konserwatywna, wewnątrz rdzenia; rodzicielski RNA pozostaje w rdzeniu http: //instruct 1. cit. cornell. edu/research/parker_lab /IMAGES/replication%20 diagram. jpg transkrypcja wczesna, na ds. RNA rodzicielskim; najpierw powstają 4 transkrypty o pełnej długości służące jako m. RNA i matryce do dobudowania nici „-” ich produkty niezbędne są do odblokowania transkrypcji pozostałych segmentów, blokowanych prawdopodobnie przez gospodarza

replikacja genomu - przez dobudowanie nici „-” transkrypcja późna - zachodzi na ds. RNA potomnym nie występują wolne segmenty „-” i wolne segmenty ds. RNA

Rodzina: Birnaviridae Rodzaj: Aquabirnavirus Avibirnavirus Entomobirnavirus ok. 60 nm, nagie, jednowarstwowy kapsyd ds. RNA - dwa segmenty (A i B), ok. 3100 i 2800 bp; na segmencie A występują 2 ORFs, na B - 1 ORF 2 daje początek jednej poliproteinie która jest kotranslacyjnie rozszczepiana (przez NS) na 3 polipeptydy

Vp 1/Vpg - RNA-zależna RNA-polimeraza wytwarza 2 nici m. RNA o pełnej długości wiadomo, że występują replikacyjne formy pośrednie, ale nie jest jasne, czy powstają wolne nici „-” nie obserwuje się syntezy białek wczesnych lub późnych
 Genomw
Genomw Genomw
Genomw Program antywirusowy definicja
Program antywirusowy definicja Wirusy komputerowe prezentacja
Wirusy komputerowe prezentacja Budowa wirusa komputerowego
Budowa wirusa komputerowego Jednostki miar w sieciach komputerowych
Jednostki miar w sieciach komputerowych Wirus komputerowy definicja
Wirus komputerowy definicja Politique d écrémage
Politique d écrémage Strategie skimming
Strategie skimming Qurtel strategie
Qurtel strategie Stratégie de coping
Stratégie de coping Strategie das
Strategie das Politique rh cours
Politique rh cours Stratégie corporate cours
Stratégie corporate cours Storytelling autismo
Storytelling autismo Stratégie déduite
Stratégie déduite Masterclass strategie implementatie
Masterclass strategie implementatie Strategie didattiche inclusive
Strategie didattiche inclusive La star stratégie
La star stratégie Strategie didattiche inclusive
Strategie didattiche inclusive Vision mission strategie pyramide
Vision mission strategie pyramide Sap strategie
Sap strategie Copy strategy def
Copy strategy def Rhetorical strategy
Rhetorical strategy Stratégie marketing
Stratégie marketing Adidas diagnostic interne
Adidas diagnostic interne Jan rauscher
Jan rauscher Vinay e darbelnet
Vinay e darbelnet Cyber risicobereidheid
Cyber risicobereidheid St strategie
St strategie Copy-strategie beispiel bier
Copy-strategie beispiel bier Grondvormen logistiek
Grondvormen logistiek Villamoo
Villamoo Outpacing strategie
Outpacing strategie Strategie top down
Strategie top down Nim spiel strategie
Nim spiel strategie Concept d'evocation
Concept d'evocation Strategie didactica
Strategie didactica Laboratorio di strategie e creatività pubblicitarie
Laboratorio di strategie e creatività pubblicitarie Stratégie émergente et délibérée
Stratégie émergente et délibérée Instrumente der ezb
Instrumente der ezb Starbucks internationale strategie
Starbucks internationale strategie Skimming is used to identify the
Skimming is used to identify the Vier gewinnt strategie algorithmus
Vier gewinnt strategie algorithmus Strategia pull
Strategia pull Lettura orientativa
Lettura orientativa Gerarchia delle strategie dell'impresa
Gerarchia delle strategie dell'impresa