Ewolucja genw i genomw Rozmiar genomu Eukaryota 1



- Slides: 33

Ewolucja genów i genomów

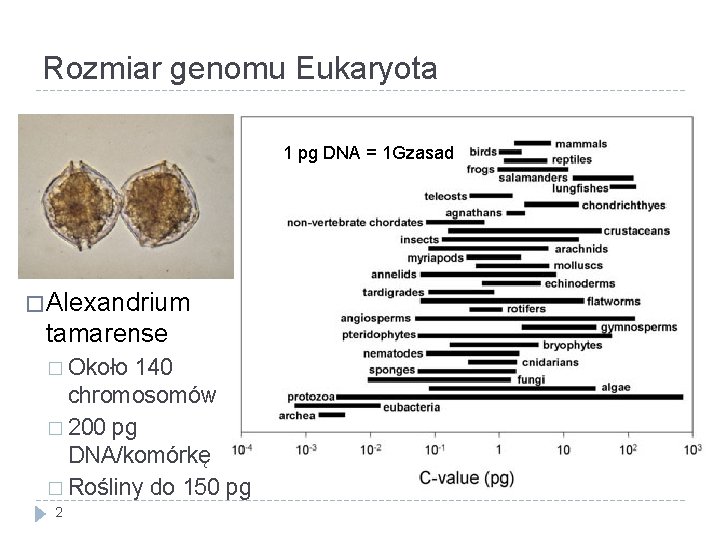
Rozmiar genomu Eukaryota 1 pg DNA = 1 Gzasad � Alexandrium tamarense � Około 140 chromosomów � 200 pg DNA/komórkę � Rośliny do 150 pg 2

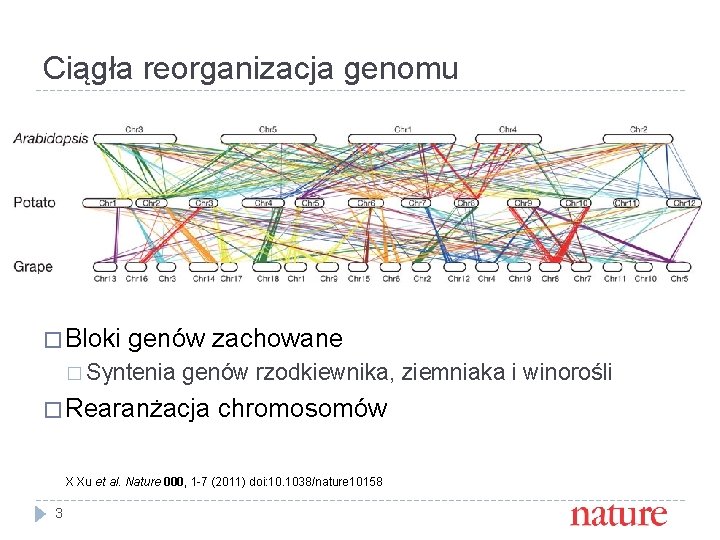
Ciągła reorganizacja genomu � Bloki genów zachowane � Syntenia genów rzodkiewnika, ziemniaka i winorośli � Rearanżacja chromosomów X Xu et al. Nature 000, 1 -7 (2011) doi: 10. 1038/nature 10158 3

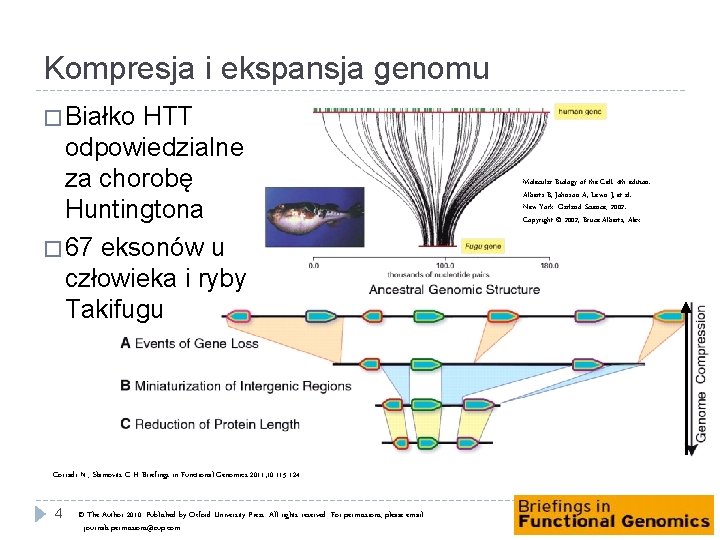
Kompresja i ekspansja genomu � Białko HTT odpowiedzialne za chorobę Huntingtona � 67 eksonów u człowieka i ryby Takifugu Corradi N , Slamovits C H Briefings in Functional Genomics 2011; 10: 115 -124 4 © The Author 2010. Published by Oxford University Press. All rights reserved. For permissions, please email: journals. permissions@oup. com Molecular Biology of the Cell. 4 th edition. Alberts B, Johnson A, Lewis J, et al. New York: Garland Science; 2002. Copyright © 2002, Bruce Alberts, Alex

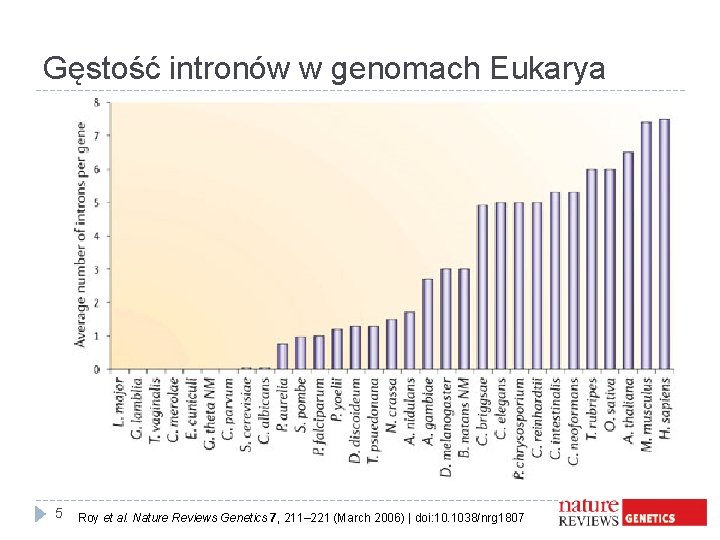
Gęstość intronów w genomach Eukarya 5 Roy et al. Nature Reviews Genetics 7, 211– 221 (March 2006) | doi: 10. 1038/nrg 1807

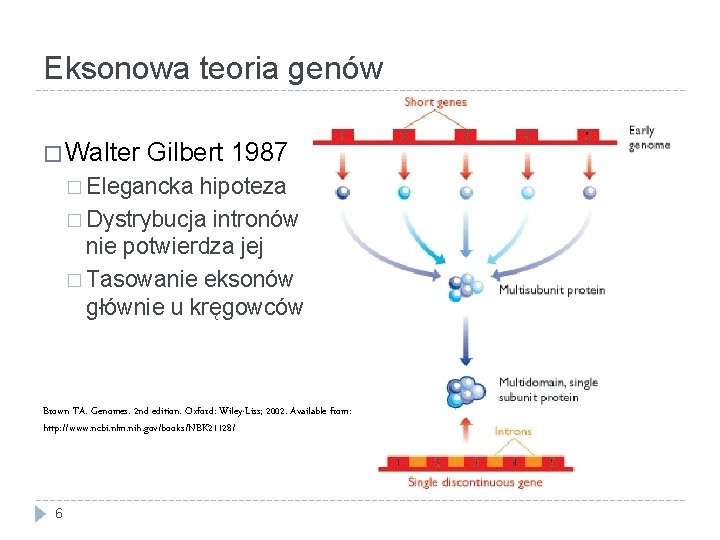
Eksonowa teoria genów � Walter Gilbert 1987 � Elegancka hipoteza � Dystrybucja intronów nie potwierdza jej � Tasowanie eksonów głównie u kręgowców Brown TA. Genomes. 2 nd edition. Oxford: Wiley-Liss; 2002. Available from: http: //www. ncbi. nlm. nih. gov/books/NBK 21128/ 6

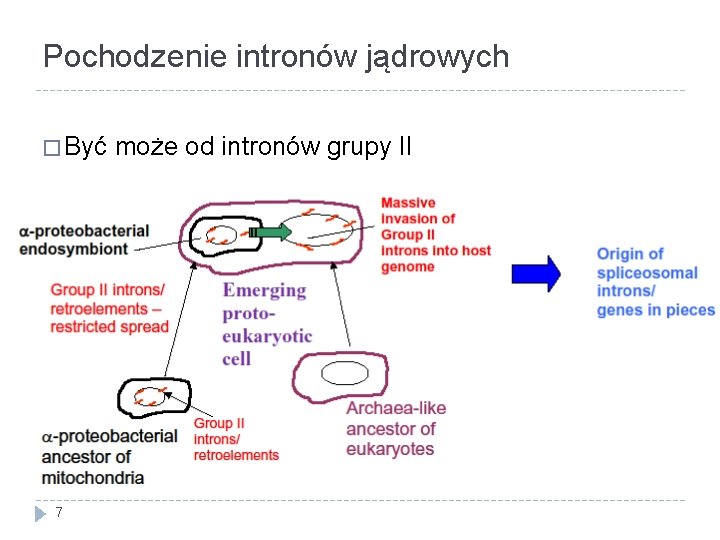
Pochodzenie intronów jądrowych � Być 7 może od intronów grupy II

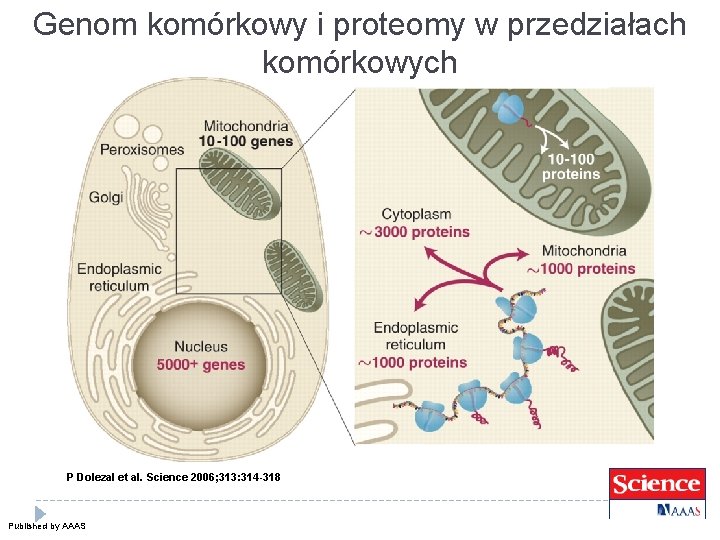
Genom komórkowy i proteomy w przedziałach komórkowych P Dolezal et al. Science 2006; 313: 314 -318 Published by AAAS

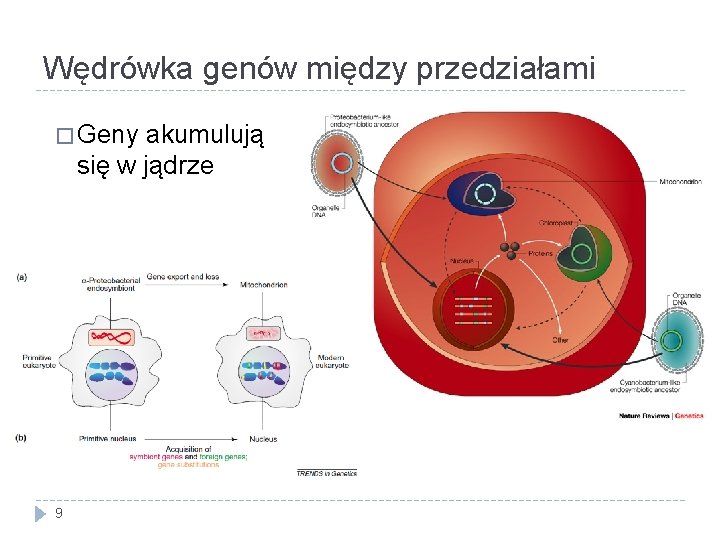
Wędrówka genów między przedziałami � Geny akumulują się w jądrze 9

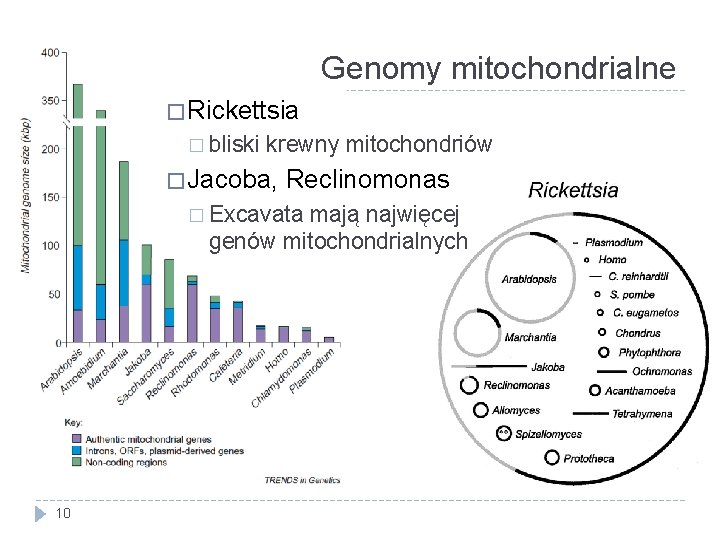
Genomy mitochondrialne � Rickettsia � bliski krewny mitochondriów � Jacoba, Reclinomonas � Excavata mają najwięcej genów mitochondrialnych 10

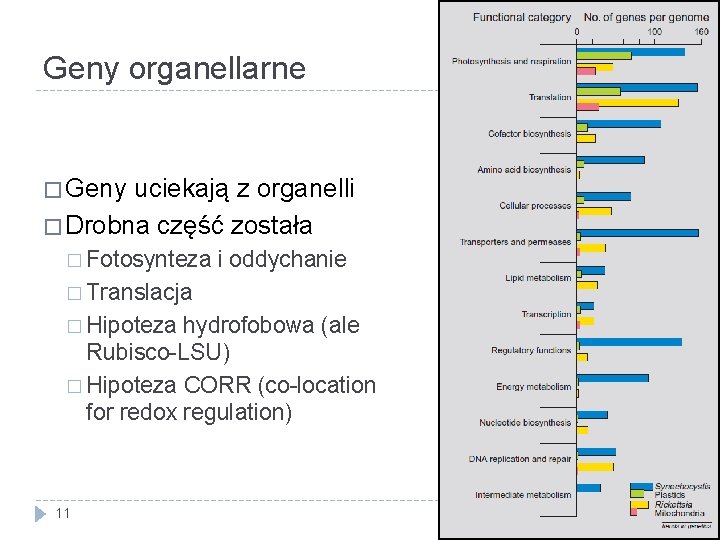
Geny organellarne � Geny uciekają z organelli � Drobna część została � Fotosynteza i oddychanie � Translacja � Hipoteza hydrofobowa (ale Rubisco-LSU) � Hipoteza CORR (co-location for redox regulation) 11

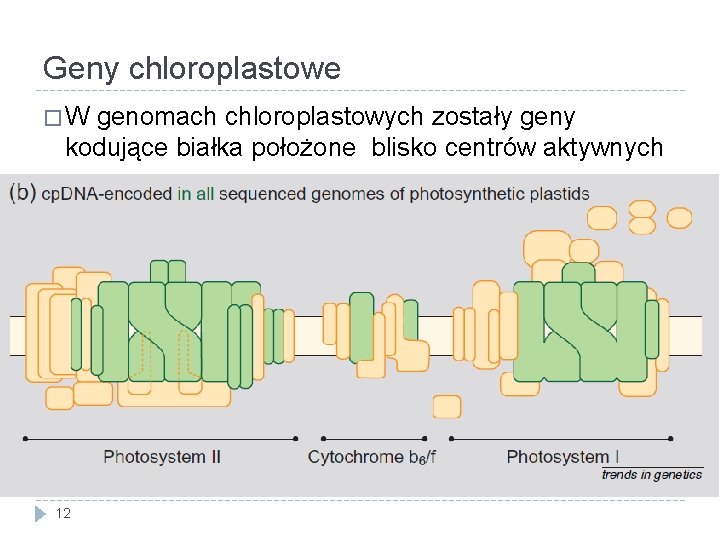
Geny chloroplastowe �W genomach chloroplastowych zostały geny kodujące białka położone blisko centrów aktywnych 12

Ewolucja przez duplikacje genów � Hemoglobina 13 Brown TA. Genomes. 2 nd edition. Oxford: Wiley-Liss; 2002.

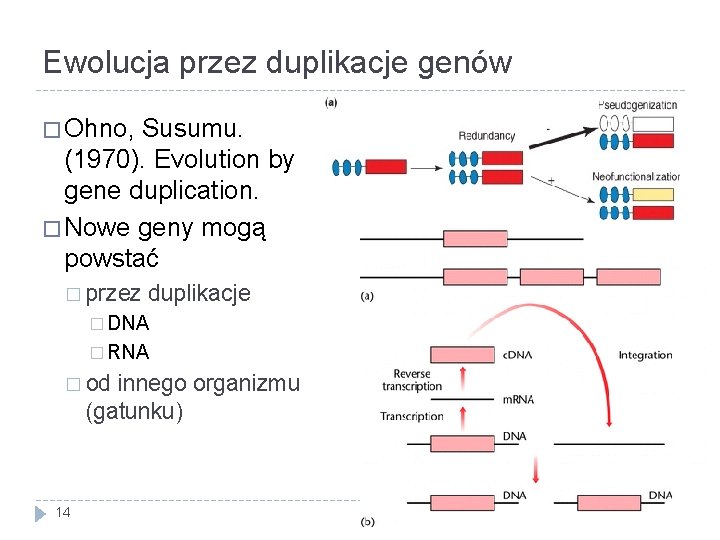
Ewolucja przez duplikacje genów � Ohno, Susumu. (1970). Evolution by gene duplication. � Nowe geny mogą powstać � przez duplikacje � DNA � RNA � od innego organizmu (gatunku) 14

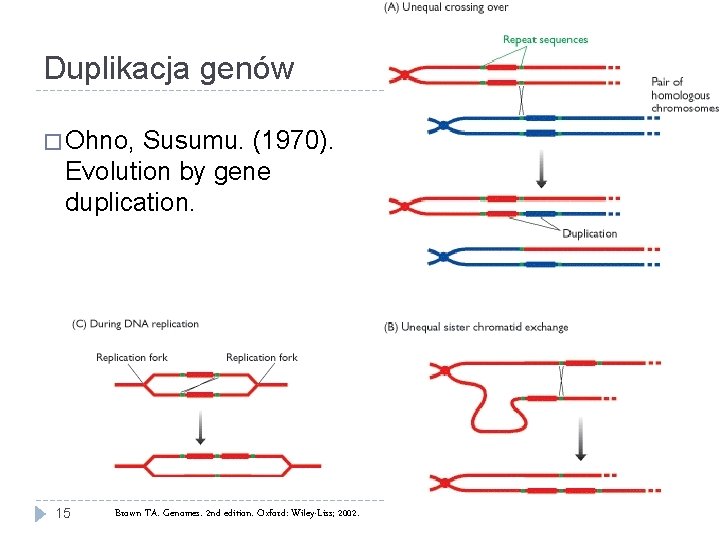
Duplikacja genów � Ohno, Susumu. (1970). Evolution by gene duplication. 15 Brown TA. Genomes. 2 nd edition. Oxford: Wiley-Liss; 2002.

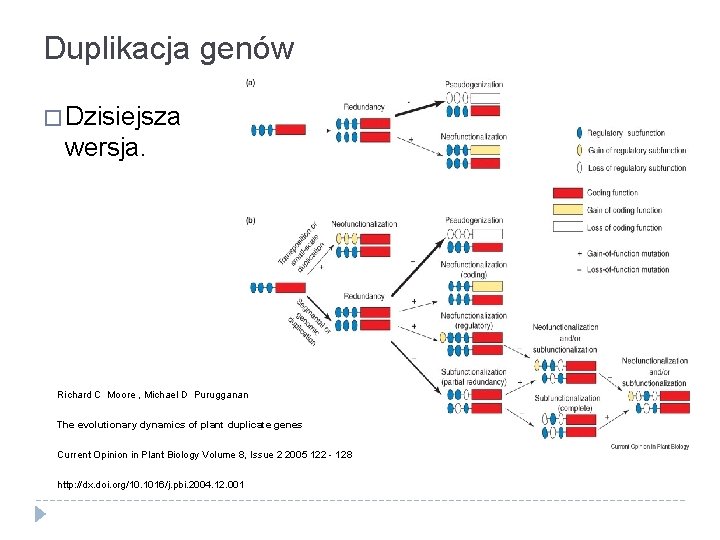
Duplikacja genów � Dzisiejsza wersja. Richard C Moore , Michael D Purugganan The evolutionary dynamics of plant duplicate genes Current Opinion in Plant Biology Volume 8, Issue 2 2005 122 - 128 http: //dx. doi. org/10. 1016/j. pbi. 2004. 12. 001

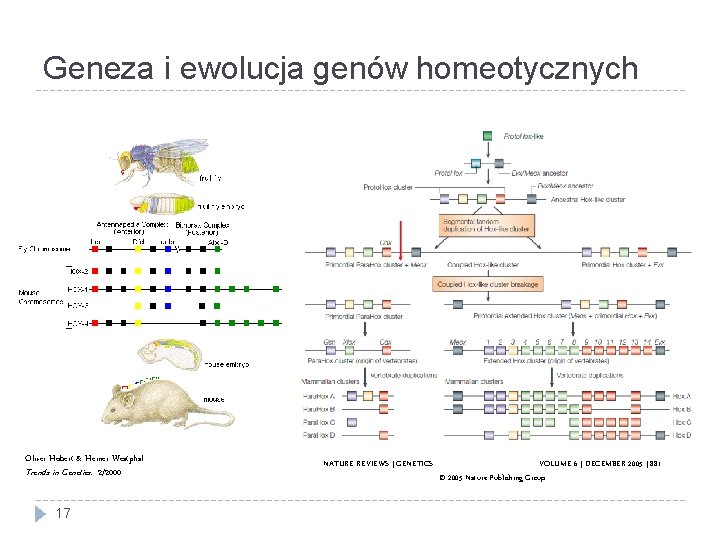
Geneza i ewolucja genów homeotycznych Oliver Hobert & Heiner Westphal Trends in Genetics: 2/2000 17 NATURE REVIEWS | GENETICS VOLUME 6 | DECEMBER 2005 | 881 © 2005 Nature Publishing Group

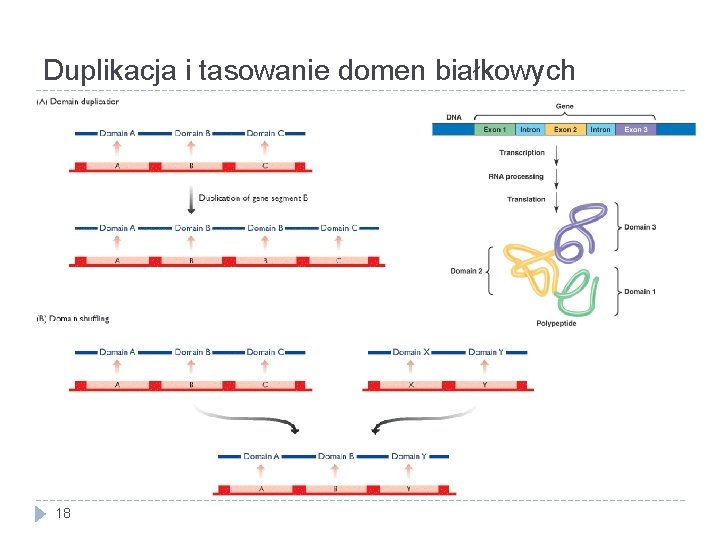
Duplikacja i tasowanie domen białkowych 18

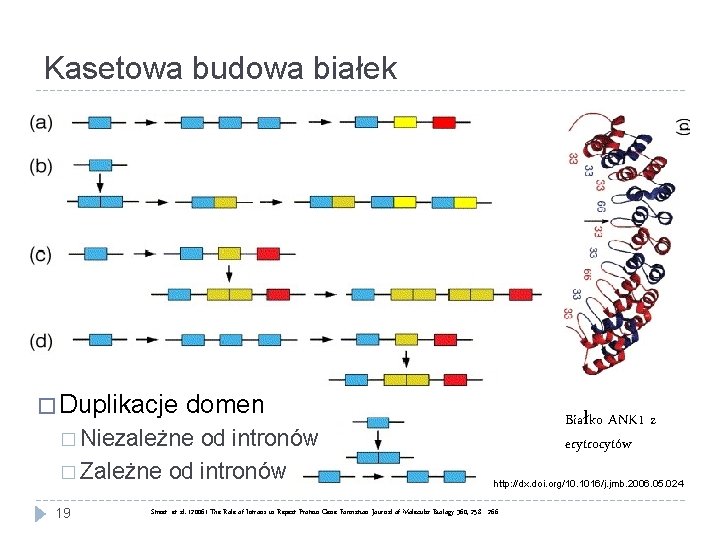
Kasetowa budowa białek � Duplikacje domen od intronów � Zależne od intronów Białko ANK 1 z erytrocytów � Niezależne 19 http: //dx. doi. org/10. 1016/j. jmb. 2006. 05. 024 Street et al. (2006) The Role of Introns in Repeat Protein Gene Formation Journal of Molecular Biology 360, 258 - 266

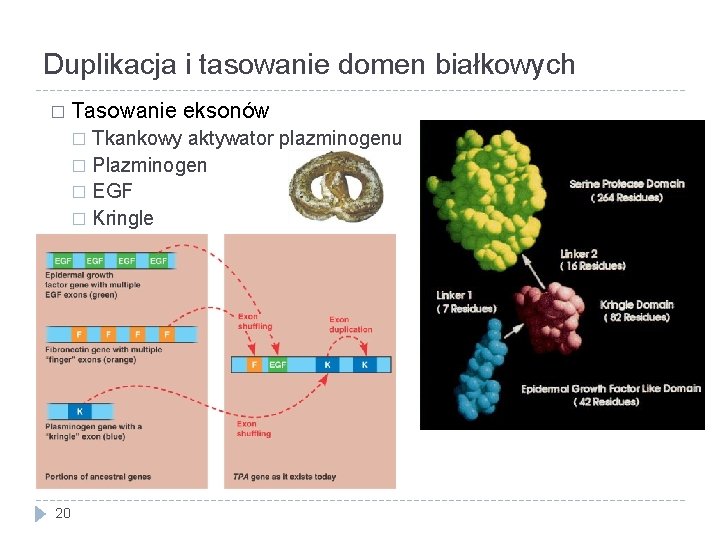
Duplikacja i tasowanie domen białkowych � Tasowanie eksonów Tkankowy aktywator plazminogenu � Plazminogen � EGF � Kringle � 20

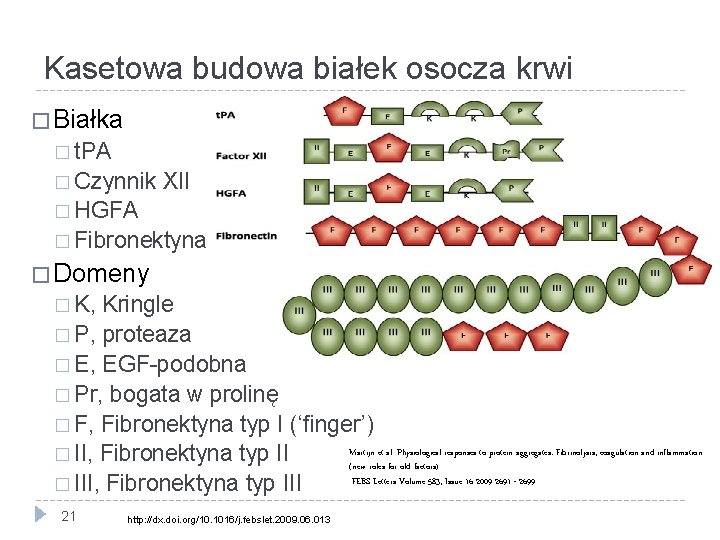
Kasetowa budowa białek osocza krwi � Białka � t. PA � Czynnik XII � HGFA � Fibronektyna � Domeny � K, Kringle � P, proteaza � E, EGF-podobna � Pr, bogata w prolinę � F, Fibronektyna typ I (‘finger’) Martijn et al. Physiological responses to protein aggregates: Fibrinolysis, coagulation and inflammation � II, Fibronektyna typ II (new roles for old factors) FEBS Letters Volume 583, Issue 16 2009 2691 - 2699 � III, Fibronektyna typ III 21 http: //dx. doi. org/10. 1016/j. febslet. 2009. 06. 013

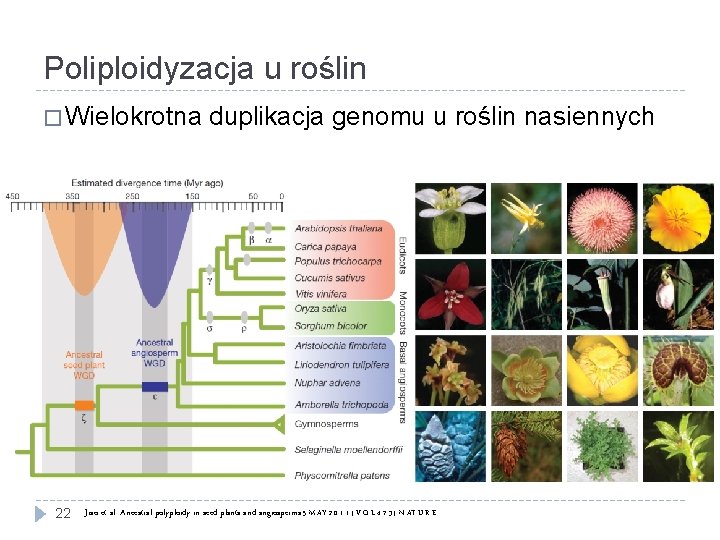
Poliploidyzacja u roślin � Wielokrotna 22 duplikacja genomu u roślin nasiennych Jiao et al. Ancestral polyploidy in seed plants and angiosperms 5 M AY 2 0 1 1 | V O L 4 7 3 | N AT U R E

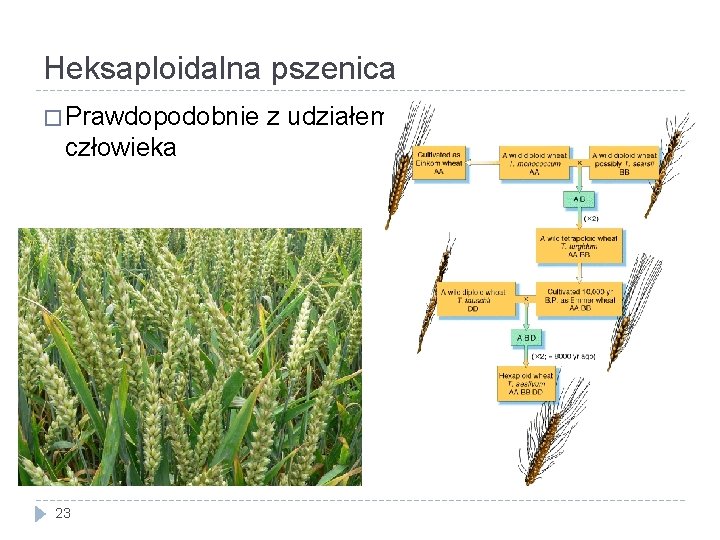
Heksaploidalna pszenica � Prawdopodobnie człowieka 23 z udziałem

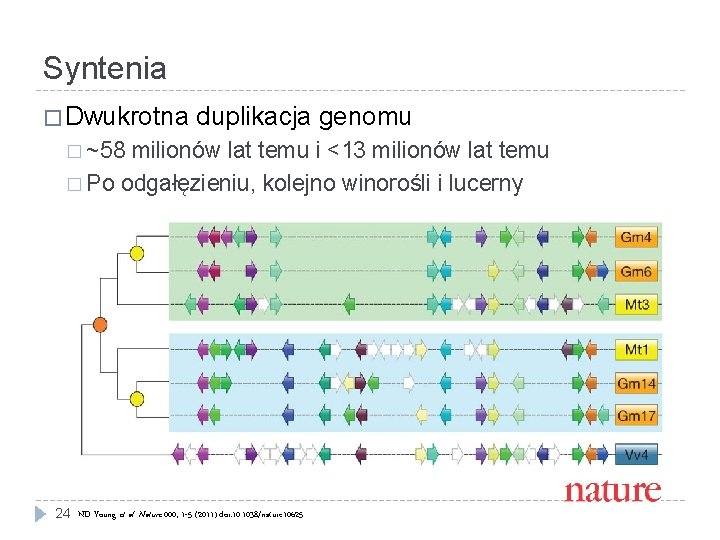
Syntenia � Dwukrotna duplikacja genomu � ~58 milionów lat temu i <13 milionów lat temu � Po odgałęzieniu, kolejno winorośli i lucerny 24 ND Young et al. Nature 000, 1 -5 (2011) doi: 10. 1038/nature 10625

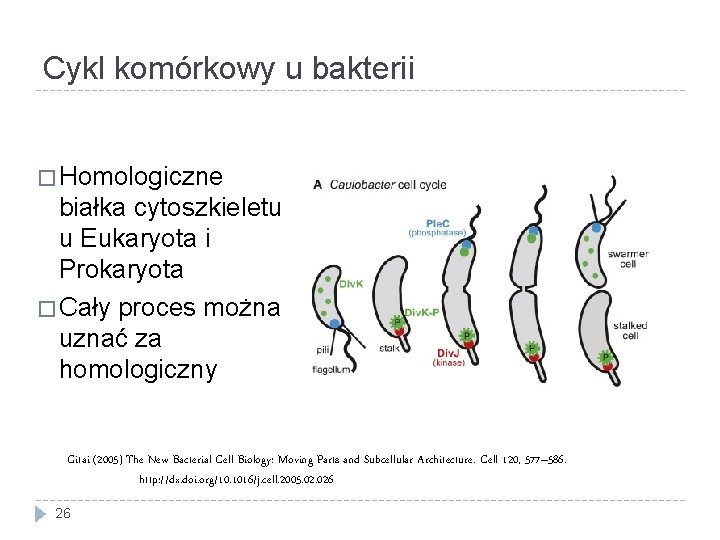
Rozmnażanie bezpłciowe � Głównie Prokaryota, ale też u Eukaryota � Skomplikowany cykl komórkowy u Eukaryota homologiczny z podziałem komórkowym bakterii 25

Cykl komórkowy u bakterii � Homologiczne białka cytoszkieletu u Eukaryota i Prokaryota � Cały proces można uznać za homologiczny Gitai (2005) The New Bacterial Cell Biology: Moving Parts and Subcellular Architecture. Cell 120, 577– 586. http: //dx. doi. org/10. 1016/j. cell. 2005. 026 26

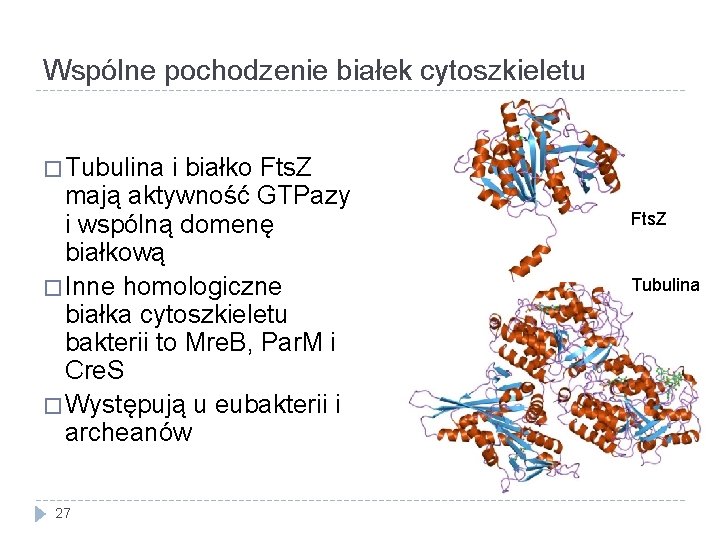
Wspólne pochodzenie białek cytoszkieletu � Tubulina i białko Fts. Z mają aktywność GTPazy i wspólną domenę białkową � Inne homologiczne białka cytoszkieletu bakterii to Mre. B, Par. M i Cre. S � Występują u eubakterii i archeanów 27 Fts. Z Tubulina

Wspólne pochodzenie białek cytoszkieletu � Struktura drugorzędowa i przyrównanie sekwencji białka Fts. Z z M. jannaschii oraz tubulin ze świni Faguy & Doolittle (1998) Cytoskeletal proteins: The evolution of cell division. Current Biology 8, R 338 - R 341 http: //dx. doi. org/10. 1016/S 0960 -9822(98)70216 -7 28

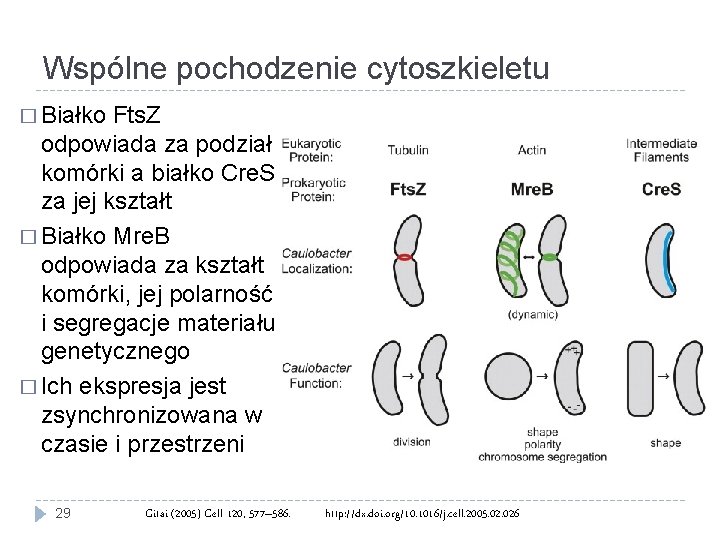
Wspólne pochodzenie cytoszkieletu � Białko Fts. Z odpowiada za podział komórki a białko Cre. S za jej kształt � Białko Mre. B odpowiada za kształt komórki, jej polarność i segregacje materiału genetycznego � Ich ekspresja jest zsynchronizowana w czasie i przestrzeni 29 Gitai (2005) Cell 120, 577– 586. http: //dx. doi. org/10. 1016/j. cell. 2005. 026

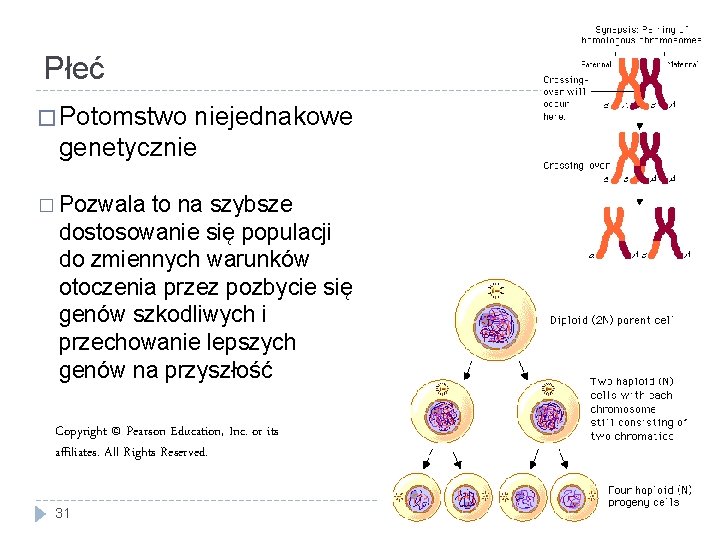
Płeć i rozmnażanie płciowe � To proces jednokomórkowy � Wiele eukariontów rozmnaża się bezpłciowo � Powstał u prokariontów i rozwinął się u wczesnych eukariontów zapewne dzięki rekombinacji � Daje przewagę ewolucyjną (w większości sytuacji) � Kosztuje � Wysiłek zlokalizowania i skłonienia partnera � Skomplikowanie cyklu komórkowego 30

Płeć � Potomstwo niejednakowe genetycznie � Pozwala to na szybsze dostosowanie się populacji do zmiennych warunków otoczenia przez pozbycie się genów szkodliwych i przechowanie lepszych genów na przyszłość Copyright © Pearson Education, Inc. or its affiliates. All Rights Reserved. 31

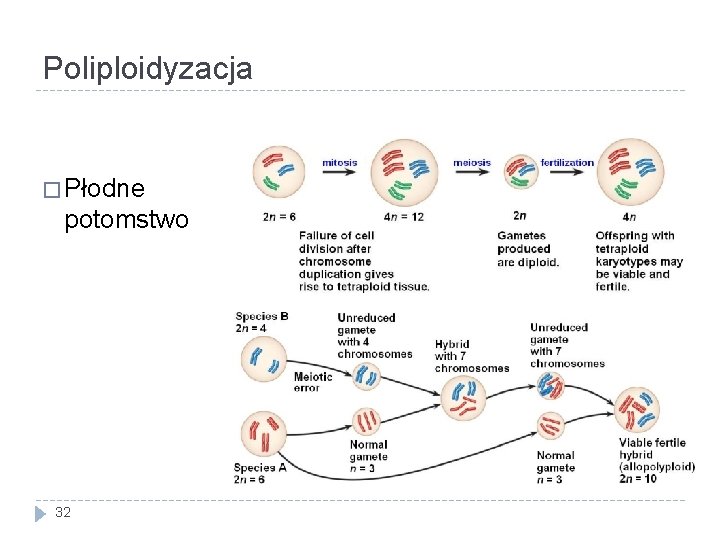
Poliploidyzacja � Płodne potomstwo 32

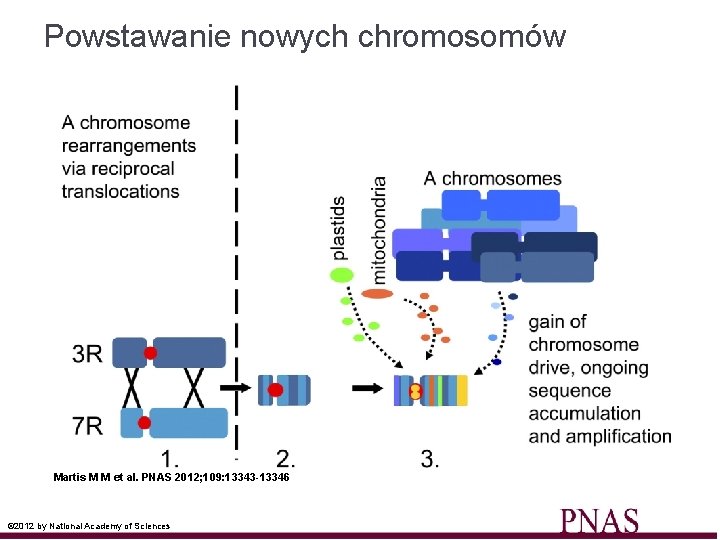
Powstawanie nowych chromosomów Martis M M et al. PNAS 2012; 109: 13343 -13346 © 2012 by National Academy of Sciences