Transkrypcja genw jdrowych u rolin i jej regulacja














- Slides: 60

Transkrypcja genów jądrowych u roślin i jej regulacja

Maszyneria transkrypcyjna - prokarionty • Mechanizmy regulacji ekspresji genów różnią się zasadniczo u eukariontów i prokariontów. • U prokariontów stan podstawowy dla transkrypcji jest nierestrykcyjny (brak ograniczenia dostępności do DNA dla kompleksu RNA polimerazy). Negatywna regulacja jest rzadka i zależy od represorów specyficznych dla konkretnych sekwencji. • U prokariontów sieć regulatorowa genów ma niską złożoność. Pojedynczy TF reguluje średnio 3 geny, a pojedynczy gen jest pod kontrolą średnio dwóch TF. Wiele promotorów regulowanych jest przez pojedynczy regulator. Regulatory te rzadko regulują transkrypcję innych TF. • U prokariontów, wiążące się z DNA, specyficzne sekwencyjnie TF na ogół rozpoznają długie sekwencje (>12 par zasad).

Maszyneria transkrypcyjna - eukarionty • • • U eukariontów stan podstawowy dla transkrypcji jest restrykcyjny, co wynika z upakowania DNA w chromatynę, która uniemożliwia rozpoznawania standardowych promotorów przez podstawową maszynerie transkrypcyjną. Wpływ struktury chromatynowej promotora na jego dostępność czyni niezbędnym udział w regulacji transkrypcji czynników modyfikujących chromatynę. Określa to w zasadniczy sposób model regulacji transkrypcji u eukariontów. W systemie regulacji uczestniczą nie tylko składniki podstawowej maszynerii transkrypcyjnej i wielka liczba TF wiążących się ze specyficznymi sekwencjami DNA, ale także bardzo liczne i rozmaite białka związane z chromatyną. U eukariontów regulatory transkrypcji działają według logiki kombinatorycznej, co skutecznie zwiększa liczbę i różnorodność aktywności regulatorowych i prowadzi do dużej złożoności sieci regulacyjnych. Sekwencje rozpoznawane przez eukariotyczne TF mają długość 5 -10 par zasad.

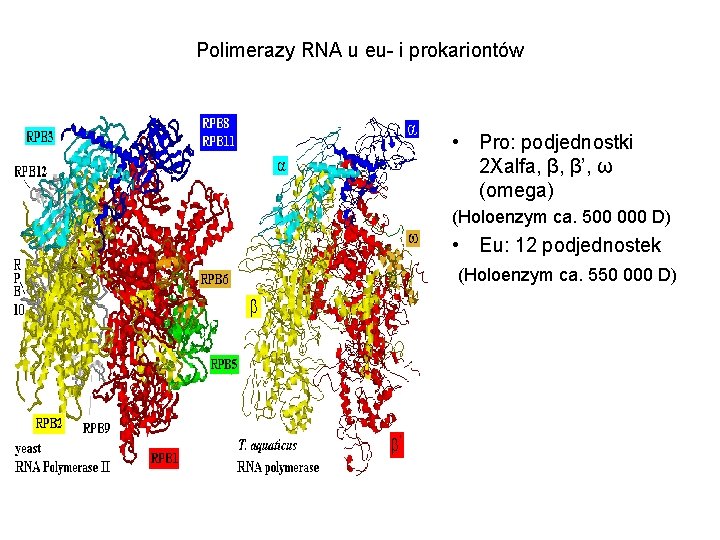
Polimerazy RNA u eu- i prokariontów • Pro: podjednostki 2 Xalfa, β, β’, ω (omega) (Holoenzym ca. 500 000 D) • Eu: 12 podjednostek (Holoenzym ca. 550 000 D)

Promotor prokariotyczny • Obejmuje dwie podstawowe sekwencje zaangażowane w kontrolę transkrypcji: TATAAT ( -10 pz) i TTGACA (-35 pz)

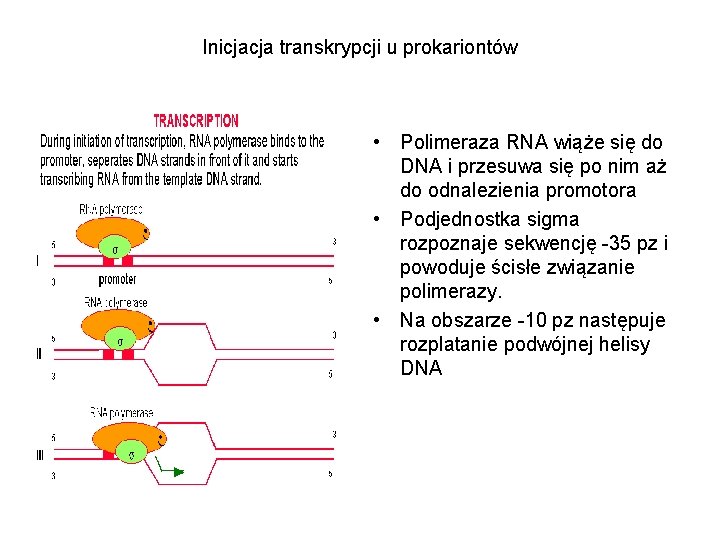
Inicjacja transkrypcji u prokariontów • Polimeraza RNA wiąże się do DNA i przesuwa się po nim aż do odnalezienia promotora • Podjednostka sigma rozpoznaje sekwencję -35 pz i powoduje ścisłe związanie polimerazy. • Na obszarze -10 pz następuje rozplatanie podwójnej helisy DNA

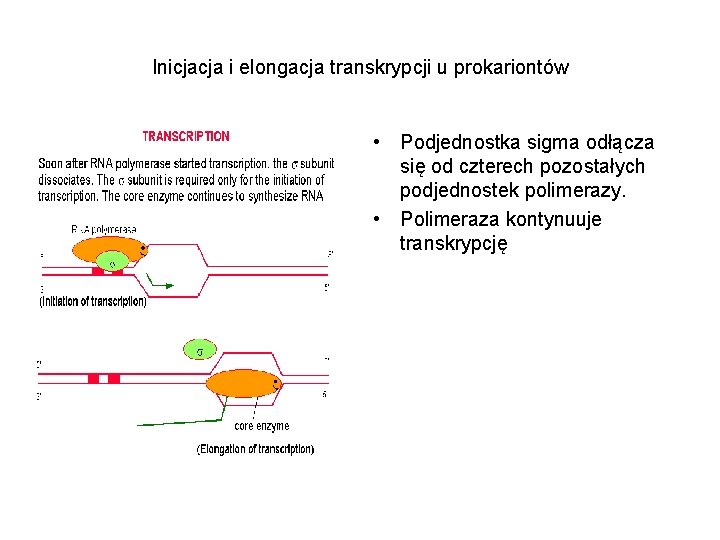
Inicjacja i elongacja transkrypcji u prokariontów • Podjednostka sigma odłącza się od czterech pozostałych podjednostek polimerazy. • Polimeraza kontynuuje transkrypcję

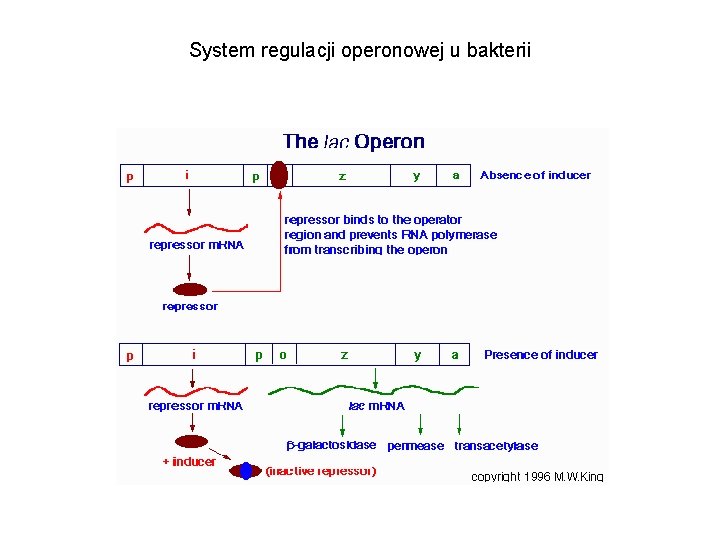
System regulacji operonowej u bakterii

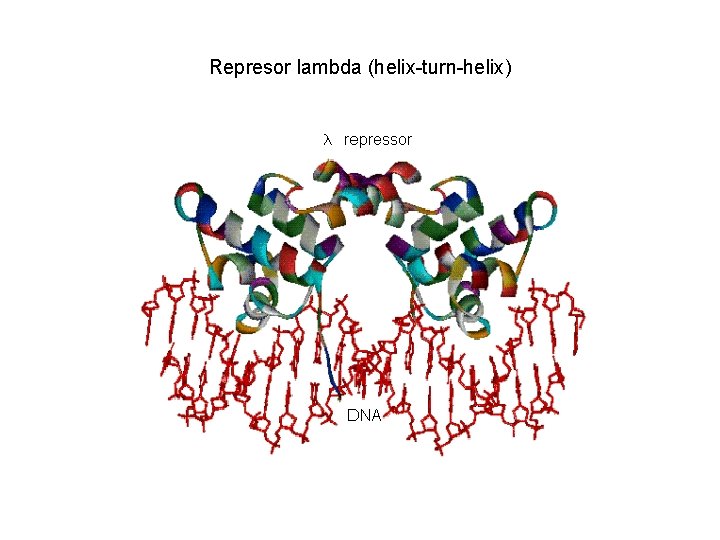
Represor lambda (helix-turn-helix)

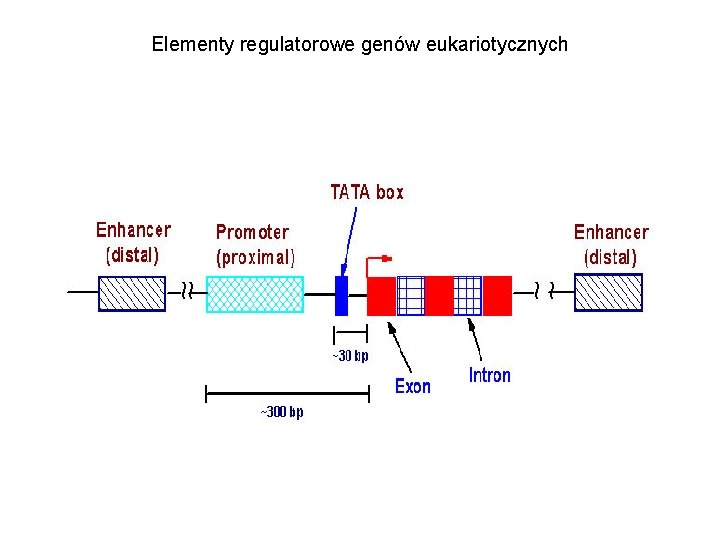
Elementy regulatorowe genów eukariotycznych

Białka związane z transkrypcją u eukariontów należą do 4 zróżnicowanych funkcjonalnie grup • 1. Podstawowy aparat transkrypcyjny i związane z nim ogólne czynniki transkrypcyjne (GTF - General Transcription Factors) • 2. Specyficzne w stosunku do sekwencji, wiążące się z DNA czynniki transkrypcyjne (TF). • 3. Duże wielo-podjednostkowe kompleksy koaktywatorów i innych kofaktorów. • 4. Białka związane z chromatyną

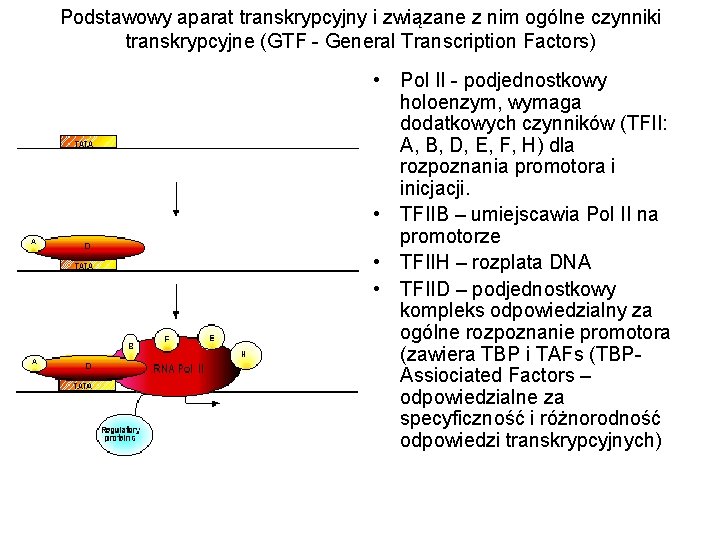
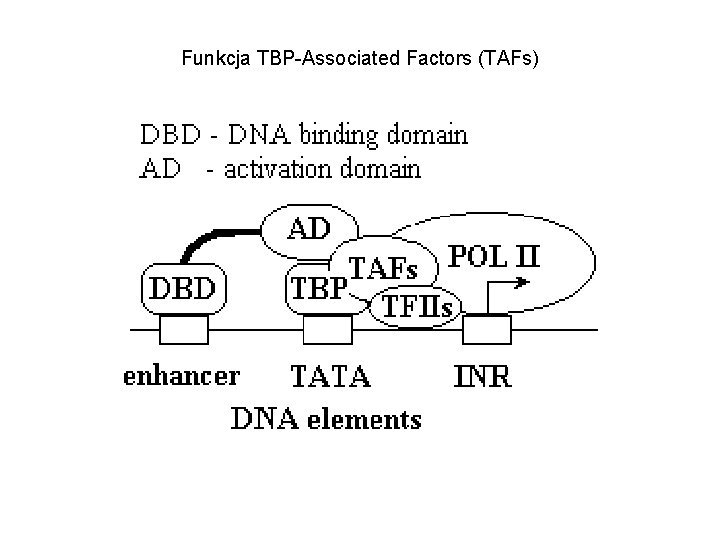
Podstawowy aparat transkrypcyjny i związane z nim ogólne czynniki transkrypcyjne (GTF - General Transcription Factors) • Pol II - podjednostkowy holoenzym, wymaga dodatkowych czynników (TFII: A, B, D, E, F, H) dla rozpoznania promotora i inicjacji. • TFIIB – umiejscawia Pol II na promotorze • TFIIH – rozplata DNA • TFIID – podjednostkowy kompleks odpowiedzialny za ogólne rozpoznanie promotora (zawiera TBP i TAFs (TBPAssiociated Factors – odpowiedzialne za specyficzność i różnorodność odpowiedzi transkrypcyjnych)

Funkcja TBP-Associated Factors (TAFs)

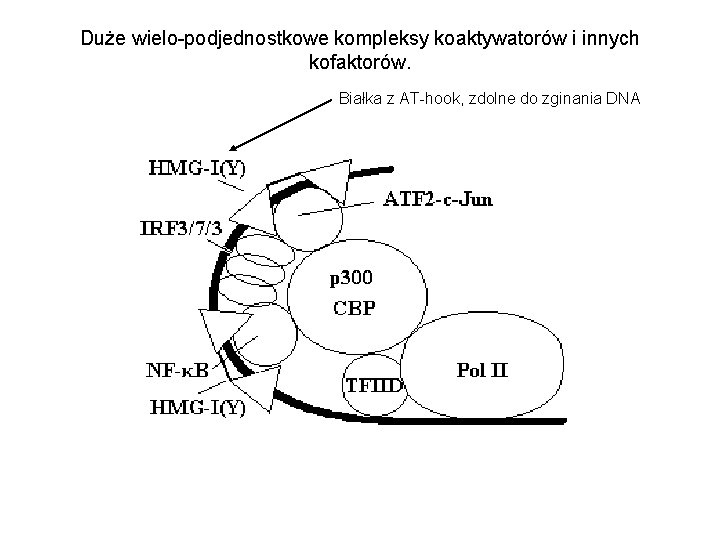
Duże wielo-podjednostkowe kompleksy koaktywatorów i innych kofaktorów. Białka z AT-hook, zdolne do zginania DNA

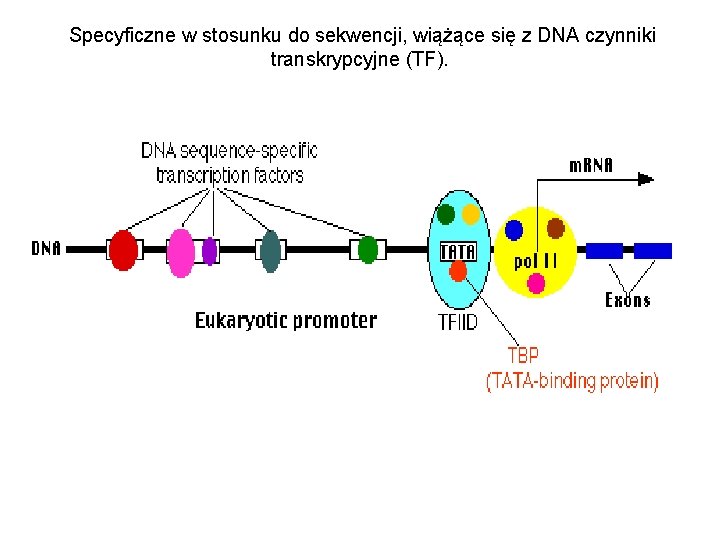
Specyficzne w stosunku do sekwencji, wiążące się z DNA czynniki transkrypcyjne (TF).

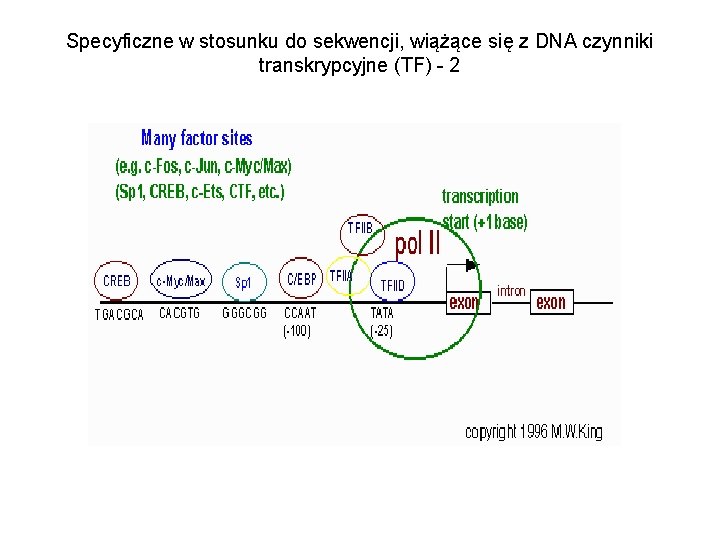
Specyficzne w stosunku do sekwencji, wiążące się z DNA czynniki transkrypcyjne (TF) - 2

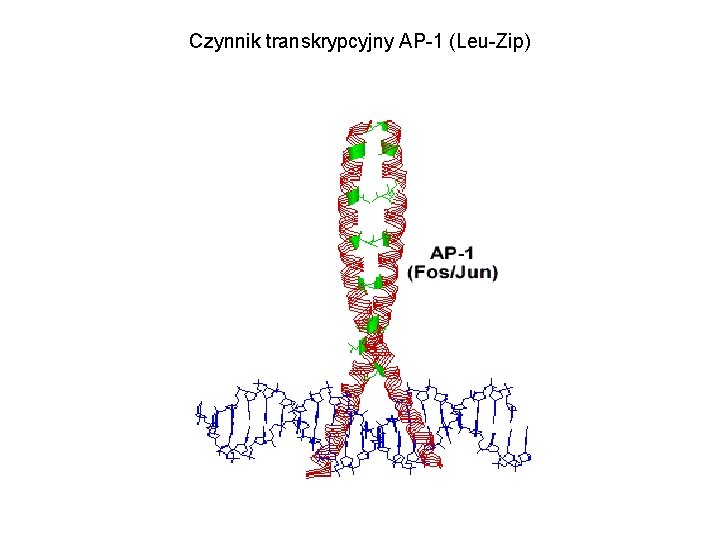
Czynnik transkrypcyjny AP-1 (Leu-Zip)

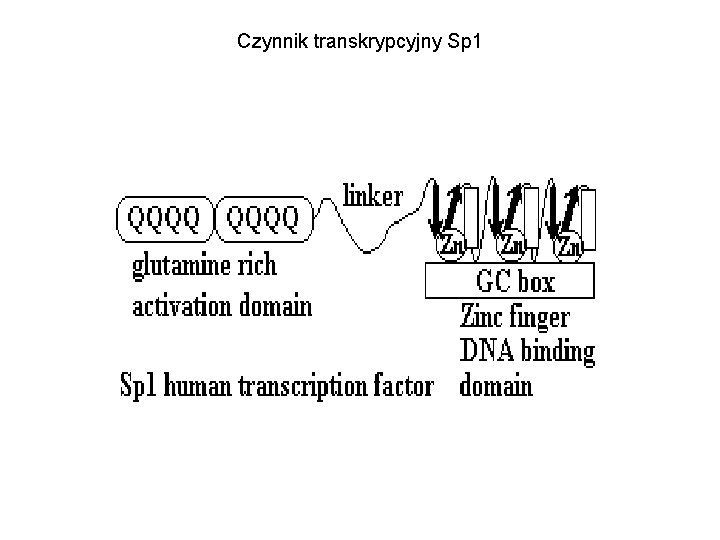
Czynnik transkrypcyjny Sp 1

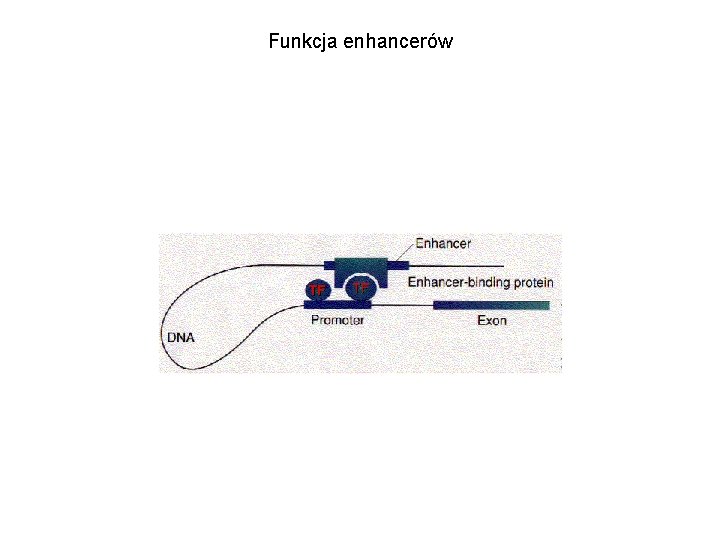
Funkcja enhancerów

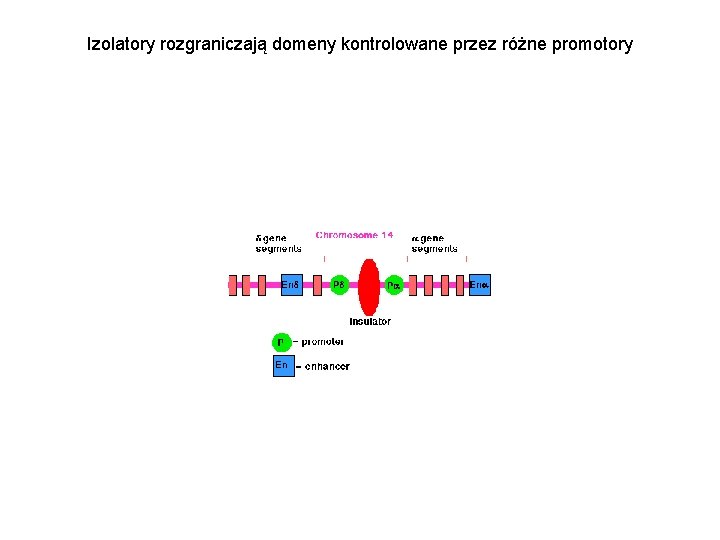
Izolatory rozgraniczają domeny kontrolowane przez różne promotory

Meyerowitz 2002

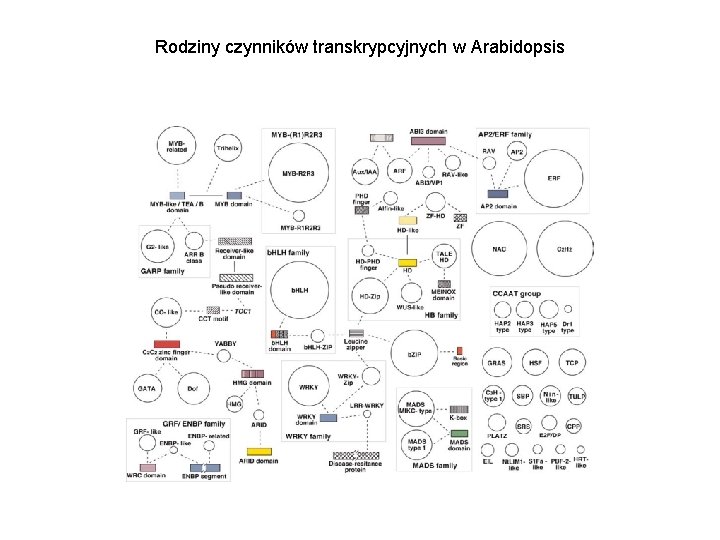
Rodziny czynników transkrypcyjnych w Arabidopsis

Rodziny Homeobox (HB) i Zinc-Finger-Homeobox (ZF-HB) w Arabidopsis • Rodzina Homeobox w Arabidopsis zawiera klasy z różnymi kombinacjami domen białkowych, różnice wynikają też z fiologenezy domeny HB. • Specyficzny układ domen (leucine zipper, PHD finger, STAR) wynika z ich mieszania charakterystycznego dla roślin, nie występuje w innych królestwach (Drosophila, C. elegans, drożdże). • Białka ZF-HB mają specyficzny tylko dla roślin motyw koordynujący cynk.

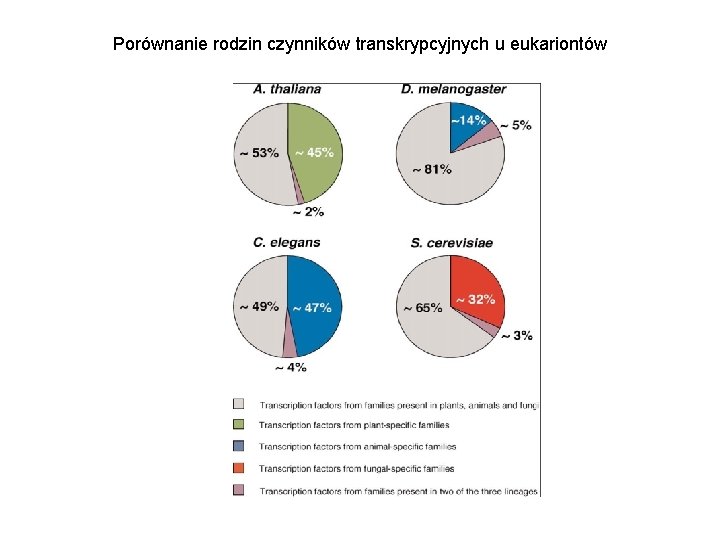
Porównanie rodzin czynników transkrypcyjnych u eukariontów

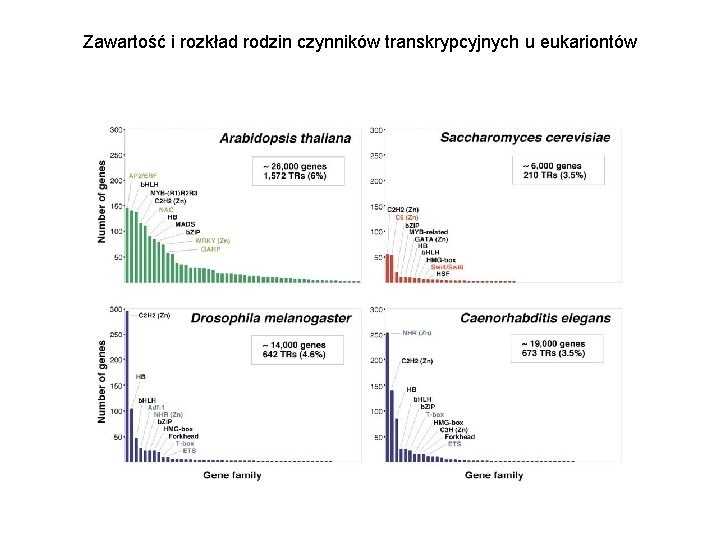
Zawartość i rozkład rodzin czynników transkrypcyjnych u eukariontów

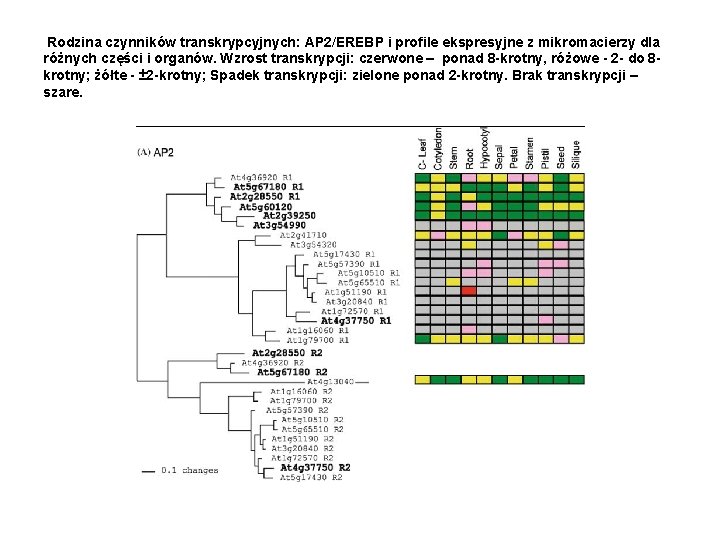
Rodzina czynników transkrypcyjnych: AP 2/EREBP i profile ekspresyjne z mikromacierzy dla różnych części i organów. Wzrost transkrypcji: czerwone – ponad 8 -krotny, różowe - 2 - do 8 krotny; żółte - ± 2 -krotny; Spadek transkrypcji: zielone ponad 2 -krotny. Brak transkrypcji – szare.


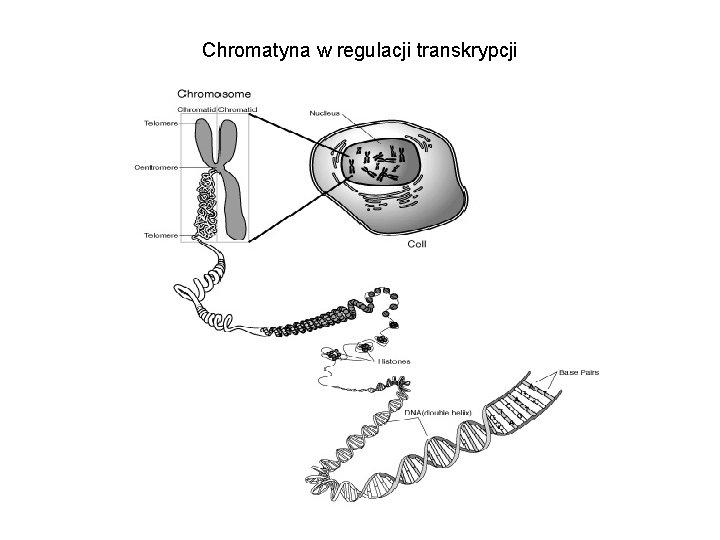
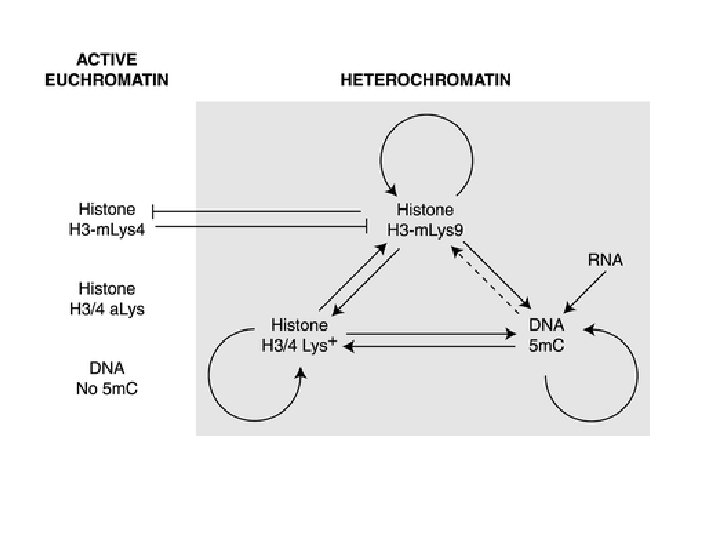
Chromatyna w regulacji transkrypcji




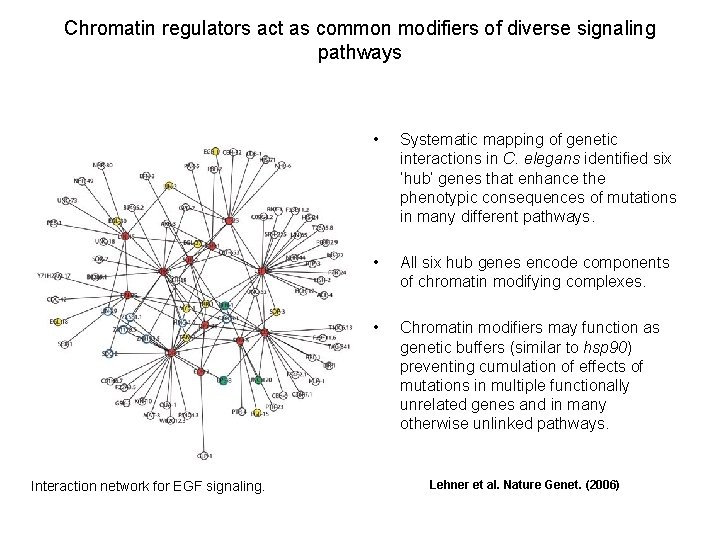
Chromatin regulators act as common modifiers of diverse signaling pathways Interaction network for EGF signaling. • Systematic mapping of genetic interactions in C. elegans identified six ‘hub’ genes that enhance the phenotypic consequences of mutations in many different pathways. • All six hub genes encode components of chromatin modifying complexes. • Chromatin modifiers may function as genetic buffers (similar to hsp 90) preventing cumulation of effects of mutations in multiple functionally unrelated genes and in many otherwise unlinked pathways. Lehner et al. Nature Genet. (2006)

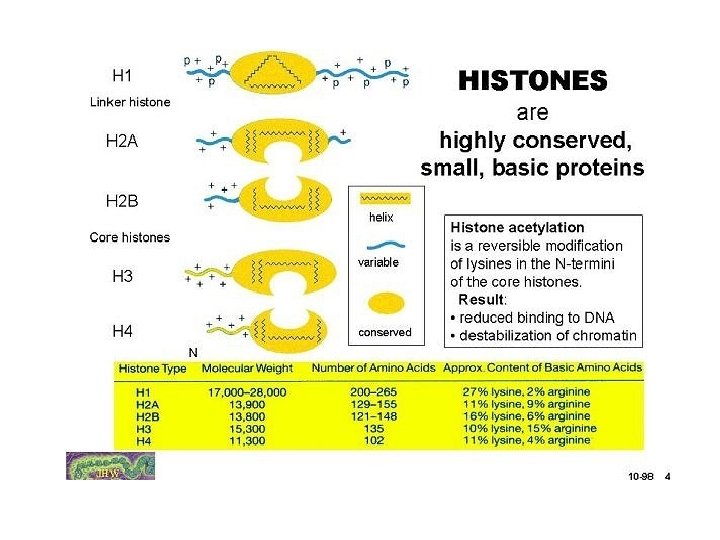
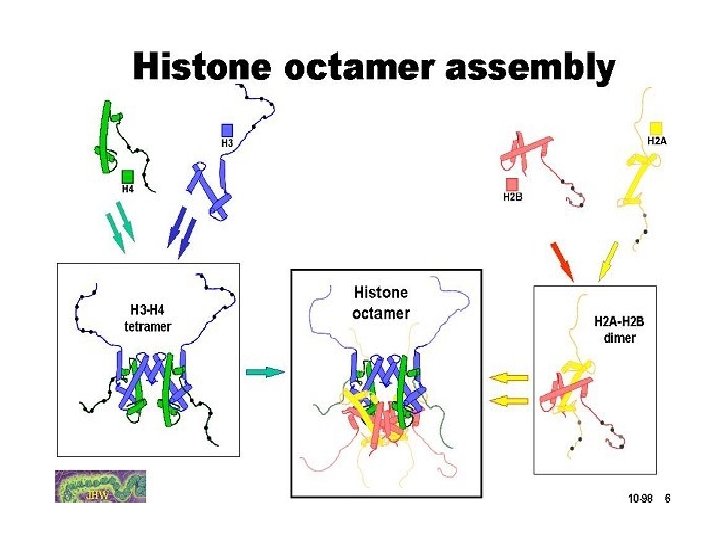
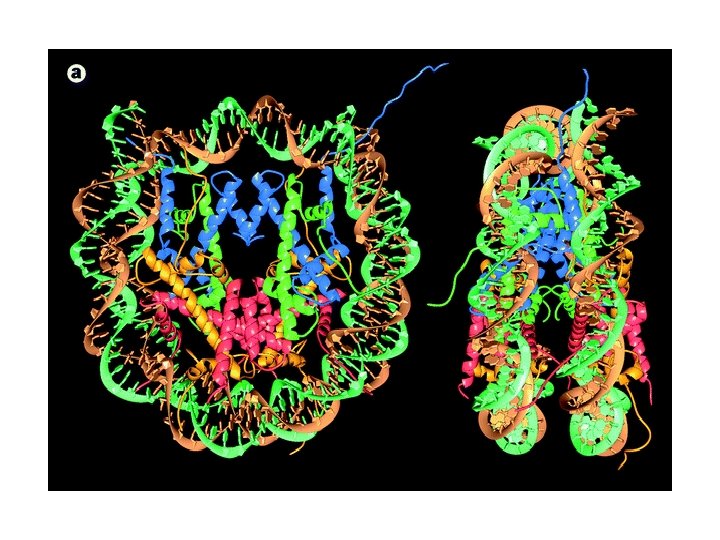
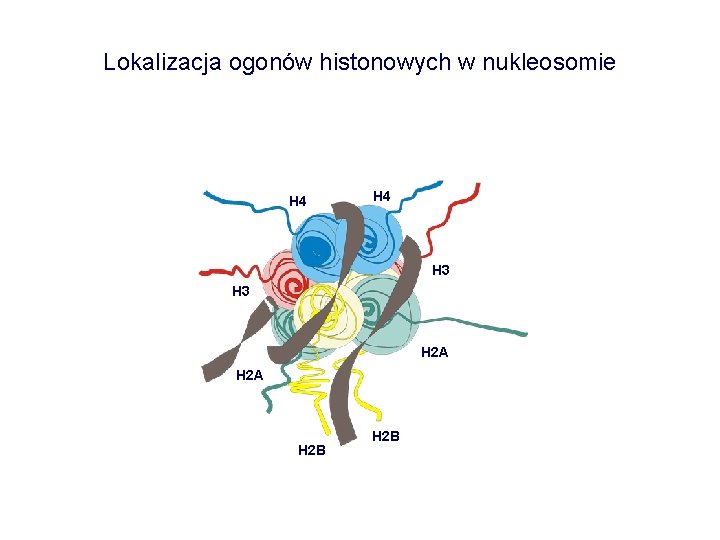
Lokalizacja ogonów histonowych w nukleosomie H 4 H 3 H 2 A H 2 B

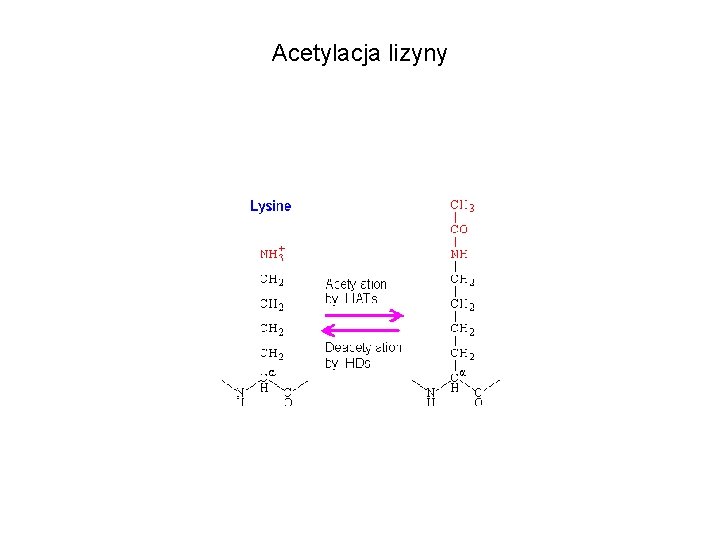
Acetylacja lizyny

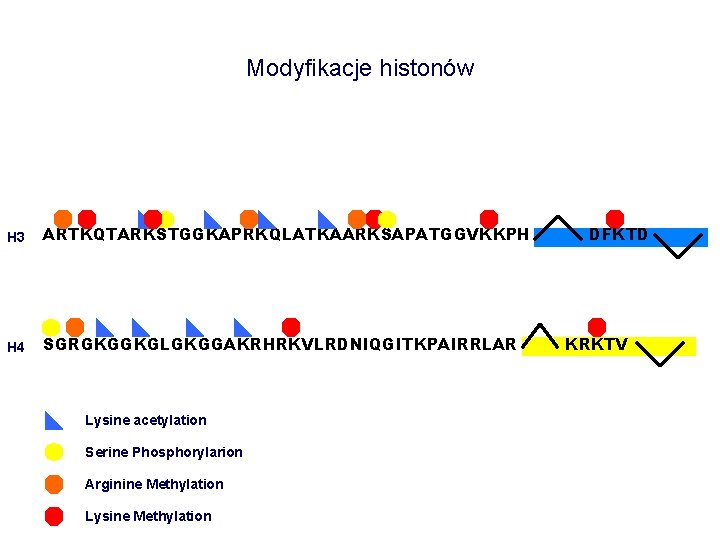
Modyfikacje histonów H 3 ARTKQTARKSTGGKAPRKQLATKAARKSAPATGGVKKPH H 4 SGRGKGGKGLGKGGAKRHRKVLRDNIQGITKPAIRRLAR Lysine acetylation Serine Phosphorylarion Arginine Methylation Lysine Methylation DFKTD KRKTV

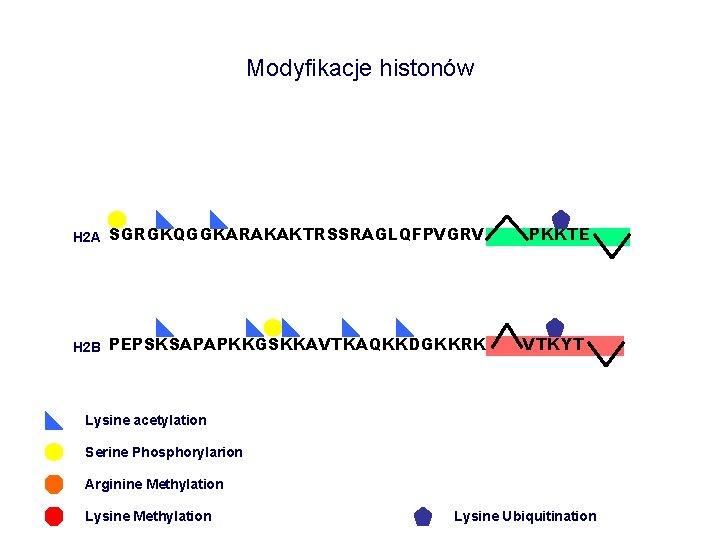
Modyfikacje histonów H 2 A SGRGKQGGKARAKAKTRSSRAGLQFPVGRV H 2 B PEPSKSAPAPKKGSKKAVTKAQKKDGKKRK PKKTE VTKYT Lysine acetylation Serine Phosphorylarion Arginine Methylation Lysine Ubiquitination

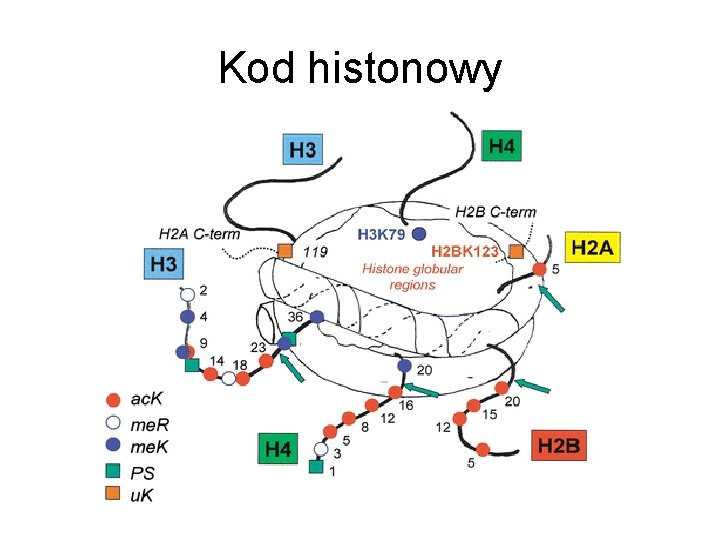
Kod histonowy

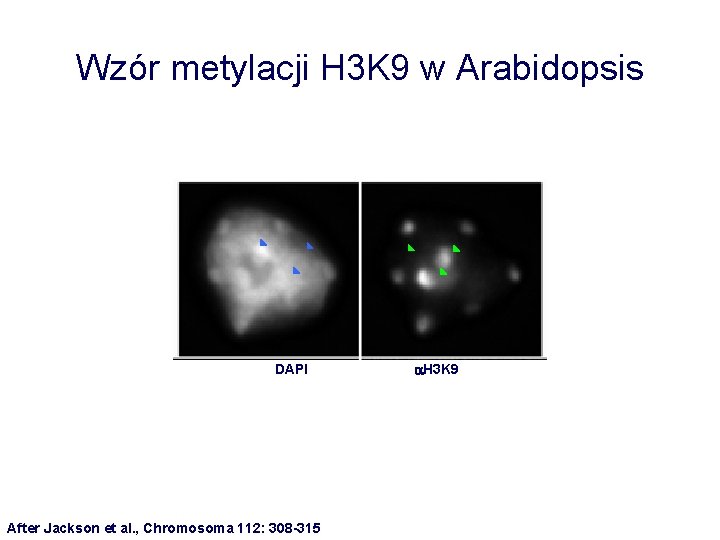
Wzór metylacji H 3 K 9 w Arabidopsis DAPI After Jackson et al. , Chromosoma 112: 308 -315 a. H 3 K 9

Analiza odpowiedzi tytoniowych komórek BY-2 na 250 m. M Na. Cl 0 5 10 15 20 30 45 60 90 120 min. anty-fosfo(S 10)-H 3 0 0 5 10 15 20 30 45 60 90 120 min. anty-fosfo(S 10)acetyl(K 14)-H 3 0 0 5 10 15 20 30 45 60 90 min. 5 10 15 20 30 45 60 90 120 min. anty-acetyl-H 4 0 min. 0 15 30 45 60 90 120 5 10 15 20 30 45 60 90 min. 3% aktyna Tsi 1 Nt. C 7 osmotyna intensywność sygnału western-blot, wyrażonaw jednostkach umownych, przypadająca na ilość białka

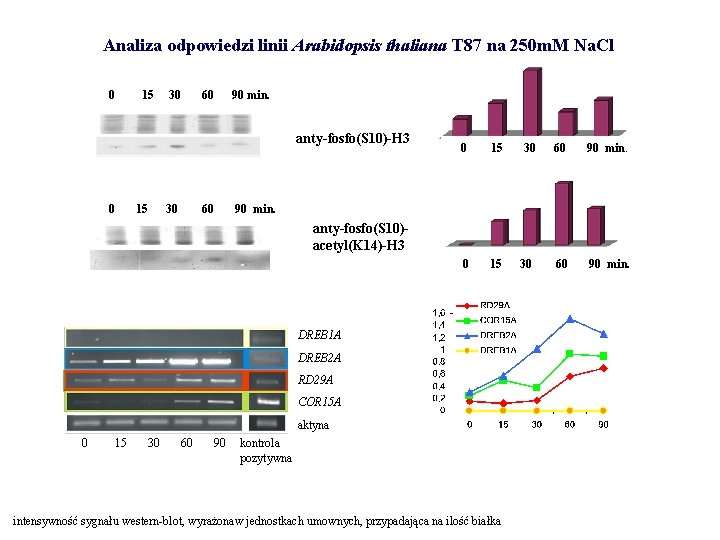
Analiza odpowiedzi linii Arabidopsis thaliana T 87 na 250 m. M Na. Cl 0 15 30 60 90 min. anty-fosfo(S 10)-H 3 0 15 30 60 90 min. anty-fosfo(S 10)acetyl(K 14)-H 3 DREB 1 A DREB 2 A RD 29 A COR 15 A aktyna 0 15 30 60 90 kontrola pozytywna intensywność sygnału western-blot, wyrażonaw jednostkach umownych, przypadająca na ilość białka 30

Metylacja cytozyn w DNA • • Reaction: Cytosine → 5 -methylcytosine (5 m. C) Enzymes: Diverse group of DNA methyltransferases (Dnmt’s) Sequence context: Cp. G – animals Cp. G (major), Cp. Np. G, Cp. Np - plants

ATP dependent Chromatin Remodeling Kingston, R. E. , Narlikar, G. J. Genes&Development 13: 2339 -2352(1999)

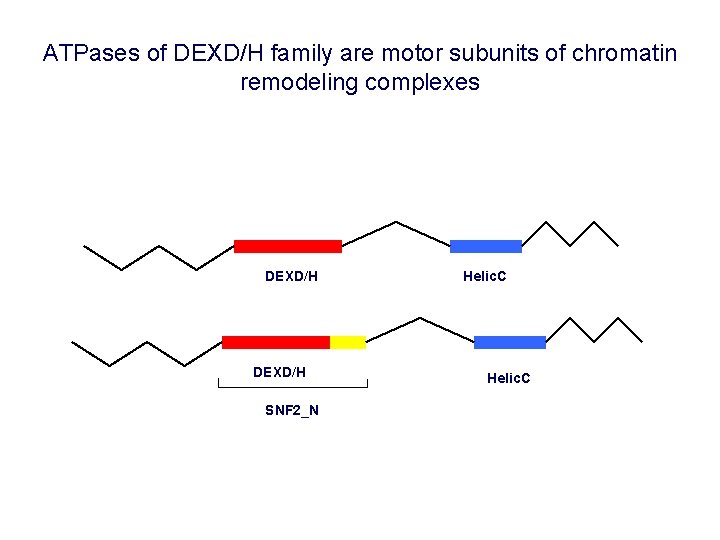
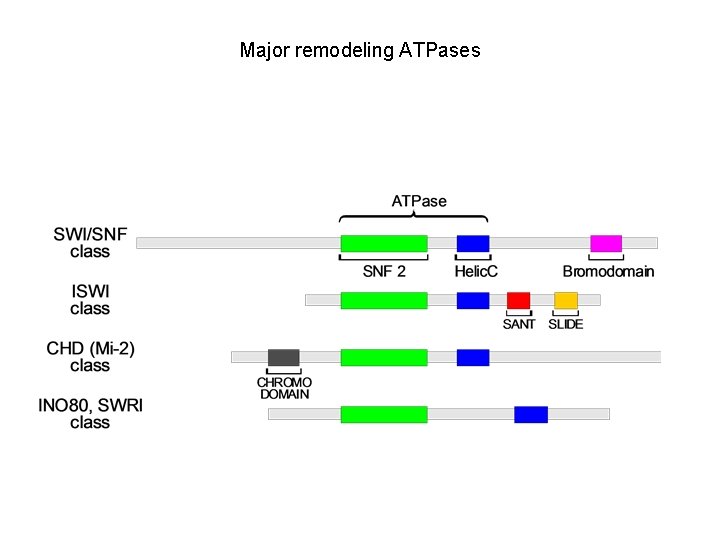
ATPases of DEXD/H family are motor subunits of chromatin remodeling complexes DEXD/H SNF 2_N Helic. C

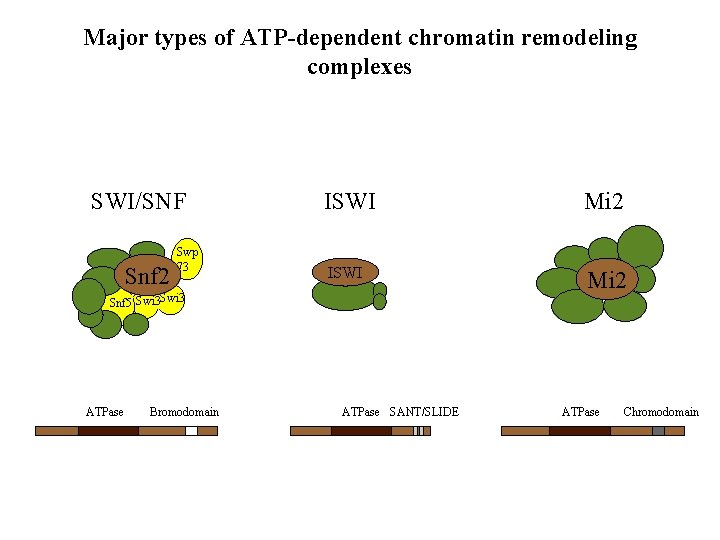
Major types of ATP-dependent chromatin remodeling complexes SWI/SNF Snf 2 Swp 73 ISWI Snf 5 Swi 3 ATPase Bromodomain ATPase SANT/SLIDE Mi 2 ATPase Chromodomain

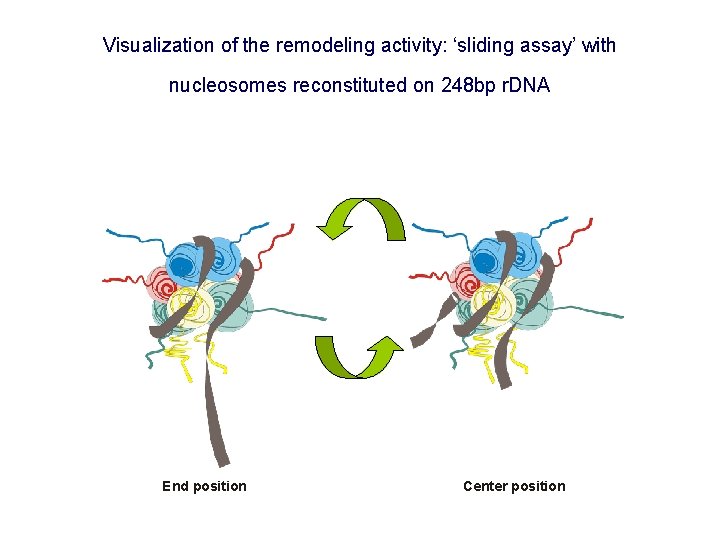
Visualization of the remodeling activity: ‘sliding assay’ with nucleosomes reconstituted on 248 bp r. DNA End position Center position

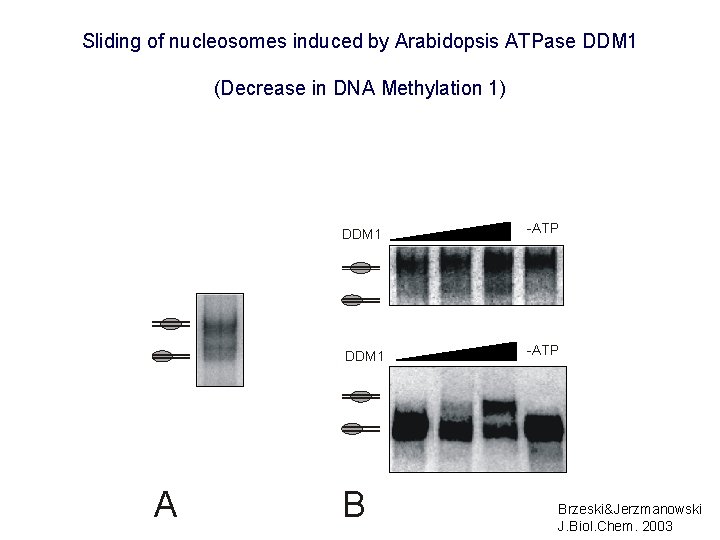
Sliding of nucleosomes induced by Arabidopsis ATPase DDM 1 (Decrease in DNA Methylation 1) A DDM 1 -ATP B Brzeski&Jerzmanowski J. Biol. Chem. 2003

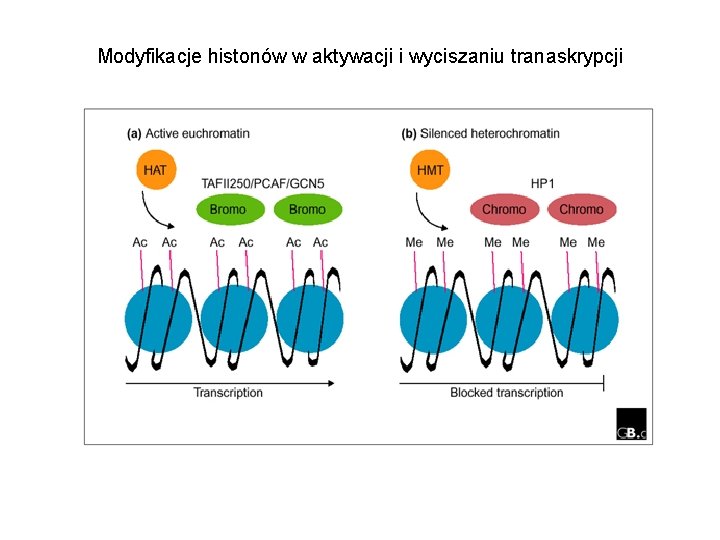
Modyfikacje histonów w aktywacji i wyciszaniu tranaskrypcji

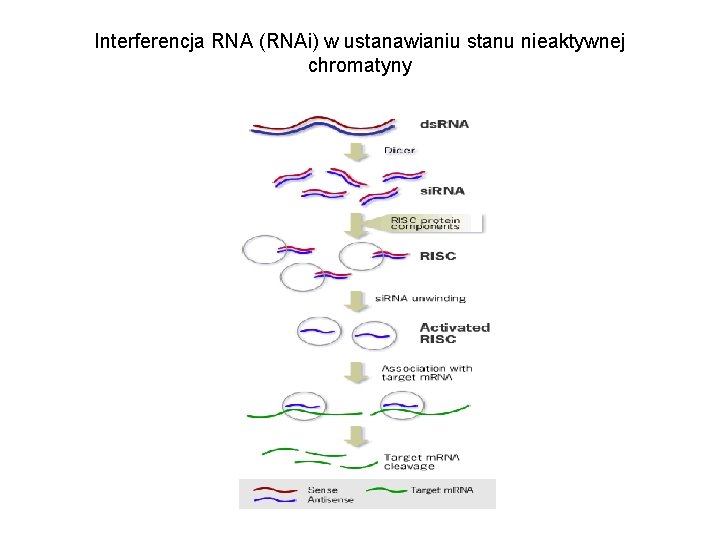
Interferencja RNA (RNAi) w ustanawianiu stanu nieaktywnej chromatyny


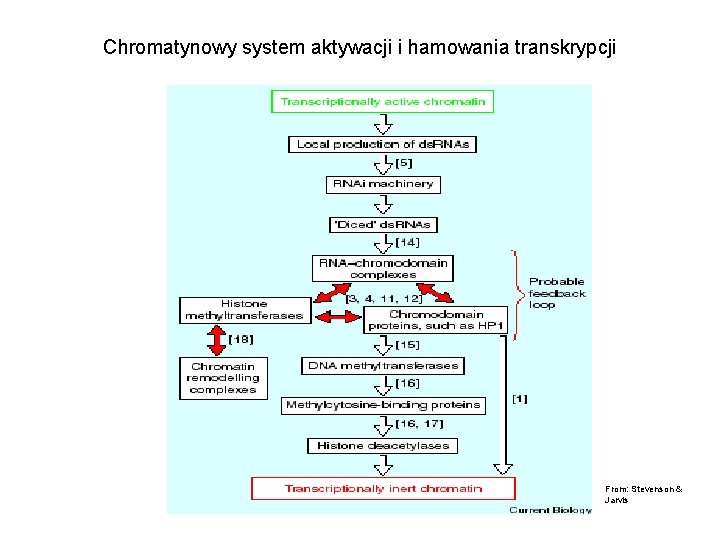
Chromatynowy system aktywacji i hamowania transkrypcji From: Stevenson & Jarvis

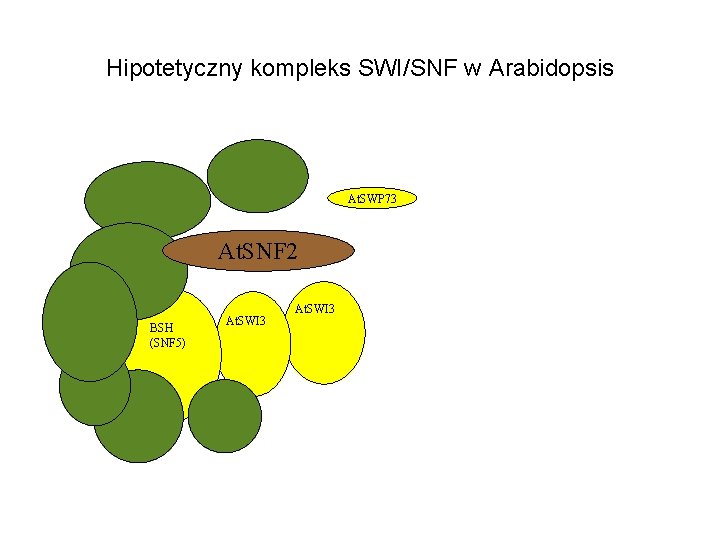
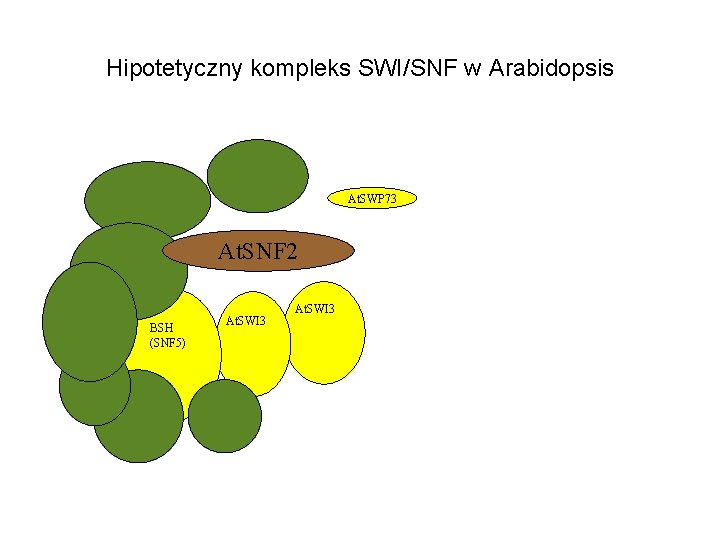
Hipotetyczny kompleks SWI/SNF w Arabidopsis At. SWP 73 At. SNF 2 BSH (SNF 5) At. SWI 3

Major remodeling ATPases

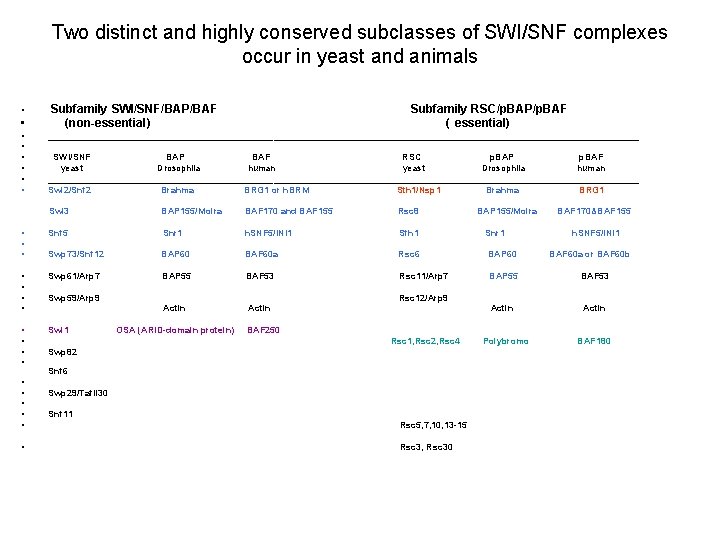
Two distinct and highly conserved subclasses of SWI/SNF complexes occur in yeast and animals • • Subfamily SWI/SNF/BAP/BAF (non-essential) Subfamily RSC/p. BAP/p. BAF ( essential) ___________________________________________________________ SWI/SNF BAP BAF RSC p. BAP p. BAF yeast Drosophila human ___________________________________________________________ Swi 2/Snf 2 Brahma BRG 1 or h. BRM Sth 1/Nsp 1 Brahma BRG 1 Swi 3 BAP 155/Moira BAF 170 and BAF 155 Rsc 8 • • • Snf 5 Snr 1 h. SNF 5/INI 1 Sfh 1 Swp 73/Snf 12 BAP 60 BAF 60 a Rsc 6 BAP 60 • • Swp 61/Arp 7 BAP 55 BAF 53 Rsc 11/Arp 7 BAP 55 BAF 53 Actin • • Swi 1 • • • Swp 59/Arp 9 BAP 155/Moira Snr 1 BAF 170&BAF 155 h. SNF 5/INI 1 BAF 60 a or BAF 60 b Rsc 12/Arp 9 OSA (ARID-domain protein) BAF 250 Rsc 1, Rsc 2, Rsc 4 Swp 82 Snf 6 Swp 29/Taf. II 30 Snf 11 Rsc 5, 7, 10, 13 -15 Rsc 3, Rsc 30 Polybromo BAF 180

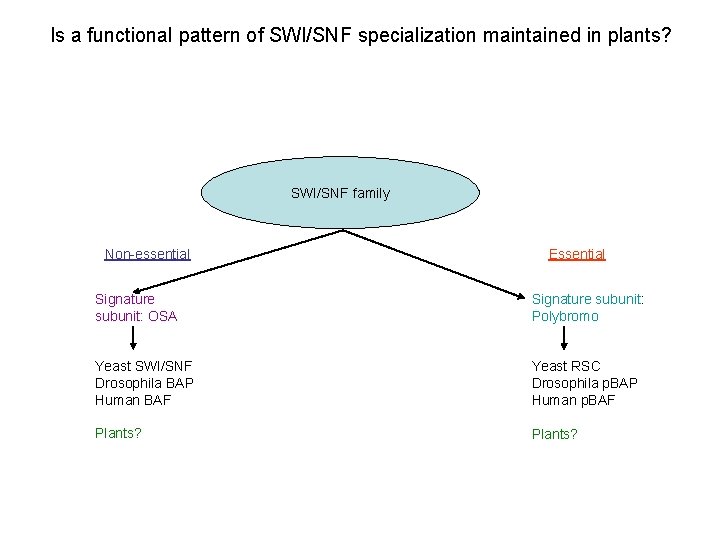
Is a functional pattern of SWI/SNF specialization maintained in plants? SWI/SNF family Non-essential Essential Signature subunit: OSA Signature subunit: Polybromo Yeast SWI/SNF Drosophila BAP Human BAF Yeast RSC Drosophila p. BAP Human p. BAF Plants?

Arabidopsis SWI/SNF complexes – the landscape of possibilities

Hypothetical organization of SWI/SNF remodeling in Arabidopsis

Hipotetyczny kompleks SWI/SNF w Arabidopsis At. SWP 73 At. SNF 2 BSH (SNF 5) At. SWI 3

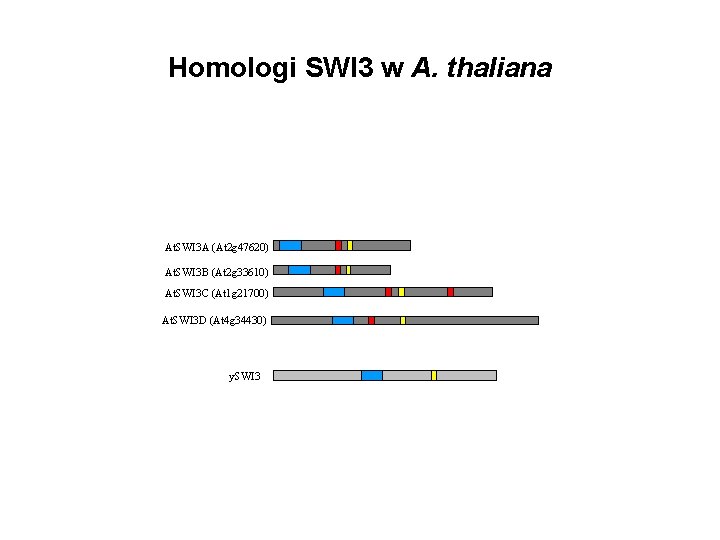
Homologi SWI 3 w A. thaliana At. SWI 3 A (At 2 g 47620) At. SWI 3 B (At 2 g 33610) At. SWI 3 C (At 1 g 21700) At. SWI 3 D (At 4 g 34430) y. SWI 3

Drzewo filogenetyczne białek typu SWI 3

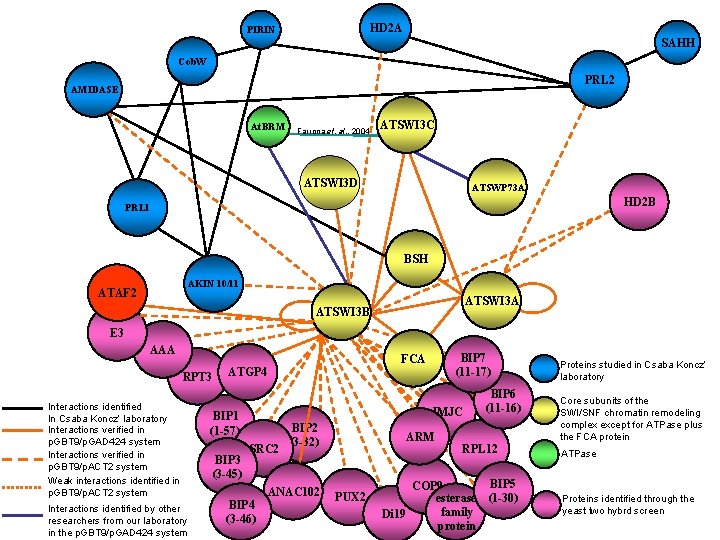
HD 2 A PIRIN SAHH Cob. W PRL 2 AMIDASE At. BRM Farrona et. al. , 2004 ATSWI 3 C ATSWI 3 D ATSWP 73 A HD 2 B PRL 1 BSH AKIN 10/11 ATAF 2 ATSWI 3 A ATSWI 3 B E 3 AAA BIP 7 (11 -17) FCA RPT 3 Interactions identified In Csaba Koncz’ laboratory Interactions verified in p. GBT 9/p. GAD 424 system Interactions verified in p. GBT 9/p. ACT 2 system Weak interactions identified in p. GBT 9/p. ACT 2 system Interactions identified by other researchers from our laboratory in the p. GBT 9/p. GAD 424 system ATGP 4 JMJC BIP 1 (1 -57) BIP 3 (3 -45) SRC 2 BIP 2 (3 -32) ANAC 102 BIP 4 (3 -46) ARM PUX 2 BIP 6 (11 -16) RPL 12 BIP 5 COP 9 esterase (1 -30) family Di 19 protein Proteins studied in Csaba Koncz’ laboratory Core subunits of the SWI/SNF chromatin remodeling complex except for ATPase plus the FCA protein ATPase Proteins identified through the yeast two hybrd screen
 Transkrypcja a transliteracja
Transkrypcja a transliteracja Regulacja dwupołożeniowa
Regulacja dwupołożeniowa Regulacja nerwowo-hormonalna prezentacja
Regulacja nerwowo-hormonalna prezentacja Regulacja dwupołożeniowa
Regulacja dwupołożeniowa Jan van eyck (1390-1441)
Jan van eyck (1390-1441) La vierge du chancelier rolin analyse
La vierge du chancelier rolin analyse La virgen del canciller rolin
La virgen del canciller rolin Kristina rolin
Kristina rolin Optysil intermag
Optysil intermag Rodina a jej funkcie
Rodina a jej funkcie Anioł pański
Anioł pański Charakteristika osoby kompozicia
Charakteristika osoby kompozicia Krzywa jotowata
Krzywa jotowata Quo vadis problematyka
Quo vadis problematyka Wiersz jana brzechwy zoo
Wiersz jana brzechwy zoo Neurodydaktyka i jej znaczenie w procesie kształcenia
Neurodydaktyka i jej znaczenie w procesie kształcenia Voda a jej skupenstva
Voda a jej skupenstva Chranime svoju obec a jej okolie
Chranime svoju obec a jej okolie Strój tadeusza soplicy
Strój tadeusza soplicy Wyznaczanie liczby gdy dany jest jej procent
Wyznaczanie liczby gdy dany jest jej procent Sumerske hymny
Sumerske hymny Neurodydaktyka definicja
Neurodydaktyka definicja Skloňovanie bicykel
Skloňovanie bicykel Funkcie rodiny
Funkcie rodiny žiarovka a jej objavenie
žiarovka a jej objavenie Mapa zeme
Mapa zeme Sylwia grzeszczak data i miejsce urodzenia
Sylwia grzeszczak data i miejsce urodzenia Bajka o wyspie
Bajka o wyspie Składanie sił
Składanie sił