Procarioti Eucarioti RNA m RNA RNA messaggero r














- Slides: 68



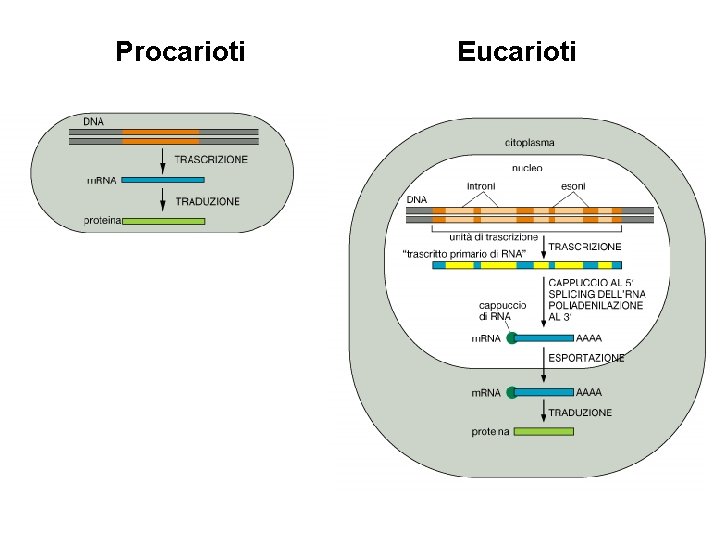
Procarioti Eucarioti




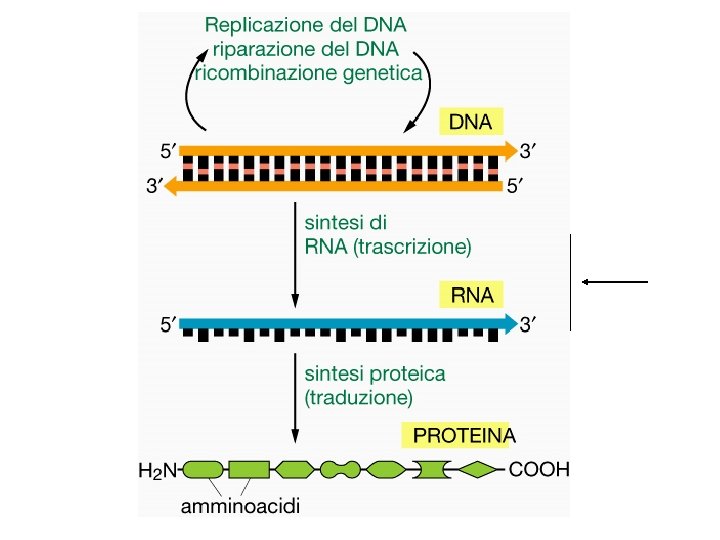
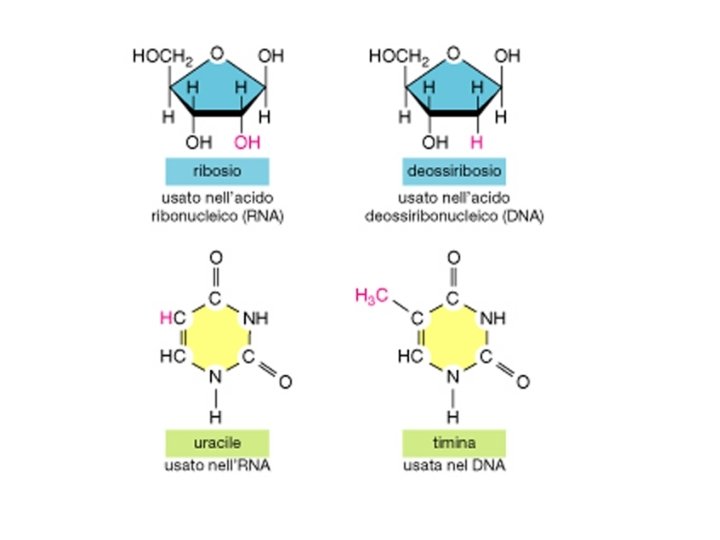
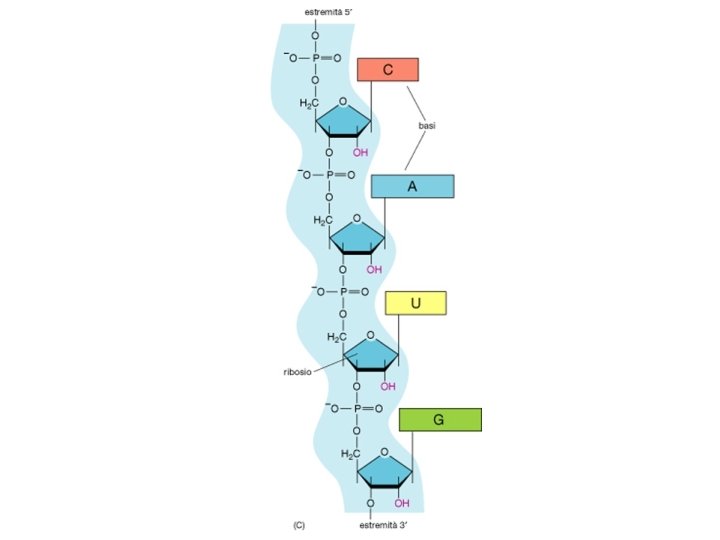
RNA • m. RNA -> RNA messaggero • r. RNA -> RNA ribosomale (piu’ abbondante) • t. RNA -> RNA transfer • sn. RNA -> piccoli RNA nucleari • micro. RNA -> regolazione • Altri

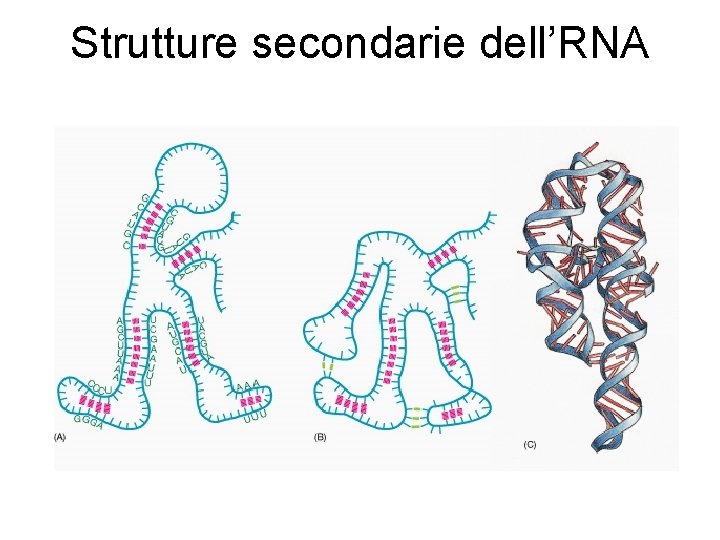
Strutture secondarie dell’RNA

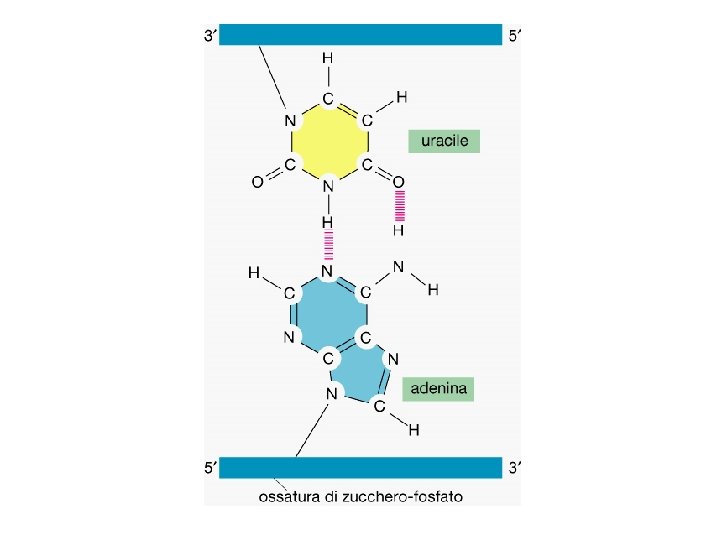
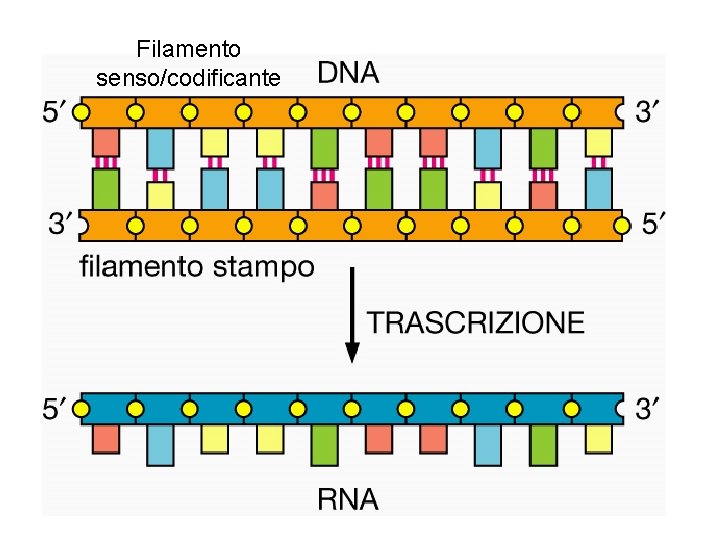
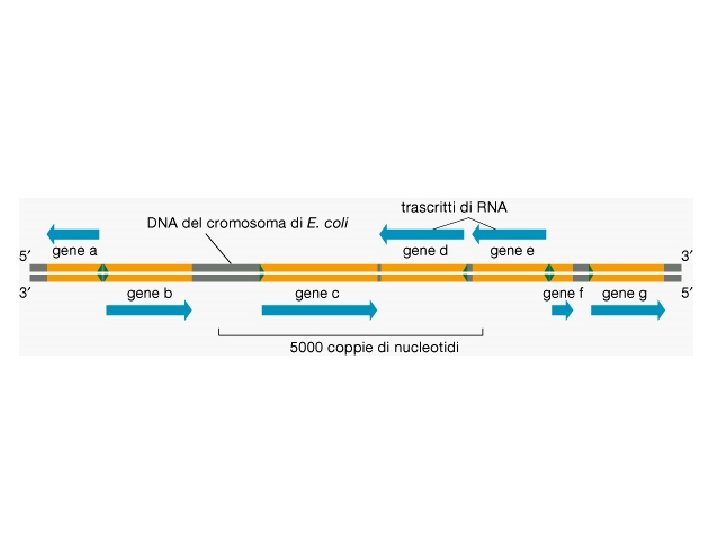
Filamento senso/codificante

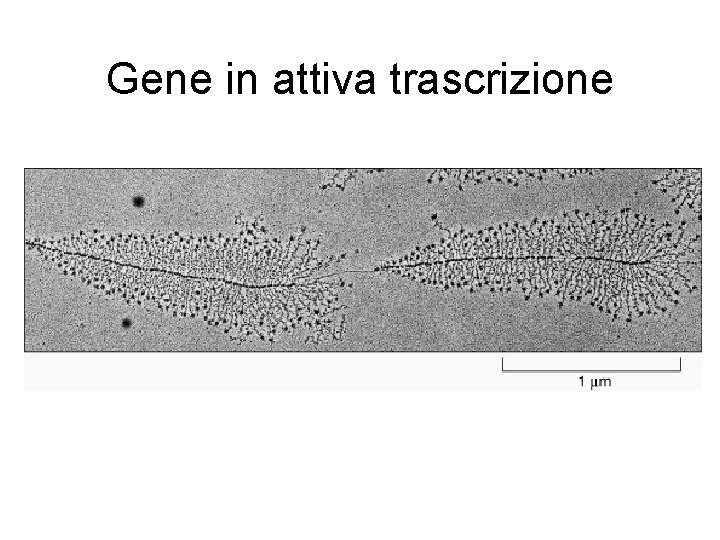
Gene in attiva trascrizione

The Nobel Prize in Chemistry 2006 "for his studies of the molecular basis of eukaryotic transcription" Photo: Stanford University Roger D. Kornberg Stanford University Stanford, CA, USA b. 1947 Arthur Kornberg Born 1918 Died 2007

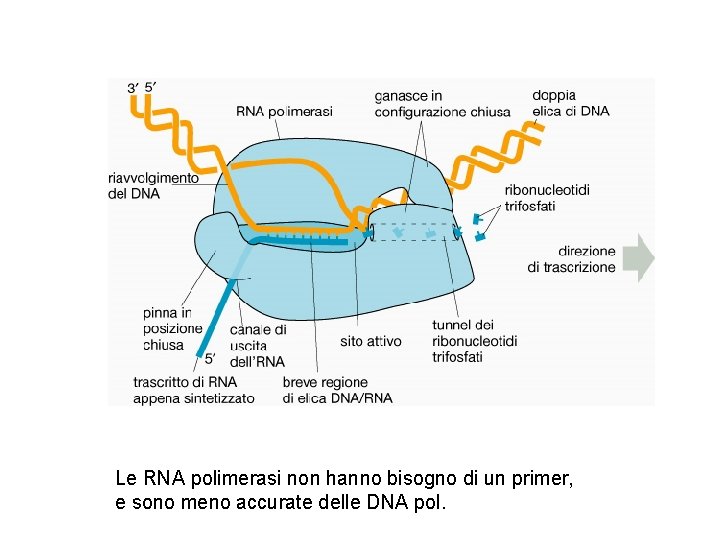
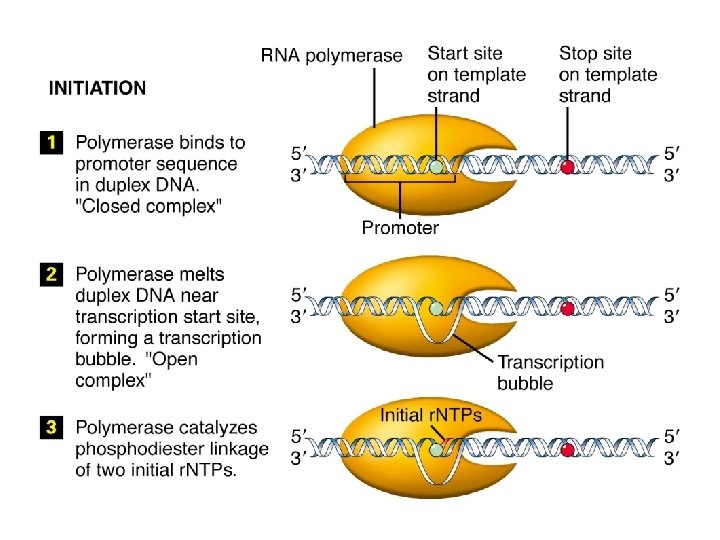
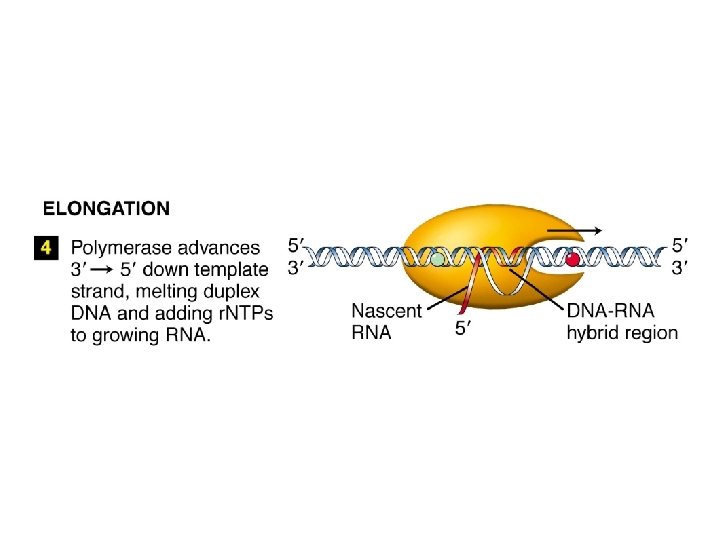
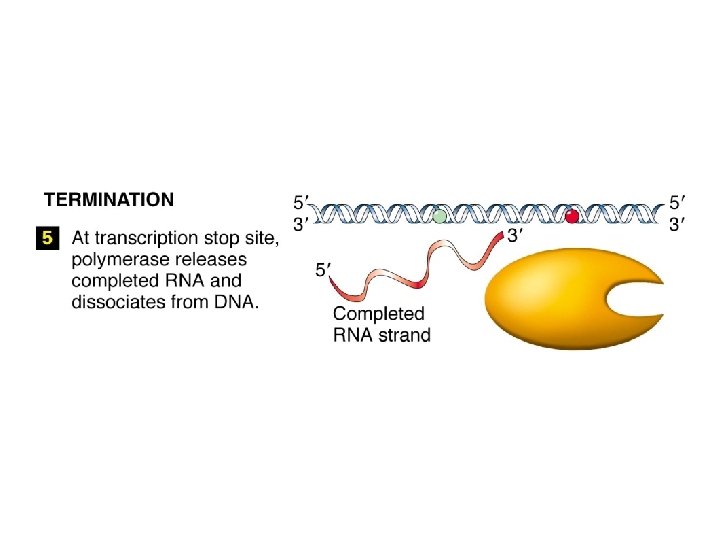
Le RNA polimerasi non hanno bisogno di un primer, e sono meno accurate delle DNA pol.

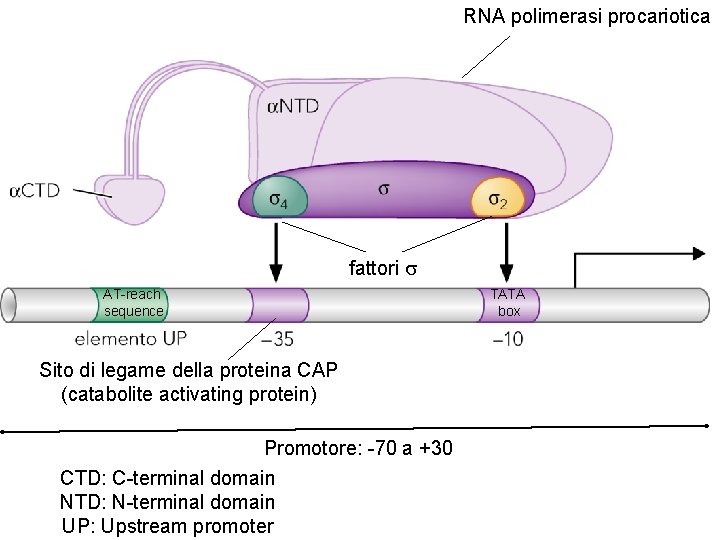
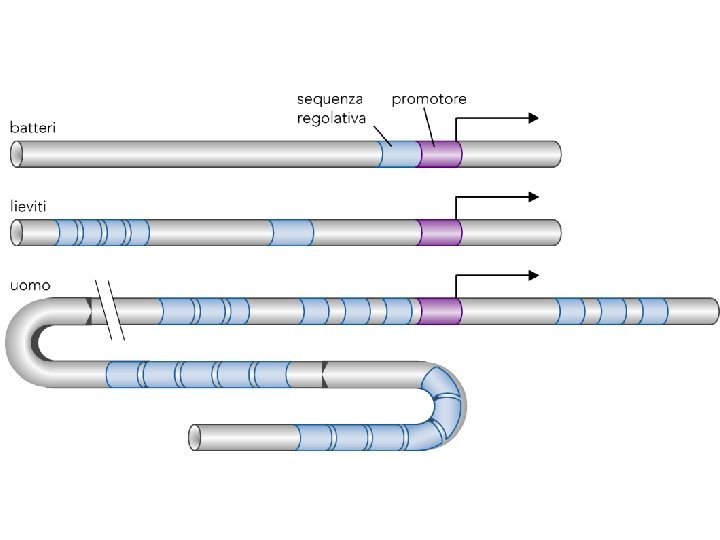
RNA polimerasi procariotica fattori s AT-reach sequence Sito di legame della proteina CAP (catabolite activating protein) Promotore: -70 a +30 CTD: C-terminal domain NTD: N-terminal domain UP: Upstream promoter TATA box



Trascrizione negli eucarioti


LE CELLULE EUCARIOTICHE HANNO 3 RNA POLIMERASI 1 --> r. RNA POLIMERASI 2 --> m. RNA POLIMERASI 3 --> t. RNA, sn. RNA,






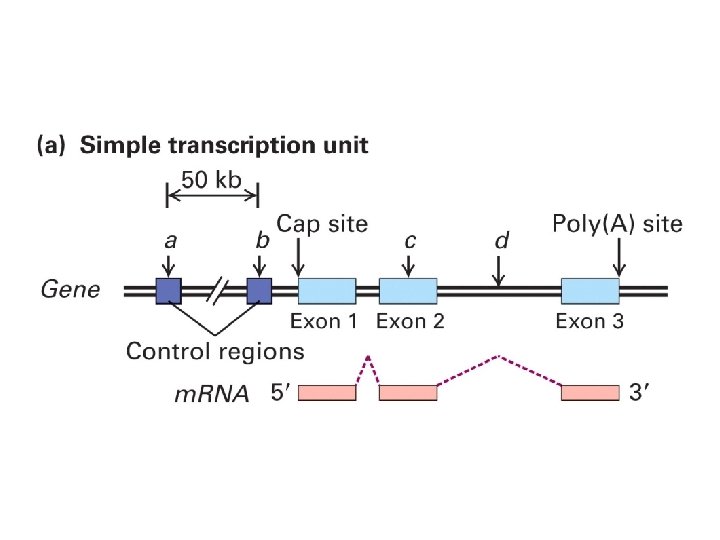
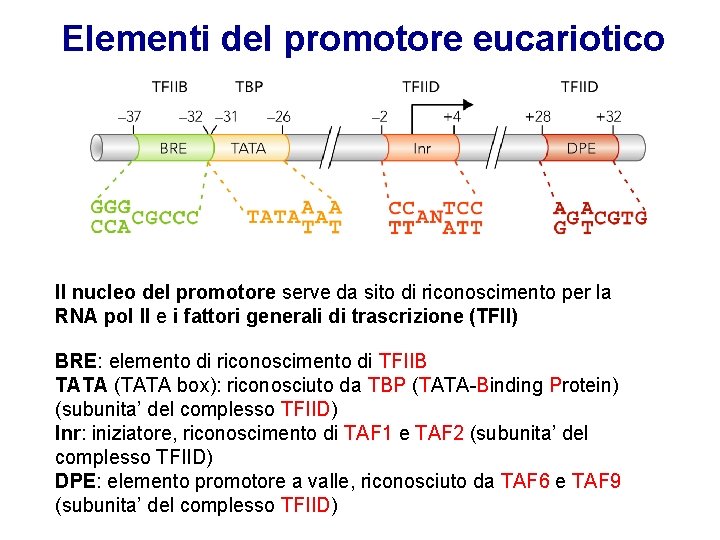
Elementi del promotore eucariotico Il nucleo del promotore serve da sito di riconoscimento per la RNA pol II e i fattori generali di trascrizione (TFII) BRE: elemento di riconoscimento di TFIIB TATA (TATA box): riconosciuto da TBP (TATA-Binding Protein) (subunita’ del complesso TFIID) Inr: iniziatore, riconoscimento di TAF 1 e TAF 2 (subunita’ del complesso TFIID) DPE: elemento promotore a valle, riconosciuto da TAF 6 e TAF 9 (subunita’ del complesso TFIID)

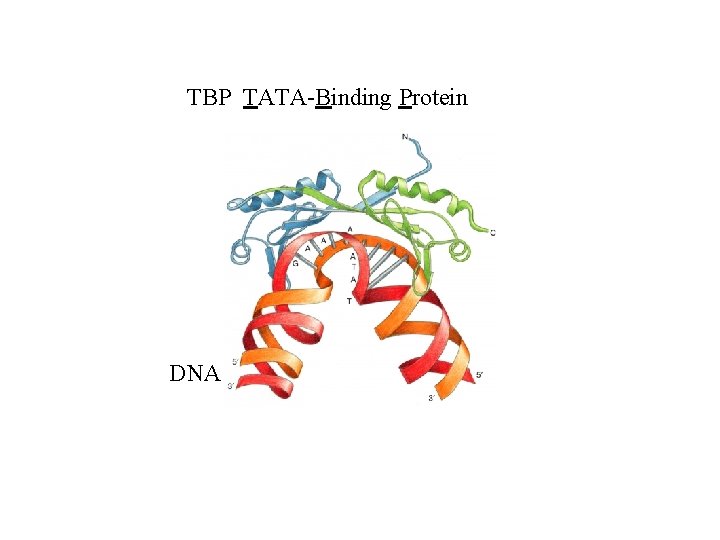
TBP TATA-Binding Protein DNA

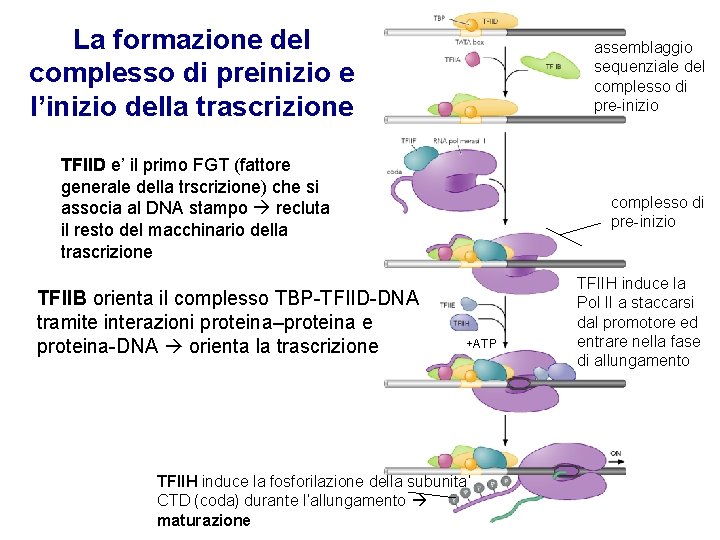
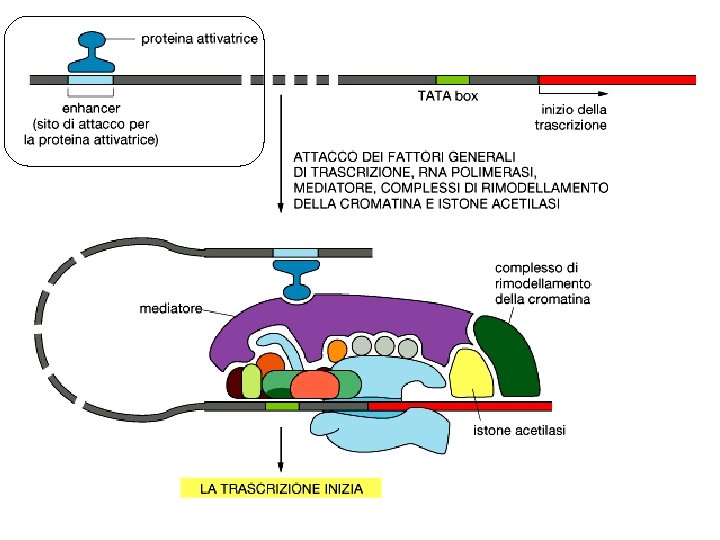
La formazione del complesso di preinizio e l’inizio della trascrizione assemblaggio sequenziale del complesso di pre-inizio TFIID e’ il primo FGT (fattore generale della trscrizione) che si associa al DNA stampo recluta il resto del macchinario della trascrizione TFIIB orienta il complesso TBP-TFIID-DNA tramite interazioni proteina–proteina e proteina-DNA orienta la trascrizione complesso di pre-inizio +ATP TFIIH induce la fosforilazione della subunita’ CTD (coda) durante l’allungamento maturazione TFIIH induce la Pol II a staccarsi dal promotore ed entrare nella fase di allungamento

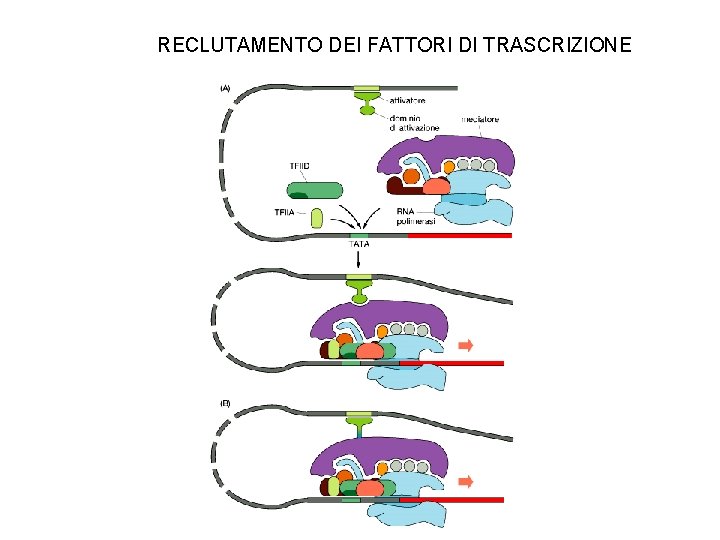
RECLUTAMENTO DEI FATTORI DI TRASCRIZIONE

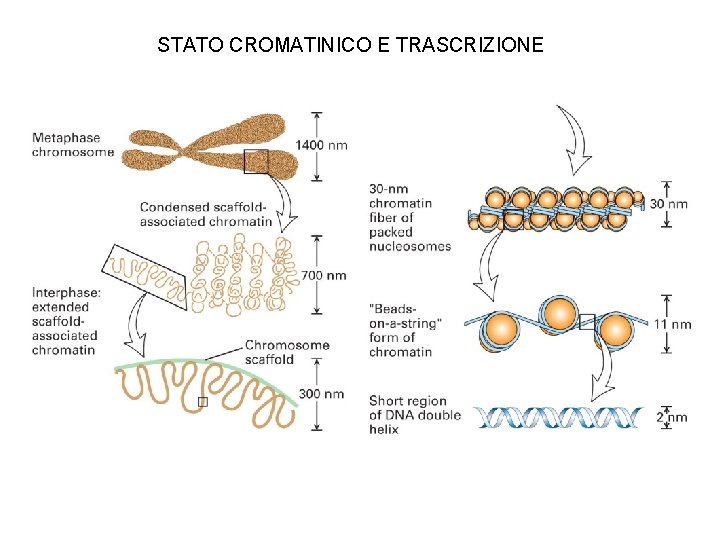
STATO CROMATINICO E TRASCRIZIONE


https: //www. youtube. com/watch? v=SMt Wv. Dbf. HLo

DNA LEARNING CENTER https: //dnalc. cshl. edu/resources/animations/


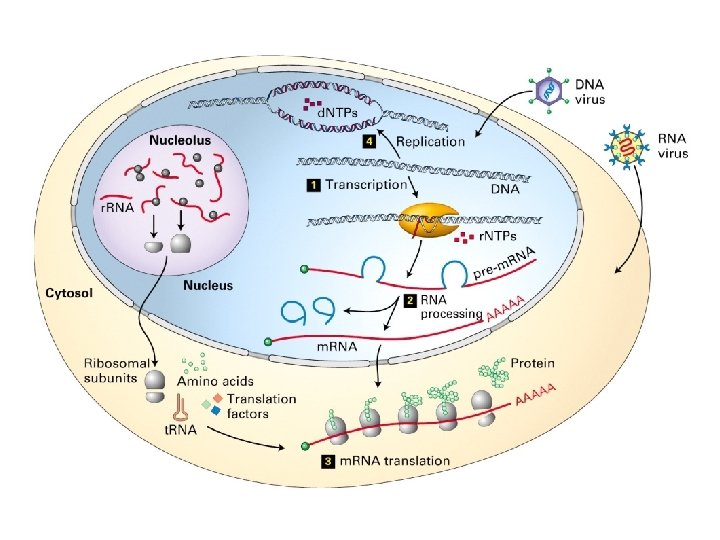
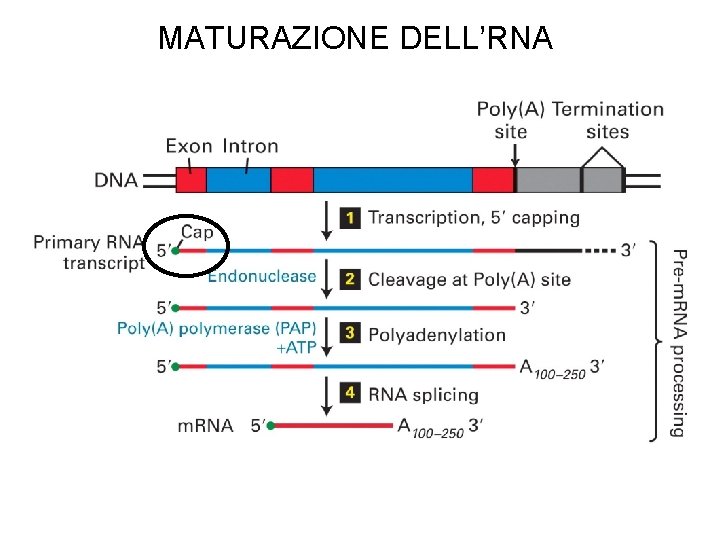
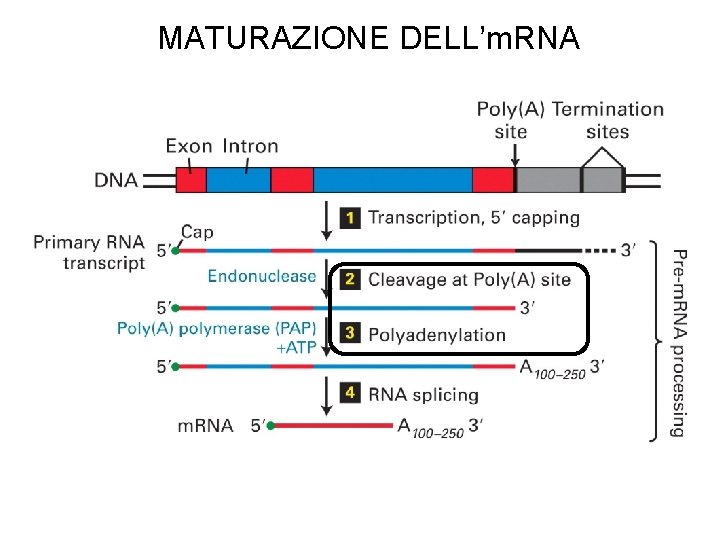
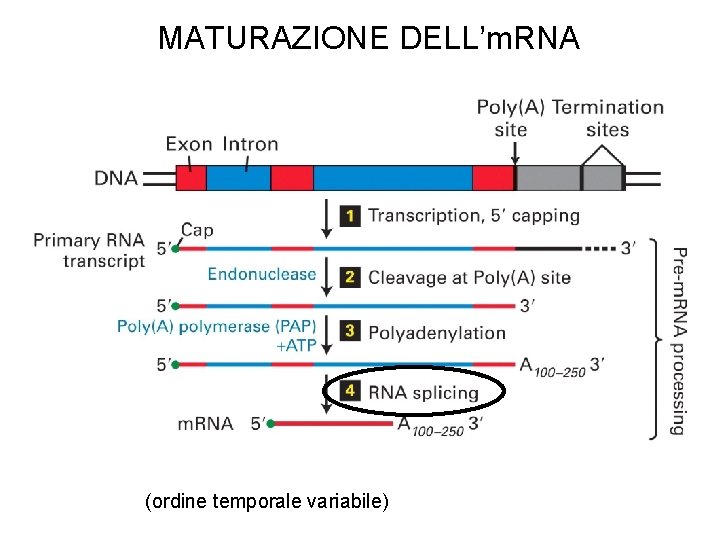
MATURAZIONE DELL’RNA

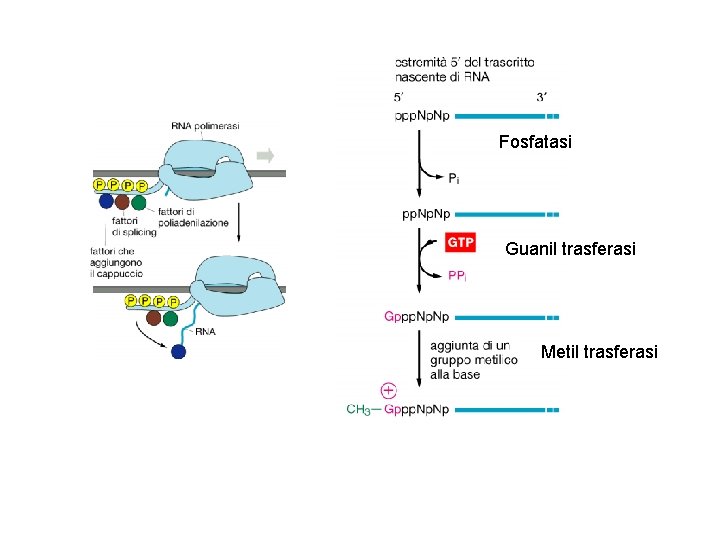
Fosfatasi Guanil trasferasi Metil trasferasi


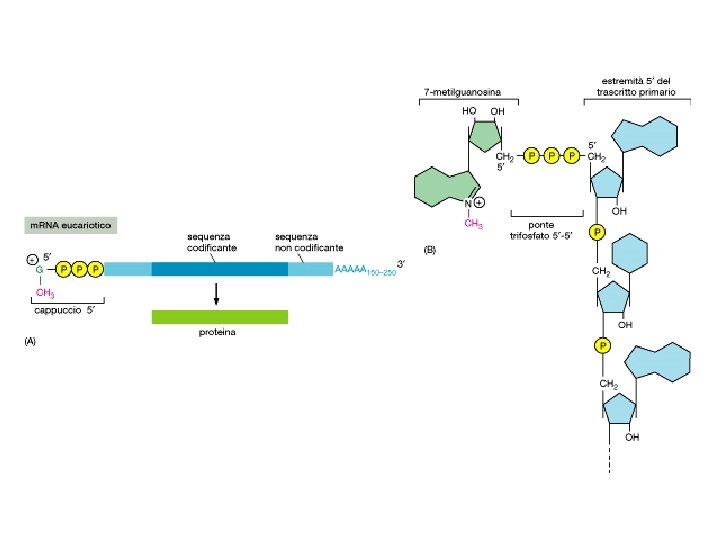
MATURAZIONE DELL’m. RNA

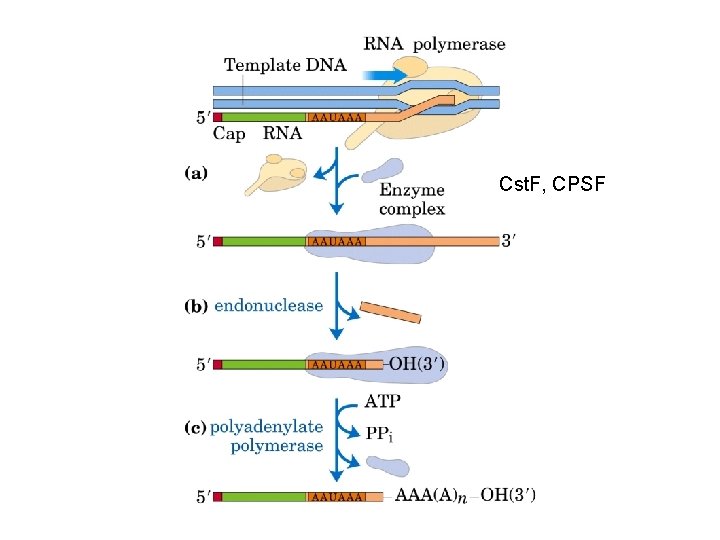
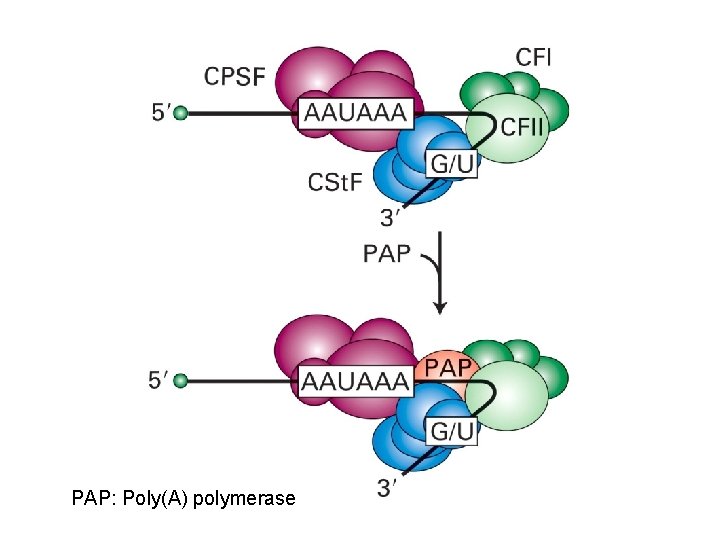
Cst. F, CPSF

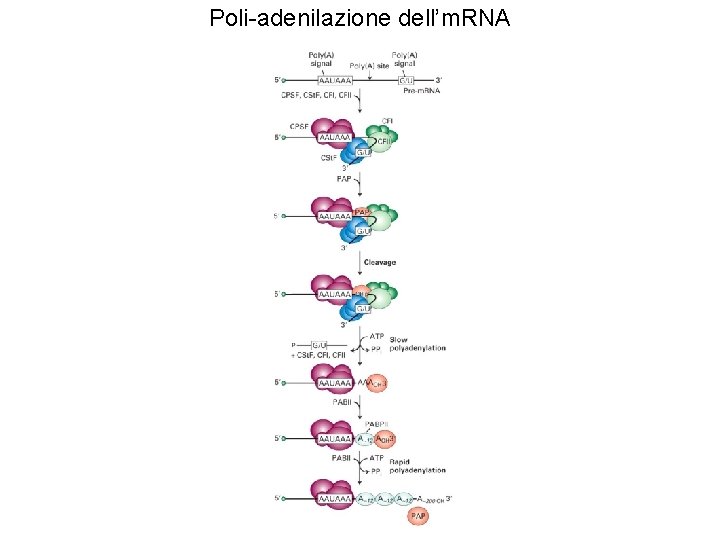
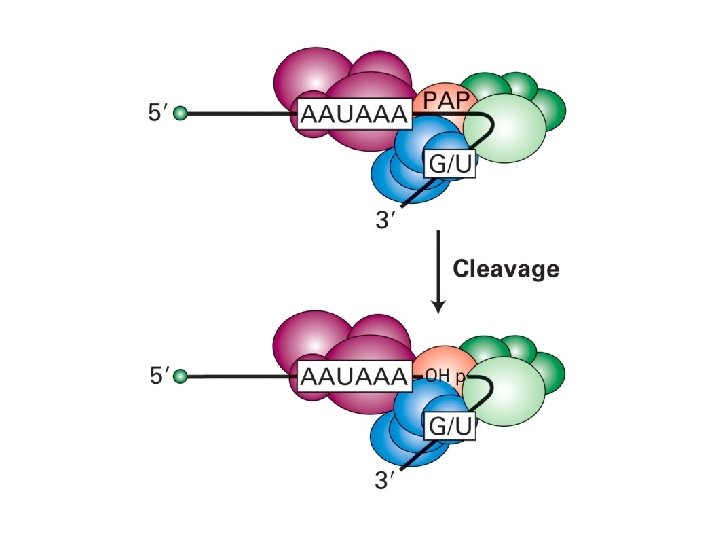
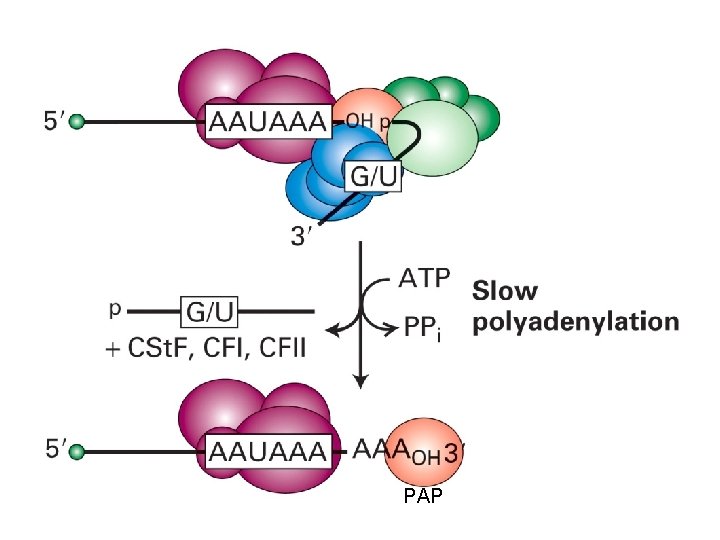
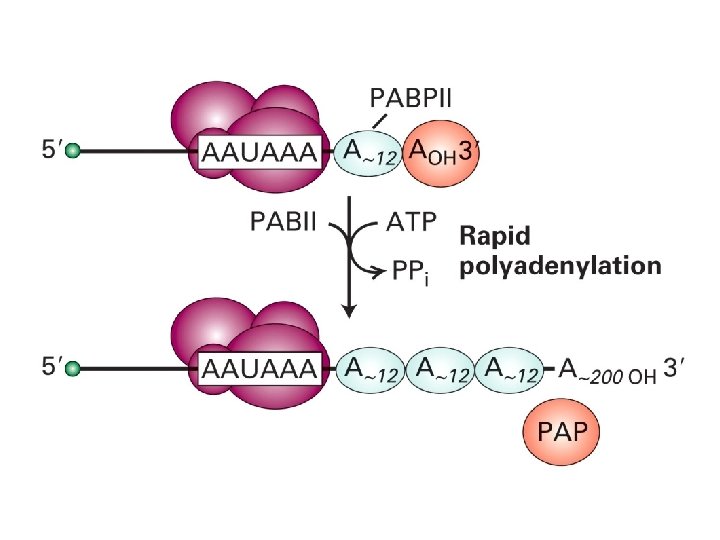
Poli-adenilazione dell’m. RNA

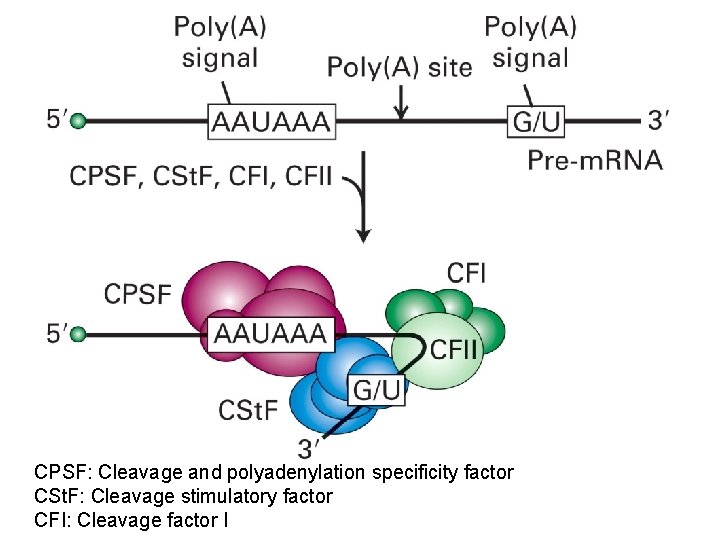
CPSF: Cleavage and polyadenylation specificity factor CSt. F: Cleavage stimulatory factor CFI: Cleavage factor I

PAP: Poly(A) polymerase


PAP

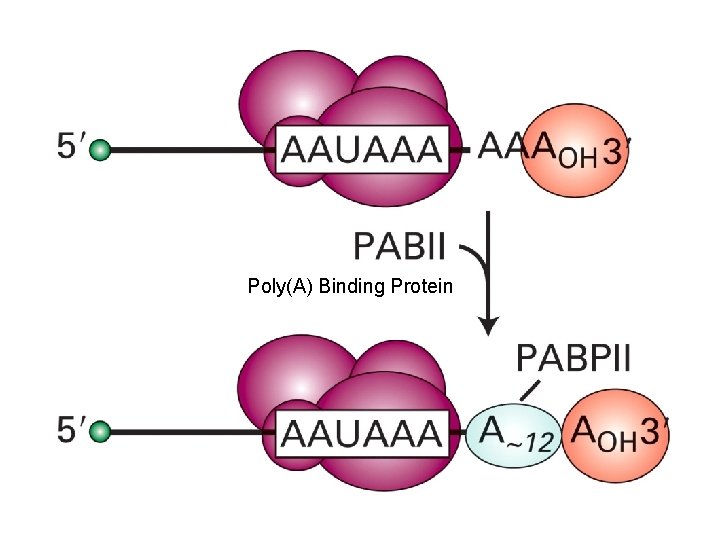
Poly(A) Binding Protein


MATURAZIONE DELL’m. RNA (ordine temporale variabile)

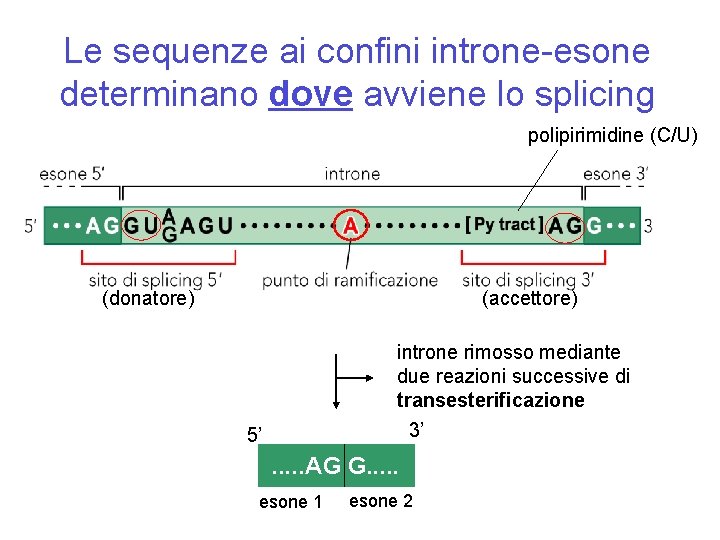
Lo splicing • Come vengono distinti gli esoni dagli introni? • Come vengono rimossi gli introni? • Come vengono uniti in maniera specifica e ordinata gli esoni?

Le sequenze ai confini introne-esone determinano dove avviene lo splicing polipirimidine (C/U) (donatore) (accettore) introne rimosso mediante due reazioni successive di transesterificazione 3’ 5’ . . . AG G. . . esone 1 esone 2

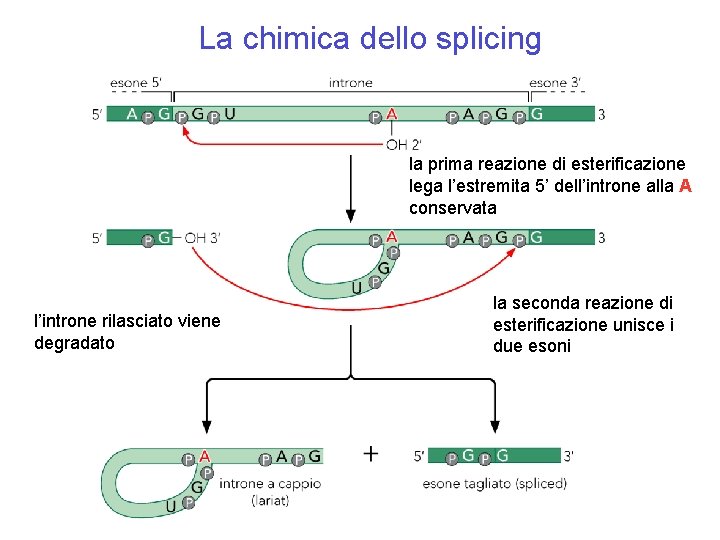
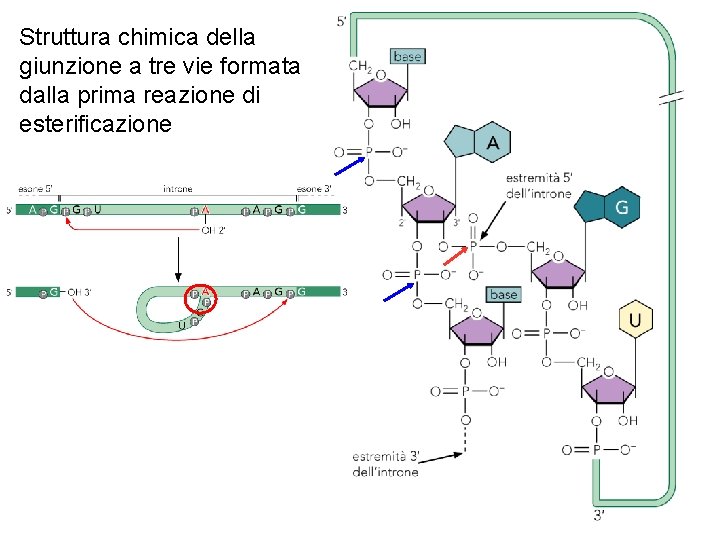
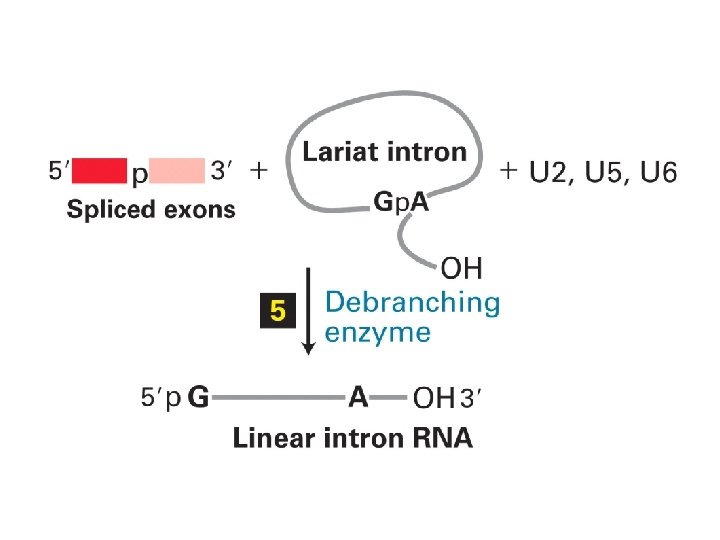
La chimica dello splicing la prima reazione di esterificazione lega l’estremita 5’ dell’introne alla A conservata l’introne rilasciato viene degradato la seconda reazione di esterificazione unisce i due esoni

Struttura chimica della giunzione a tre vie formata dalla prima reazione di esterificazione

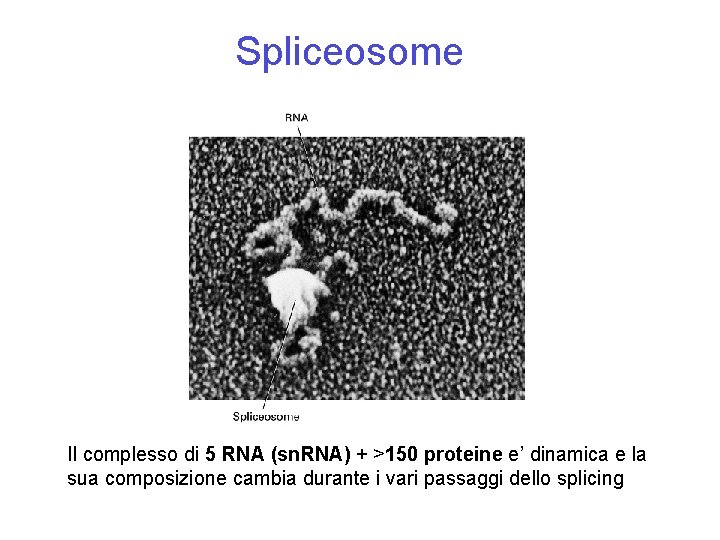
Spliceosome Il complesso di 5 RNA (sn. RNA) + >150 proteine e’ dinamica e la sua composizione cambia durante i vari passaggi dello splicing

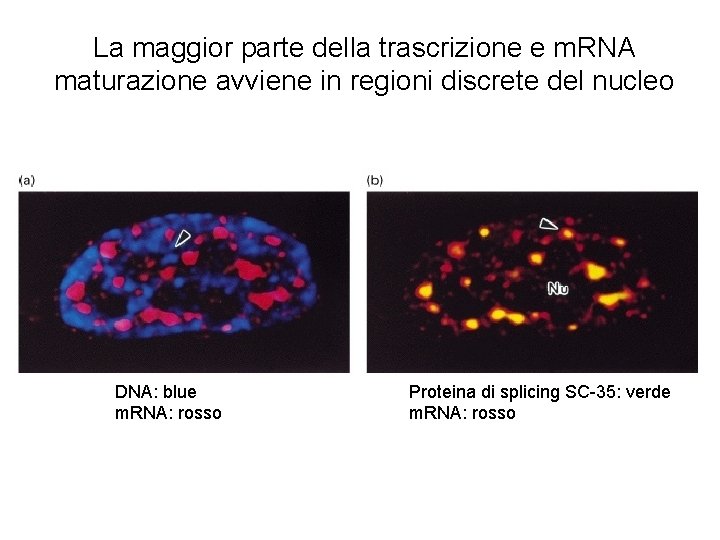
La maggior parte della trascrizione e m. RNA maturazione avviene in regioni discrete del nucleo DNA: blue m. RNA: rosso Proteina di splicing SC-35: verde m. RNA: rosso

I piccoli RNA nucleari (sn. RNA) sn. RNA: U 1, U 2, U 4, U 5, U 6 sn. RNA + proteine = small nuclear ribonuclear protein (Sn. RNP) o “snurp” Funzioni degli sn. RNP -Riconoscono il sito di splicing 5’, il punto di ramificazione, e il sito 3’ -Avvicinano questi siti -Catalizzano il taglio e la giunzione dell’RNA

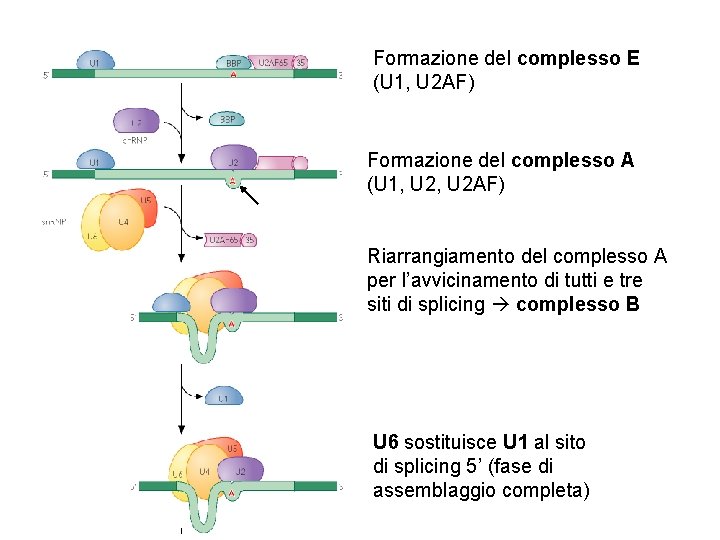
Formazione del complesso E (U 1, U 2 AF) Formazione del complesso A (U 1, U 2 AF) Riarrangiamento del complesso A per l’avvicinamento di tutti e tre siti di splicing complesso B U 6 sostituisce U 1 al sito di splicing 5’ (fase di assemblaggio completa)

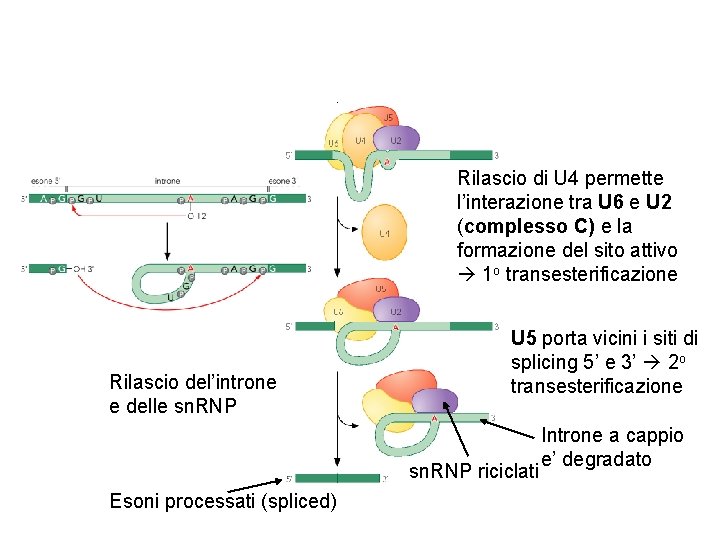
Rilascio di U 4 permette l’interazione tra U 6 e U 2 (complesso C) e la formazione del sito attivo 1 o transesterificazione Rilascio del’introne e delle sn. RNP U 5 porta vicini i siti di splicing 5’ e 3’ 2 o transesterificazione sn. RNP riciclati Esoni processati (spliced) Introne a cappio e’ degradato


https: //www. youtube. com/watch? v=a. Vg wr 0 Qp. YNE

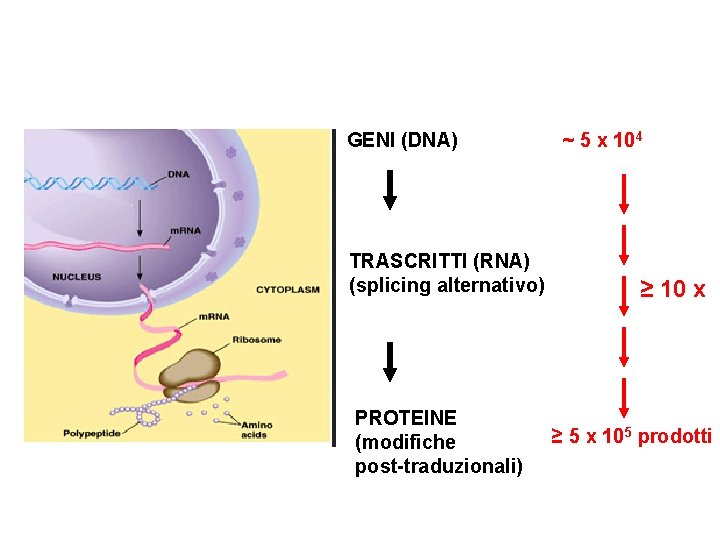
GENI (DNA) TRASCRITTI (RNA) (splicing alternativo) PROTEINE (modifiche post-traduzionali) ~ 5 x 104 ≥ 10 x ≥ 5 x 105 prodotti

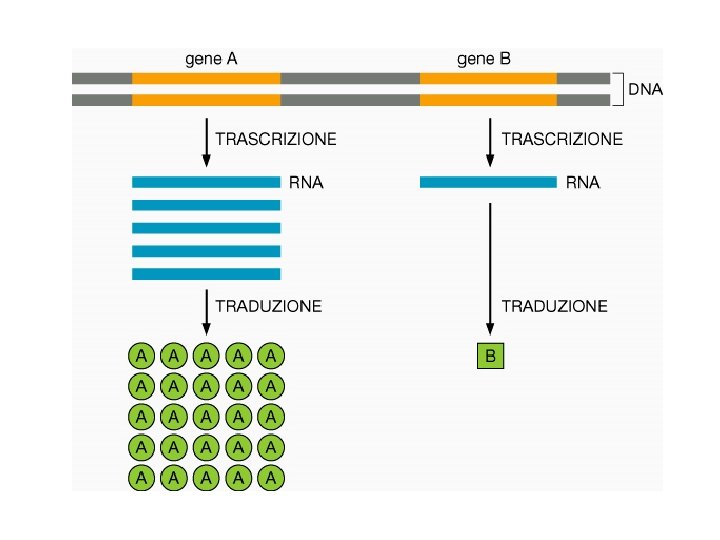
Splicing alternativo: un gene, diversi m. RNA e proteine

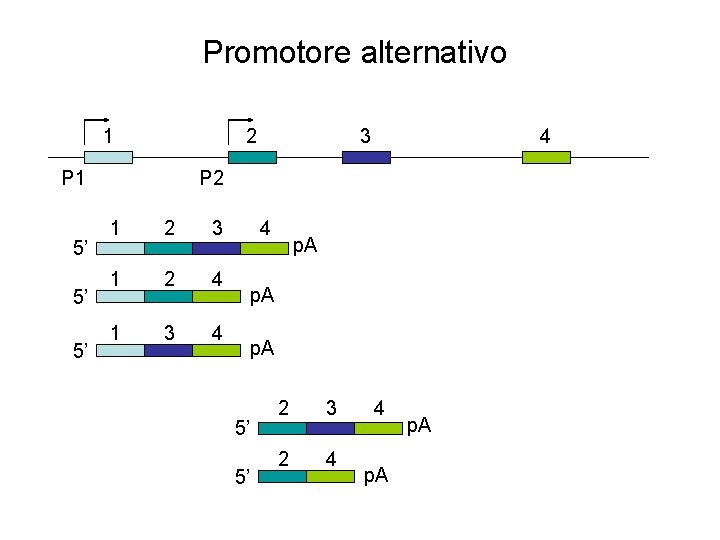
Promotore alternativo 1 2 P 1 5’ 5’ 5’ 3 4 P 2 1 2 3 1 2 4 1 3 4 4 p. A 5’ 5’ 2 3 2 4 4 p. A

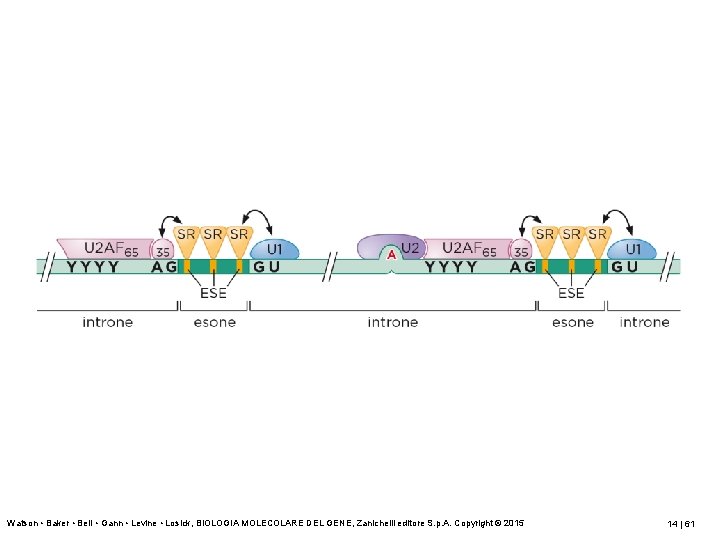
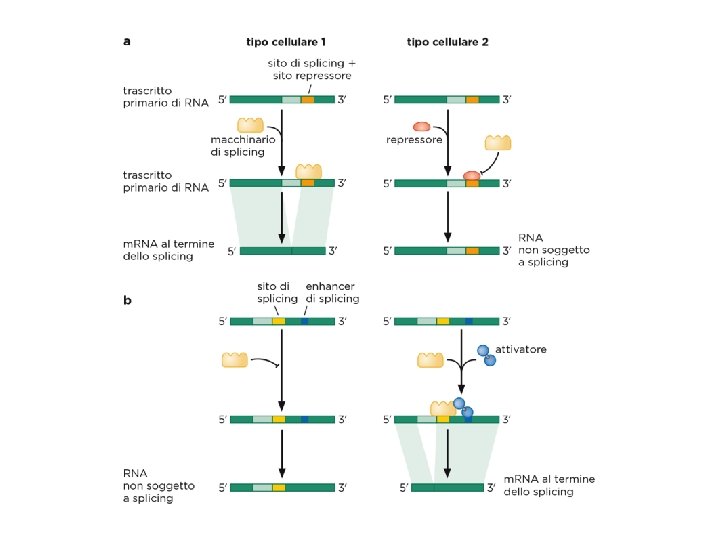
Lo splicing è altamente regolato • Attivatori di splicing (esonici e intronici, proteine SR). • Soppressori di splicing (hn. RNP). • cis elements: SRE – ESE (exonic s. enhancer) – ISE (Intronic s. enhancer) – ESS, ISS (silencers)

Watson • Baker • Bell • Gann • Levine • Losick, BIOLOGIA MOLECOLARE DEL GENE, Zanichelli editore S. p. A. Copyright © 2015 14 | 61


Errori di splicing sono causa di malattie genetiche • Mutazioni dei siti di splicing • Mutazioni di componenti dello spliceosoma

Uso terapeutico dello splicing indotto • Inibizione di splicing di esone mutato (exon skipping). (cardiomiopatia dilatativa da mutazione di Titin, Atrofia muscolare spinale)

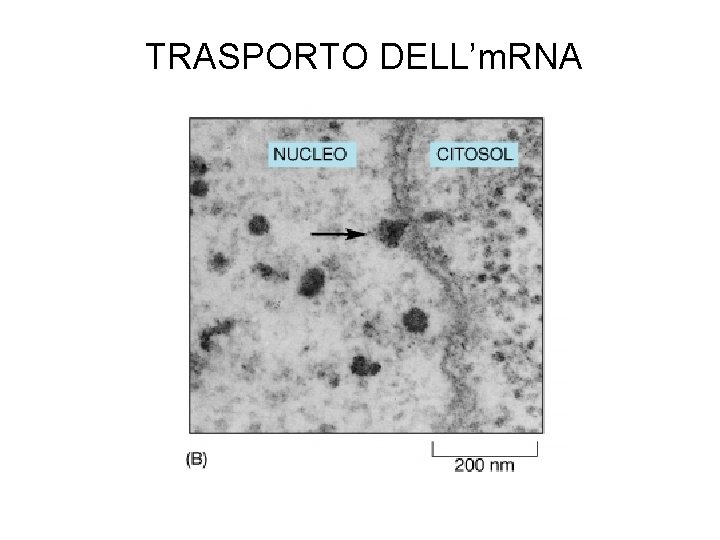
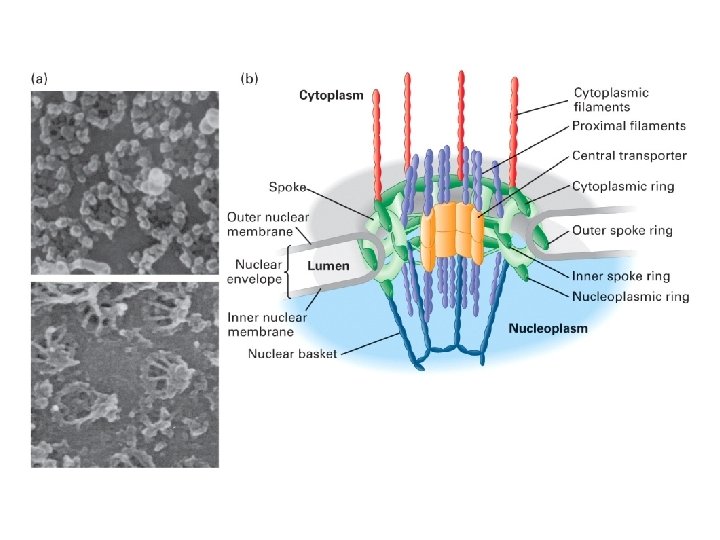
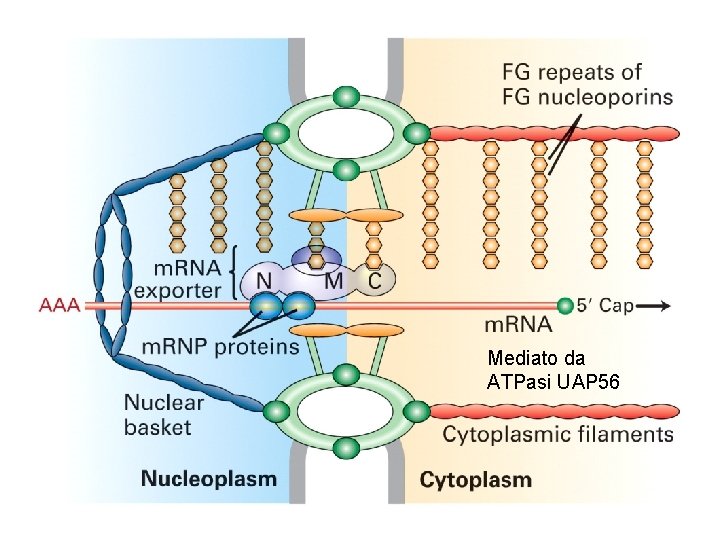
TRASPORTO DELL’m. RNA

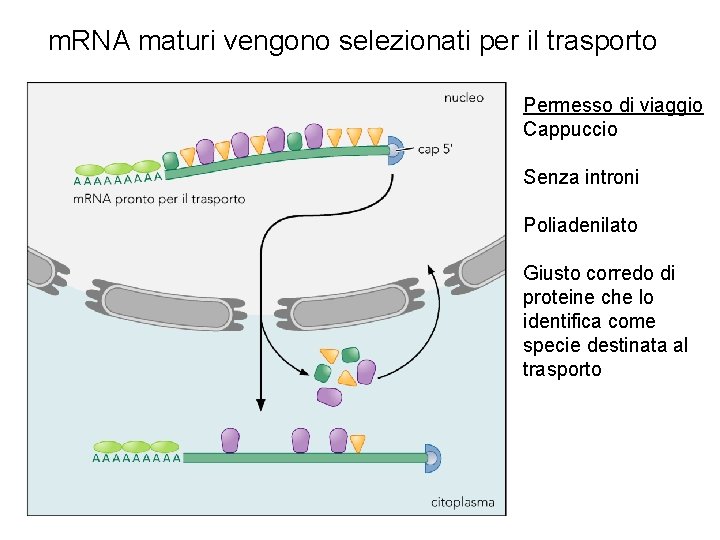
m. RNA maturi vengono selezionati per il trasporto Permesso di viaggio Cappuccio Senza introni Poliadenilato Giusto corredo di proteine che lo identifica come specie destinata al trasporto


Mediato da ATPasi UAP 56