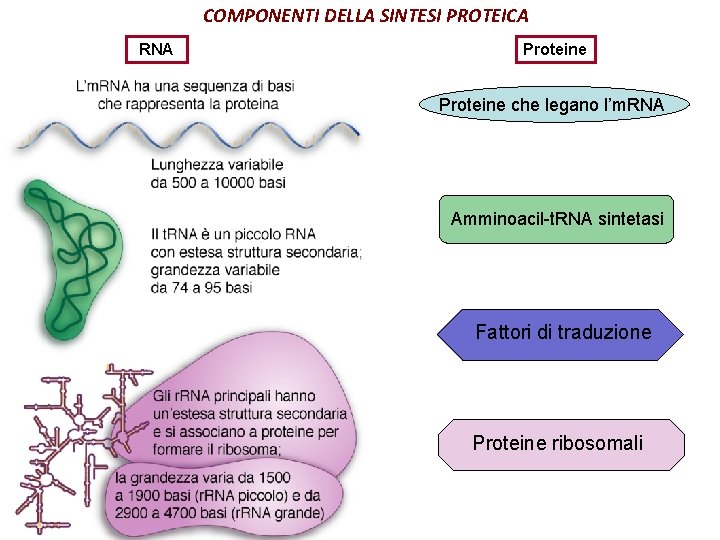
COMPONENTI DELLA SINTESI PROTEICA RNA Proteine che legano














- Slides: 41

COMPONENTI DELLA SINTESI PROTEICA RNA Proteine che legano l’m. RNA Amminoacil-t. RNA sintetasi Fattori di traduzione Proteine ribosomali

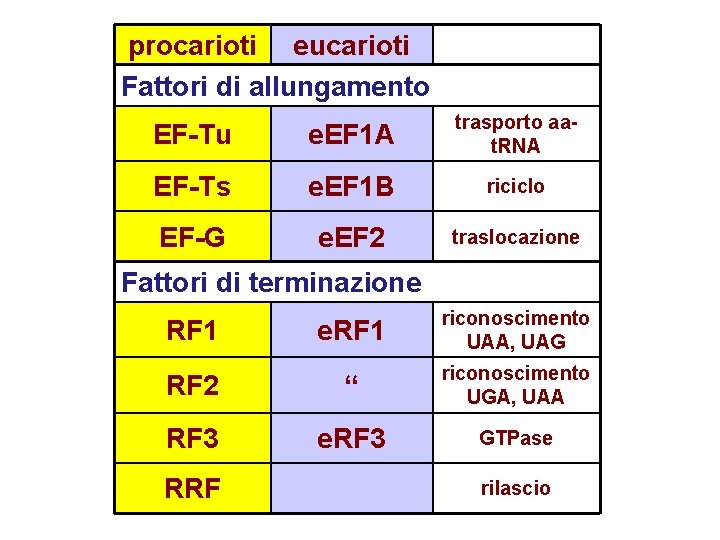
procarioti eucarioti Fattori di allungamento EF-Tu e. EF 1 A trasporto aat. RNA EF-Ts e. EF 1 B riciclo EF-G e. EF 2 traslocazione Fattori di terminazione RF 1 e. RF 1 riconoscimento UAA, UAG RF 2 “ riconoscimento UGA, UAA RF 3 e. RF 3 GTPase RRF rilascio

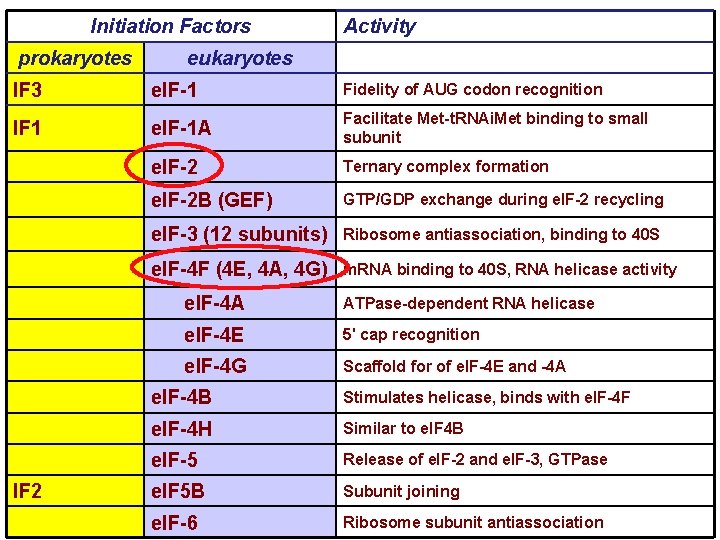
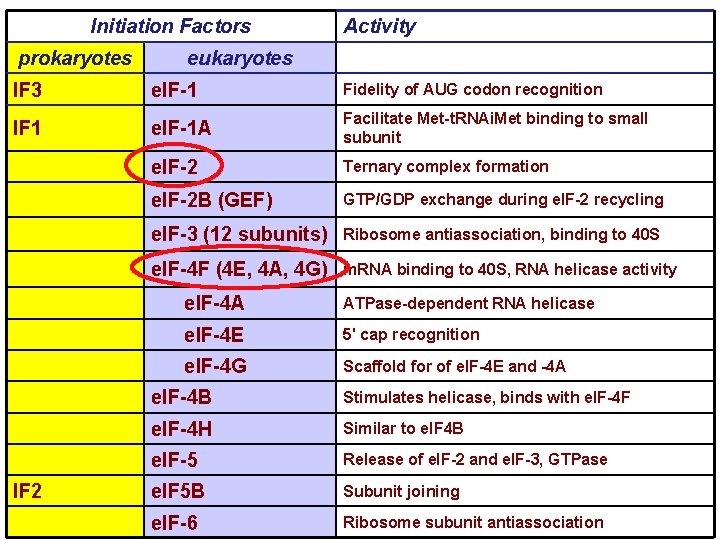
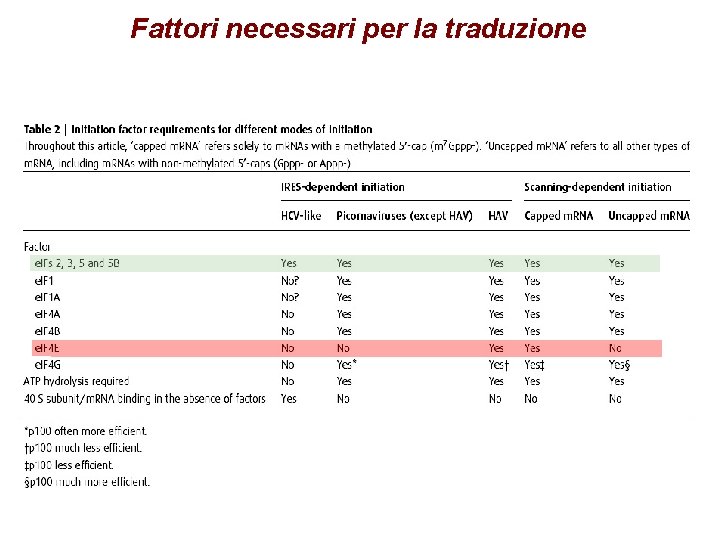
Initiation Factors prokaryotes Activity eukaryotes IF 3 e. IF-1 Fidelity of AUG codon recognition IF 1 e. IF-1 A Facilitate Met-t. RNAi. Met binding to small subunit e. IF-2 Ternary complex formation e. IF-2 B (GEF) GTP/GDP exchange during e. IF-2 recycling e. IF-3 (12 subunits) Ribosome antiassociation, binding to 40 S e. IF-4 F (4 E, 4 A, 4 G) m. RNA binding to 40 S, RNA helicase activity IF 2 e. IF-4 A ATPase-dependent RNA helicase e. IF-4 E 5' cap recognition e. IF-4 G Scaffold for of e. IF-4 E and -4 A e. IF-4 B Stimulates helicase, binds with e. IF-4 F e. IF-4 H Similar to e. IF 4 B e. IF-5 Release of e. IF-2 and e. IF-3, GTPase e. IF 5 B Subunit joining e. IF-6 Ribosome subunit antiassociation


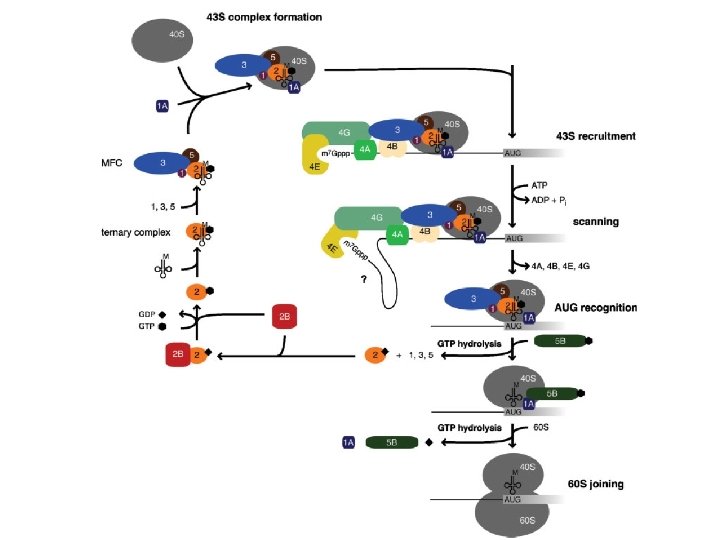
Passaggi dell’inizio di traduzione 1. Formazione complesso 43 S 2. Reclutamento del complesso 43 S sul 5’ dell’m. RNA (48 S) 3. Scanning del 5’ UTR e riconoscimento dell’AUG 4. Formazione del complesso 80 S

2

e. IF 2 § 3 subunità: a, b, g § Subunità b aiuta attività di GTPasi e modula il legame t. RNAi-e. IF 2 g § Subunità a è un regolatore della traduzione. E’ fosforilata (ser 51) da diverse chinasi in risposta a stress e. IF 2 B § 5 subunità: a, b, g, d, e § Fattore di scambio GDP-GTP (GEF) per e. IF 2 § 2 subcomplessi: d, e attività catalitica a, b, g attività regolativa

e. IF 3 § 10 -11 subunità § Nucleo di 5 subunità: e. IF 3 a, b, c, i, g § In lievito forma un complesso con e. IF 1, e. IF 2, e. IF 5, Met-t. RNAi (MFC) § Richiesto per il legame del 43 S all’m. RNA

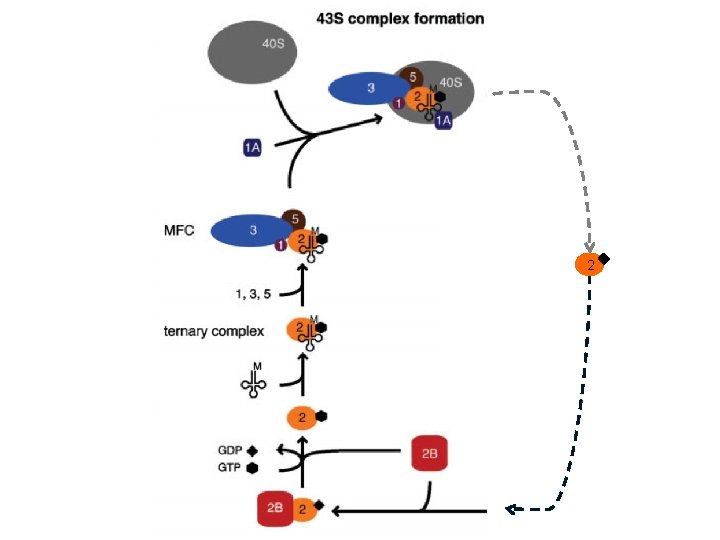
Reclutamento 43 S-m. RNA

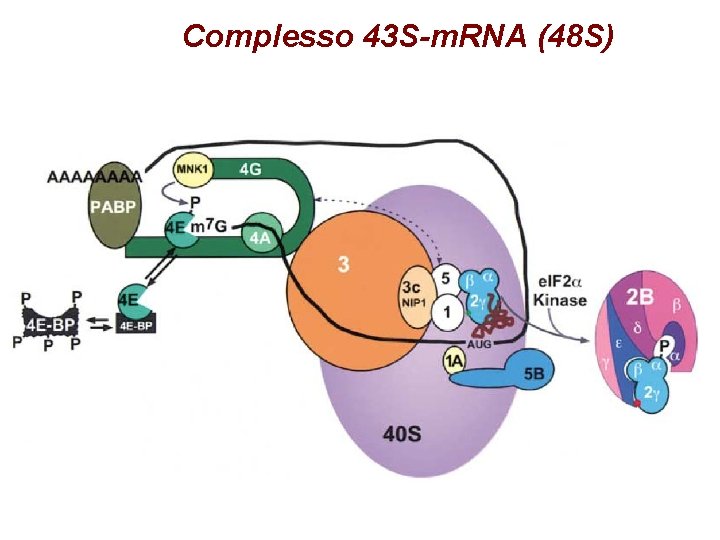
Complesso 43 S-m. RNA (48 S)

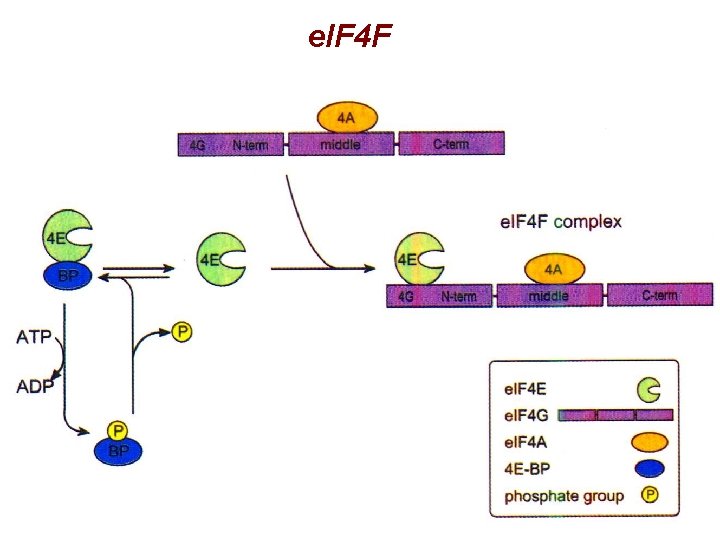
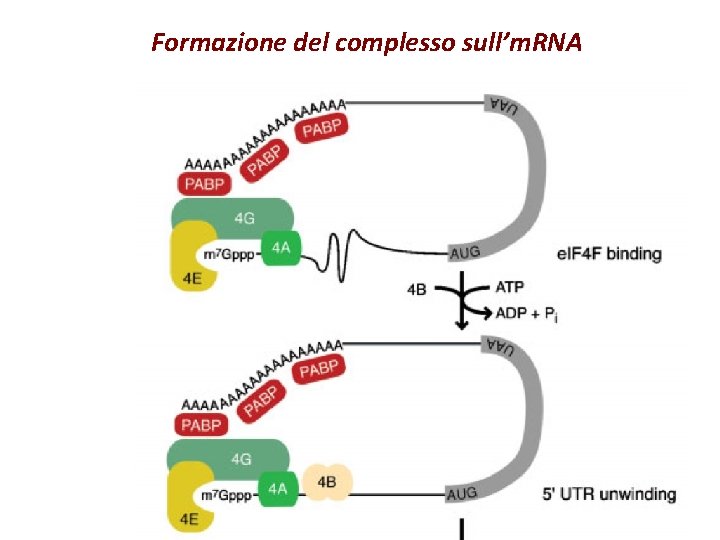
e. IF 4 F

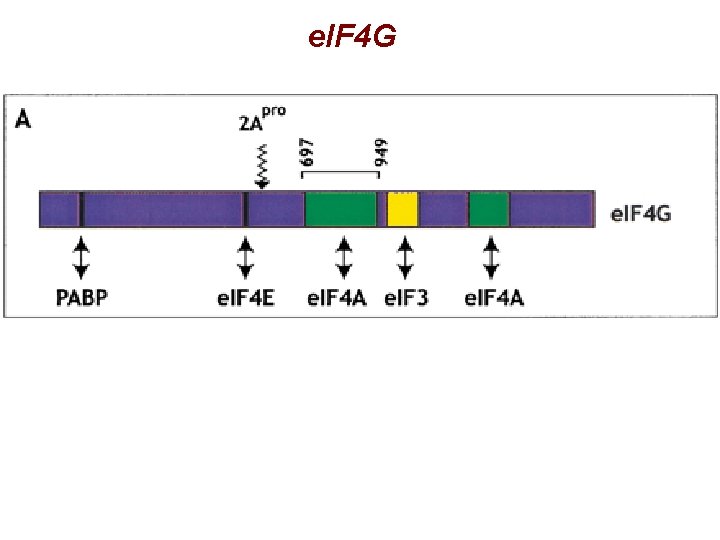
e. IF 4 F § Composto da 3 subunità e. IF 4 A: elicasi, aiutato da e. IF 4 B e. IF 4 E: cap binding protein, regolato da fosforilazione e interazione con e. IF 4 E-BP e. IF 4 G: adattatore, interagisce con diversi fattori

e. IF 4 G

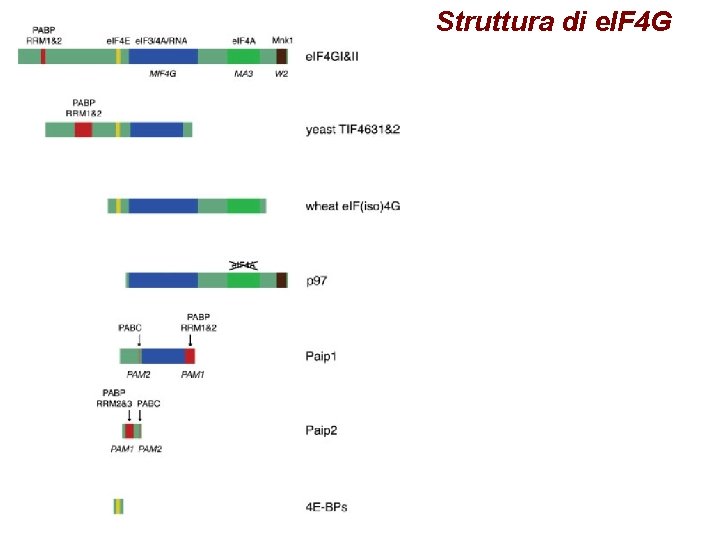
Struttura di e. IF 4 G

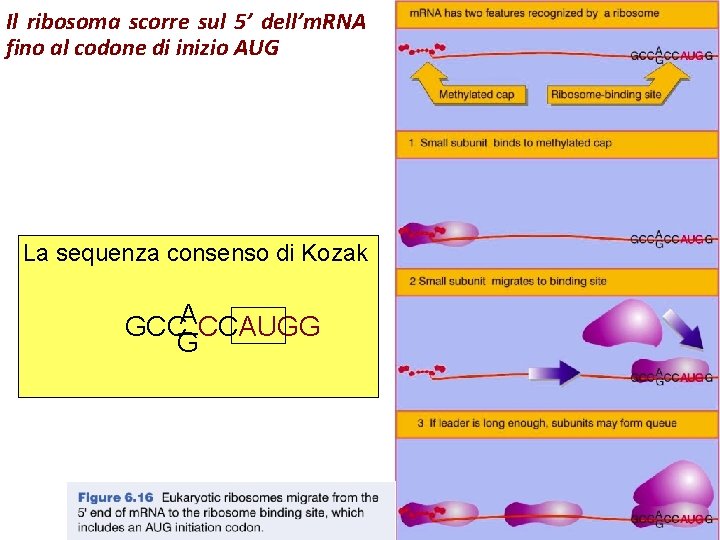
Il ribosoma scorre sul 5’ dell’m. RNA fino al codone di inizio AUG La sequenza consenso di Kozak A _ GCC CCAUGG G

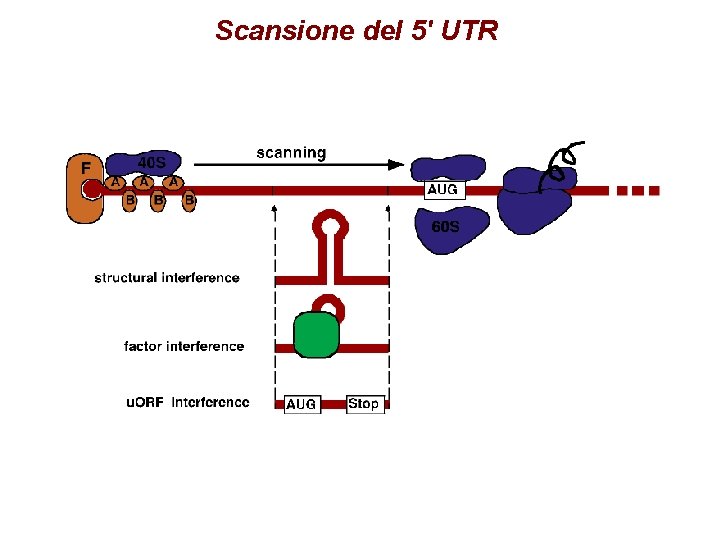
Scansione del 5' UTR

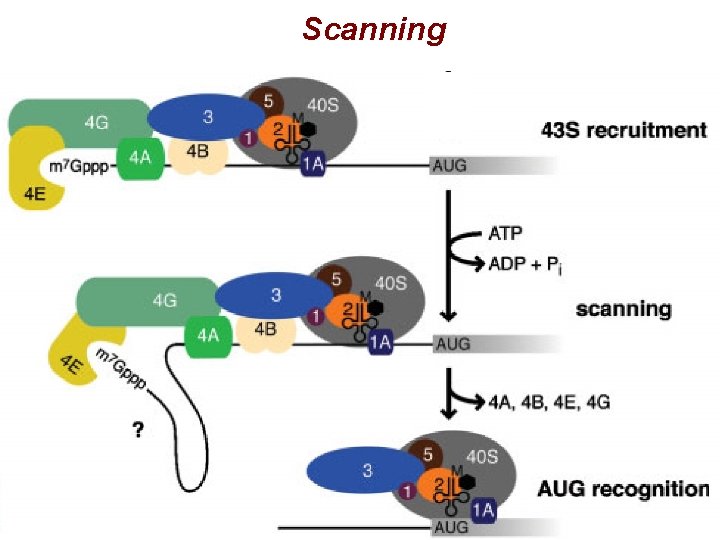
Scanning

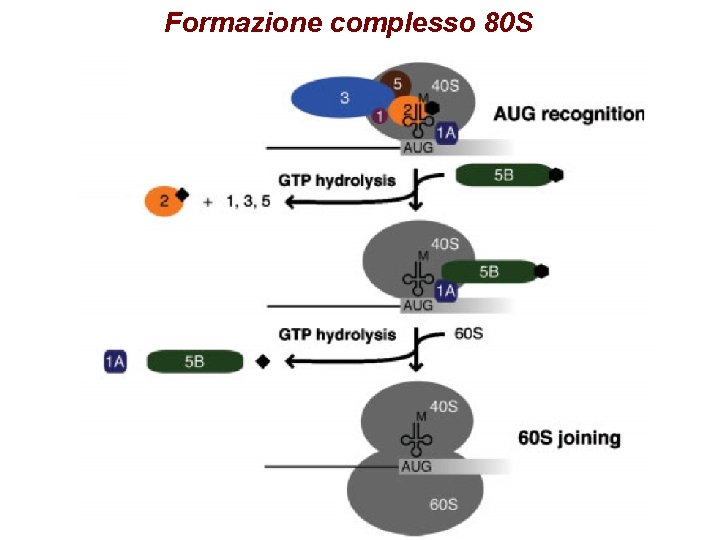
Formazione complesso 80 S

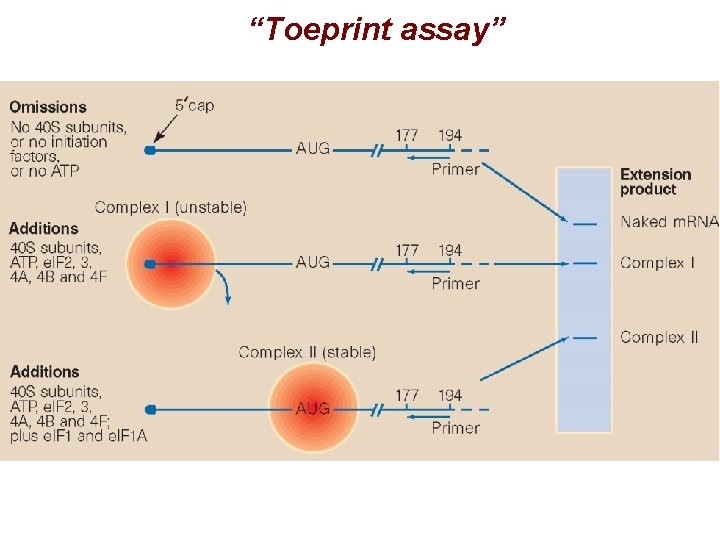
“Toeprint assay”

Scanning § 40 S, ATP, e. IF 2, e. IF 4 A, e. IF 4 B, e. IF 4 F, m. RNA sufficienti per formare complesso I (non produttivo) § e. IF 1, e. IF 1 A necessari per il complesso II (scanning fino all’AUG) § Se non ci sono strutture secondarie e. IF 4 A, 4 B, 4 F non sono necessari (in vitro)

Formazione del complesso sull’m. RNA

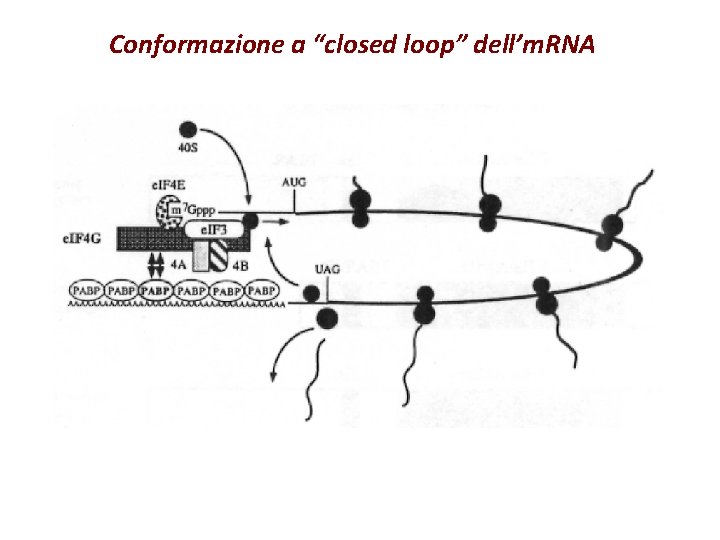
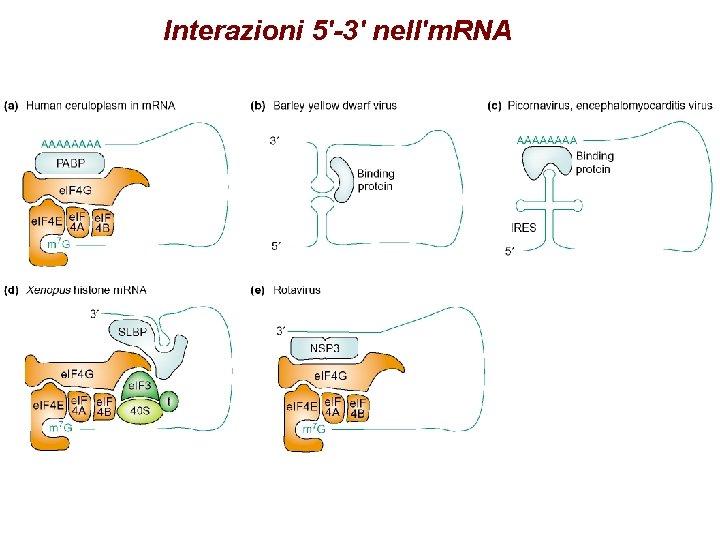
Conformazione a “closed loop” dell’m. RNA


Interazioni 5'-3' nell'm. RNA

Ruolo di PABP nella traduzione § In estratti “cell free” di lievito sinergismo tra cap e coda poli(A) § Interazione tra PABP e e. IF 4 G § e. IF 4 E, e. IF 4 G, PABP e m. RNA formano strutture circolari (in vitro) § Altre proteine che interagiscono con PABP (Paip 1, 2 e e. RF 3)


Initiation Factors prokaryotes Activity eukaryotes IF 3 e. IF-1 Fidelity of AUG codon recognition IF 1 e. IF-1 A Facilitate Met-t. RNAi. Met binding to small subunit e. IF-2 Ternary complex formation e. IF-2 B (GEF) GTP/GDP exchange during e. IF-2 recycling e. IF-3 (12 subunits) Ribosome antiassociation, binding to 40 S e. IF-4 F (4 E, 4 A, 4 G) m. RNA binding to 40 S, RNA helicase activity IF 2 e. IF-4 A ATPase-dependent RNA helicase e. IF-4 E 5' cap recognition e. IF-4 G Scaffold for of e. IF-4 E and -4 A e. IF-4 B Stimulates helicase, binds with e. IF-4 F e. IF-4 H Similar to e. IF 4 B e. IF-5 Release of e. IF-2 and e. IF-3, GTPase e. IF 5 B Subunit joining e. IF-6 Ribosome subunit antiassociation

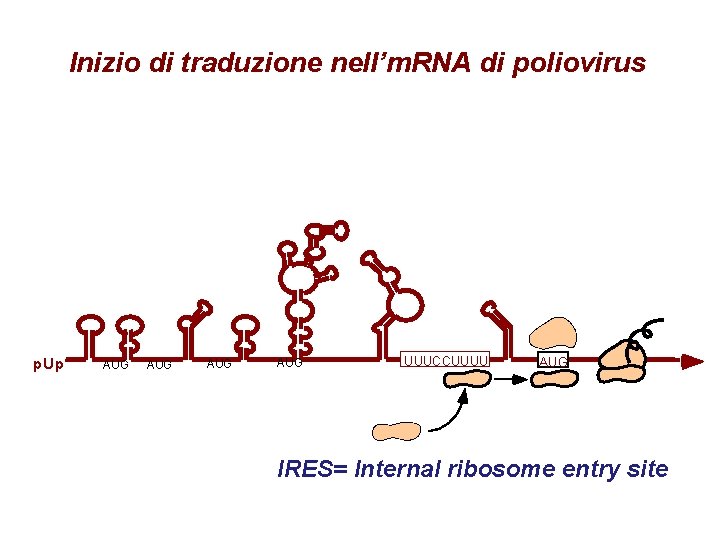
Inizio di traduzione nell’m. RNA di poliovirus p. Up AUG AUG UUUCCUUUU AUG IRES= Internal ribosome entry site

Saggio dell’m. RNA bicistronico cap luciferasi CAT +/- +++ cap CAT IRES cap CAT (+/0) luciferasi +++ (+) 4 F luciferasi IRES luciferasi +++


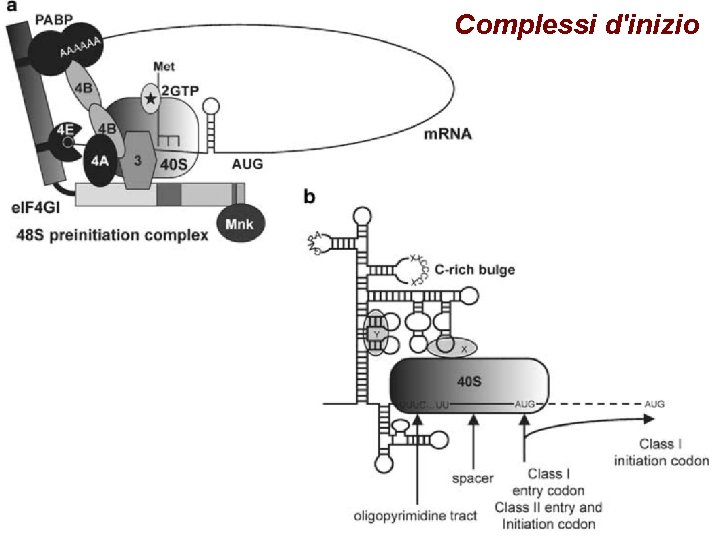
Complessi d'inizio

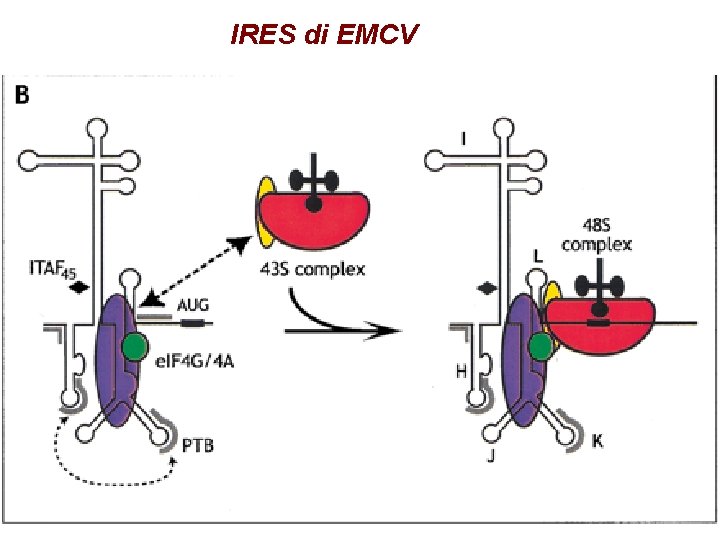
IRES di EMCV

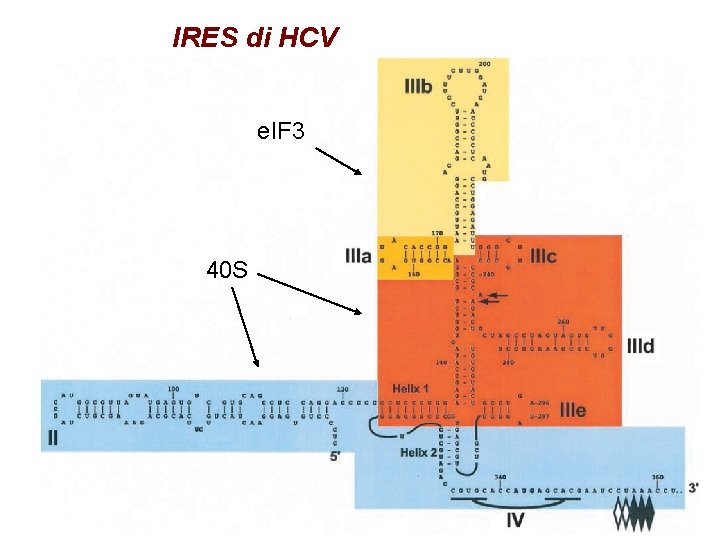
IRES di HCV e. IF 3 40 S

Fattori necessari per la traduzione

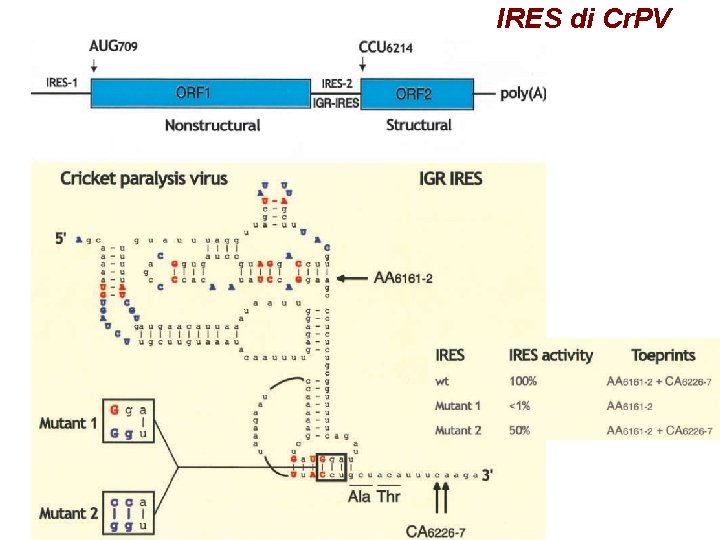
IRES di Cr. PV

Sequenze IRES virali

IRES negli m. RNA cellulari

Ruolo delle IRES

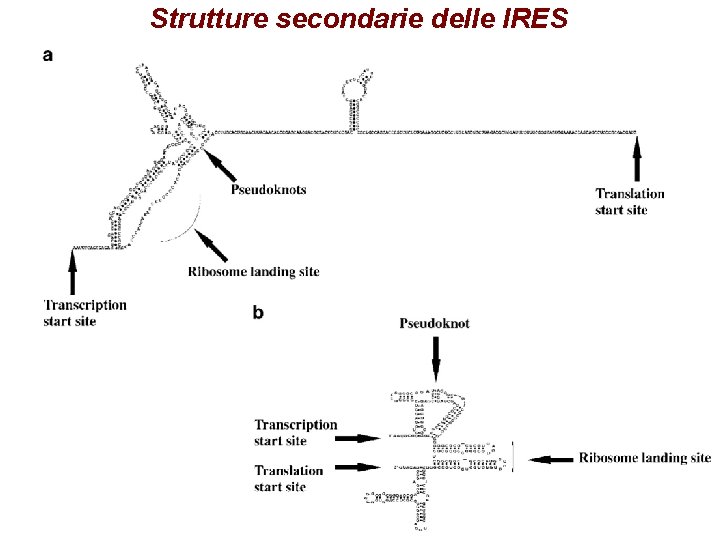
Strutture secondarie delle IRES

Fattori trans-agenti delle IRES (ITAF)

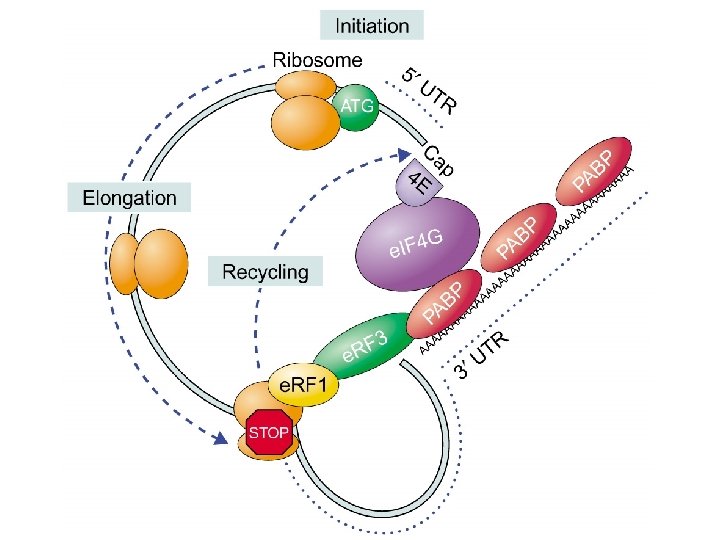
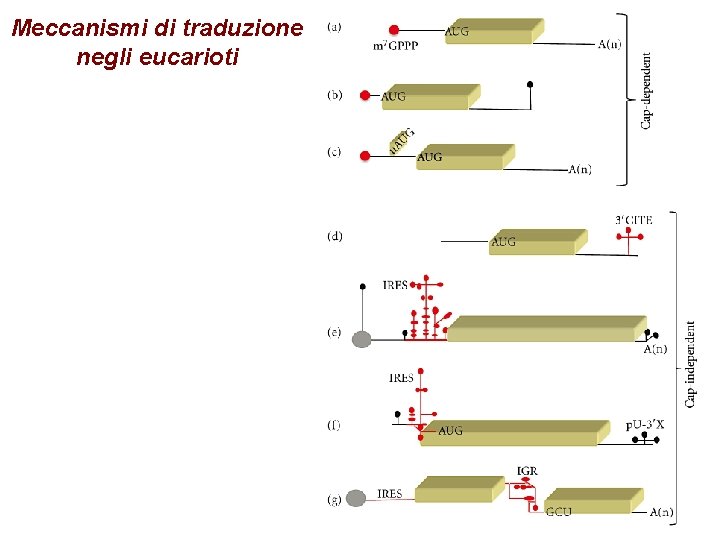
Meccanismi di traduzione negli eucarioti
 La sintesi proteica si arresta quando
La sintesi proteica si arresta quando Parte della fisica che studia il moto dei corpi
Parte della fisica che studia il moto dei corpi Componenti fortemente connesse
Componenti fortemente connesse Pressão hidrostática
Pressão hidrostática Síntese proteíca
Síntese proteíca Virus definitie
Virus definitie Enzima
Enzima Sintese proteica
Sintese proteica Repetto
Repetto Componenti principali di un computer
Componenti principali di un computer Componenti del sangue
Componenti del sangue Green village cittadella
Green village cittadella Analisi delle componenti principali
Analisi delle componenti principali Che cos'è rna
Che cos'è rna Bicistronico
Bicistronico Che che kooley
Che che kooley Facesti come quei che va di notte che porta il lume
Facesti come quei che va di notte che porta il lume Proposizione oggettiva e soggettiva
Proposizione oggettiva e soggettiva Ricordo quegli occhi pieni di vita
Ricordo quegli occhi pieni di vita Voi che per li occhi mi passate il core
Voi che per li occhi mi passate il core Semantica componenziale
Semantica componenziale Azione 17 pnsd
Azione 17 pnsd C.m. n. 8 del 6 marzo 2013 sintesi
C.m. n. 8 del 6 marzo 2013 sintesi Romanticismo sintesi
Romanticismo sintesi Come si forma un idrossido
Come si forma un idrossido Sintesi
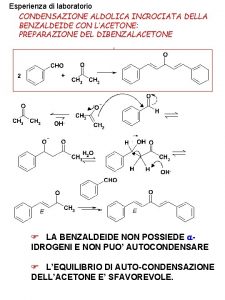
Sintesi Dibenzalacetone tlc
Dibenzalacetone tlc Abilità traspositive
Abilità traspositive Sintesi 5 maggio
Sintesi 5 maggio Sintesi portale mantova
Sintesi portale mantova Cos'è la sintesi di un testo
Cos'è la sintesi di un testo Sintesi di gabriel
Sintesi di gabriel Dpr 80 2013 sintesi
Dpr 80 2013 sintesi Autonomia scolastica sintesi
Autonomia scolastica sintesi Sintesi fenobarbital
Sintesi fenobarbital L'amai perchè provava un dolore profondo
L'amai perchè provava un dolore profondo Legge 13 luglio 2015 n. 107 sintesi
Legge 13 luglio 2015 n. 107 sintesi Brunton
Brunton Sintesi di gabriel
Sintesi di gabriel Legge 297 1994
Legge 297 1994 Curricolo orizzontale e verticale sintesi
Curricolo orizzontale e verticale sintesi 53/2003 sintesi
53/2003 sintesi