Codice genetico traduzione sintesi proteica 1 TRADUZIONE sintesi










- Slides: 33

Codice genetico, traduzione, sintesi proteica 1

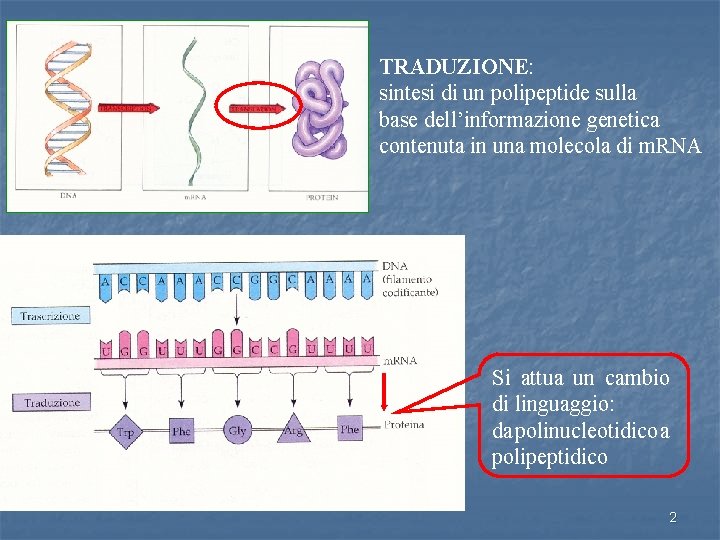
TRADUZIONE: sintesi di un polipeptide sulla base dell’informazione genetica contenuta in una molecola di m. RNA Si attua un cambio di linguaggio: da polinucleotidico a polipeptidico 2

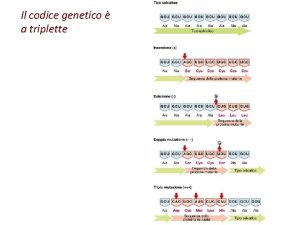
Codice genetico 4 basi (A, C, G, U) 20 aa Dobbiamo passare da un “alfabeto” a 4 lettere a uno a 20 lettere Codice a 1 lettera (41 combinazioni) 4 aa Codice a 2 lettere (42 combinazioni) 16 aa Codice a 3 lettere (43 combinazioni) 64 aa 3

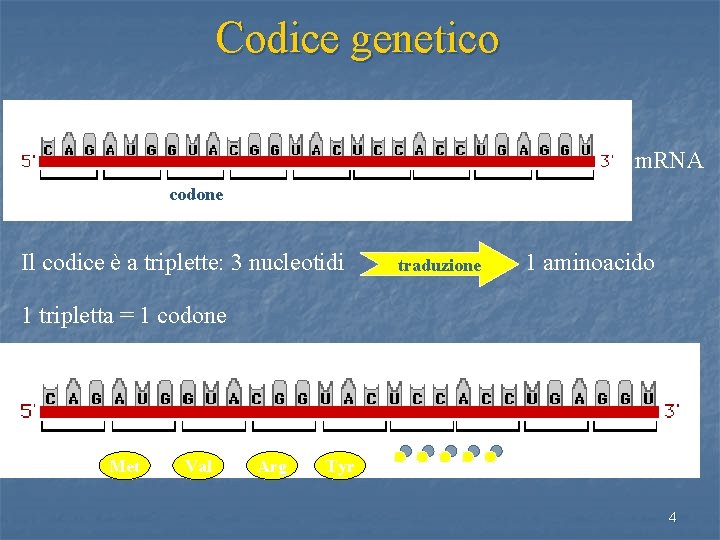
Codice genetico m. RNA codone Il codice è a triplette: 3 nucleotidi traduzione 1 aminoacido 1 tripletta = 1 codone Met Val Arg Tyr 4

Codice genetico 1961 Marshall Niremberg decodifica la prima tripletta UUU = Phe (fenilalanina) (traduzione in vitro di un poli U) 1966 la decodificazione è completata 43 possibili combinazioni: 64 codoni, di cui 61 sono codificanti e 3 sono codoni di STOP 5

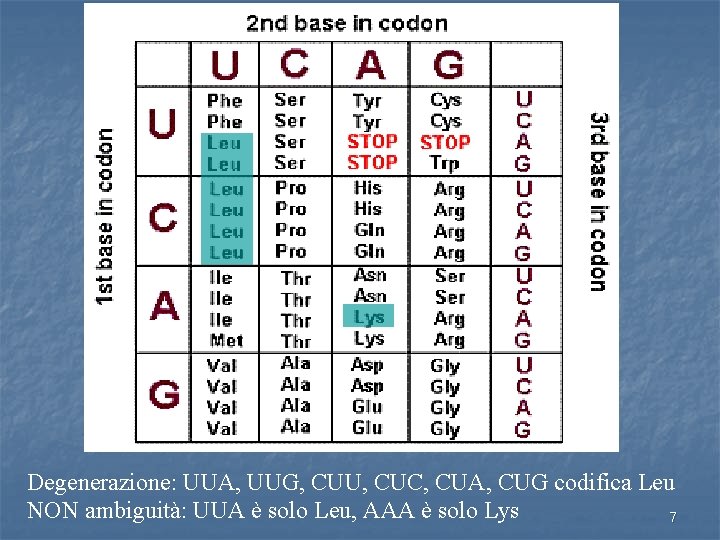
Aminoacidi e codoni 20 sono gli aminoacidi che formano le proteine 61 sono le triplette o “codoni” codificanti del codice genetico universale il codice genetico è “degenerato” (ridondante): più triplette codificano per uno stesso aa ma non è mai ambiguo (una tripletta codifica per un solo aa) 6

Degenerazione: UUA, UUG, CUU, CUC, CUA, CUG codifica Leu NON ambiguità: UUA è solo Leu, AAA è solo Lys 7

Il codice genetico è universale Il codice di traduzione è lo stesso per tutti gli organismi viventi AAA Lys 8

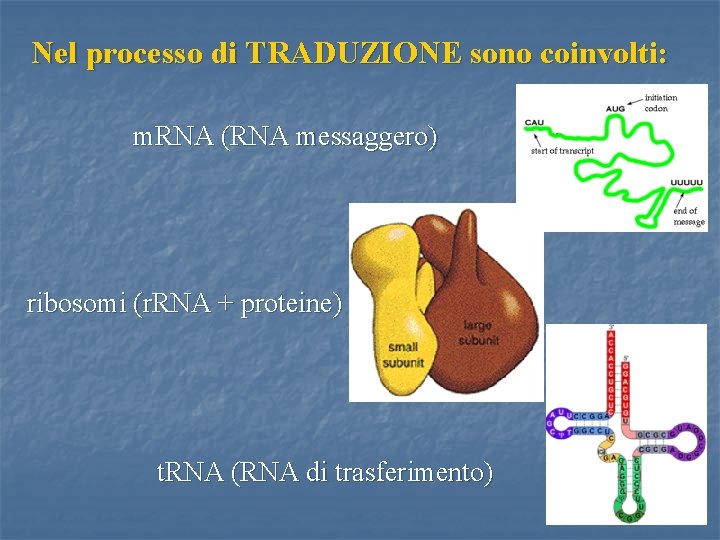
Nel processo di TRADUZIONE sono coinvolti: m. RNA (RNA messaggero) ribosomi (r. RNA + proteine) t. RNA (RNA di trasferimento) 9

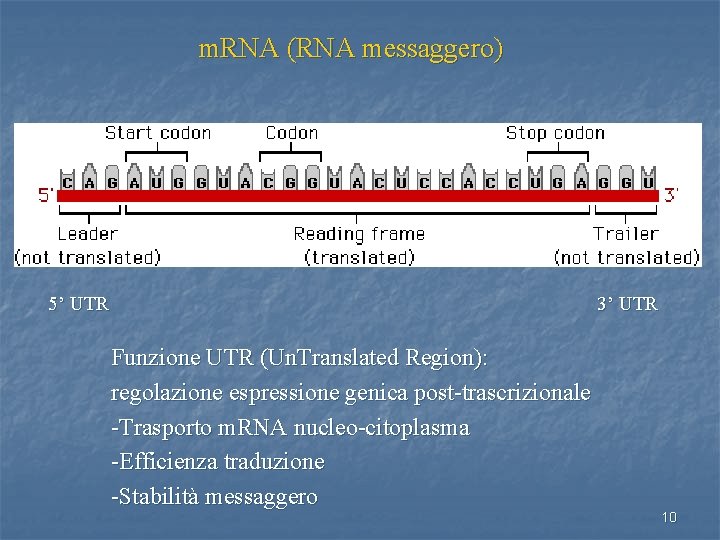
m. RNA (RNA messaggero) 5’ UTR 3’ UTR Funzione UTR (Un. Translated Region): regolazione espressione genica post-trascrizionale -Trasporto m. RNA nucleo-citoplasma -Efficienza traduzione -Stabilità messaggero 10

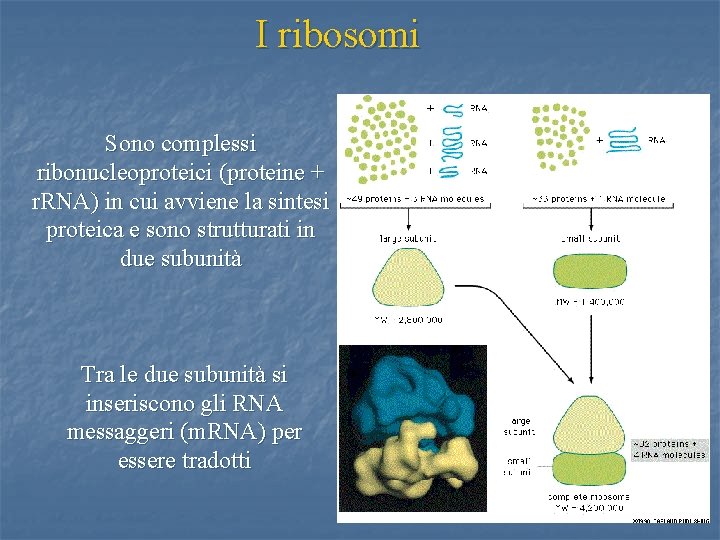
I ribosomi Sono complessi ribonucleoproteici (proteine + r. RNA) in cui avviene la sintesi proteica e sono strutturati in due subunità Tra le due subunità si inseriscono gli RNA messaggeri (m. RNA) per essere tradotti 11

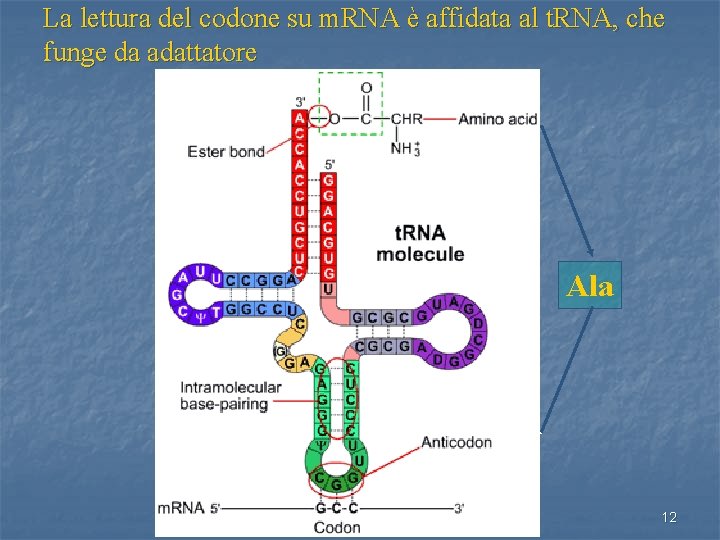
La lettura del codone su m. RNA è affidata al t. RNA, che funge da adattatore Ala 12

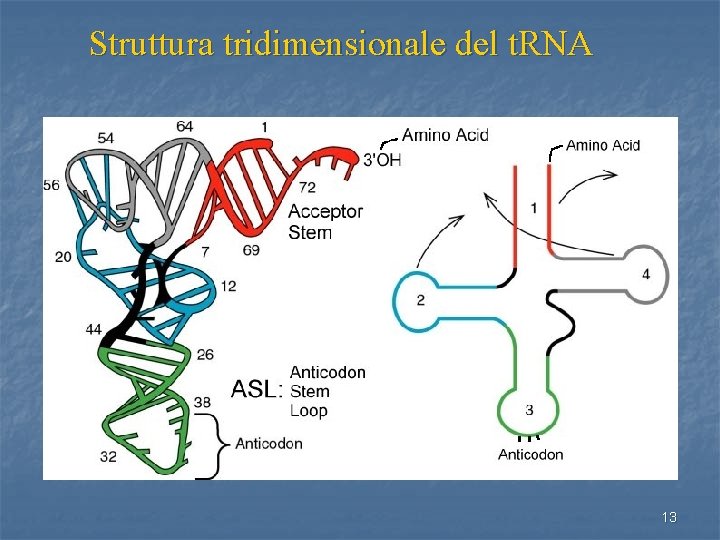
Struttura tridimensionale del t. RNA 13

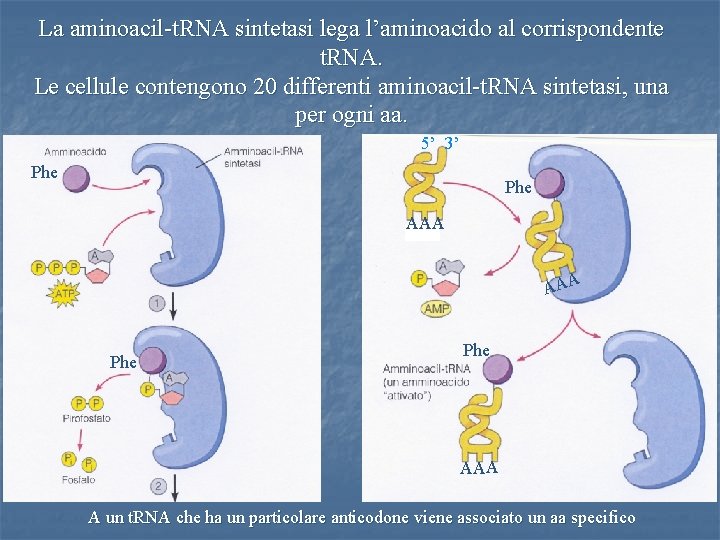
La aminoacil-t. RNA sintetasi lega l’aminoacido al corrispondente t. RNA. Le cellule contengono 20 differenti aminoacil-t. RNA sintetasi, una per ogni aa. 5’ 3’ Phe AAA 14 A un t. RNA che ha un particolare anticodone viene associato un aa specifico

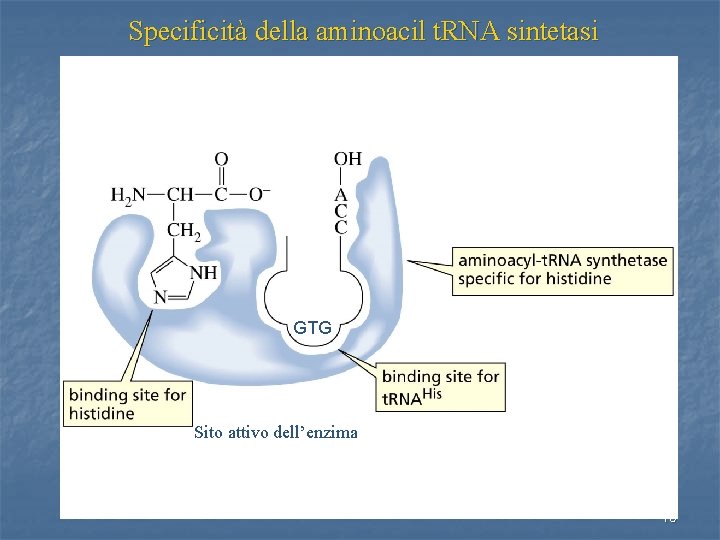
Specificità della aminoacil t. RNA sintetasi GTG Sito attivo dell’enzima 15

Aminoacidi e codoni 20 sono gli aminoacidi che formano le proteine 61 sono le triplette o “codoni” del codice genetico universale Esistono tanti t. RNA quanti sono i codoni? Se ciascuna molecola di t. RNA si appaiasse con il codone dell’m. RNA utilizzando le regole dell’appaiamento canonico tra basi complementari, sarebbero necessari 61 tipi di t. RNA. Poiché la maggior parte degli organismi ha < 45 tipi di t. RNA (48 nell’uomo, 31 nei batteri), questo significa che alcuni t. RNA sono in grado di appaiarsi con più di un codone. 16

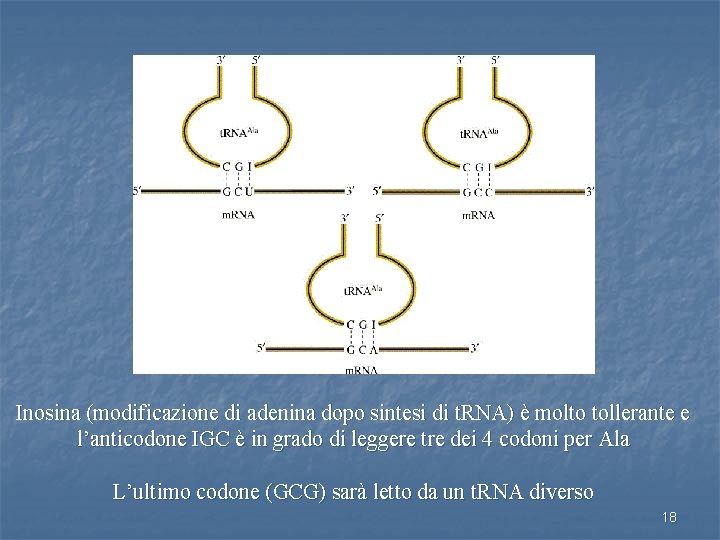
Esempio t. RNA Ala 5’-GCA-3’ 5’-GCC-3’ 5’-GCU-3’ 5’-GCG-3’ codoni Alanina 4 t. RNA con anticodoni diversi per Ala? codoni 5’-GCA-3’ 5’-GCC-3’ 5’-GCU-3’ 5’GCG-3’ 3’-CGU-5’ 3’-CGG-5’ 3’-CGA-5’ 3’CGC-5’ anticodoni 17

Inosina (modificazione di adenina dopo sintesi di t. RNA) è molto tollerante e l’anticodone IGC è in grado di leggere tre dei 4 codoni per Ala L’ultimo codone (GCG) sarà letto da un t. RNA diverso 18

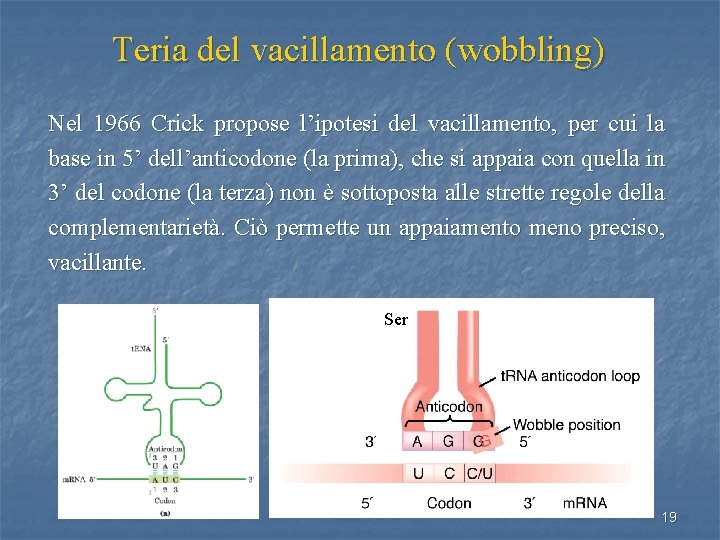
Teria del vacillamento (wobbling) Nel 1966 Crick propose l’ipotesi del vacillamento, per cui la base in 5’ dell’anticodone (la prima), che si appaia con quella in 3’ del codone (la terza) non è sottoposta alle strette regole della complementarietà. Ciò permette un appaiamento meno preciso, vacillante. Ser 19

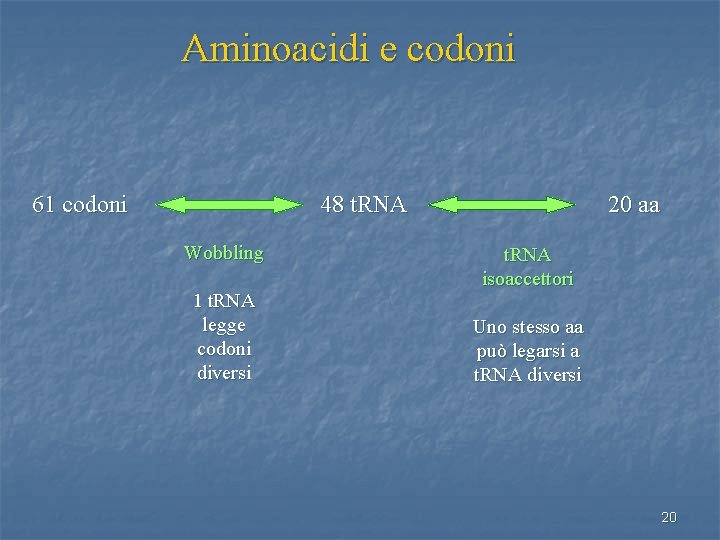
Aminoacidi e codoni 61 codoni 48 t. RNA Wobbling 1 t. RNA legge codoni diversi 20 aa t. RNA isoaccettori Uno stesso aa può legarsi a t. RNA diversi 20

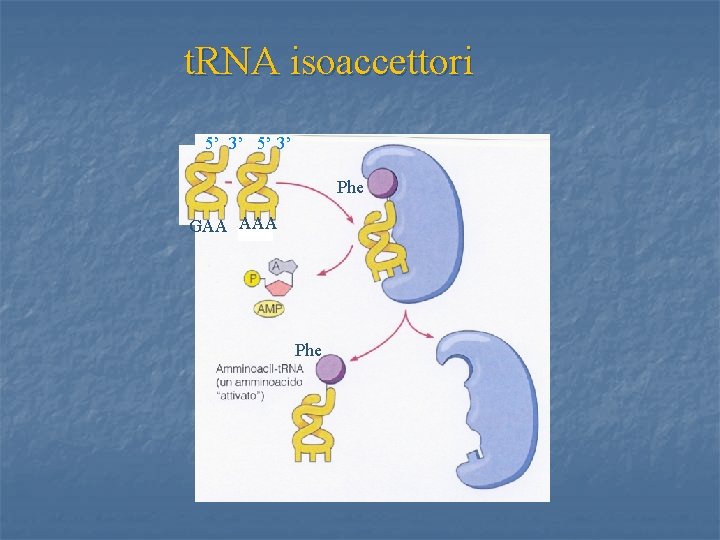
t. RNA isoaccettori 5’ 3’ Phe GAA AAA Phe 21

I codoni: UAA, UAG, UGA sono detti codoni di terminazione o codoni nonsenso perché non vengono riconosciuti da nessun anticodone complementare: Sono segnali di terminazione della traduzione 22

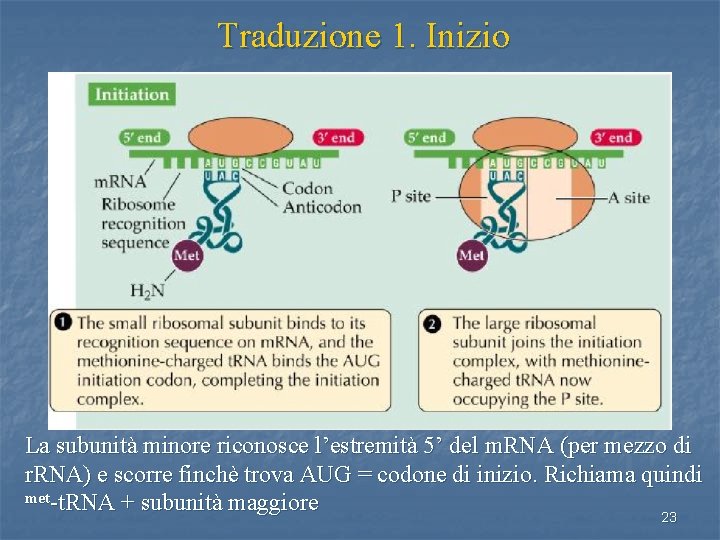
Traduzione 1. Inizio La subunità minore riconosce l’estremità 5’ del m. RNA (per mezzo di r. RNA) e scorre finchè trova AUG = codone di inizio. Richiama quindi met-t. RNA + subunità maggiore 23

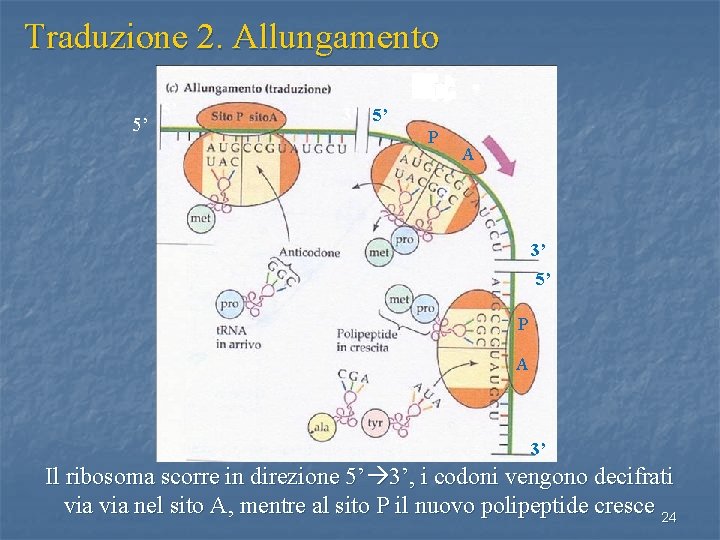
Traduzione 2. Allungamento 5’ 5’ 3’ 5’ P A 3’ Il ribosoma scorre in direzione 5’ 3’, i codoni vengono decifrati via nel sito A, mentre al sito P il nuovo polipeptide cresce 24

L’attività peptidil transferasica della subunità maggiore del ribosoma è dovuta all’ r. RNA 23 S Ribozima: RNA con attività catalitica 25

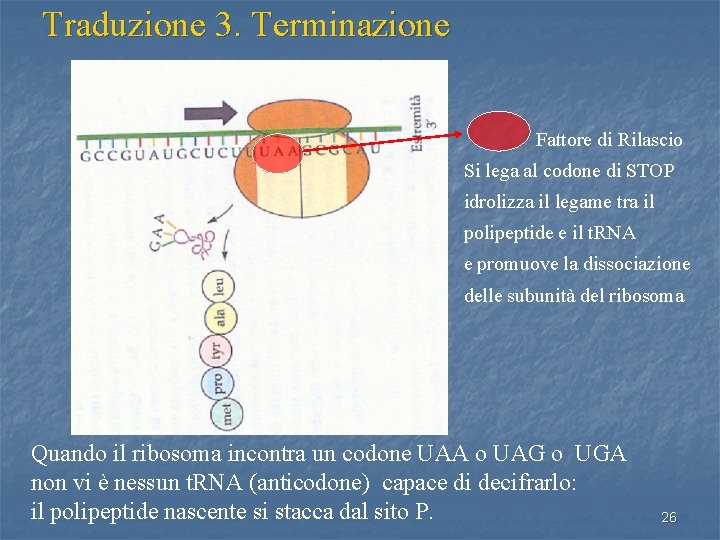
Traduzione 3. Terminazione P A Fattore di Rilascio Si lega al codone di STOP idrolizza il legame tra il polipeptide e il t. RNA e promuove la dissociazione delle subunità del ribosoma Quando il ribosoma incontra un codone UAA o UAG o UGA non vi è nessun t. RNA (anticodone) capace di decifrarlo: il polipeptide nascente si stacca dal sito P. 26

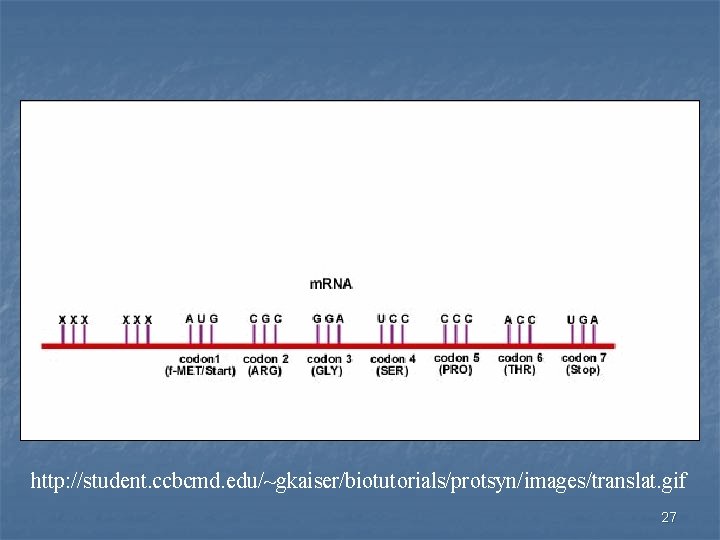
http: //student. ccbcmd. edu/~gkaiser/biotutorials/protsyn/images/translat. gif 27

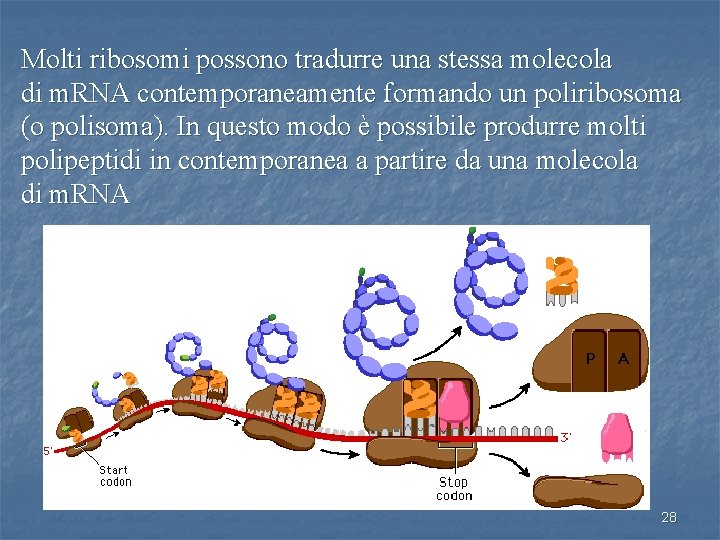
Molti ribosomi possono tradurre una stessa molecola di m. RNA contemporaneamente formando un poliribosoma (o polisoma). In questo modo è possibile produrre molti polipeptidi in contemporanea a partire da una molecola di m. RNA 28

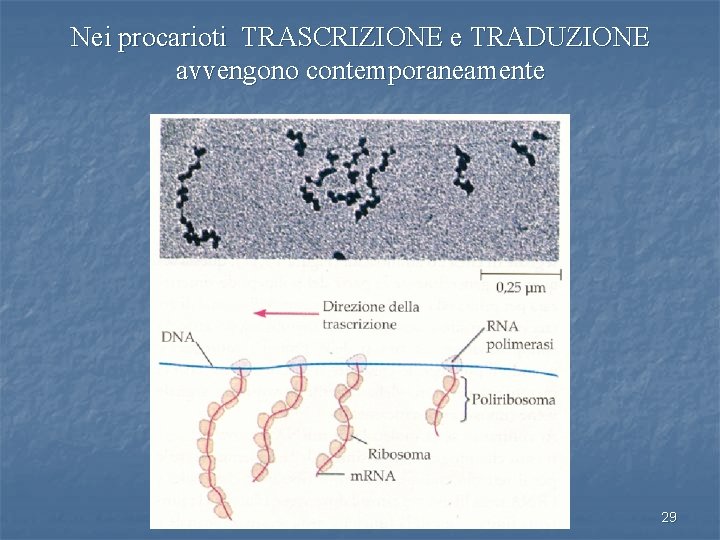
Nei procarioti TRASCRIZIONE e TRADUZIONE avvengono contemporaneamente 29

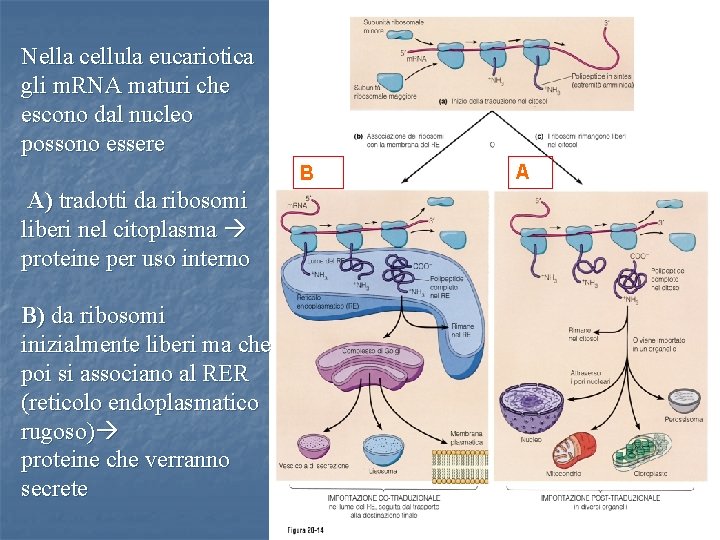
Nella cellula eucariotica gli m. RNA maturi che escono dal nucleo possono essere B A A) tradotti da ribosomi liberi nel citoplasma proteine per uso interno B) da ribosomi inizialmente liberi ma che poi si associano al RER (reticolo endoplasmatico rugoso) proteine che verranno secrete 30

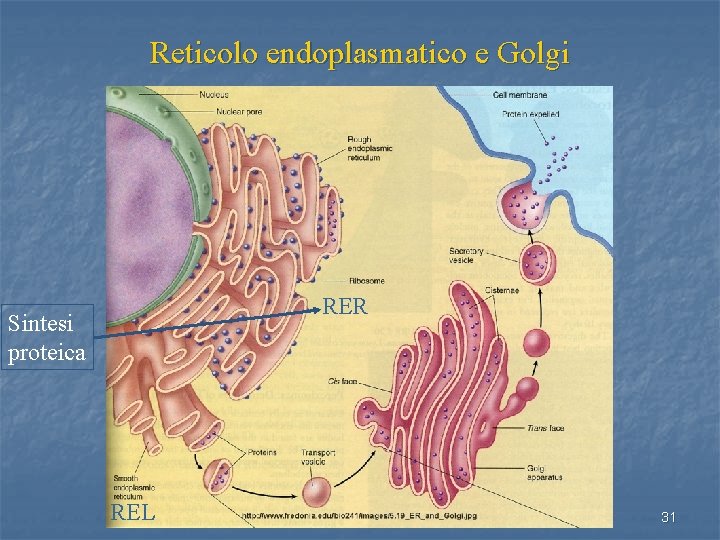
Reticolo endoplasmatico e Golgi RER Sintesi proteica REL 31

Una volta sintetizzate le catene polipeptidiche subiscono varie modificazioni post-traduzionali mediante le quali si trasformano in proteine funzionanti 33

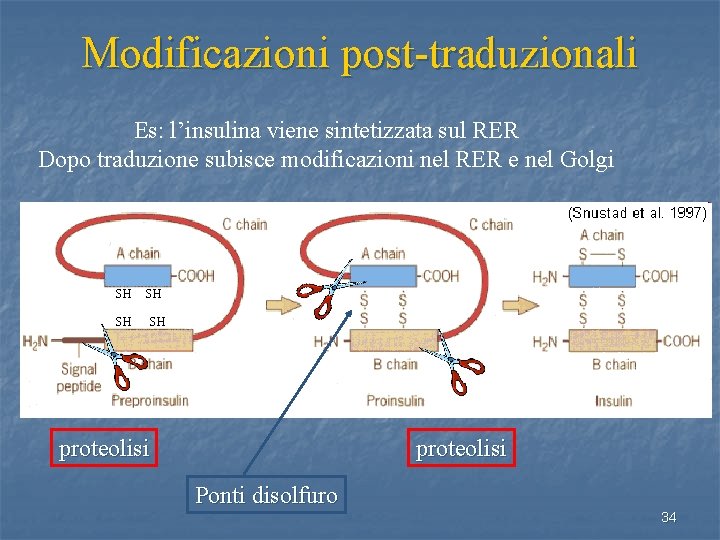
Modificazioni post-traduzionali Es: l’insulina viene sintetizzata sul RER Dopo traduzione subisce modificazioni nel RER e nel Golgi SH SH proteolisi Ponti disolfuro 34
 La sintesi proteica si arresta quando
La sintesi proteica si arresta quando Replicazione dna
Replicazione dna Virusurile definitie
Virusurile definitie Tiaminia
Tiaminia Pressão hidrostática
Pressão hidrostática Síntese proteíca
Síntese proteíca Célula pancreática
Célula pancreática Cellule eucariotiche
Cellule eucariotiche Algoritmos genéticos ejemplos resueltos python
Algoritmos genéticos ejemplos resueltos python Modelo funcionalista de influencia social
Modelo funcionalista de influencia social Codigo genetico
Codigo genetico Transcrio
Transcrio Que es un acervo genetico
Que es un acervo genetico Localização do material genetico
Localização do material genetico Las celulas procariotas tienen nucleo
Las celulas procariotas tienen nucleo Código genético
Código genético Cadena molde de adn
Cadena molde de adn Miglioramento genetico animale
Miglioramento genetico animale Codigo genetico ambiguo
Codigo genetico ambiguo Que es un acervo genetico
Que es un acervo genetico Estructura del material genético
Estructura del material genético Doping ematico e genetico
Doping ematico e genetico Linea de tiempo de la batalla de carabobo
Linea de tiempo de la batalla de carabobo Cistenil
Cistenil Flujo genético ejemplos
Flujo genético ejemplos Codigo genetico
Codigo genetico Dna
Dna Poros nucleares
Poros nucleares Melhoramento genetico
Melhoramento genetico Polimorfismo genetico
Polimorfismo genetico Flujo de genes ejemplos
Flujo de genes ejemplos Digital hub zucchetti non sincronizza
Digital hub zucchetti non sincronizza Codice palatino 418
Codice palatino 418 Codice deontologico oss slide
Codice deontologico oss slide