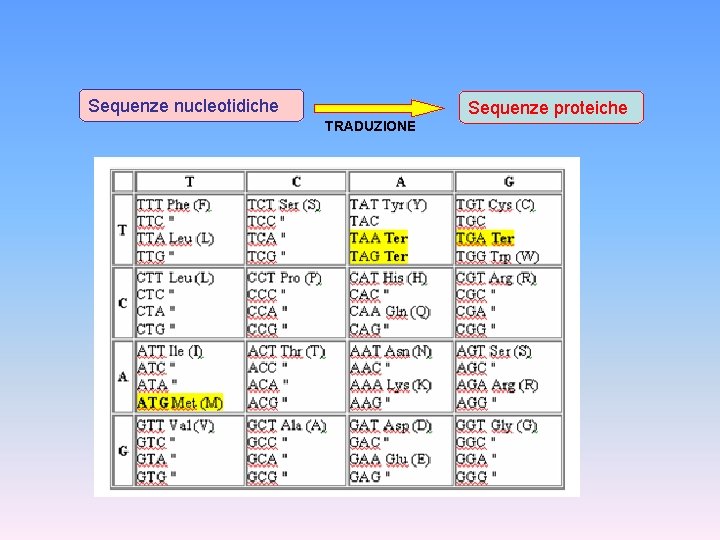
Sequenze nucleotidiche Sequenze proteiche TRADUZIONE Esercizio relativo alla





























- Slides: 51

Sequenze nucleotidiche Sequenze proteiche TRADUZIONE

Esercizio relativo alla traduzione di una sequenza nucleotidica in una sequenza amminoacidica Data la sequenza del gene della b-emoglobina umana: … ctggcccacaagtatcactac… 1)Scrivere la traduzione di questa sequenza in una sequenza amminoacidica 2)Scrivere la sequenza nucleotidica per un cambiamento di una singola base che produca una mutazione silente in questa regione (la mutazione silete è quella che lascia invariata la sequenza amminoacidica) 3)Scrivere la sequenza nucleotidica e la traduzione in sequenza amminoacidica per un cambiamento di una singola base che produca una mutazione di un amminoacido.

Ricerca di pattern e di motivi funzionali Qualche definizione necessaria ……. Un motivo di interesse biologico è costituito da un insieme di caratteri (nucleotidi o amminoacidi) non necessariamente contigui nella sequenza ma che si trovano sempre o sono spesso associati ad una precisa struttura e funzione biologica (ad esempio: promotori o hanno la stessa capacità di legare nucleotidi) La bioinformatica si occupa di sviluppare metodi per il riconoscimento di pattern di interesse biologico e di curare banche dati in cui tali pattern siano organizzati e resi disponibili per l’analisi strutturale e funzionale di nuove sequenze. Ciò deriva dal fatto che nel corso dell’evoluzione la natura ha sviluppato uno o pochi modi per erealizzare una nuova funzione (ad es. attività catalitica o altro)

Per quanto riguarda la Ricerca di pattern e motivi funzionali in sequenze nucleotidiche Ricercare i siti di giunzione tra introni ed esoni Un gene è costituito da una sequenza codificante interrotta da sequenze non codificanti (dette introni). I geni sono combinazioni di corti esoni ed introni di lunghezza variabile. Il termine esoni si applica a tutte le regioni che non sono eliminate nel corso di maturazione del RNA [cioè le regioni non tradotte al 5’ dei geni, quelle codificanti vere e proprie (CDS) e le regioni non tradotte al 3’]. Identificare i siti di giunzione tra introni ed esoni per una corretta predizione della struttura di un gene. Net. Gene: http: //genome. cbs. dtu. dk/services/Net. Gene 2/ Gen. Scan: http: //genes. mit. edu/GENSCAN. html Gen. Scan è il programma più usato per predire la struttura di un gene


Esercizio 1: Predizioni dei geni codificanti proteine in sequenze genomiche mediante Gen. Scan Ricerchiamo i geni in una sequenza genomica prodotta nell’ambito del progetto di sequenziamento del genoma di Fugu. Collegandosi al sito: http: //fugu. hgmp. mrc. ac. uk/fugu-bin/clonesearch/ si effettua la ricerca della sequenza scaffold S 004519. La sequenza così estratta può essere utilizzata per la predizione utilizzando il programma Gen. Scan. La sequenza estratta viene incollata nella box clicca su Run Gen. Scan Nell’output di Gen. Scan sono indicati tutti i geni predetti, per ciascuno dei quali viene riportata la corrispondente ipotetica sequenza amminoacidica. Queste sequenze possono essere caratterizzati: o effettuando una ricerca con BLAST contro la banca dati delle proteine o ricercando i domini o motivi funzionali attraverso il sistema Inter. Pro

Esercizio 2: Stabilire con precisione la struttura di uno specifico gene usando Genome. Scan (http: //genes. mit. edu/genomescan. html) A partire dalla sequenza genomica (S 004519) e dalla proteina omologa selezionata con la più alta percentuale di identità di sequenza Run. Genome. Scan

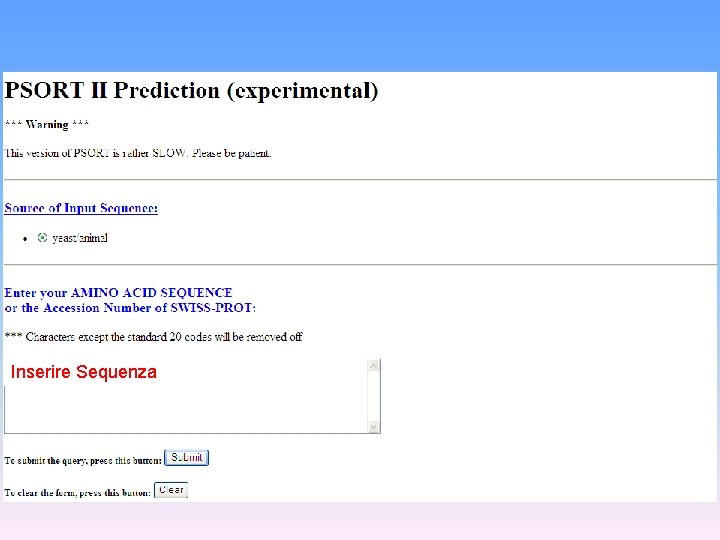
PSORT (http: //psort. nibb. ac. jp/form 2. html) è una procedura per la predizione della localizzazione delle proteine nella cellula. Riceve informazioni sottoforma di sequenze proteiche associate a localizzazioni subcellulari e ne ricava regole di associazione empiriche. Applicando queste regole ad una sequenza proteica di localizzazione ignota, PSORT giunge a predire la localizzazione, fornendo anche un indice di affidabilità della predizione.

Inserire Sequenza


Predizione della Struttura terziaria delle proteine

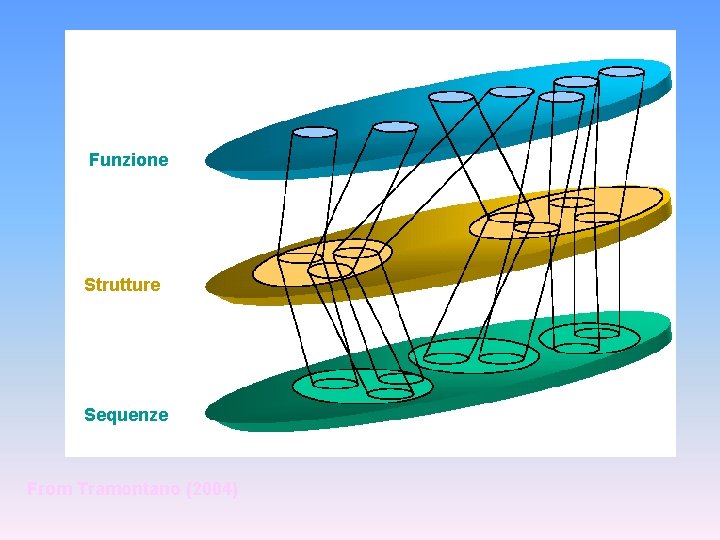
Funzione Strutture Sequenze From Tramontano (2004)




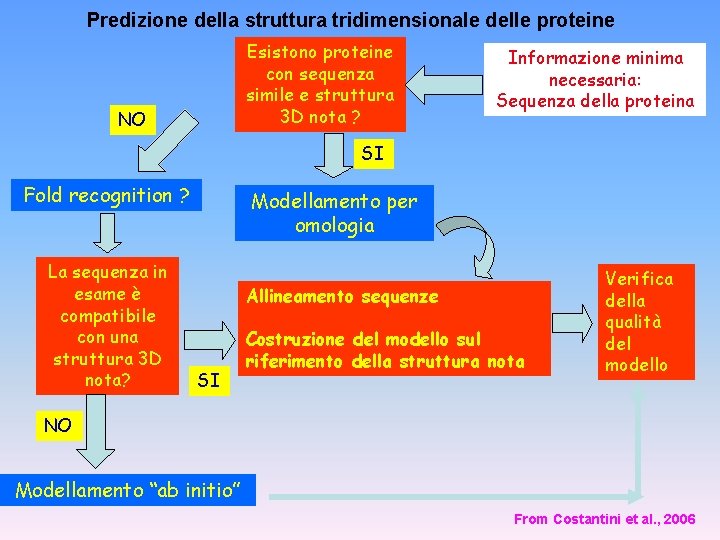
Predizione della struttura tridimensionale delle proteine Esistono proteine con sequenza simile e struttura 3 D nota ? NO Informazione minima necessaria: Sequenza della proteina SI Fold recognition ? La sequenza in esame è compatibile con una struttura 3 D nota? Modellamento per omologia Allineamento sequenze SI Costruzione del modello sul riferimento della struttura nota Verifica della qualità del modello NO Modellamento “ab initio” From Costantini et al. , 2006

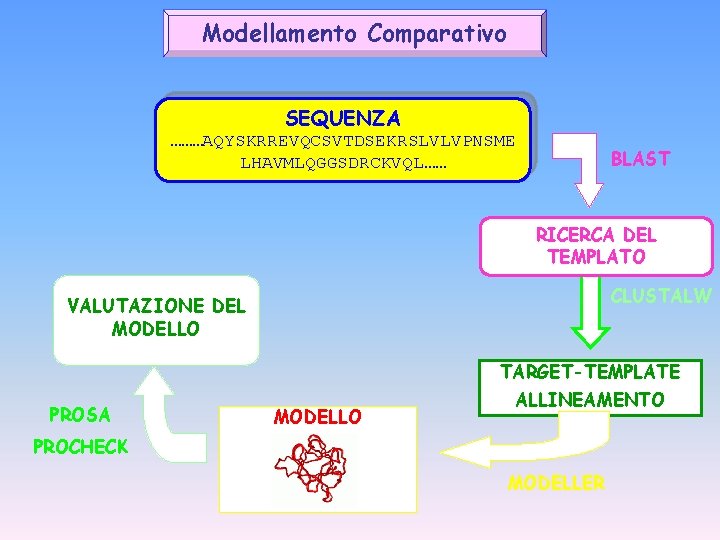
Modellamento Comparativo SEQUENZA ………AQYSKRREVQCSVTDSEKRSLVLVPNSME LHAVMLQGGSDRCKVQL…… BLAST RICERCA DEL TEMPLATO CLUSTALW VALUTAZIONE DEL MODELLO TARGET-TEMPLATE PROSA MODELLO ALLINEAMENTO PROCHECK MODELLER

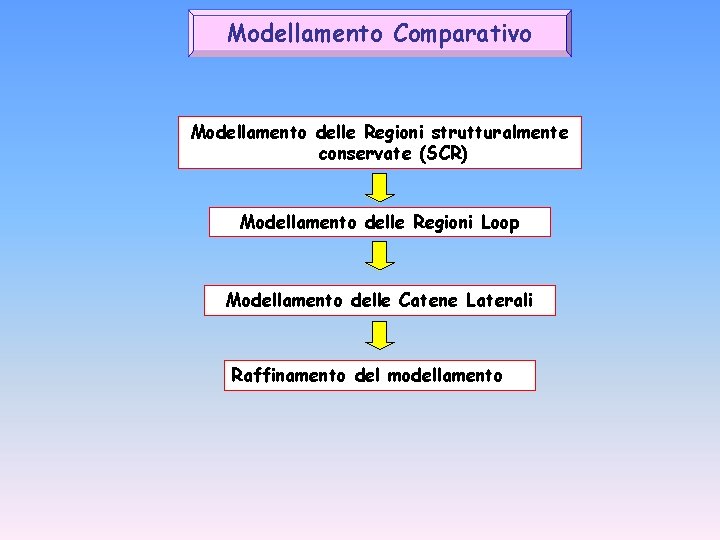
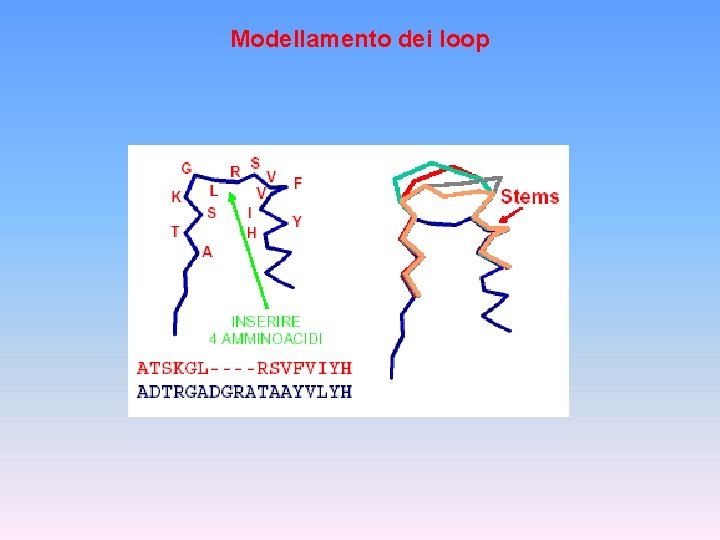
Modellamento Comparativo Modellamento delle Regioni strutturalmente conservate (SCR) Modellamento delle Regioni Loop Modellamento delle Catene Laterali Raffinamento del modellamento

Modellamento dei loop

Modellamento per omologia SWISSMODELER Server automatici: SPARKS 2 SP 3 3 D-JIGSAW Metodi per valutare un modello ottenuto per omologia


Server che utilizza Modeller

3 D-JIGSAW

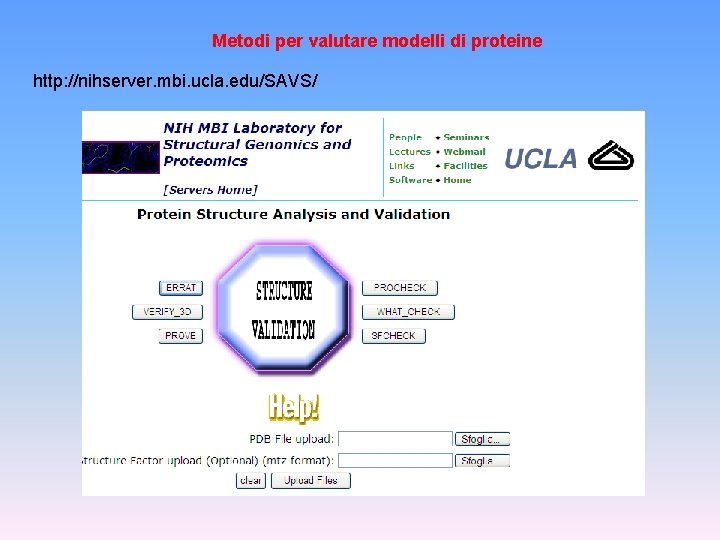
Metodi per valutare modelli di proteine http: //nihserver. mbi. ucla. edu/SAVS/

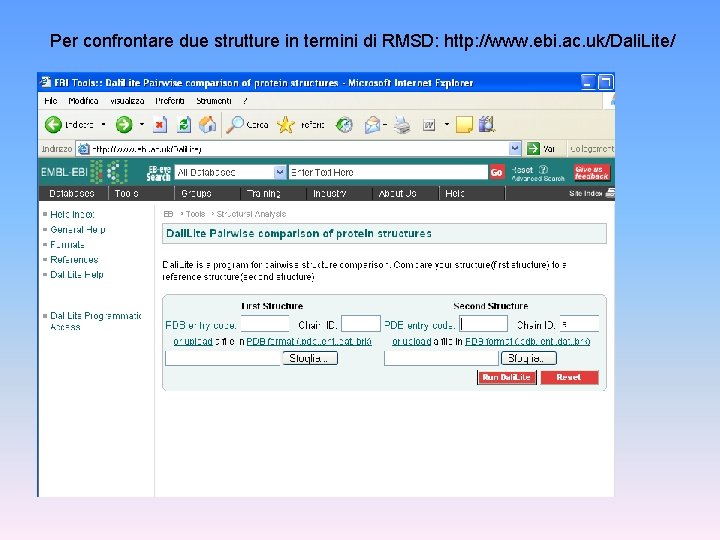
Per confrontare due strutture in termini di RMSD: http: //www. ebi. ac. uk/Dali. Lite/

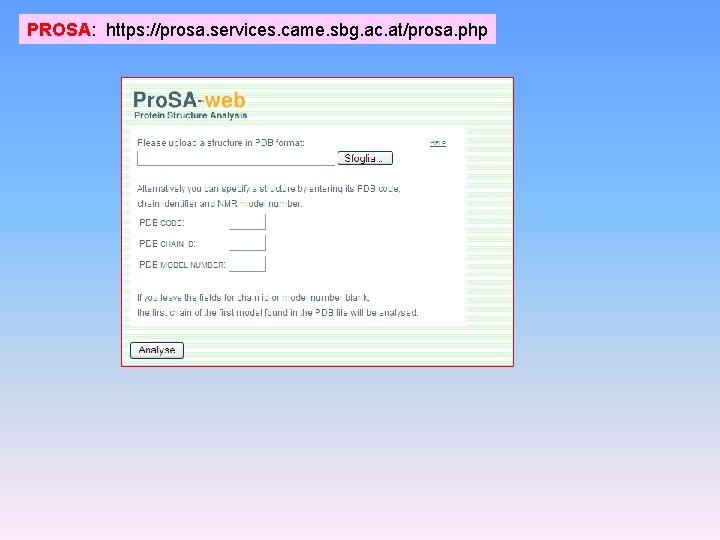
PROSA: https: //prosa. services. came. sbg. ac. at/prosa. php



Fold Recognition

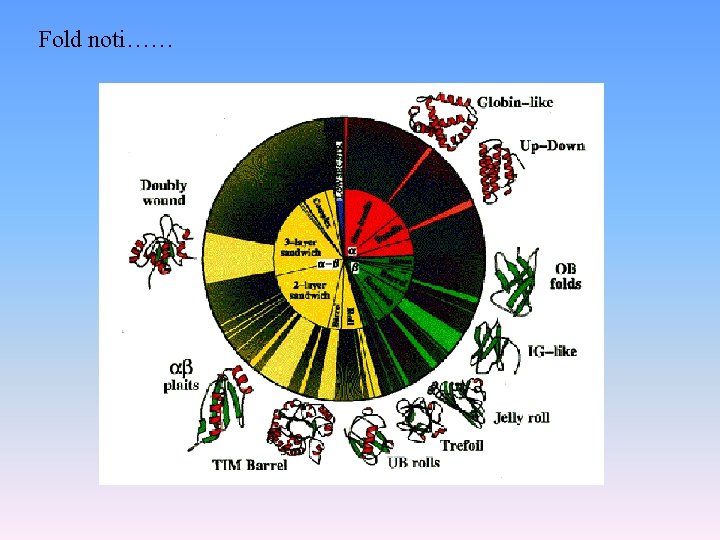
Fold noti……

Programmi per costruire modelli per riconoscimento del fold: 3 D-PSSM Based on sequence profiles, solvatation potentials and secondary structure http: //www. sbg. bio. ic. ac. uk/~3 dpssm/index 2. html FUGUE uses environment-based fold profiles that are created from structural alignments http: //www-cryst. bioc. cam. ac. uk/fugue/ SAMT 02 http: //www. soe. ucsc. edu/research/compbio/HMM-apps/T 02 -query. html The query is checked against a library of hidden Markov models. This is NOT a threading technique, it is sequence based. FFAS 03 (http: //ffas. ljcrf. edu/ffas-cgi/ffas. pl)This is a variant of FFAS, which aligns profile to profile. Profiles are generated differently from PSI-BLAST

THREADER può essere scaricato sul proprio computer


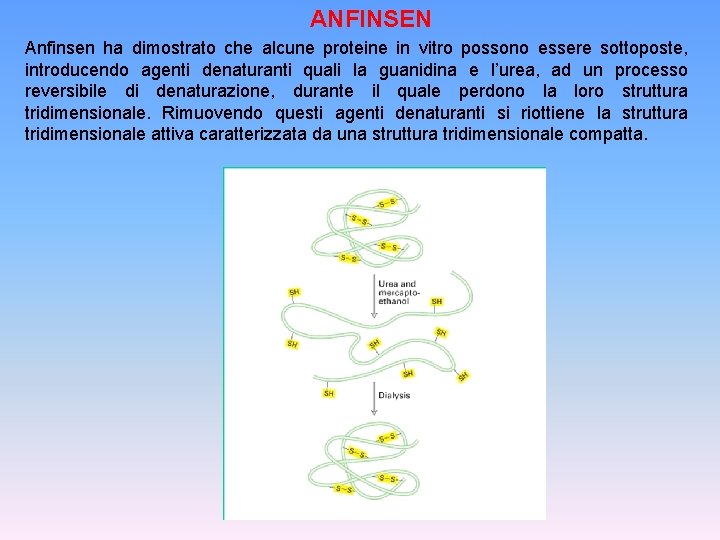
ANFINSEN Anfinsen ha dimostrato che alcune proteine in vitro possono essere sottoposte, introducendo agenti denaturanti quali la guanidina e l’urea, ad un processo reversibile di denaturazione, durante il quale perdono la loro struttura tridimensionale. Rimuovendo questi agenti denaturanti si riottiene la struttura tridimensionale attiva caratterizzata da una struttura tridimensionale compatta.

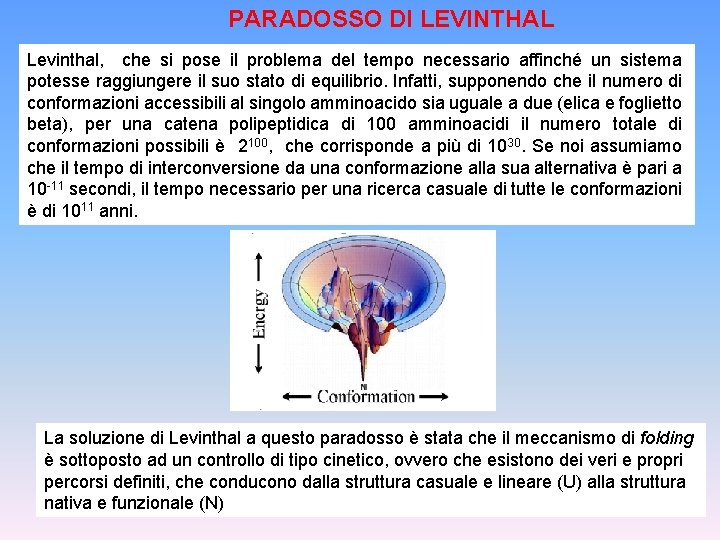
PARADOSSO DI LEVINTHAL Levinthal, che si pose il problema del tempo necessario affinché un sistema potesse raggiungere il suo stato di equilibrio. Infatti, supponendo che il numero di conformazioni accessibili al singolo amminoacido sia uguale a due (elica e foglietto beta), per una catena polipeptidica di 100 amminoacidi il numero totale di conformazioni possibili è 2100, che corrisponde a più di 1030. Se noi assumiamo che il tempo di interconversione da una conformazione alla sua alternativa è pari a 10 -11 secondi, il tempo necessario per una ricerca casuale di tutte le conformazioni è di 1011 anni. La soluzione di Levinthal a questo paradosso è stata che il meccanismo di folding è sottoposto ad un controllo di tipo cinetico, ovvero che esistono dei veri e propri percorsi definiti, che conducono dalla struttura casuale e lineare (U) alla struttura nativa e funzionale (N)

Robetta server (http: //robetta. bakerlab. org)

HMMSTR: http: //www. bioinfo. rpi. edu/~bystrc/hmmstr/server. php


DOCKING RIGIDO DOCK AUTO-DOCK

DOCKING FLESSIBILE

Ci sono vari programmi di docking: Dock, Auto. Dock, Flex. X Questi programmi si basano sulla complementarietà geometrica e chimico-fisica tra le strutture, considerando spesso anche termini energetici ed entropici.

Clus. Pro: http: //nrc. bu. edu/cluster/

Z-Dock (http: //zlab. bu. edu/zdock/) può essere scaricato sul proprio computer

Z-Dock su server: http: //zdock. bu. edu/

http: //bioinfo 3 d. cs. tau. ac. il/Patch. Dock/

http: //vakser. bioinformatics. ku. edu/resources/grammx

http: //www. csd. abdn. ac. uk/hex_server/

Protein Server valuta gli amminoacidi all’interfaccia Il programma DCOMPLEX valuta l’affinità di legame tra molecole complessate http: //sparks. informatics. iuoui. edu/czhang/complex. html



ESERCIZIO Prendendo come input il file del recettore e del ligando sul sito, provare ad eseguire un docking con i programmi visti prima.
 Letra banderita española
Letra banderita española Mas alla de mi inseguridad letra
Mas alla de mi inseguridad letra Schema sp civilistico
Schema sp civilistico Esercizio abusivo della professione
Esercizio abusivo della professione Esercizi sui fusi orari
Esercizi sui fusi orari Esercizio
Esercizio Esercizio analitico significato
Esercizio analitico significato Esercizio
Esercizio Esercizi tabella pivot
Esercizi tabella pivot Bilancio d'esercizio
Bilancio d'esercizio Esercizio fisico ipertensione arteriosa polmonare
Esercizio fisico ipertensione arteriosa polmonare Schemi bilancio oic
Schemi bilancio oic Il bilancio d'esercizio
Il bilancio d'esercizio Esercizio modello er
Esercizio modello er Nusaf traduzione
Nusaf traduzione Potams
Potams Intertextuality examples in newspapers
Intertextuality examples in newspapers Traduzione
Traduzione Ille mi par esse deo videtur traduzione
Ille mi par esse deo videtur traduzione Sao ko kelle terre
Sao ko kelle terre Plush traduzione
Plush traduzione Bootstrap traduzione informatica
Bootstrap traduzione informatica Concatenamero significato
Concatenamero significato Point of care definition
Point of care definition Dativo di effetto
Dativo di effetto Lagging indicators traduzione
Lagging indicators traduzione Ham honge kamyab
Ham honge kamyab Am 12 dezember werther traduzione
Am 12 dezember werther traduzione Praebo latino
Praebo latino Come riconoscere gerundio e gerundivo
Come riconoscere gerundio e gerundivo Sedi organi costituzionali italiani
Sedi organi costituzionali italiani Warehousing traduzione
Warehousing traduzione Screenshot traduzione italiano
Screenshot traduzione italiano Seed traduzione informatica
Seed traduzione informatica Language traduzione
Language traduzione Donna angelo di montale
Donna angelo di montale Beloved horses hateful man riassunto
Beloved horses hateful man riassunto Andra moi ennepe musa
Andra moi ennepe musa Chunking traduzione
Chunking traduzione Rna
Rna Foundation subjects traduzione
Foundation subjects traduzione Johanna monti
Johanna monti Deus, in adiutorium meum intende
Deus, in adiutorium meum intende Is ea id traduzione
Is ea id traduzione Analisi sequenziale
Analisi sequenziale Wenn die haifische menschen wären interpretation
Wenn die haifische menschen wären interpretation Vivat lingua
Vivat lingua Paginazione gerarchica
Paginazione gerarchica Traduzione
Traduzione Yes i will yes joyce
Yes i will yes joyce Aste per tende a molla
Aste per tende a molla Primus amor phoebi daphne
Primus amor phoebi daphne