A A 2015 2016 CORSO INTEGRATO DI INFORMATICA















- Slides: 35

A. A. 2015 -2016 CORSO INTEGRATO DI INFORMATICA E BIOINFORMATICA per il CLT in BIOLOGIA MOLECOLARE Scuola di Scienze, Università di Padova Docenti: Prof. Alessandro Sperduti (Informatica) Prof. Stefania Bortoluzzi (Bioinformatica)

WORKING WITH BIOSEQUENCES Alignments and similarity search

WORKING WITH BIOSEQUENCES Alignments and similarity search • Traduzione di sequenze di DNA • Ricerca di similarità • BLAST

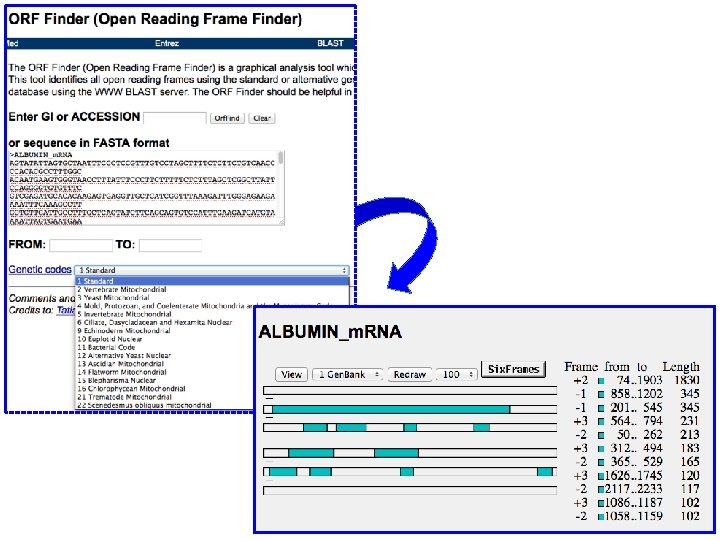
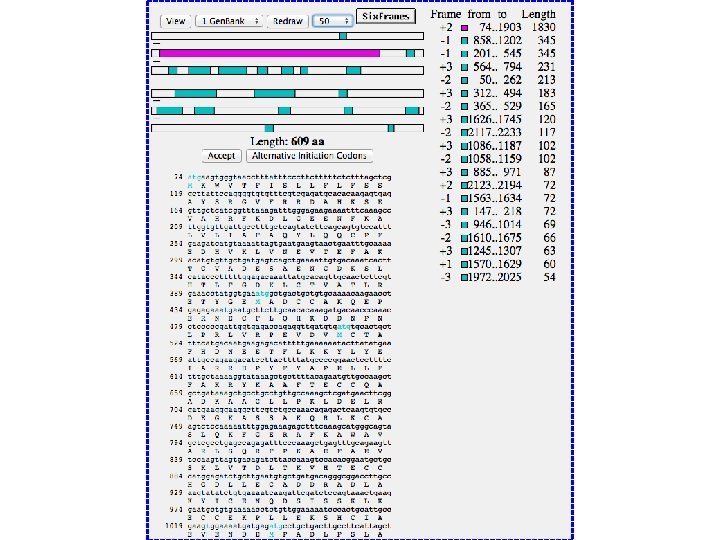
TRADUZIONE DI SEQUENZE DI DNA >ALBUMIN_m. RNA AGTATATTAGTGCTAATTTCCCTCCGTTTGTCCTAGCTTTTCTCTTCTGTCAACCCCACACGCCTTTGGC ACAATGAAGTGGGTAACCTTTATTTCCCTTCTTTTTCTCTTTAGCTCGGCTTATTCCAGGGGTGTGTTTC GTCGAGATGCACACAAGAGTGAGGTTGCTCATCGGTTTAAAGATTTGGGAGAAGAAAATTTCAAAGCCTT GGTGTTGATTGCCTTTGCTCAGTATCTTCAGCAGTGTCCATTTGAAGATCATGTAAAATTAGTGAA GTAACTGAATTTGCAAAAACATGTGTTGCTGATGAGTCAGCTGAAAATTGTGACAAATCACTTCATACCC TTTTTGGAGACAAATTATGCACAGTTGCAACTCTTCGTGAAACCTATGGTGAAATGGCTGACTGCTGTGC AAAACAAGAACCTGAGAGAAATGCTTCTTGCAACACAAAGATGACAACCCAAACCTCCCCCGATTG GTGAGACCAGAGGTTGATGTGCACTGCTTTTCATGACAATGAAGAGACATTTTTGAAAAAATACT TATATGAAATTGCCAGAAGACATCCTTACTTTTATGCCCCGGAACTCCTTTTCTTTGCTAAAAGGTATAA AGCTGCTTTTACAGAATGTTGCCAAGCTGCTGATAAAGCTGCCTGTTGCCAAAGCTCGATGAACTT CGGGATGAAGGCTTCGTCTGCCAAACAGAGACTCAAGTGTGCCAGTCTCCAAAAATTTGGAGAAA GAGCTTTCAAAGCATGGGCAGTAGCTCGCCTGAGCCAGAGATTTCCCAAAGCTGAGTTTGCAGAAGTTTC CAAGTTAGTGACAGATCTTACCAAAGTCCACACGGAATGCTGCCATGGAGATCTGCTTGAATGTGCTGAT GACAGGGCGGACCTTGCCAAGTATATCTGTGAAAATCAAGATTCGATCTCCAGTAAACTGAAGGAATGCT GTGAAAAACCTCTGTTGGAAAAATCCCACTGCATTGCCGAAGTGGAAAATGATGAGATGCCTGCTGACTT GCCTTCATTAGCTGCTGATTTTGTTGAAAGTAAGGATGTTTGCAAAAACTATGCTGAGGCAAAGGATGTC TTCCTGGGCATGTTTTTGTATGAATATGCAAGAAGGCATCCTGATTACTCTGTCGTGCTGCTGCTGAGAC TTGCCAAGACATATGAAACCACTCTAGAGAAGTGCTGTGCCGCTGCAGATCCTCATGAATGCTATGCCAA AGTGTTCGATGAATTTAAACCTCTTGTGGAAGAGCCTCAGAATTTAATCAAAATTGTGAGCTTTTT GAGCAGCTTGGAGAGTACAAATTCCAGAATGCGCTATTAGTTCGTTACACCAAGAAAGTACCCCAAGTGT CAACTCTTGTAGAGGTCTCAAGAAACCTAGGAAAAGTGGGCAGCAAATGTTGTAAACATCCTGA AGCAAAAAGAATGCCCTGTGCAGAAGACTATCCGTGGTCCTGAACCAGTTATGTGTGTTGCATGAG AAAACGCCAGTAAGTGACAGAGTCACCAAATGCTGCACAGAATCCTTGGTGAACAGGCGACCATGCTTTT CAGCTCTGGAAGTCGATGAAACATACGTTCCCAAAGAGTTTAATGCTGAAACATTCACCTTCCATGCAGA TATATGCACACTTTCTGAGAAGGAGAGACAAATCAAGAAACTGCACTTGTTGAGCTCGTGAAACAC AAGCCCAAGGCAACAAAAGAGCAACTGAAAGCTGTTATGGATGATTTCGCAGCTTTTGTAGAGAAGTGCT GCAAGGCTGACGATAAGGAGACCTGCTTTGCCGAGGAGGGTAAAAAACTTGTTGCTGCAAGTCAAGCTGC CTTAGGCTTATAACATCACATTTAAAAGCATCTCAGCCTACCATGAGAATAAGAGAAAATGAAGAT CAAAAGCTTATTCATCTGTTTTTCGTTGGTGTAAAGCCAACACCCTGTCTAAAAAACATAAATT TCTTTAATCATTTTGCCTCTTTTCTCTGTGCTTCAATTAATAAAAAATGGAAAGAATCTAATAGAGTGGT ACAGCACTGTTATTTTTCAAAGATGTGTTGCTATCCTGAAAATTCTGTAGGTTCTGTGGAAGTTCCAGTG TTCTCTCTTATTCCACTTCGGTAGAGGATTTCTAGTTTCTTGTGGGCTAATTAAATCATTAATACT CTTCTAAAAAAAAAA

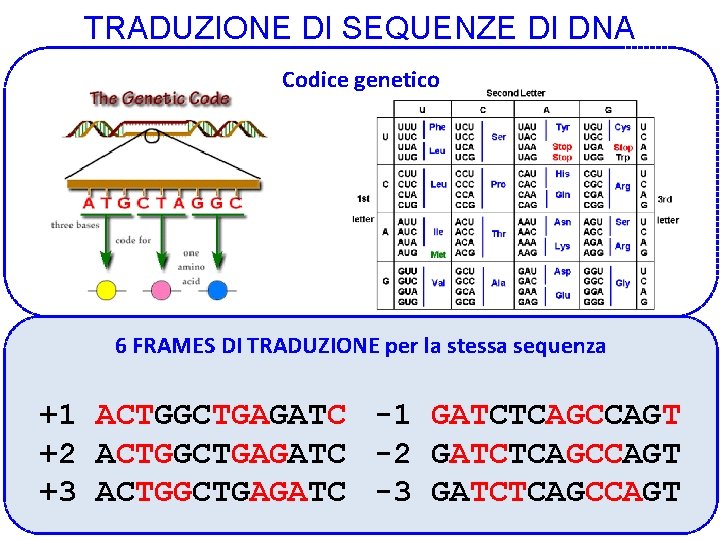
TRADUZIONE DI SEQUENZE DI DNA Codice genetico 6 FRAMES DI TRADUZIONE per la stessa sequenza +1 ACTGGCTGAGATC -1 GATCTCAGCCAGT +2 ACTGGCTGAGATC -2 GATCTCAGCCAGT +3 ACTGGCTGAGATC -3 GATCTCAGCCAGT



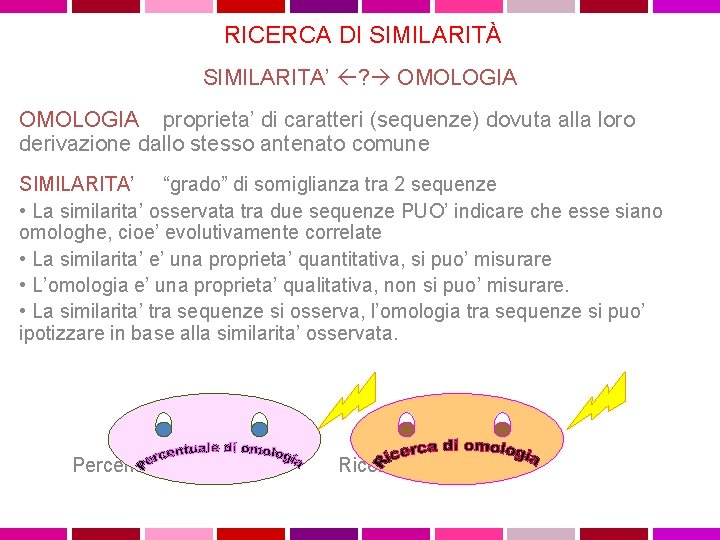
RICERCA DI SIMILARITÀ SIMILARITA’ ? OMOLOGIA proprieta’ di caratteri (sequenze) dovuta alla loro derivazione dallo stesso antenato comune SIMILARITA’ “grado” di somiglianza tra 2 sequenze • La similarita’ osservata tra due sequenze PUO’ indicare che esse siano omologhe, cioe’ evolutivamente correlate • La similarita’ e’ una proprieta’ quantitativa, si puo’ misurare • L’omologia e’ una proprieta’ qualitativa, non si puo’ misurare. • La similarita’ tra sequenze si osserva, l’omologia tra sequenze si puo’ ipotizzare in base alla similarita’ osservata. Percentuale di similarita’ Ricerca di similarita’

OMOLOGIA E OMOPLASIA Omologia similarita’ dovuta a derivazione dallo stesso antenato comune Omoplasia similarita’ dovuta a convergenza, stessa pressione selettiva due linee evolutive puo’ condurre a caratteri simili ORTOLOGIA E PARALOGIA OMOLOGIA ANTENATO COMUNE ORTOLOGIA PARALOGIA PROCESSO DI SPECIAZIONE DUPLICAZIONE GENICA Descrivo le relazioni tra geni di una famiglia intraorganismo (paralogia) o tra diversi organismi (ortologia) su


§ Dimensioni delle banche dati § Ripetitività delle ricerche § Lentezza degli algoritmi “esatti” Sistemi rapidi ma approssimati di allineamento Metodi euristici per l’allineamento • gli algoritmi esatti effettuano delle ricerche esaustive ed esplorano tutto lo spazio degli allineamenti possibili (programmazione dinamica) • si tratta comunque di algoritmi di ordine n 2, ovvero per allineare due sequenze lunghe ognuna 1000 residui, effettuano 1000 x 1000 = un milione di confronti: troppo lenti!!!! • la crescita esponenziale delle dimensioni delle banche dati di sequenze biologiche ha portato allo sviluppo di programmi (come FASTA e BLAST) in grado di effettuare velocemente ricerche di similarità, grazie a soluzioni euristiche sono basate su assunzioni non certe, ma estremamente probabili. In pratica la ricerca è resa più veloce a scapito della certezza di avere veramente trovato la soluzione migliore

BLAST Basic Local Alignment Search Tool (Altschul 1990) L’ algoritmo di BLAST e’ euristico e opera: 1 Tagliando le sequenze da comparare in piccoli pezzi (parole) 2 Ignorando tutte le coppie di parole (sequenza query/database) la cui comparazione da’ un punteggio inferiore ad un limite fissato 3 Cercando di estendere tutte le hits rimanenti sino a che l’allineamento locale raggiunge un certo punteggio Dati una SEQUENZA QUERY ed un DATABASE DI SEQUENZE, BLAST ricerca nel database “parole” di lunghezza almeno “W” con un punteggio di similarita’ di almeno “T” una volta allineate con la sequenza “query” (HSP, High Scoring Pairs). Le “parole” selezionate vengono estese, se possibile, fino a raggiungere un punteggio superiore a “S” oppure un “E-value” inferiore al limite specificato.


1 - Seeding • In sequenze di DNA W = 7 • In sequenze proteiche W = 23

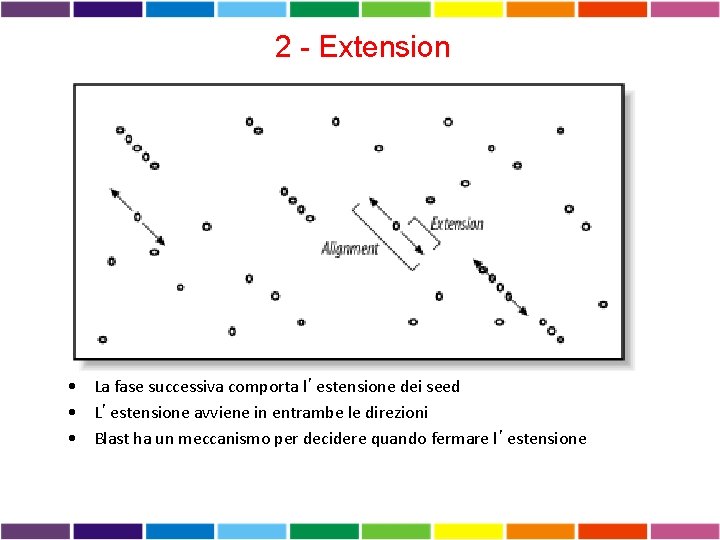
2 - Extension • La fase successiva comporta l’estensione dei seed • L’estensione avviene in entrambe le direzioni • Blast ha un meccanismo per decidere quando fermare l’estensione

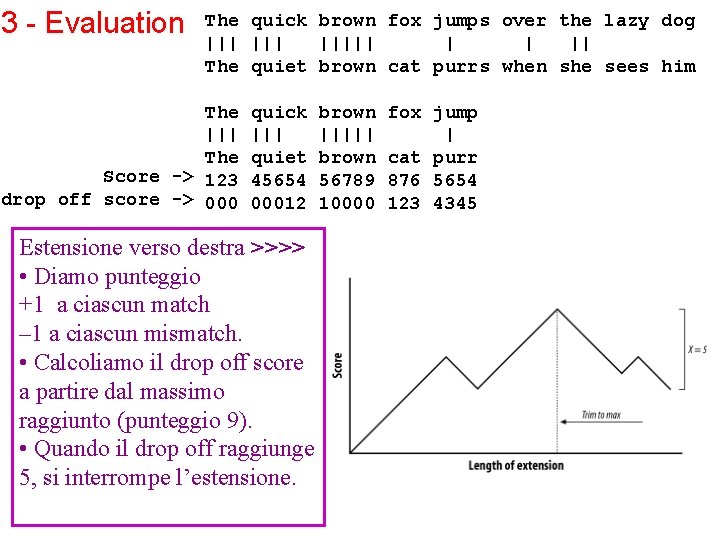
3 - Evaluation The quick brown fox jumps over the lazy dog ||| ||||| | | || The quiet brown cat purrs when she sees him The ||| The Score -> 123 drop off score -> 000 quick ||| quiet 45654 00012 brown ||||| brown 56789 10000 Estensione verso destra >>>> • Diamo punteggio +1 a ciascun match – 1 a ciascun mismatch. • Calcoliamo il drop off score a partire dal massimo raggiunto (punteggio 9). • Quando il drop off raggiunge 5, si interrompe l’estensione. fox jump | cat purr 876 5654 123 4345

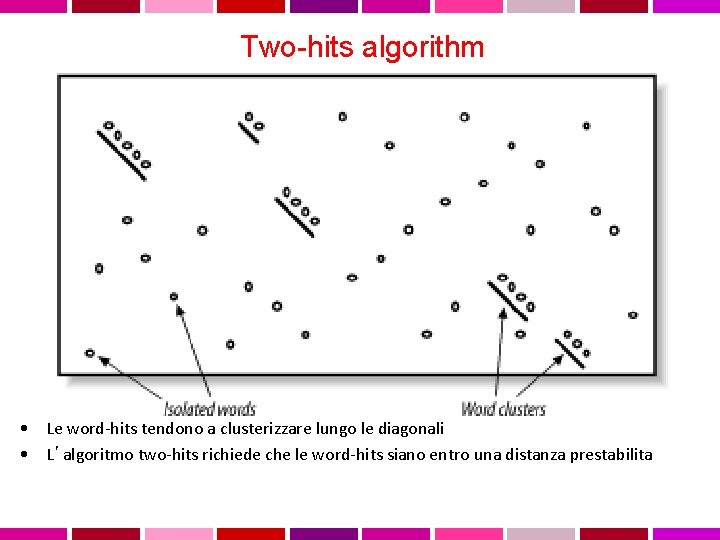
Two-hits algorithm • Le word-hits tendono a clusterizzare lungo le diagonali • L’algoritmo two-hits richiede che le word-hits siano entro una distanza prestabilita

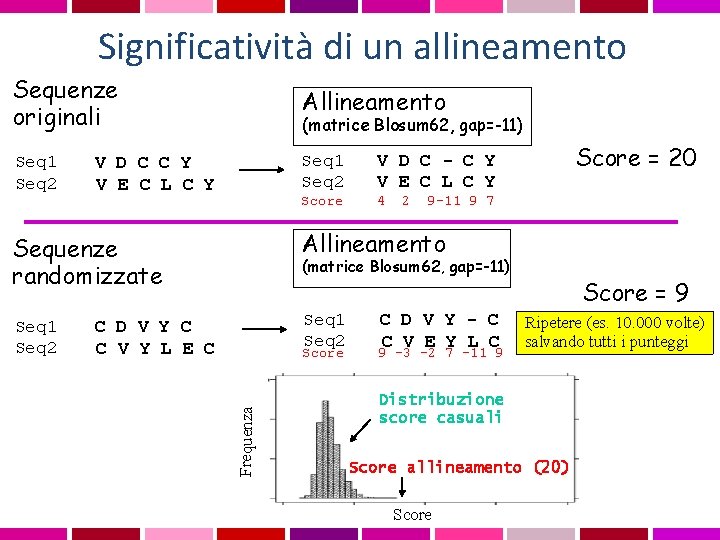
Significatività di un allineamento Sequenze allineate Osservazione Ipotesi alternative ATTGCCCACGTTCGCGATCG ATAGGGCACTTT-GCGATGA ** ***** OMOLOGIA? CASO?

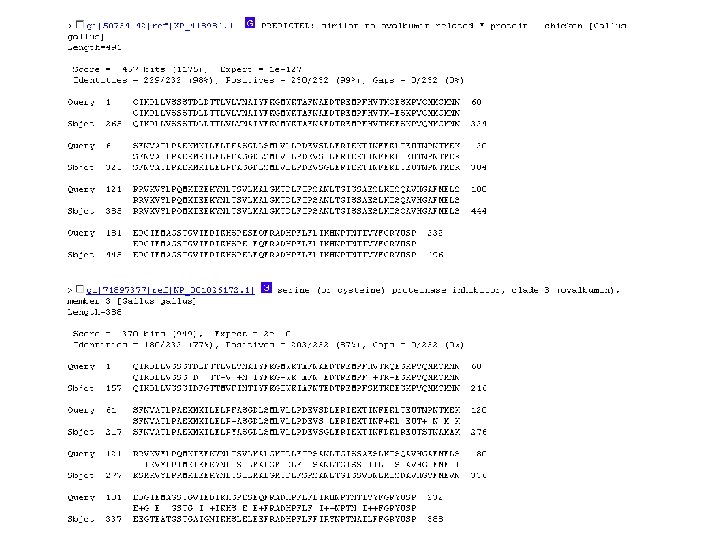
Il risultato di una ricerca di similarita’ e’ una lista dei migliori allineamenti, tra la sequenza query e le sequenze “estratte” dal database. La SIGNIFICATIVITA’ di ciascun allineamento si calcola come P value o E value P value e’ la probabilita’ di ottenere un allineamento con punteggio uguale o migliore di quello osservato Si calcola mettendo in relazione il punteggio osservato (S) con la distribuzione attesa di HSP quando si comparano sequenze random della stessa lunghezza e composizione di quella in analisi (query sequence) Piu’ il P value e’ vicino a 0 piu’ e’ significativo 2 x 10 -245 e’ meglio do 0. 001 !!! E value e’ il numero atteso di allineamenti con punteggio uguale o migliore di quello osservato Piu’ e’ basso piu’ e’ buono

Significatività di un allineamento Sequenze originali Allineamento Seq 1 Seq 2 V D C - C Y V E C L C Y Score 4 (matrice Blosum 62, gap=-11) V D C C Y V E C L C Y 2 Score = 20 9 -11 9 7 Sequenze randomizzate Allineamento Seq 1 Seq 2 (matrice Blosum 62, gap=-11) C D V Y C C V Y L E C Frequenza Score C D V Y - C C V E Y L C 9 -3 -2 7 -11 9 Score = 9 Ripetere (es. 10. 000 volte) salvando tutti i punteggi Distribuzione score casuali Score allineamento (20) Score

Usare BLAST OPZIONI Sequenza query nucleotidica proteica (sequenza in formato FASTA, Gen. Bank Accession numbers o GI numbers) Database di seq. nucleotidiche database di seq. proteiche Programma Standard BLAST (blastn) Standard protein BLAST (blastp) translated blast (blastx, tblastn, tblastx) MEGABLAST PSI-BLAST PHI-BLAST … Blast selection table http: //www. ncbi. nlm. nih. gov/BLAST/producttable. shtml

Usare BLAST database di seq. nucleotidiche nr All Gen. Bank+EMBL+DDBJ+PDB sequences (but no EST, STS, GSS, or phase 0, 1 or 2 HTGS sequences). No longer "nonredundant". est Database of Gen. Bank+EMBL+DDBJ sequences from EST division. est_human est_mouse htgs Unfinished High Throughput Genomic Sequences yeast Saccharomyces cerevisiae genomic nucleotide sequences mito Database of mitochondrial sequences vector Vector subset of Gen. Bank(R), NCBI, in month All new or revised Gen. Bank+EMBL+DDBJ+PDB sequences alu Select Alu repeats from REPBASE, suitable for masking Alu repeats from query sequences. dbsts Database of Gen. Bank+EMBL+DDBJ sequences from STS division. chromosome Searches Complete Genomes, Complete Chromosome, or contigs form the NCBI Reference Sequence project.

Usare BLAST PROGRAMMI Blastn Nucleotide query - Nucleotide db Blastp Protein query - Protein db Translating BLAST attraverso la traduzione concettuale della query sequence o dei database permette di comparare una sequenza nucleotidica con database di proteine o viceversa. Translated query - Protein db blastx Protein query - Translated db tblastn Translated query - Translated db tblastx MEGABLAST usa un algoritmo greedy (ingordo) veloce ed ottimizzato per comparare sequenze che differiscono poco Search for short nearly exact matches blastn con parametri scelti in modo da ottimizzare la ricerca di matches quasi esatti e brevi. Questi si trovano spesso per caso, percio’ utilizza alto E-value, piccola dimensione della parola e filtering PSI-BLAST Find members of a protein family or build a custom positionspecific score matrix PHI-BLAST Find proteins similar to the query around a given pattern

WWW BLAST http: //blast. ncbi. nlm. nih. gov

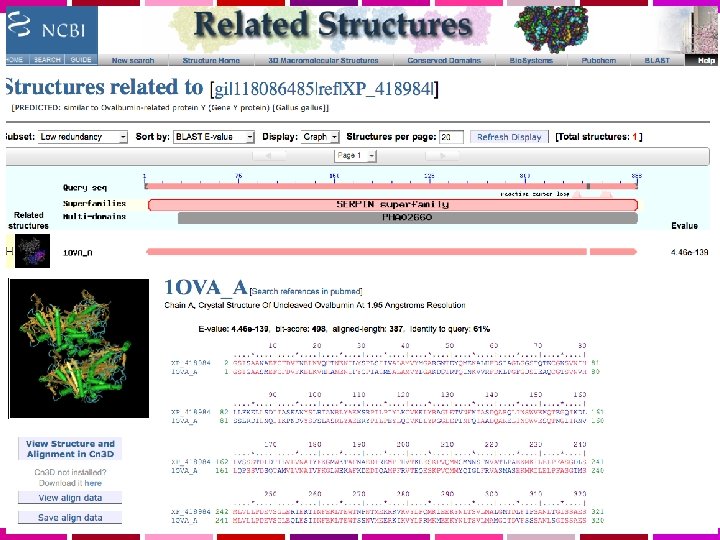
WWW Blast: Risultati …




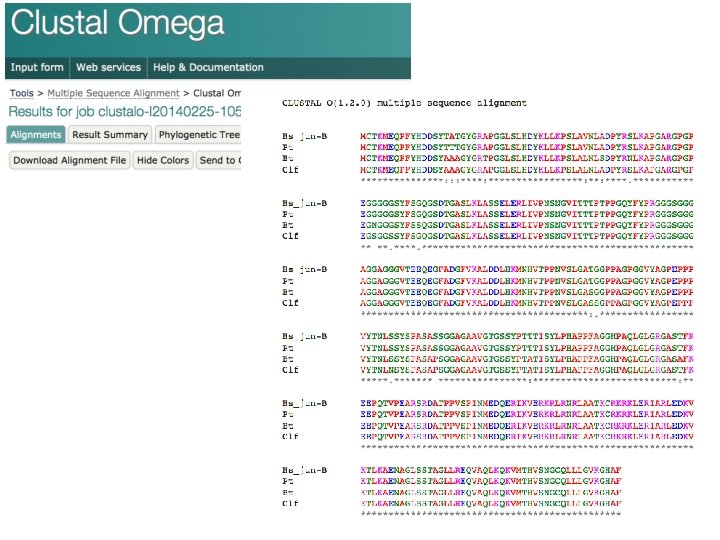
Allineamento multiplo di sequenze >Hs_jun-B MCTKMEQPFYHDDSYTATGYGRAPGGLSLHDYKLLKPSLAVNLADPYRSLKAPGARGPGPEGGGGGSYFS GQGSDTGASLKLASSELERLIVPNSNGVITTTPTPPGQYFYPRGGGSGGGAGGAGGGVTEEQEGFADGFV KALDDLHKMNHVTPPNVSLGATGGPPAGPGGVYAGPEPPPVYTNLSSYSPASASSGGAGAAVGTGSSYPT TTISYLPHAPPFAGGHPAQLGLGRGASTFKEEPQTVPEARSRDATPPVSPINMEDQERIKVERKRLRNRL AATKCRKRKLERIARLEDKVKTLKAENAGLSSTAGLLREQVAQLKQKVMTHVSNGCQLLLGVKGHAF >Pt MCTKMEQPFYHDDSYTTTGYGRAPGGLSLHDYKLLKPSLAVNLADPYRSLKAPGARGPGPEGGGGGSYFS GQGSDTGASLKLASSELERLIVPNSNGVITTTPTPPGQYFYPRGGGSGGGAGGAGGGVTEEQEGFADGFV KALDDLHKMNHVTPPNVSLGATGGPPAGPGGVYAGPEPPPVYTNLSSYSPASASSGGAGAAVGTGSSYPT TTISYLPHAPPFAGGHPAQLGLGRGASTFKEEPQTVPEARSRDATPPVSPINMEDQERIKVERKRLRNRL AATKCRKRKLERIARLEDKVKTLKAENAGLSSTAGLLREQVAQLKQKVMTHVSNGCQLLLGVKGHAF >Bt MCTKMEQPFYHDDSYAAAGYGRTPGGLSLHDYKLLKPSLALNLSDPYRNLKAPGARGPGPEGNGGGSYFS SQGSDTGASLKLASSELERLIVPNSNGVITTTPTPPGQYFYPRGGGSGGGAGGAGGGVTEEQEGFADGFV KALDDLHKMNHVTPPNVSLGASGGPPAGPGGVYAGPEPPPVYTNLSSYSPASAPSGGAGAAVGTGSSYPT ATISYLPHAPPFAGGHPAQLGLGRGASAFKEEPQTVPEARSRDATPPVSPINMEDQERIKVERKRLRNRL AATKCRKRKLERIARLEDKVKTLKAENAGLSSTAGLLREQVAQLKQKVMTHVSNGCQLLLGVKGHAF >Clf MCTKMEQPFYHDDSYAAAGYGRAPGGLSLHDYKLLKPSLALNLADPYRSLKAPGARGPGPEGSGGSSYFS GQGSDTGASLKLASSELERLIVPNSNGVITTTPTPPGQYFYPRGGGSGGGAGGAGGGVTEEQEGFADGFV KALDDLHKMNHVTPPNVSLGASSGPPAGPGGVYAGPEPPPVYTNLNSYSPASAPSGGAGAAVGTGSSYPT ATISYLPHAPPFAGGHPAQLGLGRGASTFKEEPQTVPEARSRDATPPVSPINMEDQERIKVERKRLRNRL AATKCRKRKLERIARLEDKVKTLKAENAGLSSTAGLLREQVAQLKQKVMTHVSNGCQLLLGVKGHAF

STEPS IN MULTIPLE ALIGNMENT 1. Progressive multiple alignment xxxxxxxxxxxxxxx xxxxxxxxxxxxxxx

Allineamento multiplo di sequenze Clustal Omega


one i z u r t s o c i Rico t e n e og l i f o r e Alb … Pre diz Motivi d ion ed i seque nza con ella servati stru ttur ad elle pro tein e

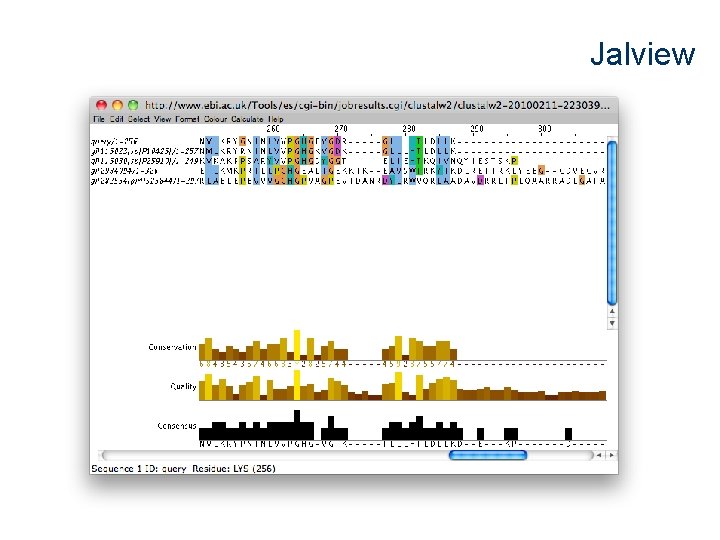
Jalview

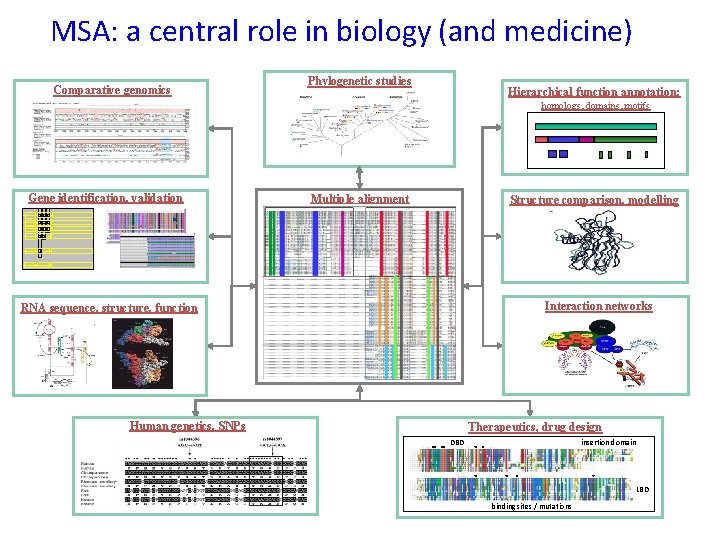
MSA: a central role in biology (and medicine) Comparative genomics Phylogenetic studies Hierarchical function annotation: homologs, domains, motifs Gene identification, validation Multiple alignment Structure comparison, modelling Interaction networks RNA sequence, structure, function Human genetics, SNPs Therapeutics, drug design insertion domain DBD Therapeutics, drug discovery LBD binding sites / mutations