Cintique cellulaire nonstructure A Garnier Gnie chimique Croissance




































- Slides: 62

Cinétique cellulaire non-structurée A. Garnier, Génie chimique

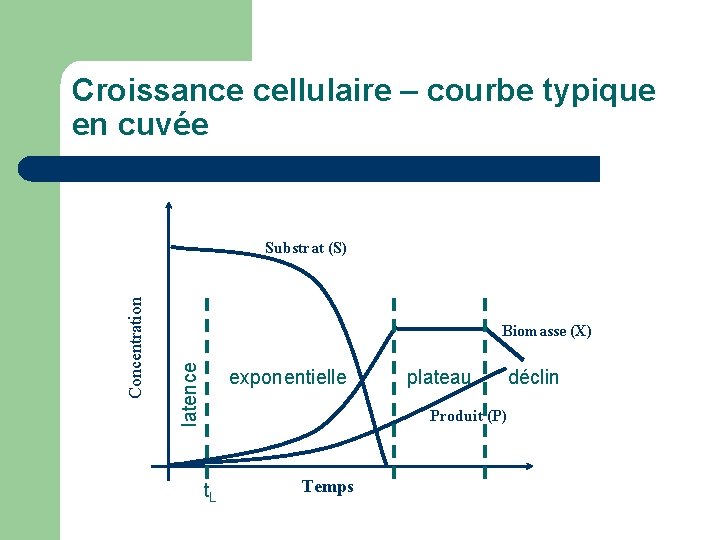
Croissance cellulaire – courbe typique en cuvée Biomasse (X) latence Concentration Substrat (S) exponentielle plateau Produit (P) t. L Temps déclin

Croissance cellulaire - variables X S P Taux de variation (kg/(m 3. s) Taux de production /consommation d. X/dt d. S/dt d. P/dt rx rs rp Taux spécifique (kg/(kg. s) Rendement (kg/kg) = rx/X qs= -rs/X qp= r. P/X Yx/s = rx/rs = /qs Yp/x, Yp/s

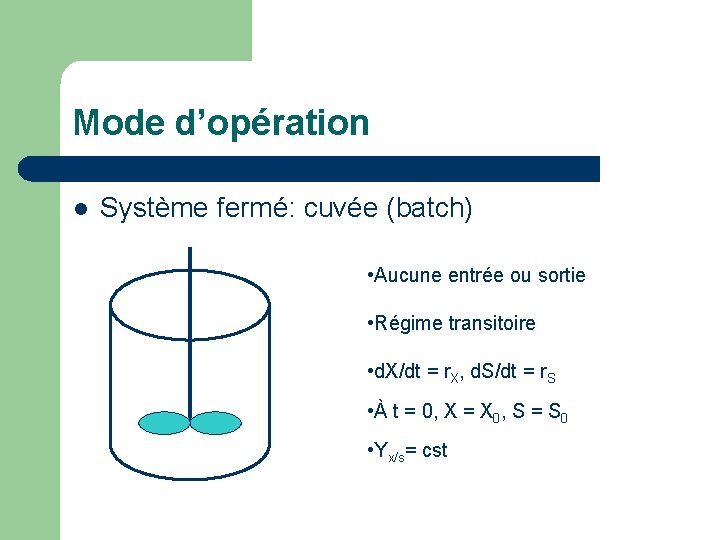
Mode d’opération l Système fermé: cuvée (batch) • Aucune entrée ou sortie • Régime transitoire • d. X/dt = r. X, d. S/dt = r. S • À t = 0, X = X 0, S = S 0 • Yx/s= cst

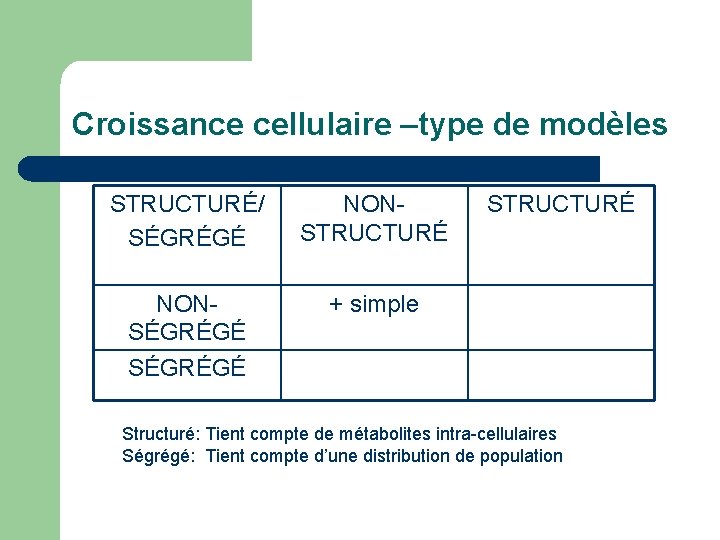
Croissance cellulaire –type de modèles STRUCTURÉ/ SÉGRÉGÉ NONSTRUCTURÉ NONSÉGRÉGÉ + simple STRUCTURÉ SÉGRÉGÉ Structuré: Tient compte de métabolites intra-cellulaires Ségrégé: Tient compte d’une distribution de population

Modèles de croissance l Du plus simple au plus compliqué – – l Phénomènes connexes – – l Exponentiel (ordre 0) Linéaire (logistique) Monod Autres Maintenance Mortalité Production – Luedeking-Piret combiné aux différents modèles

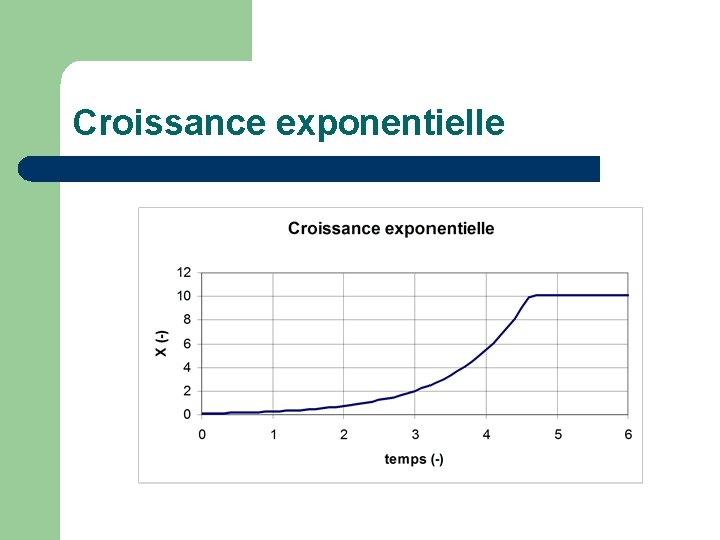
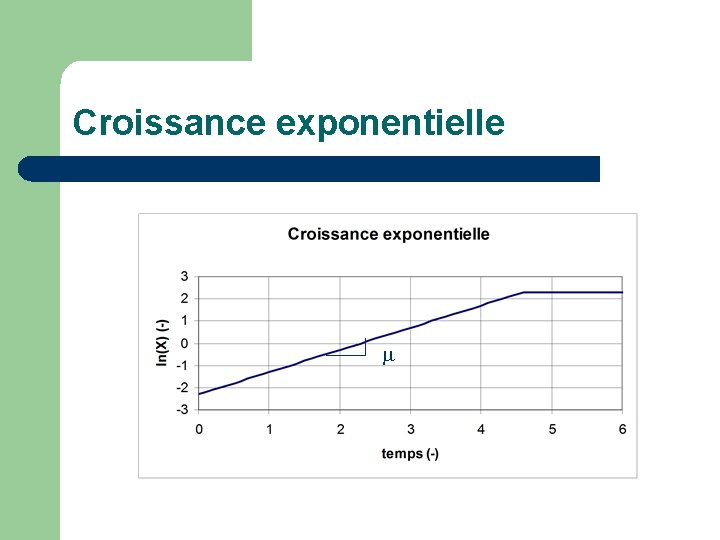
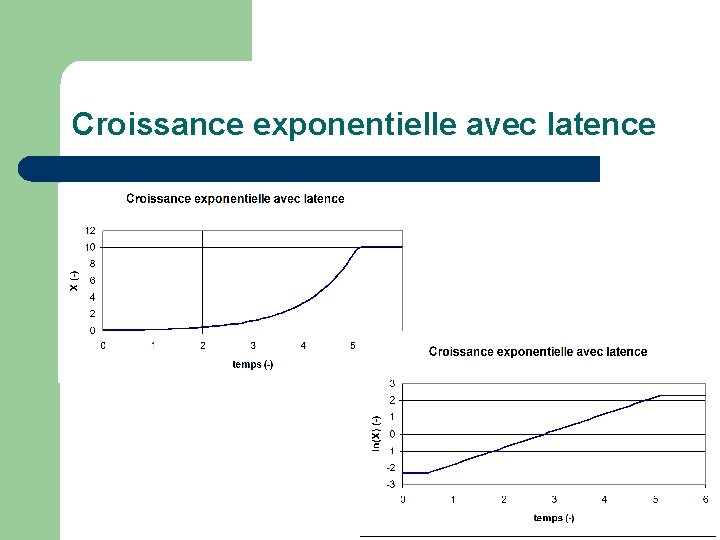
Ordre 0: Croissance exponentielle Où = cste Solution: X X X Avec latence: Pour t < t. L Jusqu’à XMAX = X 0 + YX/S*S 0 Pour t ≥ t. L

Croissance exponentielle

Croissance exponentielle

Croissance exponentielle avec latence

Modèle linéaire – courbe logistique Si Yx/s est constant: (X-Xo) = Yx/s (So-S) (XMAX-Xo) = Yx/s * So } Xmax-X = Yx/s * S On propose alors: S 1/(Yx/s) * (Xmax – X) (1 -X/Xmax)

Modèle logistique (courbe en S) l l l = k*(1 -X/Xmax) d. X/dt = k*(1 -X/Xmax)*X Solution: – Sachant que: – On obtient:

Modèle logistique (suite)

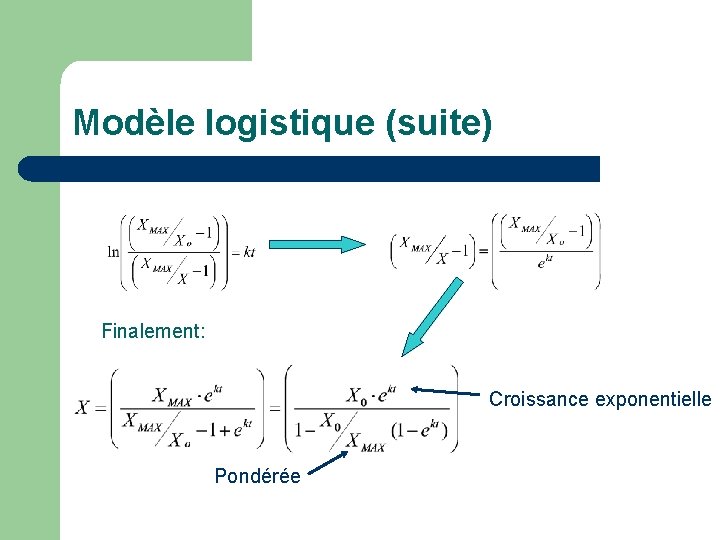
Modèle logistique (suite) Finalement: Croissance exponentielle Pondérée

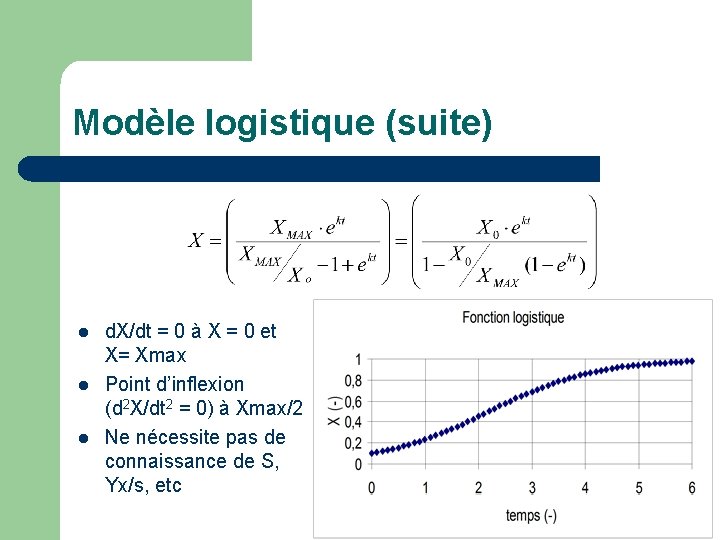
Modèle logistique (suite) l l l d. X/dt = 0 à X = 0 et X= Xmax Point d’inflexion (d 2 X/dt 2 = 0) à Xmax/2 Ne nécessite pas de connaissance de S, Yx/s, etc

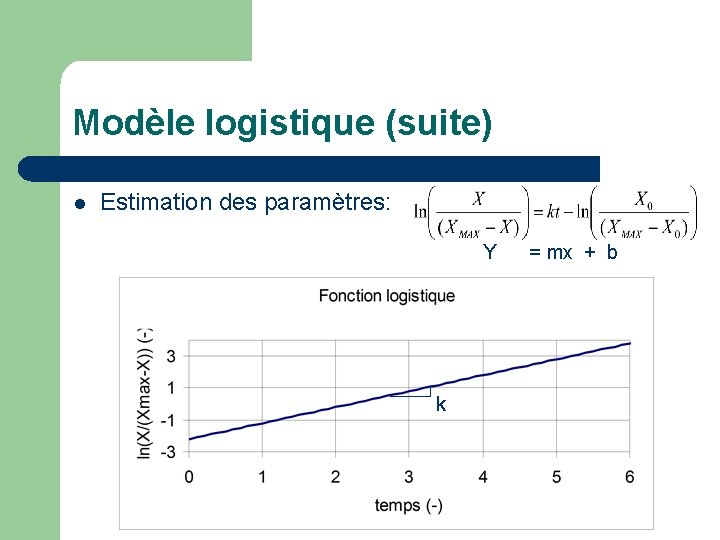
Modèle logistique (suite) l Estimation des paramètres: Y = mx + b k

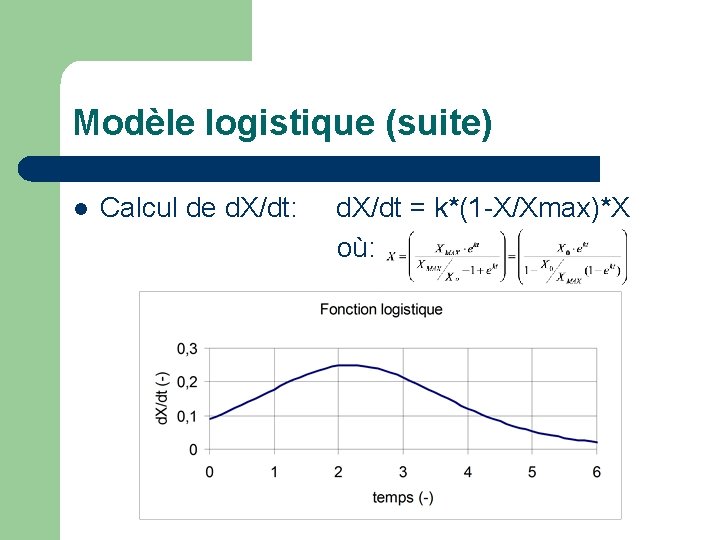
Modèle logistique (suite) Calcul de d. X/dt: d. X/dt = k*(1 -X/Xmax)*X où: l

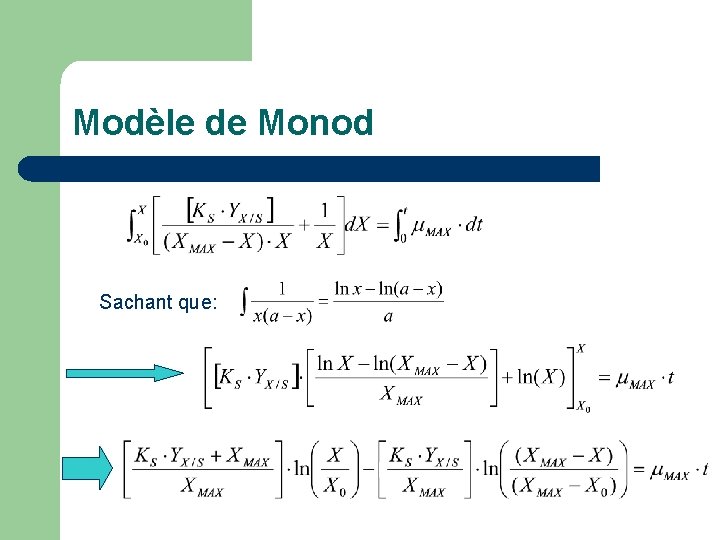
Modèle enzymatique - Monod (1) (2) (3) -Une variable indépendante, 3 variables dépendantes, trois équations = Une solution!

Modèle de Monod l Équation 3 n’est peutêtre pas nécessaire: Xmax-X = Yx/s * S Alors:

Modèle de Monod

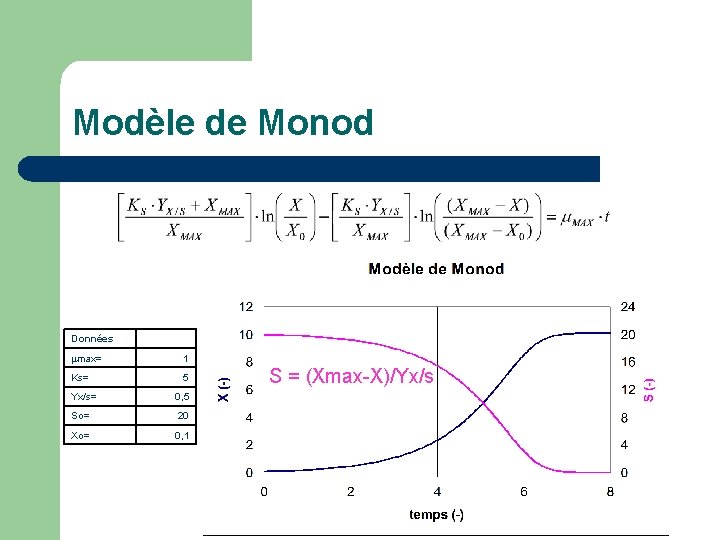
Modèle de Monod Sachant que:

Modèle de Monod Données max= 1 Ks= 5 Yx/s= 0, 5 So= 20 Xo= 0, 1 S = (Xmax-X)/Yx/s

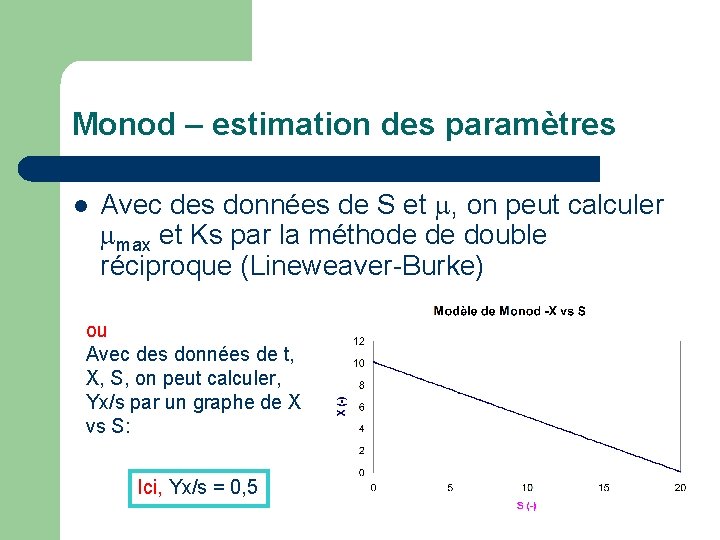
Monod – estimation des paramètres l Avec des données de S et , on peut calculer max et Ks par la méthode de double réciproque (Lineweaver-Burke) ou Avec des données de t, X, S, on peut calculer, Yx/s par un graphe de X vs S: Ici, Yx/s = 0, 5

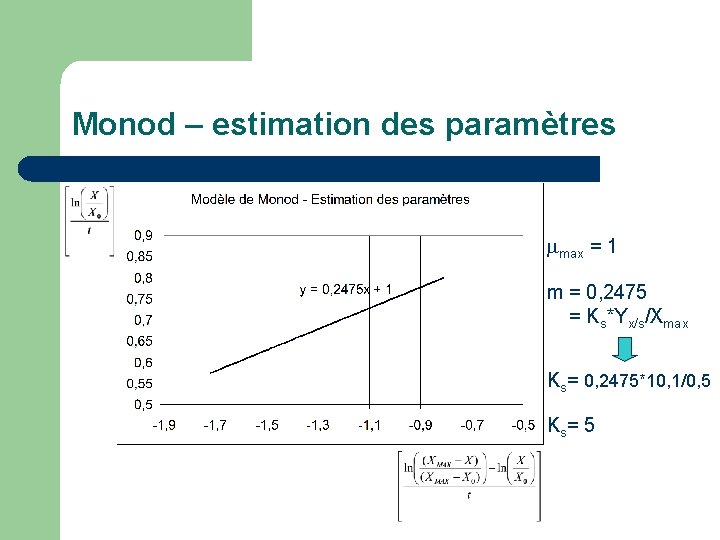
Monod – estimation des paramètres l Puis on peut reformuler l’équation de X pour isoler des termes reliés linéairement: ÷t Y = b + m X ÷t

Monod – estimation des paramètres max = 1 m = 0, 2475 = Ks*Yx/s/Xmax Ks= 0, 2475*10, 1/0, 5 Ks= 5

Autres modèles (Shuler-Kargi) l Inhibition par sous-produit toxique: ou l Tessier: l Moser: l Contois: • Haldane: Etc. . .

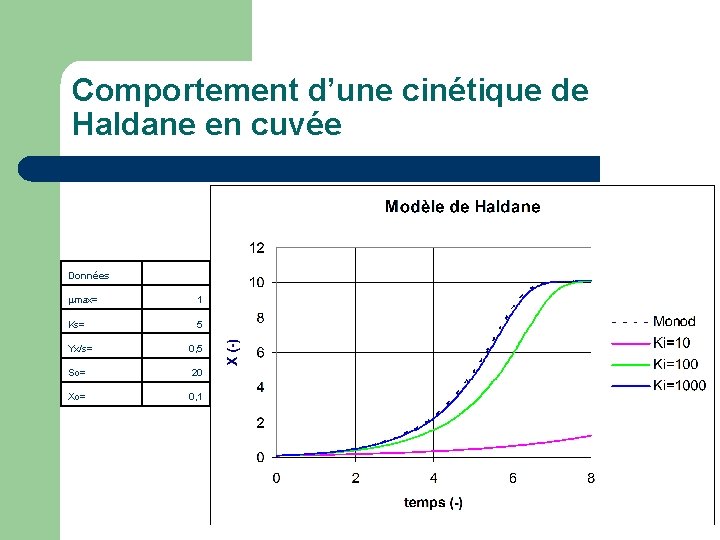
Inhibition par le substrat: Haldane En Cuvée: (1) (2) Xmax-X = Yx/s * S (3)

Haldane en cuvée

Haldane en cuvée

Comportement d’une cinétique de Haldane en cuvée Données max= 1 Ks= 5 Yx/s= 0, 5 So= 20 Xo= 0, 1

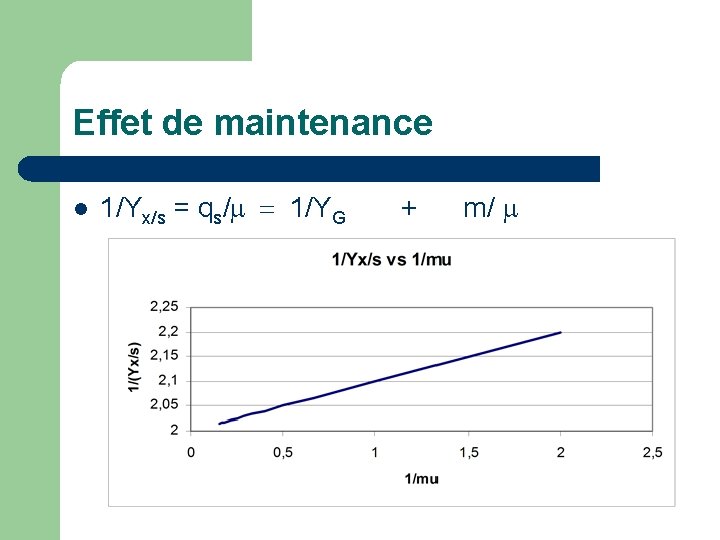
Effet de maintenance l S = S(croissance) + S(maintenance) l r. S = 1/YG* r. X + m * X l qs = 1/YG* + m l 1/Yx/s = qs/ = 1/YG + m/

Effet de maintenance l 1/Yx/s = qs/ = 1/YG + m/

Effet de mortalité l Le taux de mortalité cellulaire: rd = - kd*X où kd=cste l Donc en cuvée: d. X/dt = *X – kd*X = ( – kd)*X En général, on néglige la maintenance et la mortalité en cuvée

Cinétique de production l Le modèle le plus connu est celui de Luedeking-Piret: } } r. P = *r. X + b * X associé non-associé à la croissance q. P = * + b

Combinaison L-P/Croissance cellulaire r. P = *r. X + b * X En cuvée: d. P/dt = * d. X/dt + b * X Or nous avons déjà des relations pour X

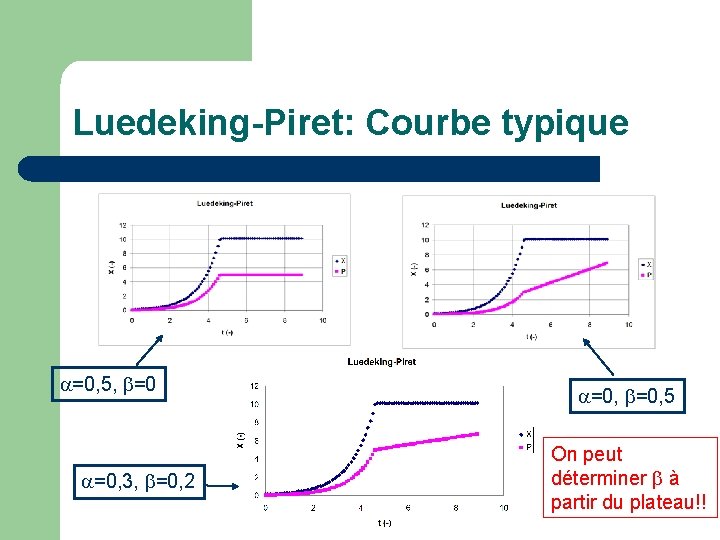
Luedeking-Piret: Courbe typique =0, 5, b=0 =0, 3, b=0, 2 =0, b=0, 5 On peut déterminer b à partir du plateau!!

Combinaison L-P/Logistique

Combinaison L-P/Logistique détermination des paramètres • Si on connaît b: Y = m * X • On trouve ! • Mais si on ne connaît pas β…

Combinaison L-P/Monod

Détermination des paramètres l Une méthode: Ou encore…

Autres modes d’opération l l Cuvée alimentée Continu avec recirculation Piston (plug flow)

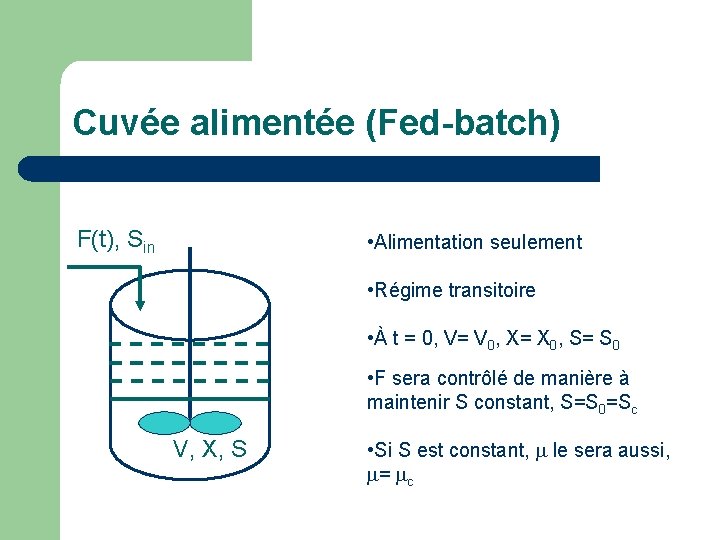
Cuvée alimentée (Fed-batch) F(t), Sin • Alimentation seulement • Régime transitoire • À t = 0, V= V 0, X= X 0, S= S 0 • F sera contrôlé de manière à maintenir S constant, S=S 0=Sc V, X, S • Si S est constant, le sera aussi, = c

Cuvée alimentée (Fed-batch) l 3 bilans seront nécessaires pour obtenir un modèle de ce système: F ….

Cuvée alimentée (Fed-batch)

Cuvée alimentée (Fed-batch) 0 F

Cuvée alimentée (Fed-batch)

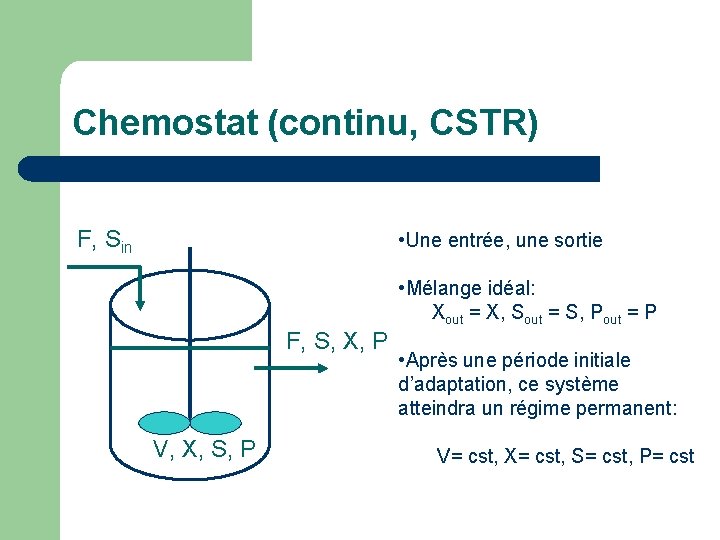
Chemostat (continu, CSTR) F, Sin • Une entrée, une sortie • Mélange idéal: Xout = X, Sout = S, Pout = P F, S, X, P V, X, S, P • Après une période initiale d’adaptation, ce système atteindra un régime permanent: V= cst, X= cst, S= cst, P= cst

Chemostat (continu, CSTR) l l Bilan sur la biomasse: Bilan sur le substrat: Où D: taux de dilution

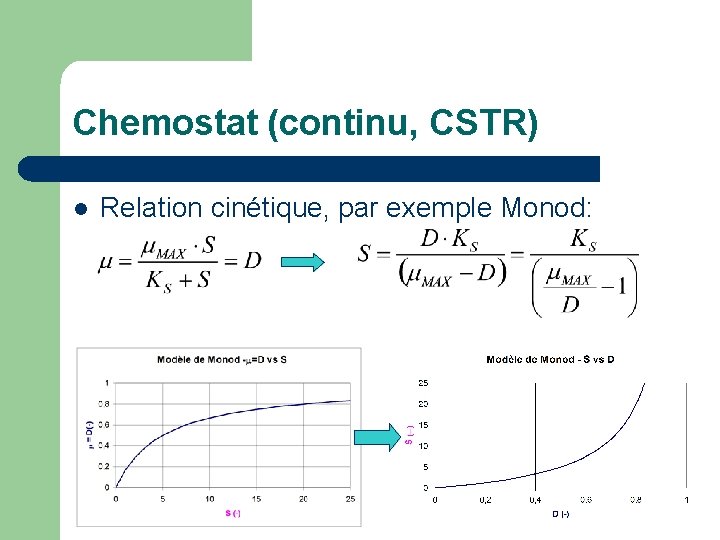
Chemostat (continu, CSTR) l Relation cinétique, par exemple Monod:

Chemostat (continu, CSTR) l Le produit:

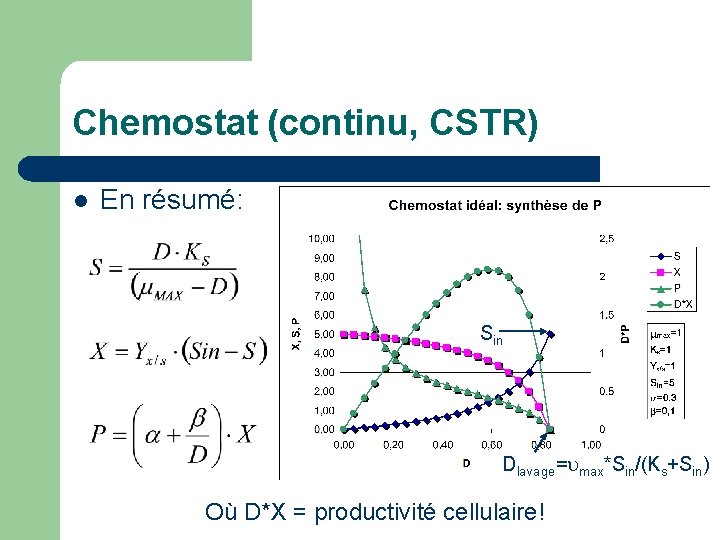
Chemostat (continu, CSTR) l En résumé: Sin Dlavage=umax*Sin/(Ks+Sin) Où D*X = productivité cellulaire!

Chemostat (continu, CSTR) l Productivité cellulaire: – D ((L/min) / L de culture) * X (g de cellule/L)= Productivité (g de cellules/(min*L de culture) – l Productivité en produit: –

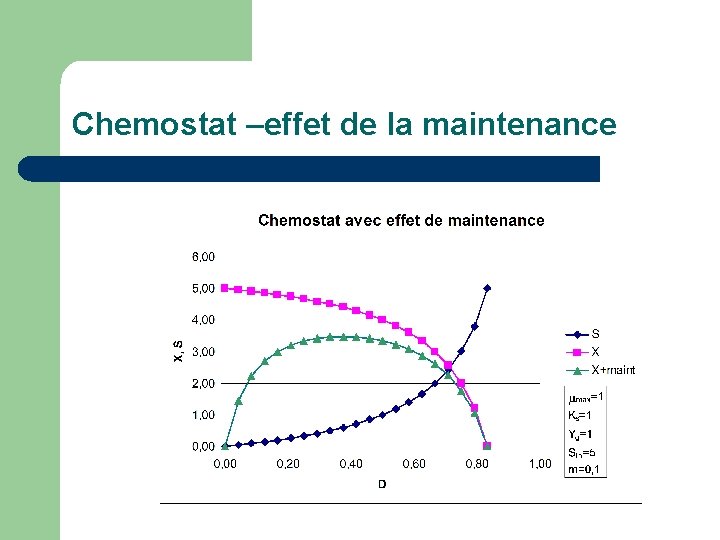
Chemostat –effet de la maintenance l l l Bilan X: = D inchangé Cinétique: inchangée Bilan S:

Chemostat –effet de la maintenance

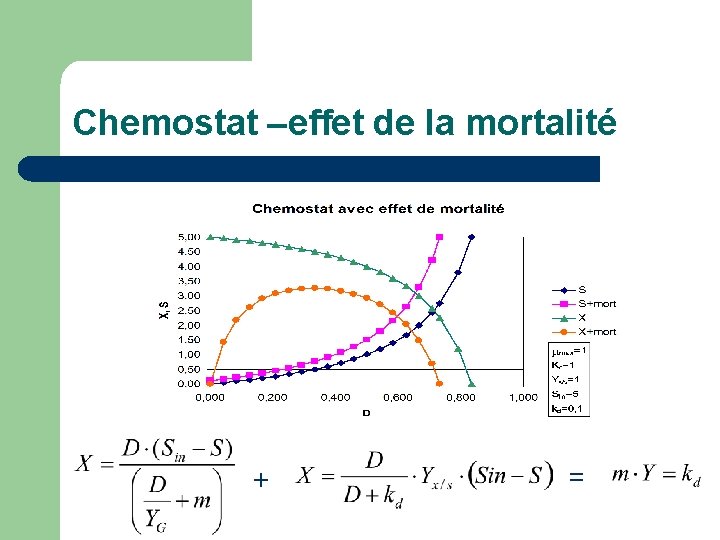
Chemostat –effet de la mortalité l Bilan X: l Cinétique: l Bilan S:

Chemostat –effet de la mortalité + =

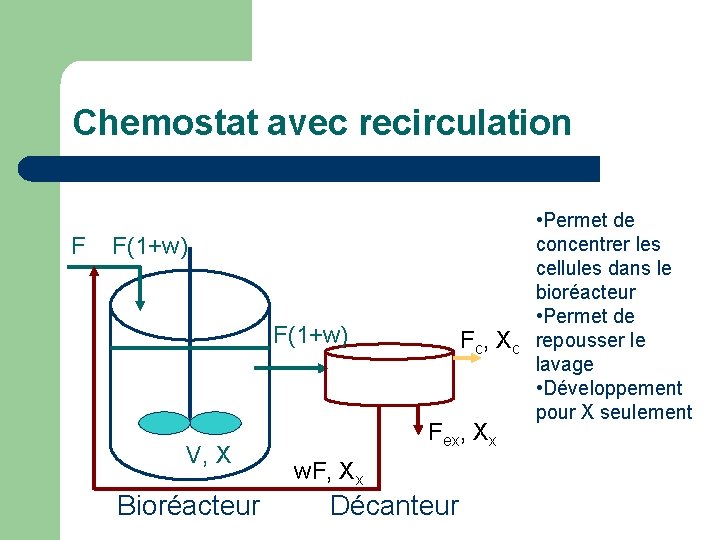
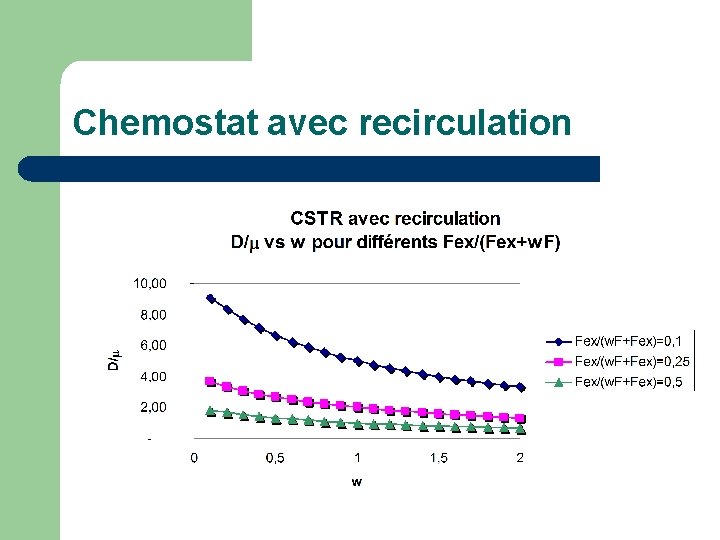
Chemostat avec recirculation F F(1+w) V, X Fc, Xc Fex, Xx w. F, Xx Bioréacteur Décanteur • Permet de concentrer les cellules dans le bioréacteur • Permet de repousser le lavage • Développement pour X seulement

Chemostat avec recirculation l Bilan sur les cellules du bioréacteur:

Chemostat avec recirculation l Bilan sur les cellules dans le séparateur: 0

Chemostat avec recirculation

Modèles non-structurés l Beaucoup de diversité et de potentiel l Mais ne répond pas à tous les besoins…

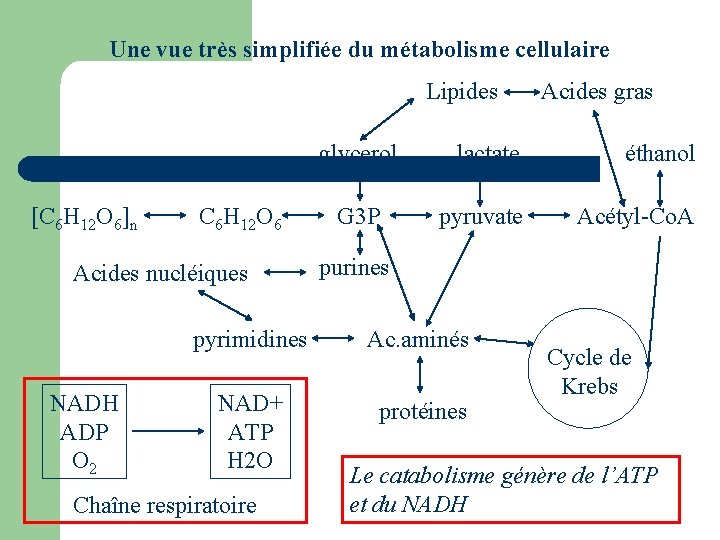
Une vue très simplifiée du métabolisme cellulaire Lipides glycerol [C 6 H 12 O 6]n C 6 H 12 O 6 Acides nucléiques NADH ADP O 2 G 3 P lactate éthanol pyruvate Acétyl-Co. A purines pyrimidines Ac. aminés NAD+ ATP H 2 O protéines Chaîne respiratoire Acides gras Cycle de Krebs Le catabolisme génère de l’ATP et du NADH
 Schema cellule animal
Schema cellule animal Psm culture cellulaire
Psm culture cellulaire Signalisation cellulaire
Signalisation cellulaire Membrane plasmique végétale
Membrane plasmique végétale Chaine de transport des électrons
Chaine de transport des électrons Garnier emboss
Garnier emboss Tracy garnier
Tracy garnier Neutralia shampoo
Neutralia shampoo Garnier
Garnier Tracy garnier
Tracy garnier Nadine garnier
Nadine garnier Charles garnier obras
Charles garnier obras Famille chimique
Famille chimique Traceur chimique
Traceur chimique Procédé chimique
Procédé chimique Formule chimique plagioclase
Formule chimique plagioclase Valence des corps chimiques
Valence des corps chimiques Composition chimique du poisson
Composition chimique du poisson L'affinité chimique
L'affinité chimique Déplacement chimique
Déplacement chimique Systme digestif
Systme digestif Elmet berango
Elmet berango Absorption intestinale
Absorption intestinale Constante dequilibre
Constante dequilibre équilibre chimique
équilibre chimique Risque chimique définition
Risque chimique définition Formule cuivre
Formule cuivre Les dérives du pétrole sont des matériaux synthétiques
Les dérives du pétrole sont des matériaux synthétiques Flu produit chimique
Flu produit chimique Procédé chimique
Procédé chimique Contribution à la croissance
Contribution à la croissance Croissance somatique
Croissance somatique Contribution à la croissance
Contribution à la croissance Betli
Betli Chorus ii équilibré croissance
Chorus ii équilibré croissance Sijori croquis
Sijori croquis Croissance intensive
Croissance intensive Abeille croissance continue ou discontinue
Abeille croissance continue ou discontinue Stagnation pondérale bébé
Stagnation pondérale bébé La croissance intensive
La croissance intensive Facteurs de croissance hématopoïétiques
Facteurs de croissance hématopoïétiques Croissance douglas
Croissance douglas Les sources de la croissance
Les sources de la croissance