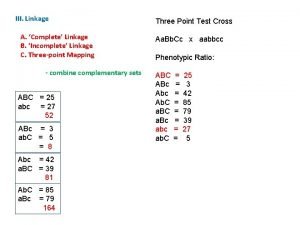
3 1 ASSOCIAZIONE LINKAGE CONCETTI FONDAMENTALI Figura 3


















- Slides: 52

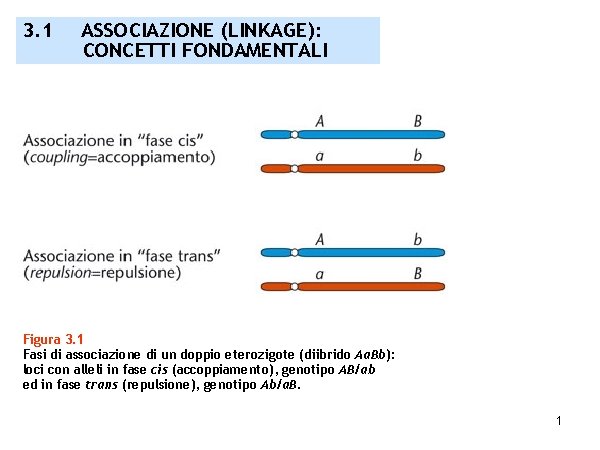
3. 1 ASSOCIAZIONE (LINKAGE): CONCETTI FONDAMENTALI Figura 3. 1 Fasi di associazione di un doppio eterozigote (diibrido Aa. Bb): loci con alleli in fase cis (accoppiamento), genotipo AB/ab ed in fase trans (repulsione), genotipo Ab/a. B. 1

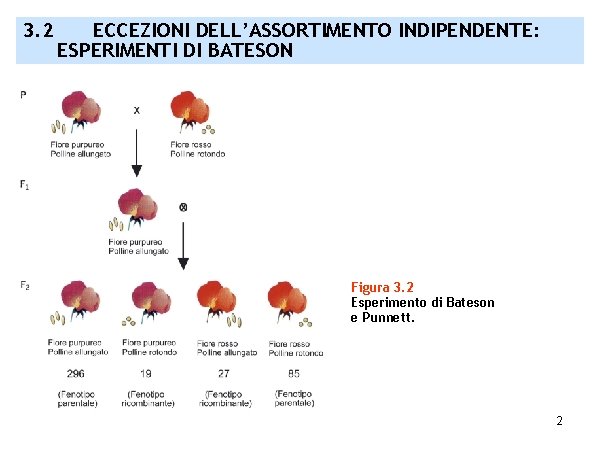
3. 2 ECCEZIONI DELL’ASSORTIMENTO INDIPENDENTE: ESPERIMENTI DI BATESON Figura 3. 2 Esperimento di Bateson e Punnett. 2

3. 2 ECCEZIONI DELL’ASSORTIMENTO INDIPENDENTE: ESPERIMENTI DI BATESON Tabella 3. 1 Frequenze fenotipiche osservate e attese nel primo degli esperimenti di Bateson e Punnett, e calcolo del chi-quadrato. 3

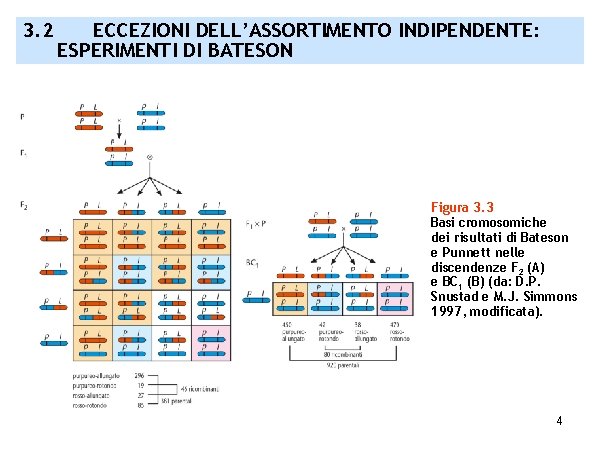
3. 2 ECCEZIONI DELL’ASSORTIMENTO INDIPENDENTE: ESPERIMENTI DI BATESON Figura 3. 3 Basi cromosomiche dei risultati di Bateson e Punnett nelle discendenze F 2 (A) e BC 1 (B) (da: D. P. Snustad e M. J. Simmons 1997, modificata). 4

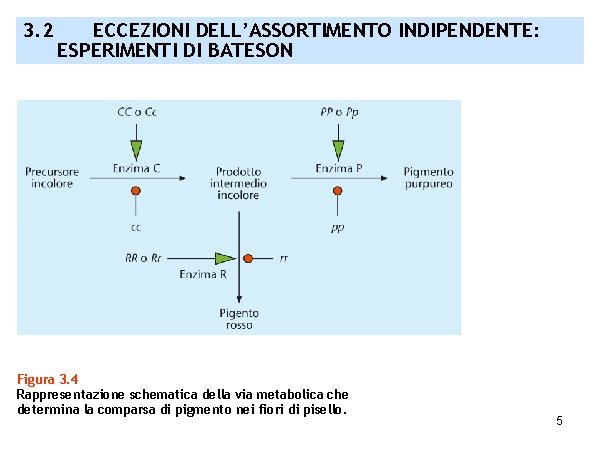
3. 2 ECCEZIONI DELL’ASSORTIMENTO INDIPENDENTE: ESPERIMENTI DI BATESON Figura 3. 4 Rappresentazione schematica della via metabolica che determina la comparsa di pigmento nei fiori di pisello. 5

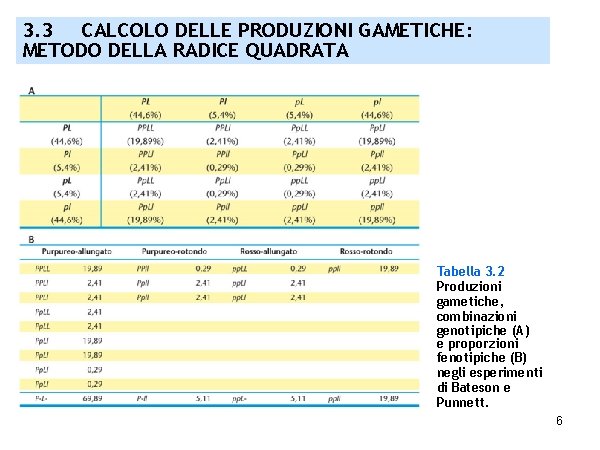
3. 3 CALCOLO DELLE PRODUZIONI GAMETICHE: METODO DELLA RADICE QUADRATA Tabella 3. 2 Produzioni gametiche, combinazioni genotipiche (A) e proporzioni fenotipiche (B) negli esperimenti di Bateson e Punnett. 6

3. 3 CALCOLO DELLE PRODUZIONI GAMETICHE: METODO DELLA RADICE QUADRATA QUADRO 3. 1 – SOSTENITORI E DENIGRATORI DEL MENDELISMO: W. BATESON, T. H. MORGAN E T. LYSENKO Figura 3. 5 Fotografia di Trofim Denisovich Lysenko (1898 -1976). 7

3. 4 SCOPERTA DELL’ASSOCIAZIONE IN DROSOPHILA: ESPERIMENTI DI MORGAN Figura 3. 6 Mutante di Drosophila ad occhi bianchi: schemi di incrocio effettuati da Morgan per stabilire l’eredità del carattere. 8

3. 4 SCOPERTA DELL’ASSOCIAZIONE IN DROSOPHILA: ESPERIMENTI DI MORGAN Figura 3. 7 Esperimento di Morgan in Drosophila: verifica dell’associazione tra i loci responsabili della forma delle ali (Vg, allele per le ali lunghe e vg, allele per le ali corte o vestigiali) e il colore del corpo (B, allele per il corpo grigio e b, allele per il corpo nero) (da: D. P. Snustad e M. J. Simmons 1997, modificata). 9

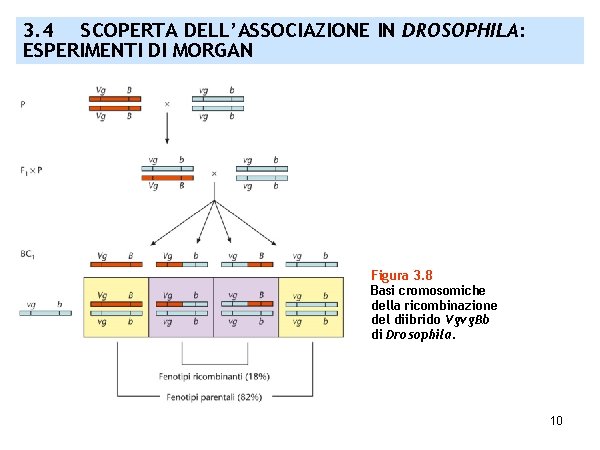
3. 4 SCOPERTA DELL’ASSOCIAZIONE IN DROSOPHILA: ESPERIMENTI DI MORGAN Figura 3. 8 Basi cromosomiche della ricombinazione del diibrido Vgvg. Bb di Drosophila. 10

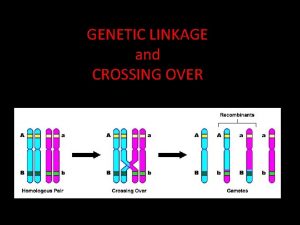
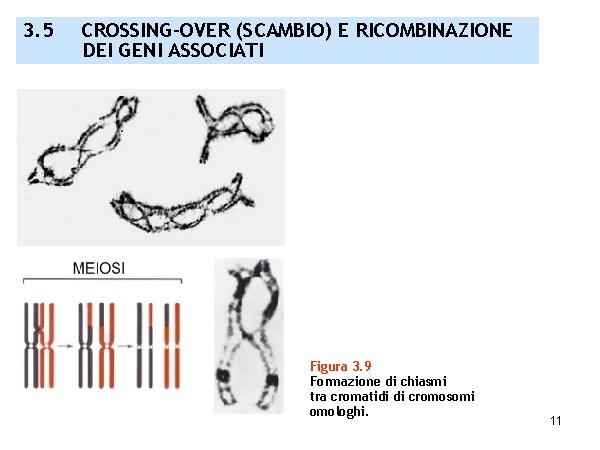
3. 5 CROSSING-OVER (SCAMBIO) E RICOMBINAZIONE DEI GENI ASSOCIATI Figura 3. 9 Formazione di chiasmi tra cromatidi di cromosomi omologhi. 11

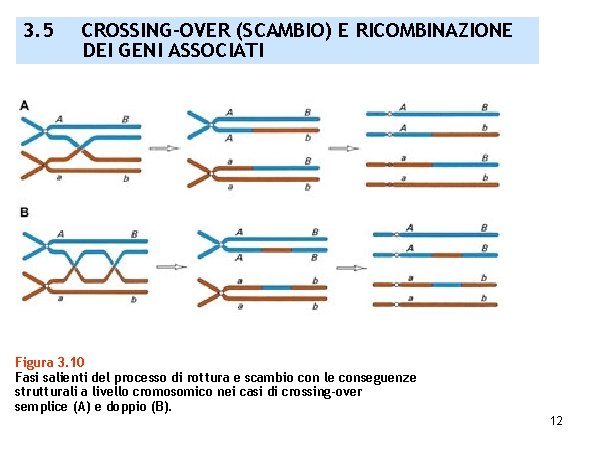
3. 5 CROSSING-OVER (SCAMBIO) E RICOMBINAZIONE DEI GENI ASSOCIATI Figura 3. 10 Fasi salienti del processo di rottura e scambio con le conseguenze strutturali a livello cromosomico nei casi di crossing-over semplice (A) e doppio (B). 12

3. 5 CROSSING-OVER (SCAMBIO) E RICOMBINAZIONE DEI GENI ASSOCIATI Figura 3. 11 Classificazione di gameti parentali e ricombinanti in relazione alla fase di associazione dei geni nel diibrido Aa. Bb. 13

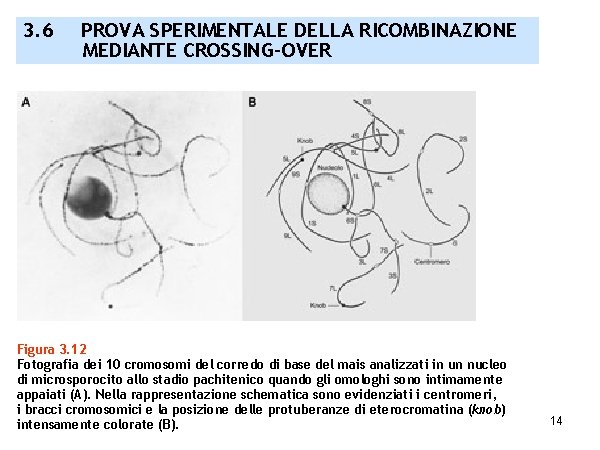
3. 6 PROVA SPERIMENTALE DELLA RICOMBINAZIONE MEDIANTE CROSSING-OVER Figura 3. 12 Fotografia dei 10 cromosomi del corredo di base del mais analizzati in un nucleo di microsporocito allo stadio pachitenico quando gli omologhi sono intimamente appaiati (A). Nella rappresentazione schematica sono evidenziati i centromeri, i bracci cromosomici e la posizione delle protuberanze di eterocromatina (knob) intensamente colorate (B). 14

3. 6 PROVA SPERIMENTALE DELLA RICOMBINAZIONE MEDIANTE CROSSING-OVER Figura 3. 13 A Esperimento di Creighton e Mc. Clintock in mais: (A) incrocio tra il mutante per il cromosoma 9 e il tipo normale; (B) conseguenze del crossing-over; (C) morfologia delle coppie di omologhi nelle piante della discendenza ricombinanti per ilcromosoma 9. 15

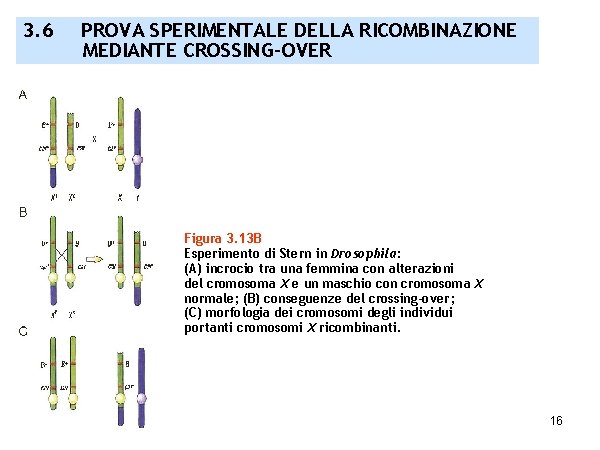
3. 6 PROVA SPERIMENTALE DELLA RICOMBINAZIONE MEDIANTE CROSSING-OVER Figura 3. 13 B Esperimento di Stern in Drosophila: (A) incrocio tra una femmina con alterazioni del cromosoma X e un maschio con cromosoma X normale; (B) conseguenze del crossing-over; (C) morfologia dei cromosomi degli individui portanti cromosomi X ricombinanti. 16

3. 6 PROVA SPERIMENTALE DELLA RICOMBINAZIONE MEDIANTE CROSSING-OVER QUADRO 3. 2 – MENDEL E IL LINKAGE Figura 3. 14 Mappa genetica di Pisum sativum (A) e possibili riarrangiamenti cromosomici (B) (Fonte: N. F. Weeden et al. (1998) A consensus linkage map for Pisum sativum. Pisum Genetics, 30: 1 -4). 17

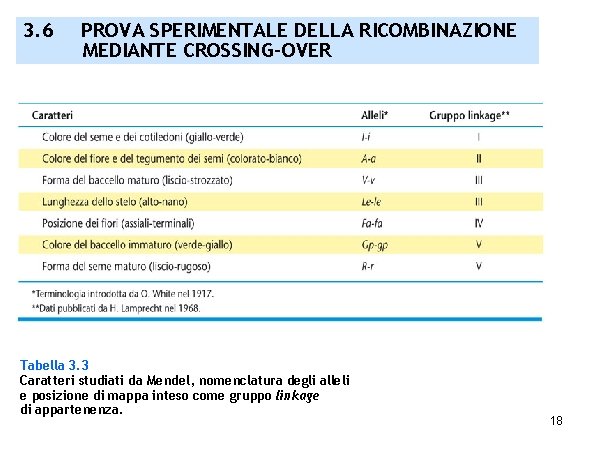
3. 6 PROVA SPERIMENTALE DELLA RICOMBINAZIONE MEDIANTE CROSSING-OVER Tabella 3. 3 Caratteri studiati da Mendel, nomenclatura degli alleli e posizione di mappa inteso come gruppo linkage di appartenenza. 18

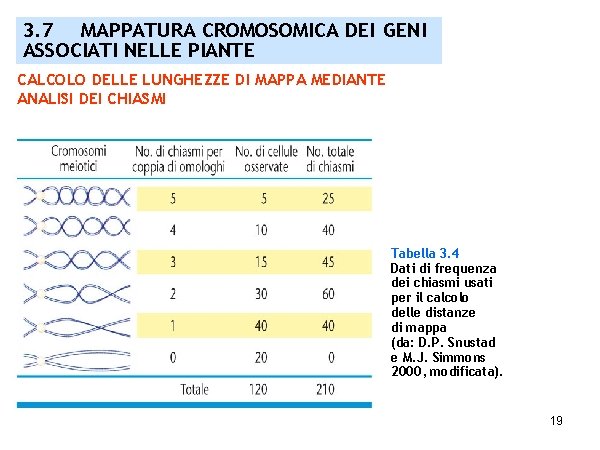
3. 7 MAPPATURA CROMOSOMICA DEI GENI ASSOCIATI NELLE PIANTE CALCOLO DELLE LUNGHEZZE DI MAPPA MEDIANTE ANALISI DEI CHIASMI Tabella 3. 4 Dati di frequenza dei chiasmi usati per il calcolo delle distanze di mappa (da: D. P. Snustad e M. J. Simmons 2000, modificata). 19

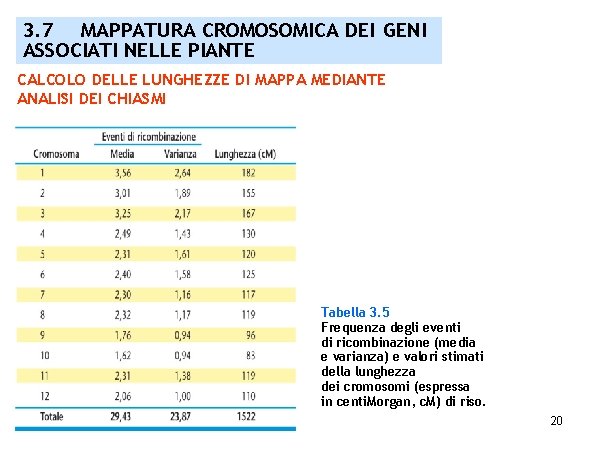
3. 7 MAPPATURA CROMOSOMICA DEI GENI ASSOCIATI NELLE PIANTE CALCOLO DELLE LUNGHEZZE DI MAPPA MEDIANTE ANALISI DEI CHIASMI Tabella 3. 5 Frequenza degli eventi di ricombinazione (media e varianza) e valori stimati della lunghezza dei cromosomi (espressa in centi. Morgan, c. M) di riso. 20

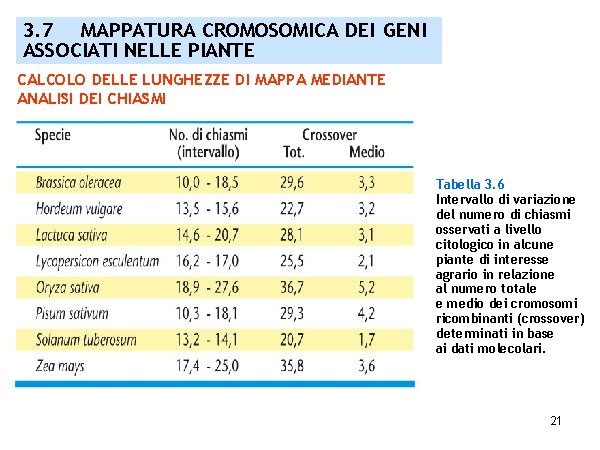
3. 7 MAPPATURA CROMOSOMICA DEI GENI ASSOCIATI NELLE PIANTE CALCOLO DELLE LUNGHEZZE DI MAPPA MEDIANTE ANALISI DEI CHIASMI Tabella 3. 6 Intervallo di variazione del numero di chiasmi osservati a livello citologico in alcune piante di interesse agrario in relazione al numero totale e medio dei cromosomi ricombinanti (crossover) determinati in base ai dati molecolari. 21

3. 7 MAPPATURA CROMOSOMICA DEI GENI ASSOCIATI NELLE PIANTE CALCOLO DELLE DISTANZE DI MAPPA MEDIANTE TEST A DUE PUNTI Figura 3. 15 Spighe di mais che mostrano segregazione per il colore dell’aleurone e per la struttura dell’endosperma. (A) Mutante con pericarpo incolore, omozigote per il gene colorless pericap (p 1 p 1), che rende visibile la segregazione del gene anthocyaninless a 1 con rapporto 3 aleurone rosso (A 1–): 1 aleurone incolore (a 1 a 1); mutanti shrunken sh 1 (B) e sh 2 (C) che manifestano cariossidi collassate, indentate o spigolose. 22

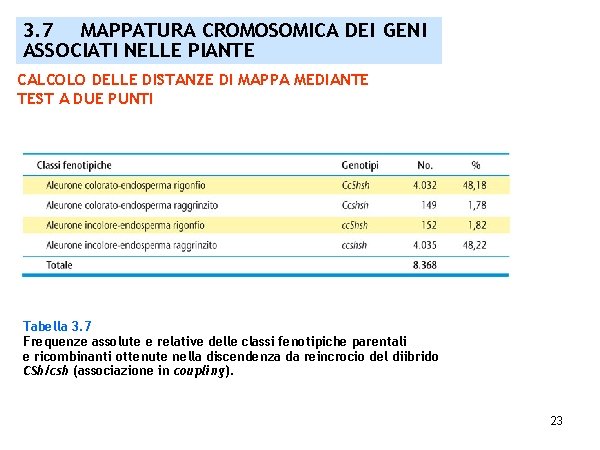
3. 7 MAPPATURA CROMOSOMICA DEI GENI ASSOCIATI NELLE PIANTE CALCOLO DELLE DISTANZE DI MAPPA MEDIANTE TEST A DUE PUNTI Tabella 3. 7 Frequenze assolute e relative delle classi fenotipiche parentali e ricombinanti ottenute nella discendenza da reincrocio del diibrido CSh/csh (associazione in coupling). 23

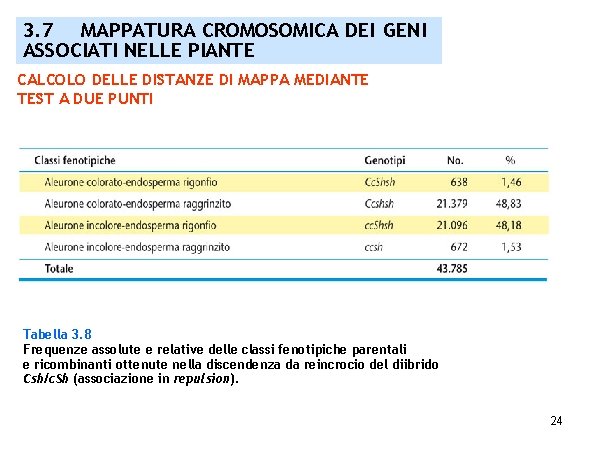
3. 7 MAPPATURA CROMOSOMICA DEI GENI ASSOCIATI NELLE PIANTE CALCOLO DELLE DISTANZE DI MAPPA MEDIANTE TEST A DUE PUNTI Tabella 3. 8 Frequenze assolute e relative delle classi fenotipiche parentali e ricombinanti ottenute nella discendenza da reincrocio del diibrido Csh/c. Sh (associazione in repulsion). 24

3. 7 MAPPATURA CROMOSOMICA DEI GENI ASSOCIATI NELLE PIANTE CALCOLO DELLE DISTANZE DI MAPPA MEDIANTE TEST A DUE PUNTI Tabella 3. 9 Dati complessivi di entrambi i reincroci effettuati da Hutchison per stabilire la distanza tra i geni C/c (coloured) e Sh/sh (shrunken) in mais. 25

3. 8 CALCOLO DELLE DISTANZE DI MAPPA NELLE POPOLAZIONI F 2 Tabella 3. 10 Combinazioni genotipiche e proporzioni fenotipiche della F 2 derivate usando le frequenze gametiche calcolate con il metodo della radice quadrata. 26

3. 8 CALCOLO DELLE DISTANZE DI MAPPA NELLE POPOLAZIONI F 2 Tabella 3. 11 Metodo di correzione delle distanze di mappa. 27

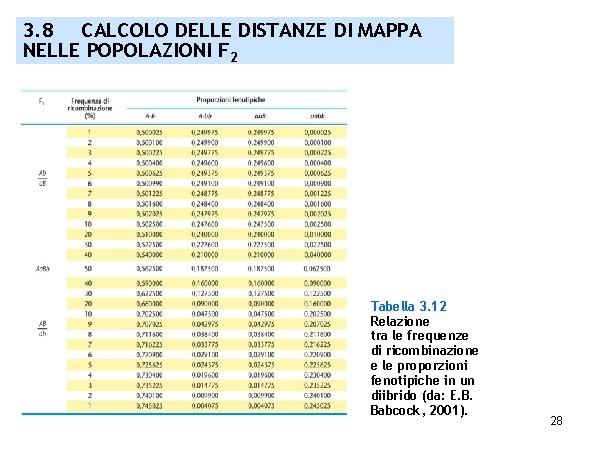
3. 8 CALCOLO DELLE DISTANZE DI MAPPA NELLE POPOLAZIONI F 2 Tabella 3. 12 Relazione tra le frequenze di ricombinazione e le proporzioni fenotipiche in un diibrido (da: E. B. Babcock, 2001). 28

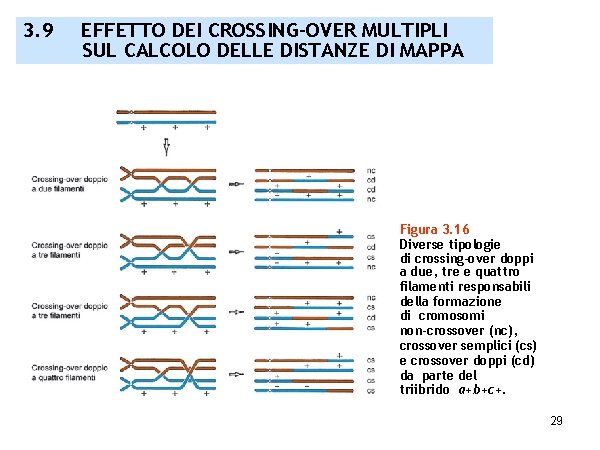
3. 9 EFFETTO DEI CROSSING-OVER MULTIPLI SUL CALCOLO DELLE DISTANZE DI MAPPA Figura 3. 16 Diverse tipologie di crossing-over doppi a due, tre e quattro filamenti responsabili della formazione di cromosomi non-crossover (nc), crossover semplici (cs) e crossover doppi (cd) da parte del triibrido a+b+c+. 29

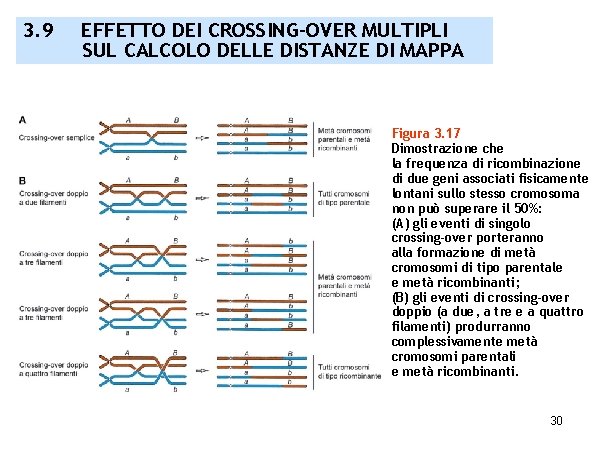
3. 9 EFFETTO DEI CROSSING-OVER MULTIPLI SUL CALCOLO DELLE DISTANZE DI MAPPA Figura 3. 17 Dimostrazione che la frequenza di ricombinazione di due geni associati fisicamente lontani sullo stesso cromosoma non può superare il 50%: (A) gli eventi di singolo crossing-over porteranno alla formazione di metà cromosomi di tipo parentale e metà ricombinanti; (B) gli eventi di crossing-over doppio (a due, a tre e a quattro filamenti) produrranno complessivamente metà cromosomi parentali e metà ricombinanti. 30

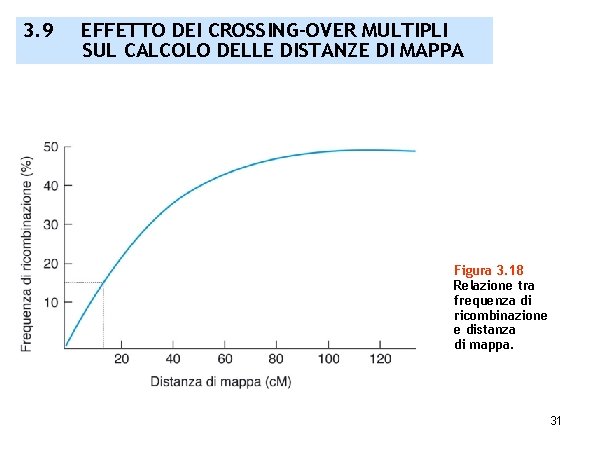
3. 9 EFFETTO DEI CROSSING-OVER MULTIPLI SUL CALCOLO DELLE DISTANZE DI MAPPA Figura 3. 18 Relazione tra frequenza di ricombinazione e distanza di mappa. 31

3. 9 EFFETTO DEI CROSSING-OVER MULTIPLI SUL CALCOLO DELLE DISTANZE DI MAPPA Figura 3. 19 Conseguenze genetiche dei crossing-over multipli (doppi, tripli e quadrupli) tra cromatidi non fratelli e di crossing-over tra cromatidi fratelli (da: D. P. Snustad e M. J. Simmons 2000, modificata). 32

3. 10 MAPPATURA MEDIANTE TEST A TRE PUNTI, INTERFERENZA E COEFFICIENTE DI COINCIDENZA Tabella 3. 13 Dati riguardanti un test a tre punti condotto da Beadle in mais usando una popolazione segregante BC 1 prodotta reincrociando un ibrido F 1 eterozigote per le mutazioni virescent (+/v), glossy (+/gl) e variable sterile (+/va) con una linea wildtype (+/+) a tutti e tre i loci (A); proporzioni dei fenotipi ordinati per classi simmetriche (B). 33

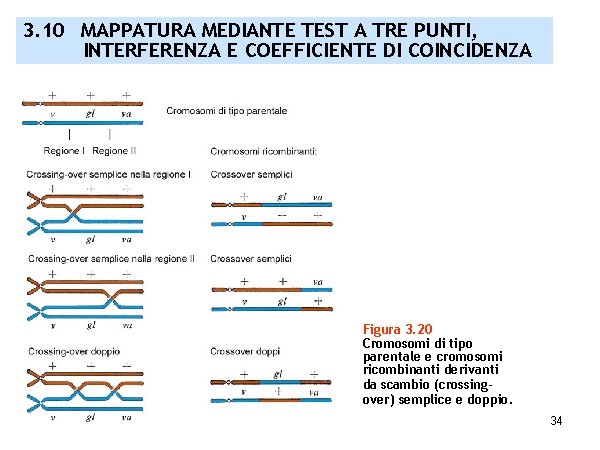
3. 10 MAPPATURA MEDIANTE TEST A TRE PUNTI, INTERFERENZA E COEFFICIENTE DI COINCIDENZA Figura 3. 20 Cromosomi di tipo parentale e cromosomi ricombinanti derivanti da scambio (crossingover) semplice e doppio. 34

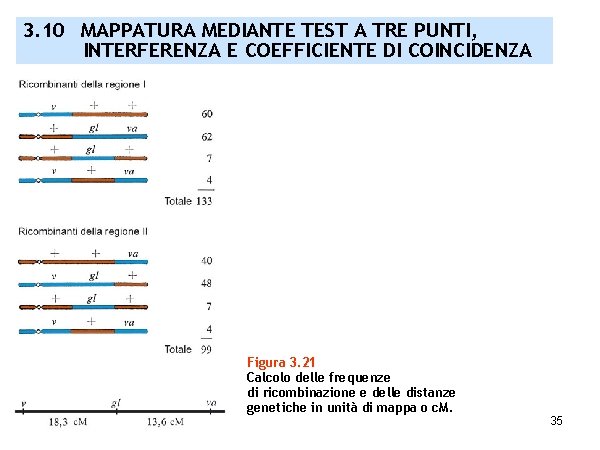
3. 10 MAPPATURA MEDIANTE TEST A TRE PUNTI, INTERFERENZA E COEFFICIENTE DI COINCIDENZA Figura 3. 21 Calcolo delle frequenze di ricombinazione e delle distanze genetiche in unità di mappa o c. M. 35

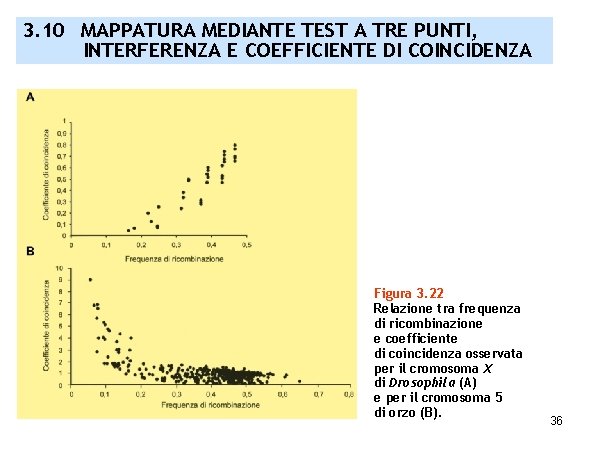
3. 10 MAPPATURA MEDIANTE TEST A TRE PUNTI, INTERFERENZA E COEFFICIENTE DI COINCIDENZA Figura 3. 22 Relazione tra frequenza di ricombinazione e coefficiente di coincidenza osservata per il cromosoma X di Drosophila (A) e per il cromosoma 5 di orzo (B). 36

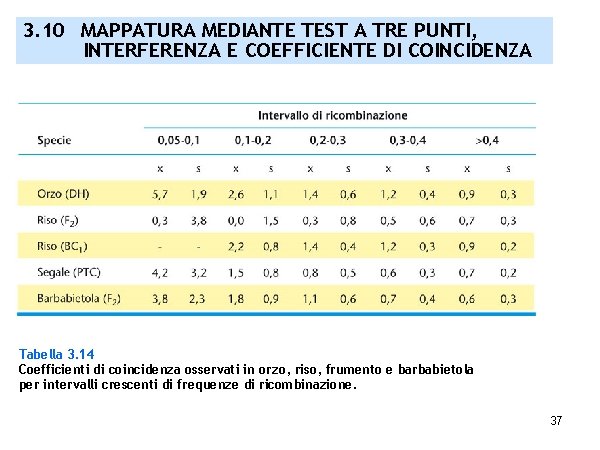
3. 10 MAPPATURA MEDIANTE TEST A TRE PUNTI, INTERFERENZA E COEFFICIENTE DI COINCIDENZA Tabella 3. 14 Coefficienti di coincidenza osservati in orzo, riso, frumento e barbabietola per intervalli crescenti di frequenze di ricombinazione. 37

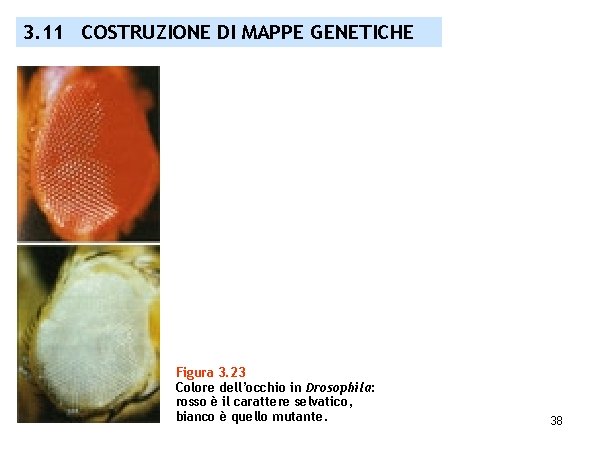
3. 11 COSTRUZIONE DI MAPPE GENETICHE Figura 3. 23 Colore dell’occhio in Drosophila: rosso è il carattere selvatico, bianco è quello mutante. 38

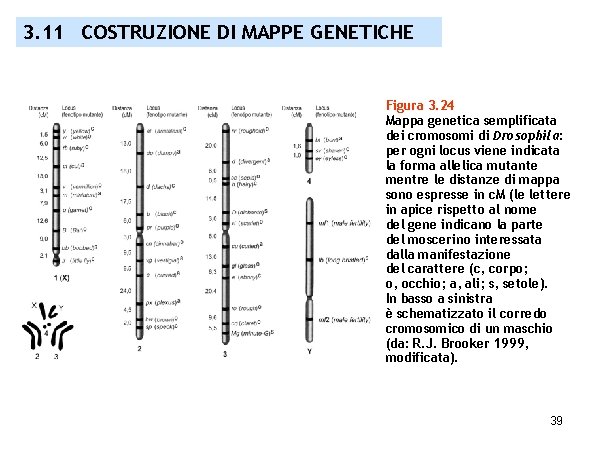
3. 11 COSTRUZIONE DI MAPPE GENETICHE Figura 3. 24 Mappa genetica semplificata dei cromosomi di Drosophila: per ogni locus viene indicata la forma allelica mutante mentre le distanze di mappa sono espresse in c. M (le lettere in apice rispetto al nome del gene indicano la parte del moscerino interessata dalla manifestazione del carattere (c, corpo; o, occhio; a, ali; s, setole). In basso a sinistra è schematizzato il corredo cromosomico di un maschio (da: R. J. Brooker 1999, modificata). 39

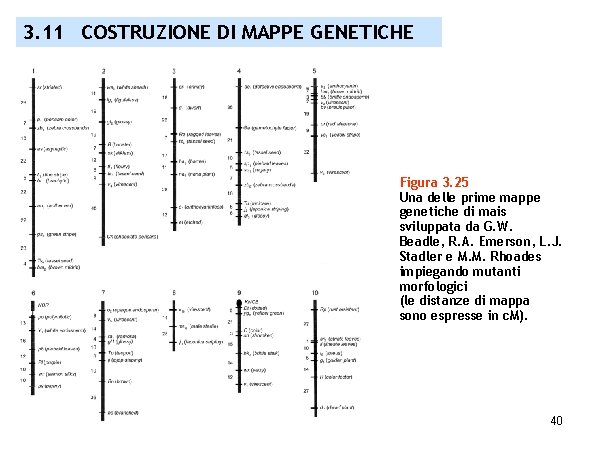
3. 11 COSTRUZIONE DI MAPPE GENETICHE Figura 3. 25 Una delle prime mappe genetiche di mais sviluppata da G. W. Beadle, R. A. Emerson, L. J. Stadler e M. M. Rhoades impiegando mutanti morfologici (le distanze di mappa sono espresse in c. M). 40

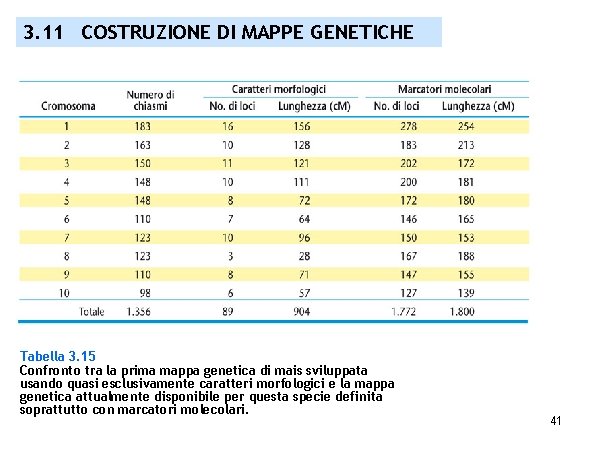
3. 11 COSTRUZIONE DI MAPPE GENETICHE Tabella 3. 15 Confronto tra la prima mappa genetica di mais sviluppata usando quasi esclusivamente caratteri morfologici e la mappa genetica attualmente disponibile per questa specie definita soprattutto con marcatori molecolari. 41

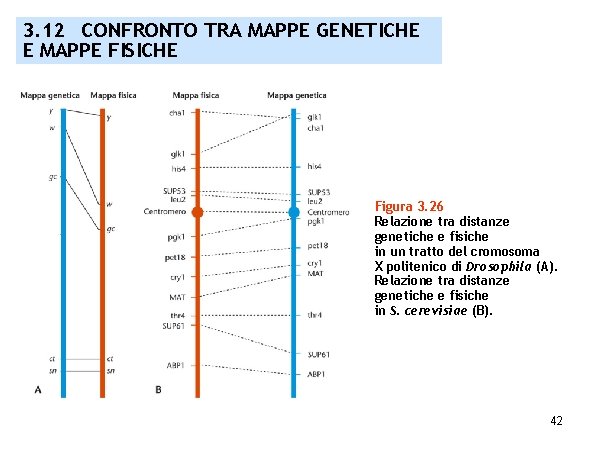
3. 12 CONFRONTO TRA MAPPE GENETICHE E MAPPE FISICHE Figura 3. 26 Relazione tra distanze genetiche e fisiche in un tratto del cromosoma X politenico di Drosophila (A). Relazione tra distanze genetiche e fisiche in S. cerevisiae (B). 42

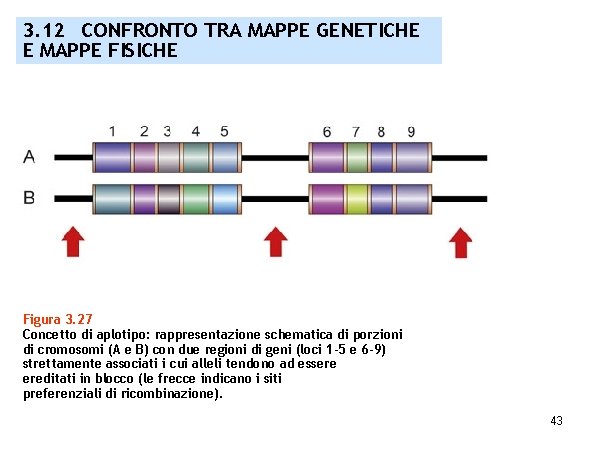
3. 12 CONFRONTO TRA MAPPE GENETICHE E MAPPE FISICHE Figura 3. 27 Concetto di aplotipo: rappresentazione schematica di porzioni di cromosomi (A e B) con due regioni di geni (loci 1 -5 e 6 -9) strettamente associati i cui alleli tendono ad essere ereditati in blocco (le frecce indicano i siti preferenziali di ricombinazione). 43

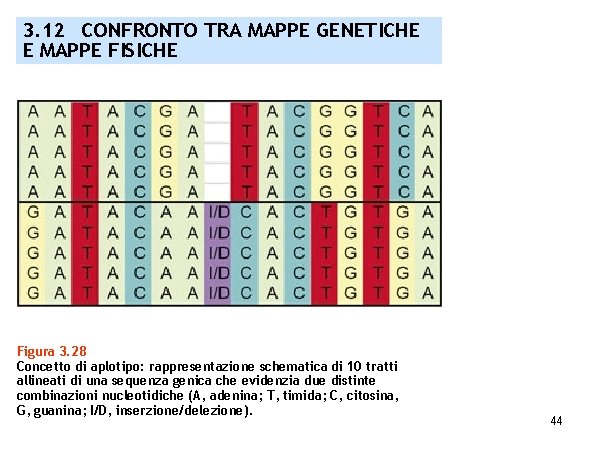
3. 12 CONFRONTO TRA MAPPE GENETICHE E MAPPE FISICHE Figura 3. 28 Concetto di aplotipo: rappresentazione schematica di 10 tratti allineati di una sequenza genica che evidenzia due distinte combinazioni nucleotidiche (A, adenina; T, timida; C, citosina, G, guanina; I/D, inserzione/delezione). 44

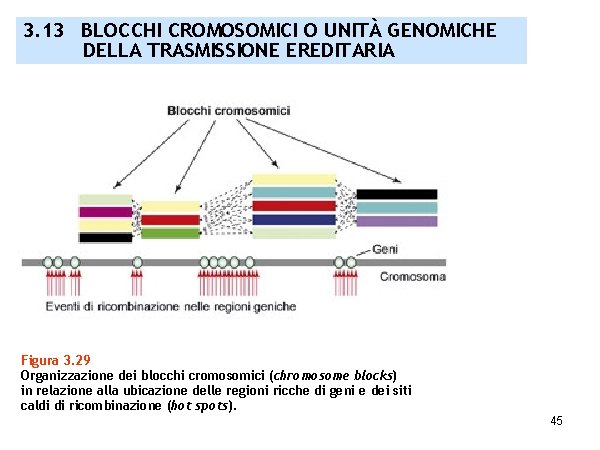
3. 13 BLOCCHI CROMOSOMICI O UNITÀ GENOMICHE DELLA TRASMISSIONE EREDITARIA Figura 3. 29 Organizzazione dei blocchi cromosomici (chromosome blocks) in relazione alla ubicazione delle regioni ricche di geni e dei siti caldi di ricombinazione (hot spots). 45

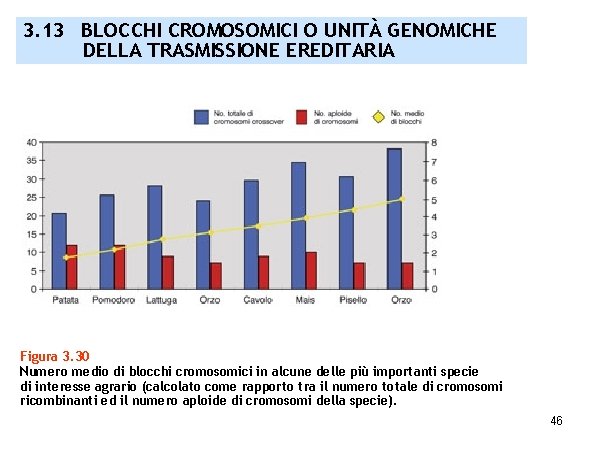
3. 13 BLOCCHI CROMOSOMICI O UNITÀ GENOMICHE DELLA TRASMISSIONE EREDITARIA Figura 3. 30 Numero medio di blocchi cromosomici in alcune delle più importanti specie di interesse agrario (calcolato come rapporto tra il numero totale di cromosomi ricombinanti ed il numero aploide di cromosomi della specie). 46

3. 13 BLOCCHI CROMOSOMICI O UNITÀ GENOMICHE DELLA TRASMISSIONE EREDITARIA Figura 3. 31 Eterosi o vigore ibrido: (A) spighe rappresentative di due linee inbred di mais e del loro ibrido F 1; (B) bacche di melanzana rappresentative della linea pura portaseme, della linea pura impollinante e del loro ibrido F 1 (Foto: Seminis Vegetable Seeds Italia). 47

3. 13 BLOCCHI CROMOSOMICI O UNITÀ GENOMICHE DELLA TRASMISSIONE EREDITARIA Figura 3. 32 Scenari ipotetici delle fasi gametiche tra gli alleli di geni a due loci associati in seguito a diversi eventi di ricombinazione e mutazione: (A) sono evidenti solo due aplotipi in uguale proporzione dimostrando totale disequilibrio di associazione per la mancanza di ricombinazione tra i geni; (B) sono evidenti tre dei quattro possibili aplotipi dimostrando parziale disequilibrio di associazione in seguito a mutazione intercorsa in uno dei geni ma in assenza di ricombinazione tra questi; (C) sono evidenti tutti e quattro i possibili aplotipi ed in uguale proporzione dimostrando completo equilibrio di associazione per la presenza di ricombinazione tra geni. 48

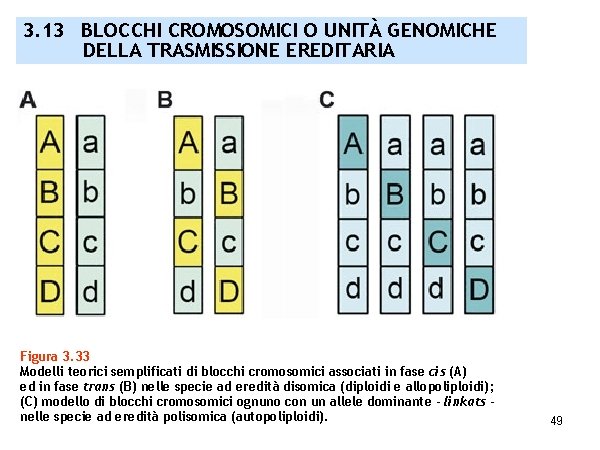
3. 13 BLOCCHI CROMOSOMICI O UNITÀ GENOMICHE DELLA TRASMISSIONE EREDITARIA Figura 3. 33 Modelli teorici semplificati di blocchi cromosomici associati in fase cis (A) ed in fase trans (B) nelle specie ad eredità disomica (diploidi e allopoliploidi); (C) modello di blocchi cromosomici ognuno con un allele dominante - linkats nelle specie ad eredità polisomica (autopoliploidi). 49

3. 14 ANALISI GENETICA DI ASSOCIAZIONE NELL’UOMO Figura 3. 34 Albero genealogico che evidenzia l’associazione tra i loci ABO e NPS 1: gli individui affetti dalla sindrome sono riportati in rosso, mentre il loro gruppo sanguigno viene indicato in lettere. Le frecce indicano i genotipi ricombinanti. 50

3. 14 ANALISI GENETICA DI ASSOCIAZIONE NELL’UOMO Figura 3. 35 Quadrato di Punnett che mostra i genotipi ottenibili dalla coppia della generazione I. 51

3. 14 ANALISI GENETICA DI ASSOCIAZIONE NELL’UOMO Figura 3. 36 Mappa genetica umana con la posizione dei geni responsabili di alcune malattie ereditarie. 52
 Pirandello temi principali
Pirandello temi principali Dynamic linking loader
Dynamic linking loader Denominador y numerador
Denominador y numerador Segmenti adiacenti
Segmenti adiacenti Virginia henderson
Virginia henderson Concetti indicatori e variabili esempi
Concetti indicatori e variabili esempi Porte logiche fondamentali
Porte logiche fondamentali Quali sono gli elementi fondamentali dello stato
Quali sono gli elementi fondamentali dello stato Principi fondamentali della sicurezza sul lavoro
Principi fondamentali della sicurezza sul lavoro Primi teoremi sui limiti
Primi teoremi sui limiti Associazione cocoon roma
Associazione cocoon roma Le leggi ponderali
Le leggi ponderali Il popolo è l'elemento
Il popolo è l'elemento Aris associazione
Aris associazione I fondamentali della pallavolo
I fondamentali della pallavolo Legge di faraday
Legge di faraday Studenti e professori uniti per
Studenti e professori uniti per Test associazione implicita
Test associazione implicita Associazione pazienti italiani colangiocarcinoma
Associazione pazienti italiani colangiocarcinoma Primitiva definizione
Primitiva definizione Kierkegaard punti fondamentali
Kierkegaard punti fondamentali Casa verde
Casa verde Elementi fondamentali dello stato
Elementi fondamentali dello stato Maurizio orlando unifi
Maurizio orlando unifi Fondamentali economici
Fondamentali economici Costituzione associazione professionale
Costituzione associazione professionale Aral crema
Aral crema Slidetodoc.com
Slidetodoc.com Enti fondamentali della geometria
Enti fondamentali della geometria Quali sono gli enti geometrici fondamentali
Quali sono gli enti geometrici fondamentali Principi fondamentali della costituzione mappa
Principi fondamentali della costituzione mappa Grandezze fondamentali e derivate
Grandezze fondamentali e derivate Nicostatin
Nicostatin Adelfi e filadelfi
Adelfi e filadelfi Formule trigonometriche
Formule trigonometriche Aicap
Aicap Anbba
Anbba My job associazione professionale
My job associazione professionale Magnetismo nella materia zanichelli
Magnetismo nella materia zanichelli Gli enti geometrici fondamentali
Gli enti geometrici fondamentali Carabinieri brugherio
Carabinieri brugherio Associazione aicap
Associazione aicap Fondo funzioni fondamentali
Fondo funzioni fondamentali Derivata arcotangente
Derivata arcotangente Struttura della fabula
Struttura della fabula Associazione tecla
Associazione tecla No desenho abaixo a figura
No desenho abaixo a figura Ackerman angle
Ackerman angle What is a linkage institution
What is a linkage institution Genetic linkage
Genetic linkage Linkage disequilibrium
Linkage disequilibrium Mindworks uidaho
Mindworks uidaho Linkage institutions examples
Linkage institutions examples