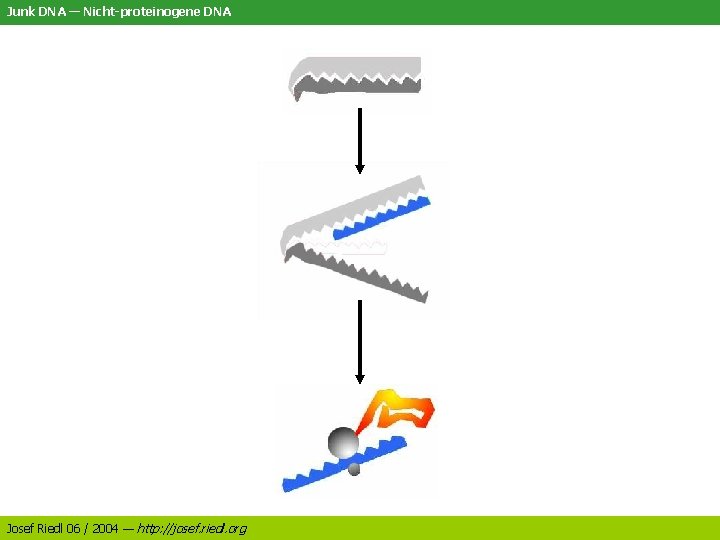
Junk DNA Nichtproteinogene DNA Josef Riedl 06 2004


































- Slides: 64

Junk DNA — Nicht-proteinogene DNA Josef Riedl 06 / 2004 — http: //josef. riedl. org

Junk DNA — Nicht-proteinogene DNA Josef Riedl 06 / 2004 — http: //josef. riedl. org

Junk DNA — Nicht-proteinogene DNA gilt für 2% der menschlichen DNA Rest — ? Josef Riedl 06 / 2004 — http: //josef. riedl. org

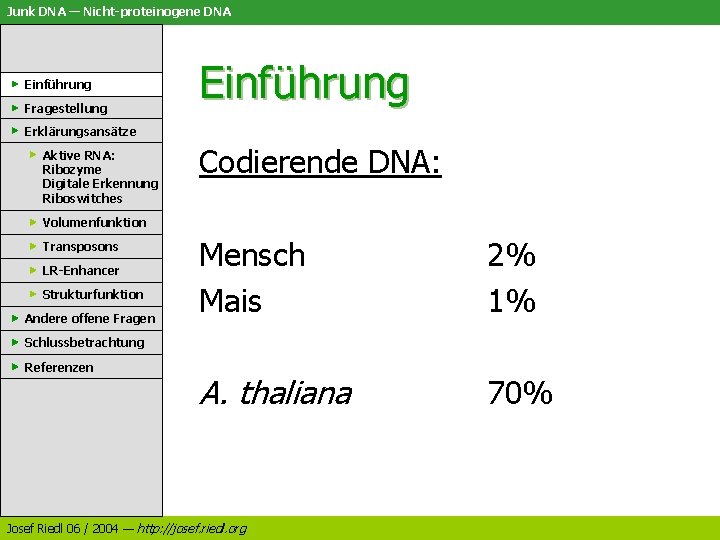
Junk DNA — Nicht-proteinogene DNA Einführung Fragestellung Erklärungsansätze Aktive RNA: Ribozyme Digitale Erkennung Riboswitches Volumenfunktion Transposons LR-Enhancer Einführung Strukturfunktion Andere offene Fragen Schlussbetrachtung Referenzen Josef Riedl 06 / 2004 — http: //josef. riedl. org

Junk DNA — Nicht-proteinogene DNA Einführung Fragestellung Einführung Erklärungsansätze Aktive RNA: Ribozyme Digitale Erkennung Riboswitches Codierende DNA: Volumenfunktion Transposons LR-Enhancer Strukturfunktion Andere offene Fragen Mensch Mais 2% 1% A. thaliana 70% Schlussbetrachtung Referenzen Josef Riedl 06 / 2004 — http: //josef. riedl. org

Junk DNA — Nicht-proteinogene DNA Einführung Fragestellung Einführung Erklärungsansätze Aktive RNA: Ribozyme Digitale Erkennung Riboswitches Volumenfunktion Transposons LR-Enhancer Strukturfunktion Andere offene Fragen Schlussbetrachtung Referenzen Rest: Junk DNA Unnötige DNA ohne erkennbare Bedeutung Nicht-proteincodierende DNA ENCODE; RNome; Transkriptionseinheit Josef Riedl 06 / 2004 — http: //josef. riedl. org

Junk DNA — Nicht-proteinogene DNA Einführung Fragestellung Erklärungsansätze Aktive RNA: Ribozyme Digitale Erkennung Riboswitches Volumenfunktion Transposons LR-Enhancer Ist Junk-DNA überflüssig? Woher kommt Junk-DNA? Strukturfunktion Andere offene Fragen Schlussbetrachtung Was beinhaltet Junk-DNA? Referenzen Josef Riedl 06 / 2004 — http: //josef. riedl. org

Junk DNA — Nicht-proteinogene DNA Einführung Fragestellung Erklärungsansätze Aktive RNA: Ribozyme Digitale Erkennung Riboswitches Volumenfunktion Transposons Indizien: • Fehlende Korrelation: LR-Enhancer Strukturfunktion Andere offene Fragen Schlussbetrachtung Referenzen DNA-Menge Entwicklungsstufe Zahl der Gene • Ungewollte Anhäufung Josef Riedl 06 / 2004 — http: //josef. riedl. org

Junk DNA — Nicht-proteinogene DNA Einführung Fragestellung Erklärungsansätze Aktive RNA: Ribozyme Digitale Erkennung Riboswitches Volumenfunktion Transposons LR-Enhancer Strukturfunktion Andere offene Fragen Schlussbetrachtung Referenzen Mechanismen: • • Genduplikation Stillegung von Genkopien Transposons Retroviren • Mangelnde Selektion Josef Riedl 06 / 2004 — http: //josef. riedl. org

Junk DNA — Nicht-proteinogene DNA Einführung Fragestellung Erklärungsansätze Aktive RNA: Ribozyme Digitale Erkennung Riboswitches Volumenfunktion Transposons LR-Enhancer Neue Überlegung: • 98% überflüssig? Strukturfunktion Andere offene Fragen Schlussbetrachtung Referenzen • Regulatoren und andere Elemente jenseits der Genebene? • Komplexität / Genanzahl? Josef Riedl 06 / 2004 — http: //josef. riedl. org

Junk DNA — Nicht-proteinogene DNA Einführung Fragestellung Erklärungsansätze Aktive RNA: Ribozyme Digitale Erkennung Riboswitches Volumenfunktion Transposons LR-Enhancer Strukturfunktion Andere offene Fragen Schlussbetrachtung • „Schätze in Junk-DNA“ • Neue Erkenntnisse (und Spekulation) in verschiedenen Richtungen Referenzen • Keine allgemeine Methode Josef Riedl 06 / 2004 — http: //josef. riedl. org

Junk DNA — Nicht-proteinogene DNA Einführung Fragestellung Erklärungsansätze Aktive RNA: Ribozyme Digitale Erkennung Riboswitches Volumenfunktion Transposons LR-Enhancer Aktive RNA Strukturfunktion Andere offene Fragen Schlussbetrachtung Referenzen Josef Riedl 06 / 2004 — http: //josef. riedl. org

Junk DNA — Nicht-proteinogene DNA Einführung Fragestellung Aktive RNA Erklärungsansätze Aktive RNA: Ribozyme Digitale Erkennung Riboswitches Ribozyme Volumenfunktion Transposons LR-Enhancer Strukturfunktion Andere offene Fragen Schlussbetrachtung - katalytisch aktiv - analoge Erkennung (wie Proteine) Referenzen Josef Riedl 06 / 2004 — http: //josef. riedl. org

Junk DNA — Nicht-proteinogene DNA Einführung Fragestellung Aktive RNA Erklärungsansätze Aktive RNA: Ribozyme Digitale Erkennung Riboswitches Volumenfunktion Transposons LR-Enhancer Strukturfunktion Andere offene Fragen Schlussbetrachtung Referenzen Josef Riedl 06 / 2004 — http: //josef. riedl. org

Junk DNA — Nicht-proteinogene DNA Einführung Fragestellung Aktive RNA Erklärungsansätze Aktive RNA: Ribozyme Digitale Erkennung Riboswitches Volumenfunktion Transposons LR-Enhancer Strukturfunktion Andere offene Fragen Schlussbetrachtung Referenzen Josef Riedl 06 / 2004 — http: //josef. riedl. org

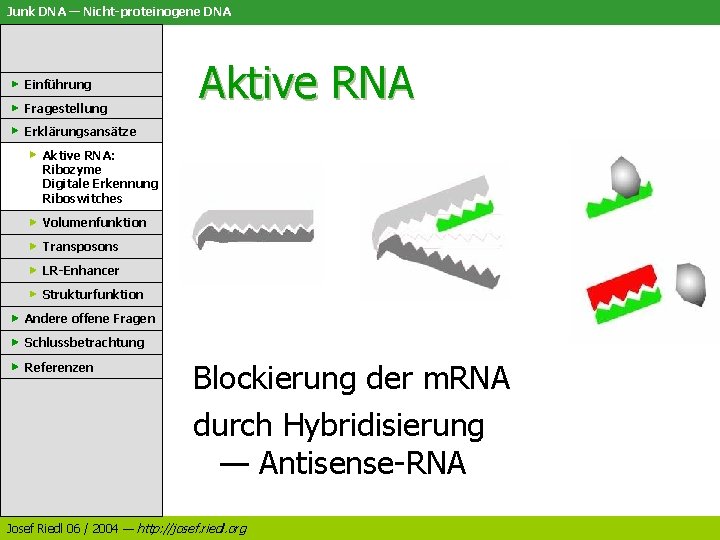
Junk DNA — Nicht-proteinogene DNA Einführung Fragestellung Aktive RNA Erklärungsansätze Aktive RNA: Ribozyme Digitale Erkennung Riboswitches Antisense-RNA Volumenfunktion Transposons LR-Enhancer Strukturfunktion Andere offene Fragen Schlussbetrachtung - regulatorisch aktiv - digitale Erkennung (Komplementärsequenz) Referenzen - wirkt allein, simpler Mechanismus Josef Riedl 06 / 2004 — http: //josef. riedl. org

Junk DNA — Nicht-proteinogene DNA Einführung Fragestellung Aktive RNA Erklärungsansätze Aktive RNA: Ribozyme Digitale Erkennung Riboswitches Volumenfunktion Transposons LR-Enhancer Strukturfunktion Andere offene Fragen Schlussbetrachtung Referenzen Blockierung der m. RNA durch Hybridisierung — Antisense-RNA Josef Riedl 06 / 2004 — http: //josef. riedl. org

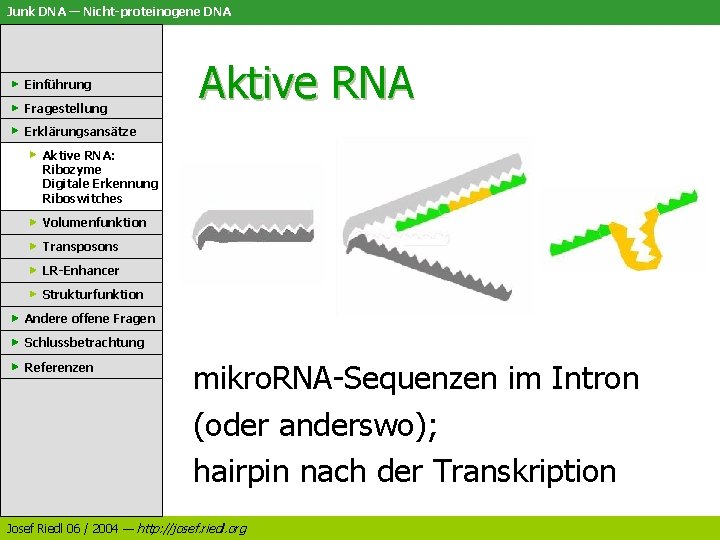
Junk DNA — Nicht-proteinogene DNA Einführung Fragestellung Aktive RNA Erklärungsansätze Aktive RNA: Ribozyme Digitale Erkennung Riboswitches Mikro-RNA und RNAi Volumenfunktion Transposons LR-Enhancer Strukturfunktion Andere offene Fragen Schlussbetrachtung - regulatorisch aktiv - digitale Erkennung (Komplementärsequenz) Referenzen - komplexe Maschinerie Josef Riedl 06 / 2004 — http: //josef. riedl. org

Junk DNA — Nicht-proteinogene DNA Einführung Fragestellung Aktive RNA Erklärungsansätze Aktive RNA: Ribozyme Digitale Erkennung Riboswitches Volumenfunktion Transposons LR-Enhancer Strukturfunktion Andere offene Fragen Schlussbetrachtung Referenzen mikro. RNA-Sequenzen im Intron (oder anderswo); hairpin nach der Transkription Josef Riedl 06 / 2004 — http: //josef. riedl. org

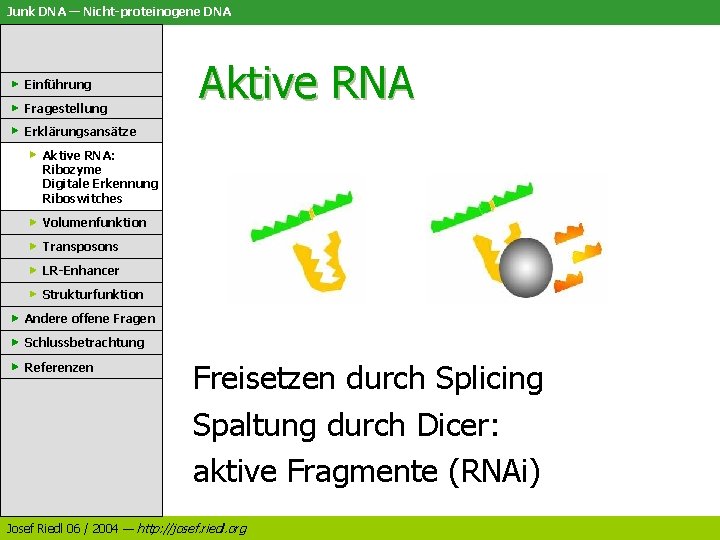
Junk DNA — Nicht-proteinogene DNA Einführung Fragestellung Aktive RNA Erklärungsansätze Aktive RNA: Ribozyme Digitale Erkennung Riboswitches Volumenfunktion Transposons LR-Enhancer Strukturfunktion Andere offene Fragen Schlussbetrachtung Referenzen Freisetzen durch Splicing Spaltung durch Dicer: aktive Fragmente (RNAi) Josef Riedl 06 / 2004 — http: //josef. riedl. org

Junk DNA — Nicht-proteinogene DNA Einführung Fragestellung Aktive RNA Erklärungsansätze Aktive RNA: Ribozyme Digitale Erkennung Riboswitches Volumenfunktion Transposons LR-Enhancer Strukturfunktion Andere offene Fragen Schlussbetrachtung Referenzen Erkennen der m. RNA: Hybridisierung Abbau durch Enzymkomplex — Andere Funktionen? Josef Riedl 06 / 2004 — http: //josef. riedl. org

Junk DNA — Nicht-proteinogene DNA Einführung Fragestellung Aktive RNA Erklärungsansätze Aktive RNA: Ribozyme Digitale Erkennung Riboswitches Volumenfunktion Transposons LR-Enhancer - regulatorisch und katalytisch aktiv Strukturfunktion Andere offene Fragen Schlussbetrachtung Referenzen - analoge Erkennung (niedermolekulare Substanzen) Josef Riedl 06 / 2004 — http: //josef. riedl. org

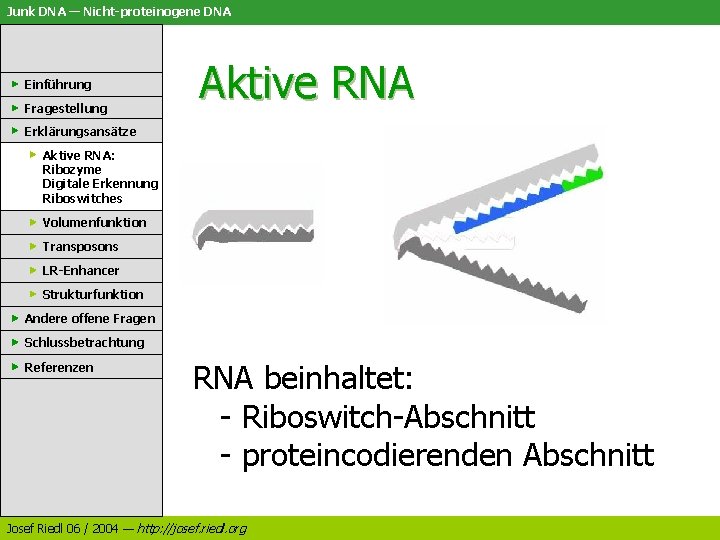
Junk DNA — Nicht-proteinogene DNA Einführung Fragestellung Aktive RNA Erklärungsansätze Aktive RNA: Ribozyme Digitale Erkennung Riboswitches Volumenfunktion Transposons LR-Enhancer Strukturfunktion Andere offene Fragen Schlussbetrachtung Referenzen RNA beinhaltet: - Riboswitch-Abschnitt - proteincodierenden Abschnitt Josef Riedl 06 / 2004 — http: //josef. riedl. org

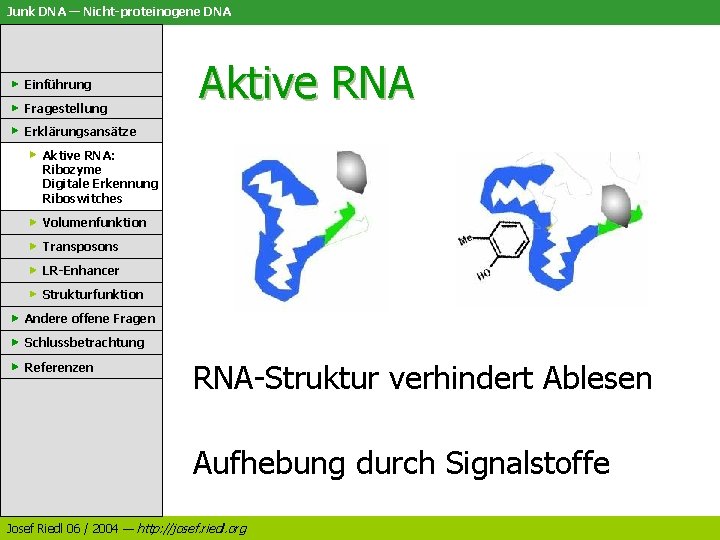
Junk DNA — Nicht-proteinogene DNA Einführung Fragestellung Aktive RNA Erklärungsansätze Aktive RNA: Ribozyme Digitale Erkennung Riboswitches Volumenfunktion Transposons LR-Enhancer Strukturfunktion Andere offene Fragen Schlussbetrachtung Referenzen RNA-Struktur verhindert Ablesen Aufhebung durch Signalstoffe Josef Riedl 06 / 2004 — http: //josef. riedl. org

Junk DNA — Nicht-proteinogene DNA Einführung Fragestellung Aktive RNA: Bewertung Erklärungsansätze Aktive RNA: Ribozyme Digitale Erkennung Riboswitches Volumenfunktion Transposons LR-Enhancer Strukturfunktion Bewertung - Schätzung Maus: 70000 Einheiten vs. 30000 Gene - Pseudogene Andere offene Fragen Schlussbetrachtung Referenzen Erforschung - Zufallsmutagenese (z. B. Maus) Josef Riedl 06 / 2004 — http: //josef. riedl. org

Junk DNA — Nicht-proteinogene DNA Einführung Fragestellung Erklärungsansätze Aktive RNA: Ribozyme Digitale Erkennung Riboswitches Volumenfunktion Transposons LR-Enhancer Volumenfunktion Strukturfunktion Andere offene Fragen Schlussbetrachtung Referenzen Josef Riedl 06 / 2004 — http: //josef. riedl. org

Junk DNA — Nicht-proteinogene DNA Einführung Fragestellung Volumenfunktion Erklärungsansätze Aktive RNA: Ribozyme Digitale Erkennung Riboswitches Volumenfunktion Keine Korrelation: DNA-Menge – Genzahl Transposons LR-Enhancer Strukturfunktion Andere offene Fragen Schlussbetrachtung Referenzen Korrelation: DNA-Menge – Kernvolumen DNA-Menge – Zellvolumen (Th. Cavalier-Smith) Josef Riedl 06 / 2004 — http: //josef. riedl. org

Junk DNA — Nicht-proteinogene DNA Einführung Fragestellung Volumenfunktion Erklärungsansätze Aktive RNA: Ribozyme Digitale Erkennung Riboswitches Untersuchungsobjekt: Volumenfunktion Transposons LR-Enhancer Cryptomonaden Strukturfunktion Andere offene Fragen Schlussbetrachtung Referenzen Josef Riedl 06 / 2004 — http: //josef. riedl. org

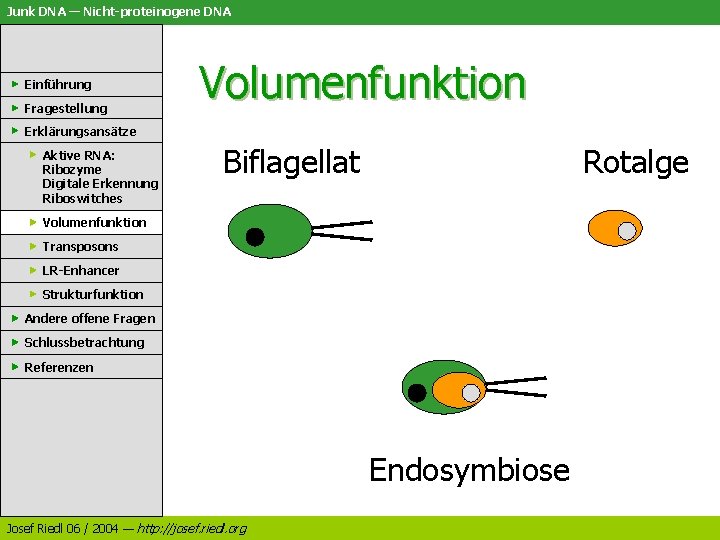
Junk DNA — Nicht-proteinogene DNA Einführung Fragestellung Volumenfunktion Erklärungsansätze Aktive RNA: Ribozyme Digitale Erkennung Riboswitches Biflagellat Rotalge Volumenfunktion Transposons LR-Enhancer Strukturfunktion Andere offene Fragen Schlussbetrachtung Referenzen Endosymbiose Josef Riedl 06 / 2004 — http: //josef. riedl. org

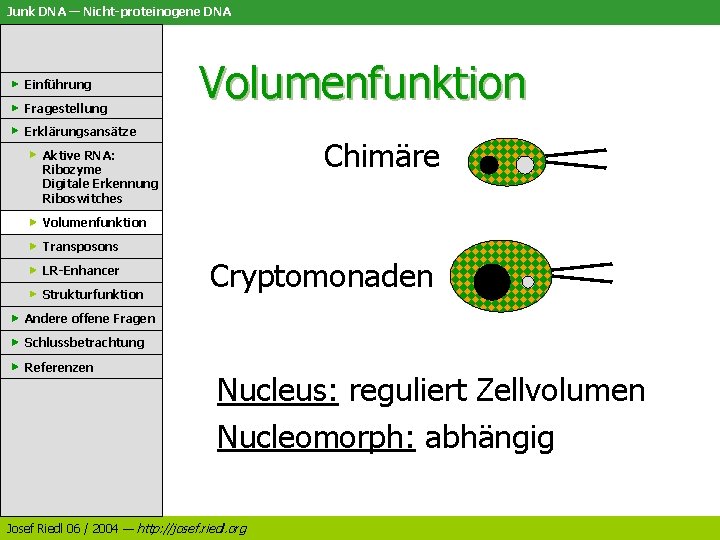
Junk DNA — Nicht-proteinogene DNA Einführung Fragestellung Volumenfunktion Erklärungsansätze Chimäre Aktive RNA: Ribozyme Digitale Erkennung Riboswitches Volumenfunktion Transposons LR-Enhancer Strukturfunktion Cryptomonaden Andere offene Fragen Schlussbetrachtung Referenzen Nucleus: reguliert Zellvolumen Nucleomorph: abhängig Josef Riedl 06 / 2004 — http: //josef. riedl. org

Junk DNA — Nicht-proteinogene DNA Einführung Fragestellung Volumenfunktion Erklärungsansätze Aktive RNA: Ribozyme Digitale Erkennung Riboswitches Volumenfunktion Transposons LR-Enhancer Strukturfunktion Andere offene Fragen Schlussbetrachtung Referenzen Unabhängige Entwicklung: • Nucleus: DNA-Gehalt ~ Zellvolumen Evolutive Vergrösserung • Nucleomorph: DNA-Gehalt unabhängig Evolutive Reduzierung Josef Riedl 06 / 2004 — http: //josef. riedl. org

Junk DNA — Nicht-proteinogene DNA Einführung Fragestellung Volumenfunktion Erklärungsansätze Aktive RNA: Ribozyme Digitale Erkennung Riboswitches Volumenfunktion Transposons LR-Enhancer Strukturfunktion Schlussfolgerung: Selektion gegen und für DNAAnsammlung ist möglich! Andere offene Fragen Schlussbetrachtung Referenzen Für „Junk-DNA“ muss es einen evolutiven Grund geben! Josef Riedl 06 / 2004 — http: //josef. riedl. org

Junk DNA — Nicht-proteinogene DNA Einführung Fragestellung Volumenfunktion Erklärungsansätze Aktive RNA: Ribozyme Digitale Erkennung Riboswitches Volumenfunktion Transposons LR-Enhancer Strukturfunktion Andere offene Fragen Schlussbetrachtung Referenzen Erklärung: Größere Zelle => mehr Proteine Mehr Proteine => mehr. Ablesen Mehr Ablesen => mehr Enzyme Mehr Enzyme => mehr Platzbedarf Mehr Platzbedarf => Volumenschaffende sekundäre DNA Josef Riedl 06 / 2004 — http: //josef. riedl. org

Junk DNA — Nicht-proteinogene DNA Einführung Fragestellung Erklärungsansätze Aktive RNA: Ribozyme Digitale Erkennung Riboswitches Volumenfunktion Transposons LR-Enhancer Strukturfunktion Volumenfunktion: Bewertung Wichtigste Erkenntnis: Selektion gegen/für Junk-DNA Andere offene Fragen Schlussbetrachtung Referenzen Josef Riedl 06 / 2004 — http: //josef. riedl. org

Junk DNA — Nicht-proteinogene DNA Einführung Fragestellung Erklärungsansätze Aktive RNA: Ribozyme Digitale Erkennung Riboswitches Volumenfunktion Transposons LR-Enhancer Transposons Strukturfunktion Andere offene Fragen Schlussbetrachtung Referenzen Josef Riedl 06 / 2004 — http: //josef. riedl. org

Junk DNA — Nicht-proteinogene DNA Einführung Fragestellung Transposons Erklärungsansätze Aktive RNA: Ribozyme Digitale Erkennung Riboswitches Volumenfunktion Transposons LR-Enhancer • „springende“ Genelemente • Mechanismus: Transposase Strukturfunktion Andere offene Fragen Schlussbetrachtung • teilweise ± stillgelegt Referenzen Josef Riedl 06 / 2004 — http: //josef. riedl. org

Junk DNA — Nicht-proteinogene DNA Einführung Fragestellung Transposons Erklärungsansätze Aktive RNA: Ribozyme Digitale Erkennung Riboswitches Volumenfunktion • erzeugen „Narben“ beim Ausschneiden Transposons LR-Enhancer Strukturfunktion • erzeugen teilweise Kopien Andere offene Fragen Schlussbetrachtung Referenzen • können Gene durch Disruption zerstören Josef Riedl 06 / 2004 — http: //josef. riedl. org

Junk DNA — Nicht-proteinogene DNA Einführung Fragestellung Transposons Erklärungsansätze Aktive RNA: Ribozyme Digitale Erkennung Riboswitches Transposons und Junk-DNA: Volumenfunktion Transposons LR-Enhancer Strukturfunktion Andere offene Fragen Schlussbetrachtung Referenzen Direkt: Transposons erzeugen wertlose Bereiche durch Disruption und Selbstvervielfältigung Josef Riedl 06 / 2004 — http: //josef. riedl. org

Junk DNA — Nicht-proteinogene DNA Einführung Fragestellung Transposons Erklärungsansätze Aktive RNA: Ribozyme Digitale Erkennung Riboswitches Transposons und Junk-DNA: Volumenfunktion Transposons LR-Enhancer Strukturfunktion Andere offene Fragen Schlussbetrachtung Referenzen Indirekt: Transposonaktivität macht Mehrfachkopien von Genen notwendig und sinnvoll Josef Riedl 06 / 2004 — http: //josef. riedl. org

Junk DNA — Nicht-proteinogene DNA Einführung Fragestellung Transposons: Bewertung Erklärungsansätze Aktive RNA: Ribozyme Digitale Erkennung Riboswitches • eindeutig nachgewiesen Volumenfunktion Transposons LR-Enhancer • fehlende Selektion—? Strukturfunktion Andere offene Fragen Schlussbetrachtung Referenzen Josef Riedl 06 / 2004 — http: //josef. riedl. org

Junk DNA — Nicht-proteinogene DNA Einführung Fragestellung Erklärungsansätze Aktive RNA: Ribozyme Digitale Erkennung Riboswitches Volumenfunktion Transposons LR-Enhancer Long-Range Enhancer Strukturfunktion Andere offene Fragen Schlussbetrachtung Referenzen Josef Riedl 06 / 2004 — http: //josef. riedl. org

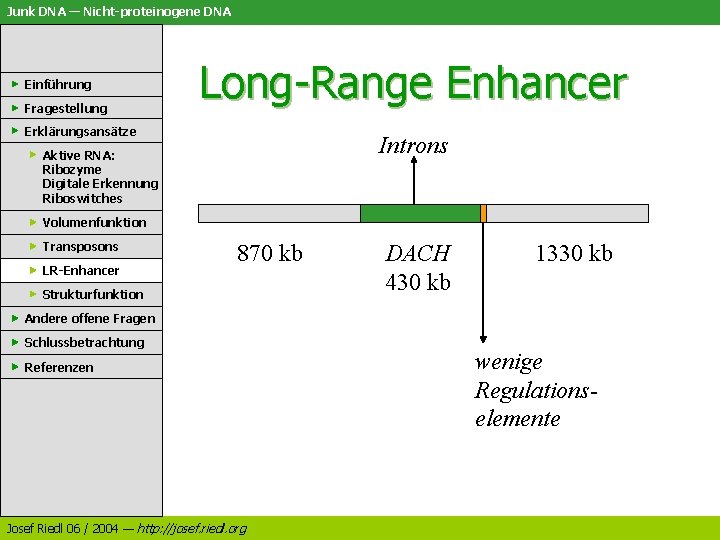
Junk DNA — Nicht-proteinogene DNA Einführung Fragestellung Long-Range Enhancer Erklärungsansätze Aktive RNA: Ribozyme Digitale Erkennung Riboswitches Experimenteller Ansatz: Volumenfunktion Transposons LR-Enhancer Scanning Human Gene Deserts Strukturfunktion Andere offene Fragen Schlussbetrachtung Referenzen Beispiel: DACH (u. a. Gehirnentwicklung) Josef Riedl 06 / 2004 — http: //josef. riedl. org

Junk DNA — Nicht-proteinogene DNA Einführung Fragestellung Long-Range Enhancer Erklärungsansätze Introns Aktive RNA: Ribozyme Digitale Erkennung Riboswitches Volumenfunktion Transposons LR-Enhancer 870 kb Strukturfunktion DACH 430 kb 1330 kb Andere offene Fragen Schlussbetrachtung Referenzen Josef Riedl 06 / 2004 — http: //josef. riedl. org wenige Regulationselemente

Junk DNA — Nicht-proteinogene DNA Einführung Fragestellung Long-Range Enhancer Erklärungsansätze Aktive RNA: Ribozyme Digitale Erkennung Riboswitches Volumenfunktion Transposons LR-Enhancer Strukturfunktion Andere offene Fragen Schlussbetrachtung Referenzen Vergleich mit anderen Spezies: 1098 nicht-codierende Sequenzen konserviert in Maus und Mensch (Kriterium: > 100 bp, > 70%) Josef Riedl 06 / 2004 — http: //josef. riedl. org

Junk DNA — Nicht-proteinogene DNA Einführung Fragestellung Long-Range Enhancer Erklärungsansätze Aktive RNA: Ribozyme Digitale Erkennung Riboswitches Volumenfunktion Transposons LR-Enhancer Strukturfunktion Andere offene Fragen Schlussbetrachtung Referenzen Vergleich mit anderen Spezies: 32 nicht-codierende Sequenzen konserviert in Maus, Mensch, Frosch, Zebrafisch, Fugu entspricht 1 Mrd. Jahre Evolution Josef Riedl 06 / 2004 — http: //josef. riedl. org

Junk DNA — Nicht-proteinogene DNA Einführung Fragestellung Long-Range Enhancer Erklärungsansätze Aktive RNA: Ribozyme Digitale Erkennung Riboswitches Volumenfunktion Transposons LR-Enhancer Strukturfunktion Andere offene Fragen Schlussbetrachtung Auswahl 9 konservierte Sequenzen (aus Introns und den beiden flankierenden „Genwüsten“) Referenzen Josef Riedl 06 / 2004 — http: //josef. riedl. org

Junk DNA — Nicht-proteinogene DNA Einführung Fragestellung Long-Range Enhancer Erklärungsansätze Aktive RNA: Ribozyme Digitale Erkennung Riboswitches Testverfahren: Enhancer? ? Volumenfunktion Transposons LR-Enhancer Strukturfunktion Andere offene Fragen ? Schlussbetrachtung b-Galactosidase Referenzen Minimal-Promoter (aus Hsp 68) Expression in transgenen Mäusen Josef Riedl 06 / 2004 — http: //josef. riedl. org

Junk DNA — Nicht-proteinogene DNA Einführung Fragestellung Long-Range Enhancer Erklärungsansätze Aktive RNA: Ribozyme Digitale Erkennung Riboswitches Volumenfunktion Transposons LR-Enhancer Strukturfunktion Ergebnis 7 konservierte Sequenzen wirken als Enhancer Andere offene Fragen Schlussbetrachtung Referenzen Josef Riedl 06 / 2004 — http: //josef. riedl. org

Junk DNA — Nicht-proteinogene DNA Einführung Fragestellung Long-Range Enhancer Erklärungsansätze Aktive RNA: Ribozyme Digitale Erkennung Riboswitches Volumenfunktion Transposons LR-Enhancer Strukturfunktion Ergebnis 7 konservierte Sequenzen wirken als Enhancer Andere offene Fragen Schlussbetrachtung Referenzen Josef Riedl 06 / 2004 — http: //josef. riedl. org

Junk DNA — Nicht-proteinogene DNA Einführung Fragestellung Erklärungsansätze Aktive RNA: Ribozyme Digitale Erkennung Riboswitches Volumenfunktion Transposons LR-Enhancer Long-Range Enhancer: Bewertung • Nicht-codierende DNA ist konserviert Strukturfunktion Andere offene Fragen Schlussbetrachtung Referenzen • Regulationsbereichen von Genen deutlich größer als angenommen! Josef Riedl 06 / 2004 — http: //josef. riedl. org

Junk DNA — Nicht-proteinogene DNA Einführung Fragestellung Erklärungsansätze Aktive RNA: Ribozyme Digitale Erkennung Riboswitches Volumenfunktion Transposons LR-Enhancer Strukturfunktion Andere offene Fragen Schlussbetrachtung Referenzen Josef Riedl 06 / 2004 — http: //josef. riedl. org

Junk DNA — Nicht-proteinogene DNA Einführung Fragestellung Strukturfunktion Erklärungsansätze Aktive RNA: Ribozyme Digitale Erkennung Riboswitches DNA in Chromosomen: Volumenfunktion Transposons LR-Enhancer • Telomere Strukturfunktion Andere offene Fragen Schlussbetrachtung • Centromere Referenzen Josef Riedl 06 / 2004 — http: //josef. riedl. org

Junk DNA — Nicht-proteinogene DNA Einführung Fragestellung Strukturfunktion Erklärungsansätze Aktive RNA: Ribozyme Digitale Erkennung Riboswitches DNA in Kernen: Volumenfunktion Transposons LR-Enhancer • Euchromatin Strukturfunktion Andere offene Fragen Schlussbetrachtung • Heterochromatin Referenzen Josef Riedl 06 / 2004 — http: //josef. riedl. org

Junk DNA — Nicht-proteinogene DNA Einführung Fragestellung Strukturfunktion Erklärungsansätze Aktive RNA: Ribozyme Digitale Erkennung Riboswitches DNA bei der Zellteilung: Volumenfunktion Transposons LR-Enhancer • Chromosomenverteilung Strukturfunktion Andere offene Fragen Schlussbetrachtung Referenzen Josef Riedl 06 / 2004 — http: //josef. riedl. org

Junk DNA — Nicht-proteinogene DNA Einführung Fragestellung Erklärungsansätze Aktive RNA: Ribozyme Digitale Erkennung Riboswitches Volumenfunktion Transposons LR-Enhancer Strukturfunktion: Bewertung • Sequenzabschnitte für Bindung und Erkennung durch Proteine? Andere offene Fragen Schlussbetrachtung Referenzen • Ausmaß schwer einzuschätzen Josef Riedl 06 / 2004 — http: //josef. riedl. org

Junk DNA — Nicht-proteinogene DNA Einführung Fragestellung Andere offene Fragen Erklärungsansätze Aktive RNA: Ribozyme Digitale Erkennung Riboswitches Volumenfunktion • Epigenetische Ebene Transposons LR-Enhancer Strukturfunktion Andere offene Fragen Schlussbetrachtung • Einfluss der chromosomalen Struktur Referenzen Variationen zwischen Zellen! Josef Riedl 06 / 2004 — http: //josef. riedl. org

Junk DNA — Nicht-proteinogene DNA Einführung Fragestellung Andere offene Fragen Erklärungsansätze Aktive RNA: Ribozyme Digitale Erkennung Riboswitches Volumenfunktion Spekulationen: z. B. Unterschied Affe / Mensch? Transposons LR-Enhancer Strukturfunktion Andere offene Fragen • 98% der Gene (!) identisch Schlussbetrachtung Referenzen • Rolle der Junk-DNA. . . ? Josef Riedl 06 / 2004 — http: //josef. riedl. org

Junk DNA — Nicht-proteinogene DNA Einführung Fragestellung Andere offene Fragen Erklärungsansätze Aktive RNA: Ribozyme Digitale Erkennung Riboswitches Aber: Volumenfunktion Transposons LR-Enhancer • 98% des Genoms identisch Strukturfunktion Andere offene Fragen Schlussbetrachtung Referenzen • Gene bereits identifiziert Junk-DNA ist nicht Lösung aller Probleme! Josef Riedl 06 / 2004 — http: //josef. riedl. org

Junk DNA — Nicht-proteinogene DNA Einführung Fragestellung Schlussbetrachtung Erklärungsansätze Aktive RNA: Ribozyme Digitale Erkennung Riboswitches Volumenfunktion Keine eindeutige Antwort Transposons LR-Enhancer Strukturfunktion Andere offene Fragen Schlussbetrachtung . . . aber: Neue Sichtweise über Gene hinaus! Referenzen Josef Riedl 06 / 2004 — http: //josef. riedl. org

Junk DNA — Nicht-proteinogene DNA Josef Riedl 06 / 2004 — http: //josef. riedl. org

Junk DNA — Nicht-proteinogene DNA Josef Riedl 06 / 2004 — http: //josef. riedl. org

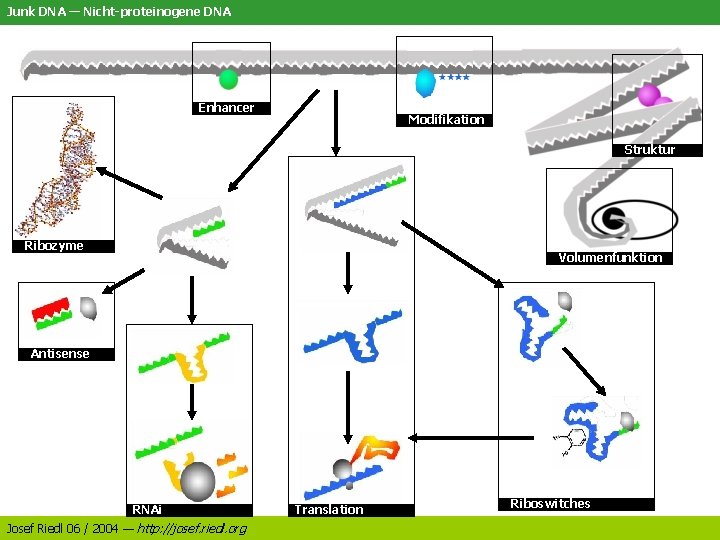
Junk DNA — Nicht-proteinogene DNA Enhancer Modifikation Struktur Ribozyme Volumenfunktion Antisense RNAi Josef Riedl 06 / 2004 — http: //josef. riedl. org Translation Riboswitches

Junk DNA — Nicht-proteinogene DNA Ende Vielen Dank für Ihre Aufmerksamkeit! Josef Riedl 06 / 2004 — http: //josef. riedl. org

Junk DNA — Nicht-proteinogene DNA Einführung Fragestellung Referenzen (Auswahl) Erklärungsansätze Aktive RNA: Ribozyme Digitale Erkennung Riboswitches Volumenfunktion • Allgemein: Preziosen im DNA-Schrott, Spektrum der Wissenschaft 02/2004, 68 • RNA: An Expanding Universe of Noncoding RNAs, Science 296 (2002), 1260 • Volumenfunktion: Eukaryotic non-coding DNA is functional (. . . ), Proc. R. Soc. Lond. B. 266, 2053 • LR-Enhancer: Scanning Human Gene Deserts (. . . ), Science 302 (2003), 413 • http: //josef. riedl. org/junk-dna. pdf Transposons LR-Enhancer Strukturfunktion Andere offene Fragen Schlussbetrachtung Referenzen Josef Riedl 06 / 2004 — http: //josef. riedl. org
 Alexander riedl
Alexander riedl Junk food vs healthy food project
Junk food vs healthy food project Which kingdom is considered the “junk drawer”?
Which kingdom is considered the “junk drawer”? Junk food science
Junk food science Red flags of junk science
Red flags of junk science Space junk the space age began
Space junk the space age began Proyecto educativo
Proyecto educativo 10 red flags of junk science
10 red flags of junk science Parallelism examples
Parallelism examples Polo y servicio meaning
Polo y servicio meaning Conclusion of healthy food and junk food
Conclusion of healthy food and junk food Riddle about food
Riddle about food Junk shuttle
Junk shuttle Expected capital gains yield formula
Expected capital gains yield formula Role play on healthy food and junk food
Role play on healthy food and junk food Fast food paragraph
Fast food paragraph Healthy food vs junk
Healthy food vs junk Introduction of fast food
Introduction of fast food Junk drawer robotics
Junk drawer robotics Junk bond rating
Junk bond rating Role play on healthy food and junk food
Role play on healthy food and junk food Hotel+olsanka+prag
Hotel+olsanka+prag Josef istler
Josef istler Josef sibille
Josef sibille Joseph stalin animal farm
Joseph stalin animal farm Josef fousek
Josef fousek Verblühter löwenzahn gedicht
Verblühter löwenzahn gedicht Josef stalin referat
Josef stalin referat Josef partykiewicz
Josef partykiewicz Josef wojciechowicz
Josef wojciechowicz Strsk
Strsk Josef mitteregger
Josef mitteregger Adam josef cüppers
Adam josef cüppers Freud
Freud Josef zítek
Josef zítek Josef mathauser
Josef mathauser Lesestile
Lesestile Joseffritzel
Joseffritzel Josef förster
Josef förster Josef kote peintre
Josef kote peintre Josef leisen
Josef leisen Stříhali dohola malého chlapečka motivy
Stříhali dohola malého chlapečka motivy Velké, širé, rodné lány rozbor
Velké, širé, rodné lány rozbor Josef weidendorfer
Josef weidendorfer Hans-josef klauck
Hans-josef klauck Josef witiska
Josef witiska New york school
New york school Josef lada prezentace
Josef lada prezentace Josef guggenmos wer bin ich lösung
Josef guggenmos wer bin ich lösung Josef weigel
Josef weigel Josef fritzl
Josef fritzl Josef hallberg
Josef hallberg Josef jungmann prezentace
Josef jungmann prezentace Franz josef gellert
Franz josef gellert Franz josef och
Franz josef och Josef lada wikipedie
Josef lada wikipedie Josef kajetán tyl prezentace
Josef kajetán tyl prezentace Franz josef mone schule
Franz josef mone schule Josef formánek
Josef formánek Josef bei potifar grundschule
Josef bei potifar grundschule Josef men
Josef men Wer bin ich josef guggenmos schmetterling
Wer bin ich josef guggenmos schmetterling Josef janko
Josef janko Josef václav sládek lumírovci
Josef václav sládek lumírovci Unternehmensmanagement definition
Unternehmensmanagement definition