Flynn SISD single instruction stream over a single






















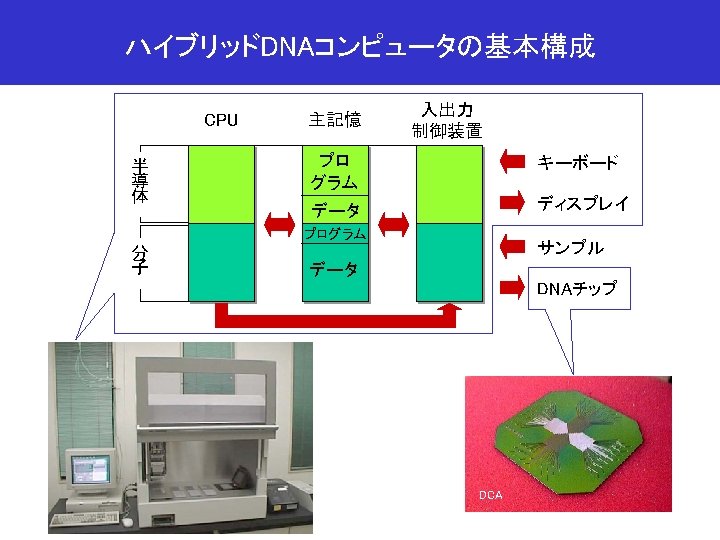
![ハイブリッドDNAコンピュータによるプログラムの実行 [Instrument] [Reset Counter] 0 [Home Position] 0 [MJ-Open Lid] ・・・ [Get 1(0)] [Get ハイブリッドDNAコンピュータによるプログラムの実行 [Instrument] [Reset Counter] 0 [Home Position] 0 [MJ-Open Lid] ・・・ [Get 1(0)] [Get](https://slidetodoc.com/presentation_image_h/7bfb9e0d3cffdf69fb08dd52b30a72d6/image-49.jpg)


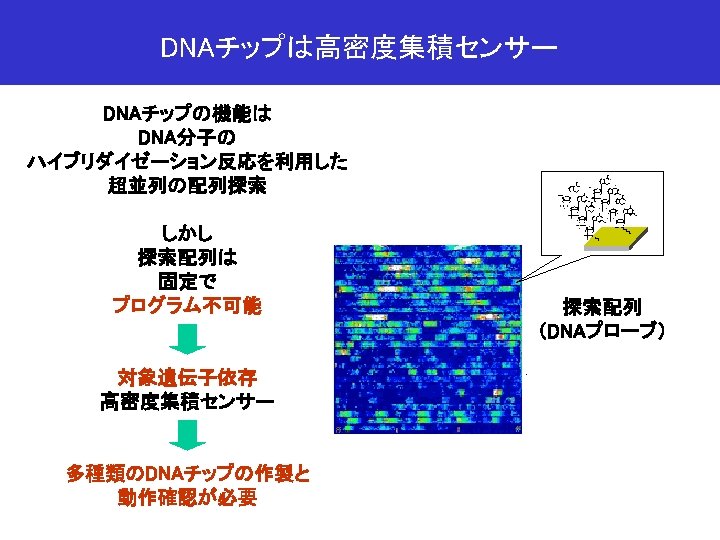
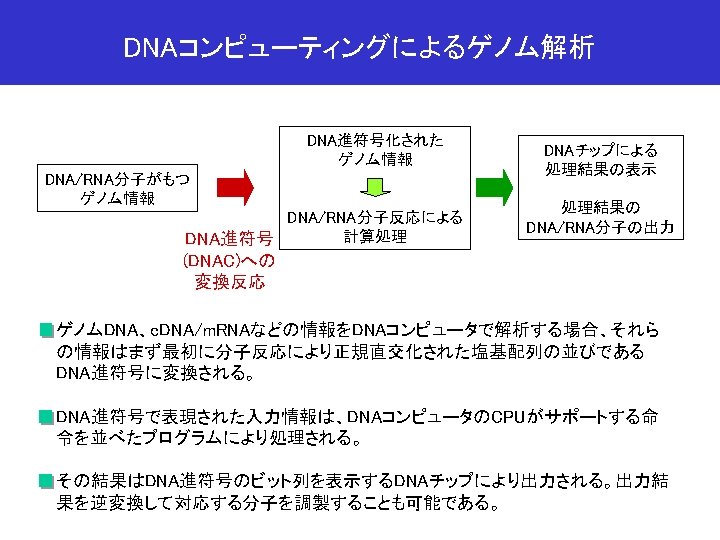








- Slides: 95



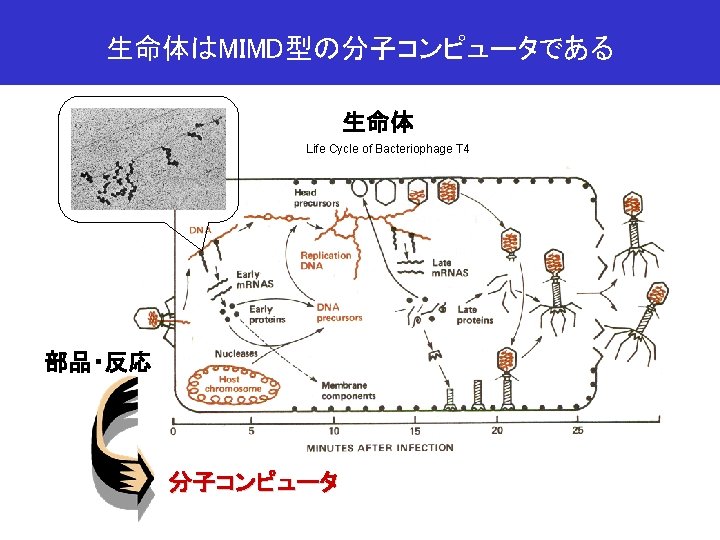
Flynnの計算機アーキテクチャの分類 SISD (single instruction stream over a single data stream) von Neumann sequential computer MISD (multiple instruction streams over a single data stream) systolic arrays, special-purpose parallel computers SIMD (single instruction stream over multiple data streams) vector computers, special-purpose parallel computers MIMD (multiple instruction streams over multiple data streams) general-purpose parallel computers







Leonard M. Adlemanの論文 (Science 266, 1021 -1024, 1994)







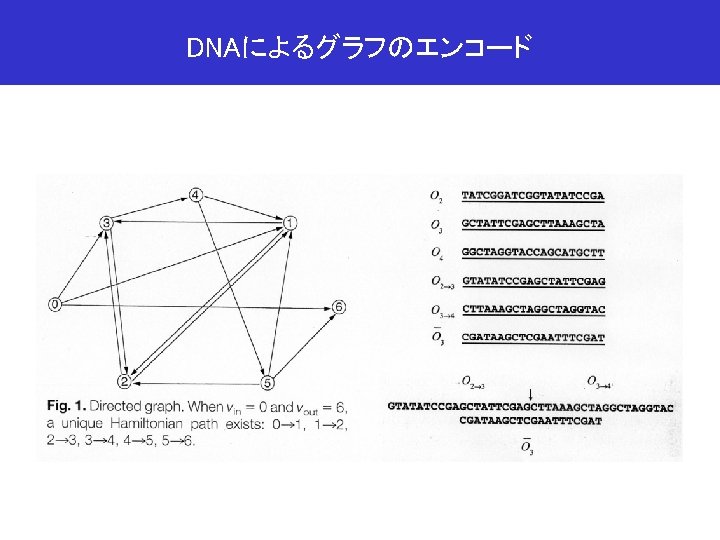
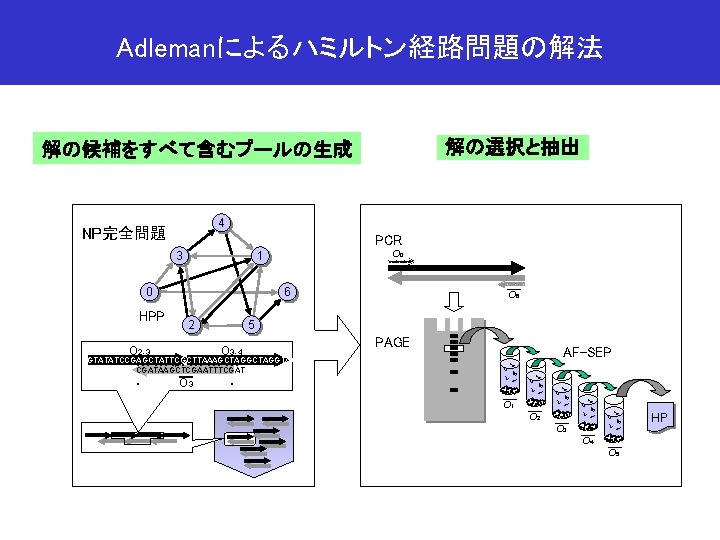
Adlemanによるハミルトン経路問題の解法 解の選択と抽出 解の候補をすべて含むプールの生成 4 NP完全問題 PCR 3 0 HPP O 2 -3 O 0 1 6 2 O 6 5 O 3 -4 PAGE AF-SEP ………………. . O 3 GTATATCCGAGCTATTCGAG CTTAAAGCTAGGTAC CGATAAGCTCGAATTTCGAT O 1 HP O 2 O 3 O 4 O 5


クリーク問題の解法 Q. Quyang et al. , Science 278, 446 -449 (1997).

3 SATの解法 Q. Liu et al. Nature 403, 175 -179 (2000).

騎士問題の解法 D. Faulhammer et al. , Proc. Natl. Acad. Sci. USA 97, 1385 -1389 (2000).

3 SATの解法 K. Sakamoto et al. Science 288, 1223 -1226 (2000).

クリーク問題の解法 D. T. Chiu et al. , Proc. Natl. Acad. Sci. USA 98, 2961 -2966 (2001).

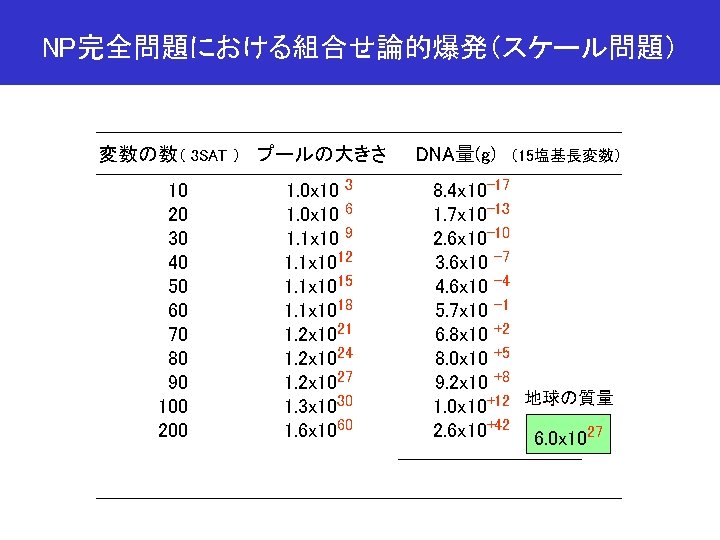
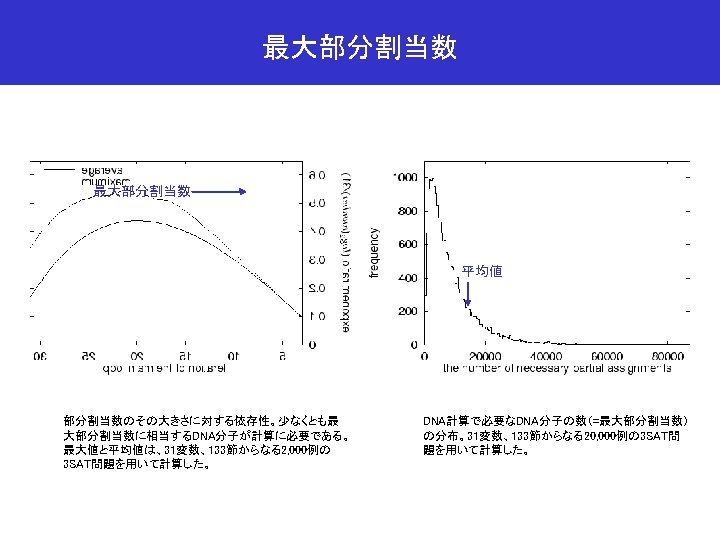
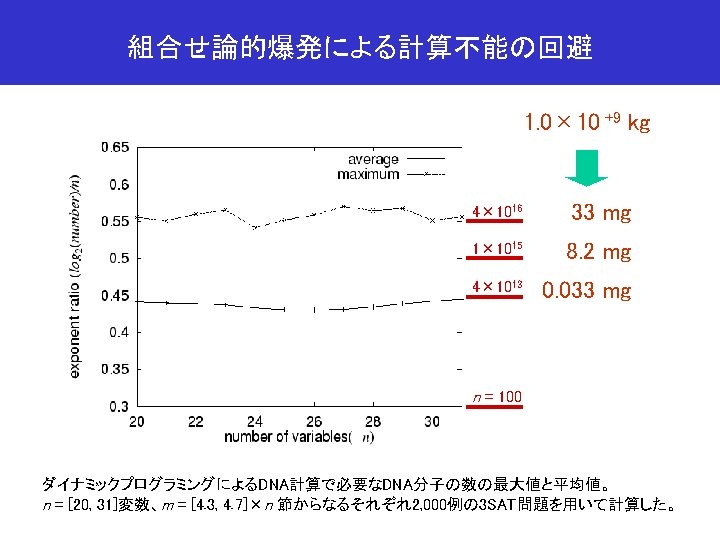
NP完全問題における組合せ論的爆発(スケール問題) 変数の数( 3 SAT ) プールの大きさ 10 20 30 40 50 60 70 80 90 100 200 1. 0 x 10 3 1. 0 x 10 6 1. 1 x 10 9 1. 1 x 1012 1. 1 x 1015 1. 1 x 1018 1. 2 x 1021 1. 2 x 1024 1. 2 x 1027 1. 3 x 1030 1. 6 x 1060 DNA量(g) (15塩基長変数) 8. 4 x 10 -17 1. 7 x 10 -13 2. 6 x 10 -10 3. 6 x 10 -7 4. 6 x 10 -4 5. 7 x 10 -1 6. 8 x 10 +2 8. 0 x 10 +5 9. 2 x 10 +8 1. 0 x 10+12 地球の質量 2. 6 x 10+42 6. 0 x 1027







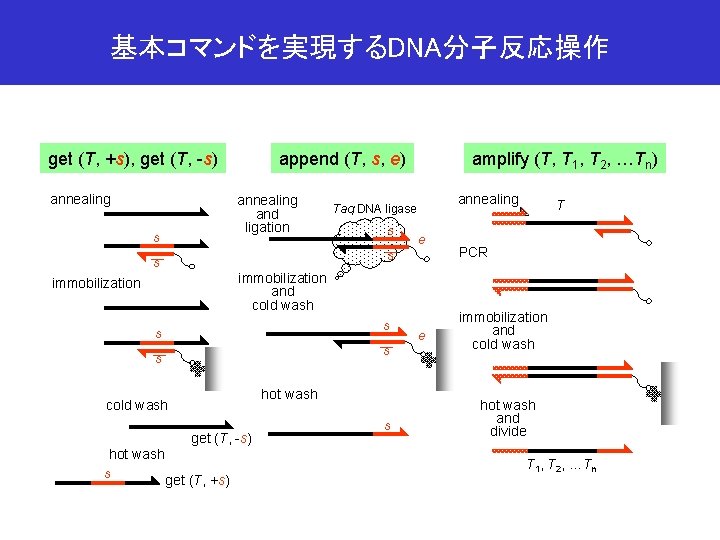
基本コマンドを実現するDNA分子反応操作 append (T, s, e) get (T, +s), get (T, -s) annealing and ligation s annealing Taq DNA ligase s s immobilization and cold wash immobilization s s hot wash cold wash get (T, -s) hot wash s amplify (T, T 1, T 2, …Tn) get (T, +s) s e T PCR immobilization and cold wash hot wash and divide T 1, T 2, …Tn




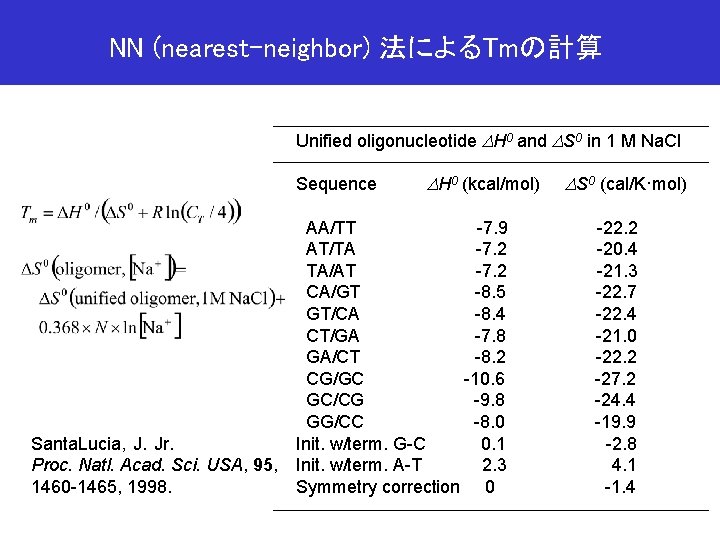
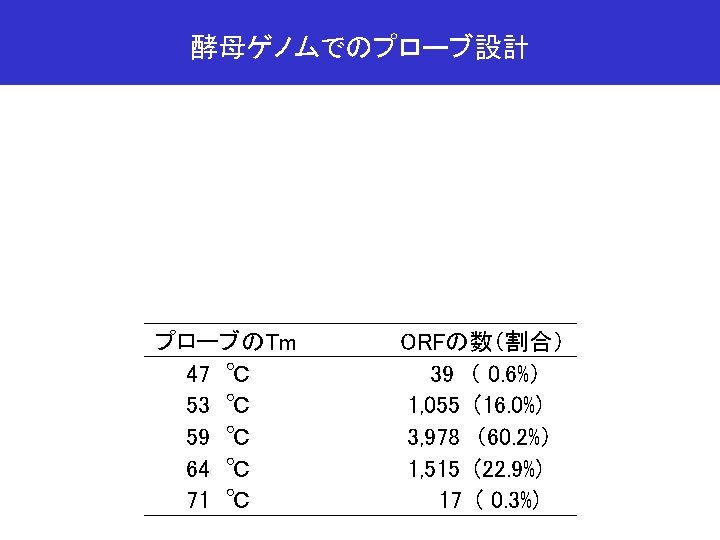
NN (nearest-neighbor) 法によるTmの計算 Unified oligonucleotide DH 0 and DS 0 in 1 M Na. Cl Sequence DH 0 (kcal/mol) DS 0 (cal/K·mol) AA/TT -7. 9 -22. 2 AT/TA -7. 2 -20. 4 TA/AT -7. 2 -21. 3 CA/GT -8. 5 -22. 7 GT/CA -8. 4 -22. 4 CT/GA -7. 8 -21. 0 GA/CT -8. 2 -22. 2 CG/GC -10. 6 -27. 2 GC/CG -9. 8 -24. 4 GG/CC -8. 0 -19. 9 Init. w/term. G-C 0. 1 -2. 8 Santa. Lucia,J.Jr. Proc. Natl. Acad. Sci. USA, 95, Init. w/term. A-T 2. 3 4. 1 Symmetry correction 0 -1. 4 1460 -1465, 1998.

ダイナミックプログラミングによる最安定 2次構造の計算 Zukerのホームページ http: //www. ibc. wustl. edu/~zuker/ Suyama, A. “RNA secondary structure and its relation to biological functions. ” Proc. of Taniguchi Symposium, 162 -172, 1985. (複数の1本鎖核酸分子の複合体の 最安定2次構造計算への拡張)

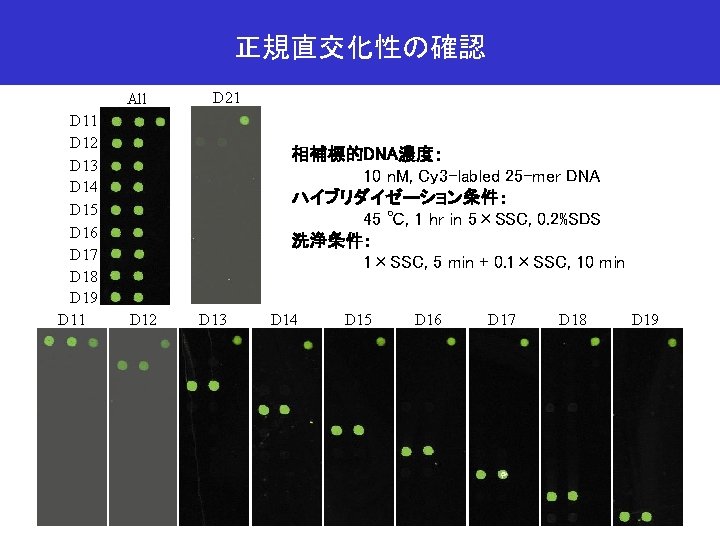
正規直交化性の確認 All D 11 D 12 D 13 D 14 D 15 D 16 D 17 D 18 D 19 D 11 D 21 相補標的DNA濃度: 10 n. M, Cy 3 -labled 25 -mer DNA ハイブリダイゼーション条件: 45 ℃, 1 hr in 5×SSC, 0. 2%SDS 洗浄条件: 1×SSC, 5 min + 0. 1×SSC, 10 min D 12 D 13 D 14 D 15 D 16 D 17 D 18 D 19

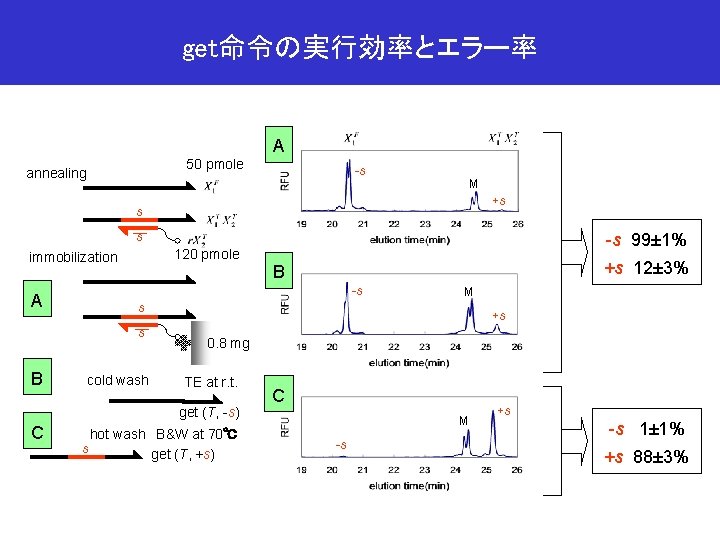
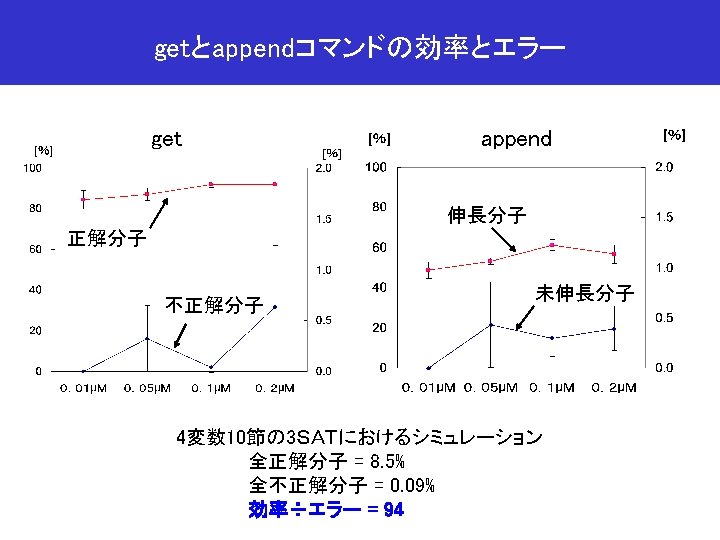
get命令の実行効率とエラー率 A 50 pmole annealing -s M +s s -s 99± 1% s 120 pmole immobilization A -s cold wash +s 0. 8 mg TE at r. t. get (T, -s) C M s s B +s 12± 3% B hot wash B&W at 70℃ s get (T, +s) C M -s +s -s 1± 1% +s 88± 3%

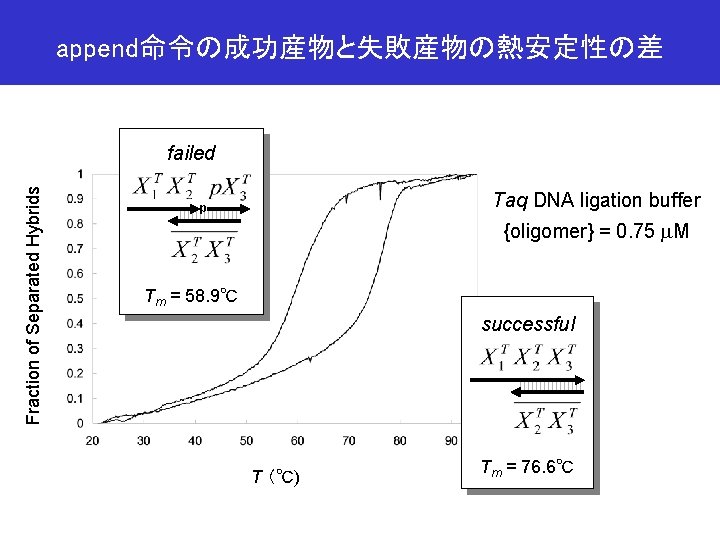
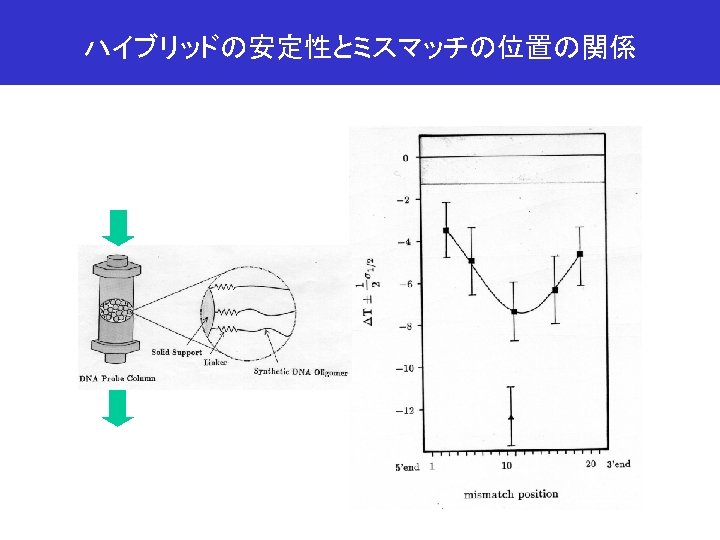
append命令の成功産物と失敗産物の熱安定性の差 Fraction of Separated Hybrids failed Taq DNA ligation buffer p {oligomer} = 0. 75 m. M Tm = 58. 9℃ successful T (℃) Tm = 76. 6℃

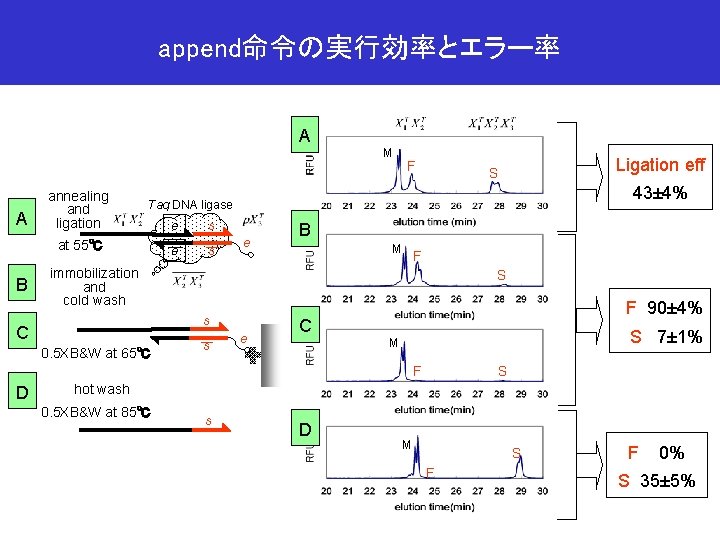
append命令の実行効率とエラー率 A A annealing and ligation F 43± 4% e e s s e B M F immobilization and cold wash S s C 0. 5 XB&W at 65℃ s e C F 90± 4% S 7± 1% M F D Ligation eff S Taq DNA ligase at 55℃ B M S hot wash 0. 5 XB&W at 85℃ s D M S F F 0% S 35± 5%



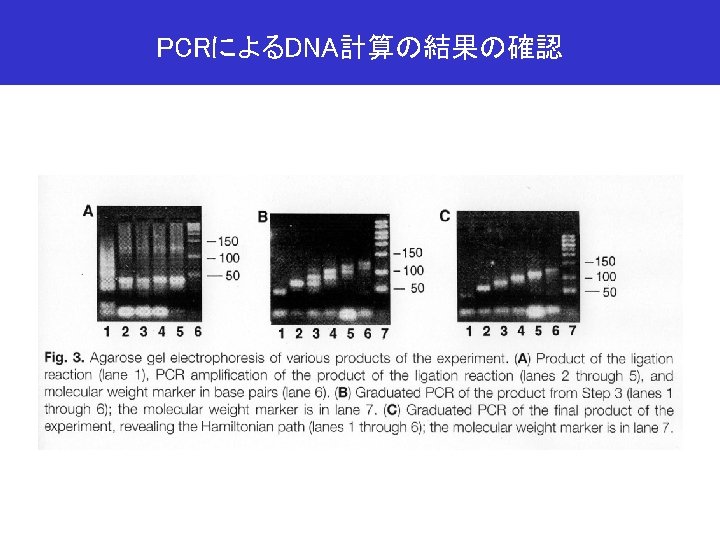
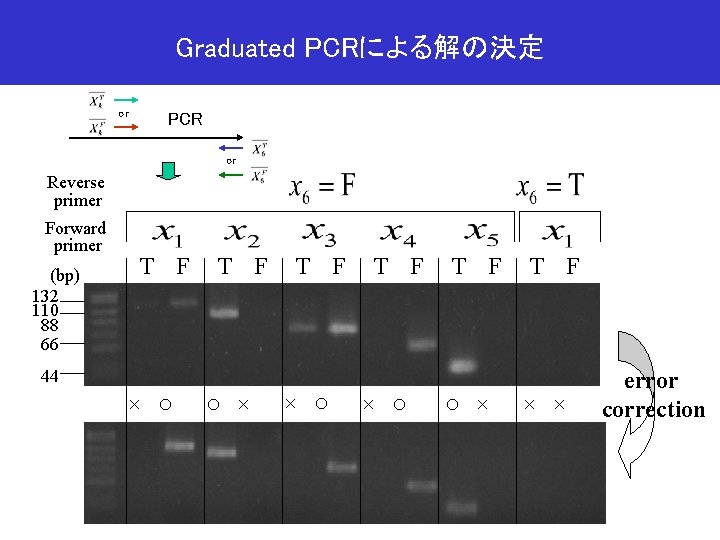
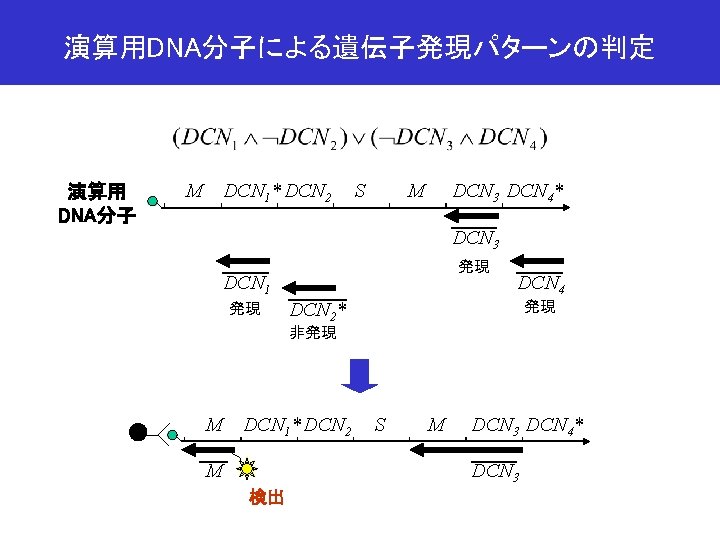
Graduated PCRによる解の決定 or PCR or Reverse primer Forward primer (bp) 132 110 88 66 T F T F 44 × ○ ○ × × × error correction



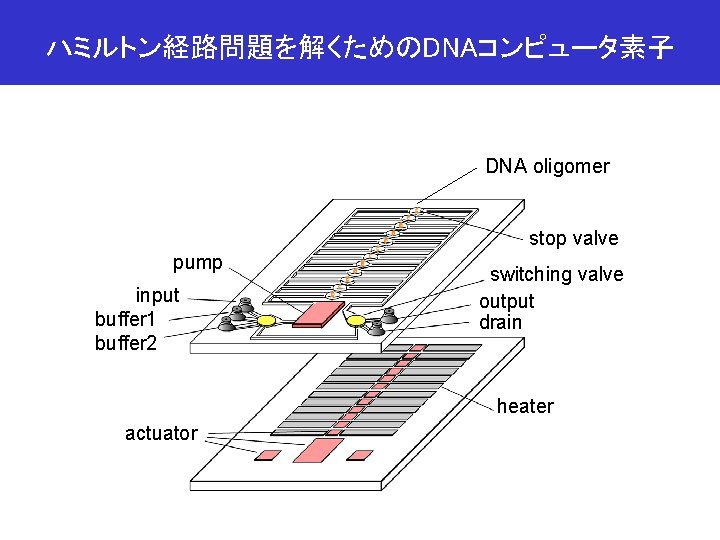
ハミルトン経路問題を解くためのDNAコンピュータ素子 DNA oligomer stop valve pump input buffer 1 buffer 2 switching valve output drain heater actuator

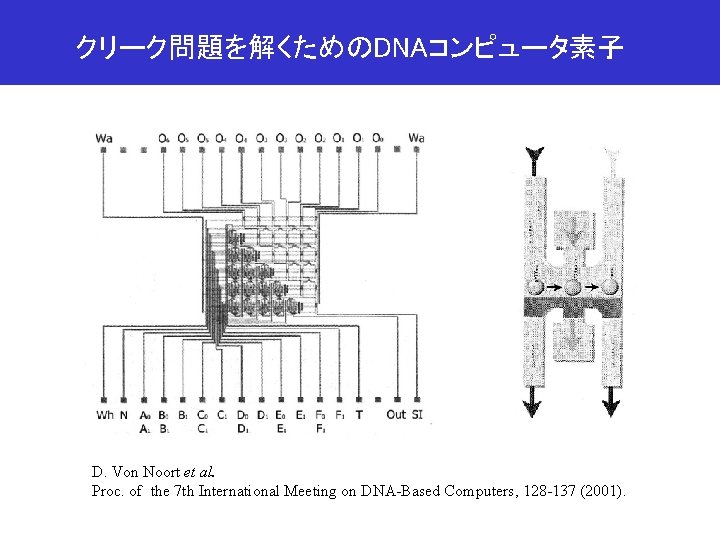
クリーク問題を解くためのDNAコンピュータ素子 D. Von Noort et al. Proc. of the 7 th International Meeting on DNA-Based Computers, 128 -137 (2001).

![ハイブリッドDNAコンピュータによるプログラムの実行 Instrument Reset Counter 0 Home Position 0 MJOpen Lid Get 10 Get ハイブリッドDNAコンピュータによるプログラムの実行 [Instrument] [Reset Counter] 0 [Home Position] 0 [MJ-Open Lid] ・・・ [Get 1(0)] [Get](https://slidetodoc.com/presentation_image_h/7bfb9e0d3cffdf69fb08dd52b30a72d6/image-49.jpg)
ハイブリッドDNAコンピュータによるプログラムの実行 [Instrument] [Reset Counter] 0 [Home Position] 0 [MJ-Open Lid] ・・・ [Get 1(0)] [Get 2(1)] [Append(2)] ・・・ [Exit] protocol-level (1 -1 -4) [MJ-Open Lid] Do 2 _SEND "LID OPEN" Do 10 _SEND "LID? " Wait_msec 500 _CMP_GSTR "OPEN" IF_Goto EQ 0 ; open Wait_msec 1000 Loop ; Time out End ; open script-level Pascal/C-level


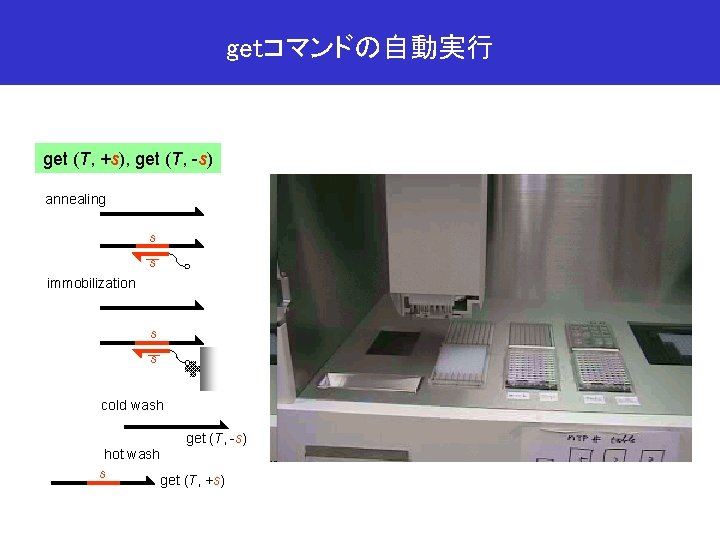
getコマンドの自動実行 get (T, +s), get (T, -s) annealing s s immobilization s s cold wash get (T, -s) hot wash s get (T, +s)



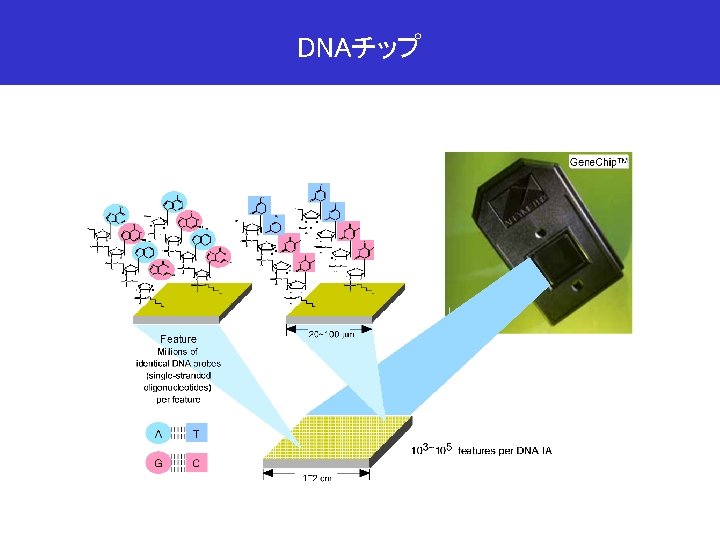
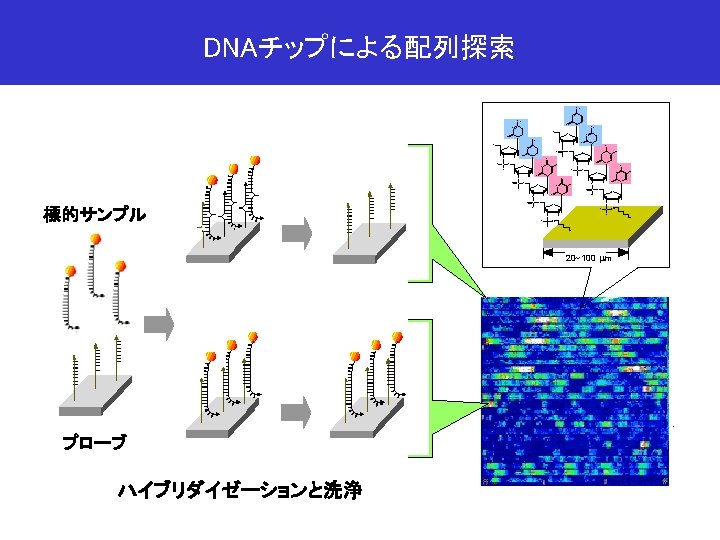
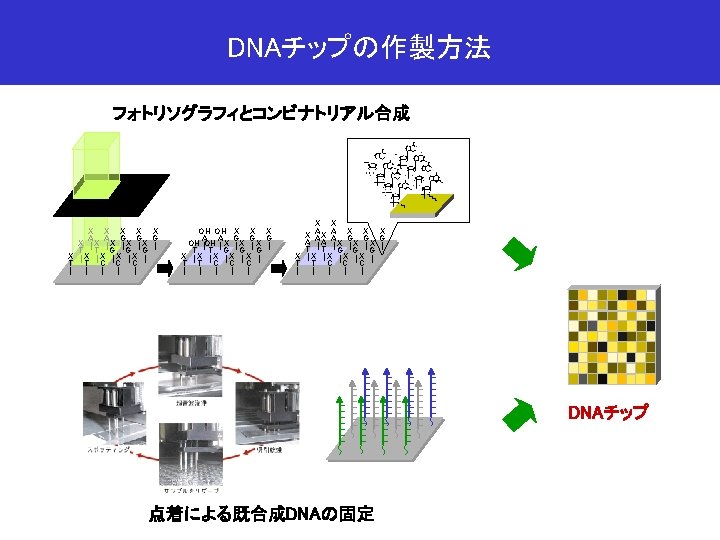
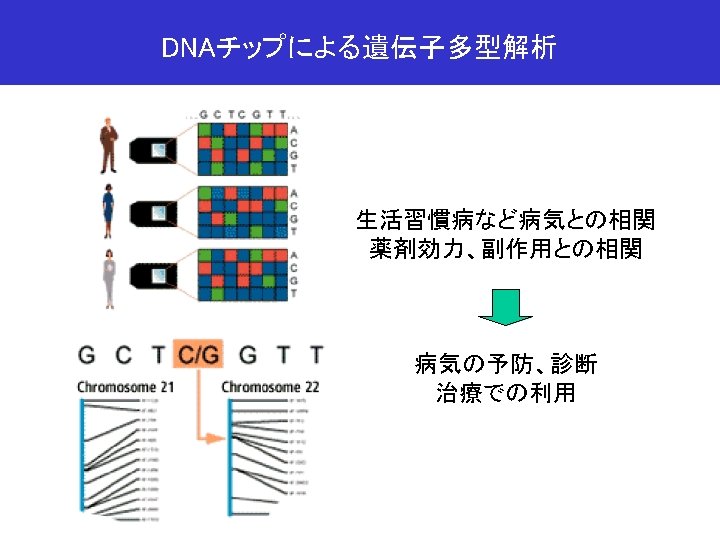
DNAチップによる配列探索 NH 2 N NH 2 O N NH 2 N HO O O N N H NH 2 H H HO H O O O N H H H O O N P O O O- H H HN H O P O H H O O O- O N H O HN H O P O O H H O O P O N O- H HN H H O H O O O- H O N HN H H O P O O H H O N O- H H H O 標的サンプル P O H H H O O P H O O O- H P O- O- O- 20~100 mm プローブ ハイブリダイゼーションと洗浄 H O-



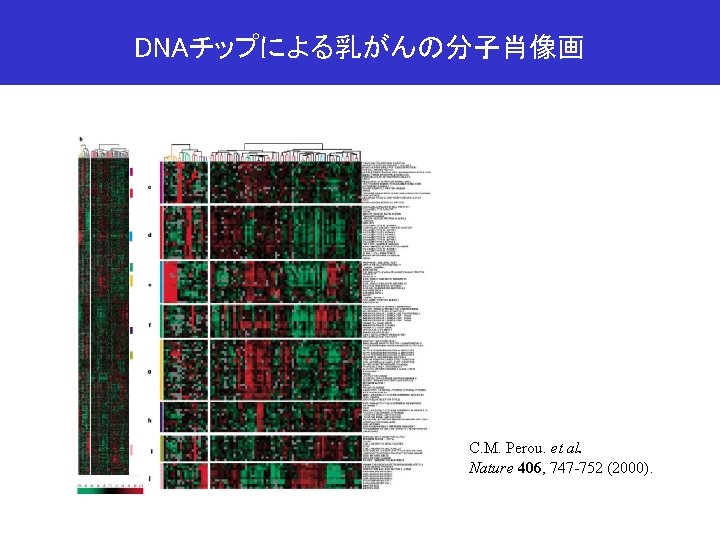
DNAチップによる乳がんの分子肖像画 C. M. Perou. et al. Nature 406, 747 -752 (2000).





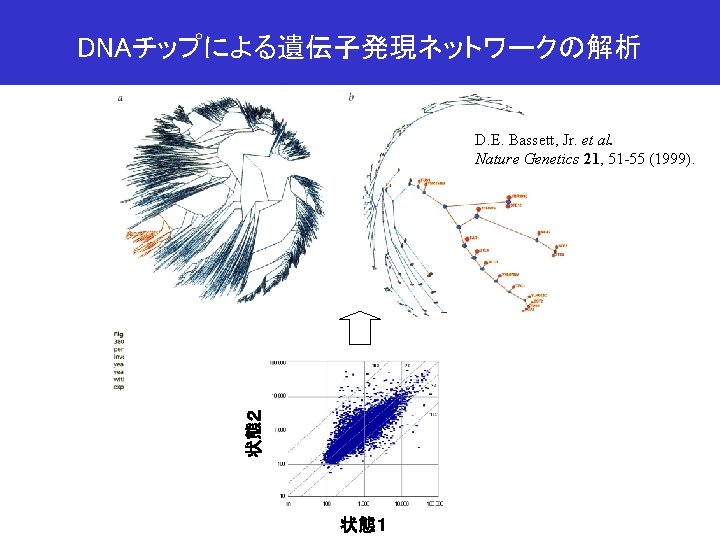
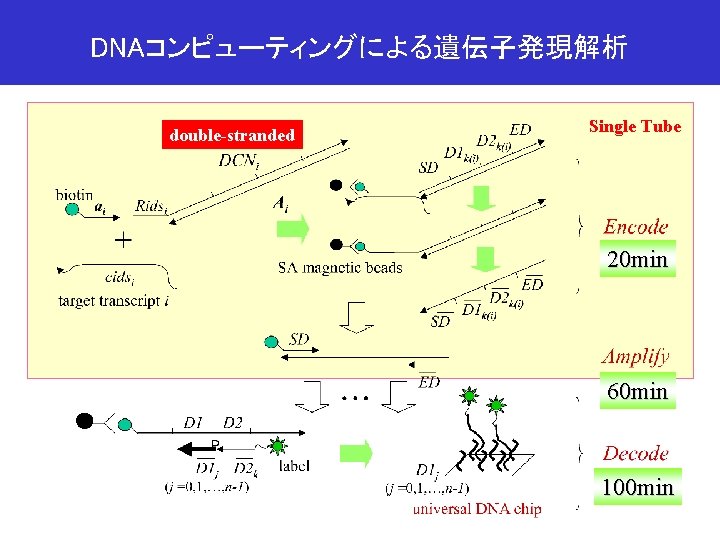
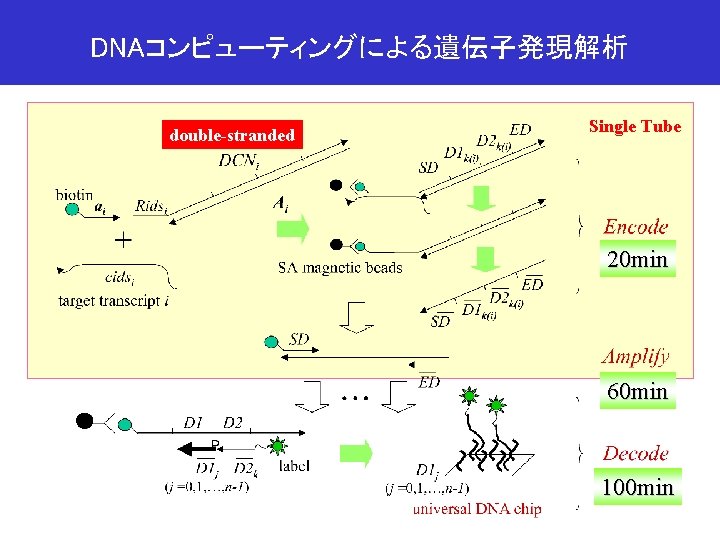
DNAコンピューティングによる遺伝子発現解析 double-stranded Single Tube 20 min 60 min 100 min




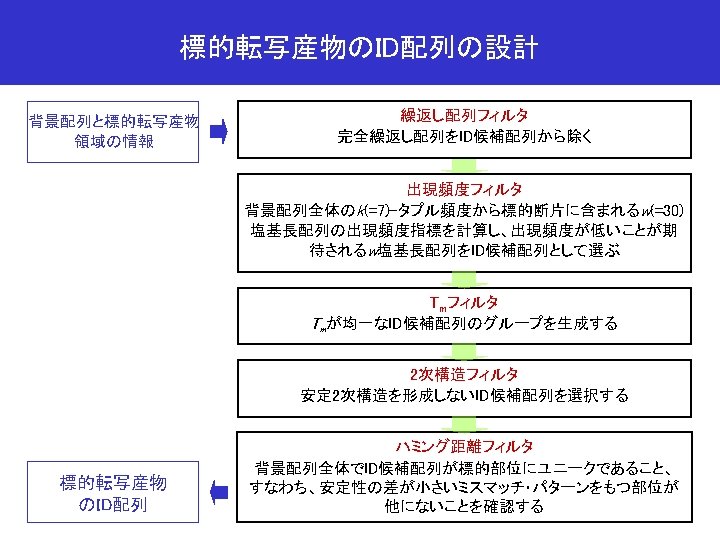
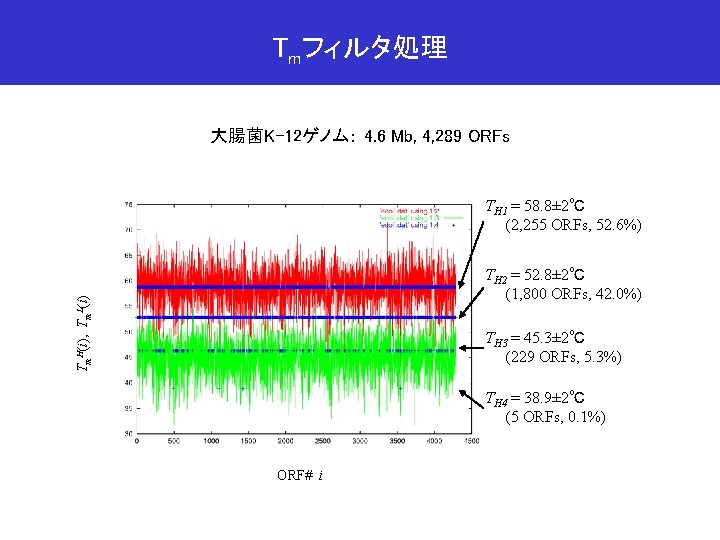
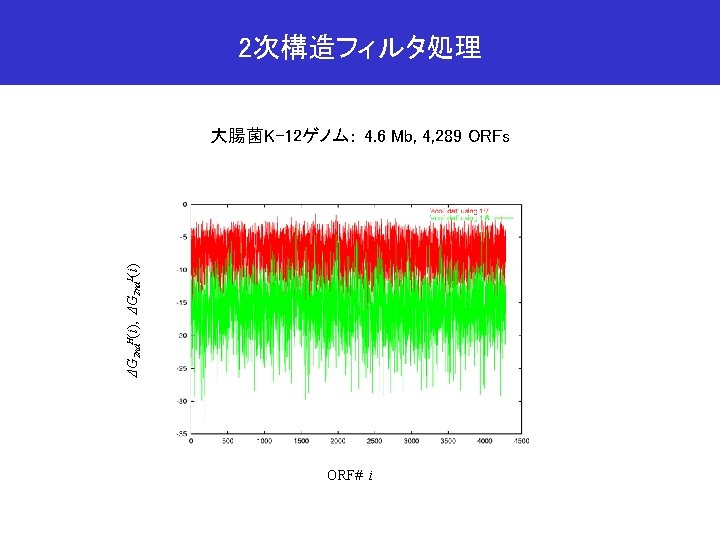
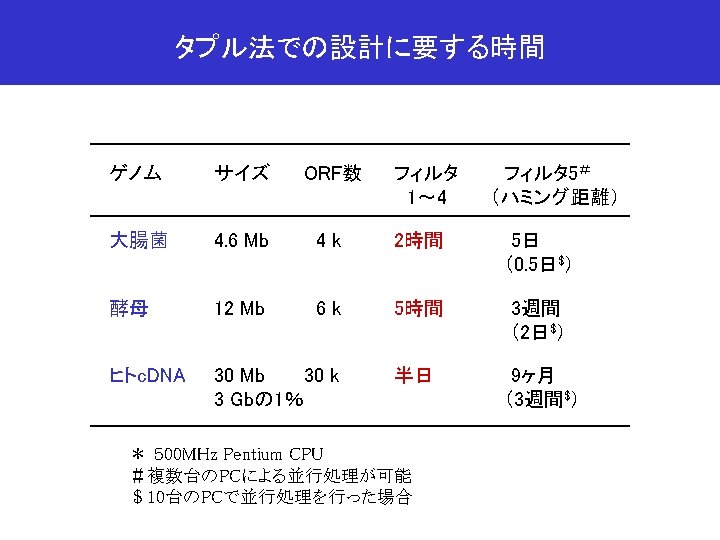
Tmフィルタ処理 大腸菌K-12ゲノム: 4. 6 Mb, 4, 289 ORFs TH 1 = 58. 8± 2℃ (2, 255 ORFs, 52. 6%) Tm. H(i), Tm. L(i) TH 2 = 52. 8± 2℃ (1, 800 ORFs, 42. 0%) TH 3 = 45. 3± 2℃ (229 ORFs, 5. 3%) TH 4 = 38. 9± 2℃ (5 ORFs, 0. 1%) ORF# i


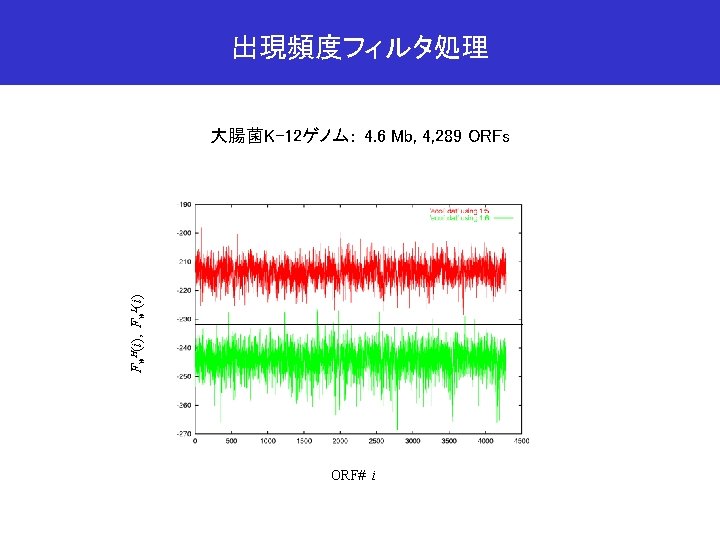
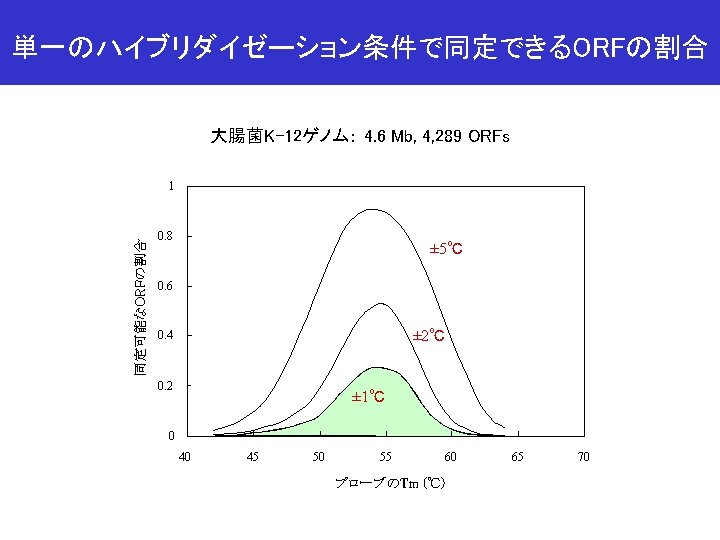
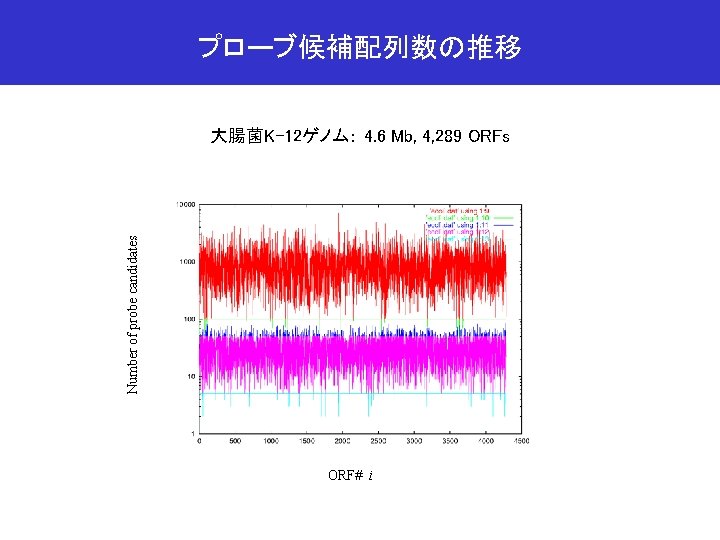
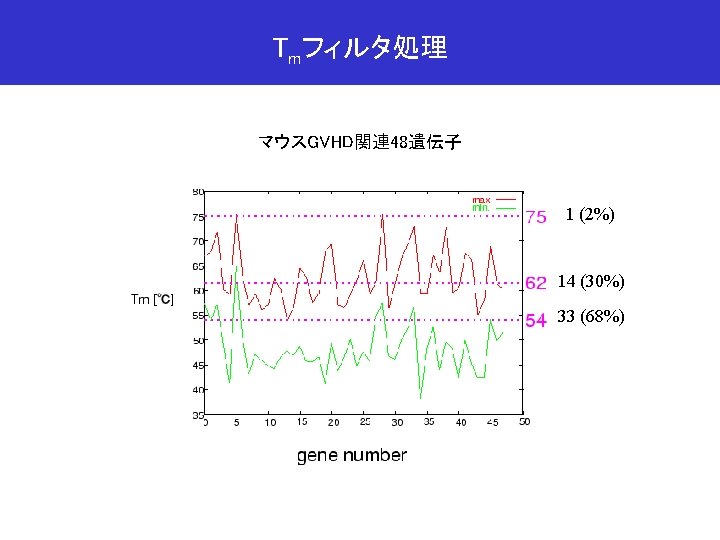
プローブ候補配列数の推移 Number of probe candidates 大腸菌K-12ゲノム: 4. 6 Mb, 4, 289 ORFs ORF# i









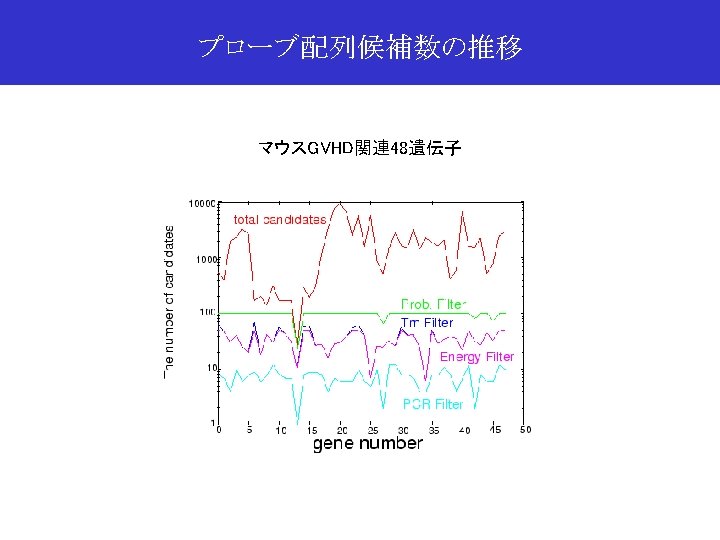
プローブの特異性(Mouse Genomic DNAのPCR) template: mouse BALB/c genomic DNA ~amol primer: 0. 2 mmol each PCR cycle: 95 -65 -72 ºC 40 cycles

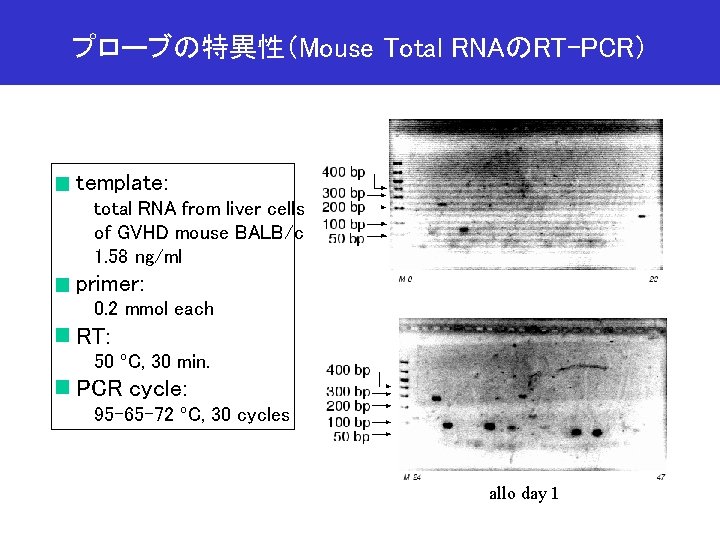
プローブの特異性(Mouse Total RNAのRT-PCR) template: total RNA from liver cells of GVHD mouse BALB/c 1. 58 ng/ml primer: 0. 2 mmol each RT: 50 ºC, 30 min. PCR cycle: 95 -65 -72 ºC, 30 cycles allo day 1

DNAコンピューティングによる遺伝子発現解析 double-stranded Single Tube 20 min 60 min 100 min

標的合成DNA分子の特異的検出 no target all targets Input: 300 p. M gene specific sequences of 30 -mer Hybridization & Washing: Cy 3 : 100 p. M each of D 1 -complements Cy 5 : output hybridization : 5×SSC, 0. 2%SDS, 45 °C, 1 hr washing : 1×SSC, 5 min; 0. 1×SSC, 10 min lower Tm group IGTP TGTP/Mg 21 Nedd 5 PRCC vitronectin Mn-SOD activin beta C

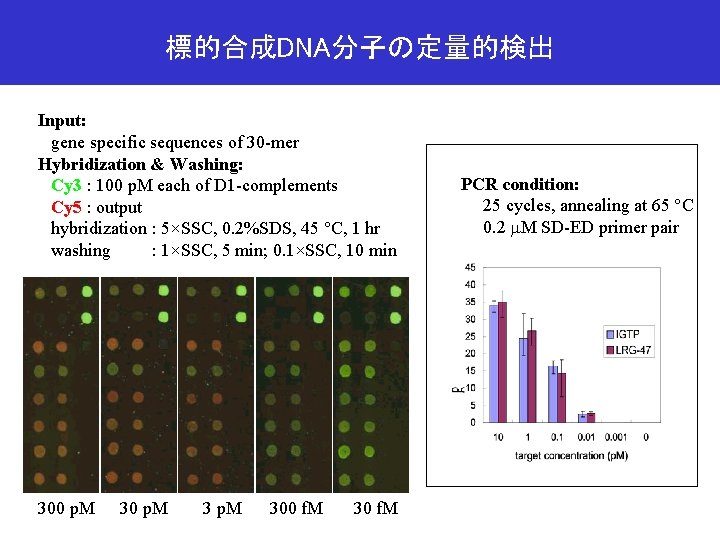
標的合成DNA分子の定量的検出 Input: gene specific sequences of 30 -mer Hybridization & Washing: Cy 3 : 100 p. M each of D 1 -complements Cy 5 : output hybridization : 5×SSC, 0. 2%SDS, 45 °C, 1 hr washing : 1×SSC, 5 min; 0. 1×SSC, 10 min 300 p. M 300 f. M 30 f. M PCR condition: 25 cycles, annealing at 65 °C 0. 2 m. M SD-ED primer pair

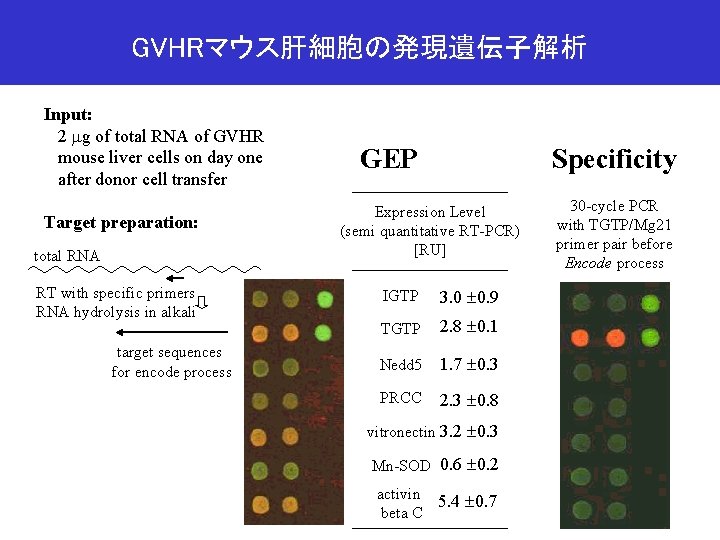
GVHRマウス肝細胞の発現遺伝子解析 Input: 2 mg of total RNA of GVHR mouse liver cells on day one after donor cell transfer Target preparation: total RNA RT with specific primers RNA hydrolysis in alkali target sequences for encode process GEP Specificity Expression Level (semi quantitative RT-PCR) [RU] IGTP 3. 0 0. 9 TGTP 2. 8 0. 1 Nedd 5 1. 7 0. 3 PRCC 2. 3 0. 8 vitronectin 3. 2 0. 3 Mn-SOD 0. 6 0. 2 activin 5. 4 0. 7 beta C 30 -cycle PCR with TGTP/Mg 21 primer pair before Encode process

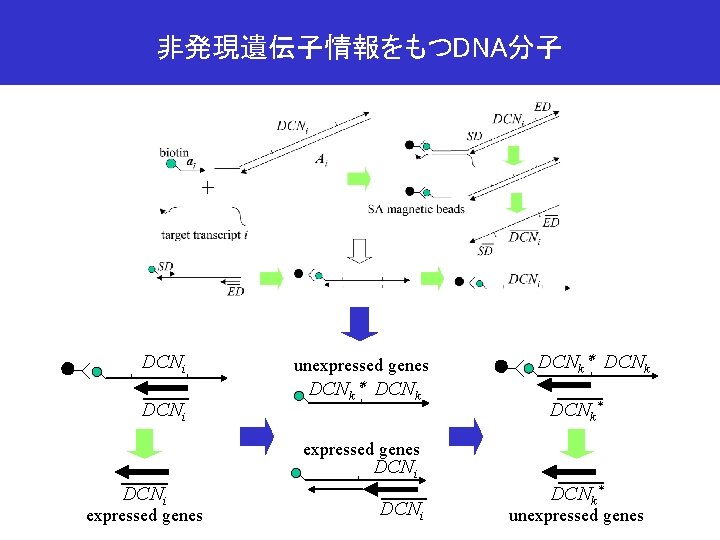
非発現遺伝子情報をもつDNA分子 DCNi unexpressed genes DCNk* DCNk* expressed genes DCNi DCNk* unexpressed genes


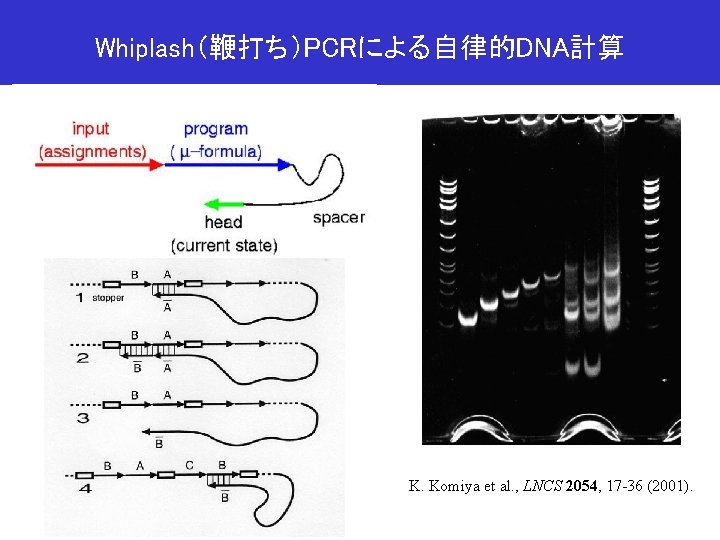
Whiplash(鞭打ち)PCRによる自律的DNA計算 K. Komiya et al. , LNCS 2054, 17 -36 (2001).






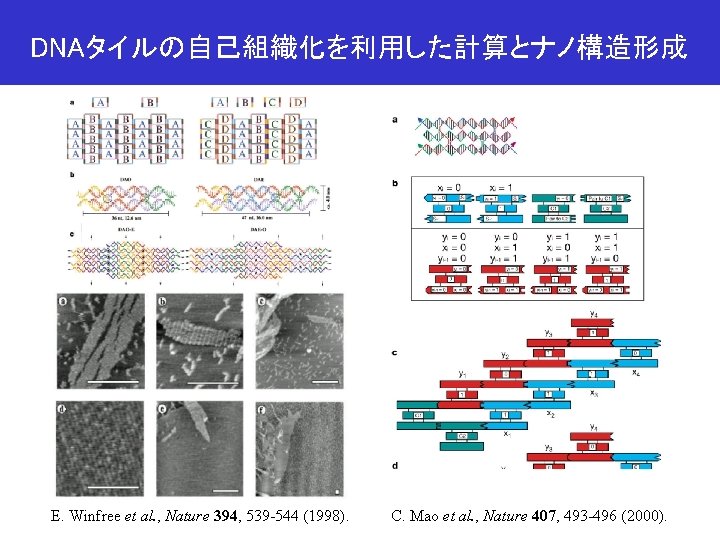
DNAタイルの自己組織化を利用した計算とナノ構造形成 E. Winfree et al. , Nature 394, 539 -544 (1998). C. Mao et al. , Nature 407, 493 -496 (2000).

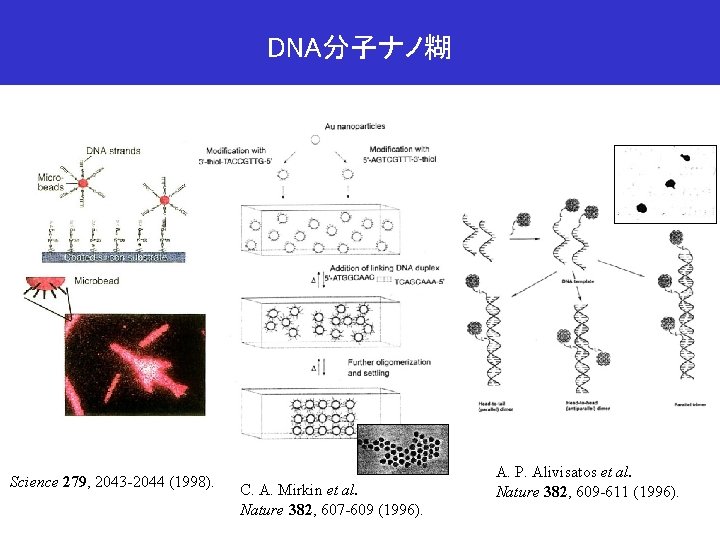
DNA分子ナノ糊 Science 279, 2043 -2044 (1998). C. A. Mirkin et al. Nature 382, 607 -609 (1996). A. P. Alivisatos et al. Nature 382, 609 -611 (1996).

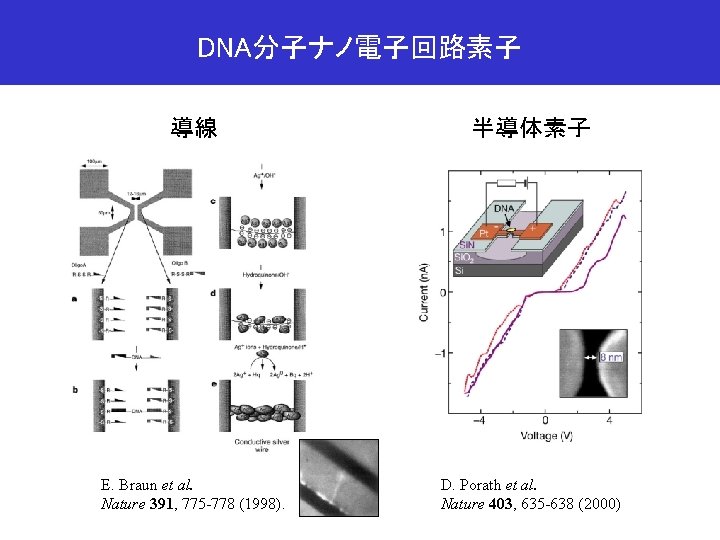
DNA分子ナノ電子回路素子 導線 半導体素子 E. Braun et al. Nature 391, 775 -778 (1998). D. Porath et al. Nature 403, 635 -638 (2000)

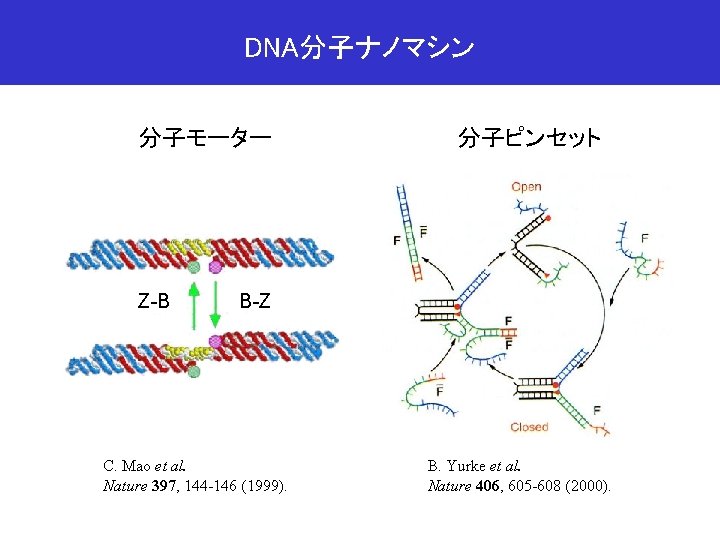
DNA分子ナノマシン 分子モーター Z-B 分子ピンセット B-Z C. Mao et al. Nature 397, 144 -146 (1999). B. Yurke et al. Nature 406, 605 -608 (2000).
