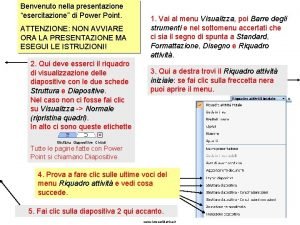
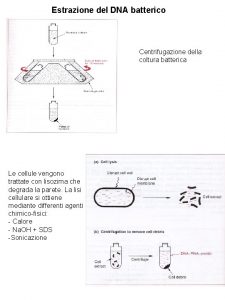
ESEMPIO DI ESERCITAZIONE ESTRAZIONE DEL DNA col metodo





















































- Slides: 64

ESEMPIO DI ESERCITAZIONE: ESTRAZIONE DEL DNA col metodo organico (PCI)






QUANTIFICAZIONE DEL DNA ESTRATTO COL METODO DELLO SPETTROFOTOMETRO











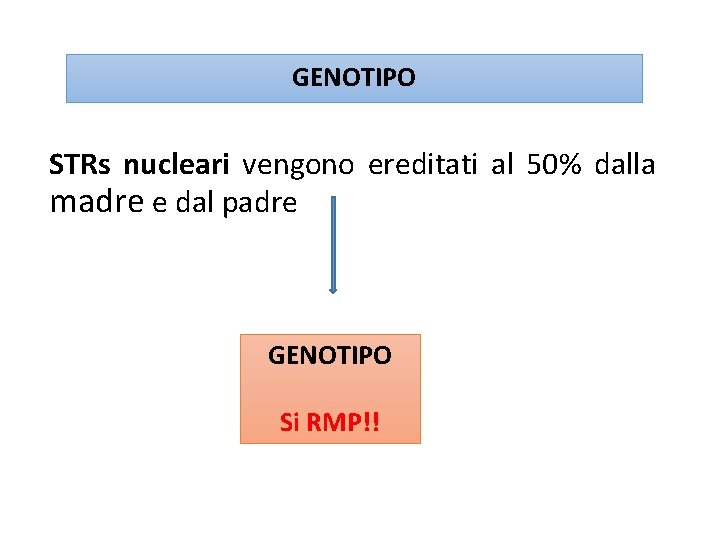
I marcatori più utilizzati in genetica forense sono i loci STRs (codominanti)… …. sottoposti Mendeliana alle regole dell’eredità Localizzati su Cromosomi AUTOSOMICI

GENOTIPO STRs nucleari vengono ereditati al 50% dalla madre e dal padre GENOTIPO Si RMP!!

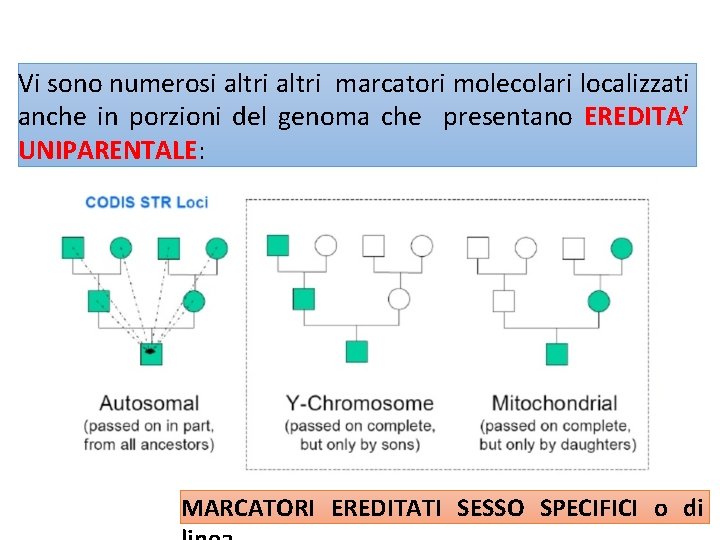
Vi sono numerosi altri marcatori molecolari localizzati anche in porzioni del genoma che presentano EREDITA’ UNIPARENTALE: MARCATORI EREDITATI SESSO SPECIFICI o di


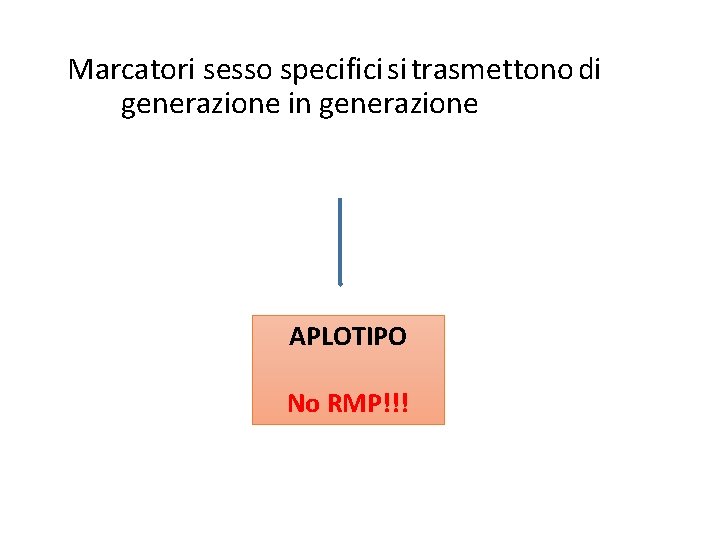
Marcatori sesso specifici si trasmettono di generazione in generazione APLOTIPO No RMP!!!

Aplotipo Combinazione di diversi stati allelici di un set di marcatori polimorfici che si trovano fisicamente associati sulla stessa molecola di DNA (es. cromosoma, regione cromosomica) Tale combinazione sono in linkage disequilibrium (LD) Gli alleli dell'Y sono sempre associati a formare aplotipi, così come gli alleli del mt. DNA

Lineage markers



DNA mitocondriale (mt. DNA) mt-EVE Eva mitocondriale (mt. EVE) è una donna dal cui mt. DNA è scaturito il mt. DNA di tutti gli esseri umani viventi. L’mt. EVE è, per così dire, la PROGENITRICE di tutti gli esseri umani in pura linea materna Visse in Africa, circa 175. 000 annni fa

Nel corso delle generazioni, dalle progenitrici sopraggiunsero sempre più mutazioni in modo tale che il suo profilo mitocondriale subì dei cambiamenti. ebbero vita gli APLOGRUPPI Con il passare del tempo l’albero genealogico diventa sempre più imponente e complesso. Si aggiungono sempre nuovi SNP nuovi sottogruppi.

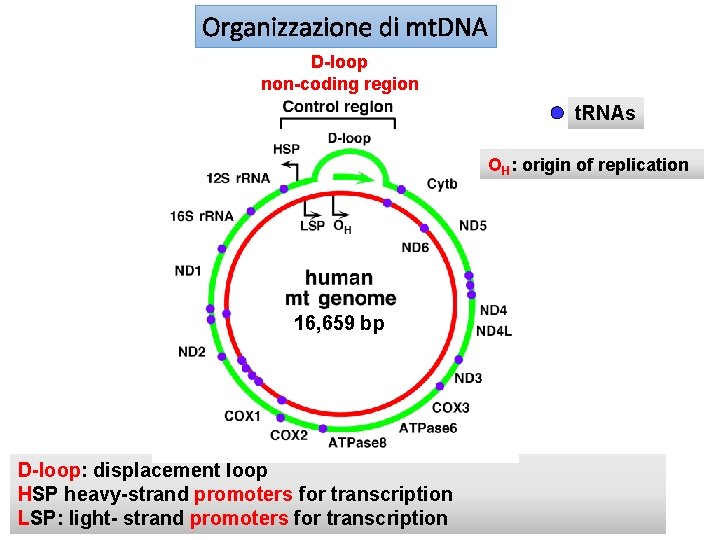
Organizzazione di mt. DNA D-loop non-coding region t. RNAs OH: origin of replication 16, 659 bp D-loop: displacement loop HSP heavy-strand promoters for transcription LSP: light- strand promoters for transcription

Mitocondri (cellule animali e vegetali): bastoncellari (O. 5 - 1 µm di spessore), fino a 10 µm di lunghezza • mt. DNA è nei mitocondri ELEVATO NUMERO DI COPIE (1000 -10000/cellula) • E’ un genoma APLOIDE, SENZA RICOMBINAZIONE • Tasso di mutazione: 6 -7 volte > g. DNA nucleare


EREDITA’ mitocondriale (mt. DNA)

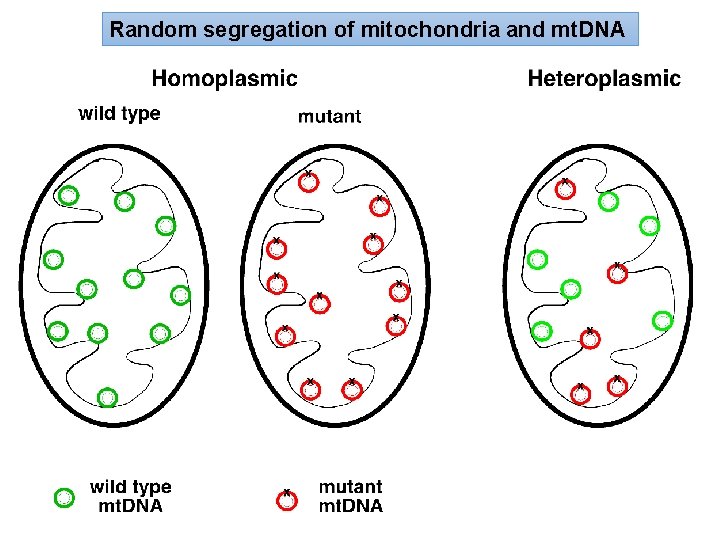
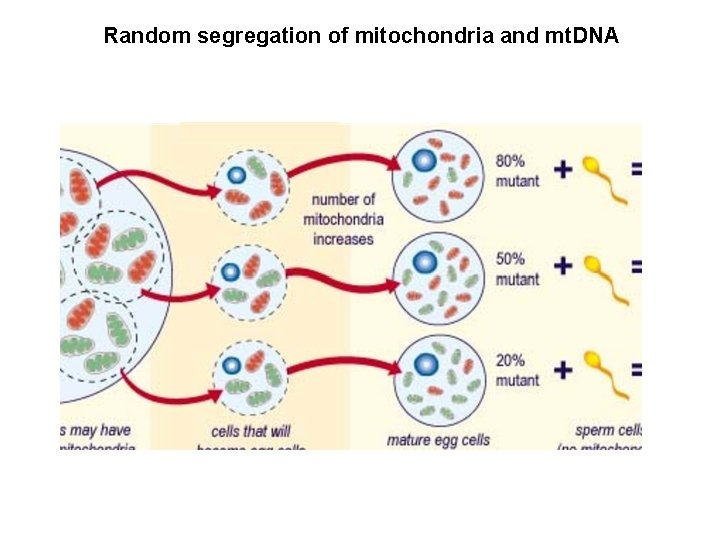
TANTI MITOCONDRI PER CELLULA: ci possono essere mitocondri con sequenza di basi differenti in una medesima posizione (eteroplasmia). Il primo caso di eteroplasmia osservata riguarda l'analisi dei resti dei Romanov (Gill et al. , 1994). I discendenti: possono ereditare una proporzione variabile delle 2 popolazioni e nel corso del tempo una popolazione può fissarsi a scapito di un'altra, ripristinando una condizione di omoplasmia.

Random segregation of mitochondria and mt. DNA

EREDITA’ MITOCONDRIALE vs GENETCA MENDELIANA

Random segregation of mitochondria and mt. DNA

IL DNA MITOCONDRIALE NELLA PRATICA FORENSE

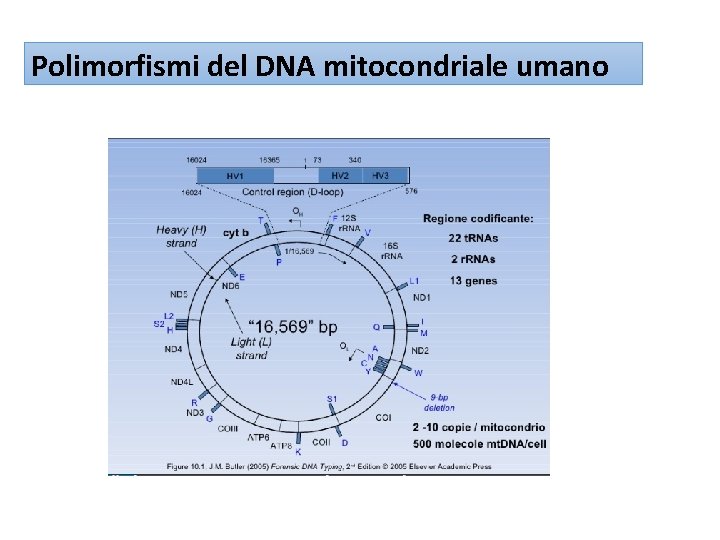
Polimorfismi del DNA mitocondriale umano

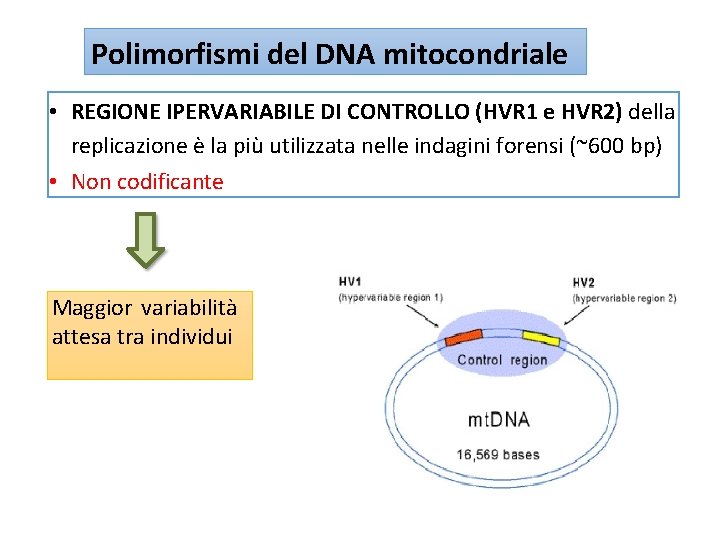
Polimorfismi del DNA mitocondriale • REGIONE IPERVARIABILE DI CONTROLLO (HVR 1 e HVR 2) della replicazione è la più utilizzata nelle indagini forensi (~600 bp) • Non codificante Maggior variabilità attesa tra individui

Polimorfismi del DNA mitocondriale Il DNA mitocondriale è meno potente per molti aspetti rispetto marcatori autosomici MA estremamente utile in genetica forense PERCHE’?


Polimorfismi del DNA mitocondriale Caratteristiche chiave per la genetica forense: Elevato numero di copie Ø Analisi di reperti con materiale cellulare ridotto o degradato Reperti antichi e resti scheletrici; Materiale biologico mal conservato; Esposizione ad agenti chimici e fisici Eredità matrilineare Ø Ricostruzione della linea parentale materna Individui imparentati presentano lo stesso aplotipo mitocondriale Non è legato al nucleo Ø Analisi di cellule ANUCLEATE (capelli telogenici) Es. dai capelli senza radice è possibile analizzare l’mt. DNA, per investigazioni scientifiche in ambito penale.


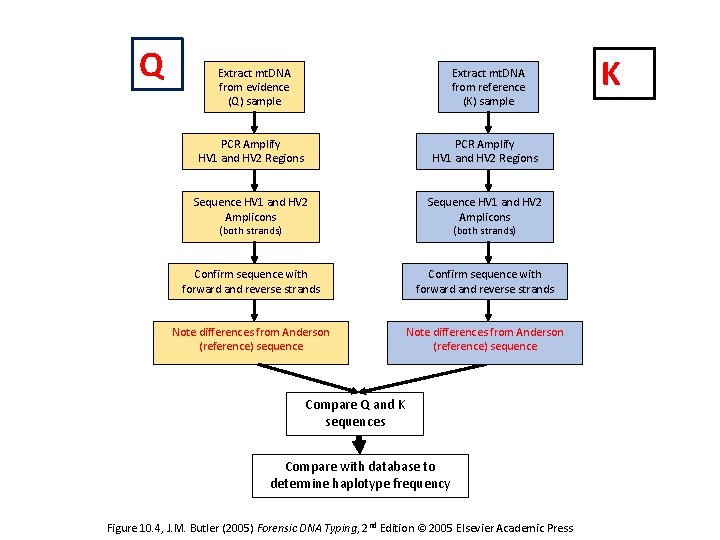
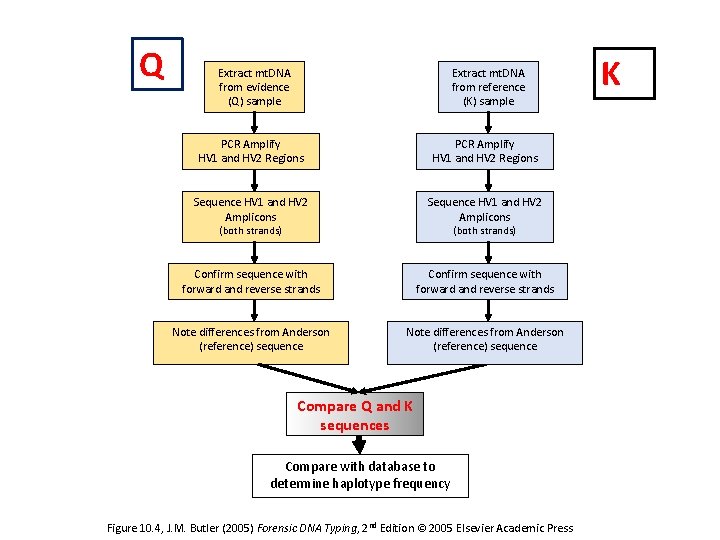
PROCEDURA d’estrazione di mt. DNA e analisi con PCR 1. Pulizia e decontaminazione dell’ambiente 2. Decontaminazione del reperto (no DNA esogeno) 3. Analisi microscopica del reperto (formazioni pilifera) ed eventuale confronto con il campione di riferimento (es. ossa, denti) 4. Estrazione dei mt. DNA (reperto e campione) e PCR 5. Purificazione e quantificazione 6. Sequenziamento automatico (Sanger): HVI, HVII (regioni controllo), SNP, STR, 7. analisi dei dati 8. Confronto col database





Q Extract mt. DNA from evidence (Q) sample Extract mt. DNA from reference (K) sample PCR Amplify HV 1 and HV 2 Regions Sequence HV 1 and HV 2 Amplicons Confirm sequence with forward and reverse strands Note differences from Anderson (reference) sequence (both strands) Compare Q and K sequences Compare with database to determine haplotype frequency Figure 10. 4, J. M. Butler (2005) Forensic DNA Typing, 2 nd Edition © 2005 Elsevier Academic Press K

CRS (Cambridge Reference Sequence) (Anderson et al, 1981) Le sequenze di mt. DNA umane sono descritte utilizzando come riferimento la prima sequenza completa di mitocondrio (CRS o sequenza di Anderson) 52

Ogni sequenza può essere archiviata annotando la posizione dei residui differenti rispetto alla CRS e il tipo di differenza. (Es. una differenza da C->T in posizione 429 può essere annotata come: 429 T).

Revised CRS (r. CRS) (Andrews et al, 1999)

Q Extract mt. DNA from evidence (Q) sample Extract mt. DNA from reference (K) sample PCR Amplify HV 1 and HV 2 Regions Sequence HV 1 and HV 2 Amplicons Confirm sequence with forward and reverse strands Note differences from Anderson (reference) sequence (both strands) Compare Q and K sequences Compare with database to determine haplotype frequency Figure 10. 4, J. M. Butler (2005) Forensic DNA Typing, 2 nd Edition © 2005 Elsevier Academic Press K

Valutazione statistica dei confronti (Q vs K) ESCLUSIONE: I 2 campioni differiscono nella sequenze di mt. DNA per più posizioni nucleotidiche si può escludere che derivino dalla stessa sorgente INCONCLUSIVO: se differiscono per 1 sola posizione nucleotidica, il dato è inconclusivo, mutazione? ? ? Conviene analizzare altri tessuti NON ESCLUSIONE: se sono identici, se 1 dei due presenta eteroplasmia in una posizione nucleotidica non si esclude che derivino dalla stessa sorgente. Tuttavia se entrambi i campioni presentano eteroplasmia nella stessa posizione, ciò rafforza che derivino dalla stessa sorgente.

Valutazione statistica dei confronti (Q vs K) E’ invece possibile stabilire la probabilità che due sequenze uguali indichino un effettivo rapporto di parentela, piuttosto che un match casuale.

Metodo usato da Gill et al. (Nature Genetics, 1994 6, 130 -135): Il grado di certezza (per l’assegnazione di una sequenza a una famiglia) si esprime come rapporto tra le probabilità di 2 ipotesi alternative: P(E)= probabilità che il campione appartenga alla famiglia rispetto alle probabilità P(E’) che il campione non appartenga alla famiglia. LR (likelihood ratio)= P(E)/P(E’)

La probabilità che due sequenze siano identiche per caso: P(E’)=F F= frequenza osservata nel database Si calcola come la probabilità che le sequenze NON abbiano subito mutazioni nell’intervallo di generazione.



Confronto col database Database di profili aplotipici e/o frequenze aplotipiche: 1. FBI Forensic mt. DNA Database: a) sezione criminale; b) sezione con accesso al pubblico 2. MITOMAP: raccolta completa delle mutazioni del umano: a) uso clinico b) uso forense 3. EMPOP: archivi disponibili di Database di popolazione: fornisce risultati che possono essere utilizzati per verificare la QUALITA’ dei dati di una sequenza mt. DNA. (16 ottobre 2006, http: //www. empop. org).

MITOMAP: Reported Mitochondrial DNA Base Substitution Diseases: Coding and Control Region Point Mutations



 Caracol col col sal de tu casita
Caracol col col sal de tu casita Kiwi dna extraction
Kiwi dna extraction Ciclo del progetto
Ciclo del progetto Implicature conversazionali esempi
Implicature conversazionali esempi Calcolo scostamenti
Calcolo scostamenti Esercitazione power point
Esercitazione power point Estrazione del lotto
Estrazione del lotto Validazione analitica
Validazione analitica Metodo abc esempio
Metodo abc esempio Metodo scientifico esempio concreto
Metodo scientifico esempio concreto Esempio di metodo sperimentale scuola primaria
Esempio di metodo sperimentale scuola primaria Induttivo e deduttivo
Induttivo e deduttivo Metodo hay esempio
Metodo hay esempio Metodo montecarlo esempio
Metodo montecarlo esempio Metodo full costing esempio
Metodo full costing esempio Estrazione separazione miscugli
Estrazione separazione miscugli Estrazione in controcorrente
Estrazione in controcorrente Cromatografia clorofilla
Cromatografia clorofilla Probabilità matematica
Probabilità matematica Estrazione con imbuto separatore
Estrazione con imbuto separatore Estrazione accelerata con solvente
Estrazione accelerata con solvente Solu zanichelli
Solu zanichelli Fenolo cloroformio alcool isoamilico
Fenolo cloroformio alcool isoamilico Estrazione solido liquido
Estrazione solido liquido Cortisone dopo estrazione dente
Cortisone dopo estrazione dente Function of dna polymerase 3
Function of dna polymerase 3 Bioflix activity dna replication nucleotide pairing
Bioflix activity dna replication nucleotide pairing Coding dna and non coding dna
Coding dna and non coding dna What are the enzymes involved in dna replication
What are the enzymes involved in dna replication Dna and genes chapter 11
Dna and genes chapter 11 Linea del tiempo de la historia
Linea del tiempo de la historia Cól miera
Cól miera Rüzgar biriktirme şekilleri
Rüzgar biriktirme şekilleri Examples of phytomastigophora
Examples of phytomastigophora Infundibulation du col vesical
Infundibulation du col vesical Mendian larrartean
Mendian larrartean Modifikasyon
Modifikasyon Distocia
Distocia Cancer du col
Cancer du col Colossians 3:12-13
Colossians 3:12-13 Cancer du col
Cancer du col Move zoppa gentil piede ineguale
Move zoppa gentil piede ineguale Frasi con past simple
Frasi con past simple Red hot root words
Red hot root words Colonel frank skimmerhorn
Colonel frank skimmerhorn Ntu col
Ntu col Agricultura 79 col escandon
Agricultura 79 col escandon Match col
Match col Douglas mcgregor theory y
Douglas mcgregor theory y Col 1:15-18
Col 1:15-18 Ensor middle
Ensor middle 1/2 col
1/2 col Nefrograma izotopica
Nefrograma izotopica Tundra biyomunda yaşayan hayvanlar
Tundra biyomunda yaşayan hayvanlar Gingiva marginal
Gingiva marginal Colosas 1:9-14
Colosas 1:9-14 Verdura col
Verdura col Col 1 nasb
Col 1 nasb Confinava col sannio
Confinava col sannio Col
Col Col 106 amit kumar
Col 106 amit kumar Col 1:21-23
Col 1:21-23 Aisha mian leaked
Aisha mian leaked Kapalı havza gölleri
Kapalı havza gölleri Col matthew hepburn
Col matthew hepburn