Taller en Anlisis filogenticos comparativos en Ecofisiologa Aplicacin



































- Slides: 56

Taller en Análisis filogenéticos comparativos en Ecofisiología Aplicación de Mesquite y R

Programa • Primero (11 Diciembre) • Introduction to Mesquite and R • Data Preparation and Manipulation • Tardes – Practical Use of Mesquite y R • Segundo (12 Diciembre) • Selection of Phylogenetic Trees • Supertrees – Assemblying Composite Trees – Sources of Phylogenetic Hypotheses • Estimation of Ancestral Character States – Categorical (Mesquite & R) – Continuous (R – ace)

Programa • Tercera (13 Diciembre) – Estimation of Phylogenetic Signal – Statistical Methods incorporating Phylogenetic information • Phylogenetic Independent Contrasts • Phylogenetic GLS • Multivariate Analyses – Bring your own Data!

Goals of Comparative Analyses • • Investigar la evolución carácter La coevolución de caracteres Control de la no independencia de las especies Hipótesis de ensayo de adaptación

Estadísticas tradicionales de asumir la independencia de las especies (unidades de muestreo)…

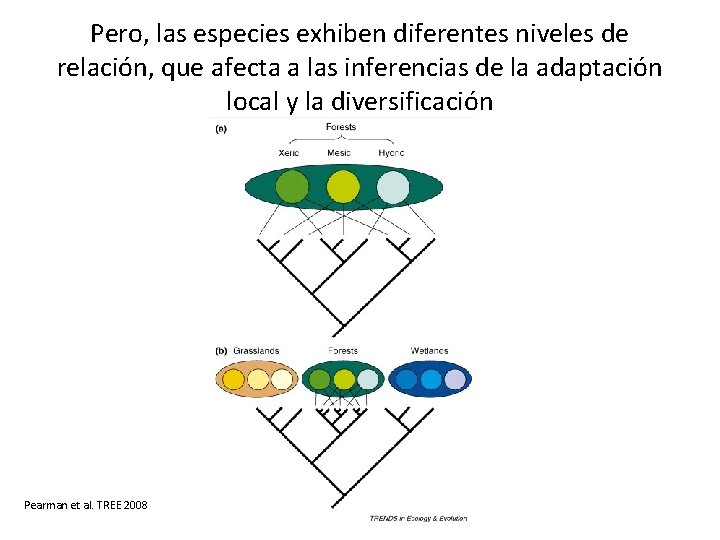
Pero, las especies exhiben diferentes niveles de relación, que afecta a las inferencias de la adaptación local y la diversificación Pearman et al. TREE 2008

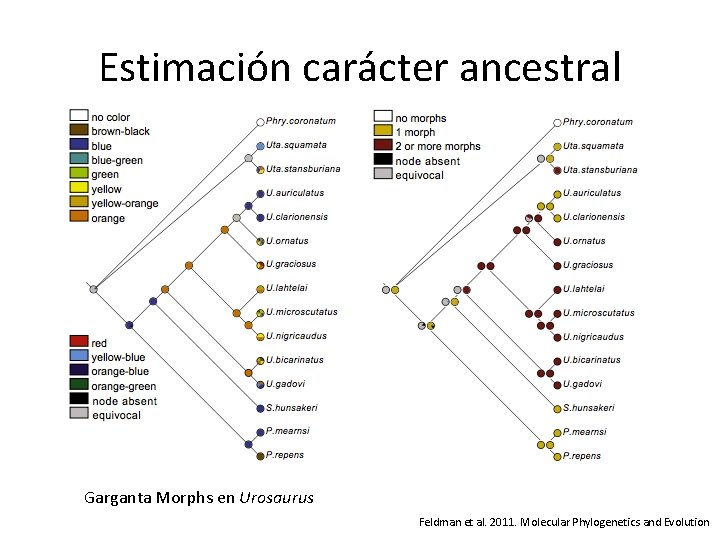
Estimación carácter ancestral Garganta Morphs en Urosaurus Feldman et al. 2011. Molecular Phylogenetics and Evolution

Dos importantes programas http: //mesquiteproject. org/mesquite. html http: //cran. r-project. org/

Objetivos de Mesquite • Manipulate Phylogenetic Trees – Estimate Ancestral Character States – Estimate Character Correlations – Inferences of Character Evolution – Multivariate Analyses

Objetivos para • How to use R to Manipulate Data • Phylogenetic Comparative Analysis • Statistical Analyses not available in Mesquite

Ventajas de • • Free Many packages available Powerful and Flexible Platform Independent – Mac. OS – Linux – Windows

Página de Inicio para R

Console de

R Studio – A GUI for http: //www. rstudio. com/


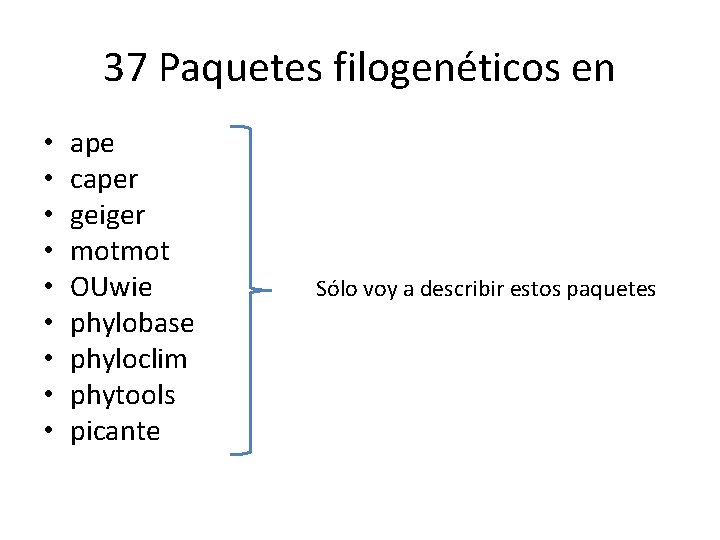
37 Paquetes filogenéticos en • • • ape caper geiger motmot OUwie phylobase phyloclim phytools picante Sólo voy a describir estos paquetes

Datos Necesarios • Phylogenetic Tree – NEXUS format – NEWICK format ((B: 0. 2, (C: 0. 3, D: 0. 4)E: 0. 5)F: 0. 1)A • Data – Continuous – Discrete – Flat Format (Texto, ASCII)

Nexus Data File Format #nexus. . . begin trees; translate 1 Phrynosoma, 2 Uta, 3 Petrosaurus, 4 Urosaurus, 5 Sceloporus ; tree one = [&U] (1, 2, (3, (4, 5)); tree two = [&U] (1, 3, (5, (2, 4)); end;

A tutorial in Mesquite • Tres elementos de Mesquite 1. Characters 2. Taxa 3. Trees

Primero Ventana de Mesquite Projects and Files – list of open projects Log – list of commands

Crear un nuevo proyecto (archivo)

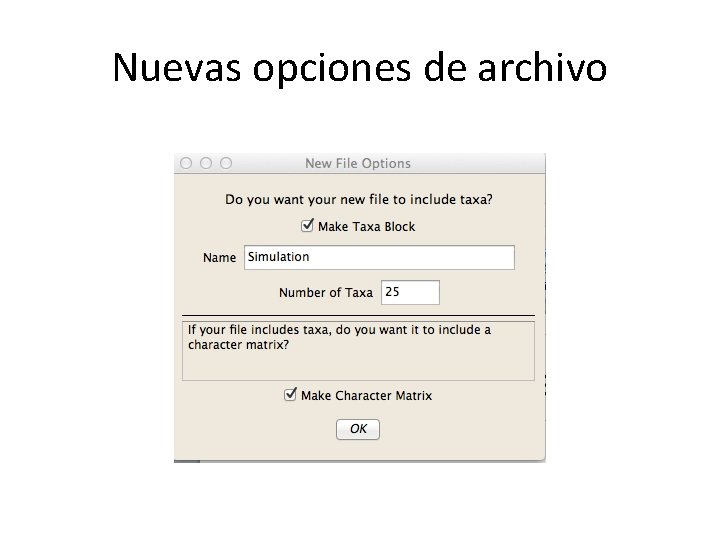
Nuevas opciones de archivo

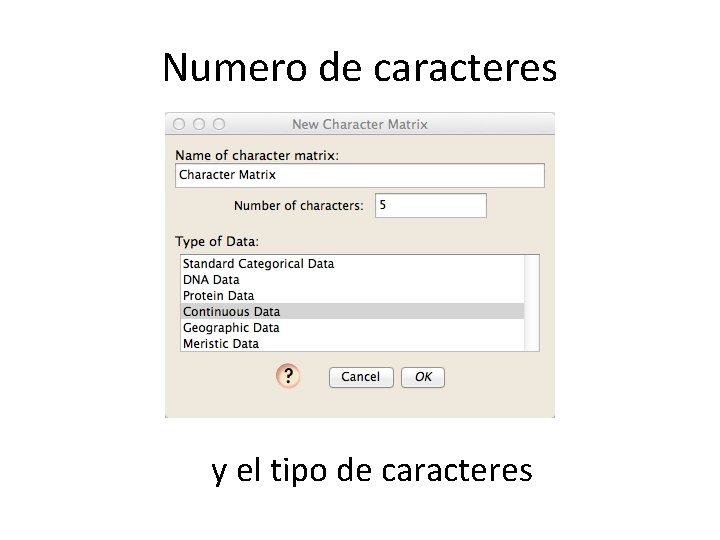
Numero de caracteres y el tipo de caracteres

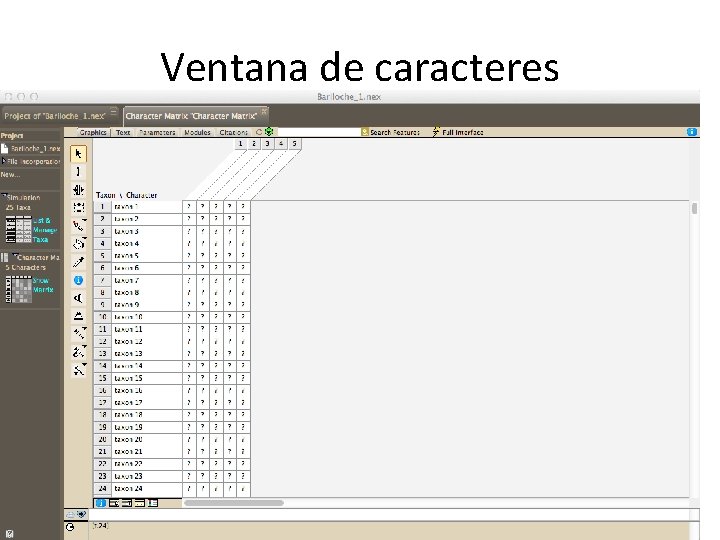
Ventana de caracteres

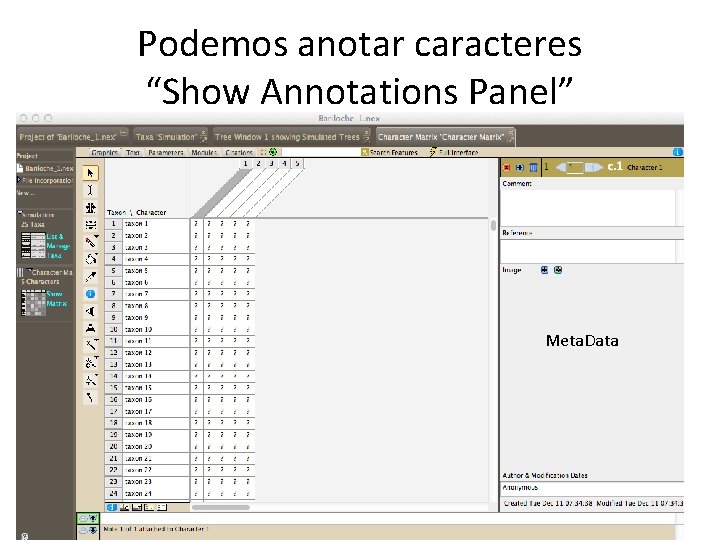
Podemos anotar caracteres “Show Annotations Panel” Meta. Data

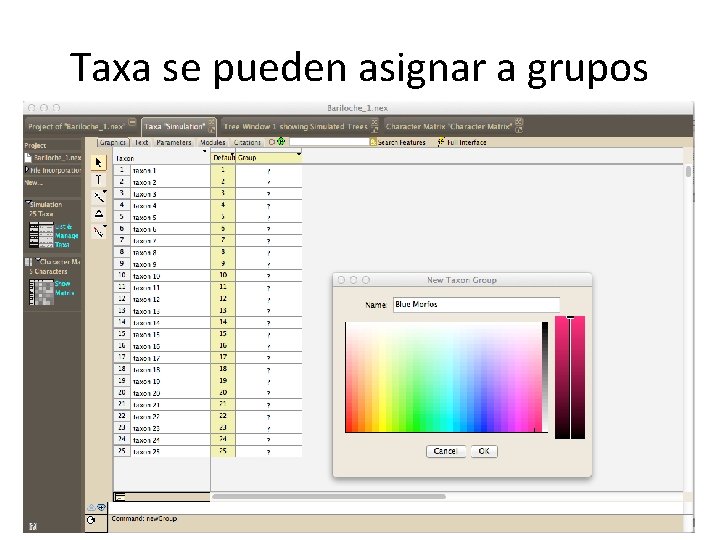
Taxa se pueden asignar a grupos

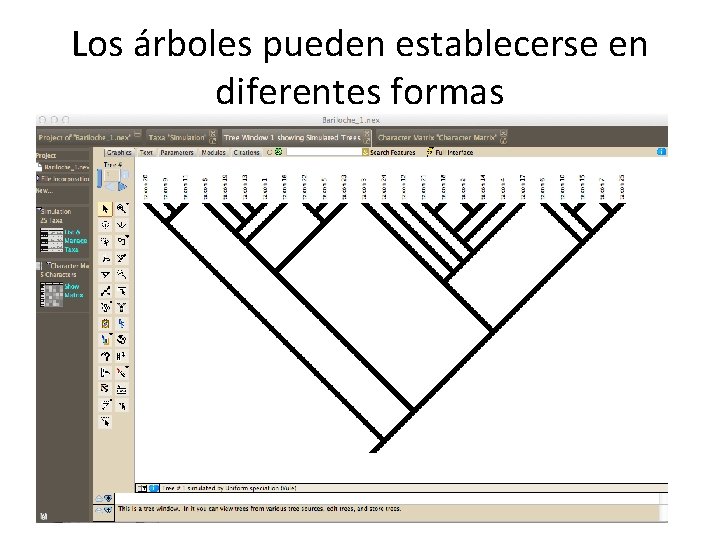
Los árboles pueden establecerse en diferentes formas



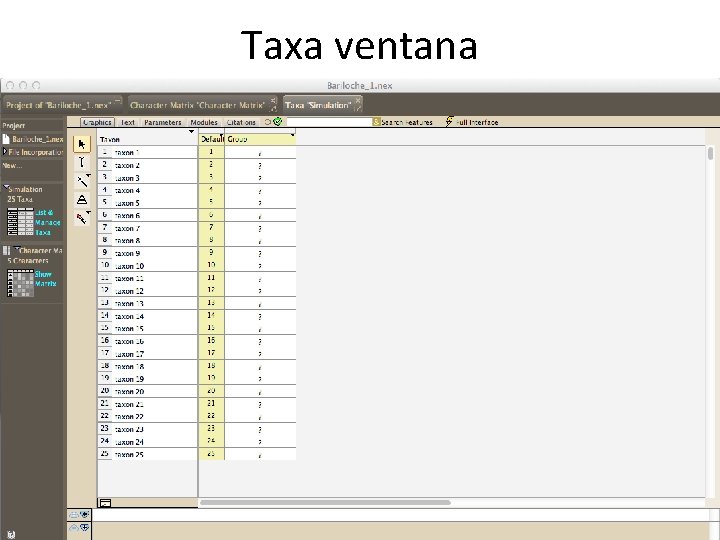
Taxa ventana

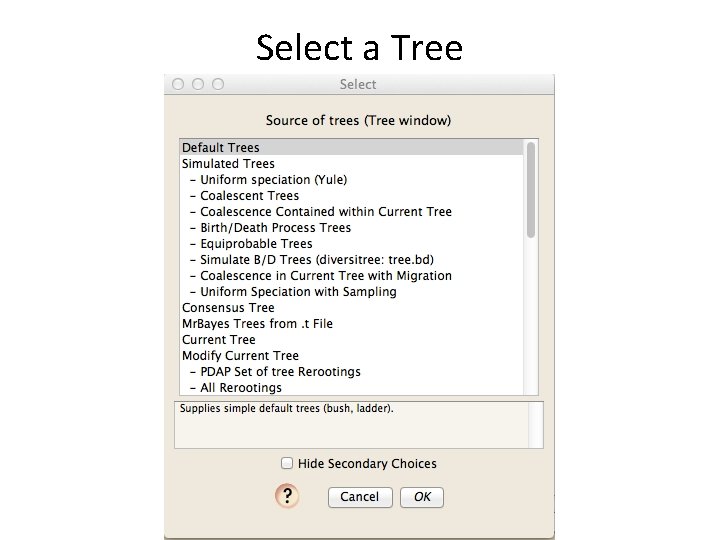
Select a Tree

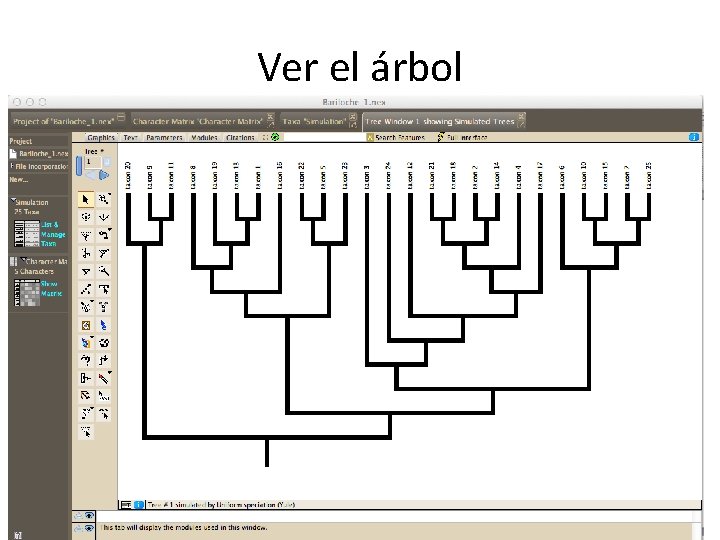
Ver el árbol

Total del Proyecto

A Gentle Introduction to

El intérprete interactivo, R

Asigna variables

Types of Variables Mode Numeric Character Factor Logical Example 10. 2, 20 “Morph”, “Substrate” Categorical TRUE, FALSE, T, F

Operators

Functions

Combinando elementos en matrices

Matrices

Operations on Matrices

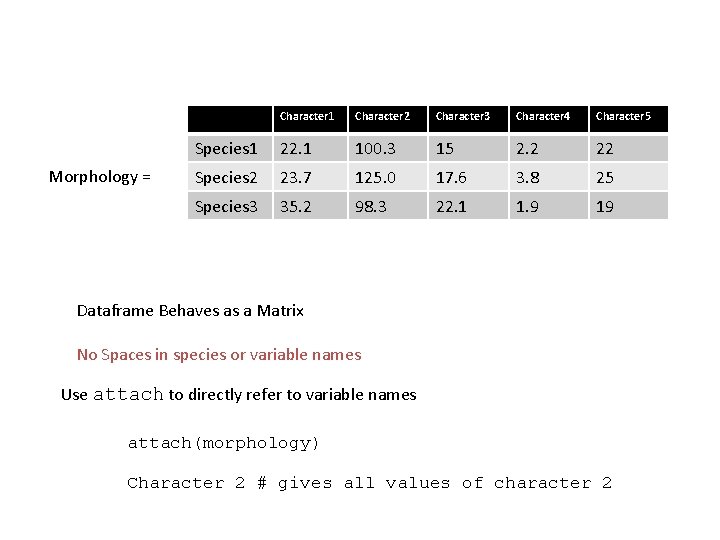
Dataframes • Rectangular table of information – Can include numbers, text • This is the form of your data when you import into R Character 1 Character 2 Species 1 Species 2 Species 3 Character 4 Character 5

Morphology = Character 1 Character 2 Character 3 Character 4 Character 5 Species 1 22. 1 100. 3 15 2. 2 22 Species 2 23. 7 125. 0 17. 6 3. 8 25 Species 3 35. 2 98. 3 22. 1 1. 9 19 Dataframe Behaves as a Matrix No Spaces in species or variable names Use attach to directly refer to variable names attach(morphology) Character 2 # gives all values of character 2

Use Help

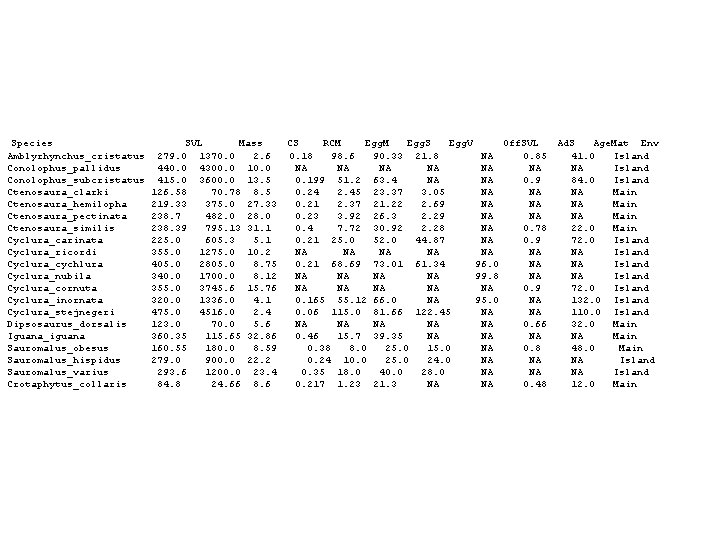
Importación de datos • Change the “working directory” – Easy in R Studio • Data should be in a clean rectangular matrix – Flat File (No formatting), ASCII text – Exported from excel • First row: Variable Names • First column: Species/Taxon Names • example: Iguana Life History Data

Species Amblyrhynchus_cristatus Conolophus_pallidus Conolophus_subcristatus Ctenosaura_clarki Ctenosaura_hemilopha Ctenosaura_pectinata Ctenosaura_similis Cyclura_carinata Cyclura_ricordi Cyclura_cychlura Cyclura_nubila Cyclura_cornuta Cyclura_inornata Cyclura_stejnegeri Dipsosaurus_dorsalis Iguana_iguana Sauromalus_obesus Sauromalus_hispidus Sauromalus_varius Crotaphytus_collaris SVL Mass 279. 0 1370. 0 2. 6 440. 0 4300. 0 10. 0 415. 0 3600. 0 13. 5 126. 58 70. 78 8. 5 219. 33 375. 0 27. 33 238. 7 482. 0 28. 0 238. 39 795. 13 31. 1 225. 0 605. 3 5. 1 355. 0 1275. 0 10. 2 405. 0 2805. 0 8. 75 340. 0 1700. 0 8. 12 355. 0 3745. 6 15. 76 320. 0 1336. 0 4. 1 475. 0 4516. 0 2. 4 123. 0 70. 0 5. 6 360. 35 115. 65 32. 86 160. 55 180. 0 8. 59 279. 0 900. 0 22. 2 293. 6 1200. 0 23. 4 84. 8 24. 66 8. 6 CS RCM Egg. S Egg. V Off. SVL 0. 18 98. 6 90. 33 21. 8 NA 0. 85 NA NA NA 0. 199 51. 2 63. 4 NA NA 0. 9 0. 24 2. 45 23. 37 3. 05 NA NA 0. 21 2. 37 21. 22 2. 69 NA NA 0. 23 3. 92 26. 3 2. 29 NA NA 0. 4 7. 72 30. 92 2. 28 NA 0. 78 0. 21 25. 0 52. 0 44. 87 NA 0. 9 NA NA NA 0. 21 68. 69 73. 01 61. 34 96. 0 NA NA NA 99. 8 NA NA NA 0. 9 0. 165 55. 12 66. 0 NA 95. 0 NA 0. 06 115. 0 81. 66 122. 45 NA NA 0. 66 0. 46 15. 7 39. 35 NA NA NA 0. 38 8. 0 25. 0 15. 0 NA 0. 8 0. 24 10. 0 25. 0 24. 0 NA NA 0. 35 18. 0 40. 0 28. 0 NA NA 0. 217 1. 23 21. 3 NA NA 0. 48 Ad. S Age. Mat Env 41. 0 Island NA Island 84. 0 Island NA Main 22. 0 Main 72. 0 Island NA Island 72. 0 Island 132. 0 Island 110. 0 Island 32. 0 Main NA Main 48. 0 Main NA Island 12. 0 Main

Importing Data • Workhorse function: read. table() iguana. lh <- read. table(file=“iguanalh. txt”, header=TRUE) • iguana. lh (dataframe name) • Check to make sure data were read in correctly • iguana. lh[1: 10, ] # look at first 10 rows

Otras formas de importar datos • Other formats: read. csv(), read. delim() • (useful if there are spaces within some fields) • Handy function: file. choose() # navigate to file iguana. lh <- read. table(file=file. choose(), header=T) attach(iguana. lh) # easy to manipulate variables

Factors • Used to represent categorical data; by default, read. table() • converts columns with characters into factors • Factors look like strings, but are treated differently by functions • species # example of a factor • Factors have levels, which are the unique values it takes • levels(species) # example of a factor • Factor levels may be ordered (e. g. , low, med, high), which is important in some analyses (see ? factor and ? ordered)

Faltan Datos • Represented by a special character in R: NA • Many functions have an argument na. rm – If TRUE, NA’s are removed – FALSE is usually default (function returns NA) – median(x, na. rm=TRUE) • read. table(file, na. strings=“NA”) • na. strings=“-999” # here, missing data are -999 • • Useful function: complete. cases(iguanlh. txt) • • Returns logical vector, T for rows without missing data • cc<- complete. cases(iguanalh. txt)

Los métodos filogenéticos en R • Start with a Tree • library(ape) # load ape package • We can create a random tree: • tree <- rtree(25) # 25 terminal taxa • Normally, read in tree(s) from file • tree <- read. nexus(file) # Nexus format • tree <- read. tree(file, …) # Newick format

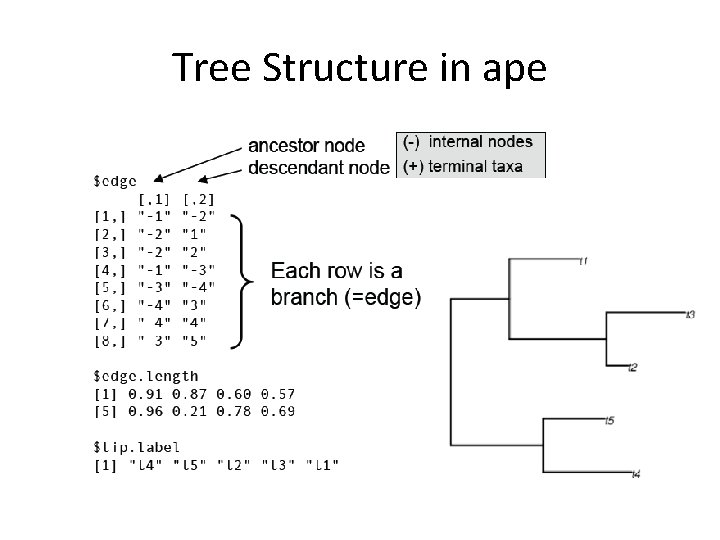
Tree Structure in ape

Plot a Tree

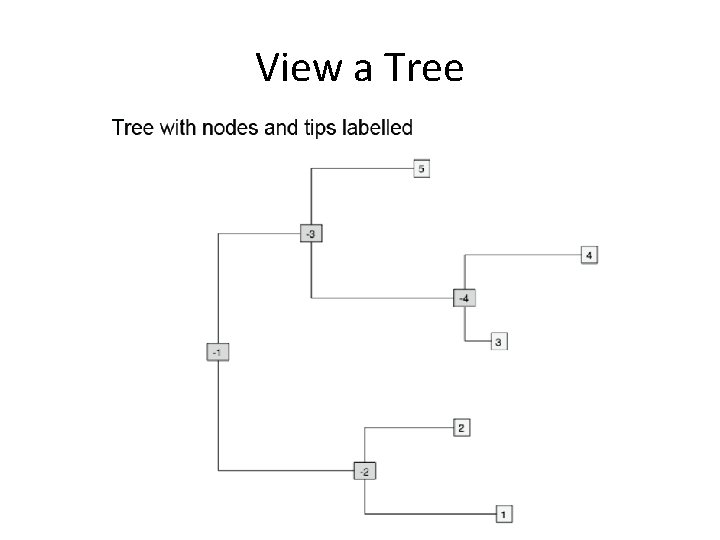
View a Tree

Manana Tree Selection Ancestor Character State Estimation
 Aplicacin
Aplicacin Master memfi
Master memfi Analisis foda maxi mini ejemplos
Analisis foda maxi mini ejemplos Anlisis de riesgos
Anlisis de riesgos Estrategias maxi maxi
Estrategias maxi maxi Amenazas foda ejemplos
Amenazas foda ejemplos Anlisis financiero
Anlisis financiero Quadros comparativos
Quadros comparativos Comparativos de desigualdad
Comparativos de desigualdad Nexos conclusivos
Nexos conclusivos Comparativos regulares e irregulares
Comparativos regulares e irregulares Socialismo y capitalismo cuadro comparativo
Socialismo y capitalismo cuadro comparativo Conectores textuales disyuntivos
Conectores textuales disyuntivos Organizadores previos
Organizadores previos Taller de liderazgo cristiano
Taller de liderazgo cristiano Competencia en el taller history
Competencia en el taller history Taller 1
Taller 1 Tecnisismos
Tecnisismos Taller
Taller Taller de textos: leer, escribir y comentar en el aula
Taller de textos: leer, escribir y comentar en el aula Gagne
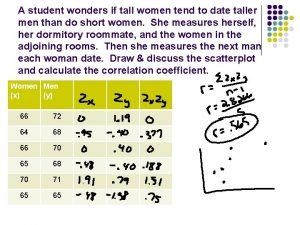
Gagne A student wonders if tall women tend to date taller men
A student wonders if tall women tend to date taller men Taller de literatura 3 medio
Taller de literatura 3 medio Serious comparative form
Serious comparative form Taller
Taller Taller teorema de tales
Taller teorema de tales Taller 1
Taller 1 Imagenes taller de lectura y redaccion
Imagenes taller de lectura y redaccion Tidy comparativo
Tidy comparativo Taller de contabilidad financiera
Taller de contabilidad financiera Taller
Taller Taller vocacional
Taller vocacional Taller manejo de emociones para padres
Taller manejo de emociones para padres Tecnica de la encuesta
Tecnica de la encuesta Taller de logica proposicional
Taller de logica proposicional Proclamadores de la palabra
Proclamadores de la palabra Taller de tilde diacrítica
Taller de tilde diacrítica Taller de esi para padres
Taller de esi para padres Taller matematicas
Taller matematicas Comparative and superlative form of fantastic
Comparative and superlative form of fantastic Taller de base de datos
Taller de base de datos Arbol de habitos taller intensivo
Arbol de habitos taller intensivo Taller de uso responsable de las redes sociales
Taller de uso responsable de las redes sociales Objetivo de solidaridad
Objetivo de solidaridad Are you taller lying down or standing
Are you taller lying down or standing Radios matra
Radios matra Taller estilos de aprendizaje
Taller estilos de aprendizaje Foda sunat
Foda sunat Taller de contabilidad
Taller de contabilidad Modelo de contingencia en la comunicación
Modelo de contingencia en la comunicación Taller de bases de datos
Taller de bases de datos Taller de lectura y redaccion
Taller de lectura y redaccion Taller sobre aplicaciones en la administración
Taller sobre aplicaciones en la administración Taller de editores
Taller de editores Taller de duelo para padres con hijos con discapacidad
Taller de duelo para padres con hijos con discapacidad Estructura de un taller educativo
Estructura de un taller educativo Comparative taller
Comparative taller