Sistema del complemento Participa de la respuesta innata


















































- Slides: 85

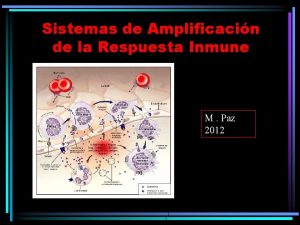
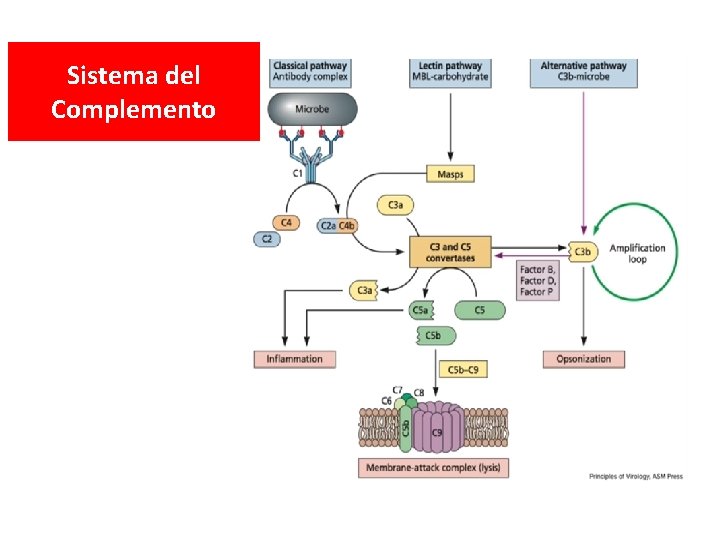
Sistema del complemento • Participa de la respuesta innata y adaptativa. • Cumple 4 acciones biológicas principales: 1)Lisis osmótica de células o virus envueltos 2)Activación de la inflamación 3)Opsonización de virus 4)Solubilización de inmunocomplejos

Sistema del Complemento

Colectinas • Son un grupo de lectinas con similitud estructural y biológica a C 1 q. • Se unen en la superficie de células o virus y pueden activar el sistema del complemento. • Pueden inhibir o potenciar la infectividad de diferentes virus.

Anticuerpos naturales • Corresponden al 2% del total de Ig. G e Ig. M séricas. • Su formación responde a la respuesta inducida contra antígenos ricos en galactosa. • Los «anticuerpos naturales» explican porque muchos virus que infectan a otros hospedadores, no infectan al hombre.

RESPUESTA ESPECÍFICA E INMUNIDAD ADAPTATIVA

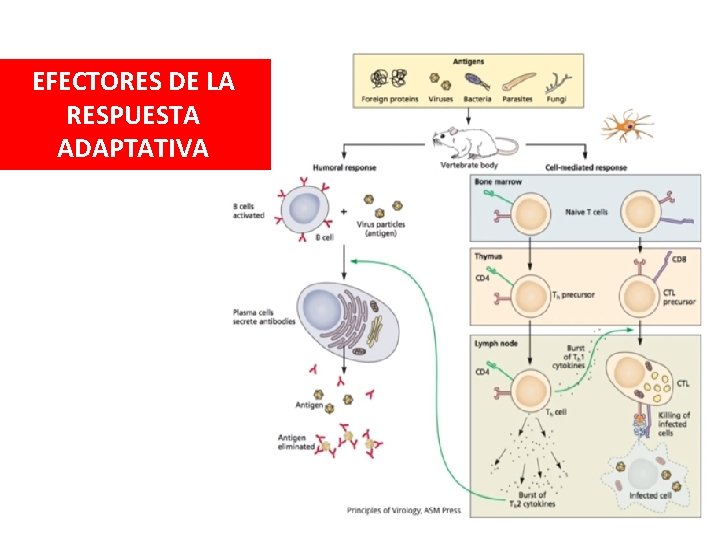
EFECTORES DE LA RESPUESTA ADAPTATIVA

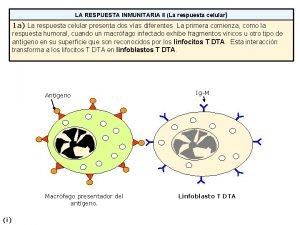
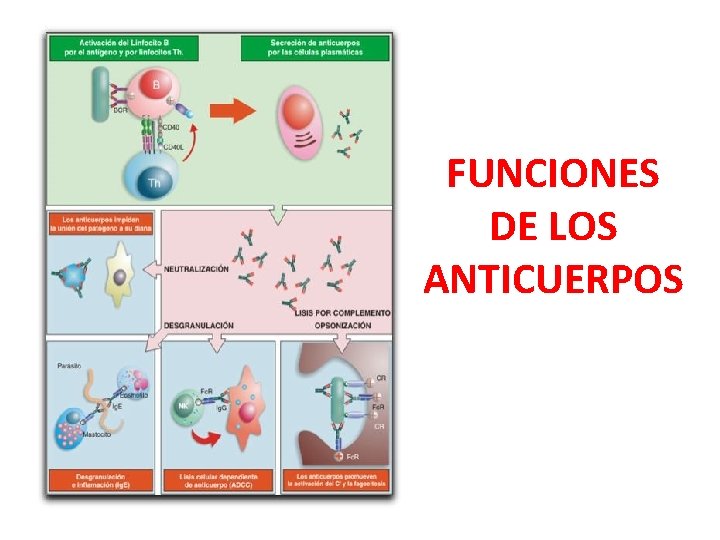
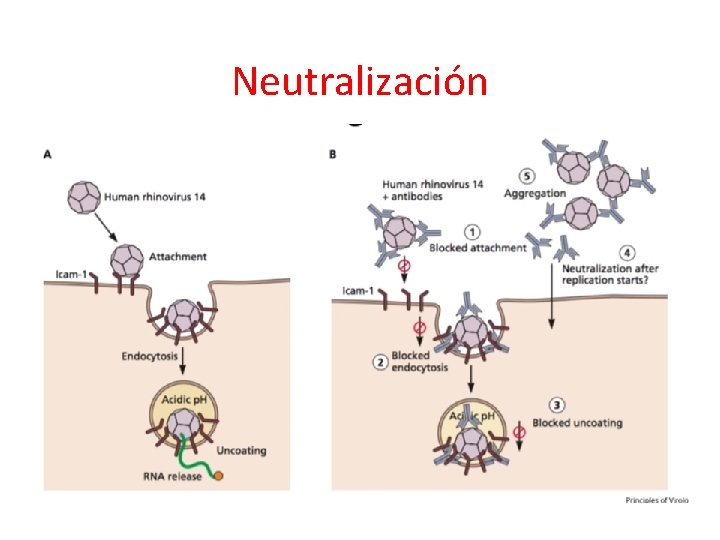
Inmunidad humoral: Anticuerpos • Controlan las infecciones causadas por virus que salen de la célula por lisis. Pueden participar en la neutralización viral si existe viremia, o en la lisis de células infectadas con participación del sistema del complemento, o de ciertas células citotóxicas con receptor para Fc (macrófagos o NK). • En el control de las infecciones virales: 1) eliminan la infección primaria, 2) limitan la viremia, y 3) limitan la enfermedad y previenen las reinfecciones.

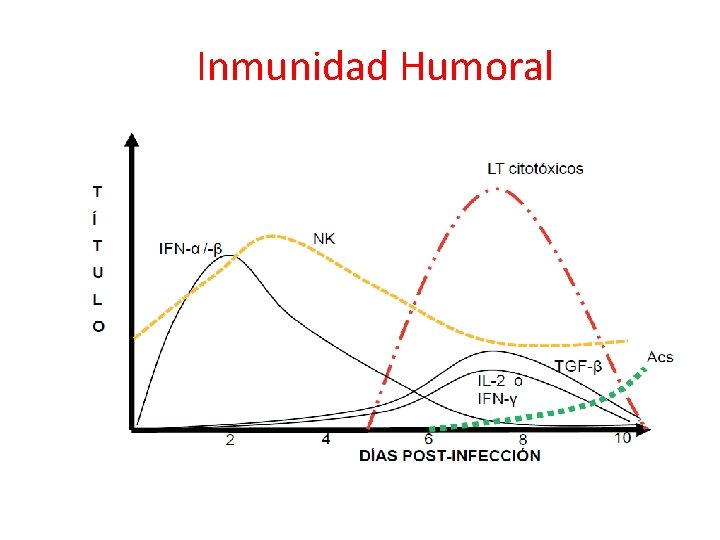
Inmunidad Humoral

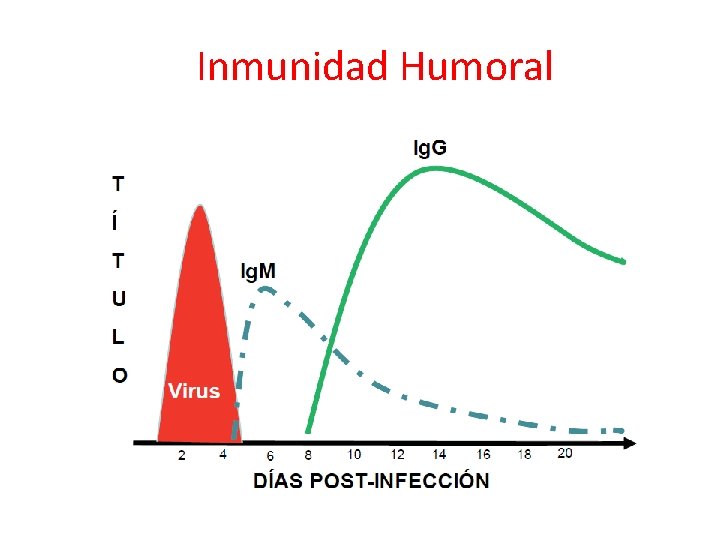
Inmunidad Humoral

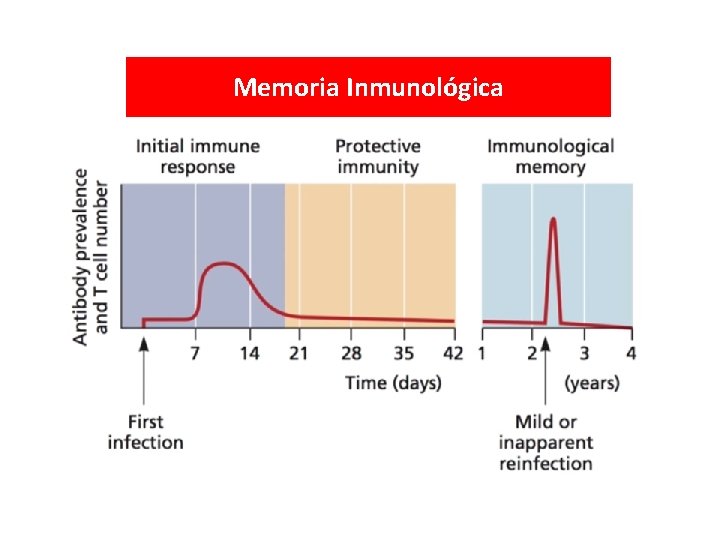
Memoria Inmunológica

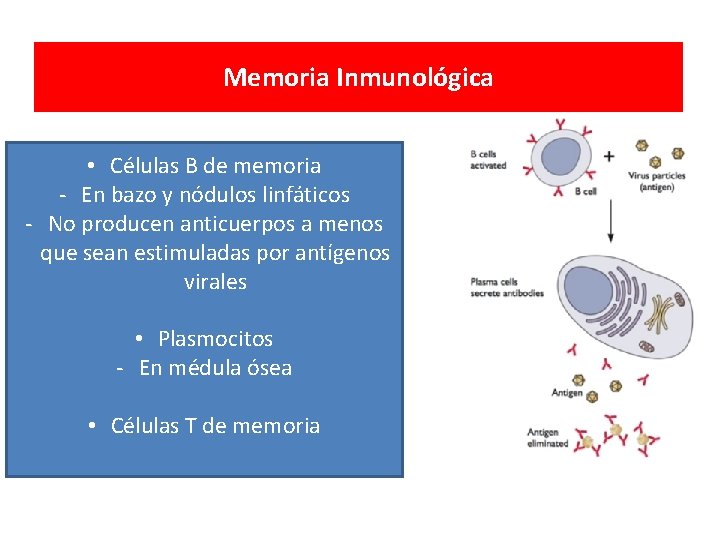
Memoria Inmunológica • Células B de memoria - En bazo y nódulos linfáticos - No producen anticuerpos a menos que sean estimuladas por antígenos virales • Plasmocitos - En médula ósea • Células T de memoria


FUNCIONES DE LOS ANTICUERPOS

Neutralización


Inmunidad Celular

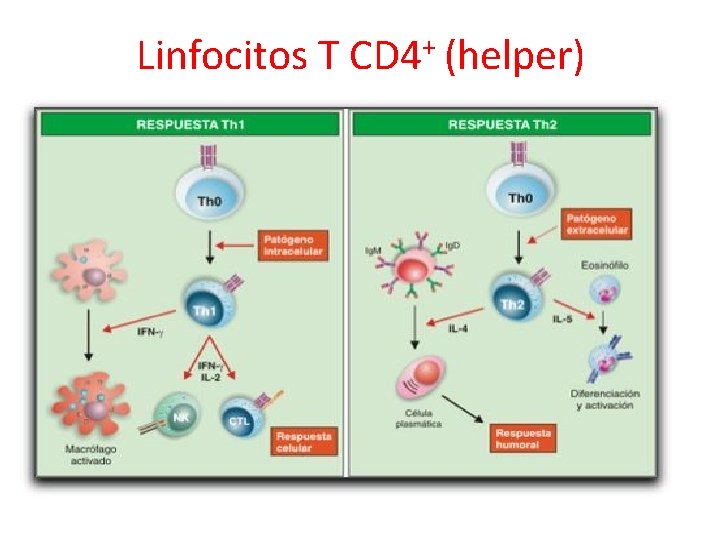
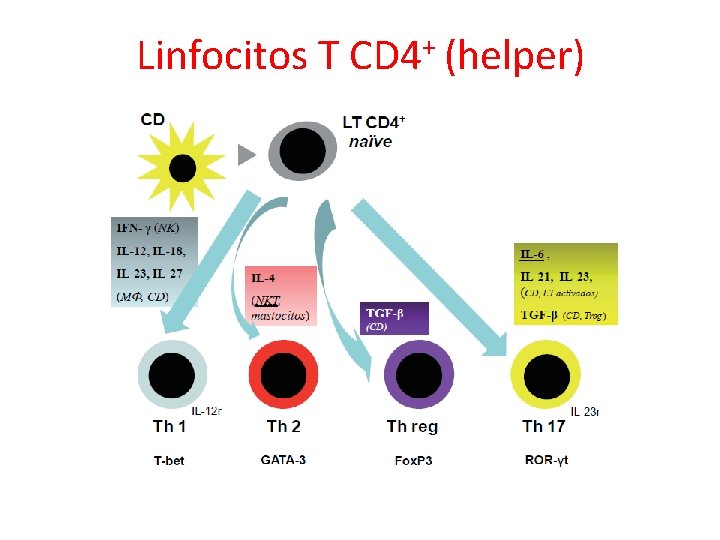
Linfocitos T + CD 4 (helper)

Linfocitos T + CD 4 (helper)

¿Qué es lo que determina que haya actividad citotóxica o producción de anticuerpos?

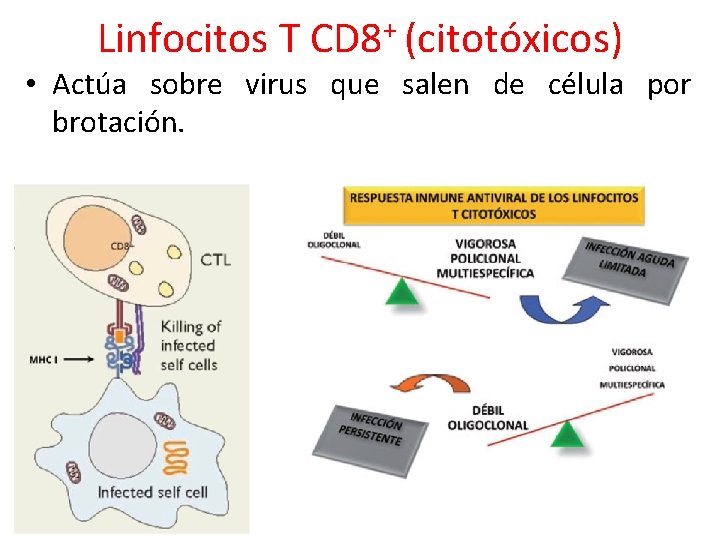
Linfocitos T CD 8+ (citotóxicos) • Actúa sobre virus que salen de célula por brotación.

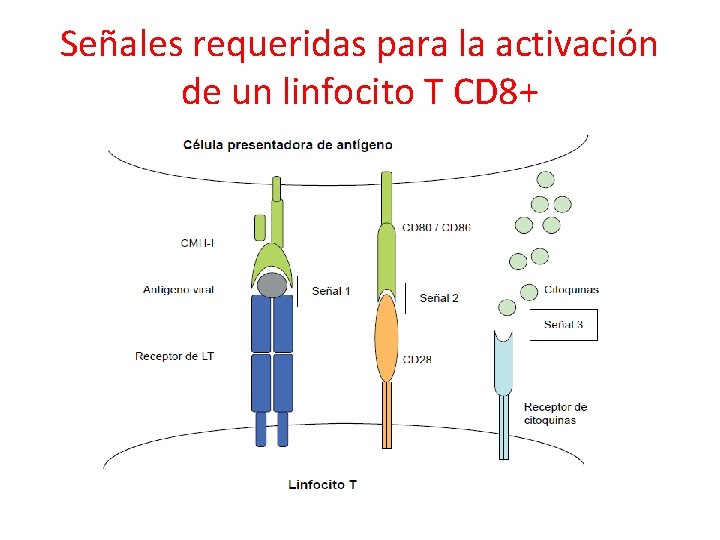
Señales requeridas para la activación de un linfocito T CD 8+

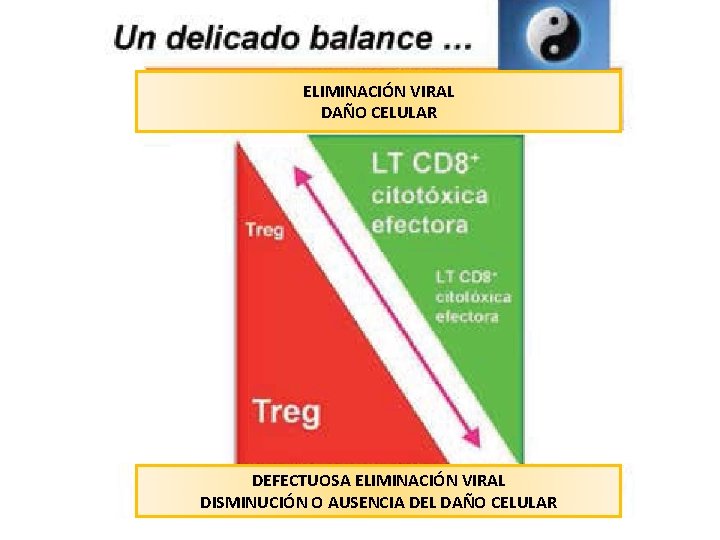
ELIMINACIÓN VIRAL DAÑO CELULAR DEFECTUOSA ELIMINACIÓN VIRAL DISMINUCIÓN O AUSENCIA DEL DAÑO CELULAR

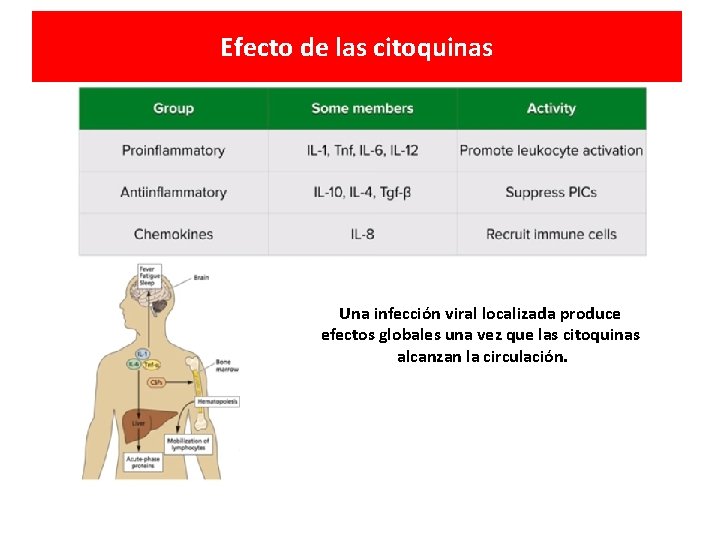
Efecto de las citoquinas Una infección viral localizada produce efectos globales una vez que las citoquinas alcanzan la circulación.

ESTO QUIERE DECIR QUE… LA RESPUESTA INFLAMATORIA CLÁSICA (CALOR, RUBOR, DOLOR, TUMOR) REFLEJA LA COMUNICACIÓN DE LOS MECANISMOS DE DEFENSA INNATOS Y ADAPTATIVOS. LA AUSENCIA DE UNA RESPUESTA INFLAMATORIA INDICA UNA INMUNIDAD ADAPTATIVA INEFICIENTE

Control de la respuesta inmune: Galectinas • Lectinas sintetizadas y almacenadas en el citosol y secretadas ante ciertos estímulos. • Presentes en todas las células del sist. inmune. • Funcionan como receptores solubles de reconocimiento de patrones (RRP) al reconocer y unirse a ligandos (PAMP) en la superficie de agentes exógenos. • Gracias a las galectinas, la respuesta inmune innata es capaz de discriminar entre una agente patógeno y otro que no lo es.

Galectina 1 Galectina 3 Inhibe fenómenos pro-inflamatorios Promueve la respuesta Th 2 Inhibe la respuesta Th 2

MECANISMO DE EVASION DE LA RESPUESTA INMUNE


Mecanismos de evasión viral EVITAR EL RECONOCIMIENTO POR EL SISTEMA INMUNE 1. Variación antigénica 2. Disminución de la expresión génica viral 3. Disociación temporal de la expresión génica viral ALTERACIÓN DE LOS MECANISMOS DE DEFENSA DEL HOSPEDADOR 1. Inhibición de la presentación antigénica 2. Inhibición o modificación de la actividad mediada por el sistema del interferón (IFN) 3. Modificación de la actividad mediada por otras citoquinas 4. Infección de células del sistema inmune y/o acción sobre las mismas a. Agotamiento del sistema inmune b. Actividad de superantígenos 5. Regulación de la apoptosis 6. Modificación de la actividad del sistema del complemento 7. Sobre-expresión de receptores para Fc y consiguiente unión de Igs 8. Interacción de proteínas virales con receptores/correceptores celulares

EVITAR EL RECONOCIMIENTO DEL SISTEMA INMUNE

1. Variación antigénica

Animal infectado

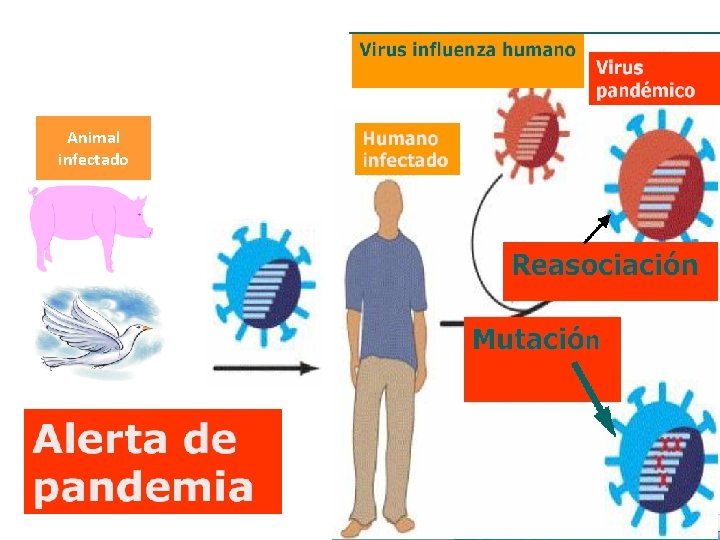
Variabilidad del virus Influenza


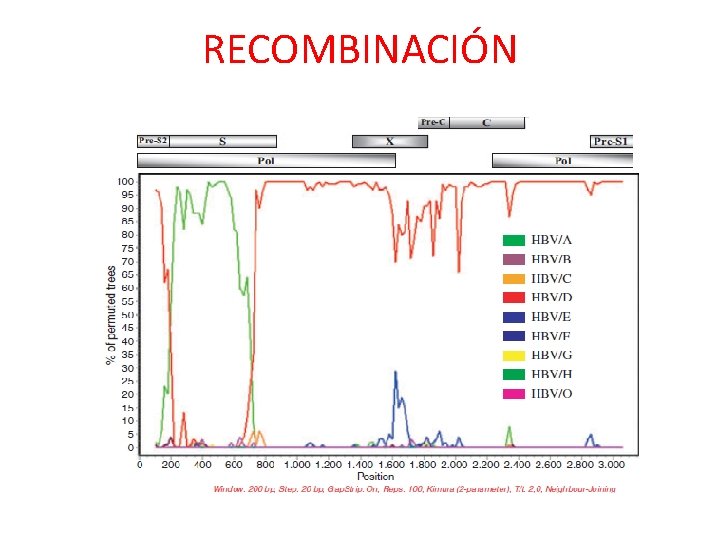
1. Variación antigénica • Reasociación genética (reordenamiento) en genomas segmentados Influenza: shift antigénico (cambios mayores) • Recombinación genética: HIV, HBV, HCV, enterovirus, rhinovirus, RSV, SARS-COV-2, etc. • Mutaciones puntuales: - Mutantes de escape a la rta. inmune humoral: HIV, HBV, HCV, Influenza - Mutantes de escape a la rta. inmune celular: HBV

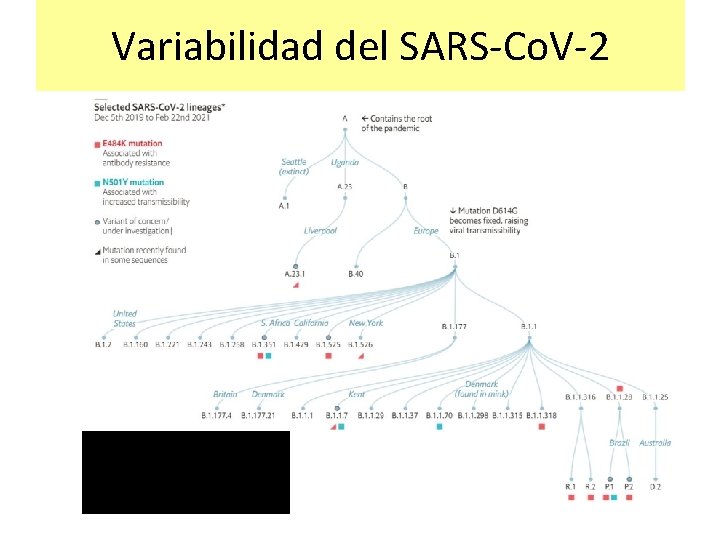
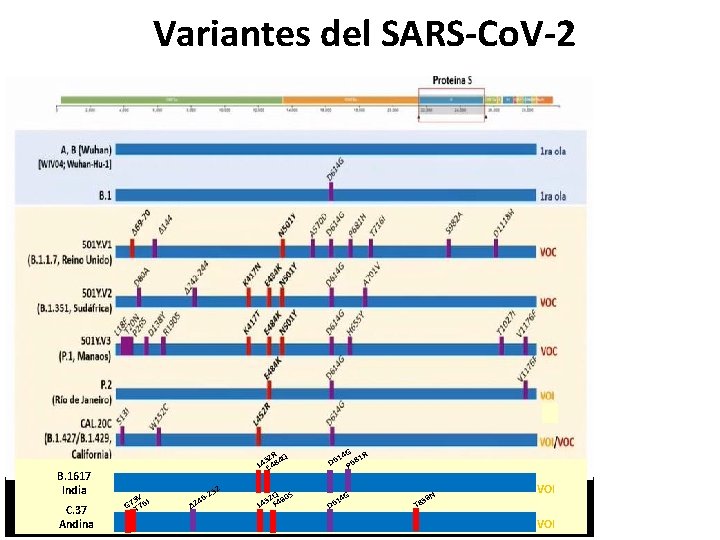
Variabilidad del SARS-Co. V-2

Variantes de interés y de preocupación Variante de interés (VOI) : Aislamiento del SARS-Co. V-2 con cambios fenotípicos con respecto al aislamiento de referencia o que posee un genoma con mutaciones que producen cambios aminoacídicos asociados con implicancias fenotípicas establecidas o sospechadas, Y ha sido identificada como causante de transmisión comunitaria/múltiples casos de COVID-19, o ha sido detectada en múltiples países. Variante de preocupación (VOC) : Una VOI que se ha demostrado estar asociada con: • Incremento en la transmisibilidad o cambio en la epidemiología de COVID 19; • Aumento en la virulencia o cambios en la presentación clínica; o • Disminución en la efectividad de las medidas sociales y de salud o métodos diagnósticos disponibles, vacunas, y tratamientos.

Variantes del SARS-Co. V-2 B. 1617 India C. 37 Andina 5 V I G 7 T 76 52 6 -2 4 ∆2 D 6 Q S 52 90 L 4 F 4 1 D 6 G R 81 P 6 R 52 Q L 4 E 484 14 4 G 9 N 5 T 8 VOI

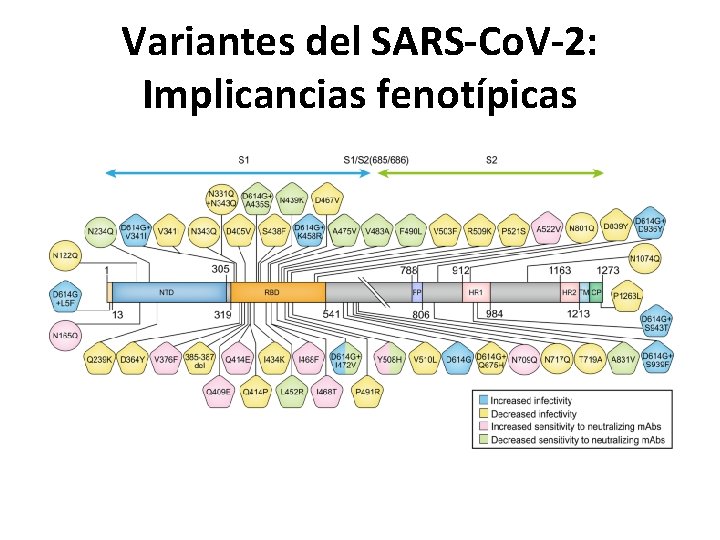
Variantes del SARS-Co. V-2: Implicancias fenotípicas

RECOMBINACIÓN

2. Disminución de la expresión génica viral • Latencia Flia. Herpesviridae (HSV, EBV, etc. ) HPV HBV en linfocitos Adenovirus en linfocitos T HIV en ciertos LT en reposo

3. Disociación temporal de la expresión génica viral • Genoma bisentido Arenavirus sólo cuando se expresa suficiente cuantía de la proteína de la nucleocápside (N) como anti-terminadora de la transcripción puede sintetizarse la glicoproteína G. La trasncripción y replicación viral están reguladas por Z.


ALTERACIÓN DE LOS MECANISMOS DE DEFENSA DEL HOSPEDADOR

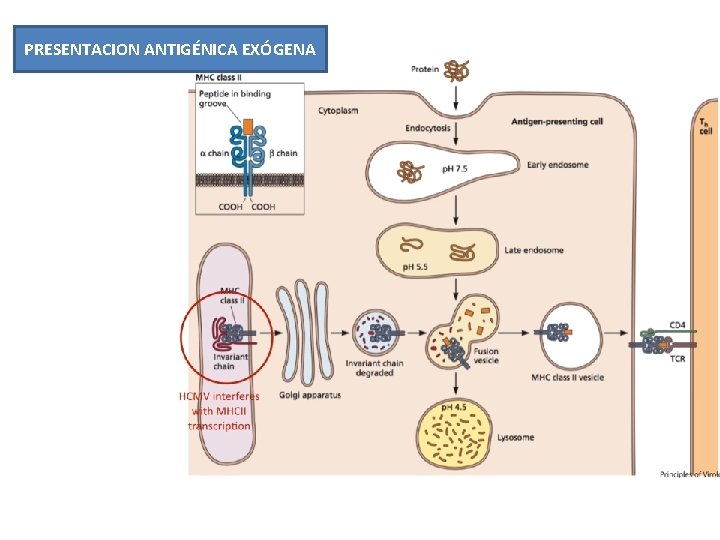
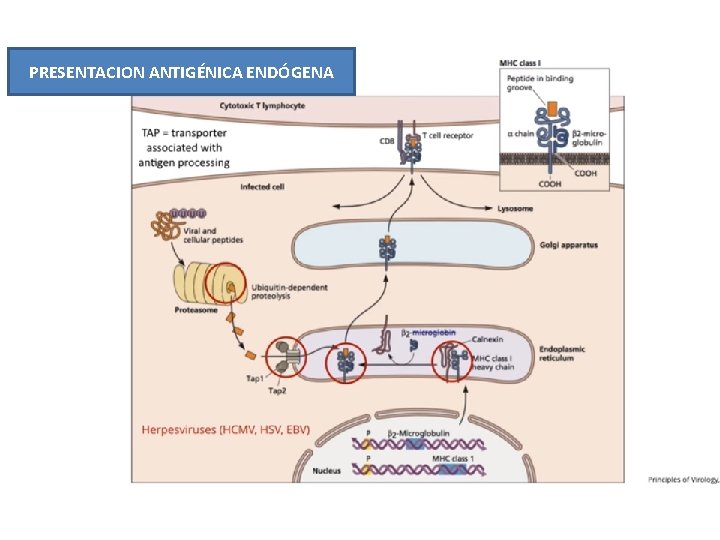
1. Inhibición de la presentación antigénica • Inhibición de la expresión de moléculas de CMH-I y/o CMH-II: - Inhibición de la expresión de moléculas de CMH-I en la superficie celular: HIV (Nef), HBV (Pol), Adenovirus (E 3), CMV (UL 18), HHV-8 (K 3 y K 5) y HCV (NS 5 B) - Inhibición de la expresión de moléculas de CMH-II en la superficie celular: VZV, EBV - Degradación de moléculas del CMH-I y CMH-II en el proteasoma: CMV (US 2) • Inhibición del procesamiento antigénico: - Inhibición de la generación del péptido antigénico: EBV (EBNA 1) - Inhibición del transporte intracelular de los antígenos virales: CMV (US 6 y US 3), HSV (ICP 47)

PRESENTACION ANTIGÉNICA EXÓGENA

PRESENTACION ANTIGÉNICA ENDÓGENA




2. Inhibición o modificación de la actividad mediada por el sistema del IFN A. Interferencia viral sobre la expresión general de los genes de la célula hospedadora B. Interferencia viral sobre los componentes específicos de los mecanismos de inducción del IFN C. Interferencia viral sobre las cascadas de transducción de señales activadas por IFN D. Interferencia viral sobre proteínas efectoras del sistema IFN

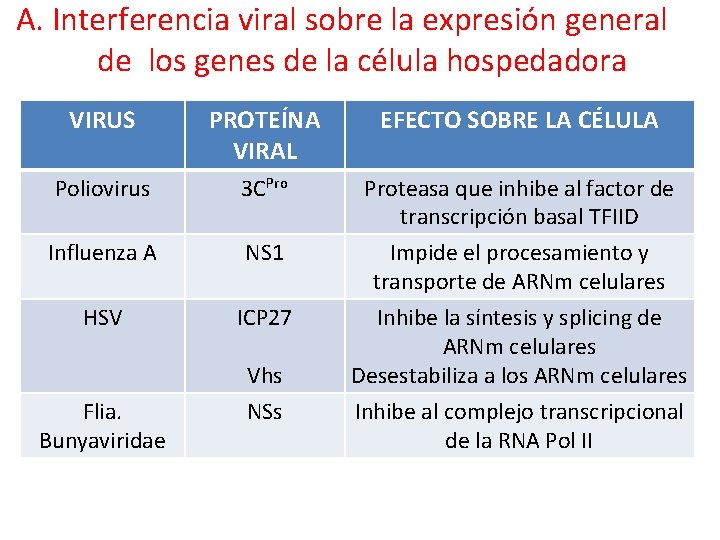
A. Interferencia viral sobre la expresión general de los genes de la célula hospedadora VIRUS PROTEÍNA VIRAL EFECTO SOBRE LA CÉLULA Poliovirus 3 CPro Proteasa que inhibe al factor de transcripción basal TFIID Influenza A NS 1 Impide el procesamiento y transporte de ARNm celulares HSV ICP 27 Inhibe la síntesis y splicing de ARNm celulares Desestabiliza a los ARNm celulares Vhs Flia. Bunyaviridae NSs Inhibe al complejo transcripcional de la RNA Pol II


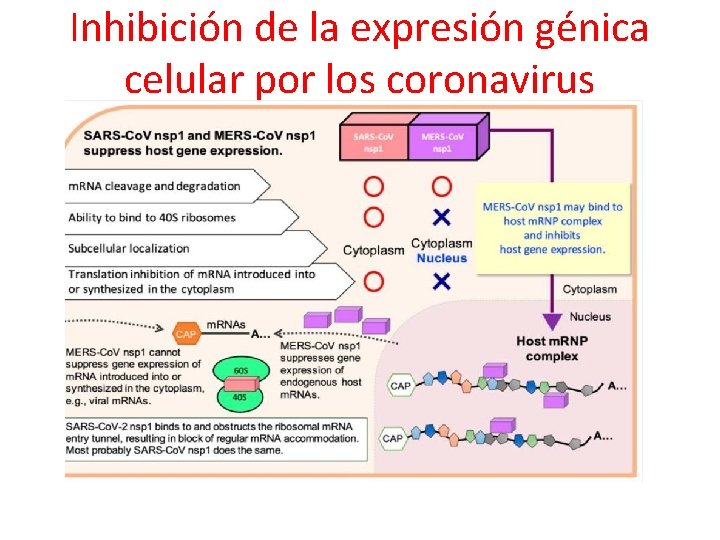
Inhibición de la expresión génica celular por los coronavirus

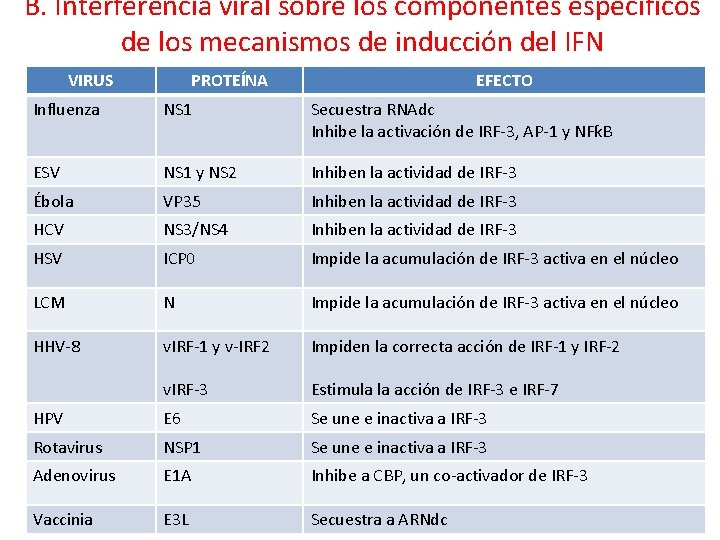
B. Interferencia viral sobre los componentes específicos de los mecanismos de inducción del IFN VIRUS PROTEÍNA EFECTO Influenza NS 1 Secuestra RNAdc Inhibe la activación de IRF-3, AP-1 y NFƙB ESV NS 1 y NS 2 Inhiben la actividad de IRF-3 Ébola VP 35 Inhiben la actividad de IRF-3 HCV NS 3/NS 4 Inhiben la actividad de IRF-3 HSV ICP 0 Impide la acumulación de IRF-3 activa en el núcleo LCM N Impide la acumulación de IRF-3 activa en el núcleo HHV-8 v. IRF-1 y v-IRF 2 Impiden la correcta acción de IRF-1 y IRF-2 v. IRF-3 Estimula la acción de IRF-3 e IRF-7 HPV E 6 Se une e inactiva a IRF-3 Rotavirus NSP 1 Se une e inactiva a IRF-3 Adenovirus E 1 A Inhibe a CBP, un co-activador de IRF-3 Vaccinia E 3 L Secuestra a ARNdc


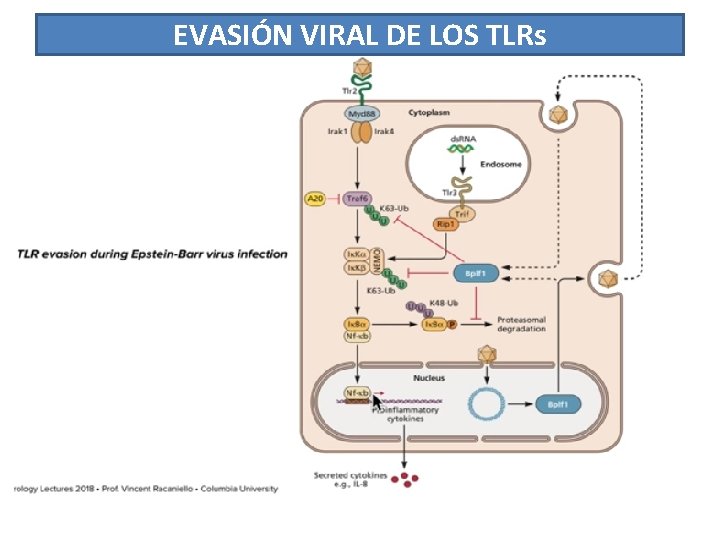
EVASIÓN VIRAL DE LOS TLRs

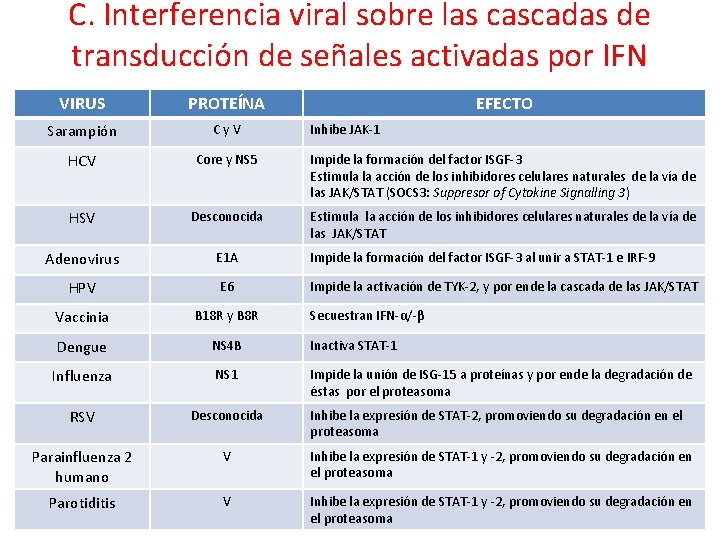
C. Interferencia viral sobre las cascadas de transducción de señales activadas por IFN VIRUS PROTEÍNA EFECTO Sarampión Cy. V HCV Core y NS 5 Impide la formación del factor ISGF-3 Estimula la acción de los inhibidores celulares naturales de la vía de las JAK/STAT (SOCS 3: Suppresor of Cytokine Signalling 3) HSV Desconocida Estimula la acción de los inhibidores celulares naturales de la vía de las JAK/STAT Adenovirus E 1 A Impide la formación del factor ISGF-3 al unir a STAT-1 e IRF-9 HPV E 6 Impide la activación de TYK-2, y por ende la cascada de las JAK/STAT Vaccinia B 18 R y B 8 R Dengue NS 4 B Inactiva STAT-1 Influenza NS 1 Impide la unión de ISG-15 a proteínas y por ende la degradación de éstas por el proteasoma RSV Desconocida Parainfluenza 2 humano V Inhibe la expresión de STAT-1 y -2, promoviendo su degradación en el proteasoma Parotiditis V Inhibe la expresión de STAT-1 y -2, promoviendo su degradación en el proteasoma Inhibe JAK-1 Secuestran IFN-α/-β Inhibe la expresión de STAT-2, promoviendo su degradación en el proteasoma


C. Interferencia viral sobre las cascadas de transducción de señales activadas por IFN

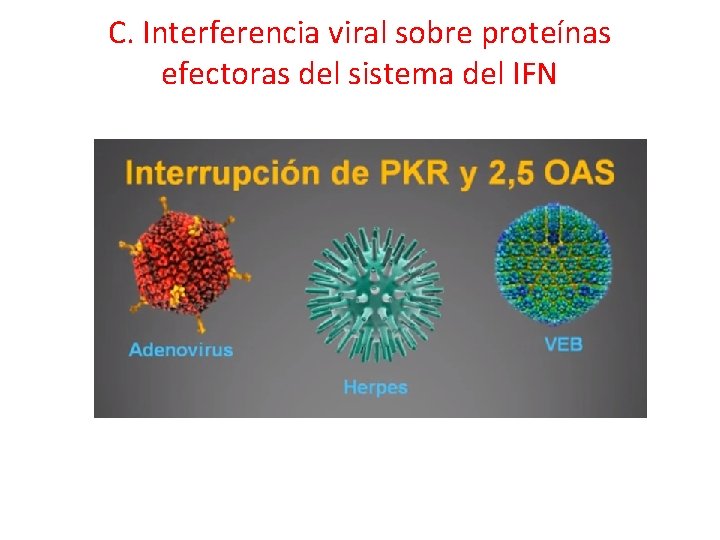
C. Interferencia viral sobre proteínas efectoras del sistema del IFN VIRUS PROTEÍNA VIRAL EFECTO HIV Tat TAR RNA (RNAp) Desconocida Inactiva a la PKR al actuar como pseudo-sustrato Impide la activación de la PKR al competir con el RNAdc Induce la síntesis de un inhibidor celular natural de la RNASA L (RLI) HCV NS 5 A E 2 IRES Inactiva a la PKR al actuar como pseudo-sustrato Impide la activación de la PKR al competir con el RNAdc Influenza NS 1 Desconocida Secuestra al RNAdc intracelular e inhibe a la PKR Estimula a un inhibidor celular natural de la PKR (p 58 IPK) Rotavirus NSP 3 Secuestra el RNAdc intracelular e inhibe la PKR HSV γ 34. 5 Desconocida Defosforila al e. IF 2α e inactiva PKR Induce la síntesis de análogos de 2’-5’OA que se une a la RNAsa L y la inactivan Secuestra al RNAdc intracelular e inhibe a la PKR y la síntesis de 2’-5’ OA sintetasa US 11 EBV EBER RNA (RNAp) Impide la activación de la PKR al competir con el RNAdc Adenovirus VAl RNA (RNAp) Impide la activación de la PKR al competir con el RNAdc HHV-8 v. IRF-2 Inactiva la PKR Vaccinia E 3 L K 3 L Secuestra al ARNdc intracelular e inhibe a la PKR Inactiva a la PKR al actuar como pseudo-sustrato


C. Interferencia viral sobre proteínas efectoras del sistema del IFN

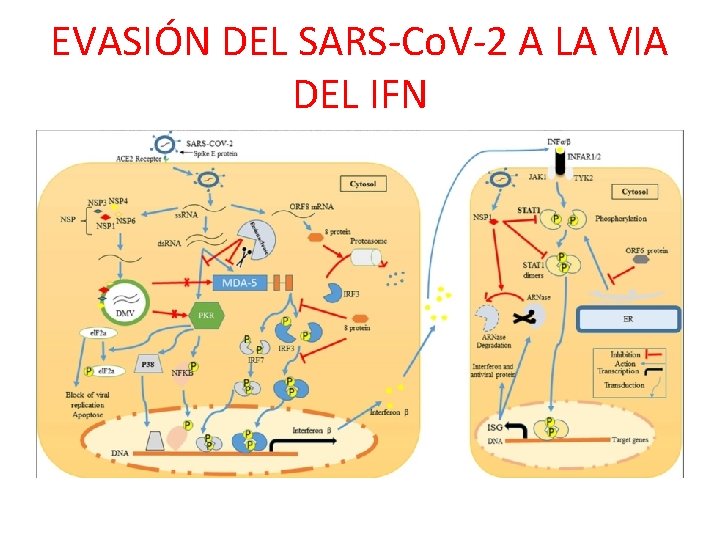
EVASIÓN DEL SARS-Co. V-2 A LA VIA DEL IFN

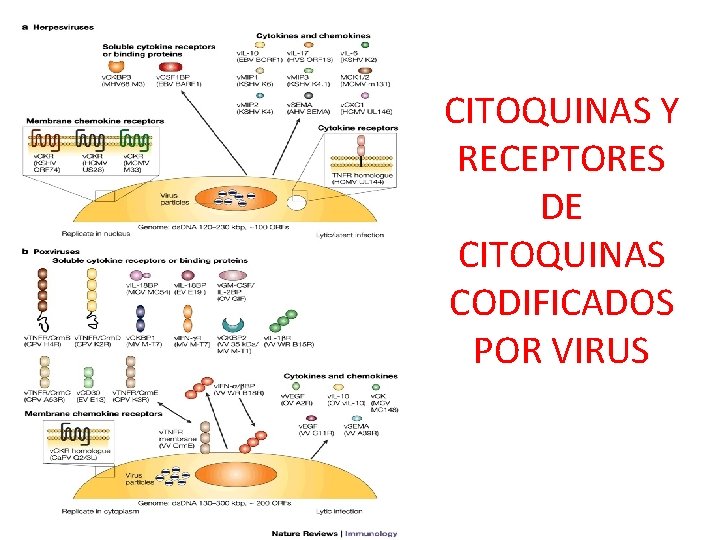
3. Modificación de la actividad mediada por otras citoquinas • • Proteína viral símil-citoquinas EBV (BCRF-1) y CMV (cmv. IL-10) Proteína viral símil-receptores para citoquinas CMV (US 27, US 28, UL 33 y UL 78) HHV-6 (U 12 y U 51) HHV-7 (U 12) HHV-8 (ORF 74 receptor símil CXCR) Vaccinia Inhibición de la síntesis de citoquinas: Vaccinia (Crm. A) Unión de citoquinas: HBV (pre-S 2)

CITOQUINAS Y RECEPTORES DE CITOQUINAS CODIFICADOS POR VIRUS





4. Infección de las células del sistema inmune y/o acción sobre las mismas • Infección de LT o LB: HIV, HBV, HCV, sarampión, adenovirus, etc. • Infección de células dendríticas y macrófagos: HIV, HBV, HCV, sarampión, etc. • Infección de células tímicas: LCM, HBV • Infección de linfocitos T mediante superantígenos: HIV • Inducción de agotamiento inmune: LCM, HIV, HCV • Inducción de apoptosis de LT: HIV, HSV, etc.


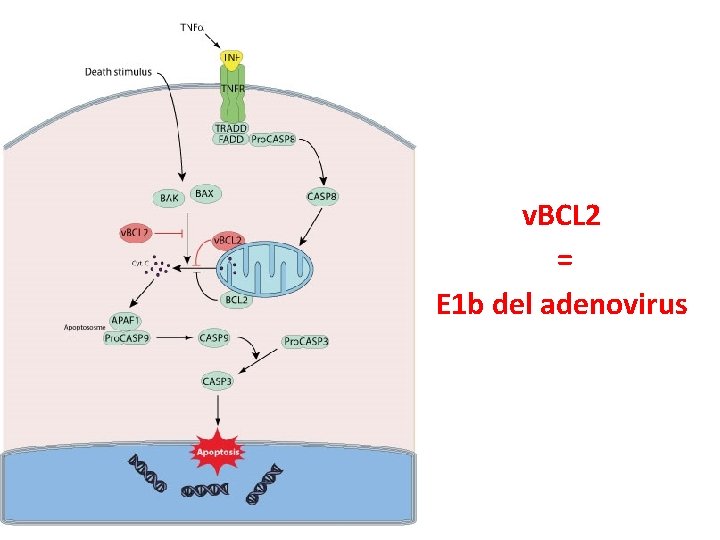
5. Regulación de la apoptosis • Inhibición de la apoptosis - Inhibición de la vía de las caspasas: Adenovirus (E 1 b), Vaccinia (Crm. A) - Dominio Bcl-2 homólogo: EBV (BHRF-1) - Interacción con dominios DED: HCV (core) - Desregulación del ciclo celular: HPV (E 6 y E 7), HBV (X) - Alteración del sistema del IFN o de sus moléculas regulatorias • Inducción de la apoptosis - HIV - Alteración de la actividad mitocondrial: Influenza (PB 1 -F 2)


v. BCL 2 = E 1 b del adenovirus


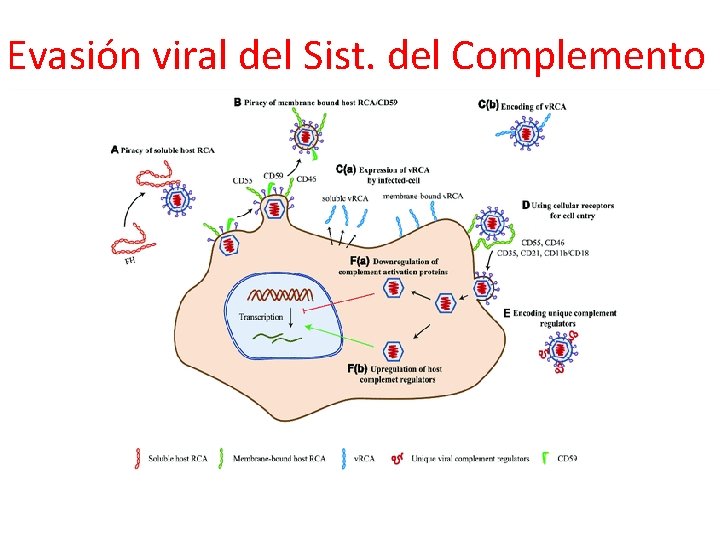
6. Modificación de la actividad del sistema del complemento • Proteína viral símil-componente del complemento HSV: gp. C se une a C 3 b e inhibe la cascada del complemento EBV: gp 340 (símil-C 3 b) se une a CD 21 e inhibe la cascada del complemento • Proteína viral que une a componentes del complemento HCV: el core se une al receptor del componente C 1 q. R en LT inhibiendo la respuesta de estas células

Evasión viral del Sist. del Complemento



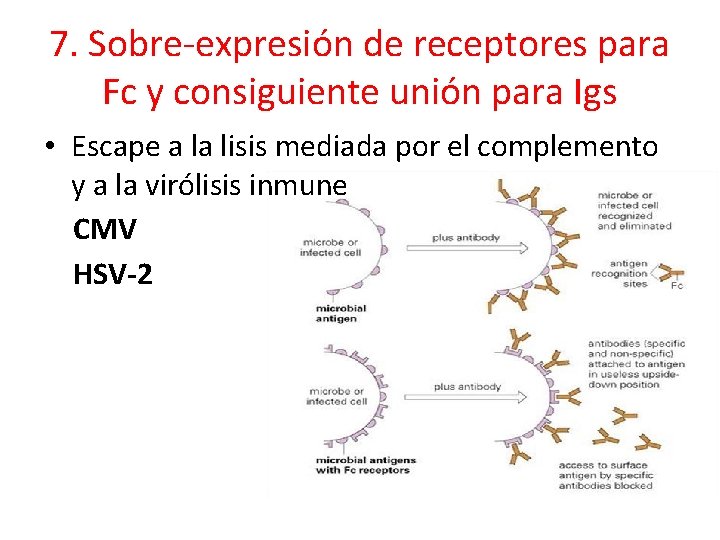
7. Sobre-expresión de receptores para Fc y consiguiente unión para Igs • Escape a la lisis mediada por el complemento y a la virólisis inmune CMV HSV-2

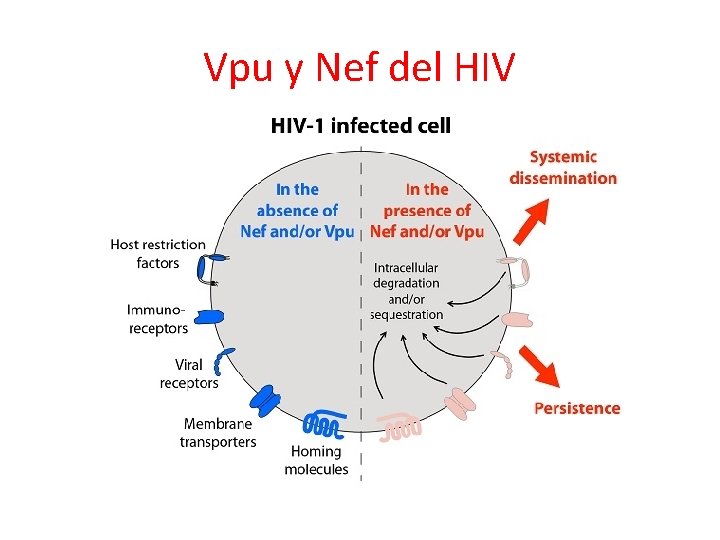
8. Interacción de proteínas virales con receptores/correceptores celulares • Inhibición de la expresión de CD 4 en la superficie celular - Aumento de la endocitosis de CD 4: HIV (Nef) - Degradación de CD 4: HIV (Vpu) • Unión de proteínas virales a CD 81 - En células NK: HCV (E 2) - En LT citotóxicos: HCV (E 2)

Vpu y Nef del HIV


 Deliberar para una convivencia democrática
Deliberar para una convivencia democrática Que es teatro
Que es teatro Adivinanzas sobre planetas
Adivinanzas sobre planetas Complemento indirecto exemplos
Complemento indirecto exemplos Complemento directo complemento indirecto
Complemento directo complemento indirecto Complemento indirecto español
Complemento indirecto español Tipo ii
Tipo ii Mac imunologia
Mac imunologia Sistema complemento
Sistema complemento Sistema de complemento
Sistema de complemento Inflammation ros
Inflammation ros Basophil markers
Basophil markers Vias de ativação do sistema complemento
Vias de ativação do sistema complemento Es la fe
Es la fe La respuesta del hombre a dios resumen
La respuesta del hombre a dios resumen Respuesta a palabra del señor
Respuesta a palabra del señor Respuesta del ejercicio
Respuesta del ejercicio Permeabilità intestinale
Permeabilità intestinale Predicato nominale
Predicato nominale Predicativi del soggetto e dell'oggetto
Predicativi del soggetto e dell'oggetto S adjetival
S adjetival Peto quaero
Peto quaero Sujeto eliptico
Sujeto eliptico Complemento del adjetivo
Complemento del adjetivo Los modificadores del predicado
Los modificadores del predicado Objeto directo ejemplos
Objeto directo ejemplos Funciones del complemento
Funciones del complemento Que es el complemento directo e indirecto
Que es el complemento directo e indirecto Complemento del nombre
Complemento del nombre Complemento circunstancial de regimen
Complemento circunstancial de regimen Immunofluorescenza indiretta
Immunofluorescenza indiretta Los complementos del sujeto
Los complementos del sujeto Placas continentales oceanicas y mixtas
Placas continentales oceanicas y mixtas Ap reproductor masculino
Ap reproductor masculino Tejidos linfoides
Tejidos linfoides Caracteristicas del sistema americano
Caracteristicas del sistema americano Sistema 1 sistema 2
Sistema 1 sistema 2 Sistema continuo y discreto
Sistema continuo y discreto Sistema semashko o sistema centralizado
Sistema semashko o sistema centralizado ¡inténtalo! elige la respuesta más lógica.
¡inténtalo! elige la respuesta más lógica. Dot respuesta
Dot respuesta Item de respuesta corta
Item de respuesta corta Gradiente dosis respuesta
Gradiente dosis respuesta Dot respuesta
Dot respuesta Respuesta pertinente
Respuesta pertinente 4 preguntas sin respuesta
4 preguntas sin respuesta Tienes una fruteria y te han repartido tres cajas respuesta
Tienes una fruteria y te han repartido tres cajas respuesta Tengo 3 manzanas si me quitas 2 cuantas tienes
Tengo 3 manzanas si me quitas 2 cuantas tienes La respuesta
La respuesta La respuesta
La respuesta Asertividad escalonada ejemplos
Asertividad escalonada ejemplos Verbo auxiliar do
Verbo auxiliar do Respuesta
Respuesta La respuesta
La respuesta Respuesta
Respuesta La respuesta
La respuesta Preguntas para miguel hidalgo
Preguntas para miguel hidalgo Curva de respuesta en frecuencia
Curva de respuesta en frecuencia La respuesta
La respuesta Preguntas sin respuesta
Preguntas sin respuesta Carotenoides
Carotenoides Respuesta
Respuesta Signos de flogosis
Signos de flogosis Respuesta
Respuesta Respuesta
Respuesta Resonante
Resonante Sistemas de segundo orden
Sistemas de segundo orden Respuesta
Respuesta Tres bellas que bellas son autor
Tres bellas que bellas son autor Estado estacionario
Estado estacionario Respuesta
Respuesta Carta remesa ejemplo
Carta remesa ejemplo Hiptesis
Hiptesis Costo de respuesta
Costo de respuesta Respuesta
Respuesta Respuesta
Respuesta Wordpad
Wordpad Vibraciones no amortiguadas
Vibraciones no amortiguadas Respuesta de
Respuesta de Ntp 399 señales de seguridad
Ntp 399 señales de seguridad Respuesta de amor
Respuesta de amor Cual es la respuesta
Cual es la respuesta Neuronas motoras
Neuronas motoras Tablas de multiplicar con respuesta
Tablas de multiplicar con respuesta Respuestas
Respuestas Respuesta
Respuesta