Profile HMMs Perfiles de Modelos ocultos de Markov


































- Slides: 38

Profile HMMs Perfiles de Modelos ocultos de Markov SONIA JIMÉNEZ GUTIÉRREZ JOSE CARLOS GARCÍA SERRANO I. T. I. SISTEMAS

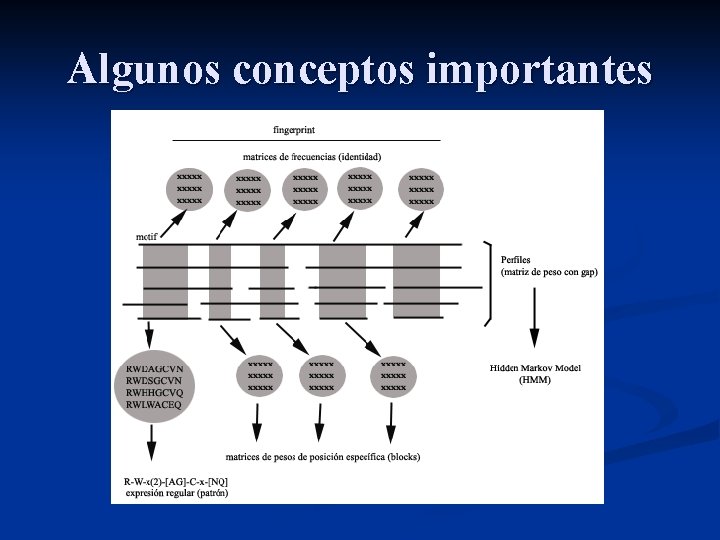
Algunos conceptos importantes n n n Surgen métodos de comparación de secuencias para la búsqueda de homologías como son los patrones, perfiles (conjunto alineado de secuencias que contiene un dominio) y HMM (modelos estadísticos de la estructura primaria de las secuencias). Motivo: si observamos un alineamiento múltiple de proteínas homólogas veremos que algunas columnas varían bastante, mientras que otras están más conservadas. Cuando observamos ciertas columnas cercanas con una alta conservación, es decir, cuando encontramos trocitos de las secuencias que se conservan más que otros y que podrían caracterizar funcionalmente a las proteínas, entonces solemos hablar de MOTIVOS. Dominio: el concepto de dominio define una unidad estructural independiente en las proteínas. Sin embargo se utiliza con cierta laxitud: por ejemplo, en estudios genéticos de deleción a veces se utiliza como sinónimo de la parte mínima de la secuencia capaz de realizar la función estudiada. En las bases de datos de dominios como PFam, un dominio se suele corresponder con el núcleo del dominio estructural, aquella zona más similar entre todas las proteínas de una familia, aunque no tiene por qué coincidir

Algunos conceptos importantes Existen distintos métodos para describir y localizar motivos: 1. Expresiones regulares o patrones: A partir de la información que contiene un alineamiento múltiple se obtiene un patrón o expresión regular utilizados para caracterizar motivos, indicando qué posiciones son más importantes y cuales pueden variar y que variaciones pueden sufrir. 2. Creación de perfiles: Es una matriz de sustitución específica para cada posición de la secuencia. A partir del alineamiento múltiple se construye dicha matriz teniendo en cuenta la frecuencia de los aminoácidos en cada posición así como sus propiedades fisicoquímicas.

Algunos conceptos importantes n Una diferencia entre los perfiles y las expresiones regulares o patrones es que no solo se limita a pequeñas regiones con un alto índice de similitud, sino que presenta una mayor utilidad a la hora de definir regiones o dominios más extensos que puedan caracterizar familias de proteínas más que motivos. El perfil puede cubrir tanto regiones conservadas como variables del alineamiento.

Algunos conceptos importantes

Algunos conceptos importantes 3. Perfiles HMMs: Se muestran como una forma más sensible, incluyendo los patrones reguladores y perfiles convencionales, de búsqueda de homólogos remotos y dominios conservados basados en una descripción estadística de la estructura primaria consenso de una familia de proteínas. n En el modelo HMMs que vamos a analizar consideramos tres estados posibles correspondientes a la probabilidad de encontrar en dicha posición un determinado residuo, la probabilidad de inserción y de deleción

Introducción n Los modelos de ocultos de Markov (HMM) surgieron como una herramienta aplicada al procesamiento del habla, un modelos estadístico que, a través de un algoritmo de aprendizaje, extraía las principales características estocásticas de una cadena de habla. Con la ingente cantidad de datos proveniente del secuenciamiento de distintos genomas, aparece un problema adjunto -> cómo extraer de estos datos la información subyacente. Solución: los HMM.

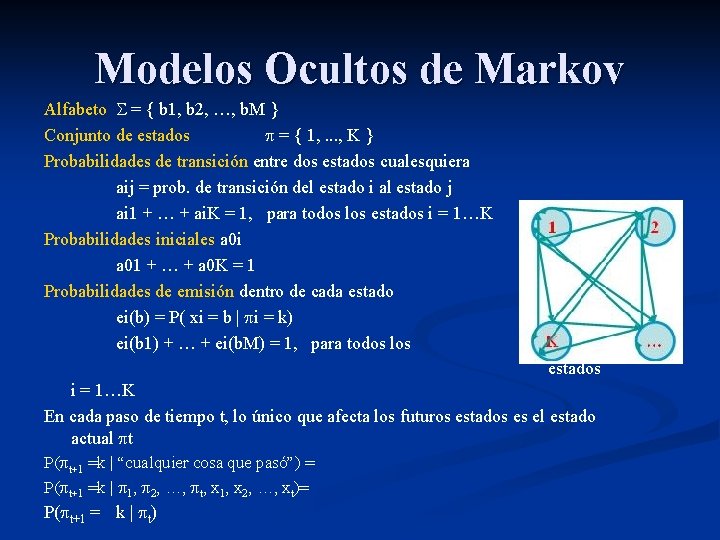
Modelos Ocultos de Markov Un modelo oculto de Markov (HMM) es un conjunto finito de estados. Las transiciones entre estados están dadas por un conjunto de probabilidades de transición. En cualquier estado particular, la observación puede ser generada, de acuerdo a la distribución de probabilidades de emisión. Sólo el resultado observable, no el estado, es visible a un observador externo por lo que los estados están “ocultos”.

Modelos Ocultos de Markov Alfabeto = { b 1, b 2, …, b. M } Conjunto de estados = { 1, . . . , K } Probabilidades de transición entre dos estados cualesquiera aij = prob. de transición del estado i al estado j ai 1 + … + ai. K = 1, para todos los estados i = 1…K Probabilidades iniciales a 0 i a 01 + … + a 0 K = 1 Probabilidades de emisión dentro de cada estado ei(b) = P( xi = b | i = k) ei(b 1) + … + ei(b. M) = 1, para todos los estados i = 1…K En cada paso de tiempo t, lo único que afecta los futuros estados es el estado actual t P( t+1 =k | “cualquier cosa que pasó”) = P( t+1 =k | 1, 2, …, t, x 1, x 2, …, xt)= P( t+1 = k | t)

Las 3 grandes preguntas sobre HMM n n n Evaluación Dado un HMM M y una secuencia x, encontrar Prob[ x|M] Decodificación Dado un HMM M, y una secuencia x, encontrar la secuencia de estados que maximiza P[ x, | M ] Aprendizaje Dado un HMM M, con probabilidad transición/emisión desconocidas, y una secuencia x, encontrar los parámetros = (ei(. ), aij) que maximizan P[ x | ]

Decodificación n Dada una secuencia de observaciones X, encuentre la secuencia de est. . 1 1 … DNA coding (C) vs non-coding (N) x = …AACCTTCCGCGCAATATAGGTAACCCCGG… = …NNCCCCCCCCCNNNN… 2 2 2 … … … K K K … … Queremos encontrar = 1, ……, N, tal que P[ x, ] esté maximizado x x 2 x 3 * 1 = argmax P[ x, ] Podemos usar programación dinámica Sea Vk(i) = max{ 1, …, i-1} P[x 1…xi-1, 1, …, i-1, xi, i = k] = Probabilidad de la secuencia de estados más verosímil que termina en el estado i = k 2 … K x. K

Algoritmo de Viterbi Es similar a “alinear” un conjunto de estados de una secuencia. Complejidad temporal: O(K 2 N) K=nº estados Complejidad espacial: O(KN) N=longitud

Agoritmos de Viterbi y Forward VITERBI Inicialización: V 0(0) = 1 Vk(0) = 0, para todo k > 0 FORWARD Inicialización: f 0(0) = 1 fk(0) = 0, para todo k > 0 Iteración: Vj(i) = ej(xi) maxk Vk(i-1) akj Iteración: fl(i) = el(xi) k fk(i-1) akl Terminación: P(x, *) = maxk Vk(N) Terminación P(x) = k fk(N) ak 0

Algoritmos de entrenamiento n n n Tenemos un conjunto de secuencias de ejemplo del tipo de las queremos que el modelo ajuste (secuencias de entrenamiento), que suponemos independientes. Si conociéramos el camino de estados que recorrió el modelo, los estados no están ocultos (el HMM se transforma en una cadena de Markov), en la cual los estimadores de máximoa verosimilitud para las frecuencias de emisión y transición se obtienen a partir de las frecuencias de observaciones. Si tenemos información (biológica o física) que nos aporte información previa a la distribución de probabilidades podemos agregársela al modelo como pseudocuentas.

Algoritmos de entrenamiento n n n Objetivo: Dada una secuencia de observaciones, encontrar el modelo más probable que genere esa secuencia Problema: No conocemos las frecuencias relativas de los estados ocultos visitados. No se conocen soluciones analíticas Nos acercamos a la solución por sucesivas aproximaciones. El problema ahora es la optimización, por lo que se pueden usar muchas heurísticas (simulated annealing, algoritmos genéticos, etc)

Algoritmo de Baum-Welch n n n Este es el algoritmo de Expectation-Maximization (EM) para la estimación de parámetros. Aplicable a cualquier proceso estocástico Encuentra las frecuencias esperadas de los posibles valores de las variables ocultas. Calcula las distribuciones de máxima verosimilitud de las variables ocultas en base a las probabilidades forward y backward. Repite estos pasos hasta satisfacer algún criterio de convergencia. Complejidad temporal: nº iteraciones*O(N 2 T)

Aplicaciones de los HMM n n Los modelos probabilísticos están tomando una mayor importancia en el análisis biológico, particularmente en problemas de análisis con muchos parámetros. Puesto que muchos problemas en biología computacional se reducen al análisis de secuencias lineales cortas, los modelos basados en HMM han sido aplicados a muchos problemas Búsqueda de genes, mapas híbridos de radiación, unión de mapas genéticos, análisis filogenético y predicción de la estructura secundaria de las proteínas. Las aplicaciones más exitosas son los perfiles HMM y HMM-based gene finders.

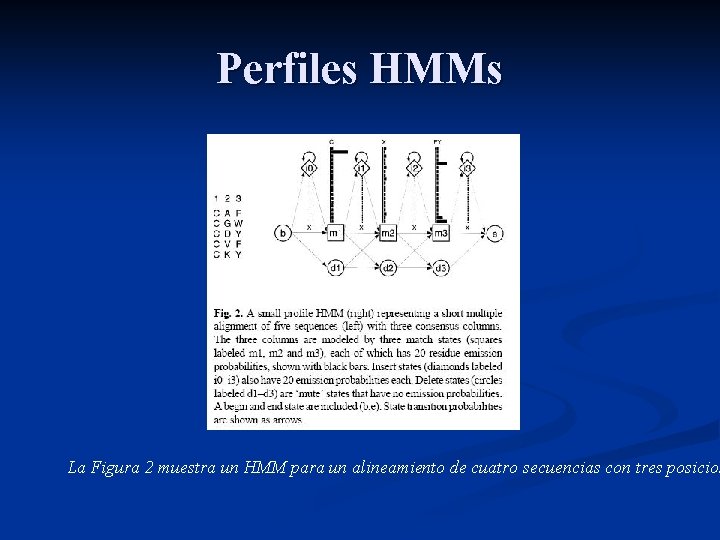
Perfiles HMMs n n n A partir de un HMM entrenado con un conjunto de secuencias previamente alineadas (CLUSTAW) se puede obtener las características estocásticas (profile) de una familia de secuencias de ADN o proteínas. En las proteínas se observan regiones de longitud considerable donde no participan gaps ni inserciones de residuos. Se puede construir un modelo donde sólo participen los estados de match, con probabilidad 1 de transición entre un estado y el siguiente y con probabilidades de emisión de residuos calculadas a partir de su frecuencia de aparición.

Perfiles HMMs n n n En M 1 se emiten los símbolos de los aminoácidos (A 1. . Al) con las probabilidades de emisión que resultan de la frecuencia de aparición de éstos en la columna 1 de las secuencias presentadas como datos Se fuerzan a 1 las probabilidades de transición entre un estado y el siguiente. En las secuencias de aminoácidos se observan porciones donde es posible hallar consenso (estados de match) y otras donde o bien aparecen insert o gaps (estados delete).

Perfiles HMMs La Figura 2 muestra un HMM para un alineamiento de cuatro secuencias con tres posicion

SOFTWARE PARA PERFILES HMM Hay múltiples paquetes de software que están disponibles para implementar perfiles HMM:

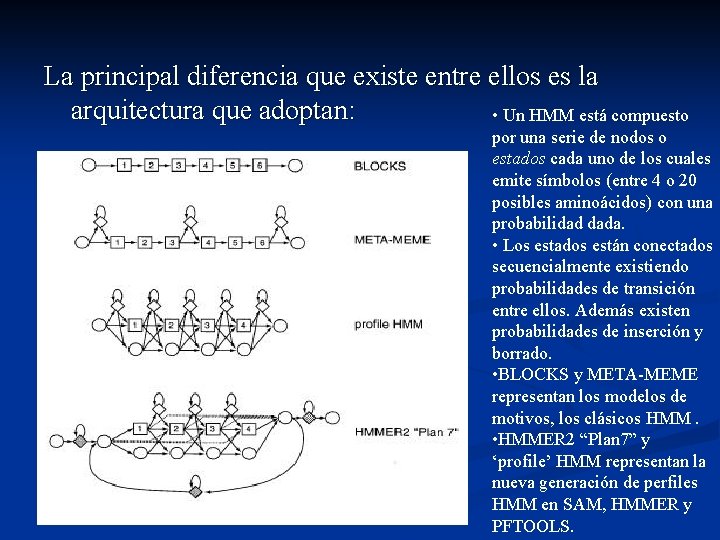
La principal diferencia que existe entre ellos es la arquitectura que adoptan: • Un HMM está compuesto por una serie de nodos o estados cada uno de los cuales emite símbolos (entre 4 o 20 posibles aminoácidos) con una probabilidad dada. • Los estados están conectados secuencialmente existiendo probabilidades de transición entre ellos. Además existen probabilidades de inserción y borrado. • BLOCKS y META-MEME representan los modelos de motivos, los clásicos HMM. • HMMER 2 “Plan 7” y ‘profile’ HMM representan la nueva generación de perfiles HMM en SAM, HMMER y PFTOOLS.

Hay dos modelos diferenciados para el autor: n n Modelos de perfiles: modelos con estados de inserción y borrado asociados con cada estado encontrado, permitiendo inserciones y borrados en la secuencia seleccionada. Modelos de motivos: modelos dominados por cadenas de estados encontrados (modelando bloques sin huecos de secuencias consenso), separados por un pequeño número de estados insertados modelando los espacios entre los bloques sin huecos.

n n n SAM, HMMER, PFTOOLS y HMMpro implementan modelos basados al menos en una parte en los perfiles originales HMM de Krogh (1994). Estos paquetes están argumentados en un simple modelo que trata con múltiples dominios, secuencias alineadas y alineamientos locales. El alineamiento local o global no es necesariamente esencial en el algoritmo, pero esto demuestra que la probabilística es una parte del modelo de arquitectura.

Programas que utilizan HMM

SAM y HMMER Usan mezclas Dirichet en muchas distribuciones para ayudar al numero de parámetro libres. Si adoptan el hibrido HMM/neural network techniques esto se acentúa. n HMMER y PFTOOLS Son usados en primer lugar para construir bases de datos de búsqueda de modelos donde están presentes los alineamientos. n PROBE, META-MEME y BLOCKS Asumen distintos modelos de motivos, los alineamientos consisten en uno o mas bloques sin huecos, separados por secuencias ‘intervening’ que son asumidas para ser aleatorias. PROBE y METAMEME adoptan modelos probabilísticos para los huecos. n

GENEWISE Es una sofisticada aplicación de búsqueda por ventanas que puede tomar un HMMER de modelo de proteína. n PSI-BLAST No es una aplicación HMM, pero usa los principios de los modelos probabilísticos para construir ‘HMMlike models’ para múltiples alineamientos. n

LIBRERIAS PARA PERFILES HMM El software para perfiles HMM esta bien para: n n Modelar una secuencia en particular de una familia de interés. Buscar secuencias homologas en una base de datos. Ahora necesitamos buscar una secuencia simple en una librería de perfiles HMM. Construir una librería requiere un largo número de múltiples alineamientos de comunes dominios.

Dos largas colecciones de perfiles HMMs están disponibles: n Pfam n PROSITE Ambas bases de datos están disponibles en la web:

Pfam Es una base de datos compuesta por los perfiles HMMs obtenidos para distintos dominios o regiones conservadas de proteínas. Contiene múltiples alineamientos de proteínas y perfiles-HMMs de esas familias de proteínas. Es una base de datos semiautomática, cuyo objetivo es ser completa y exacta. n PROSITE Es una base de datos que contiene información detallada sobre todos los motivos de secuencia de proteína conocidos. Los motivos son descritos mediante patrones regulares. n

n n n PROSITE contiene perfiles para 290 dominios de proteínas, y Pfam contiene 1313. Hay muchas discusiones sobre el número de familias de proteínas que hay, el número de 1000 fue citado en alguna ocasión, otros defienden que todas las familias tienen aproximadamente el mismo número. Ninguno de estos servidores de perfiles están maduros, ambas bases de datos para perfiles software están rápidamente cambiando.

BLOCKS Base de datos compuesta por perfiles HMMs obtenidos para distintos dominios o regiones conservadas en las proteínas. El método HMMs también es utilizado Es una base de datos compuesta por pequeños segmentos de alineamientos múltiples correspondientes a entradas en PROSITE. De hecho BLOCKS es un sistema de detección de motivos más que una base de datos propiamente dicha. n PRODOM Es una BD de dominios de proteínas generado automáticamente desde SWISS-PROT y Tr. EMBL, consiste en una compilación automática de dominios homólogos. Construido utilizando un procedimiento mejorado basado en PSI-BLAST. n

n PRINTS La base de datos PRINTS es similar en concepto, pero se usa para descargar bloques llamados "huellas dactilares", fingerprints. Ha sido recientemente incluido como un servicio on-line de BLAST y un software de búsqueda, proporcionando mayor eficacia y mejora estadística para la estimación de la seguridad de las parejas recuperadas. Es un grupo de motivos conservados para caracterizar una familia de proteínas.

HMMs PARA RECONOCIMIENTO DE PLIEGUES Los scores de los perfiles son calculados con estructuras de datos en lugar de secuencias. Ej. ‘ 3 D/1 D profiles’. Di Francesco usó perfiles HMM para modelar la segunda estructura de secuencias, modificando el SAM code para emitir un alfabeto de estructura secundaria de proteína. ¿Como puede HMM asumir su posición de independiente y esperar ser un modelo realista de estructura de proteína?

Algunos métodos de reconocimiento utilizan algoritmos de programación dinámica, que son usados para encontrar la secuencia/estructura optima alineada. La sección de reconocimiento de CASP (Current Assement of Struccture Prediction) es uno de los mas interesantes métodos de reconocimiento de cómo HMMs se desarrollan. En CASP las secuencias de proteínas son solventadas a través de criptografía o NMR, que esta disponible para grupos computacionales de predicción de estructuras.

CONCLUSIÓN n n El proyecto del genoma humano amenaza con abrumarnos en un diluvio de secuencias de datos. Las populares anotaciones de largas secuencias son muy difíciles para muchas personas. El desarrollo de métodos robustos para automatizar la clasificación y anotación de secuencias es imperativo. Surge la esperanza de que desarrollando métodos de perfiles HMM, se pueda suministrar una segunda lista de éstos que sean sólidos, sensatos y estadísticamente basados en herramientas de análisis, que completen los análisis BLAST y FASTA.

n n La combinación del poder del software HMM y las largas secuencias alineadas en bases de datos para conservar dominios de proteínas, debería de hacer de esta esperanza una realidad. Los modelos ocultos de Markov (HMM) proporcionan una herramienta para la modelización de secuencias de ADN, ARN y proteínas, el descubrimiento de zonas de secuencias cuyas propiedades estadísticas son distintas a las esperadas por azar (background) como es el caso de posibles genes y actualmente están empezando a ser utilizados para modelización de estructuras tridimensionales.

n Computacionalmente tienen un costo aceptable, O(MT), comparados con los algoritmos de búsqueda y alineación (múltiple) y una potencia ya comprobada en toda la teoría de procesamiento del habla. Existen algoritmos de entrenamiento para los HMM como Viterbi y Baum-Welch, ampliamente utilizados que convergen en aproximadamente 10 a 15 iteraciones para la modelización de proteínas o ADN. También es posible encontrar paquetes públicos para la aplicación de HMM y bases de datos de profile HMM como Pfam.