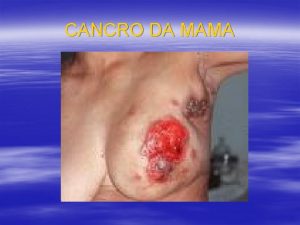
Oncogeni ed Oncosoppressori Cosa causa il cancro Cellula
































- Slides: 83

Oncogeni ed Oncosoppressori

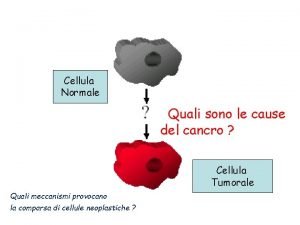
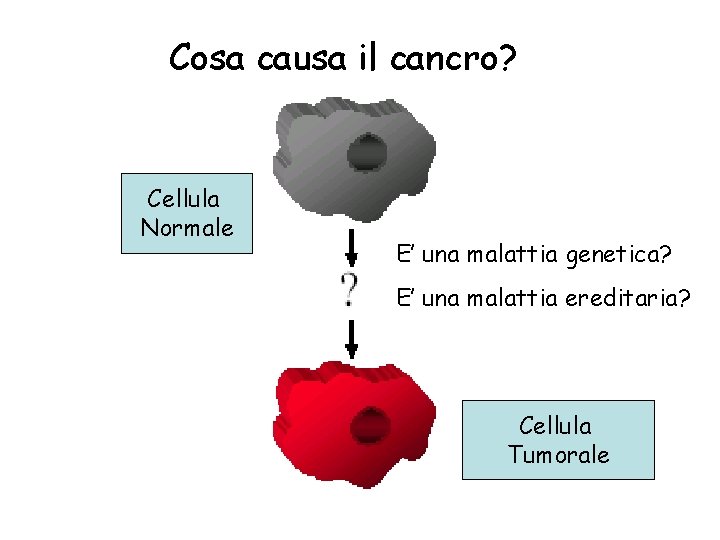
Cosa causa il cancro? Cellula Normale E’ una malattia genetica? E’ una malattia ereditaria? Cellula Tumorale

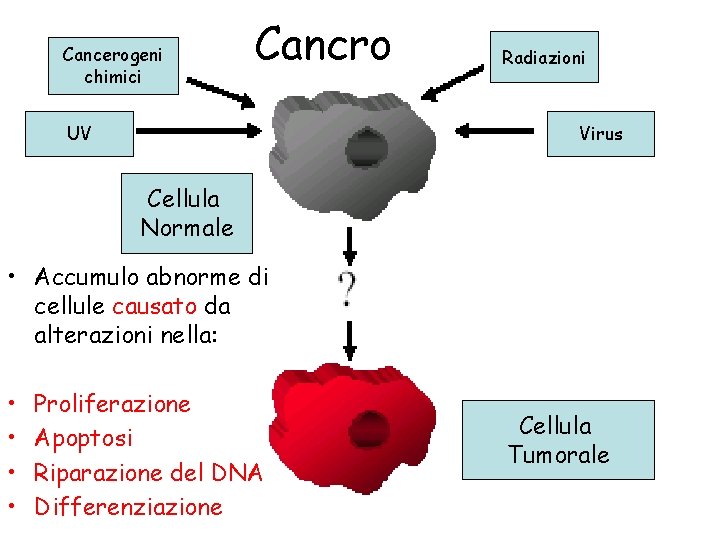
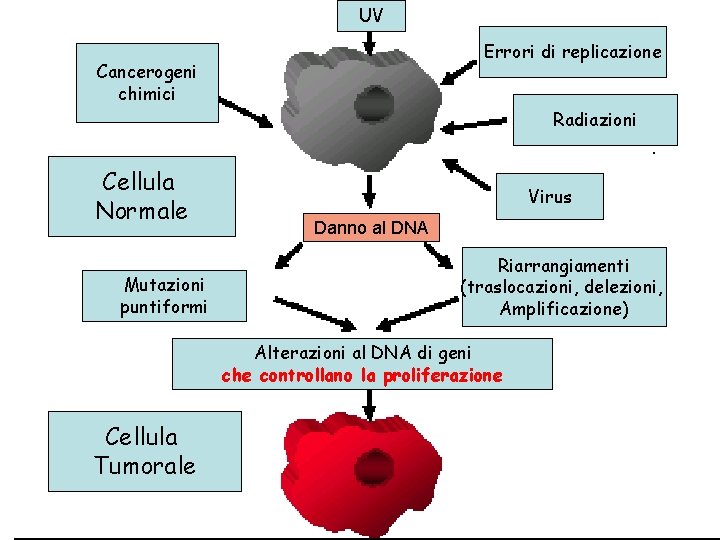
Cancerogeni chimici Cancro UV Radiazioni Virus Cellula Normale • Accumulo abnorme di cellule causato da alterazioni nella: • • Proliferazione Apoptosi Riparazione del DNA Differenziazione Cellula Tumorale

Alterata proliferazione cellulare e cancro • Ogni cellula contiene ~ 30 -40, 000 geni • Alcuni geni sono responsabili del controllo della proliferazione cellulare • Se in questi geni avviene una mutazione, la proliferazione diventa anomala • L’anomala proliferazione è una caratteristica del cancro

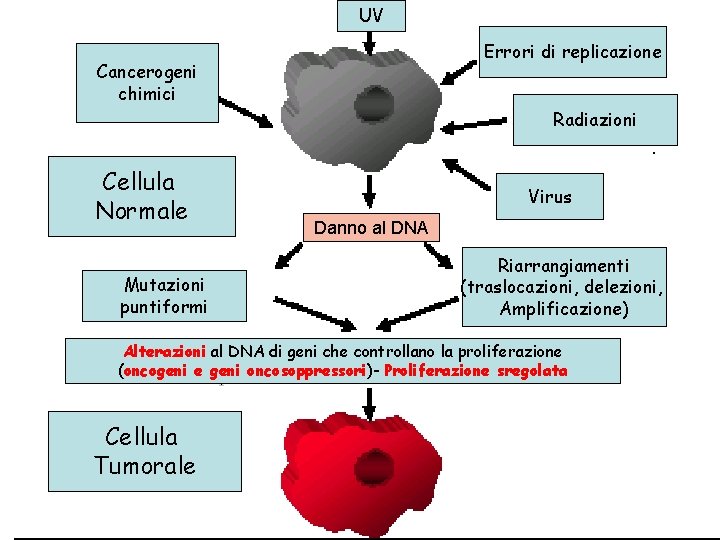
UV Errori di replicazione Cancerogeni chimici Radiazioni Cellula Normale Mutazioni puntiformi Virus Danno al DNA Riarrangiamenti (traslocazioni, delezioni, Amplificazione) Alterazioni al DNA di geni che controllano la proliferazione Cellula Tumorale

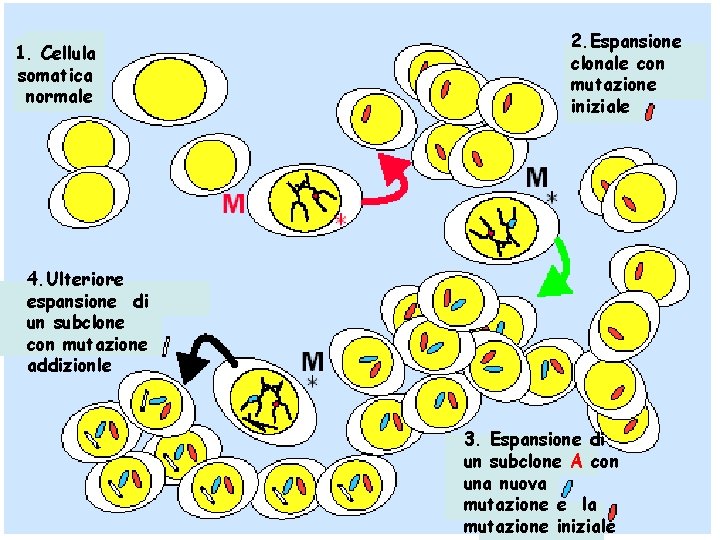
1. Cellula somatica normale 2. Espansione clonale con mutazione iniziale 4. Ulteriore espansione di un subclone con mutazione addizionle 3. Espansione di un subclone A con una nuova mutazione e la mutazione iniziale

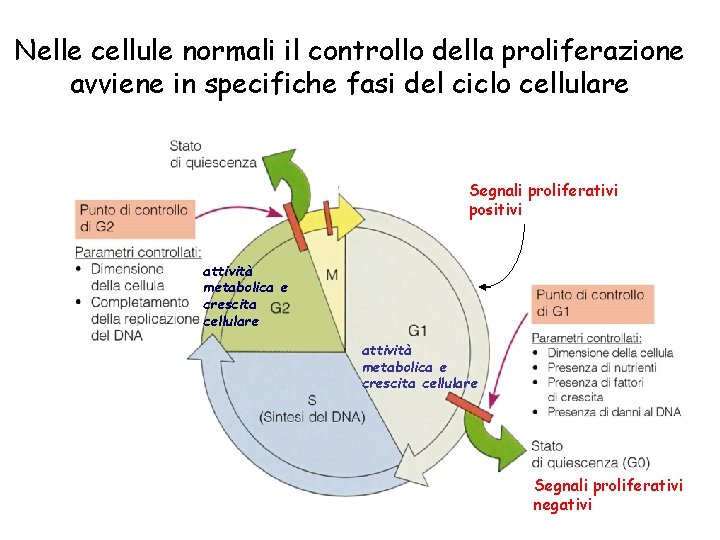
Nelle cellule normali il controllo della proliferazione avviene in specifiche fasi del ciclo cellulare Segnali proliferativi positivi attività metabolica e crescita cellulare Segnali proliferativi negativi

Come è controllata la proliferazione cellulare? • Il ciclo cellulare e la divisione cellulare sono controllati dalla regolata espressione di geni • L’integrità genomica di tutte le cellule dipende dall’attenta attività dei geni riparativi soprattutto in cellule più esposte all’azione di genotossici • Coinvolge l’equilibrio tra l’espressione di geni responsabili per la regolazione positiva della proliferazione e l’espressione di geni responsabili per la regolazione negativa della proliferazione

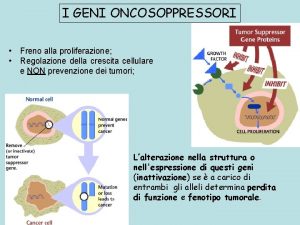
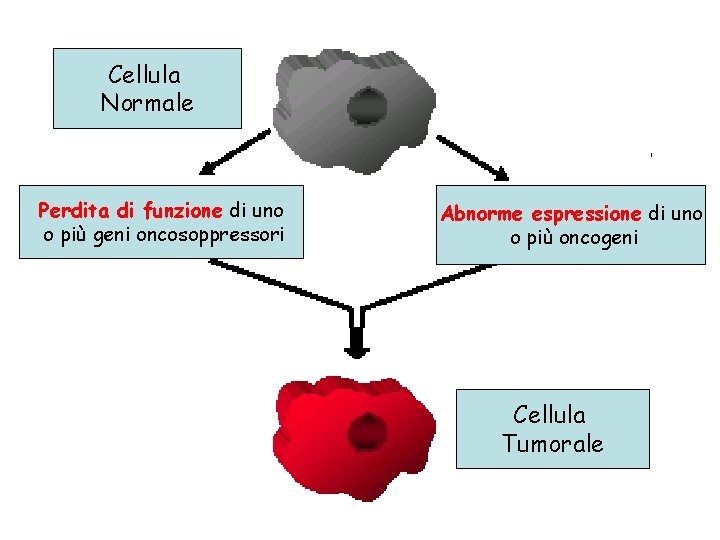
Cellula Normale Perdita di funzione di uno o più geni oncosoppressori Abnorme espressione di uno o più oncogeni Cellula Tumorale

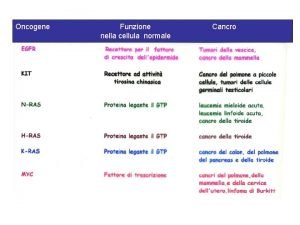
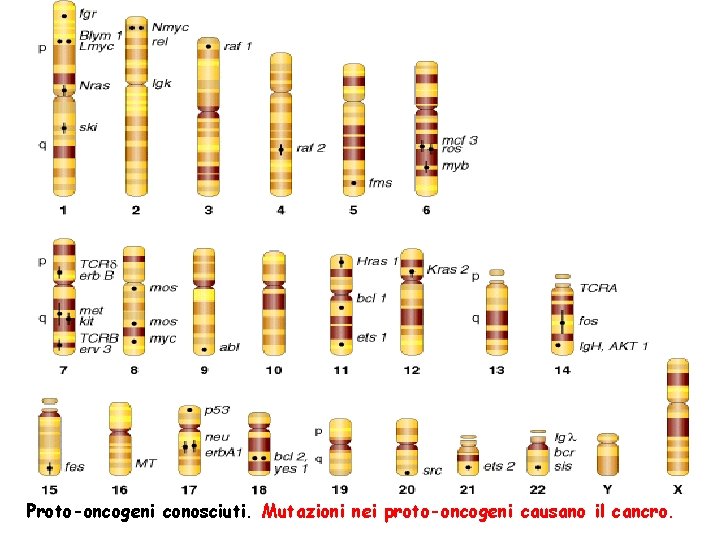
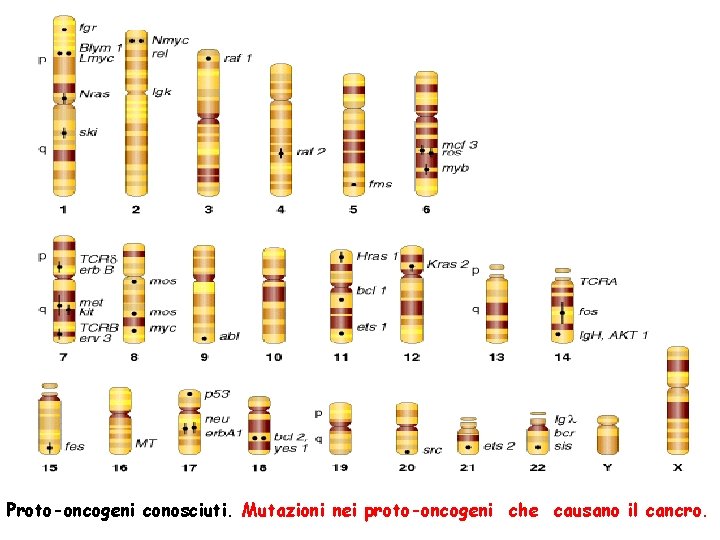
Proto-oncogeni conosciuti. Mutazioni nei proto-oncogeni causano il cancro.

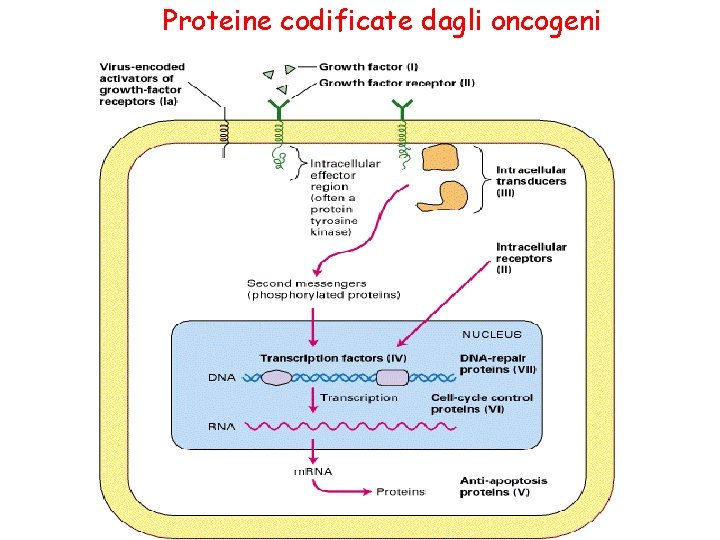
Proteine codificate dagli oncogeni

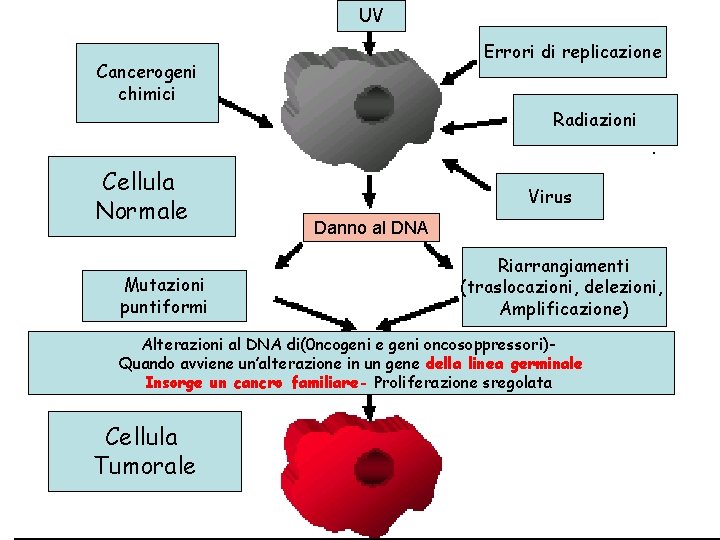
UV Errori di replicazione Cancerogeni chimici Radiazioni Cellula Normale Mutazioni puntiformi Virus Danno al DNA Riarrangiamenti (traslocazioni, delezioni, Amplificazione) Alterazioni al DNA di geni che controllano la proliferazione (oncogeni e geni oncosoppressori)- Proliferazione sregolata Cellula Tumorale

La scoperta dei “geni del cancro” nasce da due osservazioni (fine anni ‘ 70) • Sequenze geniche di retrovirus oncògeni (v-onc) sono omologhe a sequenze di normali geni cellulari (c-onc) da questi “catturati” • Segmenti genici estratti da tumori trasformano cellule normali in cellule neoplastiche

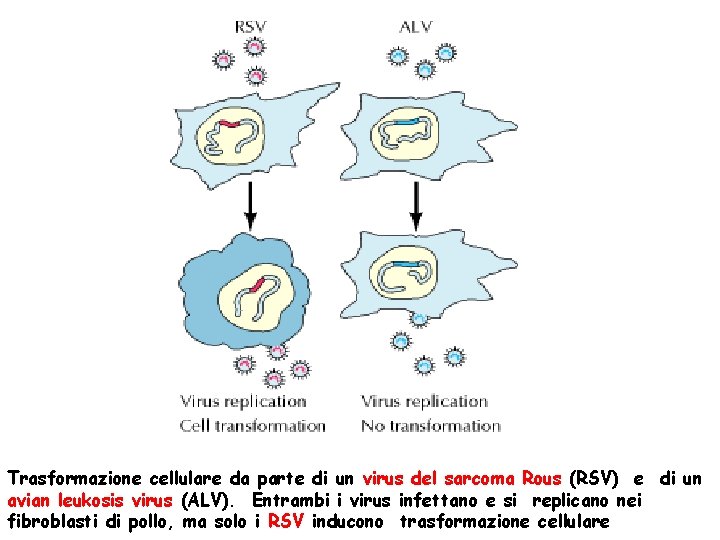
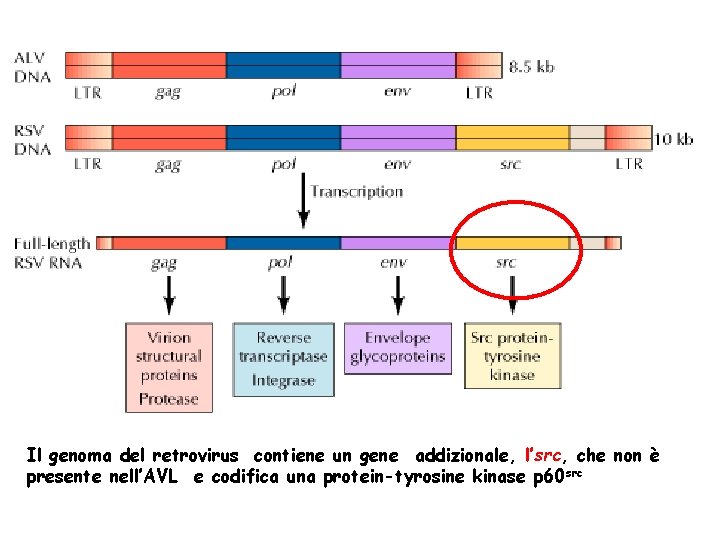
Trasformazione cellulare da parte di un virus del sarcoma Rous (RSV) e di un avian leukosis virus (ALV). Entrambi i virus infettano e si replicano nei fibroblasti di pollo, ma solo i RSV inducono trasformazione cellulare

Il genoma del retrovirus contiene un gene addizionale, l’src, che non è presente nell’AVL e codifica una protein-tyrosine kinase p 60 src

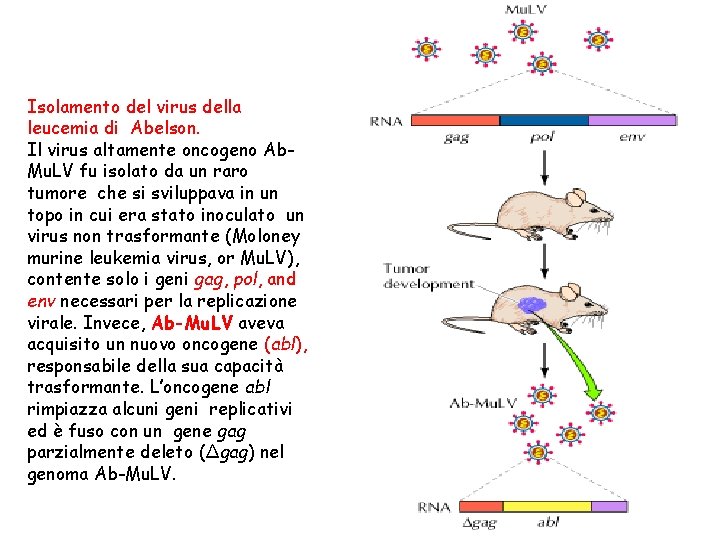
Isolamento del virus della leucemia di Abelson. Il virus altamente oncogeno Ab. Mu. LV fu isolato da un raro tumore che si sviluppava in un topo in cui era stato inoculato un virus non trasformante (Moloney murine leukemia virus, or Mu. LV), contente solo i geni gag, pol, and env necessari per la replicazione virale. Invece, Ab-Mu. LV aveva acquisito un nuovo oncogene (abl), responsabile della sua capacità trasformante. L’oncogene abl rimpiazza alcuni geni replicativi ed è fuso con un gene gag parzialmente deleto (Δgag) nel genoma Ab-Mu. LV.

Questo scenario suggeriva l’ipotesi che gli oncogeni retrovirali derivano da geni della cellula ospite, e che occasionalmente tale gene cellulare viene incorporato in un genoma virale, producendo un nuovo virus altamente oncogenico come prodotto di un evento di ricombinazione virus-ospite

Le cellule normali contengono geni che sono strettamente correlati agli oncogeni retrovirali

• I geni delle cellule normali da cui originano gli oncogeni retrovirali sono i proto-oncogeni. Sono importanti geni regolatori, codificanti proteine coinvolte nei pathways di trasduzione del segnale che controllano la normale proliferazione cellulare (es. : src, ras, and raf). • Gli oncogeni sono espressi in maniera abnorme o sono forme mutate dei corrispondenti proto -oncogeni. • Come conseguenza di tali alterazioni gli oncogeni inducono l’ abnorme proliferazione cellulare e lo sviluppo tumorale.

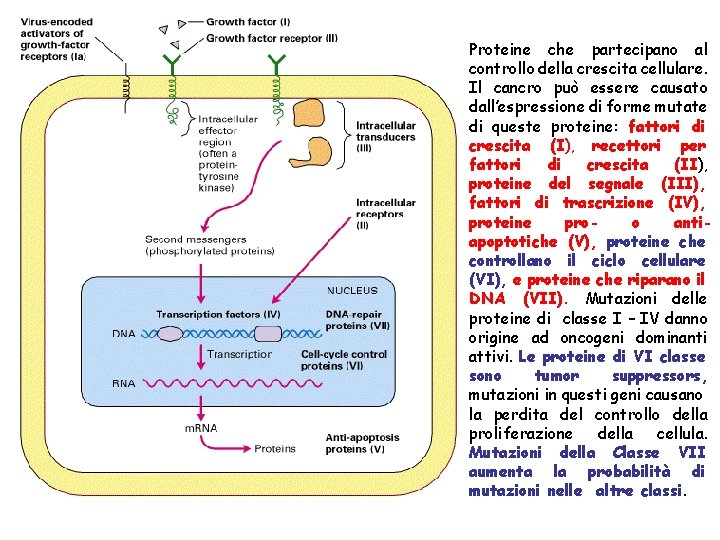
Proteine che partecipano al controllo della crescita cellulare. Il cancro può essere causato dall’espressione di forme mutate di queste proteine: fattori di crescita (I), recettori per fattori di crescita (II), proteine del segnale (III), fattori di trascrizione (IV), proteine proo antiapoptotiche (V), proteine che controllano il ciclo cellulare (VI), e proteine che riparano il DNA (VII). Mutazioni delle proteine di classe I – IV danno origine ad oncogeni dominanti attivi. Le proteine di VI classe sono tumor suppressors, mutazioni in questi geni causano la perdita del controllo della proliferazione della cellula. Mutazioni della Classe VII aumenta la probabilità di mutazioni nelle altre classi.

Proto-oncogeni conosciuti. Mutazioni nei proto-oncogeni che causano il cancro.

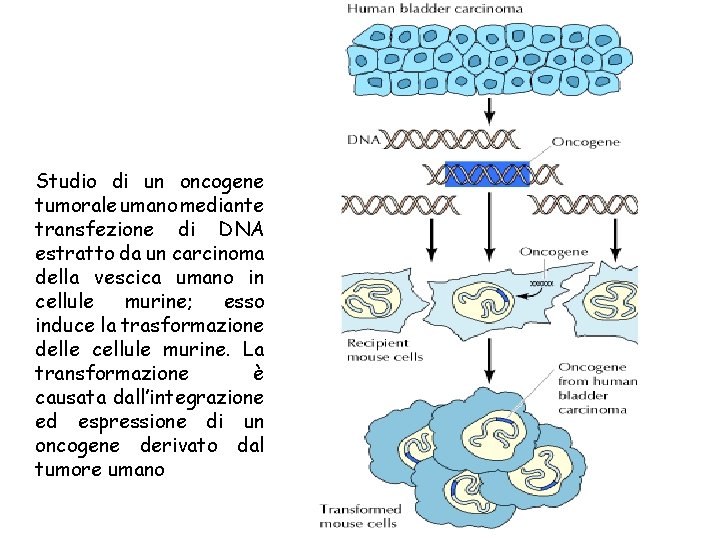
Studio di un oncogene tumorale umano mediante transfezione di DNA estratto da un carcinoma della vescica umano in cellule murine; esso induce la trasformazione delle cellule murine. La transformazione è causata dall’integrazione ed espressione di un oncogene derivato dal tumore umano

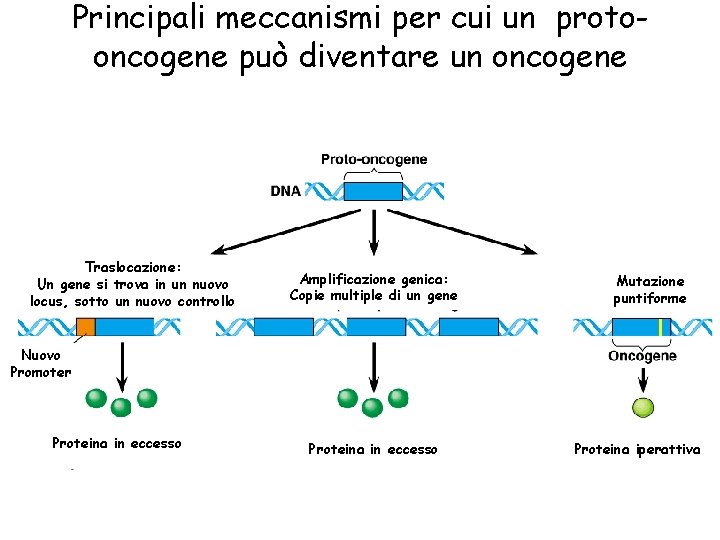
Principali meccanismi per cui un protooncogene può diventare un oncogene Traslocazione: Un gene si trova in un nuovo locus, sotto un nuovo controllo Amplificazione genica: Copie multiple di un gene Mutazione puntiforme Nuovo Promoter Proteina in eccesso Proteina iperattiva

Attivazione di un oncogene per mutazione puntiforme

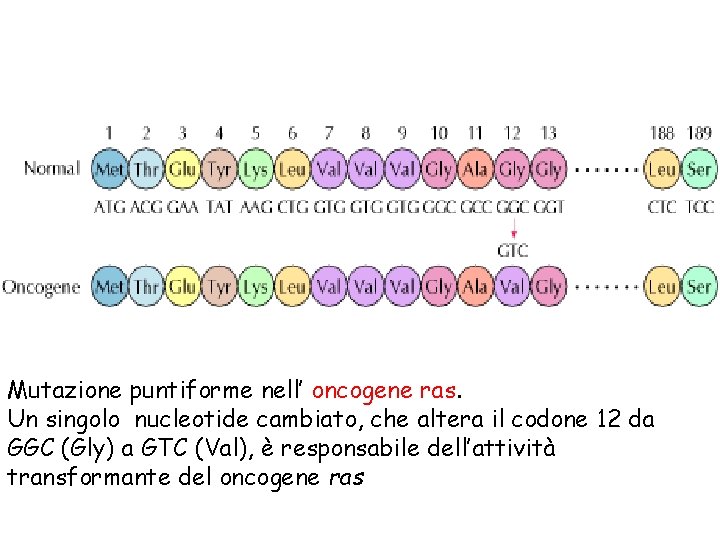
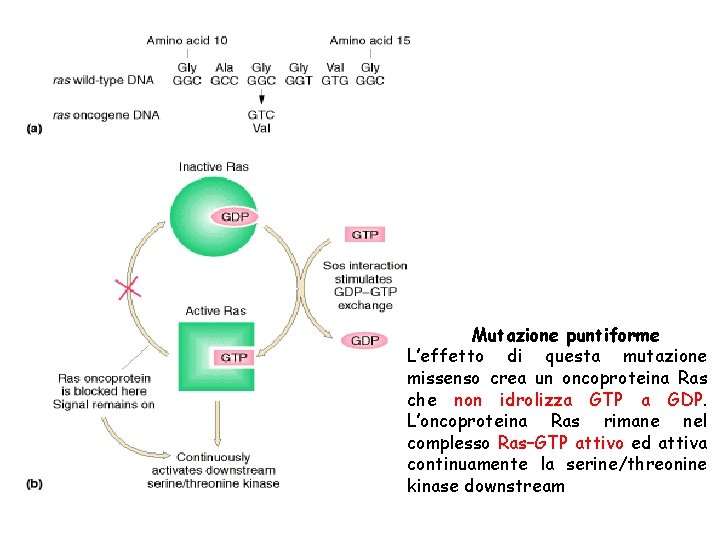
Mutazione puntiforme nell’ oncogene ras. Un singolo nucleotide cambiato, che altera il codone 12 da GGC (Gly) a GTC (Val), è responsabile dell’attività transformante del oncogene ras


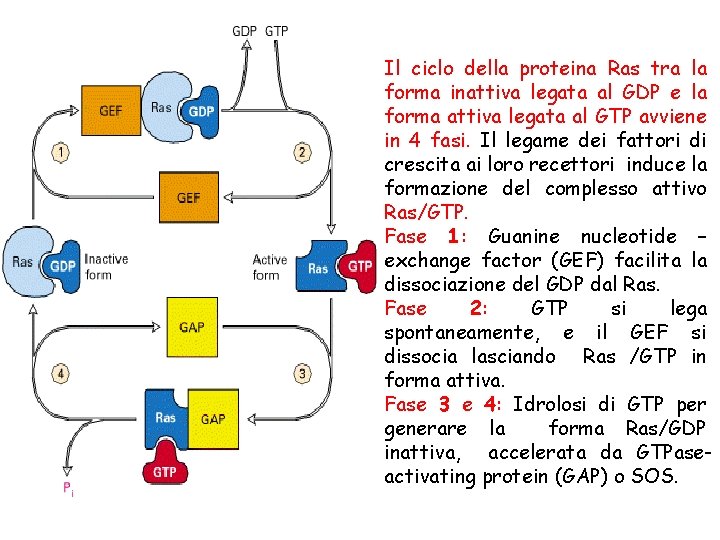
Il ciclo della proteina Ras tra la forma inattiva legata al GDP e la forma attiva legata al GTP avviene in 4 fasi. Il legame dei fattori di crescita ai loro recettori induce la formazione del complesso attivo Ras/GTP. Fase 1: Guanine nucleotide – exchange factor (GEF) facilita la dissociazione del GDP dal Ras. Fase 2: GTP si lega spontaneamente, e il GEF si dissocia lasciando Ras /GTP in forma attiva. Fase 3 e 4: Idrolosi di GTP per generare la forma Ras/GDP inattiva, accelerata da GTPaseactivating protein (GAP) o SOS.

Mutazione puntiforme L’effetto di questa mutazione missenso crea un oncoproteina Ras che non idrolizza GTP a GDP. L’oncoproteina Ras rimane nel complesso Ras–GTP attivo ed attiva continuamente la serine/threonine kinase downstream

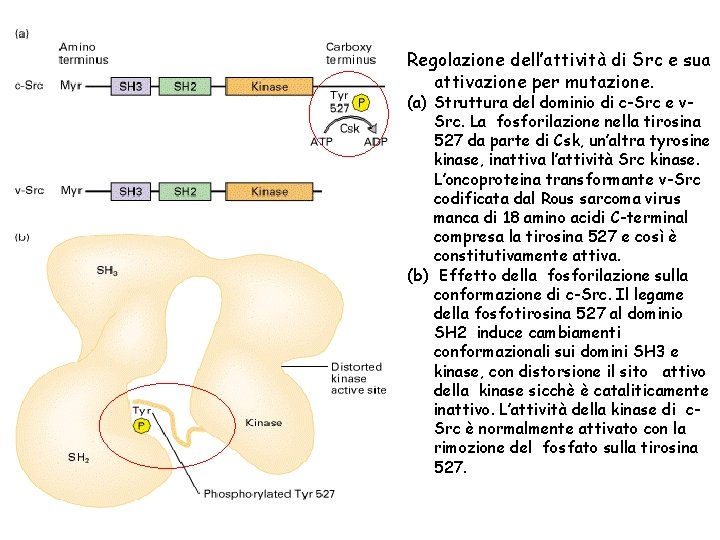
Regolazione dell’attività di Src e sua attivazione per mutazione. (a) Struttura del dominio di c-Src e v. Src. La fosforilazione nella tirosina 527 da parte di Csk, un’altra tyrosine kinase, inattiva l’attività Src kinase. L’oncoproteina transformante v-Src codificata dal Rous sarcoma virus manca di 18 amino acidi C-terminal compresa la tirosina 527 e così è constitutivamente attiva. (b) Effetto della fosforilazione sulla conformazione di c-Src. Il legame della fosfotirosina 527 al dominio SH 2 induce cambiamenti conformazionali sui domini SH 3 e kinase, con distorsione il sito attivo della kinase sicchè è cataliticamente inattivo. L’attività della kinase di c. Src è normalmente attivato con la rimozione del fosfato sulla tirosina 527.

Attivazione di un oncogene per delezione

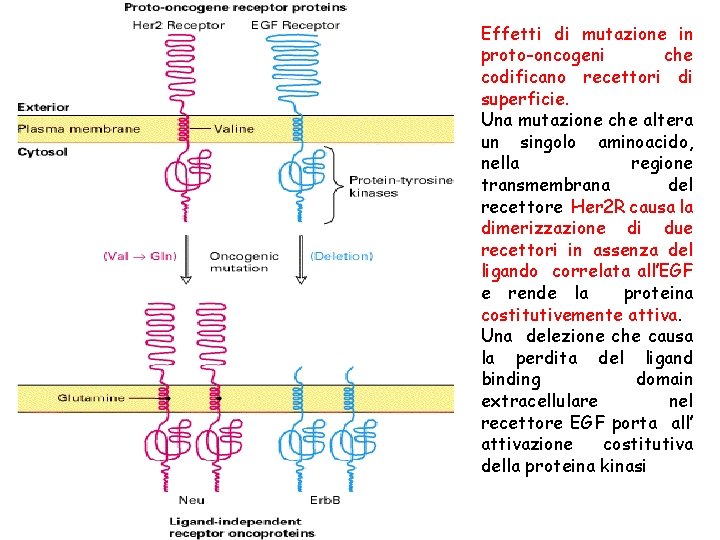
Effetti di mutazione in proto-oncogeni che codificano recettori di superficie. Una mutazione che altera un singolo aminoacido, nella regione transmembrana del recettore Her 2 R causa la dimerizzazione di due recettori in assenza del ligando correlata all’EGF e rende la proteina costitutivemente attiva. Una delezione che causa la perdita del ligand binding domain extracellulare nel recettore EGF porta all’ attivazione costitutiva della proteina kinasi

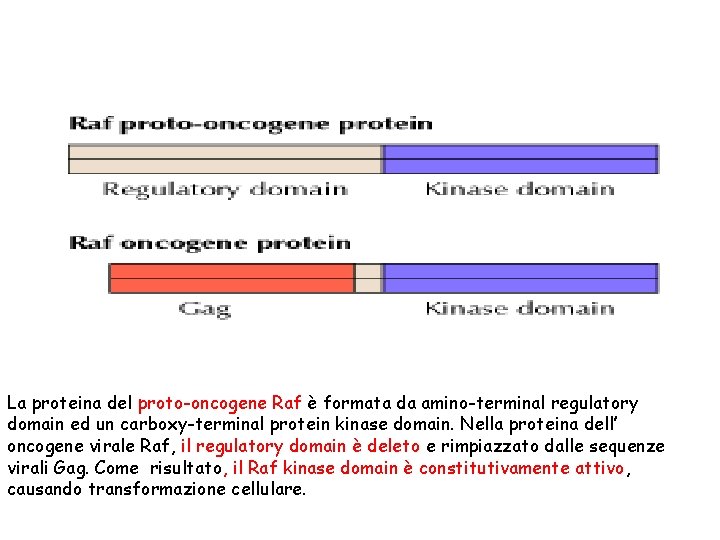
La proteina del proto-oncogene Raf è formata da amino-terminal regulatory domain ed un carboxy-terminal protein kinase domain. Nella proteina dell’ oncogene virale Raf, il regulatory domain è deleto e rimpiazzato dalle sequenze virali Gag. Come risultato, il Raf kinase domain è constitutivamente attivo, causando transformazione cellulare.

Attivazione di un oncogene per traslocazione

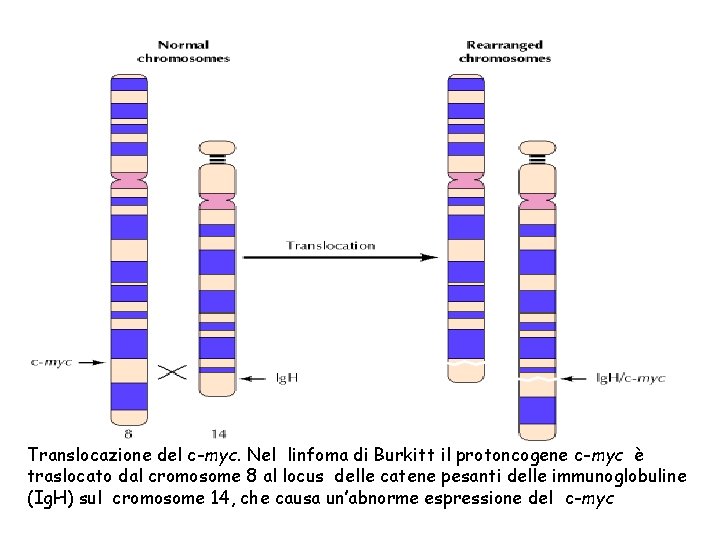
Translocazione del c-myc. Nel linfoma di Burkitt il protoncogene c-myc è traslocato dal cromosome 8 al locus delle catene pesanti delle immunoglobuline (Ig. H) sul cromosome 14, che causa un’abnorme espressione del c-myc

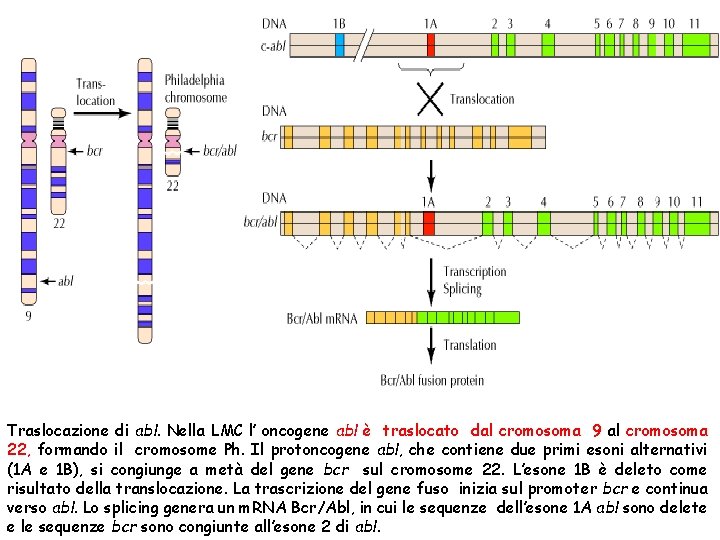
Traslocazione di abl. Nella LMC l’ oncogene abl è traslocato dal cromosoma 9 al cromosoma 22, formando il cromosome Ph. Il protoncogene abl, che contiene due primi esoni alternativi (1 A e 1 B), si congiunge a metà del gene bcr sul cromosome 22. L’esone 1 B è deleto come risultato della translocazione. La trascrizione del gene fuso inizia sul promoter bcr e continua verso abl. Lo splicing genera un m. RNA Bcr/Abl, in cui le sequenze dell’esone 1 A abl sono delete e le sequenze bcr sono congiunte all’esone 2 di abl.

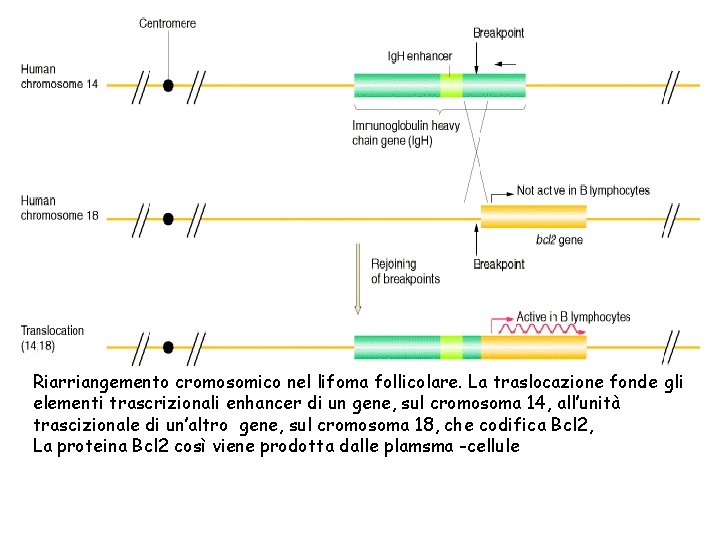
Riarriangemento cromosomico nel lifoma follicolare. La traslocazione fonde gli elementi trascrizionali enhancer di un gene, sul cromosoma 14, all’unità trascizionale di un’altro gene, sul cromosoma 18, che codifica Bcl 2, La proteina Bcl 2 così viene prodotta dalle plamsma -cellule

Attivazione di un oncogene per inserzione

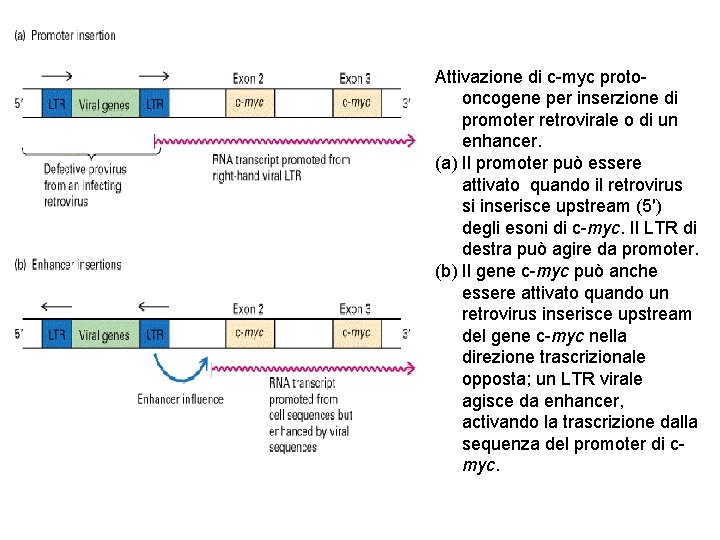
Attivazione di c-myc protooncogene per inserzione di promoter retrovirale o di un enhancer. (a) Il promoter può essere attivato quando il retrovirus si inserisce upstream (5′) degli esoni di c-myc. Il LTR di destra può agire da promoter. (b) Il gene c-myc può anche essere attivato quando un retrovirus inserisce upstream del gene c-myc nella direzione trascrizionale opposta; un LTR virale agisce da enhancer, activando la trascrizione dalla sequenza del promoter di cmyc.

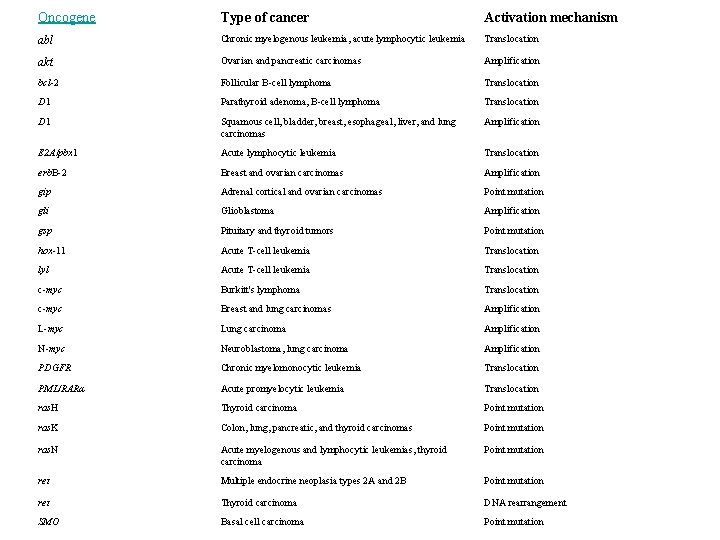
Oncogene Type of cancer Activation mechanism abl Chronic myelogenous leukemia, acute lymphocytic leukemia Translocation akt Ovarian and pancreatic carcinomas Amplification bcl-2 Follicular B-cell lymphoma Translocation D 1 Parathyroid adenoma, B-cell lymphoma Translocation D 1 Squamous cell, bladder, breast, esophageal, liver, and lung carcinomas Amplification E 2 A/pbx 1 Acute lymphocytic leukemia Translocation erb. B-2 Breast and ovarian carcinomas Amplification gip Adrenal cortical and ovarian carcinomas Point mutation gli Glioblastoma Amplification gsp Pituitary and thyroid tumors Point mutation hox-11 Acute T-cell leukemia Translocation lyl Acute T-cell leukemia Translocation c-myc Burkitt's lymphoma Translocation c-myc Breast and lung carcinomas Amplification L-myc Lung carcinoma Amplification N-myc Neuroblastoma, lung carcinoma Amplification PDGFR Chronic myelomonocytic leukemia Translocation PML/RARα Acute promyelocytic leukemia Translocation ras. H Thyroid carcinoma Point mutation ras. K Colon, lung, pancreatic, and thyroid carcinomas Point mutation ras. N Acute myelogenous and lymphocytic leukemias, thyroid carcinoma Point mutation ret Multiple endocrine neoplasia types 2 A and 2 B Point mutation ret Thyroid carcinoma DNA rearrangement SMO Basal cell carcinoma Point mutation

Oncogeni che codificano per fattori di crescita

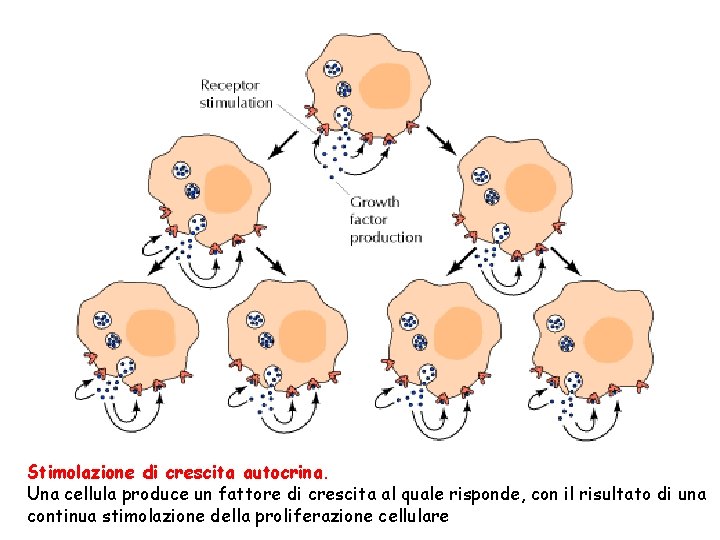
Stimolazione di crescita autocrina. Una cellula produce un fattore di crescita al quale risponde, con il risultato di una continua stimolazione della proliferazione cellulare

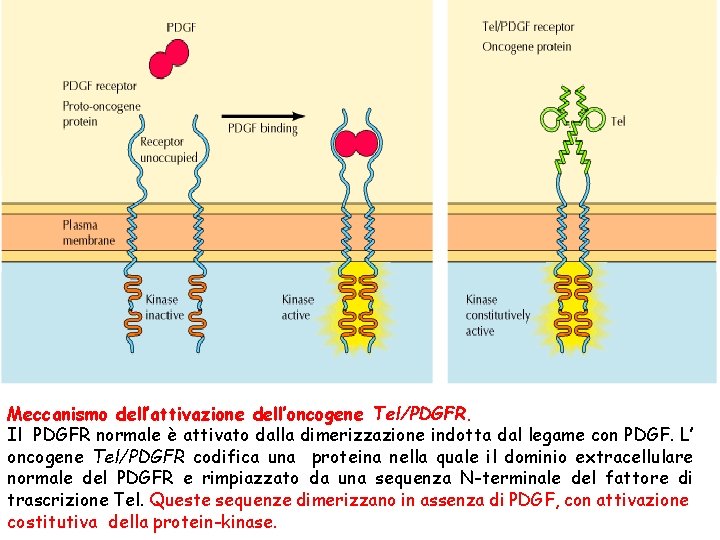
Meccanismo dell’attivazione dell’oncogene Tel/PDGFR. Il PDGFR normale è attivato dalla dimerizzazione indotta dal legame con PDGF. L’ oncogene Tel/PDGFR codifica una proteina nella quale il dominio extracellulare normale del PDGFR e rimpiazzato da una sequenza N-terminale del fattore di trascrizione Tel. Queste sequenze dimerizzano in assenza di PDGF, con attivazione costitutiva della protein-kinase.

Oncogeni che codificano per recettori di membrana

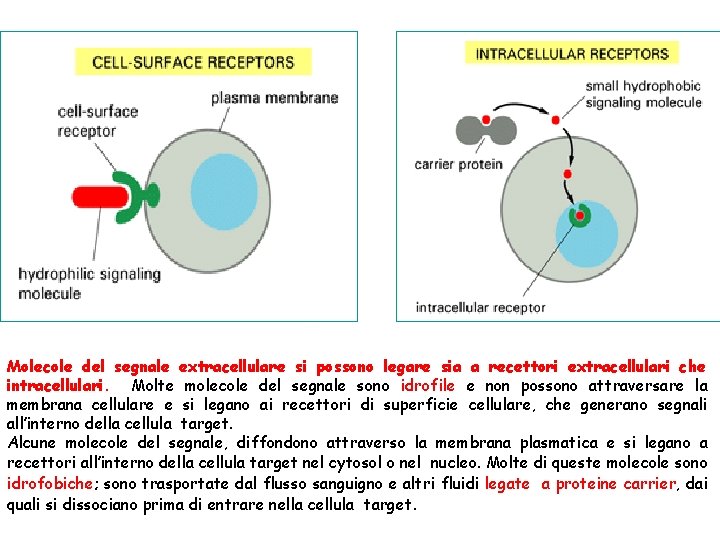
Molecole del segnale extracellulare si possono legare sia a recettori extracellulari che intracellulari. Molte molecole del segnale sono idrofile e non possono attraversare la membrana cellulare e si legano ai recettori di superficie cellulare, che generano segnali all’interno della cellula target. Alcune molecole del segnale, diffondono attraverso la membrana plasmatica e si legano a recettori all’interno della cellula target nel cytosol o nel nucleo. Molte di queste molecole sono idrofobiche; sono trasportate dal flusso sanguigno e altri fluidi legate a proteine carrier, dai quali si dissociano prima di entrare nella cellula target.

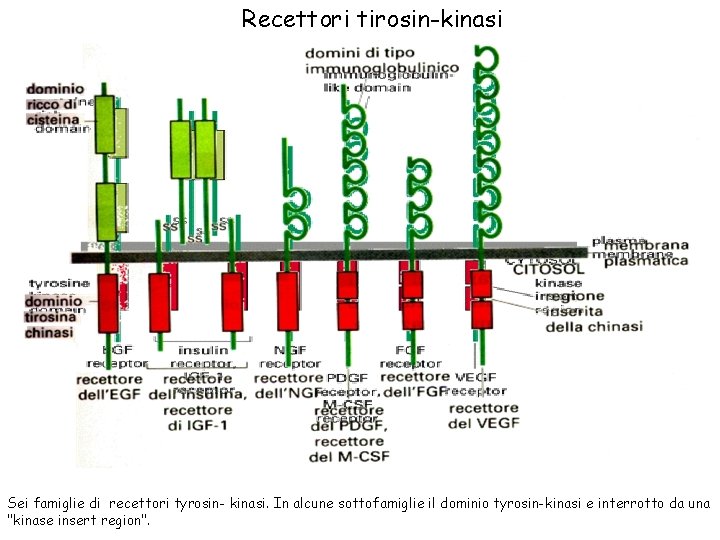
Recettori tirosin-kinasi Sei famiglie di recettori tyrosin- kinasi. In alcune sottofamiglie il dominio tyrosin-kinasi e interrotto da una "kinase insert region".

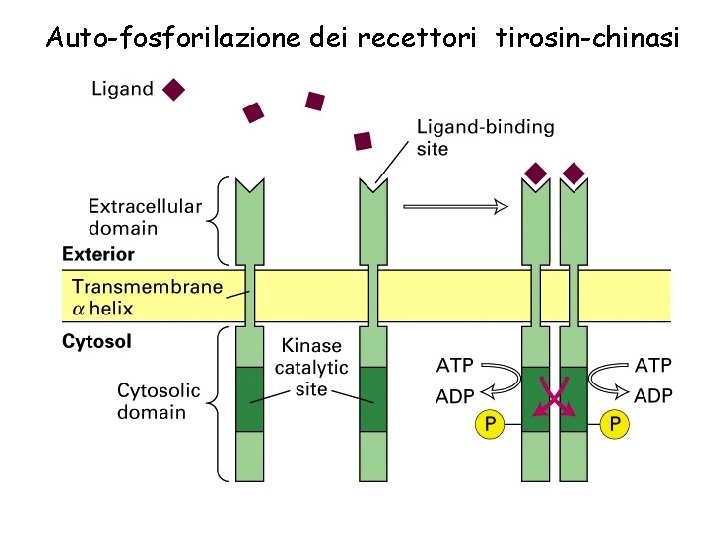
Auto-fosforilazione dei recettori tirosin-chinasi

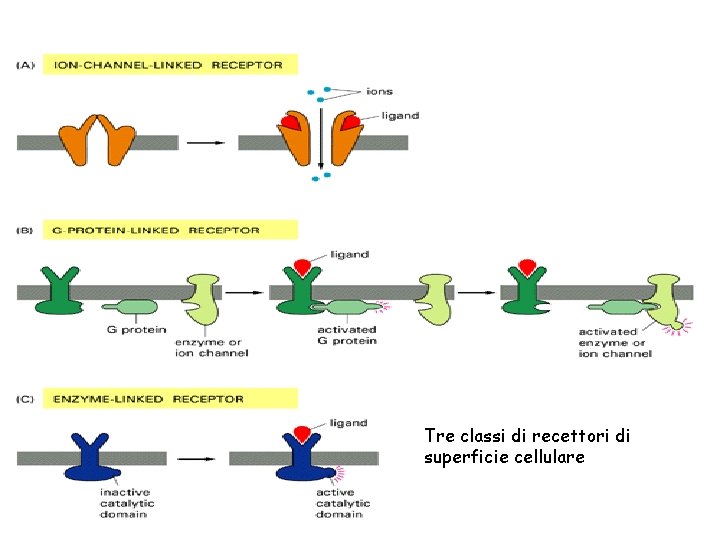
Tre classi di recettori di superficie cellulare

Recettori Enzyme-linked • (1) recettori guanilil cyclasi, che catalizzano la produzione del c. GMP nel cytosol; • (2) recettori tirosin- kinasi, che fosforilano specifici residui tirosina su un piccolo gruppo di proteine del segnale intracellulari; • (3) recettori tirosin-kinasi-associati, che sono associati a proteine con attività tyrosin-kinasi; • (4) recettori tirosin- fosfatasi, che rimuovono i gruppi fosfato dai residui di tirosina di specifiche proteine del segnale intracellulari; • (5) recettori serine/treonine kinasi, che fosforilano specifici residui di serina o treonina da alcune proteine intracellulari.

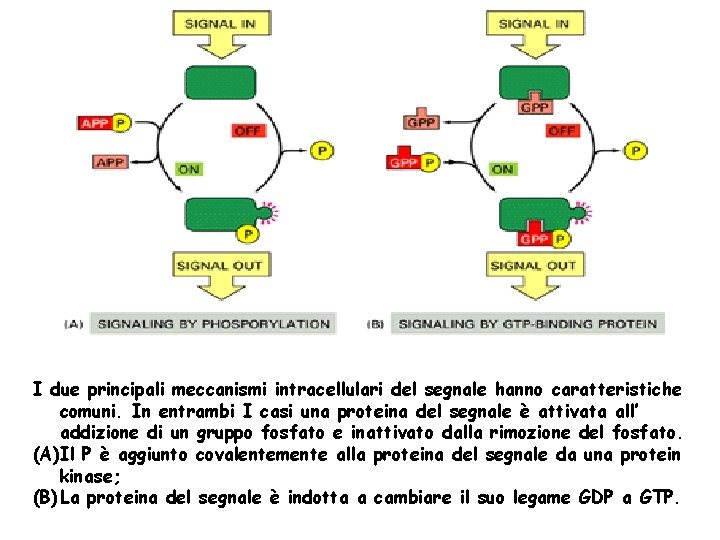
I due principali meccanismi intracellulari del segnale hanno caratteristiche comuni. In entrambi I casi una proteina del segnale è attivata all’ addizione di un gruppo fosfato e inattivato dalla rimozione del fosfato. (A)Il P è aggiunto covalentemente alla proteina del segnale da una protein kinase; (B) La proteina del segnale è indotta a cambiare il suo legame GDP a GTP.

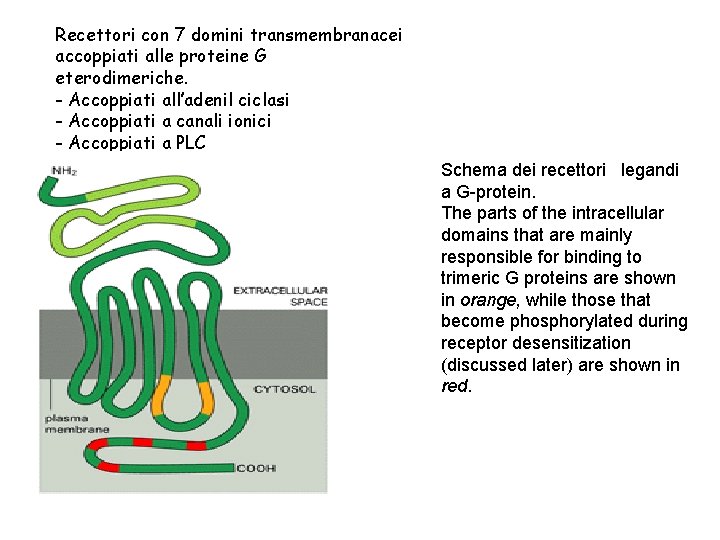
Recettori con 7 domini transmembranacei accoppiati alle proteine G eterodimeriche. - Accoppiati all’adenil ciclasi - Accoppiati a canali ionici - Accoppiati a PLC Schema dei recettori legandi a G-protein. The parts of the intracellular domains that are mainly responsible for binding to trimeric G proteins are shown in orange, while those that become phosphorylated during receptor desensitization (discussed later) are shown in red.

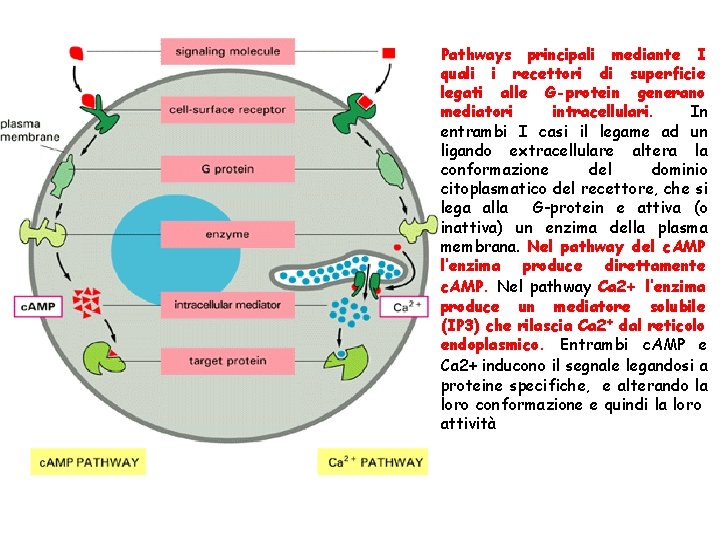
Pathways principali mediante I quali i recettori di superficie legati alle G-protein generano mediatori intracellulari. In entrambi I casi il legame ad un ligando extracellulare altera la conformazione del dominio citoplasmatico del recettore, che si lega alla G-protein e attiva (o inattiva) un enzima della plasma membrana. Nel pathway del c. AMP l’enzima produce direttamente c. AMP. Nel pathway Ca 2+ l’enzima produce un mediatore solubile (IP 3) che rilascia Ca 2+ dal reticolo endoplasmico. Entrambi c. AMP e Ca 2+ inducono il segnale legandosi a proteine specifiche, e alterando la loro conformazione e quindi la loro attività



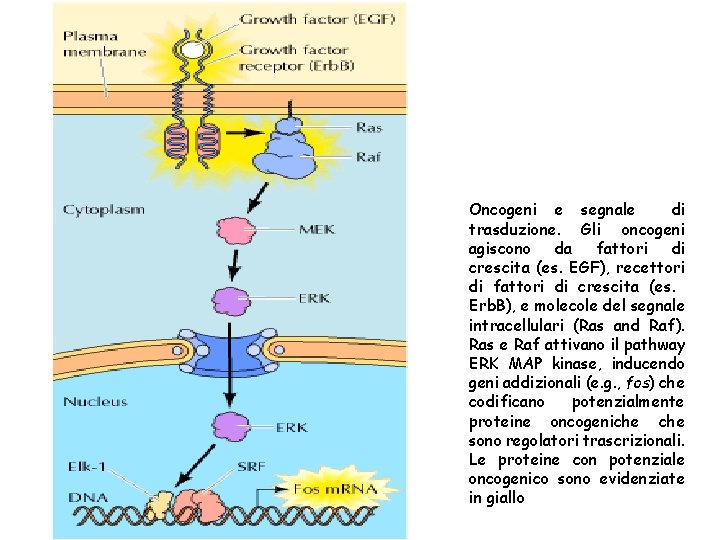
Oncogeni e segnale di trasduzione. Gli oncogeni agiscono da fattori di crescita (es. EGF), recettori di fattori di crescita (es. Erb. B), e molecole del segnale intracellulari (Ras and Raf). Ras e Raf attivano il pathway ERK MAP kinase, inducendo geni addizionali (e. g. , fos) che codificano potenzialmente proteine oncogeniche sono regolatori trascrizionali. Le proteine con potenziale oncogenico sono evidenziate in giallo

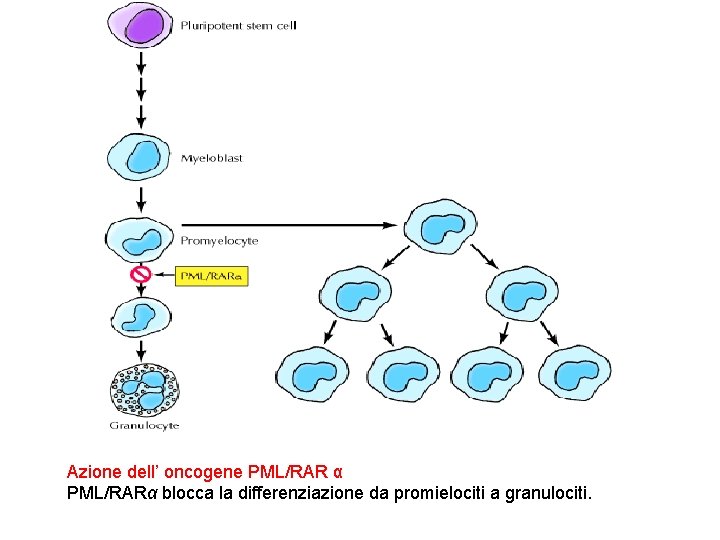
Azione dell’ oncogene PML/RAR α PML/RARα blocca la differenziazione da promielociti a granulociti.

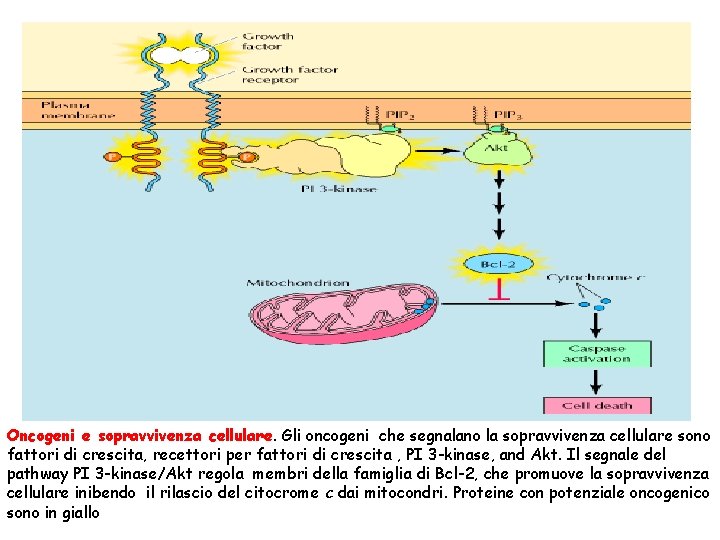
Oncogeni e sopravvivenza cellulare. Gli oncogeni che segnalano la sopravvivenza cellulare sono fattori di crescita, recettori per fattori di crescita , PI 3 -kinase, and Akt. Il segnale del pathway PI 3 -kinase/Akt regola membri della famiglia di Bcl-2, che promuove la sopravvivenza cellulare inibendo il rilascio del citocrome c dai mitocondri. Proteine con potenziale oncogenico sono in giallo

Il Cancro è una malattia ereditaria? -15% dei casi di tumore hanno una componente ereditaria

Geni adibiti al riparo del DNA (caretakers) 1. La loro mutazione è spesso alla base di sindromi neoplastiche familiari 2. Prototipo sono i geni h. MLH 1, h. PMS 1 e h. PMS 2, mutati nell’Hereditary Non Polyposis Colon Cancer (HNPCC); i geni BRCA-1 e BRCA-2, mutati nel carcinoma della mammella; il gene ATM (atassia-teleangectasia) 3. La mutazione di questi geni non influenza direttamente la crescita cellulare né l’apoptosi ma favorisce la trasformazione e accelera la progressione neoplastica. 4. Gli individui affetti presentano instabilità di particolari regioni del DNA (coppie ripetute di 1 -6 nucleotidi) denominate microsatelliti

Cancro Familiare • L’ereditarietà di una mutazione predisponente in un oncogene: - RET sindromi tumorali – Carcinoma Familiare della Midollare della tiroide (FMTC) e Neoplasia Endocrina Multipla Tipo 2 A (MEN 2 A) - MET nel cancro ereditario papillare renale - CDK 4 nel melanoma familiare • L’ereditarietà di una mutazione predisponente in un gene oncosoppressore • L’ereditarietà di una mutazione predisponente in un gene che ripara il DNA

Cancro Familiare • Una singola mutazione è insufficiente a causare il cancro • Ulteriori alterazioni in altri geni necessarie all’insorgenza del cancro sono


UV Errori di replicazione Cancerogeni chimici Radiazioni Cellula Normale Mutazioni puntiformi Virus Danno al DNA Riarrangiamenti (traslocazioni, delezioni, Amplificazione) Alterazioni al DNA di(0 ncogeni e geni oncosoppressori)Quando avviene un’alterazione in un gene della linea germinale Insorge un cancro familiare- Proliferazione sregolata Cellula Tumorale

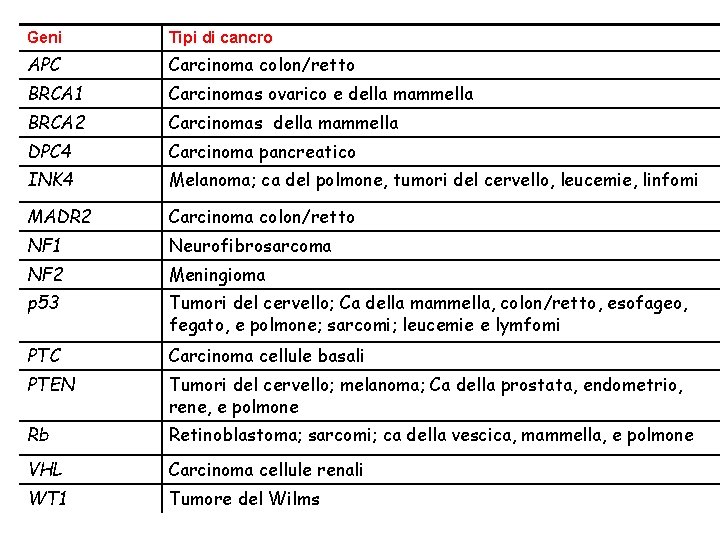
Geni Tipi di cancro APC Carcinoma colon/retto BRCA 1 Carcinomas ovarico e della mammella BRCA 2 Carcinomas della mammella DPC 4 Carcinoma pancreatico INK 4 Melanoma; ca del polmone, tumori del cervello, leucemie, linfomi MADR 2 Carcinoma colon/retto NF 1 Neurofibrosarcoma NF 2 Meningioma p 53 Tumori del cervello; Ca della mammella, colon/retto, esofageo, fegato, e polmone; sarcomi; leucemie e lymfomi PTC Carcinoma cellule basali PTEN Tumori del cervello; melanoma; Ca della prostata, endometrio, rene, e polmone Rb Retinoblastoma; sarcomi; ca della vescica, mammella, e polmone VHL Carcinoma cellule renali WT 1 Tumore del Wilms


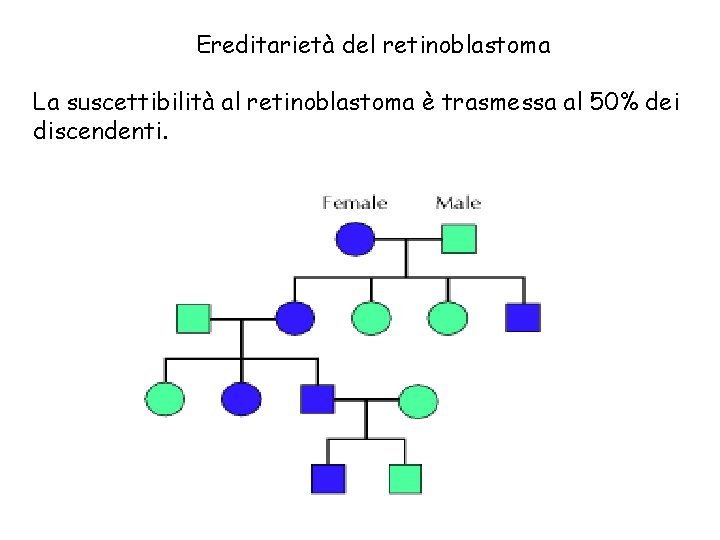
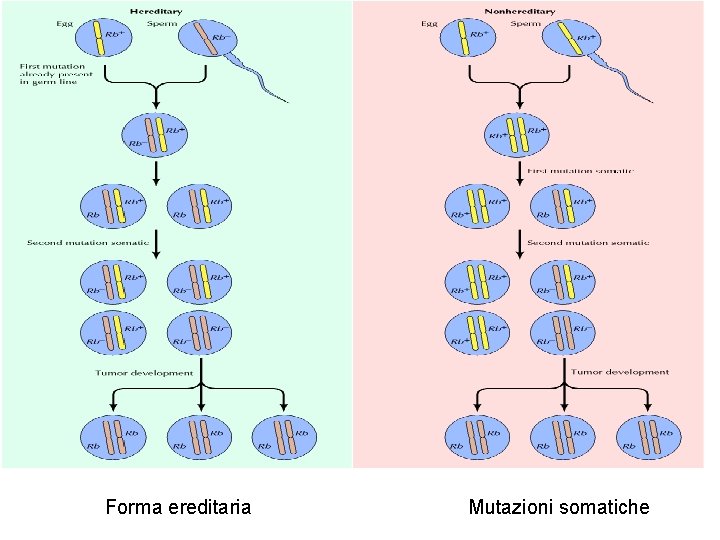
Ereditarietà del retinoblastoma La suscettibilità al retinoblastoma è trasmessa al 50% dei discendenti.

Forma ereditaria Mutazioni somatiche

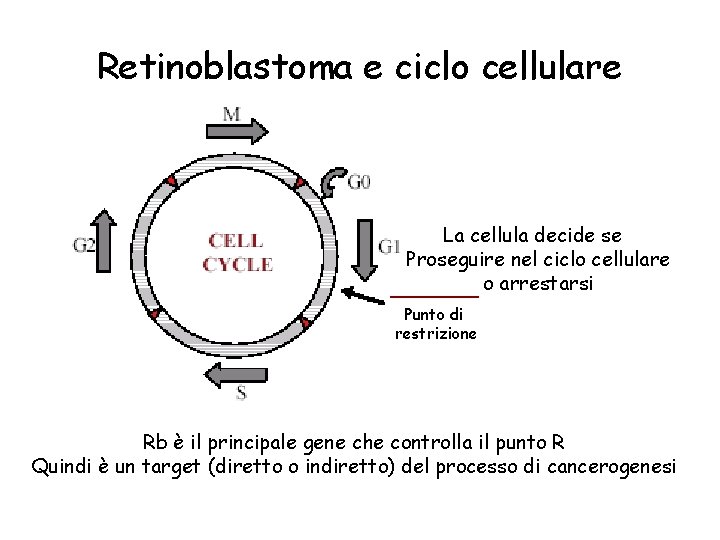
Retinoblastoma e ciclo cellulare La cellula decide se Proseguire nel ciclo cellulare o arrestarsi Punto di restrizione Rb è il principale gene che controlla il punto R Quindi è un target (diretto o indiretto) del processo di cancerogenesi


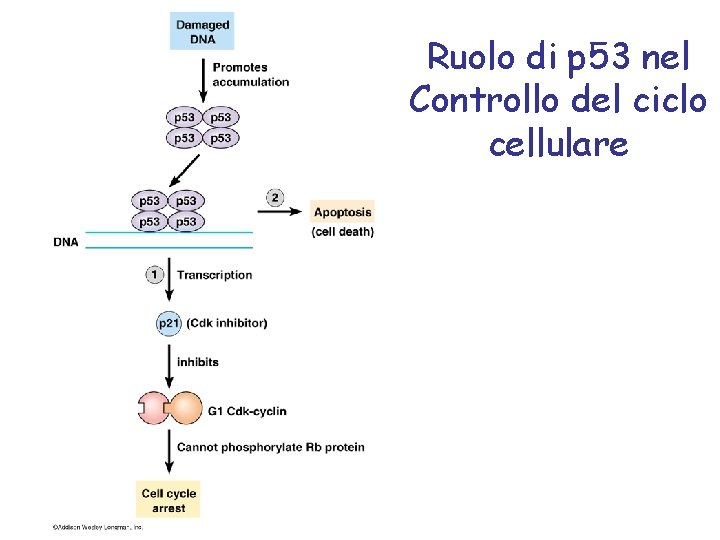
Ruolo di p 53 nel Controllo del ciclo cellulare

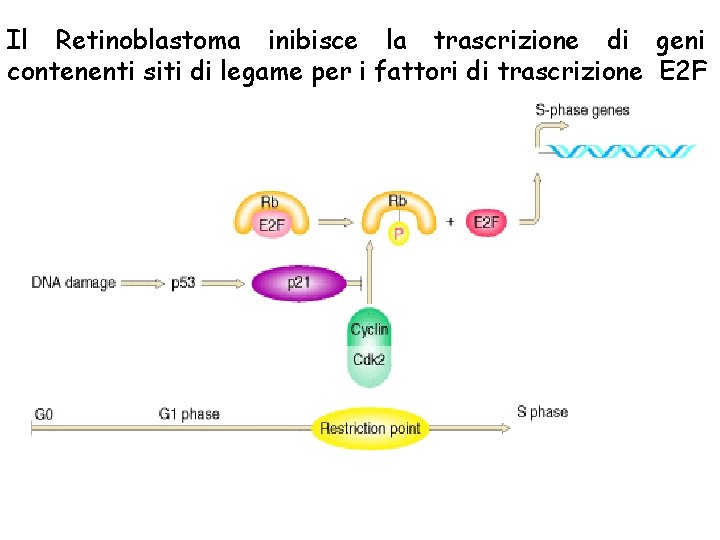
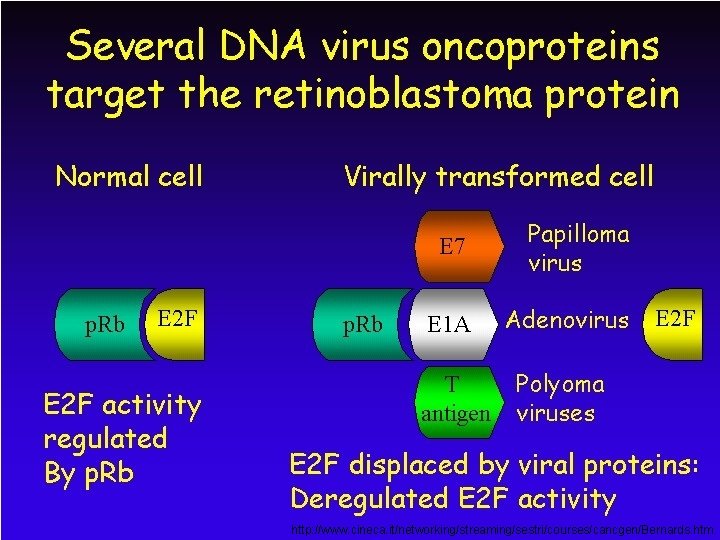
Il Retinoblastoma inibisce la trascrizione di geni contenenti siti di legame per i fattori di trascrizione E 2 F

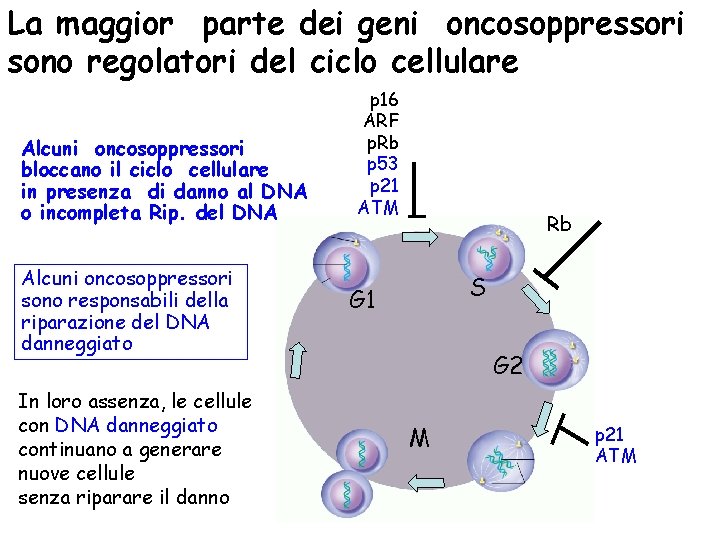
La maggior parte dei geni oncosoppressori sono regolatori del ciclo cellulare Alcuni oncosoppressori bloccano il ciclo cellulare in presenza di danno al DNA o incompleta Rip. del DNA Alcuni oncosoppressori sono responsabili della riparazione del DNA danneggiato In loro assenza, le cellule con DNA danneggiato continuano a generare nuove cellule senza riparare il danno p 16 ARF p. Rb p 53 p 21 ATM Rb S G 1 G 2 M p 21 ATM

Geni che riparano il DNA • Il cancro insorge anche per difettiva riparazione del DNA • Mutazioni nei geni (BRCA 1, BRCA 2) induce suscettibilità per il Ca della mammella • Geni (MSH 2, MLH 1, PMS 2, MSH 6) nella suscettibilità del Hereditary Non-polyposis Colon Cancer (HPNCC)

P 16 (Hereditary Melanoma) • p 16 è mutato o deleto nelle leucemie e nel cancro della testa e del collo. E’ mutato nel melanoma comprese le forme familiari della malattia. • p 16 è un membro della famiglia INK 4 degli inibitori cyclin dependent kinase inhibitor • p 16 protegge l’ Rb dall’iperfosforilazione ed inattivazione da parte di cyclin D 1/CDK 4 • Quando p 16 è mutato o deleto non è capace di eseguire questa funzione




Conclusioni • Il Cancro è una malattia multifattoriale, fattori ambientali e genetici contribuiscono allo sviluppo dei tumori • Alterazioni nei proto-oncogeni e nei geni oncosoppressori causano la predisposizione all’insorgenza di tumori. Ma non causano il cancro • Lo sviluppo del cancro è un processo multifasico: Addizionali mutazioni sono necessarie affinché si possa sviluppare un tumore • Possono essere necessari fino a 6 -12 mutazioni per lo sviluppo del cancro • I cambiamenti genici possono avvenire negli oncogeni, nei geni oncosoppressori, nei geni che regolano l’apoptosi che riparano il DNA e che controllano la differenziazione • La predisposizione ereditaria avviene circa nel 15% dei tumori per mutazioni ereditarie in: • Geni oncosoppressori • Geni che riparano il DNA • Raramente oncogeni

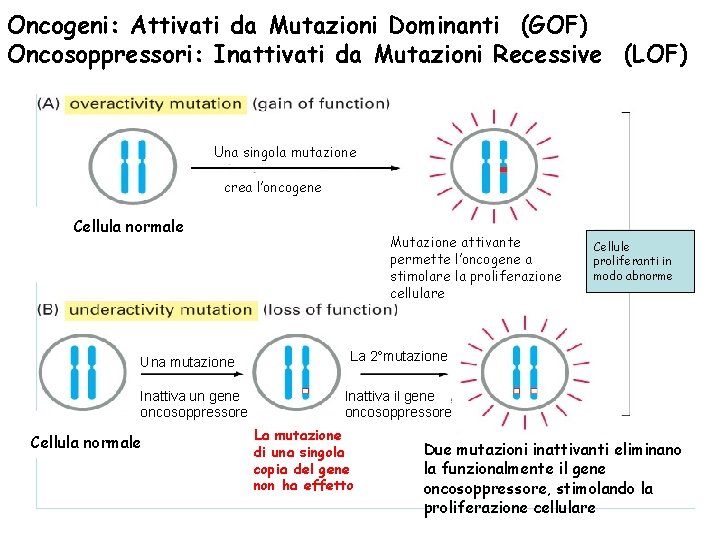
Oncogeni: Attivati da Mutazioni Dominanti (GOF) Oncosoppressori: Inattivati da Mutazioni Recessive (LOF) Una singola mutazione crea l’oncogene Cellula normale Una mutazione Inattiva un gene oncosoppressore Cellula normale Mutazione attivante permette l’oncogene a stimolare la proliferazione cellulare Cellule proliferanti in modo abnorme La 2°mutazione Inattiva ilungene oncosoppressore La mutazione di una singola copia del gene non ha effetto Due mutazioni inattivanti eliminano la funzionalmente il gene oncosoppressore, stimolando la proliferazione cellulare

Perdita di Eterozigosi La seconda mutazioneinattivante di una normale copia del gene può avvenire mediante alcuni meccanismi Loss of Heterozygosity (LOH) Second Mutation

La natura multifasica del cancro • • Lo sviluppo del cancro è un processo multifasico che coinvolge 6 -12 cambiamenti in geni regolatori. • Lo sviluppo del cancro dipende da alterazioni in vari pathways della trasduzione del segnale nella cellula. Esempio: il cancro Colon-rettale. • Lo sviluppo del cancro dipende dall’alterazione di molti geni in un pathway di transduzione. In molti casi queste alterazioni possono essere ridondanti.

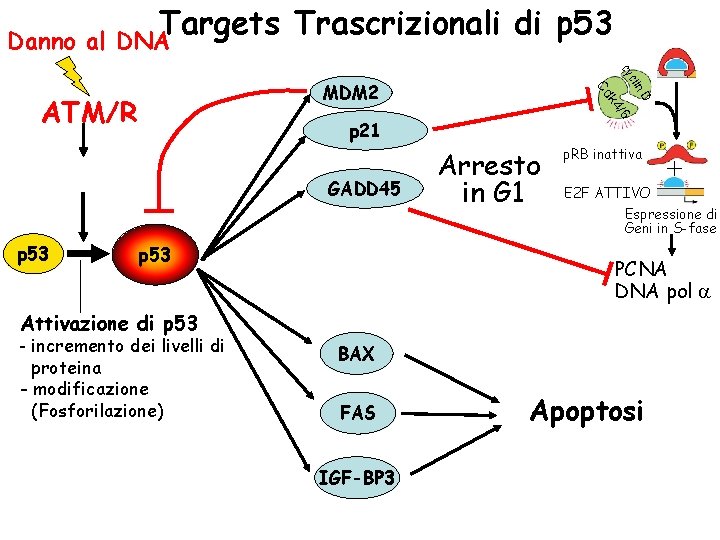
Danno al Targets Trascrizionali di p 53 DNA cy n. D /6 p 53 cli p 21 GADD 45 p 53 k 4 ATM/R Cd MDM 2 Arresto in G 1 p. RB inattiva E 2 F ATTIVO Espressione di Geni in S-fase PCNA DNA pol a Attivazione di p 53 - incremento dei livelli di proteina - modificazione (Fosforilazione) + BAX FAS IGF-BP 3 Apoptosi

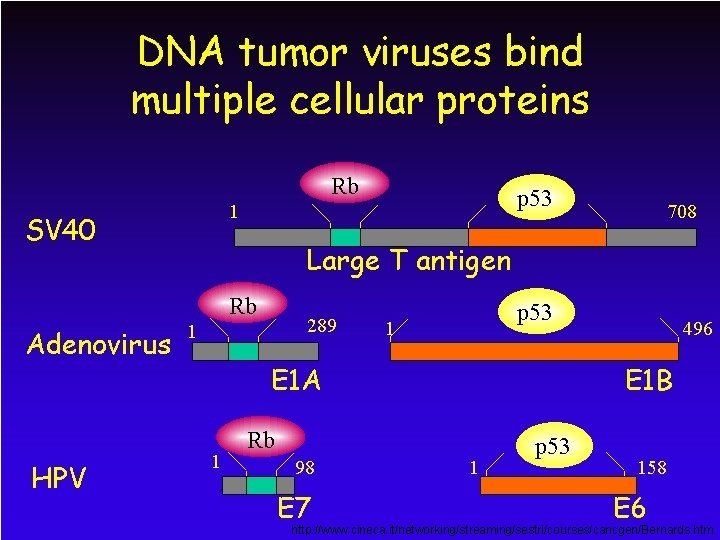
http: //www. cineca. it/networking/streaming/sestri/courses/cancgen/Bernards. htm

http: //www. cineca. it/networking/streaming/sestri/courses/cancgen/Bernards. htm
 Geni oncosoppressori
Geni oncosoppressori Epatociti
Epatociti Causa inmediata y causa basica
Causa inmediata y causa basica Cancro duro fotos
Cancro duro fotos Cancro granchio
Cancro granchio Cosa sono i fusi orari
Cosa sono i fusi orari Scienze umane cosa sono
Scienze umane cosa sono Cellula animale e vegetale
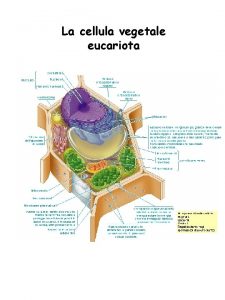
Cellula animale e vegetale Cellula animale e vegetale
Cellula animale e vegetale Giunzioni aderenti
Giunzioni aderenti Hooke scientist cell theory
Hooke scientist cell theory Tela nervosa
Tela nervosa Cellula vegetale immagine
Cellula vegetale immagine Parete secondaria cellula vegetale
Parete secondaria cellula vegetale Cellula adiposa
Cellula adiposa La cellula definizione
La cellula definizione Vacuolo cellula vegetale
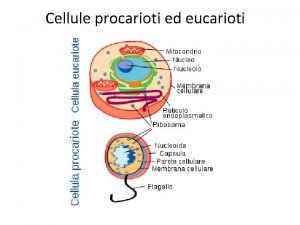
Vacuolo cellula vegetale Cellula procariote ed eucariote
Cellula procariote ed eucariote Espansina
Espansina Procariote
Procariote Cellula parenchimatica
Cellula parenchimatica Rel celula animal o vegetal
Rel celula animal o vegetal Differenza tra cellula animale e vegetale
Differenza tra cellula animale e vegetale Isoprenile
Isoprenile Cap
Cap Cellula eucariote
Cellula eucariote Trasduzione specializzata
Trasduzione specializzata Metabolismo cellulare mappa
Metabolismo cellulare mappa Cellula eucariote zanichelli
Cellula eucariote zanichelli Cellula eucariote
Cellula eucariote Plastidi cellula vegetale
Plastidi cellula vegetale Tallo fungino
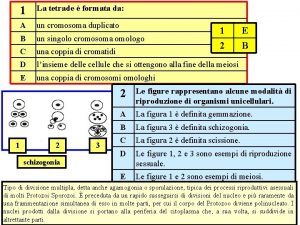
Tallo fungino Tetrade
Tetrade Cellula vegetale
Cellula vegetale Diffusione facilitata
Diffusione facilitata Osserviamo la cellula zanichelli
Osserviamo la cellula zanichelli Vincenzo busiello
Vincenzo busiello Enriquecimiento sin causa
Enriquecimiento sin causa Não te indignes por causa dos malfeitores
Não te indignes por causa dos malfeitores Elementi essenziali del contratto di lavoro
Elementi essenziali del contratto di lavoro Argumento de causa y efecto
Argumento de causa y efecto Gasometra
Gasometra Es algo mas que la distancia que el amor y la nostalgia
Es algo mas que la distancia que el amor y la nostalgia 5 partes del cuento
5 partes del cuento Hipercalemia causa
Hipercalemia causa Causa serviliana
Causa serviliana Acidosis metabólica fisiopatología
Acidosis metabólica fisiopatología Ostras perlas dolor
Ostras perlas dolor Dr. rolando estrada
Dr. rolando estrada Alcalosis metabolica posthipercapnica
Alcalosis metabolica posthipercapnica Meningitis viral antibiótico
Meningitis viral antibiótico Causas material
Causas material Viri magni nostri maiores
Viri magni nostri maiores árbol de problemas ejemplos educación
árbol de problemas ejemplos educación Que es esto
Que es esto No hay maldicion sin causa
No hay maldicion sin causa Causa efficiente
Causa efficiente Oración
Oración Poliuria
Poliuria Negozio giuridico
Negozio giuridico Conectores de causa efecto
Conectores de causa efecto Argumentos por signos
Argumentos por signos Causa effetto lannaronca
Causa effetto lannaronca Argumentos de autoridad
Argumentos de autoridad Kwasinski
Kwasinski Silogismo exemplos
Silogismo exemplos Causa necesaria y suficiente ejemplos
Causa necesaria y suficiente ejemplos Uma causa nobre mir4 completo
Uma causa nobre mir4 completo Conflicto en el cielo
Conflicto en el cielo Causa
Causa Teori indvidualisir birkmeyer
Teori indvidualisir birkmeyer Cuales fueron las causas de la primera guerra mundial
Cuales fueron las causas de la primera guerra mundial Tipos de argumentos causa efecto
Tipos de argumentos causa efecto Diagrama de obesidad
Diagrama de obesidad Causa complexa
Causa complexa Manejo hipernatremia
Manejo hipernatremia Causa raiz de un accidente
Causa raiz de un accidente Nao te indignes por causa
Nao te indignes por causa Diagrama de ishikawa 6m ejemplos
Diagrama de ishikawa 6m ejemplos Babakr al-zibari
Babakr al-zibari Lhermitte’s sign
Lhermitte’s sign Causa complexa
Causa complexa Es necesario que atraves de muchas tribulaciones
Es necesario que atraves de muchas tribulaciones Diretoria de ensino botucatu
Diretoria de ensino botucatu