Controle epigentico da expresso gnica em plantas Alterao














- Slides: 39

Controle epigenético da expressão gênica em plantas Alteração da expressão gênica sem alterações na sequência de DNA Caso clássico: Alterações epigenéticas que afetam propriedades de pigmentação Duas possibilidades: permanecem após a meiose x não permanecem após a meiose Mudancas epigenenéticas típicas das plantas: normalmente transmitidas na meiose

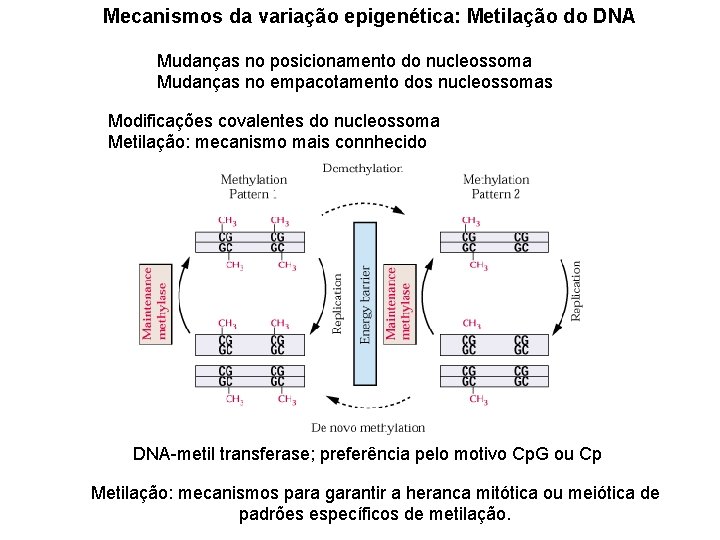
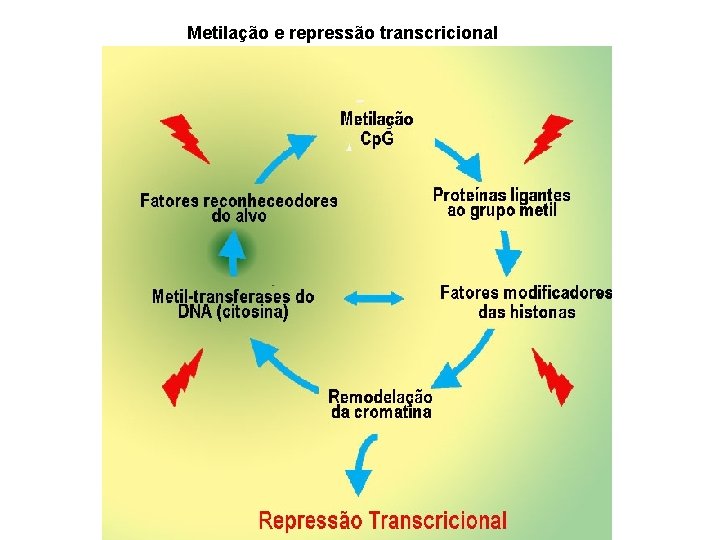
Mecanismos da variação epigenética: Metilação do DNA Mudanças no posicionamento do nucleossoma Mudanças no empacotamento dos nucleossomas Modificações covalentes do nucleossoma Metilação: mecanismo mais connhecido DNA-metil transferase; preferência pelo motivo Cp. G ou Cp Metilação: mecanismos para garantir a heranca mitótica ou meiótica de padrões específicos de metilação.

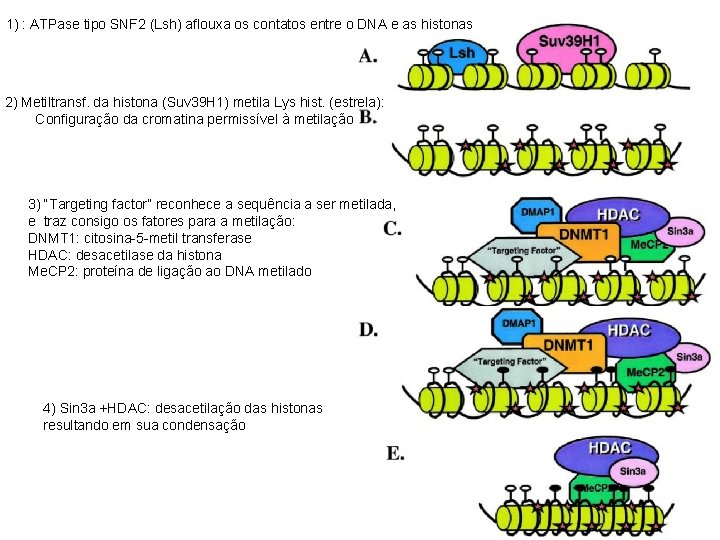
1) : ATPase tipo SNF 2 (Lsh) aflouxa os contatos entre o DNA e as histonas 2) Metiltransf. da histona (Suv 39 H 1) metila Lys hist. (estrela): Configuração da cromatina permissível à metilação 3) “Targeting factor” reconhece a sequência a ser metilada, e traz consigo os fatores para a metilação: DNMT 1: citosina-5 -metil transferase HDAC: desacetilase da histona Me. CP 2: proteína de ligação ao DNA metilado 4) Sin 3 a +HDAC: desacetilação das histonas resultando em sua condensação

Metilação e repressão transcricional

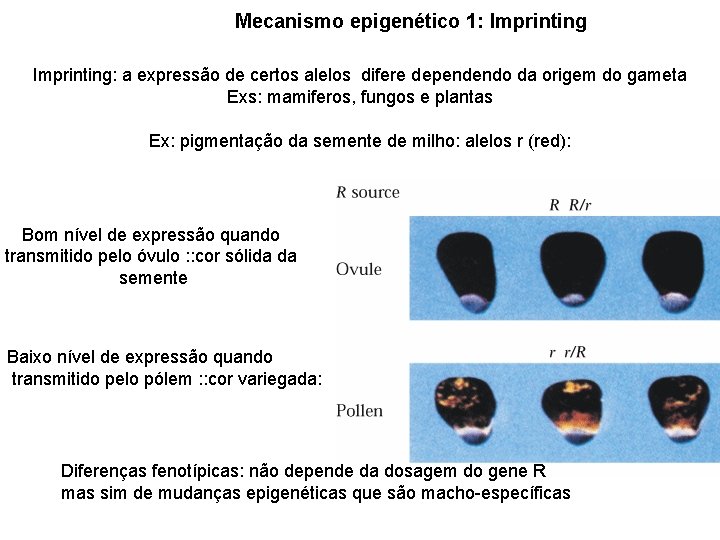
Mecanismo epigenético 1: Imprinting: a expressão de certos alelos difere dependendo da origem do gameta Exs: mamiferos, fungos e plantas Ex: pigmentação da semente de milho: alelos r (red): Bom nível de expressão quando transmitido pelo óvulo : : cor sólida da semente Baixo nível de expressão quando transmitido pelo pólem : : cor variegada: Diferenças fenotípicas: não depende da dosagem do gene R mas sim de mudanças epigenéticas que são macho-específicas

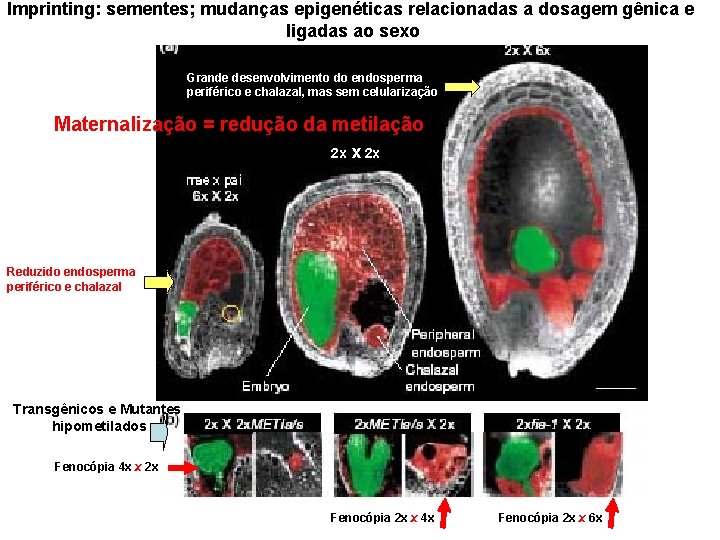
Imprinting: sementes; mudanças epigenéticas relacionadas a dosagem gênica e ligadas ao sexo Grande desenvolvimento do endosperma periférico e chalazal, mas sem celularização Maternalização = redução da metilação 2 x x 2 x Reduzido endosperma periférico e chalazal Transgênicos e Mutantes hipometilados Fenocópia 4 x x 2 x Fenocópia 2 x x 4 x Fenocópia 2 x x 6 x

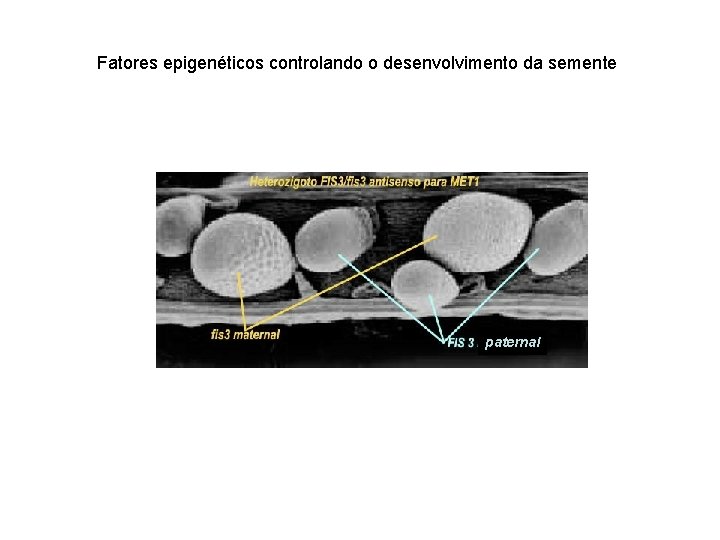
Fatores epigenéticos controlando o desenvolvimento da semente paternal

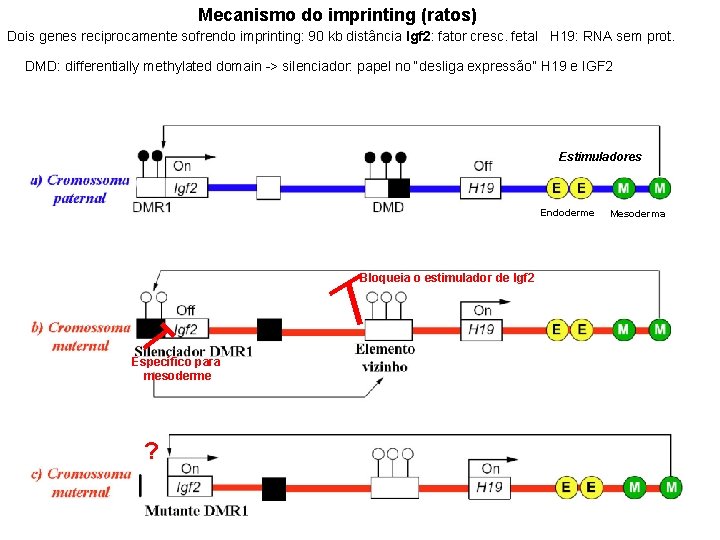
Mecanismo do imprinting (ratos) Dois genes reciprocamente sofrendo imprinting: 90 kb distância Igf 2: fator cresc. fetal H 19: RNA sem prot. DMD: differentially methylated domain -> silenciador: papel no “desliga expressão” H 19 e IGF 2 Estimuladores Endoderme Bloqueia o estimulador de Igf 2 Específico para mesoderme ? Mesoderma

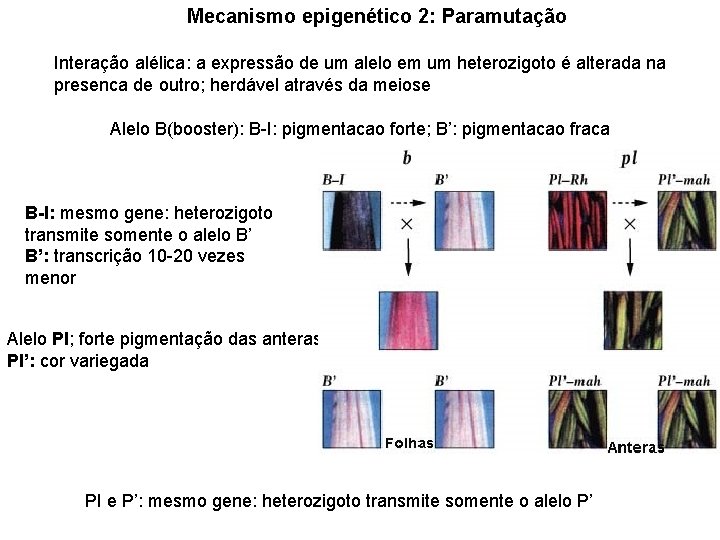
Mecanismo epigenético 2: Paramutação Interação alélica: a expressão de um alelo em um heterozigoto é alterada na presenca de outro; herdável através da meiose Alelo B(booster): B-I: pigmentacao forte; B’: pigmentacao fraca B-I: mesmo gene: heterozigoto transmite somente o alelo B’ B’: transcrição 10 -20 vezes menor Alelo Pl; forte pigmentação das anteras; Pl’: cor variegada PI e P’: mesmo gene: heterozigoto transmite somente o alelo P’

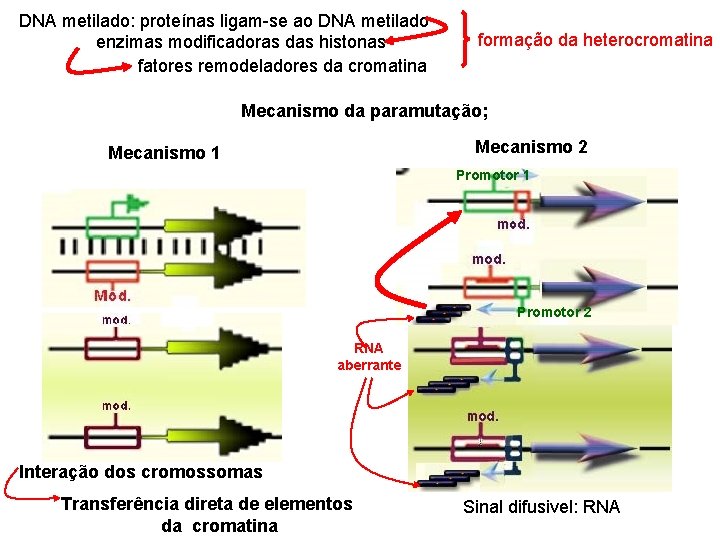
DNA metilado: proteínas ligam-se ao DNA metilado enzimas modificadoras das histonas fatores remodeladores da cromatina formação da heterocromatina Mecanismo da paramutação; Mecanismo 2 Mecanismo 1 Promotor 2 RNA aberrante Interação dos cromossomas Transferência direta de elementos da cromatina Sinal difusivel: RNA

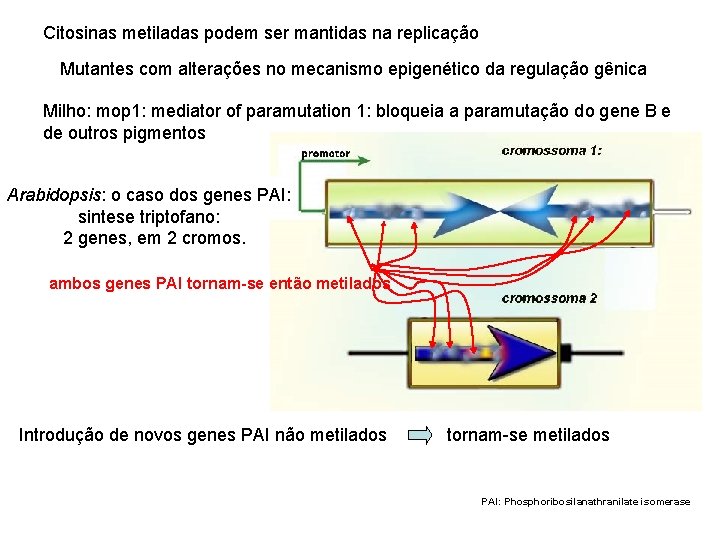
Citosinas metiladas podem ser mantidas na replicação Mutantes com alterações no mecanismo epigenético da regulação gênica Milho: mop 1: mediator of paramutation 1: bloqueia a paramutação do gene B e de outros pigmentos Arabidopsis: o caso dos genes PAI: sintese triptofano: 2 genes, em 2 cromos. ambos genes PAI tornam-se então metilados Introdução de novos genes PAI não metilados tornam-se metilados PAI: Phosphoribosilanathranilate isomerase

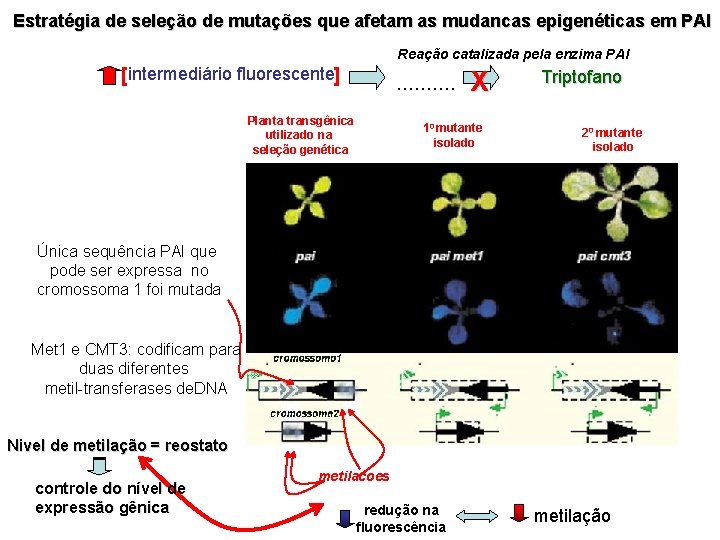
Estratégia de seleção de mutações que afetam as mudancas epigenéticas em PAI Reação catalizada pela enzima PAI [intermediário fluorescente] ………. Planta transgênica utilizado na seleção genética X 10 mutante isolado Triptofano 20 mutante isolado Única sequência PAI que pode ser expressa no cromossoma 1 foi mutada Met 1 e CMT 3: codificam para duas diferentes metil-transferases de. DNA Nivel de metilação = reostato controle do nível de expressão gênica metilacoes redução na fluorescência metilação

Mecanismo epigenético 3: Silenciamento Detecção: plantas trangênicas; Arabidopsis, Petunia e tabaco Repetição da presença de genes : : silenciamento de outras sequências Estrutura ou organização do cromossoma como causa do silenciamento Petunia mutata : sem cor (sem o gene A 1): Introdução gene A 1 do milho: cópia simples: Flor torna-se pigmentada Adição da segunda cópia de A 1: transcrição silenciada: herdável geneticamente Variegação: Silenciamento da segunda cópia Gene silenciado A 1 pode induzir silenciamento epigenético de outro gene A 1 novo, somente quando esse gene A 1 expresso é inserido próximo do primeiro.

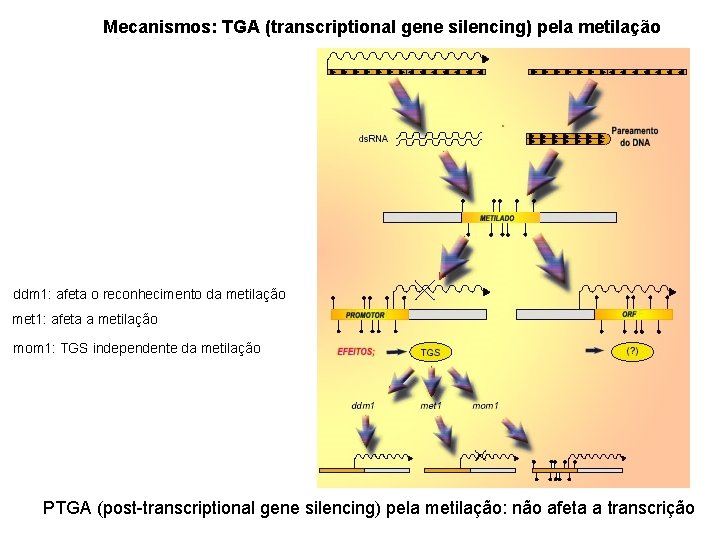
Mecanismos: TGA (transcriptional gene silencing) pela metilação ddm 1: afeta o reconhecimento da metilação met 1: afeta a metilação mom 1: TGS independente da metilação PTGA (post-transcriptional gene silencing) pela metilação: não afeta a transcrição

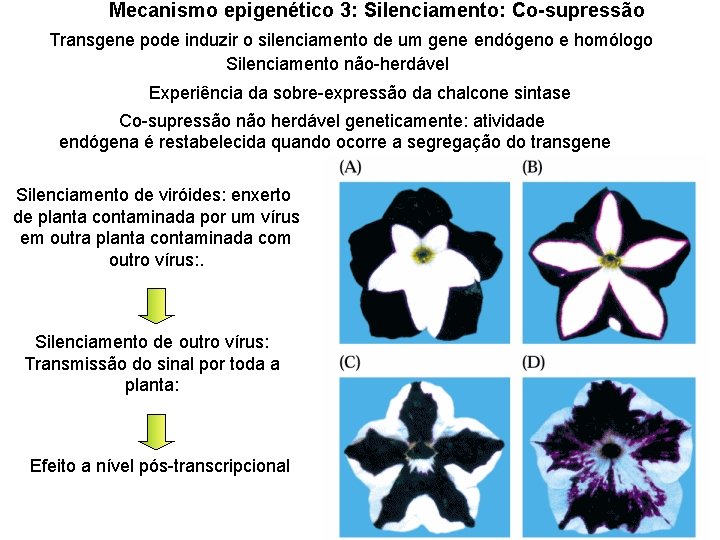
Mecanismo epigenético 3: Silenciamento: Co-supressão Transgene pode induzir o silenciamento de um gene endógeno e homólogo Silenciamento não-herdável Experiência da sobre-expressão da chalcone sintase Co-supressão não herdável geneticamente: atividade endógena é restabelecida quando ocorre a segregação do transgene Silenciamento de viróides: enxerto de planta contaminada por um vírus em outra planta contaminada com outro vírus: . Silenciamento de outro vírus: Transmissão do sinal por toda a planta: Efeito a nível pós-transcripcional

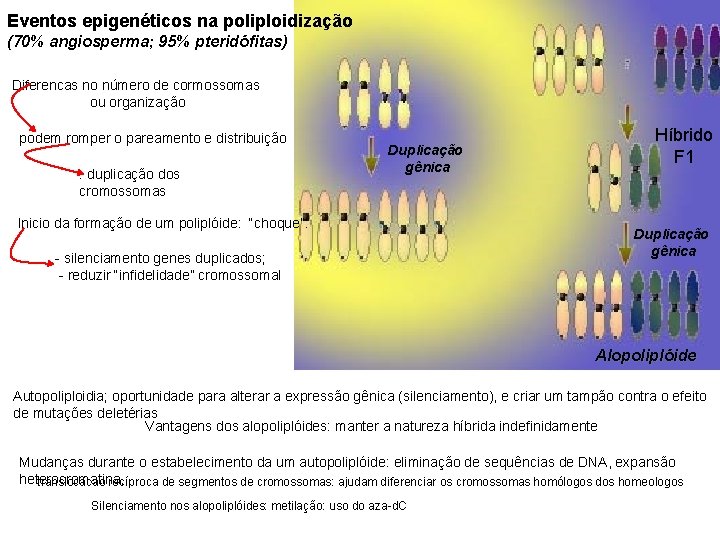
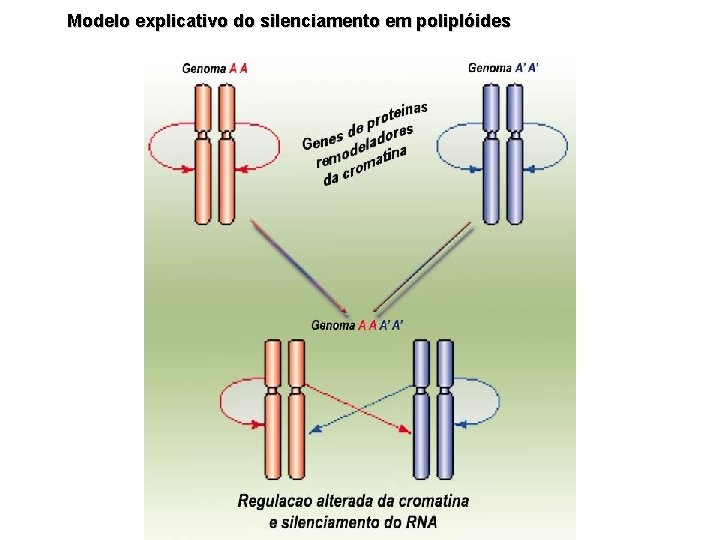
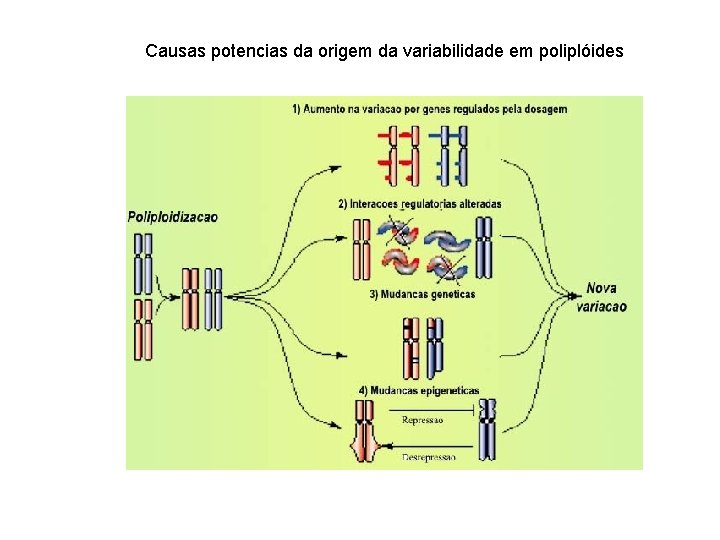
Eventos epigenéticos na poliploidização (70% angiosperma; 95% pteridófitas) Diferencas no número de cormossomas ou organização podem romper o pareamento e distribuição Duplicação gênica : duplicação dos cromossomas Inicio da formação de um poliplóide: “choque”: - silenciamento genes duplicados; - reduzir “infidelidade” cromossomal Híbrido F 1 Duplicação gênica Autopoliploide Alopoliplóide Autopoliploidia; oportunidade para alterar a expressão gênica (silenciamento), e criar um tampão contra o efeito de mutações deletérias Vantagens dos alopoliplóides: manter a natureza híbrida indefinidamente Mudanças durante o estabelecimento da um autopoliplóide: eliminação de sequências de DNA, expansão heterocromatina, translocacao recíproca de segmentos de cromossomas: ajudam diferenciar os cromossomas homólogos dos homeologos Silenciamento nos alopoliplóides: metilação: uso do aza-d. C

Modelo explicativo do silenciamento em poliplóides

Causas potencias da origem da variabilidade em poliplóides

Seleção de mutações que afetam o controle das alterações epigenéticas ddm 1: mutação no gene (SNW/SNF 2) : fator de remodelação da cromatina; Mamíferos mudanças na estrutura da cromatina devem anteceder a metilação Sem fenótipo visível; autofecundação: Paradoxo? progenia anormal: : desestabilização da programação epigenética reduzido nível de metilação : : nova metilação e silenciamento de genes que são normalmente expressos e não metilados (SUPERMAN): flores anormais Plantas antisenso para MET 1; redução em 30% no nível de metilação Hipermetilação do gene SUPERMAN e do gene AG Hipermetilação do gene SUPERMAN Problema para a obtenção de plantas transgênicas: - súbito silenciamento do transgenes 1 a defesa para superar este problema: selecionar planta transgênica com cópia unica

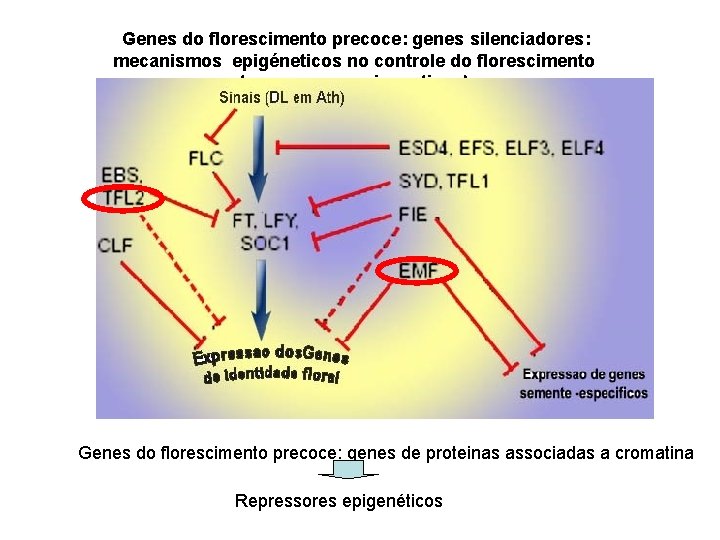
Genes do florescimento precoce: genes silenciadores: mecanismos epigéneticos no controle do florescimento (repressores epigeneticos) Genes do florescimento precoce: genes de proteinas associadas a cromatina Repressores epigenéticos

Mutantes tfl 2, emf: 1) florescem precocemente, 2) florescem sob DC, flor terminal, 3) proteínas de reserva da semente são expressas antes 35 S: TFL 1 4 sépalas nova influorescência 35 S: EMF 1 + DL duas folhas caulinares flor terminal (2 silíquas) novo broto terminal 35 S: EMF 1 + DC flores e brotos originados na mesma regiao Importância da remodelagem da cromatina no controle da expressão gênica e dos processos de desenvolvimento Pouco compeendido como esses fatores de remodelagem da cromatina funcionam em plantas


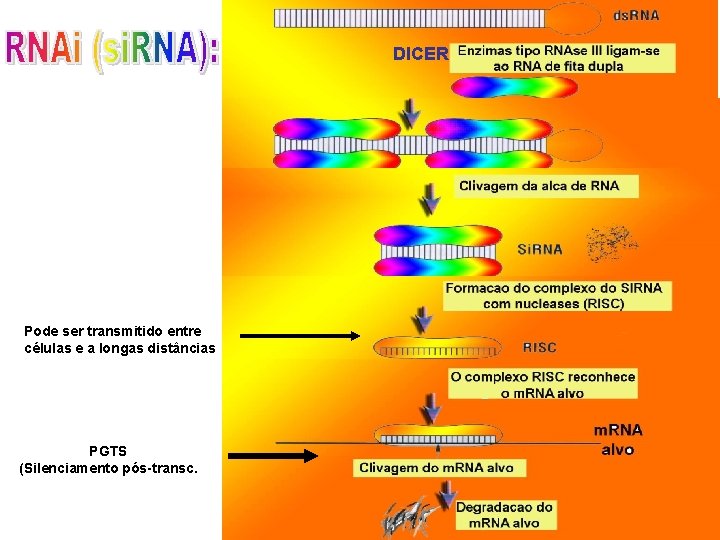
PGTS (post-transcriptional gene silencing): primeiramente descoberto em plantas Primeiro gene regulatório para PGTS foi descoberto em Neurospora Mediado por moléculas de RNA pequenas (<70 nucleotídeos) PGTS: um poderoso meio para manipular a expressao em sistemas animais: : RNAi PGTS: mais conhecido em Drosophila e levedura (Saccharomyces) Plantas usam as mudancas epigenéticas para se adaptar a mudancas genômicas ou ambientais: : flexibilidade Flexibilidade: reprogramação dos estágios de desenvolvimento Funções: 1) proteção contra efeitos deletérios da transposição de transposons 2) Resistência contra viroses (Produzem ds. RNA)

DICER Pode ser transmitido entre células e a longas distâncias PGTS (Silenciamento pós-transc.

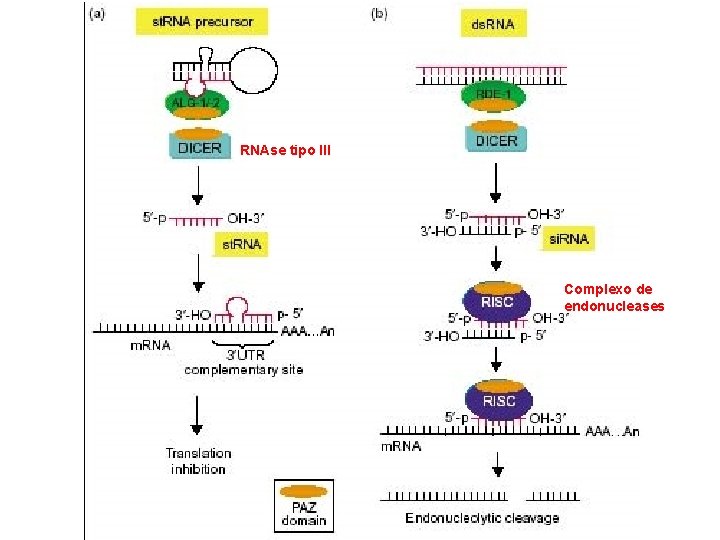
RNA curto de interferência (si. RNA) Dupla fita Micro RNA (mi. RNA) Perfeitamente complementar ao RNA alvo Direciona a clivagem endonucleolítica RNA dupla fita perfeitamente ou quase perfeitamente pareado Não conservado entre as espécies Originado de um RNA percursor de maior tamanho Cromossomal ou citoplásmico Comprimento médio de 25 nucleotídeos Fita simples Parcialmente complementar ao RNA alvo Mecanismo de ação desconhecido RNA dupla fita pareado, mas com alças Frequentemente conservado entre as espécies e filos Originado de um RNA percursor de maior tamanho Cromossomal (IGR) Tamanho médio de 70 nucleotídeos RNA curto temporal (st. RNA) Fita simples Parcialmente complementar ao RNA alvo Inibe a tradução do m. RNA dupla fita pareado, mas com alças Altamente conservado entre as espécies e filos Originado de um RNA percursor de maior tamanho Cromossomal (IGR) Tamanho médio de 70 nucleotídeos


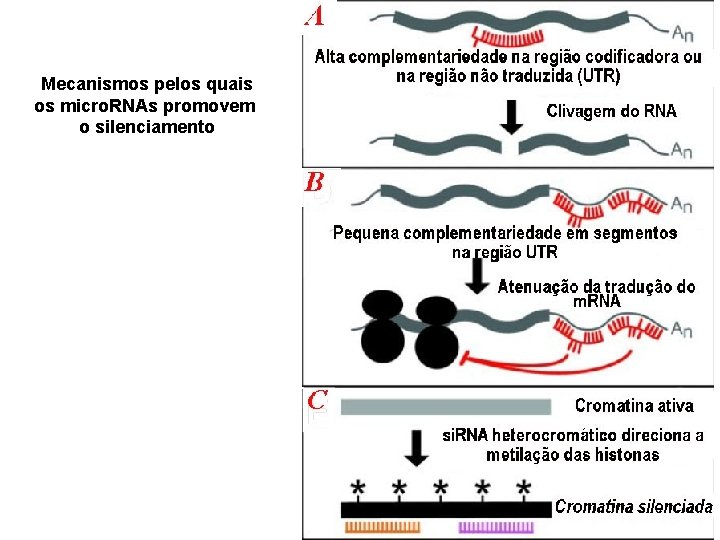
Mecanismos pelos quais os micro. RNAs promovem o silenciamento


RNAse tipo III Complexo de endonucleases


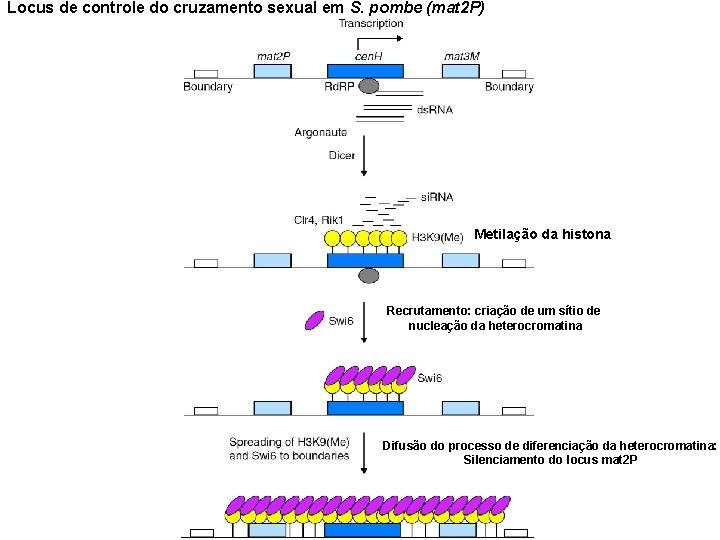
Locus de controle do cruzamento sexual em S. pombe (mat 2 P) Metilação da histona Recrutamento: criação de um sítio de nucleação da heterocromatina Difusão do processo de diferenciação da heterocromatina: Silenciamento do locus mat 2 P


61 alvos potenciais: 41 são fatores transcricionais Desen. floral Dicer Separação de orgãos Dif. Merist. Axilar e da folha Det. Da especificade de RISC: mutantes: arquit. mod Desen. floral Identidade floral

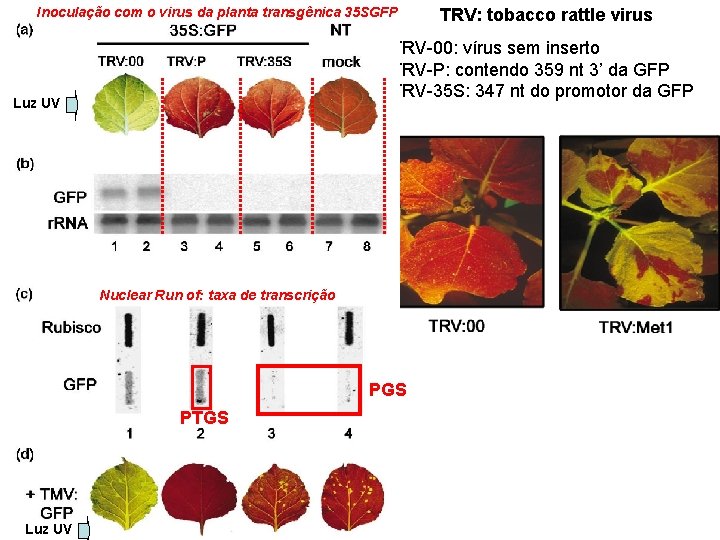
Inoculação com o virus da planta transgênica 35 SGFP TRV-00: vírus sem inserto TRV-P: contendo 359 nt 3’ da GFP TRV-35 S: 347 nt do promotor da GFP Luz UV Nuclear Run of: taxa de transcrição PGS PTGS Luz UV TRV: tobacco rattle virus

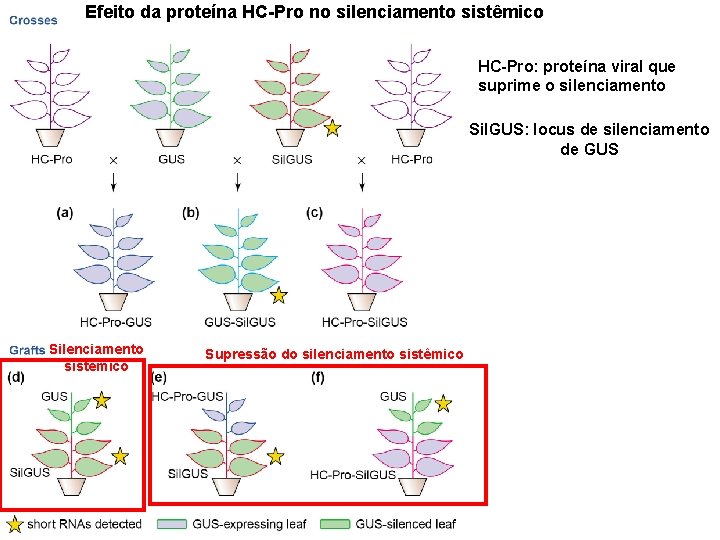
Efeito da proteína HC-Pro no silenciamento sistêmico HC-Pro: proteína viral que suprime o silenciamento Sil. GUS: locus de silenciamento de GUS Silenciamento sistêmico Supressão do silenciamento sistêmico

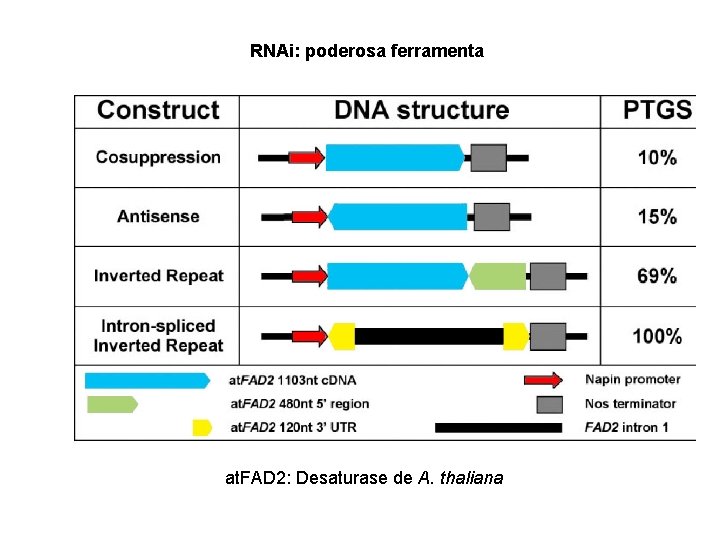
RNAi: poderosa ferramenta at. FAD 2: Desaturase de A. thaliana

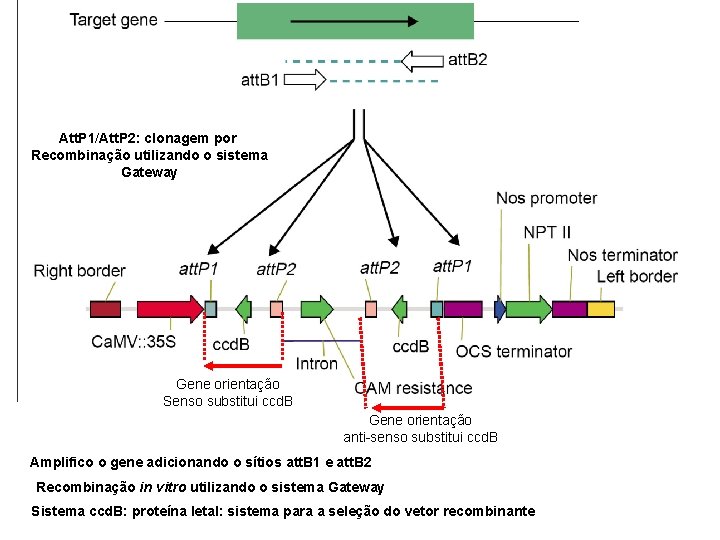
Att. P 1/Att. P 2: clonagem por Recombinação utilizando o sistema Gateway Gene orientação Senso substitui ccd. B Gene orientação anti-senso substitui ccd. B Amplifico o gene adicionando o sítios att. B 1 e att. B 2 Recombinação in vitro utilizando o sistema Gateway Sistema ccd. B: proteína letal: sistema para a seleção do vetor recombinante

RNAi

 Epigentico
Epigentico Epigentica
Epigentica Tcoffee expresso
Tcoffee expresso Plantas sin semillas briofitas
Plantas sin semillas briofitas Gnica
Gnica Gnica
Gnica Gnica
Gnica Gnica
Gnica Gnica
Gnica Gnica
Gnica Gnica
Gnica Control pretranscripcional
Control pretranscripcional Gnica
Gnica Arn polimerasa
Arn polimerasa Gnica
Gnica Gnica
Gnica Catabolica
Catabolica Gnica
Gnica Flui
Flui Controle
Controle Controle pid
Controle pid Liste des bureaux de contrôle agréés en tunisie 2021
Liste des bureaux de contrôle agréés en tunisie 2021 Sondage audit
Sondage audit Automao
Automao Controle orçamentário
Controle orçamentário Integration terrain non controlé
Integration terrain non controlé Conceito de controle administrativo
Conceito de controle administrativo Facteur rare controle de gestion
Facteur rare controle de gestion Masse salariale
Masse salariale Liste des produits soumis au contrôle technique
Liste des produits soumis au contrôle technique Sistema de controle
Sistema de controle Sistemas de controle
Sistemas de controle Contrôle de gestion
Contrôle de gestion Controle do desenvolvimento microbiano nos alimentos
Controle do desenvolvimento microbiano nos alimentos Objetivos de la temperatura corporal
Objetivos de la temperatura corporal Instrumentos de planejamento e controle financeiro
Instrumentos de planejamento e controle financeiro Contrôle financier
Contrôle financier Transitria
Transitria Sistemas de controle
Sistemas de controle Equação diferencial
Equação diferencial