Introduccin al Diseo de Primers Lic Giorgio Martn


































- Slides: 46

Introducción al Diseño de Primers Lic. Giorgio Martín Curso: Introducción a la Bioinformática. Laboratorio de Biotecnología Molecular. Módulo de Bioquímica y Farmacia. Facultad de Ciencias Exactas, Químicas y Naturales. UNa. M. Agosto – Septiembre 2010

Introducción al Diseño de Primers – Dogma central de la biología. – Código genético. – Técnica de la Reacción en Cadena de la Polimerasa (PCR). – Diseño de cebadores.

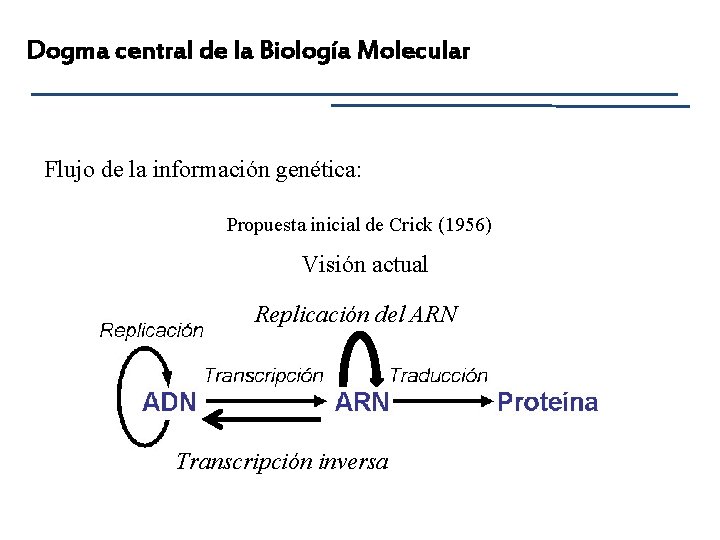
Dogma central de la Biología Molecular Flujo de la información genética: Propuesta inicial de Crick (1956) Visión actual Replicación del ARN Transcripción inversa

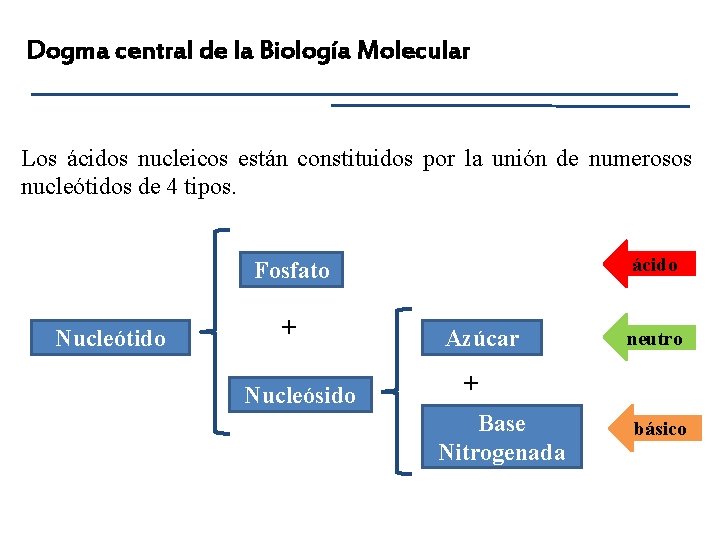
Dogma central de la Biología Molecular Los ácidos nucleicos están constituidos por la unión de numerosos nucleótidos de 4 tipos. ácido Fosfato Nucleótido + Nucleósido Azúcar neutro + Base Nitrogenada básico

Dogma central de la Biología Molecular

Dogma central de la Biología Molecular

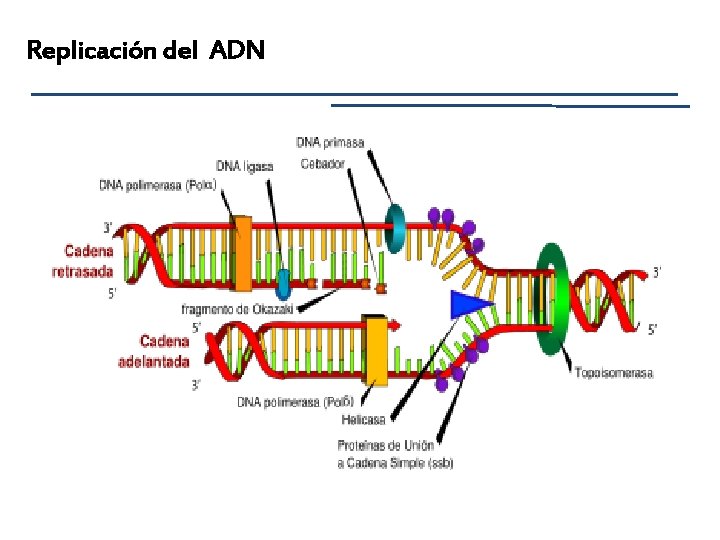
Replicación del ADN

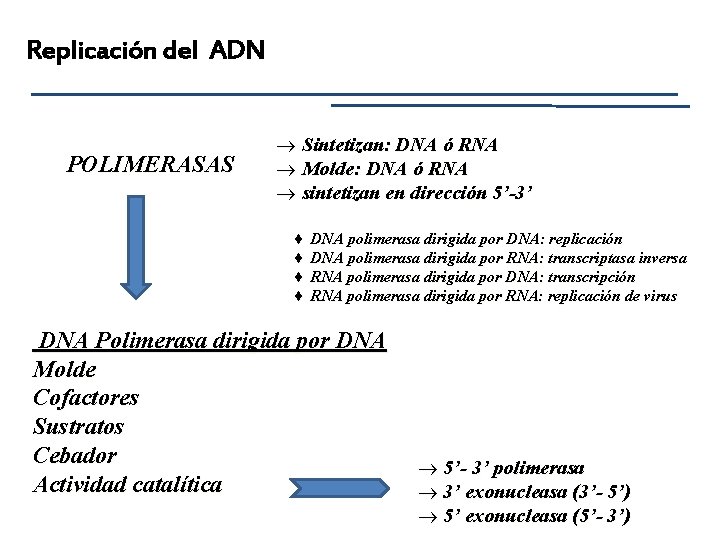
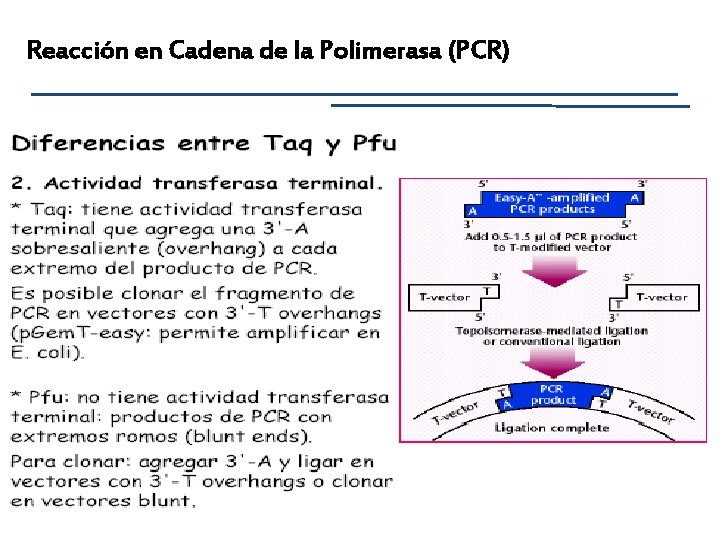
Replicación del ADN POLIMERASAS ® Sintetizan: DNA ó RNA ® Molde: DNA ó RNA ® sintetizan en dirección 5’-3’ ¨ DNA polimerasa dirigida por DNA: replicación ¨ DNA polimerasa dirigida por RNA: transcriptasa inversa ¨ RNA polimerasa dirigida por DNA: transcripción ¨ RNA polimerasa dirigida por RNA: replicación de virus DNA Polimerasa dirigida por DNA Molde Cofactores Sustratos Cebador Actividad catalítica ® 5’- 3’ polimerasa ® 3’ exonucleasa (3’- 5’) ® 5’ exonucleasa (5’- 3’)

Reacción en Cadena de la Polimerasa (PCR) – Es una técnica que permite amplificar in vitro segmentos de ADN específicos, mediante ciclos repetidos de síntesis dirigida por cebadores o primers. – Inventada en 1983 por Kary B. Mullis. – Se usa: Producción de Sondas. Secuenciación. Clonación. Estudios Clínicos: Detección de Infecciones Bacterianas y Virales. Diagnóstico de Enfermedades Hereditarias. Estudios de Evolución Molecular y Filogenia. Estudios de Medicina Forense. Estudios de Filiación.

Reacción en Cadena de la Polimerasa (PCR)

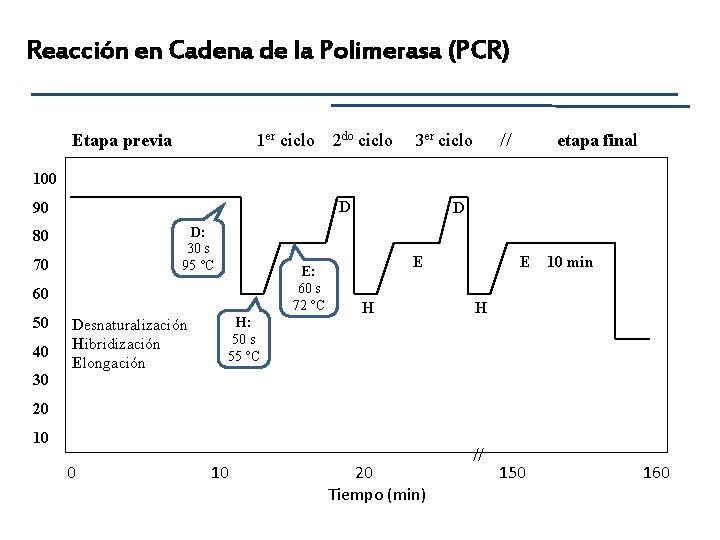
Reacción en Cadena de la Polimerasa (PCR) Etapa previa 1 er ciclo 2 do ciclo // 3 er ciclo etapa final 100 D 90 D: 30 s 95 °C 80 70 E: 60 s 72 °C 60 50 40 30 Desnaturalización Hibridización Elongación H: 50 s 55 °C D E H E 10 min H 20 10 20 Tiempo (min) // 150 160

Reacción en Cadena de la Polimerasa (PCR) Si repetimos este ciclo (desnaturalización, hibridización, extensión) muchas veces logramos una amplificación exponencial

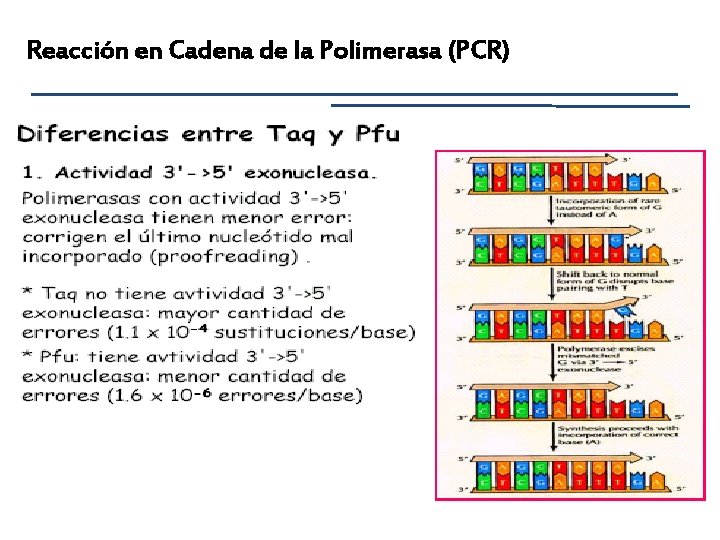
Reacción en Cadena de la Polimerasa (PCR) Especificidad: generación de un único producto de amplificación. Eficiencia: máxima producción de amplificación en función del número de ciclos. Fidelidad: número de errores que comete la ADN polimerasa durante la copia. Sensibilidad: mínima cantidad de ADN que se necesita para obtener una gran cantidad de copias.

Reacción en Cadena de la Polimerasa (PCR)

Reacción en Cadena de la Polimerasa (PCR)

Reacción en Cadena de la Polimerasa (PCR)

Diseño de Primers – El diseño cuidadoso de primers es uno de los aspectos más importantes de la PCR. – Primers mal diseñados pueden amplificar otros fragmentos de ADN: amplificación inespecífica. – Que el producto tenga el tamaño correcto no necesaria// mente indica que la secuencia es correcta.

Diseño de Primers En el diseño de los mismos algunas reglas se han demostrado como útiles, por ejemplo: I – Cada primer debe contar con una longitud de 18 -24 bases. II – Se debe mantener un contenido de G: C entre 40 y 60 %. III – Los dos primers del par deben de tener temperatura de fusión “Tm” cercanos, dentro de los 5 °C. IV – La secuencia de los primers individuales debe iniciarse y terminarse con 1 o 2 bases púricas. V – Evitar regiones con potencialidad para formar estructuras secundarias internas. VI – Evitar poli X.

Diseño de Primers VII – Secuencias adicionales pueden ser agregadas en el extremo 5’ del primer (no incluir cuando se estima la Tm del primer). VIII – Se pueden agregar degeneraciones en algunas posiciones del primer: a - Se incrementa el riesgo de amplificación inespecífica. b - Se disminuye la concentración en la mezcla de cada uno de los primers posibles. c - No se recomienda utilizar más de 64 primers diferentes en la mezcla. d - Código IUPAC/IUB:

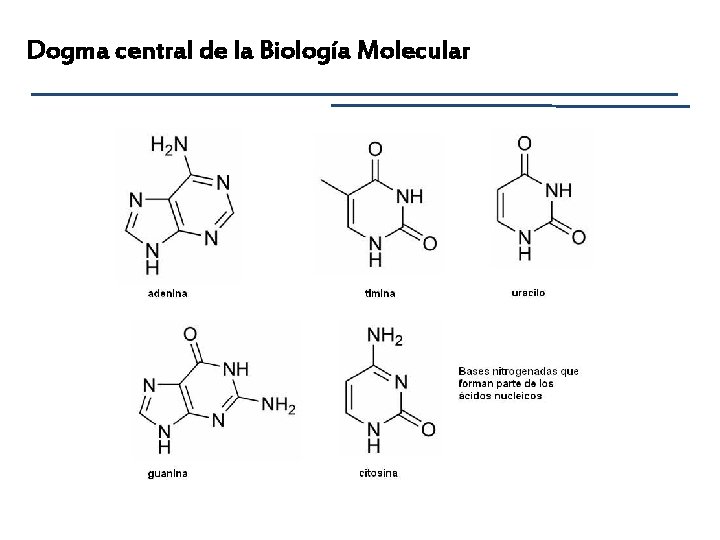
Diseño de Primers Código IUPAC/IUB: A C G T N M V adenina citosina guanina timidina en el ADN uracilo en el ARN Ao. Co. Go. T Ao. C A o C o G; no T R W S Y K H B D Ao. G Ao. T Co. G Co. T Go. T A o C o T; no G C o G o T; no A A o G o T; no C

Diseño de Primers I – Longitud del primer. 18 – 24 nucleótidos Tm óptima. Muy largos: Fallas en el hibridización > bajo rendimiento. II – Tm (T melting). Tm: Temperatura a la cual la mitad de las bases del primer están apareadas con el ADN molde. Tm = 2 (A + T) + (G + C) > Tm aumenta con la longitud del primer y el contenido de (G + C). Tm: 55 – 60 °C – 2 primers: Tm similares. T hibridización (T annealing) depende de la Tm de los primers > 5 °C por debajo de la Tm más baja del par de primer (T annealing: al menos 50 °C).

Diseño de Primers III – Especificidad. Reconocer una secuencia única dentro del ADN templado. Especificidad de un primer depende de la longitud (más largo > menos probabilidad de encontrar 2 secuencias iguales en el ADN templado).

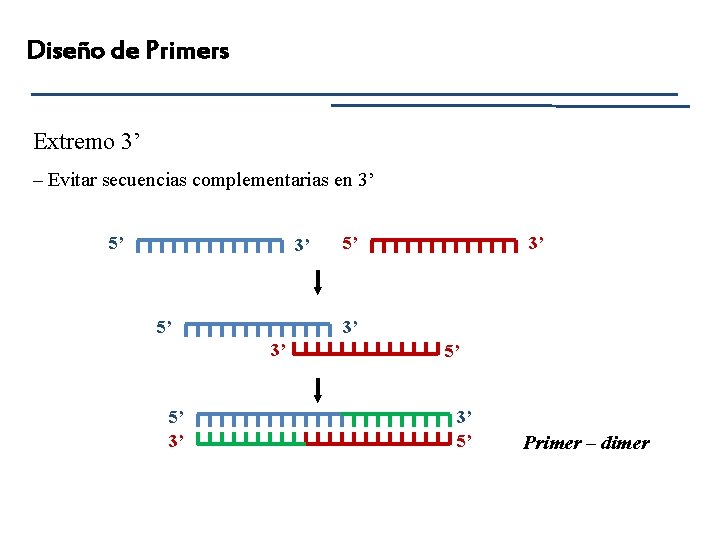
Diseño de Primers IV – Complementariedad de Secuencias. Sin complementariedad intra– o inter– primers. * Complementariedad intra – primer (más de 3 bases): Se pliega (estructuras de doble cadena) > Interfiere con el anneling al ADN templado. * Complementariedad inter – primer: Dímeros disminuye la formación de productos por competencia (peor en extremos 3’, donde se une la ADN polimerasa).

Diseño de Primers IV – Complementariedad de Secuencias. Extremo 3’. – Evitar más de dos G y/o C en los últimos nucleótidos. G o C asegura la unión del primer al molde en el extremo 3’ (unión más fuerte que A/T) > Aumenta la eficiencia de la reacción. – Evitar una T en la última base del extremo 3’. – Evitar mismatches en el extremo 3’.

Diseño de Primers Extremo 3’ – Evitar secuencias complementarias en 3’ 5’ 3’ 3’ 3’ 5’ 5’ 3’ 5’ Primer – dimer

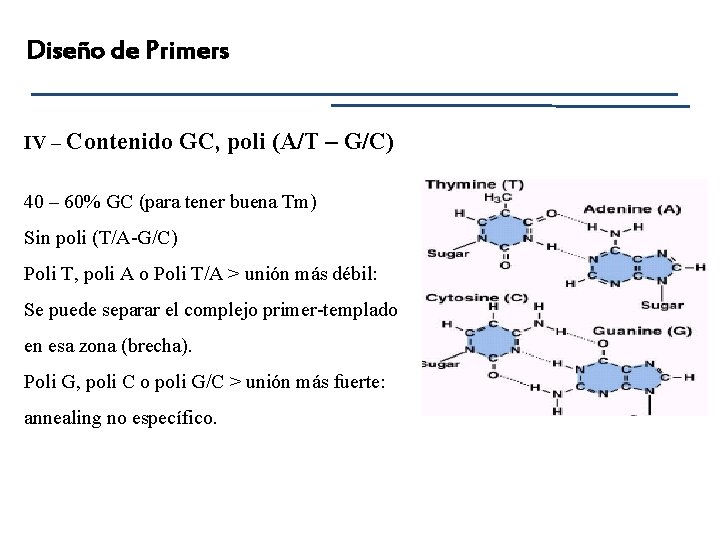
Diseño de Primers IV – Contenido GC, poli (A/T – G/C) 40 – 60% GC (para tener buena Tm) Sin poli (T/A-G/C) Poli T, poli A o Poli T/A > unión más débil: Se puede separar el complejo primer-templado en esa zona (brecha). Poli G, poli C o poli G/C > unión más fuerte: annealing no específico.

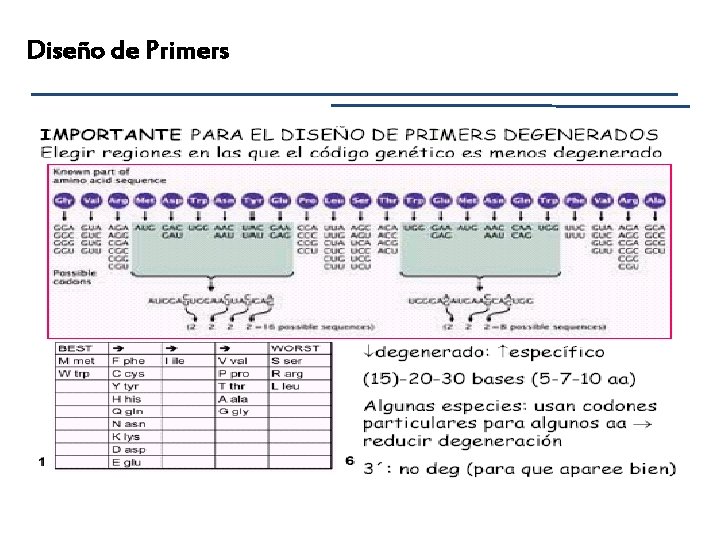
Diseño de Primers degenerados: – Se usan cuando quiero amplificar un gen y solo conozco su secuencia de aminoácidos. – Se usan secuencias que estén conservadas para una familia de proteínas. – Se usa secuencia con menor cantidad de codones posibles. – Se realizan todos los primers posibles para unirlos a la secuencia de interés – Se incluyen sitios de restricción para clonar posteriormente.

Diseño de Primers

Diseño de Primers

Diseño de Primers Ejemplo de primers degenerados: CTTCCTGAACTTGCTCTTCG CTTCCTGAACTTGGTCTTCG CTTCCTAAAATTGGTCTTCG ****** ** ****** 5’ – CTT CCT RAA MTT GST CTT CG – 3’ (primer sentido) R: G, A M: C, A S: C, G

Diseño de Primers Ejemplo de primers degenerados: – Índice de degeneración o degeneración general: Producto de la degeneración en cada nucleótido del primer – En el caso del ejemplo anterior: Índice de degeneración: 8. Solamente 1/8 de los primers van a hibridizar exactamente con el templado

Diseño de Primers – Se pueden utilizar apareamiento de bases “wobble” para evitar agregar demasiadas degeneraciones – Son apareamientos entre bases diferentes de los tradicionales Watson – Creek Ejemplos: G: T G: U

Diseño de Primers Apareamiento de bases “Wobble”

Diseño de Primers En el caso del ejemplo anterior, podemos reducir el índice de 8 a 4 agregando un nucleótido “wobble” (siempre cercanos al extremo 5’): 5’ – CTT CCT RAA MTT GST CTT CG – 3’ (primer sentido) R: G, A M: C, A S: C, G 5’ – CTT CCT GAA MTT GST CTT CG – 3’

Diseño de Primers – Si tengo C o T en la hebra utilizada como templado: En este caso elijo G en el primer, porque G puede hibridizar con C o con T – Si tengo A o G en la hebra utilizada como templado: Elijo T en el primer, porque T puede hibridizar con A o con G

Diseño de primers partir de una secuencia única de nucleótidos – Utilizaremos Primer 3 – Necesitaremos cortar y pegar una secuencia de nucleótidos – Tendremos que seleccionar el tamaño del producto deseado: máximo, óptimo, mínimo – Seleccionar las características de los primers


Diseño de primers Ejemplo de diseño con Primer 3 La secuencia corresponde al ARN mensajero que codifica para el gen Gurken en Drosophila melanogaster, número de acceso NM_057220. 3.

Diseño de primers partir de un alineamiento múltiple de secuencias proteicas – Utilizaremos el programa CODEHOP – Necesitaremos un alineamiento múltiple de secuencias proteicas – CODEHOP usa bloques de alineamiento múltiple de secuencias de aminoácidos que son distantes – Divide al primer en dos bloques: – Un bloque 5´ no degenerado – Un bloque 3´ degenerado


Diseño de primers partir de un alineamiento múltiple de secuencias proteicas 5´ ------------========= 3´ non–degenerate consensus clamp degenerate core

Diseño de primers

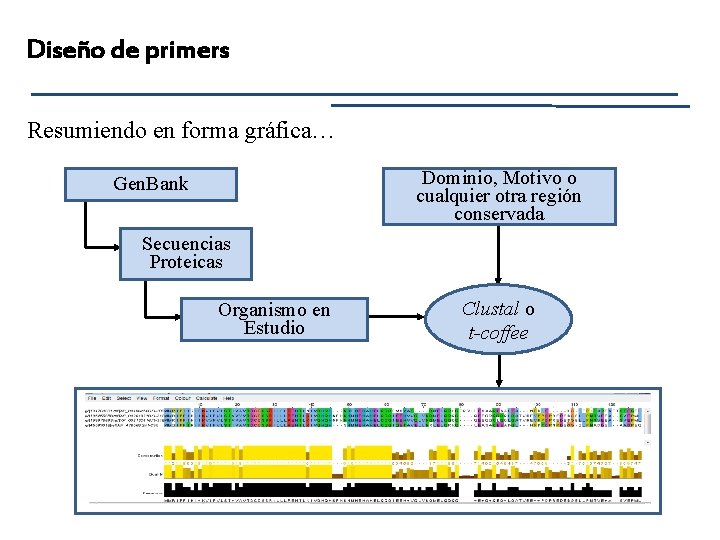
Diseño de primers Resumiendo en forma gráfica… Dominio, Motivo o cualquier otra región conservada Gen. Bank Secuencias Proteicas Organismo en Estudio Clustal o t-coffee

Diseño de primers Resumen… – Longitud de 18 a 24 nucleótidos. – Específicos. – Tm = 55 a 75 °C, con Tm Similares entre sí. – Diseñar primers con cantidad aproximadamente 50% de G: C y distribución al azar. – Evitar primers con secuencias de polipurinas y/o polipirimidinas (poly GC: disminuye especificidad – poly AT: disminuye eficiencia). – Evitar secuencias que formen estructuras secundarias. – Controlar que los primers no hibridicen entre sí. – Evitar más de 3 CG seguidas en el extremo 3´ – Comenzar y terminar con purinas – Existen programas como el Primer 3 y CODEHOP para el diseño de primers.

Diseño de primers – Para evaluar las características del los cebadores diseñados se pueden analizar con los programas Fast. PCR y VNTRI de Invitrogen

Diseño de primers Preguntas…? Dudas…? MUCHAS GRACIAS POR SU ATENCIÓN!!!
 Las seis cuerdas federico garcia lorca wikipedia
Las seis cuerdas federico garcia lorca wikipedia Test de kostick
Test de kostick Diego martn
Diego martn 05.html?title=
05.html?title= Costos mixtos
Costos mixtos Diseo de interiores
Diseo de interiores Portafolio de diseo
Portafolio de diseo Diseo grafico
Diseo grafico Introduccin
Introduccin Induccin
Induccin Introduccin
Introduccin Introducción de las enzimas
Introducción de las enzimas Introduccin
Introduccin Introduccin
Introduccin Minipcr antibiotic resistance lab answers
Minipcr antibiotic resistance lab answers Dna primers
Dna primers Diseño de primers degenerados
Diseño de primers degenerados Edip a colonna
Edip a colonna Nombres naturals i enters
Nombres naturals i enters Els primers freds
Els primers freds Temperatura de melting primers
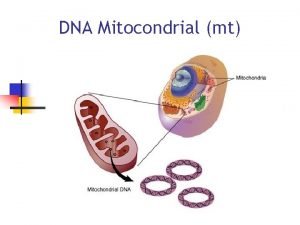
Temperatura de melting primers Las mitocondrias
Las mitocondrias Dna primers
Dna primers Dimeros de primers
Dimeros de primers Giorgio casadei
Giorgio casadei Segundo john rawls a noção de justiça comporta
Segundo john rawls a noção de justiça comporta Vitreolisi farmacologica
Vitreolisi farmacologica Giorgio turchetti
Giorgio turchetti Lo conosci a giorgio
Lo conosci a giorgio I and the village chagall
I and the village chagall Giorgio zanetti unimore
Giorgio zanetti unimore Retinoschisi giovanile x-linked
Retinoschisi giovanile x-linked 5000794318
5000794318 Piazza d'italia giorgio de chirico analysis
Piazza d'italia giorgio de chirico analysis Istituto comprensivo castel san giorgio lanzara
Istituto comprensivo castel san giorgio lanzara Barbara giorgio psychologist
Barbara giorgio psychologist Giorgio joyce
Giorgio joyce Giorgio cannavicci
Giorgio cannavicci Giorgio ganis
Giorgio ganis Giorgio caproni litania
Giorgio caproni litania Giorgio letta
Giorgio letta Dowell curves
Dowell curves Giorgio ghelli
Giorgio ghelli Da vinci de giorgio
Da vinci de giorgio Giorgio gribaudo
Giorgio gribaudo Vasari dante
Vasari dante Giorgio donnarumma
Giorgio donnarumma