CAPTULO 5 EL TRATAMIENTO DE DATOS EN ANTROPOGENTICA

















- Slides: 38

CAPÍTULO 5. EL TRATAMIENTO DE DATOS EN ANTROPOGENÉTICA La ley de equilibrio Hardy-Weinberg La población subdividida y el efecto Wahlund La similaridad genética El análisis estadístico de la similaridad Otros métodos estadísticos útiles en Antropogenética Bases de datos Programas de interés en Antropogenética

Bibliografía complementaria Calderon R, Vidales C, Peña JA, Perez-Miranda A, Dugoujon JA 1998 Immunoglobulin allotypes (GM and KM) in Basques from Spain: Approach to the Origin of the Basque Population. Human Biology, 70: 667 -698 (JAP 790) Calò, C. M. , Piras, I. S. , Moral, P. , Falchi, A. , Ghiani, M. E. , Varesi, L. , Vona, G. , 2005, Analisi molecolare delle popolazioni del Mediterraneo attraverso 11 inserzioni Alu. Antropo, 9, 1 -12. www. didac. ehu. es/antropo Cerny V. , Hajek M. , Bruzek J. , Cmejla R. , Brdicka R. , 2004, Relations génétiques des populations de langues tchadiques parmi les populations péri-sahariennes révélées par l’étude des séquences de l’ADN mitochondriale. Antropo, 7, 123 -131. www. didac. ehu. es/antropo Chen et a 2000 mt. DNA Variation in the South African Kung and Khwe and Their Genetic Relationships to Other African Populations Am. J. Hum. Genet. , 66: 1362 -1383 (JAP 1323) Coudray, C. , Guitard, E. , Lemaire, O. , Cherkaoui, M. , Baali, A. , Hilali, K. , Sevin, A. , Kandil, M. , Harich, N. , Melhaoui, M. , Larrouy, G. , Moral, P. , Dugoujon, J. M. , 2004, Les allotypes Gm des immunoglobulines chez les Berbères du Maroc. Antropo, 6, 63 -69. www. didac. ehu. es/antropo Goddard et al 2000 Linkage Disequilibrium and Allele-Frequency Distributions for 114 Single-Nucleotide Polymorphisms in Five Populations Am J Hum Genet 66: 216 -234 (JAP 1325) Jorde et al 2000 The distribution of human genetic diversity: a comparison of mitochondrial, autosomal, and Ychromosome data Am J Hum Genet 66: 979 -988 (JAP 783) Peña et al 1997 New method for comparing levels of microdifferentiation: Application to migration matrices of two populations from the Basque Country (Spain) Human Biology 69: 329 -344 (JAP 787) Peña, J. A. , Alfonso-Sánchez, M. A. , García-Obregón, S. , Pérez-Miranda, A. , 2002, Persistencia de actividad lactasa en población residente en el País Vasco. Antropo, 3, 51 -60. www. didac. ehu. es/antropo Peña et al 2002 Microsatellite DNA markers from HLA region (D 6 S 105, D 6 S 265 and TNFa) in autochthonous Basques from Northern Navarre (Spain) Annals of Human Biology 29: 176 -191 Peña, J. A. , Alfonso-Sánchez, M. A. , Pérez-Miranda, A. , García-Obregón, S. , 2004, Flujo génico y presiones selectivas en Europa. Una aproximación a partir de clinas de ADN codificante y no codificante. Antropo, 8, 117 -122. www. didac. ehu. es/antropo Roewer et al 2005 Signature of recent historical events in the European Y-chromosomal STR haplotype distribution Human Genetics (JAP 1045) Tishkoff et al 2007 History of Click-Speaking Populations of Africa Inferred from mt. DNA and Y Chromosome Genetic Variation Mol Biol Evol 24: 2180 -2195 (JAP 1322)

Equilibrio Hardy-Weinberg Genotipo AA Aa aa Frecuencia p 2 2 pq q 2 Total 1

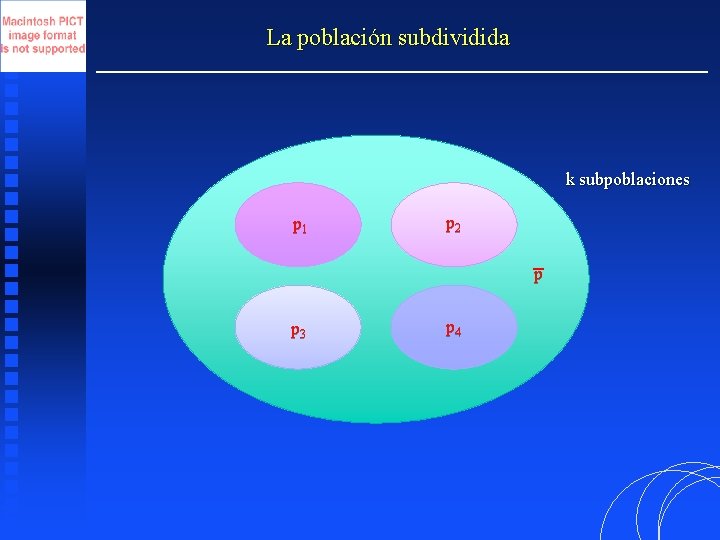
La población subdividida k subpoblaciones p 1 p 2 p p 3 p 4

La población subdividida El efecto Wahlund

La población subdividida Variación con el tiempo de de FST

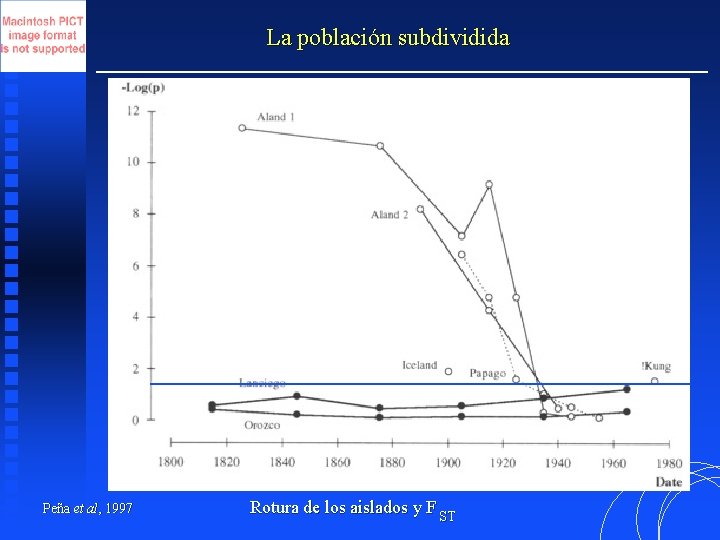
La población subdividida Peña et al, 1997 Rotura de los aislados y F ST

La similaridad genética RST = ∑i wi rii estima de Coeficiente de parentesco (matriz R)

La similaridad genética G 2 de Sanghvi Jxx=1 -hx Jyy=1 -hy D de Nei Matriz R FST de Reynolds et al Coeficientes de distancia

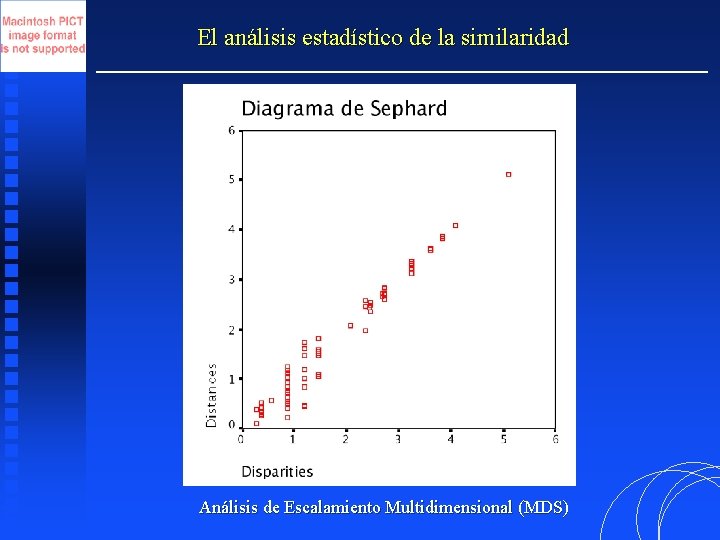
El análisis estadístico de la similaridad Con el fin de ajustar la matriz de disimilaridades D obtenida entre k muestras, a una representación euclídea en s dimensiones ( s ≤ k - 1 ), se asocia una matriz de distancias, de modo que dadas dos disimilaridades tal que dij < di'j' debe cumplirse, siempre que sea posible que las distancias representadas sean d*ij < d*i'j' De este modo, situando los M elementos de la matriz D en orden creciente di 1 j 1 < di 2 j 2 <. . . < di. Mj. M sus distancias asociadas deben quedar como d*i 1 j 1 < d*i 2 j 2 <. . . < d*i. Mj. M Pueden representarse las disimilaridades frente a sus distancias asociadas en el denominado diagrama de Sephard. Análisis de Escalamiento Multidimensional (MDS)

El análisis estadístico de la similaridad Análisis de Escalamiento Multidimensional (MDS)

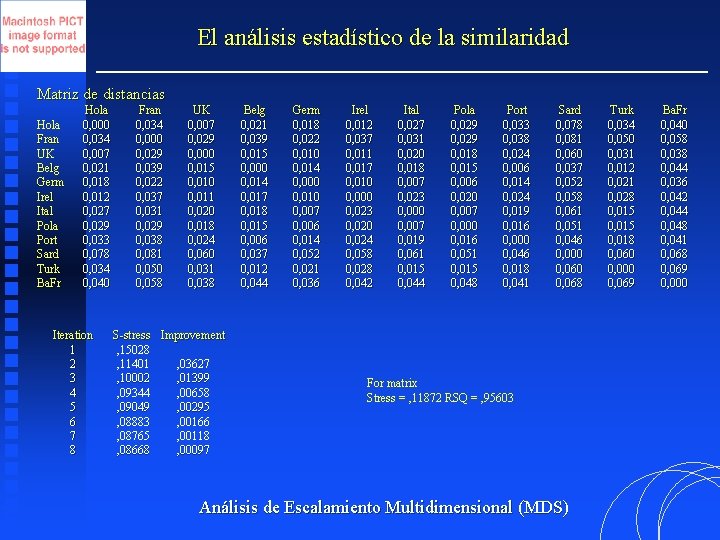
El análisis estadístico de la similaridad Matriz de distancias Hola Fran UK Belg Germ Irel Ital Pola Port Sard Turk Ba. Fr Hola 0, 000 0, 034 0, 007 0, 021 0, 018 0, 012 0, 027 0, 029 0, 033 0, 078 0, 034 0, 040 Iteration 1 2 3 4 5 6 7 8 Fran 0, 034 0, 000 0, 029 0, 039 0, 022 0, 037 0, 031 0, 029 0, 038 0, 081 0, 050 0, 058 UK 0, 007 0, 029 0, 000 0, 015 0, 010 0, 011 0, 020 0, 018 0, 024 0, 060 0, 031 0, 038 S-stress Improvement , 15028 , 11401 , 03627 , 10002 , 01399 , 09344 , 00658 , 09049 , 00295 , 08883 , 00166 , 08765 , 00118 , 08668 , 00097 Belg 0, 021 0, 039 0, 015 0, 000 0, 014 0, 017 0, 018 0, 015 0, 006 0, 037 0, 012 0, 044 Germ 0, 018 0, 022 0, 010 0, 014 0, 000 0, 010 0, 007 0, 006 0, 014 0, 052 0, 021 0, 036 Irel 0, 012 0, 037 0, 011 0, 017 0, 010 0, 000 0, 023 0, 020 0, 024 0, 058 0, 028 0, 042 Ital 0, 027 0, 031 0, 020 0, 018 0, 007 0, 023 0, 000 0, 007 0, 019 0, 061 0, 015 0, 044 Pola 0, 029 0, 018 0, 015 0, 006 0, 020 0, 007 0, 000 0, 016 0, 051 0, 015 0, 048 Port 0, 033 0, 038 0, 024 0, 006 0, 014 0, 024 0, 019 0, 016 0, 000 0, 046 0, 018 0, 041 Sard 0, 078 0, 081 0, 060 0, 037 0, 052 0, 058 0, 061 0, 051 0, 046 0, 000 0, 068 For matrix Stress = , 11872 RSQ = , 95603 Análisis de Escalamiento Multidimensional (MDS) Turk 0, 034 0, 050 0, 031 0, 012 0, 021 0, 028 0, 015 0, 018 0, 060 0, 000 0, 069 Ba. Fr 0, 040 0, 058 0, 038 0, 044 0, 036 0, 042 0, 044 0, 048 0, 041 0, 068 0, 069 0, 000

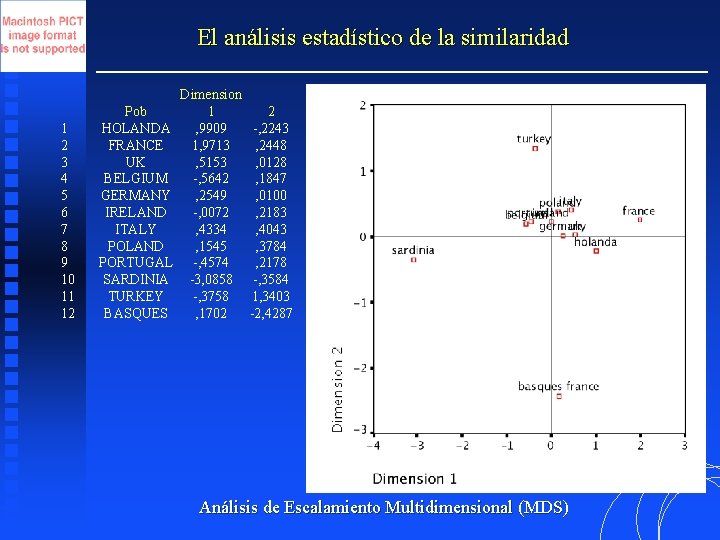
El análisis estadístico de la similaridad 1 2 3 4 5 6 7 8 9 10 11 12 Pob HOLANDA FRANCE UK BELGIUM GERMANY IRELAND ITALY POLAND PORTUGAL SARDINIA TURKEY BASQUES Dimension 1 , 9909 1, 9713 , 5153 -, 5642 , 2549 -, 0072 , 4334 , 1545 -, 4574 -3, 0858 -, 3758 , 1702 2 -, 2243 , 2448 , 0128 , 1847 , 0100 , 2183 , 4043 , 3784 , 2178 -, 3584 1, 3403 -2, 4287 Análisis de Escalamiento Multidimensional (MDS)

El análisis estadístico de la similaridad Ejemplo práctico 1 2 3 4 1 0 2 3 0, 4 0, 6 0 0, 2 0 4 0, 3 0, 2 0, 3 0 4 1 2 Análisis de Escalamiento Multidimensional (MDS) 3

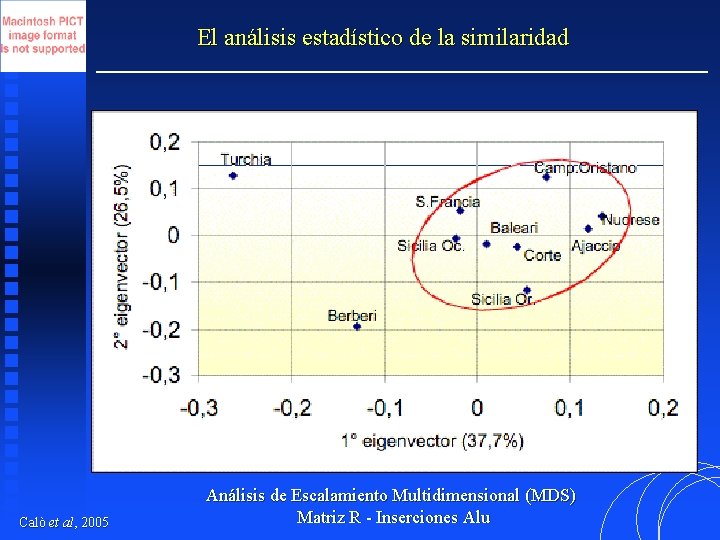
El análisis estadístico de la similaridad Calò et al, 2005 Análisis de Escalamiento Multidimensional (MDS) Matriz R - Inserciones Alu

El análisis estadístico de la similaridad Análisis Factorial de correspondencias (AFC) Distancia Chi 2

Los Bereberes Individuos beres

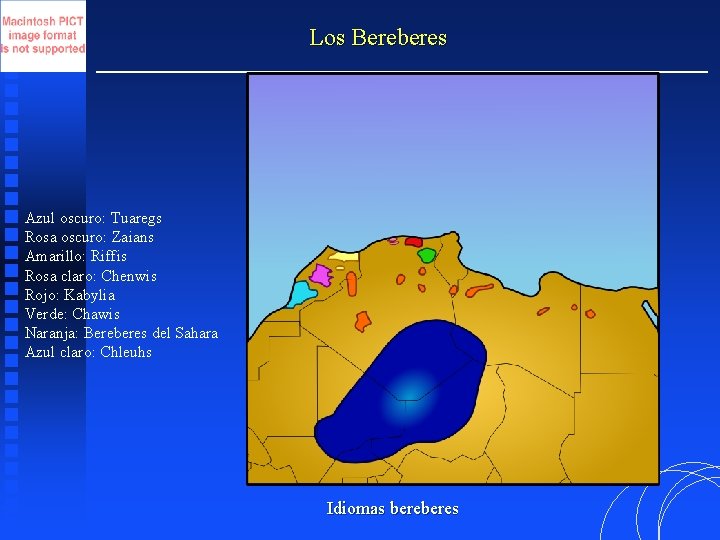
Los Bereberes Azul oscuro: Tuaregs Rosa oscuro: Zaians Amarillo: Riffis Rosa claro: Chenwis Rojo: Kabylia Verde: Chawis Naranja: Bereberes del Sahara Azul claro: Chleuhs Idiomas beres

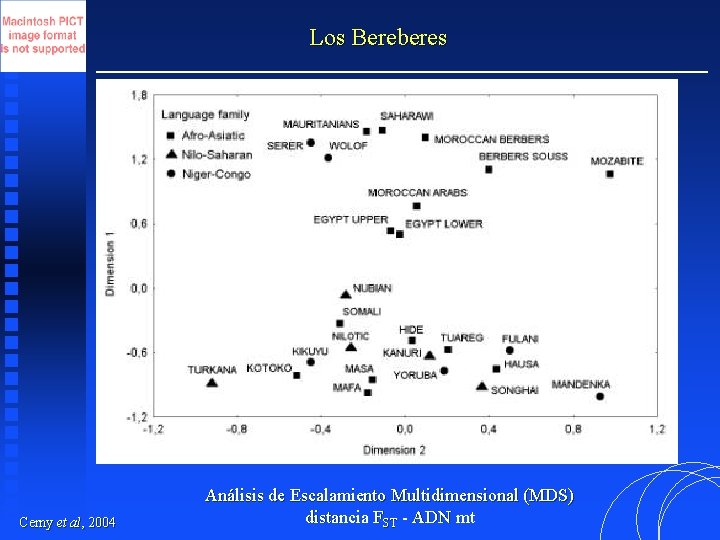
Los Bereberes Cerny et al, 2004 Análisis de Escalamiento Multidimensional (MDS) distancia FST - ADN mt

Los Bereberes

Los Bereberes 1 : Arabes de Doukkala (Maroc) ; 2 : Berbères de Amizmiz (Maroc) ; 3 : Berbères de Asni (Maroc) ; 4 : Berbères de Khenifra (Maroc) ; 5 : Berbères de Bourhia (Maroc) ; 6 : Berbères de Basse Kabylie ; 7 : Berbères de Haute Kabylie ; 8 : Berbères Mzab (Algérie) ; 9 : Berbères de Takrouna: Jeradou (Tunisie) ; 10 : Berbères de Douiret: Chenini (Tunisie) ; 11 : Berbères de Djerba (Tunisie) ; 12 : Touaregs Issequamarenes (Algérie) ; 13 : Amhara Tigrai (Ethiopie) ; 14 : Issas (Djibouti) ; 15 : Bwa (Mali) ; 16 : Mendenka (Sénégal) ; 17 : Fulani (Sénégal) ; 18 : Baoule (Côte d’Ivoire) ; 19 : Yorubas (Nigeria) ; 20 : Pygmées Aka (République Centrafrique). Coudray et al, 2004 Análisis Factorial de Correspondencias frecuencias GM

El análisis estadístico de la similaridad Método del mínimo Método del máximo Método de la media Dendrogramas

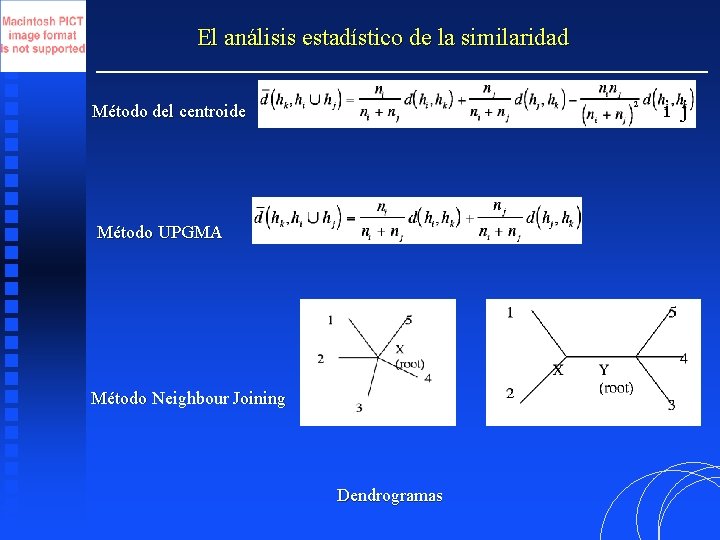
El análisis estadístico de la similaridad i j Método del centroide Método UPGMA Método Neighbour Joining Dendrogramas

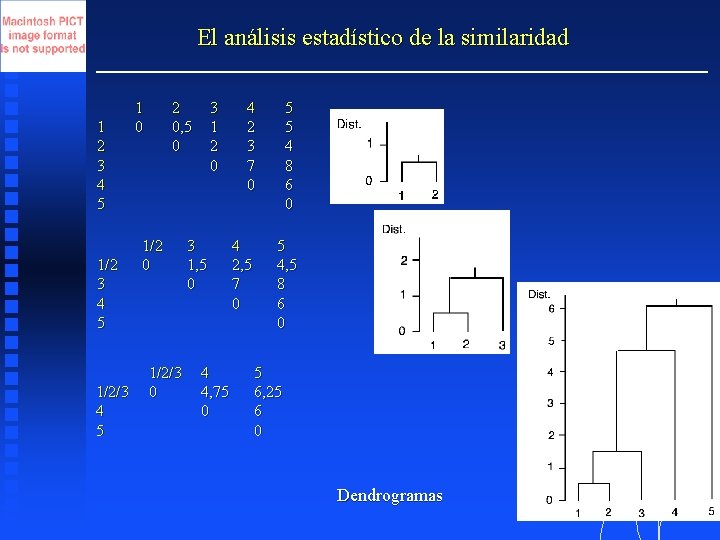
El análisis estadístico de la similaridad 1 2 3 4 5 1/2/3 4 5 1 0 2 0, 5 0 1/2/3 0 3 1 2 0 3 1, 5 0 4 4, 75 0 4 2 3 7 0 5 5 4 8 6 0 4 2, 5 7 0 5 4, 5 8 6 0 5 6, 25 6 0 Dendrogramas

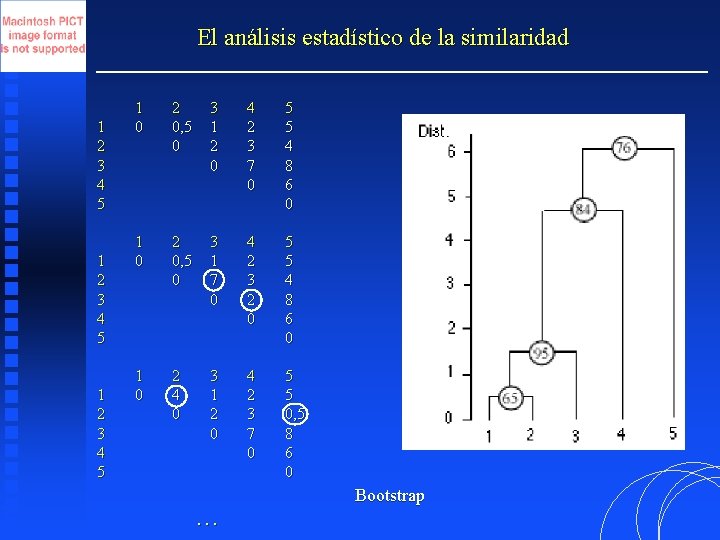
El análisis estadístico de la similaridad 1 2 3 4 5 1 0 2 0, 5 0 3 1 2 0 4 2 3 7 0 5 5 4 8 6 0 1 0 2 0, 5 0 3 1 7 0 4 2 3 2 0 5 5 4 8 6 0 1 0 2 4 0 3 1 2 0 4 2 3 7 0 5 5 0, 5 8 6 0 … Bootstrap

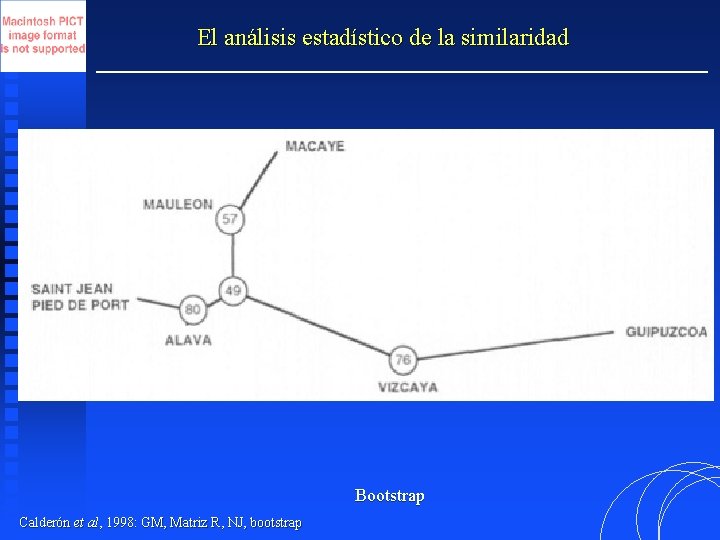
El análisis estadístico de la similaridad Bootstrap Calderón et al, 1998: GM, Matriz R, NJ, bootstrap

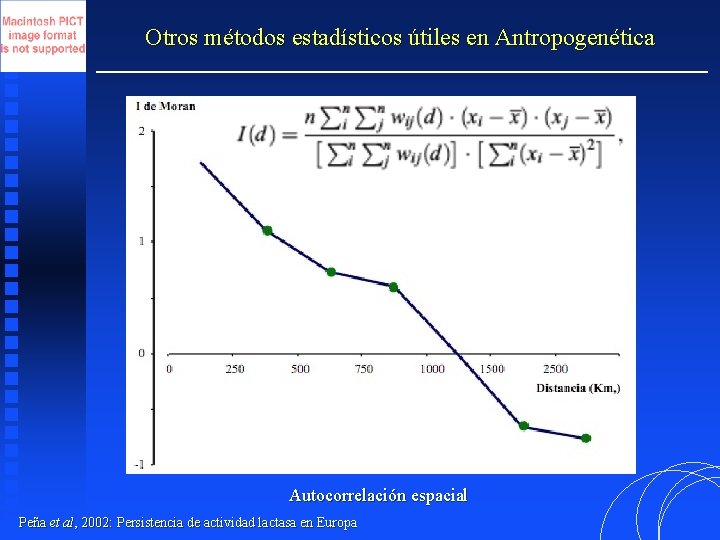
Otros métodos estadísticos útiles en Antropogenética Autocorrelación espacial Peña et al, 2002: Persistencia de actividad lactasa en Europa

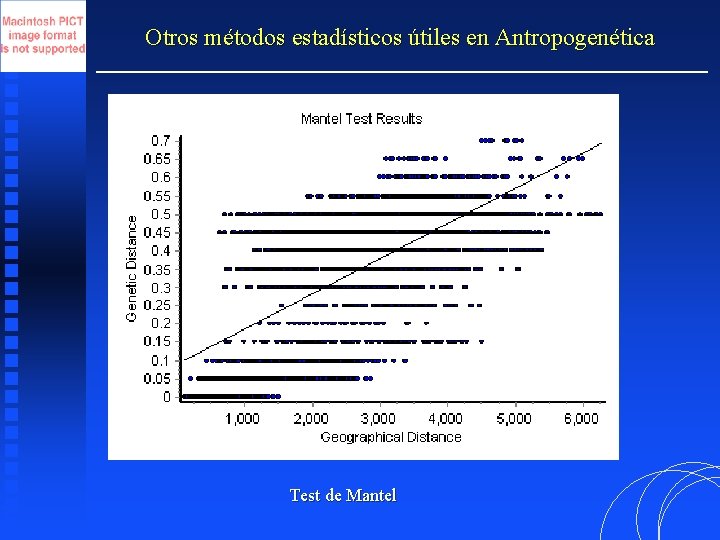
Otros métodos estadísticos útiles en Antropogenética Test de Mantel

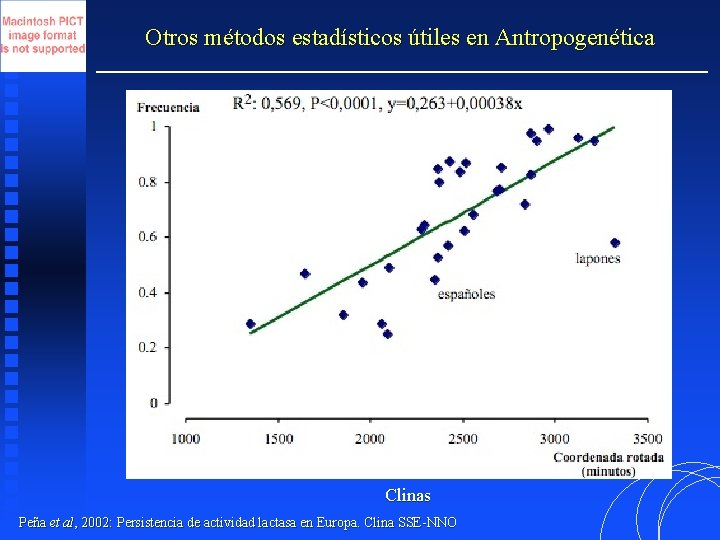
Otros métodos estadísticos útiles en Antropogenética Clinas Peña et al, 2002: Persistencia de actividad lactasa en Europa. Clina SSE-NNO

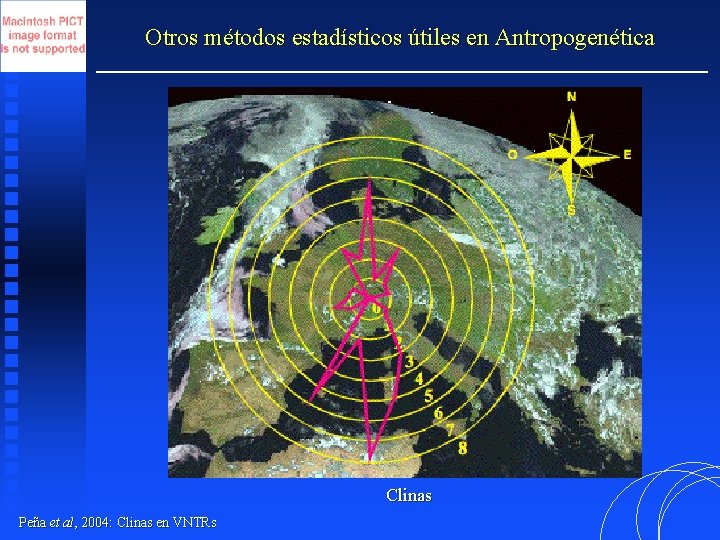
Otros métodos estadísticos útiles en Antropogenética Clinas Peña et al, 2004: Clinas en VNTRs

Otros métodos estadísticos útiles en Antropogenética Mapas sintéticos Roewer et al, 2005: Y-STRs

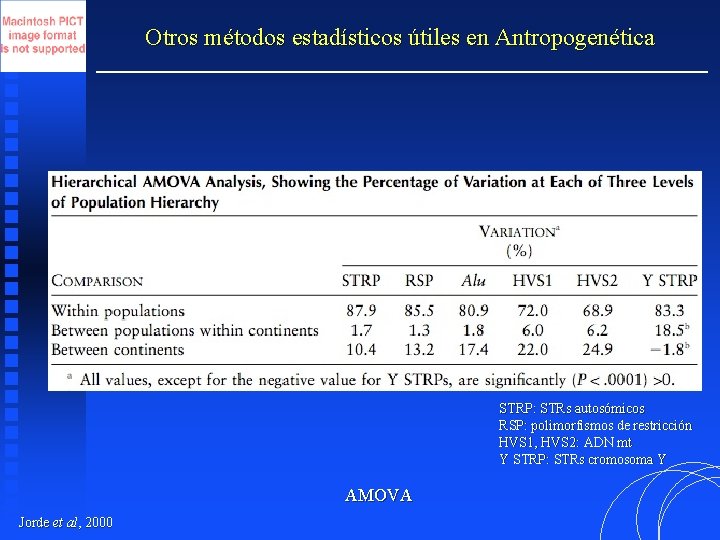
Otros métodos estadísticos útiles en Antropogenética STRP: STRs autosómicos RSP: polimorfismos de restricción HVS 1, HVS 2: ADN mt Y STRP: STRs cromosoma Y AMOVA Jorde et al, 2000

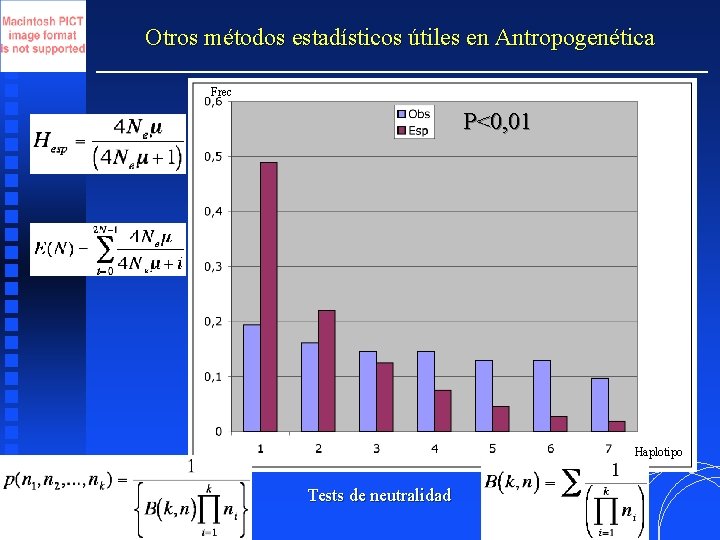
Otros métodos estadísticos útiles en Antropogenética Frec P<0, 01 Haplotipo Tests de neutralidad

Otros métodos estadísticos útiles en Antropogenética A 1 no A 1 Total B 8 376 91 467 no B 8 235 1265 1500 Total 611 1356 1967 Locus A Locus B A 1 B 8 +/+ a 376 +/b 235 -/+ c 91 -/d 1265 0, 311 x 0, 237+0, 077=0, 151 Desequilibrio de ligamiento Total n 1967

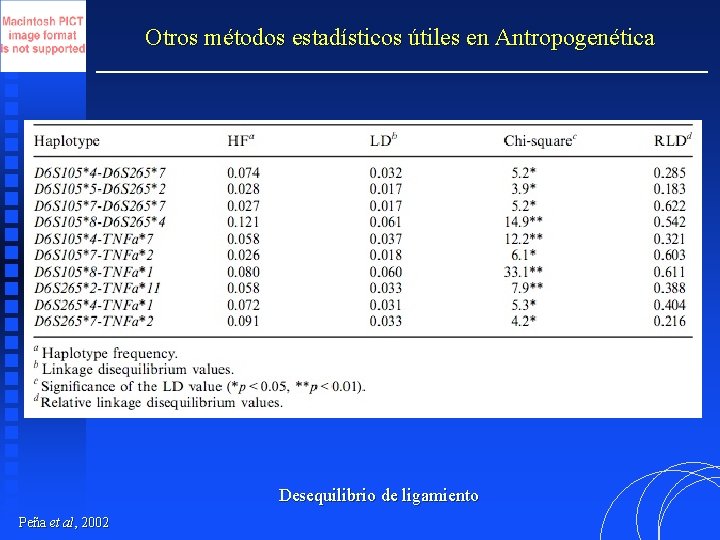
Otros métodos estadísticos útiles en Antropogenética Desequilibrio de ligamiento Peña et al, 2002

Mapa de ligamiento de 114 SNPs en varios grupos étnicos Goddard et al, 2000

Otros métodos estadísticos útiles en Antropogenética Probabilidad de coincidencia (Matching probability) Capacidad de discriminación (Power of discrimination) Capacidad de exclusión (Power of exclusion) Genética Forense

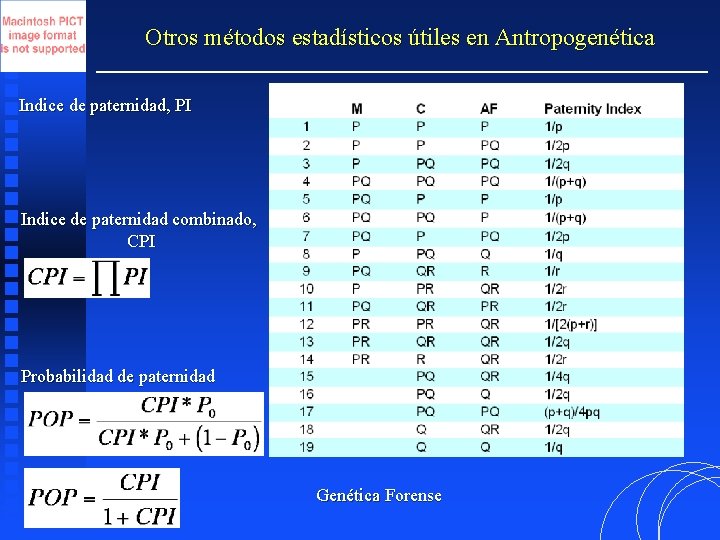
Otros métodos estadísticos útiles en Antropogenética Indice de paternidad, PI Indice de paternidad combinado, CPI Probabilidad de paternidad Genética Forense
 Https://slidetodoc.com/captulo-2-o-sudeste-e-o-sul-do/
Https://slidetodoc.com/captulo-2-o-sudeste-e-o-sul-do/ Conceito de judaismo
Conceito de judaismo Propagacion de incertidumbre
Propagacion de incertidumbre Que son datos objetivos en enfermeria
Que son datos objetivos en enfermeria Direktang sipi
Direktang sipi Glucorticoides
Glucorticoides Absceso de mama tratamiento
Absceso de mama tratamiento Diverticulitis tratamiento farmacologico
Diverticulitis tratamiento farmacologico Tratamiento targa
Tratamiento targa Toxoplasmosis cerebral tratamiento
Toxoplasmosis cerebral tratamiento Epistaxis tratamiento
Epistaxis tratamiento Tratamiento de edas
Tratamiento de edas Ectima
Ectima Hymenolepis nana hospedero definitivo
Hymenolepis nana hospedero definitivo Hemorragia esteriorizada
Hemorragia esteriorizada Pancreatitis aguda clasificacion
Pancreatitis aguda clasificacion Glandula tiroides
Glandula tiroides Dra. lucila esmeralda ruiz jáuregui
Dra. lucila esmeralda ruiz jáuregui Tratamiento de la icc
Tratamiento de la icc Anillo schatzki tratamiento
Anillo schatzki tratamiento Diferencias entre pml y tecnología al final del tubo
Diferencias entre pml y tecnología al final del tubo Aumentar en lenguaje algebraico
Aumentar en lenguaje algebraico Criterios centor
Criterios centor Prescribir el tratamiento contable
Prescribir el tratamiento contable Alcalosis respiratoria
Alcalosis respiratoria Salmonelosis sintomas
Salmonelosis sintomas Tratamiento para las drogas
Tratamiento para las drogas Tratamiento antihipertensivo
Tratamiento antihipertensivo Tratamiento disenteria en niños
Tratamiento disenteria en niños Gastroenteritis bacteriana tratamiento
Gastroenteritis bacteriana tratamiento Corrimiento uretral
Corrimiento uretral Algoritmo tratamiento scasest
Algoritmo tratamiento scasest Bronquiectasias tratamiento
Bronquiectasias tratamiento Fibrilacion ventricular
Fibrilacion ventricular Tratamiento de asma en pediatria
Tratamiento de asma en pediatria Tratamiento upp grado 2
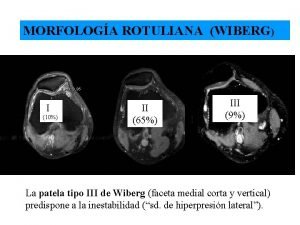
Tratamiento upp grado 2 Rotulas wiberg
Rotulas wiberg Tratamiento integrado de las lenguas
Tratamiento integrado de las lenguas Diagnosis
Diagnosis