ANALZA DNA SEKVENC Kde zskat sekvence Sangerovo sekvenovn

















![Shluková analýza - UPGMA: d[(BŠČ)G] = {d(BG)+d(ŠG)+d(ČG)}/3 WPGMA: d[(BŠČ)G] = {d[(BŠ)G] + d(ČG)}/2 Š Shluková analýza - UPGMA: d[(BŠČ)G] = {d(BG)+d(ŠG)+d(ČG)}/3 WPGMA: d[(BŠČ)G] = {d[(BŠ)G] + d(ČG)}/2 Š](https://slidetodoc.com/presentation_image_h/04364ebf9887d197cb274bb0887d1426/image-37.jpg)





- Slides: 45

ANALÝZA DNA SEKVENCÍ

Kde získat sekvence? • Sangerovo sekvenování –. ab 1 files • Gen. Bank či jiná databáze • Dryad – publikované datasety • NGS – FASTQ (obsahuje i informaci o kvalitě sekvence) – specifická analýza dat

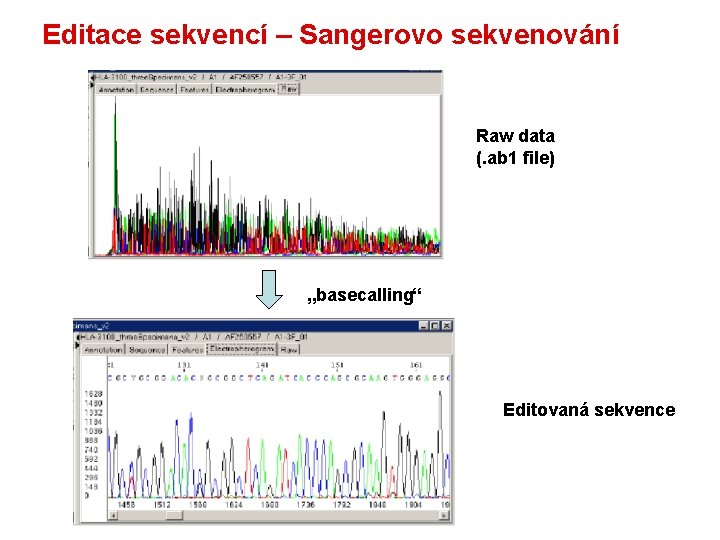
Editace sekvencí – Sangerovo sekvenování Raw data (. ab 1 file) „basecalling“ Editovaná sekvence

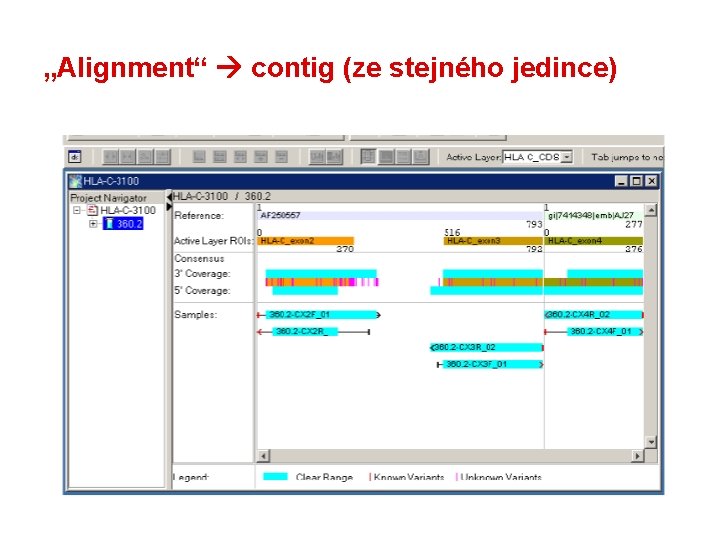
„Alignment“ contig (ze stejného jedince)

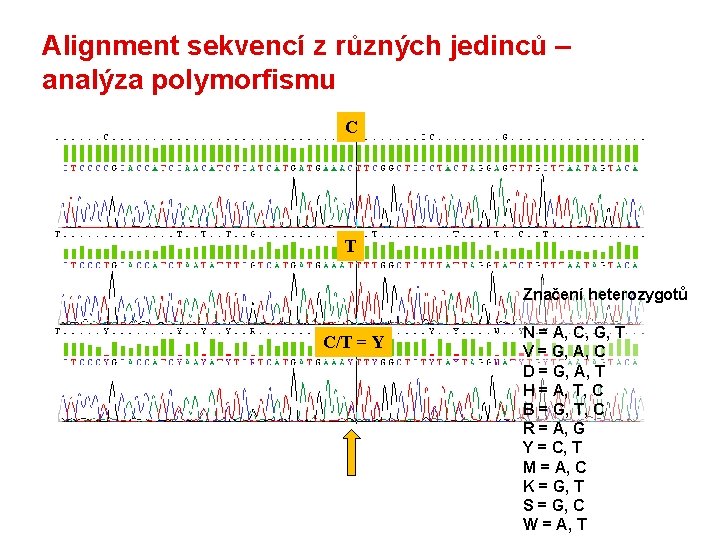
Alignment sekvencí z různých jedinců – analýza polymorfismu C T Značení heterozygotů C/T = Y N = A, C, G, T V = G, A, C D = G, A, T H = A, T, C B = G, T, C R = A, G Y = C, T M = A, C K = G, T S = G, C W = A, T

Nukleotidové a proteinové sekvence: H_sapiens MTPMRKINPLMKLINHSFIDLPTPSNISAWWNFGS báze = stav znaku P_troglod ATGACCCCGACACGCAAAATTAACCCACTAATAAA pozice (site) = znak

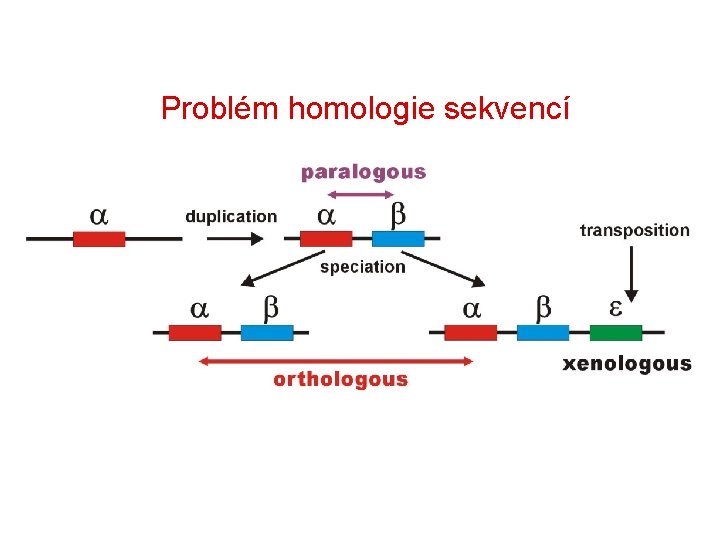
Problém homologie sekvencí

Práce se sekvencemi DNA databáze: EMBL (European Molecular Biology Laboratory) – European Bioinformatics Institute, Hinxton, UK: http: //www. ebi. ac. uk/embl/ Gen. Bank – NCBI (National Center for Biotechnology Information), Bethesda, Maryland, USA: http: //www. ncbi. nlm. nih. gov/Genbank/ DDBJ (DNA Data Bank of Japan) – National Institute of Genetics, Mishima, Japan: http: //www. ddbj. nig. ac. jp/ Proteinové databáze: SWISS-PROT – University of Geneve & Swis Institute of Bioinformatics: http: //www. expasy. ch/sprot/ a http: //www. ebi. ac. uk/swissprot/ PIR (Protein Information Resource) – NBRF (National Biomedical Research Foundation, Washington, D. C. , USA) & Tokyo University & JIPID (Japanese International Protein Information Database, Tokyo) & MIPS (Martinsried Institute for Protein Sequences, Martinsried, Germany): http: //www-nbrf. georgetown. edu/ PRF/SEQDB (Protein Resource Foundation) – Ósaka, Japan: http: //www. prf. or. jp/en/os. htm PDB (Protein Data Bank) – University of New Jersey, San Diego & Super-computer Center, University of California & National Institute of Standards and Technology: http: //www. rcsb. org/pdb/

Formáty souborů FASTA: >H_sapiens ATGACCCCAATACGCAAAATTAACCCCCTAATAAAATTAACCACTCATCGACCTCCCCACCC CATCCAACATCTCCGCATGATGAAACTTCGGCTCACTCCTTGGCGCCTGATCCTCCAAATCACCAC AGGACTATTCCTAGCCATACACTACTCACCAGACGCCTCAACCGCCTTTTCATCAATCGCCCACATCACT CGAGACGTAAATTATGGCTGAATCATCCGCTACCTTCACGCCAATGGCGCCTCAATATTCTTTATCTGCC TCTTCCTACACATCGGGCGAGGCCTATATTACGGATCATTTCTCTACTCAGAAACCTGAAACATCGGCAT. . . >P_troglod ATGACCCCGACACGCAAAATTAACCCACTAATAAAATTAATCACTCATTTATCGACCTCCCCACCC CATCCAACATTTCCGCATGATGGAACTTCGGCTCACTTCTCGGCGCCTAATCCTTCAAATTACCAC AGGATTATTCCTAGCTATACACTACTCACCAGACGCCTCAACCGCCTTCTCGTCGATCGCCCACATCACC CGAGACGTAAACTATGGTTGGATCATCCGCTACCTCCACGCTAACGGCGCCTCAATATTTTTTATCTGCC TCTTCCTACACATCGGCCGAGGTCTATATTACGGCTCATTTCTCTACCTAGAAACCTGAAACATTGGCAT. . . >P_paniscus ATGACCCCAACACGCAAAATCAACCCACTAATAAAATTAATCACTCATTTATCGACCTCCCCACCC CATCCAATATTTCCACATGATGAAACTTCGGCTCACTTCTCGGCGCCTAATCCTTCAAATCACCAC AGGACTATTCCTAGCTATACACTACTCACCAGACGCCTCAACCGCCTTCTCATCGCCCACATTACC CGAGACGTAAACTATGGTTGAATCATCCGCTACCTTCACGCTAACGGCGCCTCAATACTTTTCATCTGCC TCTTCCTACACGTCGAGGCCTATATTACGGCTCATTTCTCTACCTAGAAACCTGAAACATTGGCAT. . .

Formáty souborů Gen. Bank: ORIGIN 1 61 121 181 241 301 361 421 481 541 601 661 721 781 841 901 961 1021 // tgaaatgaag cacccaaagc tagtacattt ttaatcaatg ttatactaat tacagtcata taccatcctc attaaacttg catcaaatgc ctaatcagcc cctactttca tacggtgaag ttagacataa cttaatgcca ccctatgtcc aaaattttac aatattaaca taattttatc atattctctt tggcattcta atgtatatcg atataggcca acatcaaatt aactcttctc cgtgaaacca ggggtagcta gttatcgccc catgaccaac tcaacatagc aatcattagt atgctactca aaccccaaaa tgatcgattc aaaatcatgc agttaatgta cca ctcaagacat attaaactac tacattaaac taaaacaatt aatgctttaa ttccatatga acaacccgcc aactgaaact atacgttccc ataactgtgg cgtcaaggca ccgcaaaacc ataccaaatt acactaagaa tagtagttcc tccgtgaacc gcttaataac caagaagaag ttcttgtgta tattttcccc atcaacataa agacatatct ctatcccctt caccaatgcc ttatcagaca cttaaataag tgtcatgcat tgaaaggaca caatcaccta ttaactctcc cttgaaagac caaaatatga aaaactctaa aaagc gaactactcc cataaattta aagcatataa actgatacaa gtgttatctg ccccatttgg cctcttctcg tctggttctt acatctcgat ttggtatttt gcacacagtc aggctaatta aaacccccca atatattatt ctcatatttt tcacactcta actgaaaatg ccaccaccag catagtacaa gcaagtacat accatgaata acatacacca tctattaatc ctccgggccc acttcagggc ggtatcgggt tttattttgg tagacgcacc ttcatgcttg accccctcct aactatcaaa agtacttgta ttacgcaata cttagatgga

Formáty souborů PHYLIP (“interleaved” format): 6 1120 H_sapiens P_troglod P_paniscus G_gorilla P_pygmaeus H_lar ATGACCCCAA ATGACCCCGA ATGACCCCAA ATGACCCCTA ATGACCCCAA ATGACCCCCC TACGCAAAAT CACGCAAAAT TACGCAAAAC TGCGCAAAAC TAACCCCCTA TAACCCACTA CAACCCACTA TAACCCACTA ATAAAATTAA GCAAAACTAA ATAAAATTAA ATAAAACTAA TTAACCACTC TTAATCACTC TTAACCACTC TCAACCACTC ATTCATCGAC ATTTATCGAC ATTCATTGAC ACTCATCGAC ACTTATCGAC CTCCCCACCC CTCCCTACCC CTCCCCACCC CTTCCAGCCC CATCCAACAT CATCCAATAT CGTCCAACAT CATCAAACAT CATCCAACAT CTCCGCATGA TTCCACATGA CTCTGCATGA TTCTATATGA TGAAACTTCG TGGAACTTCG TGAAACTTTG

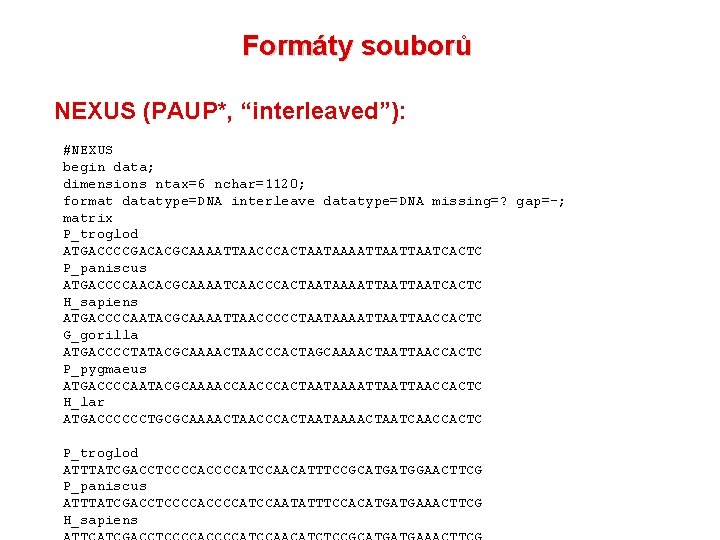
Formáty souborů NEXUS (PAUP*, “interleaved”): #NEXUS begin data; dimensions ntax=6 nchar=1120; format datatype=DNA interleave datatype=DNA missing=? gap=-; matrix P_troglod ATGACCCCGACACGCAAAATTAACCCACTAATAAAATTAATCACTC P_paniscus ATGACCCCAACACGCAAAATCAACCCACTAATAAAATTAATCACTC H_sapiens ATGACCCCAATACGCAAAATTAACCCCCTAATAAAATTAACCACTC G_gorilla ATGACCCCTATACGCAAAACTAACCCACTAGCAAAACTAATTAACCACTC P_pygmaeus ATGACCCCAATACGCAAAACCCACTAATAAAATTAACCACTC H_lar ATGACCCCCCTGCGCAAAACTAACCCACTAATAAAACTAATCAACCACTC P_troglod ATTTATCGACCTCCCCATCCAACATTTCCGCATGATGGAACTTCG P_paniscus ATTTATCGACCTCCCCATCCAATATTTCCACATGATGAAACTTCG H_sapiens

Formáty souborů Clustal: P_troglod P_paniscus H_sapiens G_gorilla P_pygmaeus H_lar ATGACCCCGACACGCAAAATTAACCCACTAATAAAATTAATCACTCATTTATCGAC ATGACCCCAACACGCAAAATCAACCCACTAATAAAATTAATCACTCATTTATCGAC ATGACCCCAATACGCAAAATTAACCCCCTAATAAAATTAACCACTCATCGAC ATGACCCCTATACGCAAAACTAACCCACTAGCAAAACTAATTAACCACTCATTGAC ATGACCCCAATACGCAAAACCCACTAATAAAATTAACCACTCATCGAC ATGACCCCCCTGCGCAAAACTAACCCACTAATAAAACTAATCAACCACTTATCGAC ******* *** ****** *** P_troglod P_paniscus H_sapiens G_gorilla P_pygmaeus H_lar CTCCCCATCCAACATTTCCGCATGATGGAACTTCGGCTCACTTCTCGGCGCCTGC CTCCCCATCCAATATTTCCACATGATGAAACTTCGGCTCACTTCTCGGCGCCTGC CTCCCCATCCAACATCTCCGCATGATGAAACTTCGGCTCACTCCTTGGCGCCTGC CTCCCTACCCCGTCCAACATCTCCACATGATGAAACTTCGGCTCACTCCTTGGTGCCTGC CTCCCCATCAAACATCTCTGCATGATGGAACTTCGGCTCACTTCTAGGCGCCTGC CTTCCAGCCCCATCCAACATTTCTATATGATGAAACTTTGGTTCACTCCTAGGCGCCTGC ** ** ***** ** ******

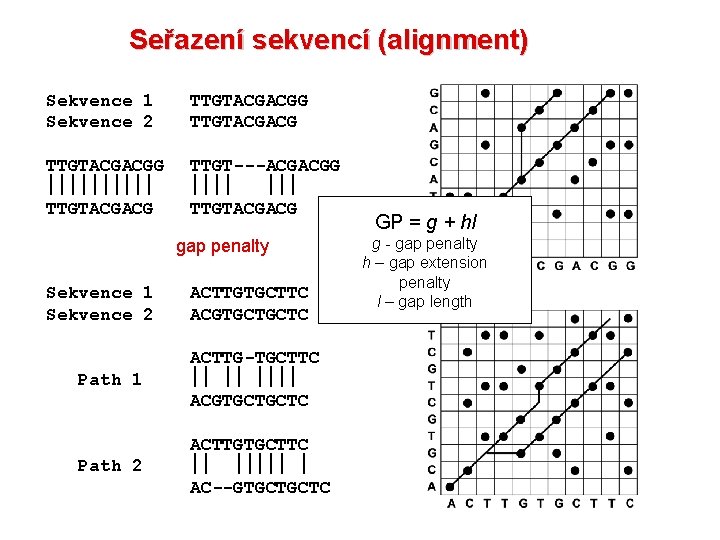
Seřazení sekvencí (alignment) Sekvence 1 Sekvence 2 TTGTACGACGG TTGTACGACG TTGT---ACGACGG TTGTACGACG gap penalty Sekvence 1 Sekvence 2 ACTTGTGCTTC ACGTGCTGCTC Path 1 ACTTG-TGCTTC ACGTGCTGCTC Path 2 ACTTGTGCTTC AC--GTGCTGCTC GP = g + hl g - gap penalty h – gap extension penalty l – gap length

Gen. Bank (http: //www. ncbi. nlm. nih. gov/genbank/) BLAST (http: //blast. ncbi. nlm. nih. gov/Blast. cgi? PROGRAM=blastn&PA GE_TYPE=Blast. Search&LINK_LOC=blasthome) Bio. Edit konverze formátů (zejména pro fylogenetickou analýzu): ALTER (http: //sing. ei. uvigo. es/ALTER/)

Dna. SP • např. tvorba haplotypového souboru (jednotlivé sekvence seřazeny do haplotypů – pro vytváření haplotypové sítě) • Phase – separace heterozygotů do haplotypů (MCMC algorithm)

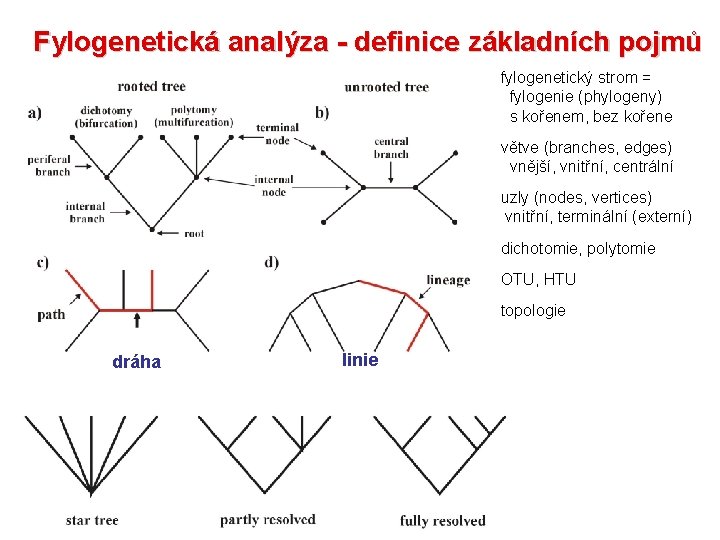
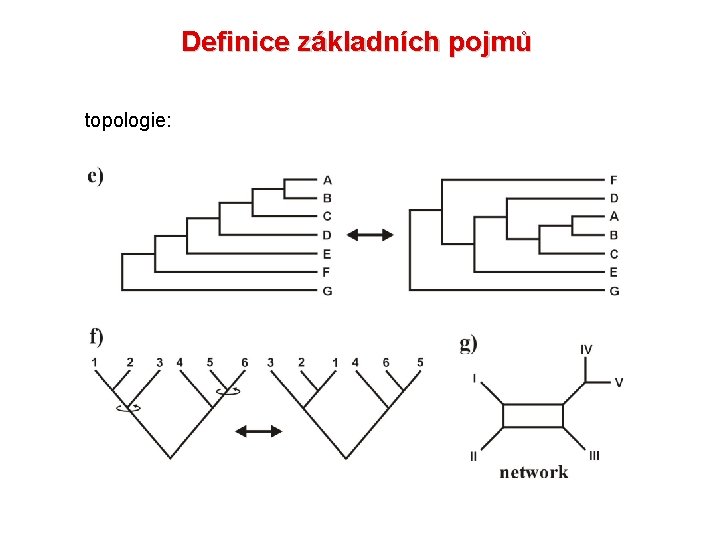
Fylogenetická analýza - definice základních pojmů fylogenetický strom = fylogenie (phylogeny) s kořenem, bez kořene větve (branches, edges) vnější, vnitřní, centrální uzly (nodes, vertices) vnitřní, terminální (externí) dichotomie, polytomie OTU, HTU topologie dráha linie

Definice základních pojmů topologie: linie

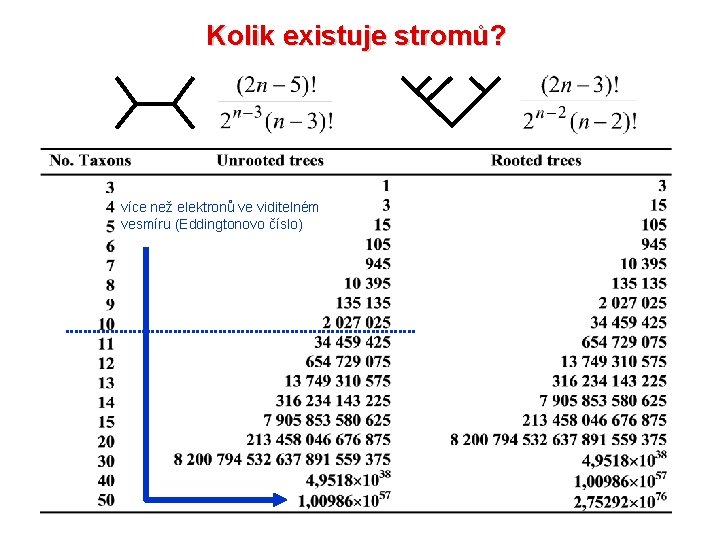
Kolik existuje stromů? více než elektronů ve viditelném vesmíru (Eddingtonovo číslo)

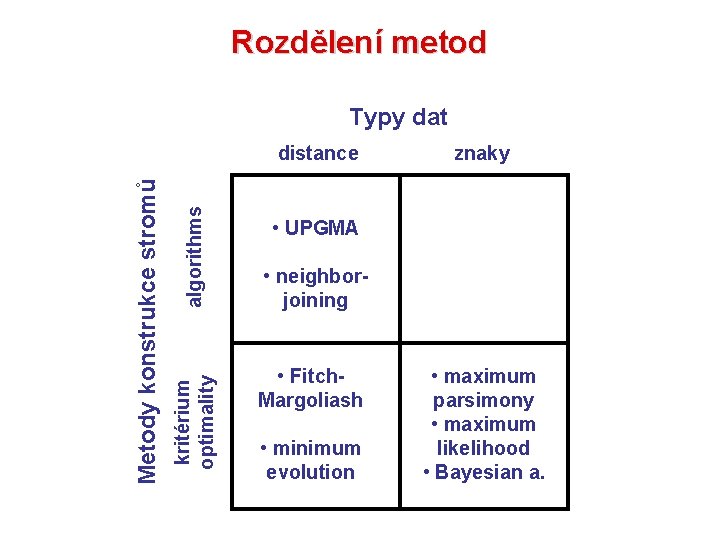
Rozdělení metod Typy dat algorithms kritérium optimality Metody konstrukce stromů distance znaky • UPGMA • neighborjoining • Fitch. Margoliash • minimum evolution • maximum parsimony • maximum likelihood • Bayesian a.

Jak hodnotit jednotlivé metody? výkonnost (efficiency): jak rychlá je metoda? síla (power): kolik znaků je třeba? konzistence (consistency): vede zvyšující se počet znaků ke správnému stromu? robustnost (robustness): jak metoda funguje při neplatnosti předpokladů? falzifikovatelnost (falsifiability): umožňuje testování platnosti předpokladů?

(1) Maximální úspornost (maximum parsimony, MP) - snaha minimalizovat počet analogických stavů I II III A 1 0 1 B 0 0 1 C 1 0 0 D 0 1 0 E 1 0 1 2 kroky William of Occam (c. 1285 - c. 1349): Occamova břitva minimální počet kroků = 3 (pro každý znak jedna změna) skutečný počet kroků = 5 2 extra kroky analogie = homoplasie stejný stav znaku vzniká vícekrát nezávisle 1 krok 2 kroky

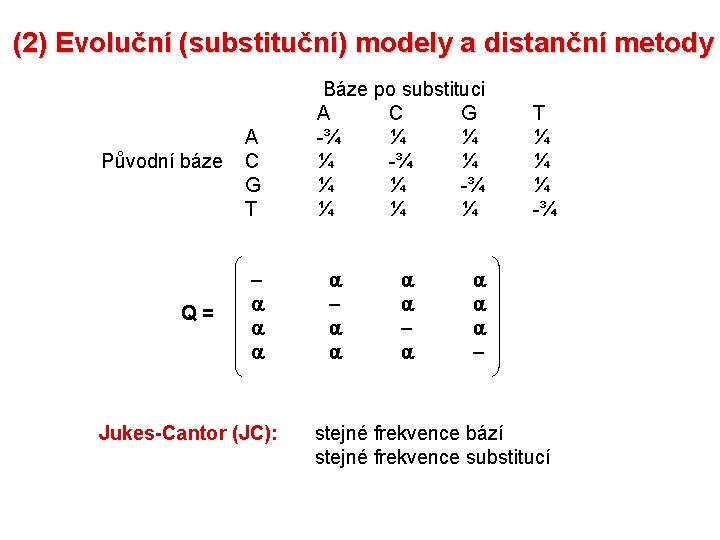
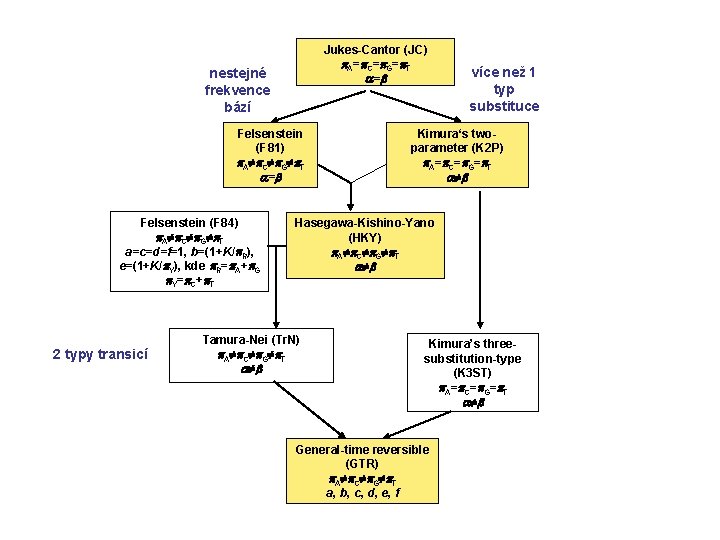
(2) Evoluční (substituční) modely a distanční metody Původní báze Q= A C G T Jukes-Cantor (JC): Báze po substituci A C G -¾ ¼ ¼ ¼ T ¼ ¼ ¼ -¾ stejné frekvence bází stejné frekvence substitucí

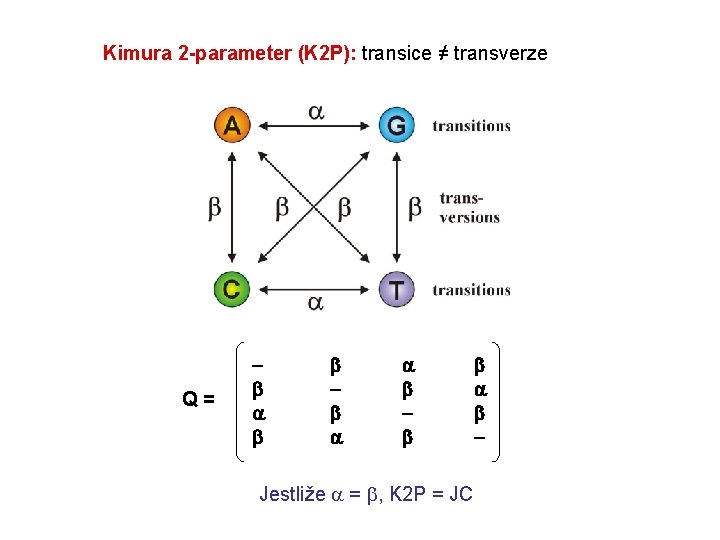
Kimura 2 -parameter (K 2 P): transice ≠ transverze Q= Jestliže = , K 2 P = JC

Felsenstein (F 81): různé frekvence bází Q= A A A C C C G G G T T T Jestliže A = C = G = T, F 81 = JC Hasegawa-Kishino-Yano (HKY): Q= A A A General time-reversible (GTR): C C C různé frekvence bází transice ≠ transverze G G G T T T různé frequence bází různé frekvence všech substitucí

Jukes-Cantor (JC) A= C= G= T = nestejné frekvence bází Felsenstein (F 81) A C G T = Felsenstein (F 84) A C G T a=c=d=f=1, b=(1+K/ R), e=(1+K/ Y), kde R= A+ G Y= C+ T 2 typy transicí více než 1 typ substituce Kimura‘s twoparameter (K 2 P) A= C= G= T Hasegawa-Kishino-Yano (HKY) A C G T Tamura-Nei (Tr. N) A C G T Kimura’s threesubstitution-type (K 3 ST) A= C= G= T General-time reversible (GTR) A C G T a, b, c, d, e, f

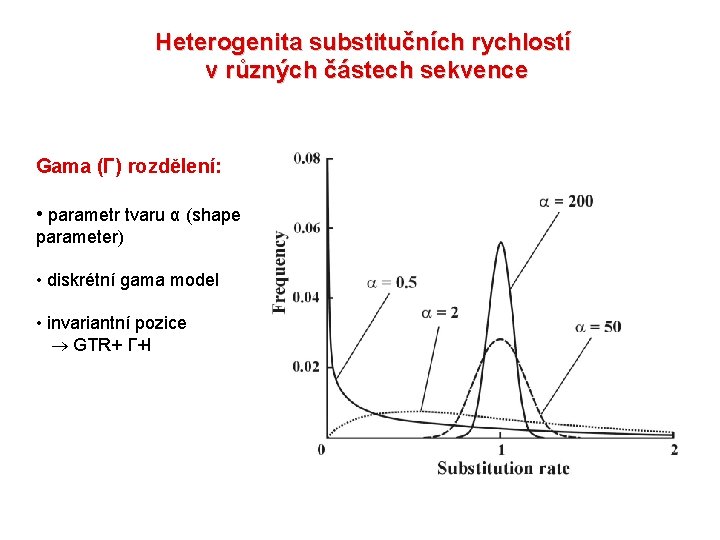
Heterogenita substitučních rychlostí v různých částech sekvence Gama (Γ) rozdělení: • parametr tvaru α (shape parameter) • diskrétní gama model • invariantní pozice GTR+ Γ+I

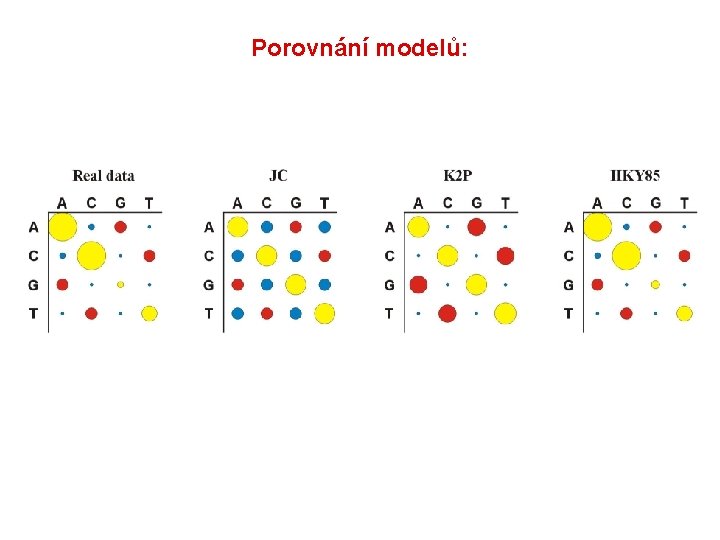
Porovnání modelů:

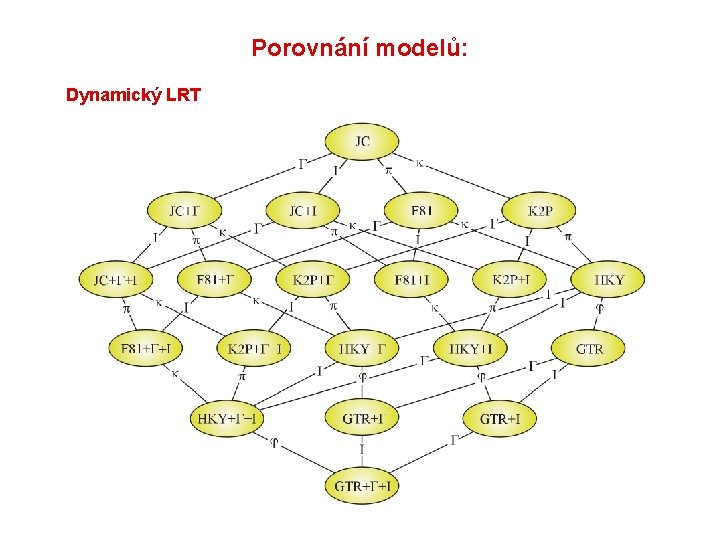
Porovnání modelů: Který model vybrat? Likelihood ratio test (LRT): nested models LR = 2(ln. L 2 – ln. L 1) Chi-square, p 2 – p 1 d. f. Akaike information criterion (AIC): nonnested models AIC = -2 ln. L + 2 p, where p = number of free parameters better model smaller AIC Bayesian information criterion (BIC): nonested models BIC = -2 ln. L + pln. N, where N = sample size

Porovnání modelů: Hierarchický LRT – Model. Test (Crandall and Posada)

Porovnání modelů: Dynamický LRT

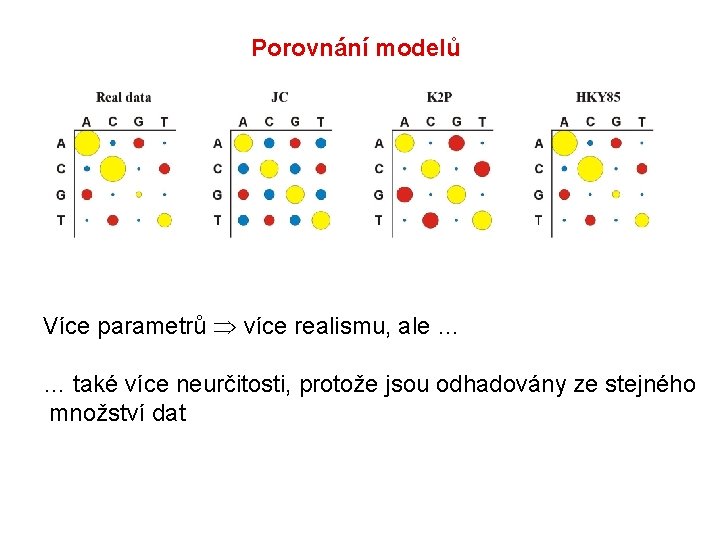
Porovnání modelů Více parametrů více realismu, ale … … také více neurčitosti, protože jsou odhadovány ze stejného množství dat

Distance • počítány pro každý pár taxonů, z matice distancí (nebo podobností) konstruován strom • distanční metody založeny na předpokladu, že pokud bychom znali skutečné distance mezi všemi studovanými taxony, mohli bychom velmi jednoduše rekonstruovat správnou fylogenii • výhoda: velmi rychlé a jednoduché (lze i na kalkulačce)

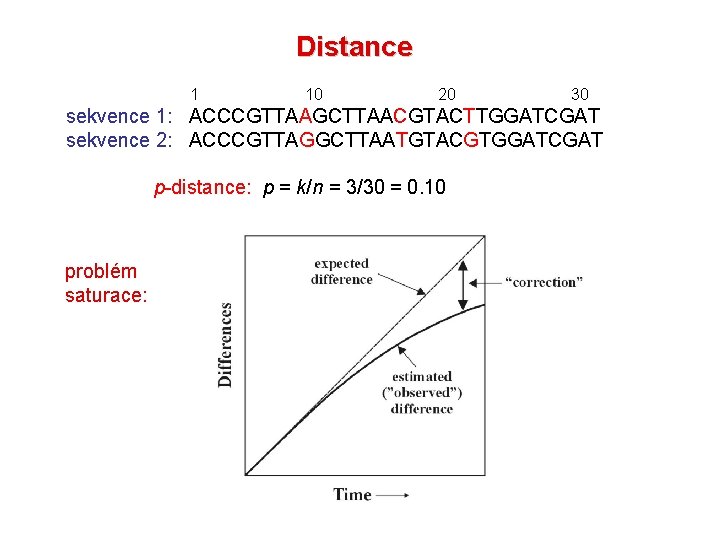
Distance 10 20 30 sekvence 1: ACCCGTTAAGCTTAACGTACTTGGATCGAT sekvence 2: ACCCGTTAGGCTTAATGTACGTGGATCGAT p-distance: p = k/n = 3/30 = 0. 10 problém saturace:

Distance pro některé modely: Příklad v MEGA

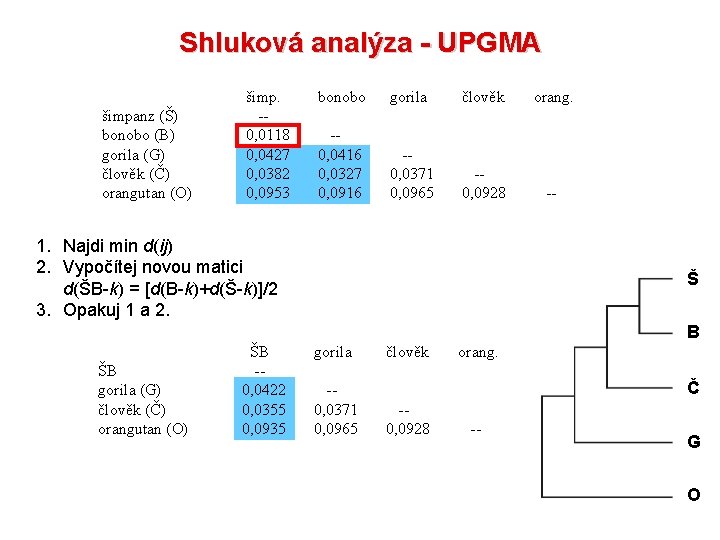
Shluková analýza - UPGMA šimpanz (Š) bonobo (B) gorila (G) člověk (Č) orangutan (O) šimp. -0, 0118 0, 0427 0, 0382 0, 0953 bonobo gorila člověk orang. -0, 0416 0, 0327 0, 0916 -0, 0371 0, 0965 -0, 0928 -- 1. Najdi min d(ij) 2. Vypočítej novou matici d(ŠB-k) = [d(B-k)+d(Š-k)]/2 3. Opakuj 1 a 2. Š B ŠB gorila (G) člověk (Č) orangutan (O) ŠB -0, 0422 0, 0355 0, 0935 gorila -0, 0371 0, 0965 člověk orang. Č -0, 0928 -- G O
![Shluková analýza UPGMA dBŠČG dBGdŠGdČG3 WPGMA dBŠČG dBŠG dČG2 Š Shluková analýza - UPGMA: d[(BŠČ)G] = {d(BG)+d(ŠG)+d(ČG)}/3 WPGMA: d[(BŠČ)G] = {d[(BŠ)G] + d(ČG)}/2 Š](https://slidetodoc.com/presentation_image_h/04364ebf9887d197cb274bb0887d1426/image-37.jpg)
Shluková analýza - UPGMA: d[(BŠČ)G] = {d(BG)+d(ŠG)+d(ČG)}/3 WPGMA: d[(BŠČ)G] = {d[(BŠ)G] + d(ČG)}/2 Š single-linkage B complete-linkage Č G O

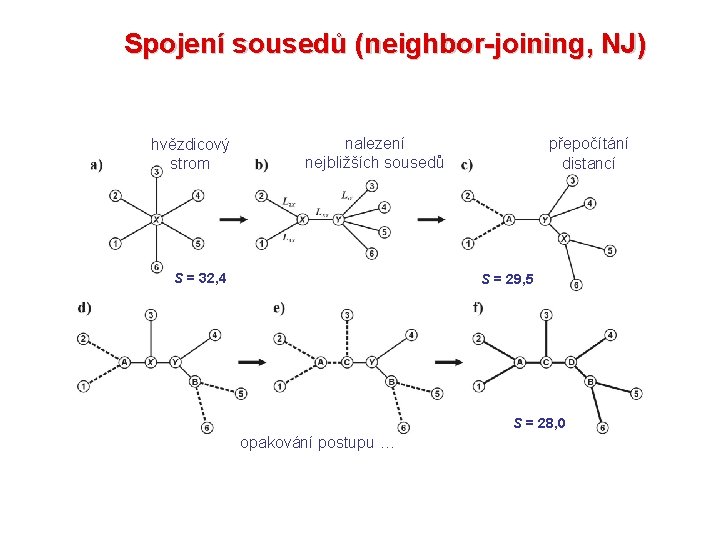
Spojení sousedů (neighbor-joining, NJ) • Algoritmická metoda • Princip minimální evoluce minimalizuje součet délek větví S • Každý pár uzlů adjustován na základě divergence od ostatních • Konstrukce jediného aditivního stromu

Spojení sousedů (neighbor-joining, NJ) hvězdicový strom nalezení nejbližších sousedů S = 32, 4 přepočítání distancí S = 29, 5 S = 28, 0 opakování postupu …

Nevýhody distančních dat: 1. ztráta části informace během transformace 2. jakmile data transformována na distance, nelze se vrátit zpět (odlišné sekvence mohou dát stejné distance) 3. nelze sledovat evoluci na různých částech sekvence 4. obtížná biologická interpretace délek větví 5. nelze kombinovat různé distanční matice

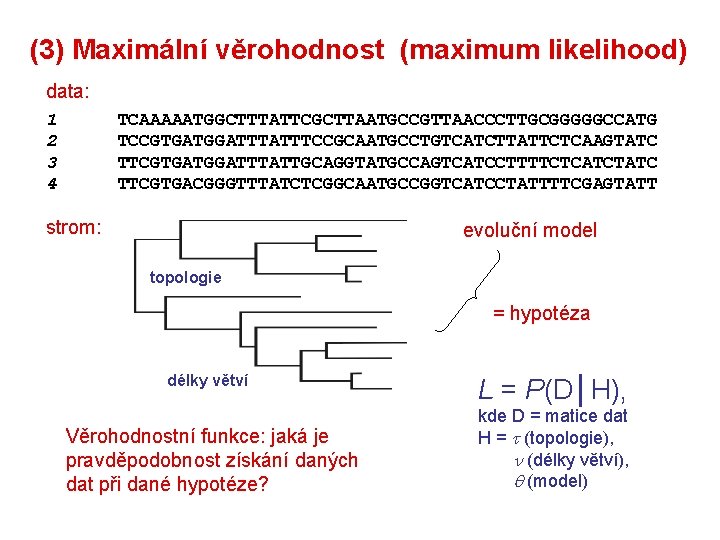
(3) Maximální věrohodnost (maximum likelihood) data: 1 2 3 4 TCAAAAATGGCTTTATTCGCTTAATGCCGTTAACCCTTGCGGGGGCCATG TCCGTGATGGATTTCCGCAATGCCTGTCATCTTATTCTCAAGTATC TTCGTGATGGATTTATTGCAGGTATGCCAGTCATCCTTTTCTCATCTATC TTCGTGACGGGTTTATCTCGGCAATGCCGGTCATCCTATTTTCGAGTATT strom: evoluční model topologie = hypotéza délky větví Věrohodnostní funkce: jaká je pravděpodobnost získání daných dat při dané hypotéze? L = P(D│H), kde D = matice dat H = (topologie), (délky větví), (model)

(4) Bayesovská analýza aposteriorní pravděpodobnost (posterior probability) = pr. platnosti hypotézy při získaných datech: P(H D) a. p. je funkcí věrohodnosti P(D H) a apriorní pravděpodobnosti (prior prob. ) prior vyjadřuje náš apriorní předpoklad nebo znalost Aposteriorní pravděpodobnost je dána Bayesovou rovnicí: věrohodnost prior P(D H) P(H) P(H D) = [P(D Hi) P(Hi)] suma čitatelů pro všechny alternativní hypotézy

Fylogenetické programy: alignment: Clustal. X http: //inn-prot. weizmann. ac. il/software/Clustal. X. html Bio. Edit Ali. View PAUP* PHYLIP MEGA. . . MP, NJ, ML RAx. ML. . . ML Mr. Bayes. . . BA http: //evolution. genetics. washington. edu/phylip/software. html#methods práce se stromy: Tree. View http: //taxonomy. zoology. gla. ac. uk/rod/treeview. html Fig. Tree

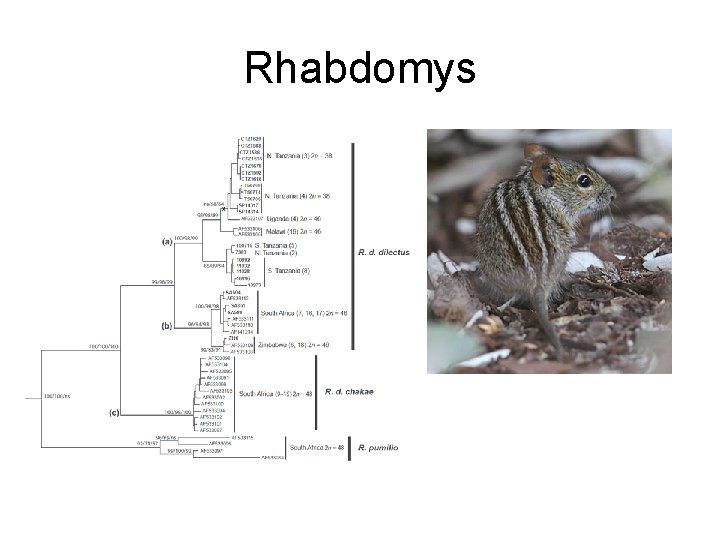
Příklad • určit druh zvířete, které jsme osekvenovali na mt. DNA (BLAST) • určit jeho fylogenetickou pozici v rámci rodu (alignment, NJ tree)

Rhabdomys
 Ribozomy prokaryot
Ribozomy prokaryot Shine dalgarnova sekvence
Shine dalgarnova sekvence Shine dalgarnova sekvence
Shine dalgarnova sekvence Chapter 11 dna and genes
Chapter 11 dna and genes Bioflix activity dna replication dna replication diagram
Bioflix activity dna replication dna replication diagram Coding dna and non coding dna
Coding dna and non coding dna Enzyme involved in dna replication
Enzyme involved in dna replication Function of dna polymerase 3
Function of dna polymerase 3 Ssreg
Ssreg Analza
Analza Finann
Finann Swot analza
Swot analza Analza
Analza Swot analza
Swot analza Analza
Analza Korelan
Korelan Analzy
Analzy Swot analza
Swot analza Analza
Analza Analuza
Analuza Post hoc definition
Post hoc definition Swot analza
Swot analza Analza
Analza Analuza
Analuza Analza
Analza Suroviny
Suroviny Cobb douglasova produkční funkce
Cobb douglasova produkční funkce Swot analza
Swot analza Swot analysis objectives
Swot analysis objectives Analza
Analza Analza
Analza Analza
Analza Analzy
Analzy Slept analyza
Slept analyza Analza
Analza Swot analza
Swot analza Kde leží těžiště pneumatiky
Kde leží těžiště pneumatiky Kde jsme
Kde jsme Gnome architecture
Gnome architecture Rieky ameriky
Rieky ameriky Kde měl daidalos domov
Kde měl daidalos domov Zdokonalený pluh
Zdokonalený pluh Kde je sever
Kde je sever 703 kar 5:070
703 kar 5:070 Skalnica horska spolocenstvo
Skalnica horska spolocenstvo Glykemický index výpočet
Glykemický index výpočet