Molekler Markr Verileri ile Genetik eitlilik Analizi Genetik













![Ortalama beklenen heterozigotluk (He) (Nei’e göre genetik çeşitlilik [D]) �Genetik çeşitlilik populasyondan rasgele çekilen Ortalama beklenen heterozigotluk (He) (Nei’e göre genetik çeşitlilik [D]) �Genetik çeşitlilik populasyondan rasgele çekilen](https://slidetodoc.com/presentation_image_h2/935089b624f3eb5a07324798d86cdf42/image-15.jpg)











- Slides: 31

Moleküler Markör Verileri ile Genetik Çeşitlilik Analizi Genetik çeşitlilik ölçütleri

Temel Genetik Çeşitlilik Analizi 1. Populasyon içi-arası, bölge içiarası vb. varyasyonların tanımlanması. 2. Bireyler, populasyonlar, bölgeler arası ilişkinin belirlenmesi.

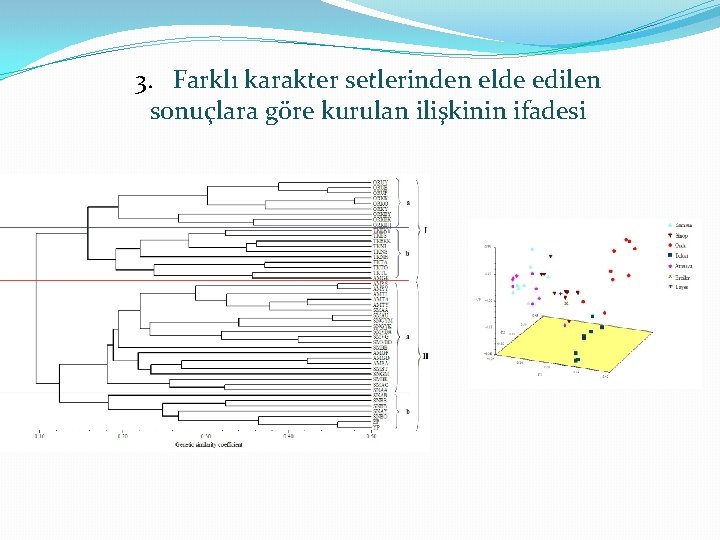
3. . Farklı karakter setlerinden elde edilen sonuçlara göre kurulan ilişkinin ifadesi

GENETİK ÇEŞİTLİLİĞİN BELİRLENMESİ � 1. Çeşitliliğin tanımlanması Populasyon içinde ve populasyonlar arısında ya da daha büyük ünitelerde (alanlar, bölgeler gibi) � 2. Üniteler arasındaki ilişkinin hesaplanması Çalışmaya konu her bir çift arasında genetik ya da geometrik uzaklıkların hesaplanması � 3. Bu ilişkilerin kümeleme ve/veya çok boyutlu uzayda gösterim (ordinasyon) metodlarıyla ifadelenmesi Bu yöntemlerin bazıları moleküler çalışmanın sonuçlarının diğer verilerle (örneğin coğrafik) mukayesesine imkan verir.


Değişken çeşitleri Kalitatif (Nitel): Karakter ya da özellikler; kategorik, binary, var-yok: (Örn: Boynuzluluk-boynuzsuzluk) • Binary; sadece iki değer alır (1 -var, 0 -yok) • Kategorik; bir çok olasılık arasından tek bir değerin alınması Ordinal : Belli bir sıra düzenindeki kategoriler (Örn. Kuyruk: 1 -uzun, 2 kısa, 3 -yağlı) Nominal: İlişkisiz kategoriler (Örn. Tüy rengi: 1 -Beyaz, 2 -Kahverengi, 3 Siyah, 4 -Kırçıl) Kantitatif (Nicel): Kesikli ya da sürekli olabilen sayısal değerlerdir. Sürekli: Belli bir aralıktan alınan değer (Örn. Canlı ağırlık (kg), Boynuz uzunluğu (cm)) Kesikli: Tam ya da ondalık sayıların alınması (Örn. Batın genişliği: tekiz, ikiz, üçüz)

�Kategorik değişkenler binary değişkenlere dönüştürülebilirler. � Sınırlandırıcıları vardır. Bazı benzerlik katsayıları belli bir karakter kategorisine değerlendirmeye alınan diğer karakterlerden daha fazla ağırlık verir. Birden fazla kategorinin aynı değişkeni içermesinden dolayı bazı değişkenlerin sonuç üzerine olması gerekenden daha az fazla etki yapması mümkün olabilir. Kategorik örnek; �Kısa kuyruk: var (1), yok (0) �Uzun kuyruk: var (1), yok (0) �Yağlı kuyruk: var (1), yok (0) � �Kantitatif değişkenler de binary değişkenlere dönüştürülebilirler. �Örneğin; � 0 -50 yumurta : var (1), yok (0) � 51 -100 yumurta : var (1), yok (0) � 101 -150 yumurta : var (1), yok (0) � 151 -200 yumurta : var (1), yok (0) �. . .

Populasyon içi genetik çeşitliliğin ölçülmesi �Çeşitli varyantlara dayanır • Polimorfizm* ya da polimorfizm oranı (Pj) *Çok biçimlilik, ortak yapının farklı formlara sahip olması. • Polimorfik lokusların oranı • Allelik varyant zenginliği (A)v • Lokus başına ortalama allel sayısı �Varyantların frekansına dayanır • Etkili allel sayısı (Ae) • Ortalama beklenen heterozigotluk (He; Nei’e göre genetik çeşitlilik (D))

Polimorfizm ya da polimorfizm oranı (Pj) � Bir genin allellerinin frekansı 0, 95 ya da 0, 99’dan küçük ya da az ise polimorfik olarak kabul edilir. Pj = q ≤ 0. 95 ya da Pj = q ≤ 0. 99 � Pj = polimorfizm oranı � q = allel frekansı � • Bu ölçü bir genin varyasyon gösterdiğini ifade eden bir kriterdir. � • Gene ait allellerin doğrudan varlığının gözlenmesi ile hesaplanır. � • Kodominant markörler ile kullanılabilir, çok sınırlı olarak da dominant markörler ile kullanılabilir. Bunun nedeni dominant marköre göre yapılan tahminin gerçek değerden daha düşük olmasıdır. � Polimorfik bir genin genellikle en yaygın bulunan allelinin frekansı 0, 95’den azdır. � 0, 005’den daha düşük frekansa sahip olan alleller genellikle “nadir alleller” olarak tanımlanırlar. 0, 95 (veya 0, 99) olarak ayarlanan alel frekansı sınırı araştırıcının isteğine kalmıştır. Bunun amacı sadece allelik varyasyonun yaygın olduğu genlerin belirlenmesine yardımcı olmaktır.

P = npj/ntoplam �P: Polimorfik lokusların oranı �npj = polimorfik lokusların sayısı �ntoplam = monomorfik + polimorfik lokusların toplam sayısı • Bir populasyondaki allelik varyasyon gösteren lokusların yüzdesini ifade eder. • Hesaplanması doğrudan polimorfik ve monomorfik lokusların sayılmasına dayanır. • Kodominant markörler ve çok sınırlı olarak da dominant markörler ile kullanılabilir.

Allelik varyantların zenginliği (A) �Bir örnekteki variant sayısını ifade eder �Çeşitliliğin ölçüsü (A - 1) varyanttır. Monomorfik bir populasyon içinde çeşitlilik derecesi 0’dır (A-1=0). • Bir örnekteki verilen bir gen için bu ölçü kaç tane allelik varyantın bulunabileceğini ifade eder. • Örnek sayısına hassastır. • Allellerin dağılımı değil sayısı önemlidir. • Ölçüm sadece kodominant markörler ile yapılır.

Lokus başına ortalama allel sayısı Tüm lokuslardaki tespit edilen allellerin toplamının toplam lokus sayısına bölünmesi ile elde edilir. K = Lokus sayısı ni = lokus başına tespit edilen allel sayısı • Bu ölçü polimorfizm bilgisi için tamamlayıcı bilgi sağlar. • Her bir lokustaki allel sayılarının toplanıp ortalamanın hesaplanmasıdır. • En iyi kodominant markörler ile uygulanabilir. Dominant markörler tüm allellerin tespitine imkan vermez.

Etkili allel sayısı (Ae) Ae = 1/(1 – h) = 1/ pi 2 � Populasyonda bulunabilecek tüm allellerin sayısıdır. pi = Bir lokustaki i’nci allelin frekansıdır. Bir lokustaki heterozigotluk: h= 1 - pi 2 • Bu ölçü her bir populasyon için belirli bir lokusta beklenen allel sayısını verir. • Bir lolustaki homozigotluğun tersi alınarak hesaplanır. • Kodominant markör verisi kullanılarak hesaplanabilir. • Örnek sayısından etkilenir. *Bu çeşitlilik ölçüsü, örnekleme stratejileri oluşturmak için bilgilendirici olabilir. Örneğin, belirli bir örneklemde tespit ettikten sonra bu değeri daha farklı bir örnek ya da örnek koleksiyonunda doğruluyoruz. İkinci seferde elde edilen rakam ilk tahmin edilen rakamdan azsa, bu, örnekleme stratejimizin yeniden gözden geçirilmesi gerektiği anlamına gelebilir.

Etkili allel sayısı (Ae) Hesaplama
![Ortalama beklenen heterozigotluk He Neie göre genetik çeşitlilik D Genetik çeşitlilik populasyondan rasgele çekilen Ortalama beklenen heterozigotluk (He) (Nei’e göre genetik çeşitlilik [D]) �Genetik çeşitlilik populasyondan rasgele çekilen](https://slidetodoc.com/presentation_image_h2/935089b624f3eb5a07324798d86cdf42/image-15.jpg)
Ortalama beklenen heterozigotluk (He) (Nei’e göre genetik çeşitlilik [D]) �Genetik çeşitlilik populasyondan rasgele çekilen belirli bir lokustaki herhangi iki allelin birbirinden farklı olması ihtimalidir. Hesaplanması: • İki allelli bir j lokusunda: hj = 1 – p 2 – q 2 • i sayıda alleli olan j lokusunda : hj = 1 – pi 2 • Birden çok lokus için : H = j. Lhj/L hj =lokus başına heterozigotluk p ve q = allel frekansları H = birden çok lokus için ortalama heterozigotluk L = Toplam lokus sayısı

�Ortalama beklenen heterozigotluk bir lokustaki beklenen homozigotların frekansının toplamı 1’den çıkarılarak bulunabilir. Her lokus içi bu tekrarlanır ve ortalaması alınır. �Hem kodominant hem de dominant markörlerde kullanılabilir. �Tahmin edilen değer yüksek frekanslara sahip allellerin varlığından etkilenebilir. � 0 ile 1 arasında bir değer alır. �Eşit frekanslara sahip çok sayıda allelin varlığında en yüksek değerine ulaşır. �Minimum of 30 lokus ve populasyon başına 20 bireyin analiz edilmesi istatistiki eğilimleri önlemek açısından önerilmektedir.

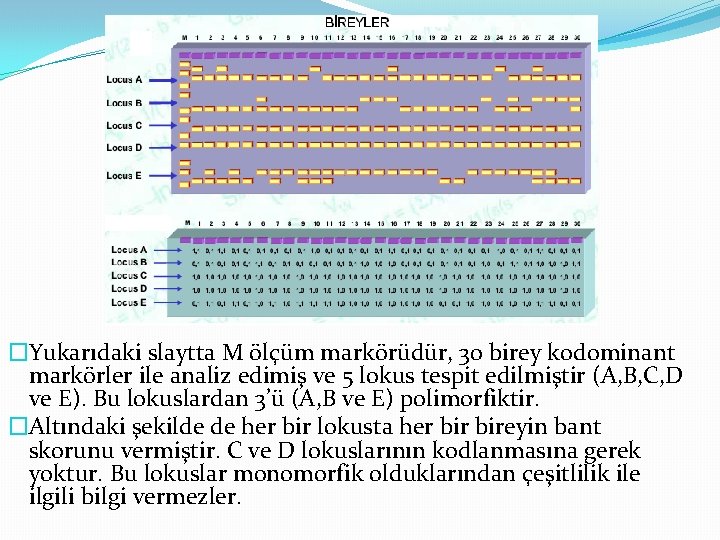
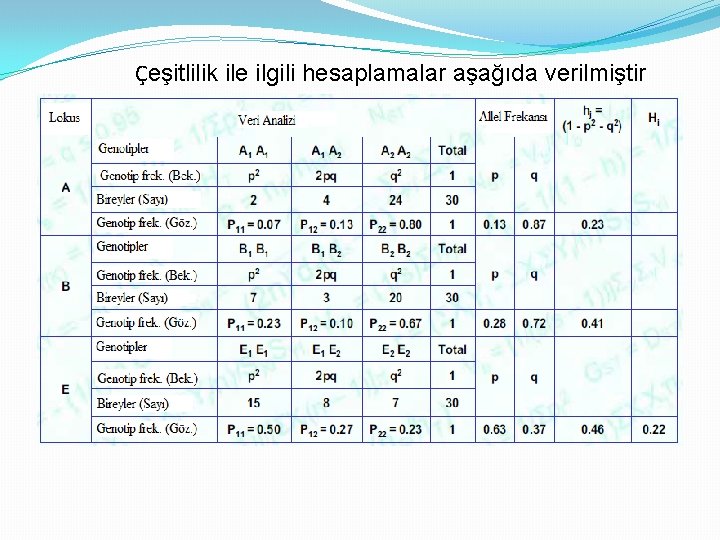
�Yukarıdaki slaytta M ölçüm markörüdür, 30 birey kodominant markörler ile analiz edimiş ve 5 lokus tespit edilmiştir (A, B, C, D ve E). Bu lokuslardan 3’ü (A, B ve E) polimorfiktir. �Altındaki şekilde de her bir lokusta her bireyin bant skorunu vermiştir. C ve D lokuslarının kodlanmasına gerek yoktur. Bu lokuslar monomorfik olduklarından çeşitlilik ile ilgili bilgi vermezler.

Çeşitlilik ile ilgili hesaplamalar aşağıda verilmiştir

� Buna göre: � 1. A, B ve E lokusları polimorfiktir çünkü allel frekansları 0, 99’un altındadır. C ve D lokusları ise monomorfiktir. � 2. Polimorfik lokusların oranı P=(3/5)=0, 6=%60. Bu değer polimorfik lokusların oranının analiz edilen tüm lokuslara bölünmesiyle elde edilir. � 3. Ortalama heterozigotluk (H 0) hesaplanırken; a. Toplamın içinde kaç lokusun heterozigot olduğu hesaplanır. Örneğin 1 numaralı birey 1 heterozigot lokusa sahiptir (A), 2 numaralı birey de 1 heterozigot lokusa sahiptir (A), 27 numaralı birey 2 heterozigot lokusa sahiptir (Ave E), …. Toplamda 16 birey monomorfik (5 lokusun her birinde tek band var), 13 birey 1 heterozigot lokusa sahip ve 1 birey de 2 heterozigot lokusa sahiptir. b. Ortalama gözlenen heterozigotluk : Ho = [16(0/5) + 13(1/5) + 1(2/5)]/(30) = 0. 1 Lokus içi gen çeşitliliği (hj)= 1 -p 2 -q 2 buna gore; A lokusu=0, 23 B lokusu=0, 41 ve E lokusu=0, 46 Ortalama beklenen heterozigotluk (Hi)= j. Lhj/L Hi = (0. 23 + 0. 41 + 0. 46)/5 = 0. 22

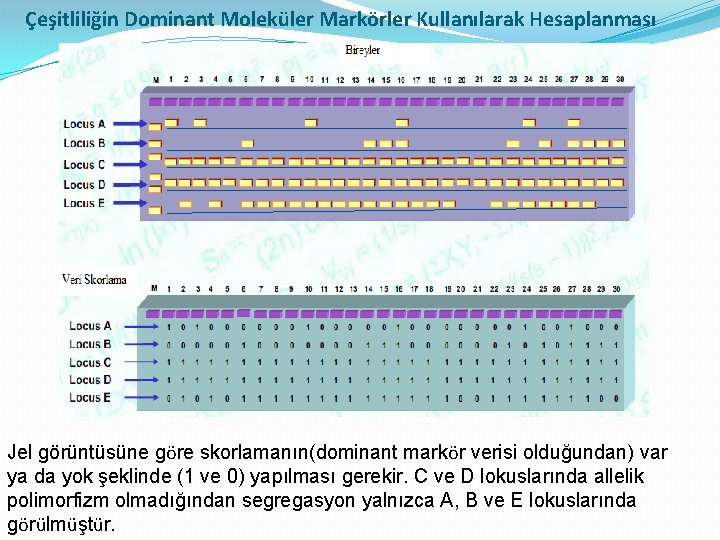
Çeşitliliğin Dominant Moleküler Markörler Kullanılarak Hesaplanması Jel görüntüsüne göre skorlamanın(dominant markör verisi olduğundan) var ya da yok şeklinde (1 ve 0) yapılması gerekir. C ve D lokuslarında allelik polimorfizm olmadığından segregasyon yalnızca A, B ve E lokuslarında görülmüştür.

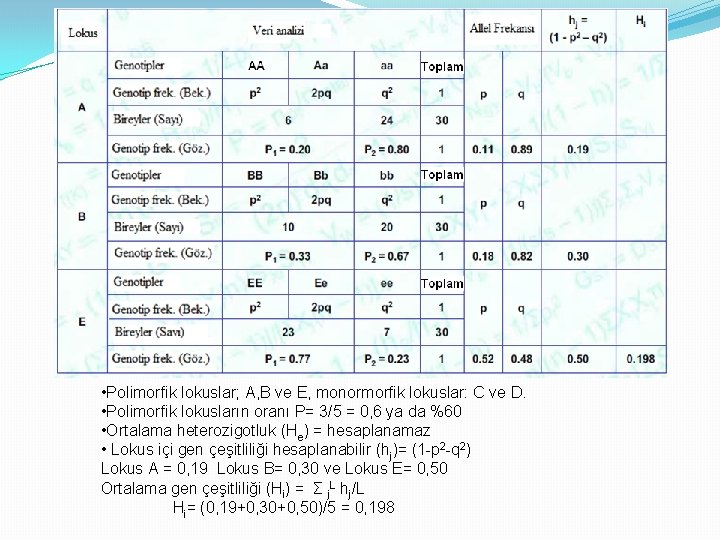
• Polimorfik lokuslar; A, B ve E, monormorfik lokuslar: C ve D. • Polimorfik lokusların oranı P= 3/5 = 0, 6 ya da %60 • Ortalama heterozigotluk (He) = hesaplanamaz • Lokus içi gen çeşitliliği hesaplanabilir (hj)= (1 -p 2 -q 2) Lokus A = 0, 19 Lokus B= 0, 30 ve Lokus E= 0, 50 Ortalama gen çeşitliliği (Hi) = Σ j. L hj/L Hi= (0, 19+0, 30+0, 50)/5 = 0, 198

POPULASYONLAR ARASI GENETİK ÇEŞİTLİLİĞİN ÖLÇÜLMESİ �Bir lokus bakımından populasyonlar arası farklılık (g. ST) �Birden fazla lokus bakımından populasyonlar arası farklılık (GST) �Populasyonun toplam genetik çeşitliliğe katkısı �F istatistikleri (Wright) �AMOVA (Moleküler varyans analizi)

� Genetik farklılaşma (differentiation); populasyonların genetik yapısında değişik düzeylerdeki (birey ve populasyon düzeyinde) polimorfik farklılıkları ifade eder. �Bir lokus bakımından populasyonlar arası farklılık : g. ST= 1 - (hs/ht) h. S = populasyon çeşitliliği h. T = toplam çeşitlilik hs = (ñ/(ñ - 1)[1 – (1/s)ΣΣXij 2 – (ho/2ñ)] h. T = 1 - Σ [(1/s) ΣXij]2 + (hs/ñs) – (ho/2ñs) ñ = populasyon büyüklüklerinin harmonik ortalaması s= populasyon sayısı ho = ortalama gözlenen heterozigotluk Xij = j’inci populasyondaki i’nci allelin beklenen frekansı

g. ST= 1 - (hs/ht) formülü; �Her bir lokus başına düşen alleller bağlamında iki ya da daha fazla populasyonun genetik farklılıklarının ölçülmesini sağlar. � 0 -1 arasında değişir. �Negatif değer alması örnekleme hatasından ya da hatalı markör sistemi kullanılmasından kaynaklanabilir. �Bileşenlerin hesaplanmasının oldukça karmaşık olması özelleşmiş bilgisayar programları kullanılmasını gerektirir. �Kodominant markörlerle, sınırlı olarak da dominant markörlerle kullanılabilir. Gerçek değerin doğru bir şekilde hesaplanabilmesi için birkaç jenerasyona ihtiyaç vardır.

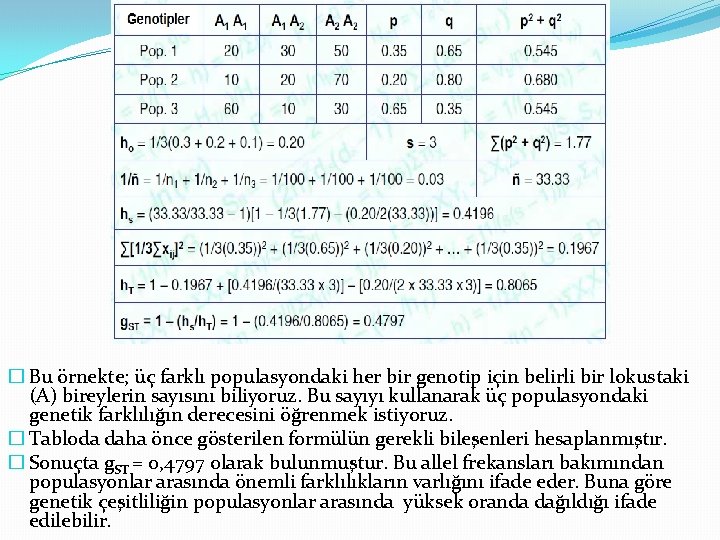
� Bu örnekte; üç farklı populasyondaki her bir genotip için belirli bir lokustaki (A) bireylerin sayısını biliyoruz. Bu sayıyı kullanarak üç populasyondaki genetik farklılığın derecesini öğrenmek istiyoruz. � Tabloda daha önce gösterilen formülün gerekli bileşenleri hesaplanmıştır. � Sonuçta g. ST = 0, 4797 olarak bulunmuştur. Bu allel frekansları bakımından populasyonlar arasında önemli farklılıkların varlığını ifade eder. Buna göre genetik çeşitliliğin populasyonlar arasında yüksek oranda dağıldığı ifade edilebilir.

Birden fazla lokus için populasyonlar arası farklılık (GST) �GST gen farklılığının katsayısıdır. Burada; HT= Toplam gen çeşitliliği = HS+ DST HS= Populasyon içi gen çeşitliliği DST=Populasyonlar arası çeşitlilik (HT/HT) =(HS/HT) + (DST/HT) = 1

�GST tüm populasyonlarda gen çeşitliliği oranının dağılımını ölçer. �Çok sayıda lokus kullanılmalıdır. �Eşitlikler oldukça kompleks yapıdadır bu yüzden hesaplamada özelleşmiş programlara gereksinim vardır. �Örneğin; HT = 0, 263 HS = 0, 202 olsun DST = 0, 263 -0. 202 = 0, 061 �GST = DST/HT * 100 = (0, 061/0, 263) * 100 = %23, 19 �Bu şu anlama gelir; çalışılan bu türün populasyonları arasındaki farklılık %23’dür.

Populasyonun toplam gen çeşitliliğine katkısı �Bu değer bir populasyonun çıkarılmasıyla toplam gen çeşitliliğin nasıl değiştiği tespit edilerek hesaplanır. Burada; CT(K) = K’nın toplam çeşitliliğe katkısı CS(K) = K’nın populasyon içi çeşitliliğe katkısı CST(K) = K’nın populasyonlar arası çeşitliliğe katkısı HT = toplam gen çeşitliliği HS = populasyon içi gen çeşitliliği DST = populasyonlar arası farklılık HT/K= K populasyonu çıkarıldıktan sonra gen çeşitliliği HS/K = K populasyonu çıkarıldıktan sonra populasyon içi gen çeşitliliği DST/K= K populasyonu çıkarıldıktan sonra populasyonlar arası gen çeşitliliği Bu ölçüm bir populasyonun eklenmesi ya da çıkarılması durumunda toplam gen çeşitliliğinde meydana gelen değişimin anlaşılmasını sağlar. Sadece kodominant markör sistemleri kullanılarak hesaplanır.

Wright’ın Fiksasyon indeksi (FST) Sewall Wright 1889 -1988

F istatistikleri (Wright) F istatistikleri alt populasyonlar düzeyinde genetik yapının analizi hakkında bilgi verir. Ayrıca alt populasyonlar arasındaki genetik uzaklığın hesaplanmasında kullanılabilir. Burada temel varsayım toplam populasyonu oluşturan ve aralarında çiftleşmenin gerçekleşemediği alt populasyonların farklı allel frekanslarına sahip olabileceğidir. � Genetik mesafe endogami durumunda olduğu gibi eş allelerin bir araya gelme olasılığının da ölçülmesini sağlar. Populasyonların genetik yapıları ile ilgili eşitlik: HT= Populasyonun tamamında tüm allel frekansları dikkate alınarak hesaplanan toplam gen çeşitliliği ya da beklenen heterozigotluk (=2 pq). HI= alt populasyonlar içinde her birey için ortalama gözlenen heterozigotluk HS = Alt populasyonlar içi ortalama beklenen heterozigotluk FIS = Her bir populasyonda ortalama heterozigotların fazlalık ya da azlığı (Akrabalı yetiştirme katsayısı) FST = Allel frekansları bağlamında populasyonlar arası gen farklılığının düzeyi FIT= Bir grup populasyonda ortalama heterozigotluğun azlık ya da çokluğu

FST değerlerinin yorumlanması Genetik farklılık yok Farklı alt populasyonlarda alternatif alleller sabitlenmiş FST; 0 ile 0, 05 arasındaysa genetik farklılık düşük 0, 05 ile 0, 15 arasındaysa genetik farklılık orta 0, 15 ile 0, 25 arasındaysa genetik farklılık yüksek >0, 25 ise genetik farklılık çok yüksek
 Verileri ışık hızıyla ileten kablo türü
Verileri ışık hızıyla ileten kablo türü Sünnimärkide uuring
Sünnimärkide uuring Genetik grundlagen
Genetik grundlagen Apa itu genetik
Apa itu genetik Canlılarda kalıtımı sağlayan genetik birim
Canlılarda kalıtımı sağlayan genetik birim Ragam genetik adalah
Ragam genetik adalah Genetik dan kesihatan
Genetik dan kesihatan Organel yang memilih memodifikasi dan membungkus protein
Organel yang memilih memodifikasi dan membungkus protein Biokemi og genetik
Biokemi og genetik Genetik kopyalama
Genetik kopyalama Korsningsschema ögonfärg
Korsningsschema ögonfärg Biokemi og genetik
Biokemi og genetik Allelen
Allelen Poliploidi nedir
Poliploidi nedir Allele genetik
Allele genetik Substansi genetik dna dan rna mempunyai kesamaan yaitu
Substansi genetik dna dan rna mempunyai kesamaan yaitu Kepribadian genetik atau hasil belajar
Kepribadian genetik atau hasil belajar Spiral genetik
Spiral genetik Dasar genetik tanaman menyerbuk silang
Dasar genetik tanaman menyerbuk silang Kata serapan baku
Kata serapan baku är kaniner färgblinda
är kaniner färgblinda Partenogenez
Partenogenez Elefatiasis
Elefatiasis Laktosin
Laktosin Makalah strukturalisme genetik
Makalah strukturalisme genetik Gömlek giyme beceri analizi
Gömlek giyme beceri analizi Artık kareler toplamı
Artık kareler toplamı Balık kılçığı diyagramı
Balık kılçığı diyagramı Makas tutma beceri analizi
Makas tutma beceri analizi Kalitatif risk değerlendirme yöntemleri
Kalitatif risk değerlendirme yöntemleri Farkn
Farkn Dikey yüzde analizi
Dikey yüzde analizi