Trascrizione e traduzione La finalit dellespressione genica quella


















- Slides: 47

Trascrizione e traduzione

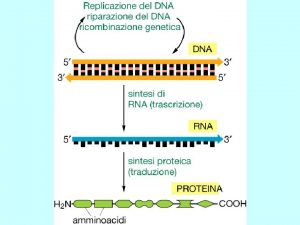
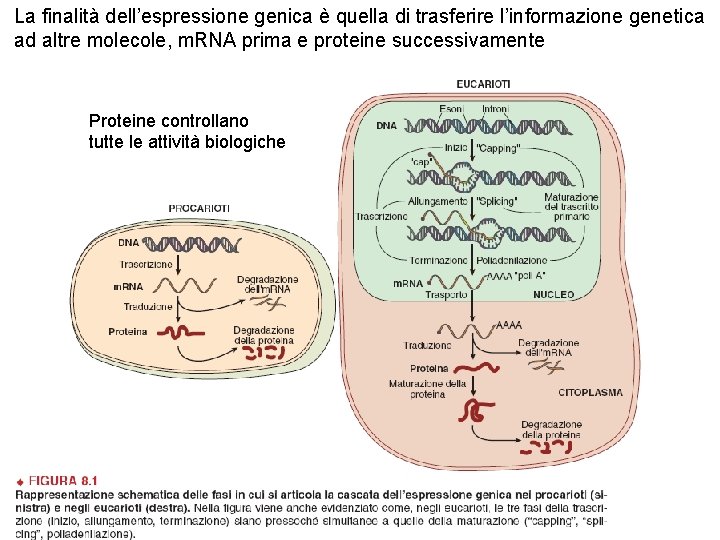
La finalità dell’espressione genica è quella di trasferire l’informazione genetica ad altre molecole, m. RNA prima e proteine successivamente Proteine controllano tutte le attività biologiche

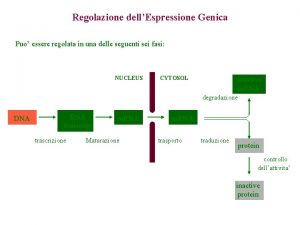
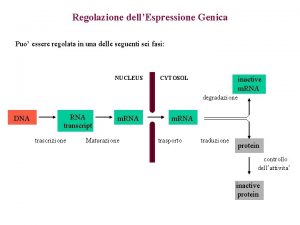
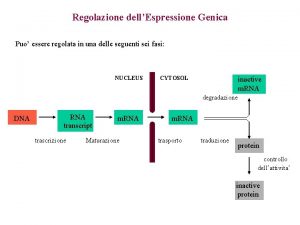
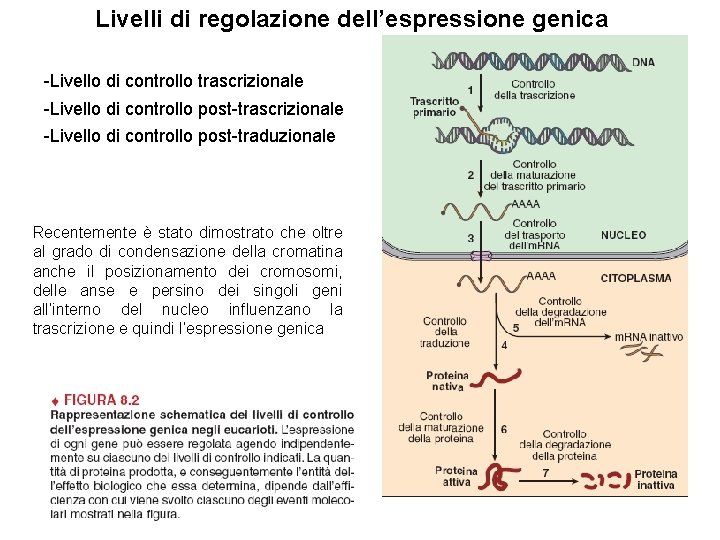
Livelli di regolazione dell’espressione genica La regolazione dell’espressione genica è un processo cruciale al fine di controllare qualitativamente e quantitativamente gli effetti biologici modulati dalle varie proteine - Diversificazione biologica, differenziamento cellulare di un organismo pluricellulare - Diversità di stati funzionali che una cellula, un tessuto o un organismo possono realizzare in diversi momenti della loro vita Livello di controllo trascrizionale Livello di controllo post-traduzionale

Livelli di regolazione dell’espressione genica -Livello di controllo trascrizionale -Livello di controllo post-traduzionale Recentemente è stato dimostrato che oltre al grado di condensazione della cromatina anche il posizionamento dei cromosomi, delle anse e persino dei singoli geni all’interno del nucleo influenzano la trascrizione e quindi l’espressione genica

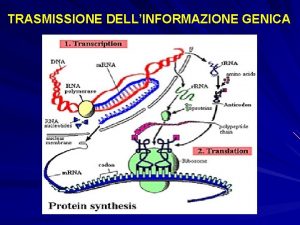
Trascrizione reazione biosintetica catalizzata dall’enzima RNA polimerasi attraverso la quale viene prodotta una molecola di RNA a partire da una sequenza di DNA -RNA è costituito da unità monomeriche di nucleotidi -I singoli nucleotidi sono uniti fra loro attraverso legami fosfodiestere che si formano tra lo zucchero di un nucleotide e il fosfato del nucleotide successivo. -L’RNA è composto da ribonucleotidi, il DNA da deossiribonucleotidi; -Le molecole di m. RNA consistono di una sola catena polinucleotidica; -Le molecole di DNA sono a doppio filamento, con le due catene polinucleotidiche tenute assieme da legami idrogeno che si formano tra le basi azotate. I due filamenti di DNA hanno direzione opposta. -la timina, contenuta nel DNA, è sostituita con l’uracile

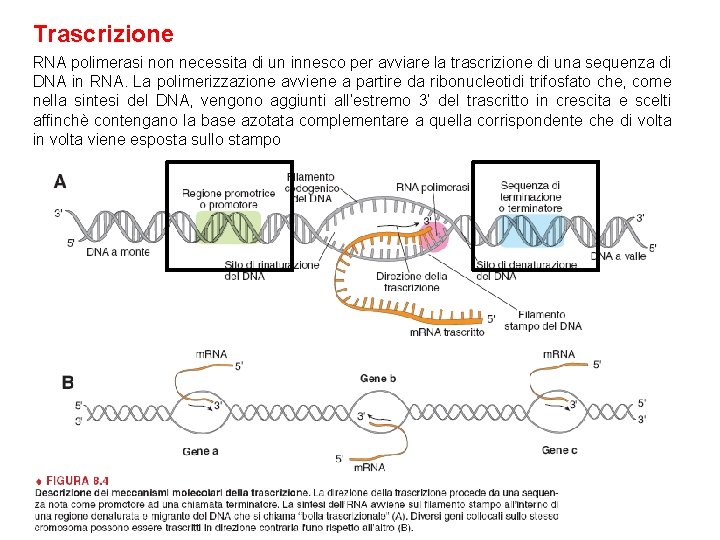
Trascrizione RNA polimerasi non necessita di un innesco per avviare la trascrizione di una sequenza di DNA in RNA. La polimerizzazione avviene a partire da ribonucleotidi trifosfato che, come nella sintesi del DNA, vengono aggiunti all’estremo 3’ del trascritto in crescita e scelti affinchè contengano la base azotata complementare a quella corrispondente che di volta in volta viene esposta sullo stampo

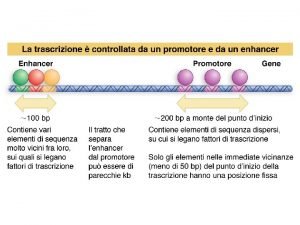
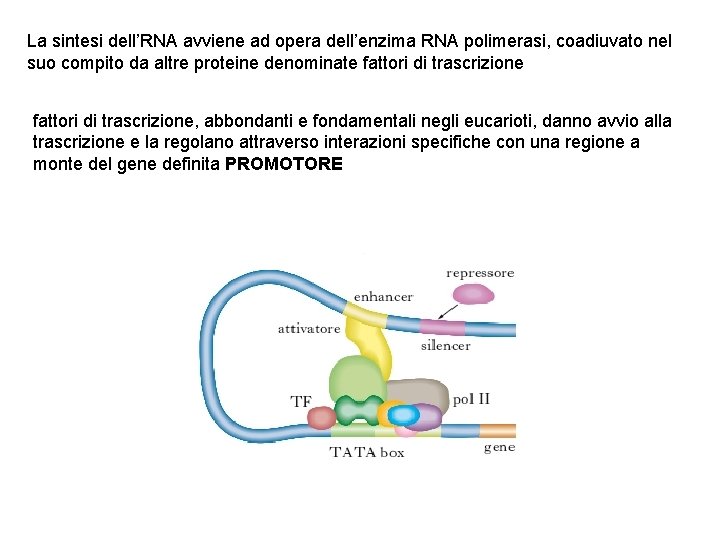
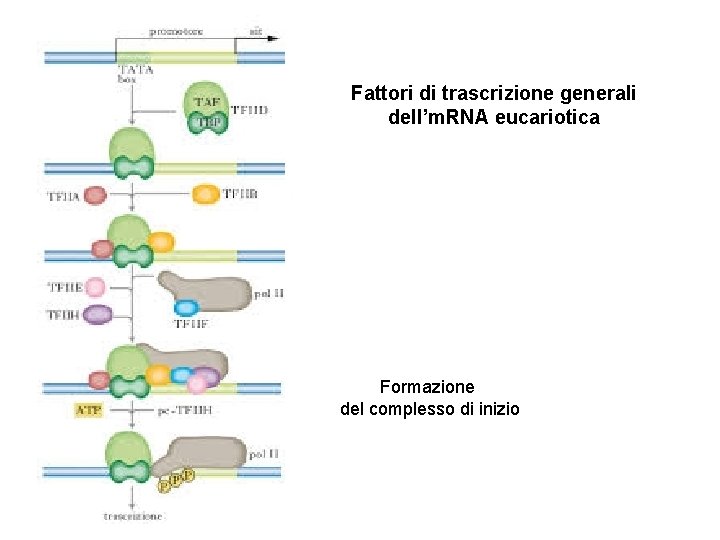
La sintesi dell’RNA avviene ad opera dell’enzima RNA polimerasi, coadiuvato nel suo compito da altre proteine denominate fattori di trascrizione, abbondanti e fondamentali negli eucarioti, danno avvio alla trascrizione e la regolano attraverso interazioni specifiche con una regione a monte del gene definita PROMOTORE

La trascrizione si articola in tre fasi: -Inizio -Allungamento -Terminazione

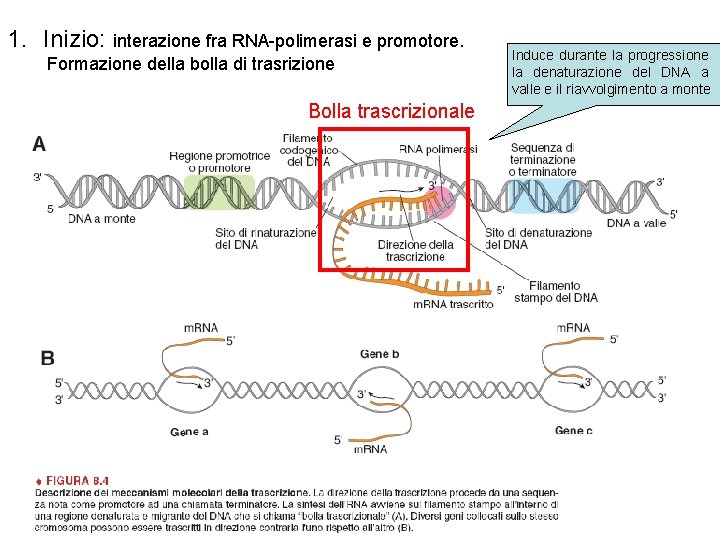
1. Inizio: interazione fra RNA-polimerasi e promotore. Formazione della bolla di trasrizione Bolla trascrizionale Induce durante la progressione la denaturazione del DNA a valle e il riavvolgimento a monte

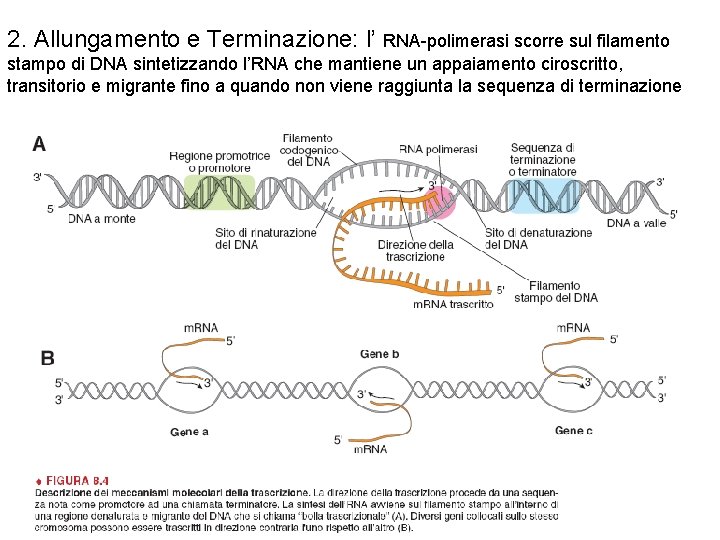
2. Allungamento e Terminazione: l’ RNA-polimerasi scorre sul filamento stampo di DNA sintetizzando l’RNA che mantiene un appaiamento ciroscritto, transitorio e migrante fino a quando non viene raggiunta la sequenza di terminazione

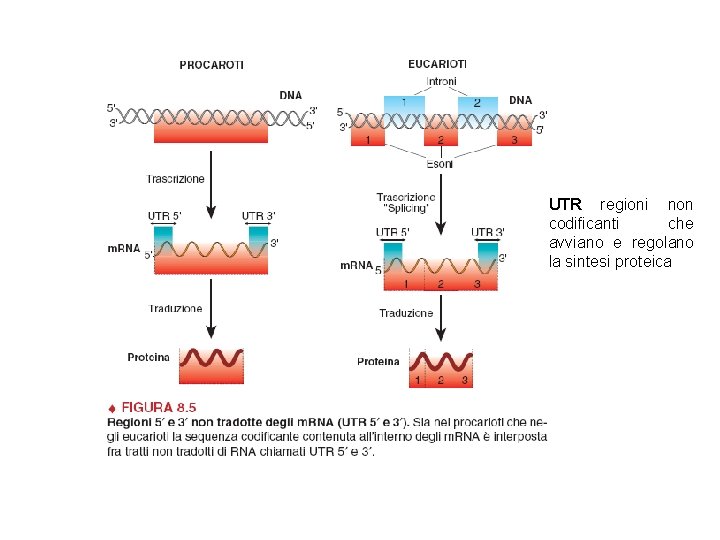
UTR regioni non codificanti che avviano e regolano la sintesi proteica

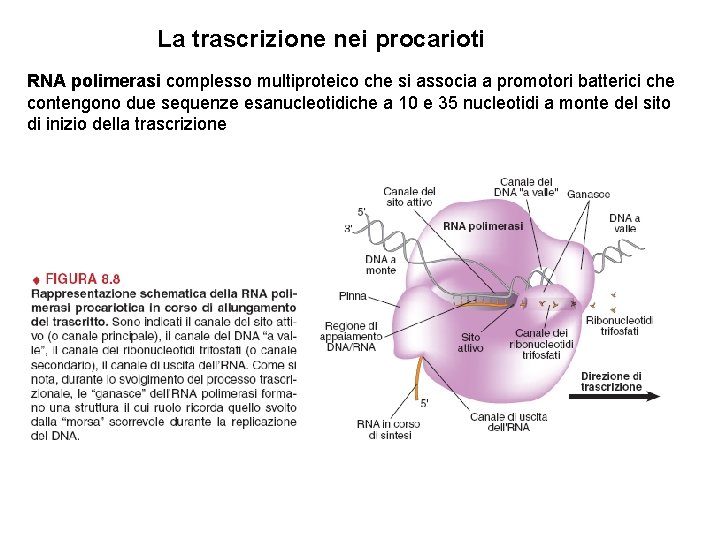
La trascrizione nei procarioti RNA polimerasi complesso multiproteico che si associa a promotori batterici che contengono due sequenze esanucleotidiche a 10 e 35 nucleotidi a monte del sito di inizio della trascrizione

La subunità s media il legame con le regioni -35 e -10 Denaturazione del DNA autonoma Rilascio del promotore, Terminatori intrinseci, sequenza palindromica ripetuta invertita


La trascrizione negli eucarioti 80% r. RNA 15% RNA non codificante 5% m. RNA

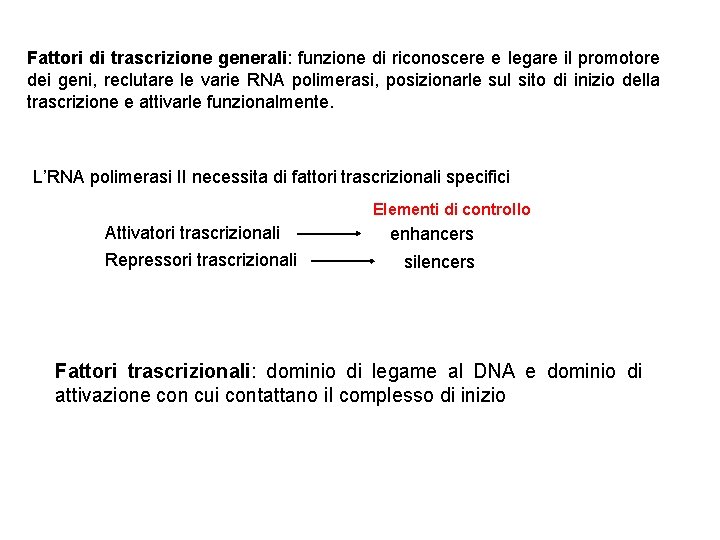
Fattori di trascrizione generali: funzione di riconoscere e legare il promotore dei geni, reclutare le varie RNA polimerasi, posizionarle sul sito di inizio della trascrizione e attivarle funzionalmente. L’RNA polimerasi II necessita di fattori trascrizionali specifici Elementi di controllo Attivatori trascrizionali Repressori trascrizionali enhancers silencers Fattori trascrizionali: dominio di legame al DNA e dominio di attivazione con cui contattano il complesso di inizio

Fattori di trascrizione generali dell’m. RNA eucariotica Formazione del complesso di inizio

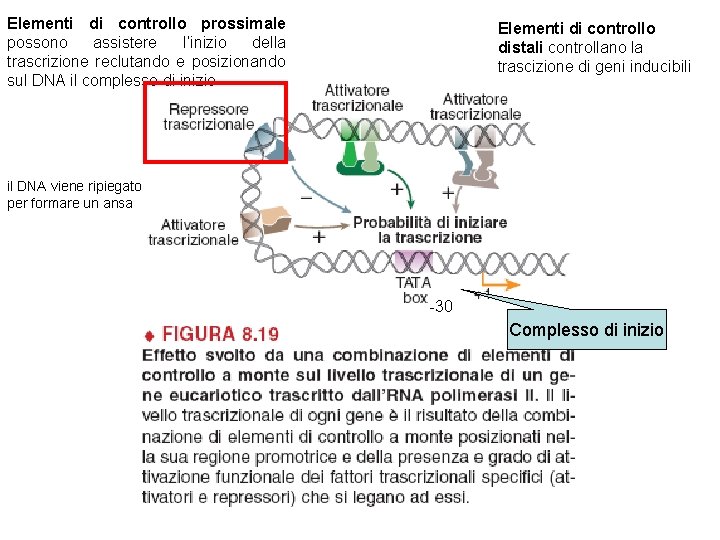
Elementi di controllo prossimale possono assistere l’inizio della trascrizione reclutando e posizionando sul DNA il complesso di inizio Elementi di controllo distali controllano la trascizione di geni inducibili il DNA viene ripiegato per formare un ansa -30 Complesso di inizio

Meccanismi che regolano l’attività di un fattore di trascrizione: -Eterodimerizzazione -Modificazioni post-traduzionali -Localizzazione subcellulare -Interazione con un ligando

L’attività inibitoria dei repressori viene esercitata attraverso una attività di interferenza nei confronti degli attivatori che può svolgersi con diversi meccanismi: -Il dominio di legame del repressore compete con quello per il legame di una sequenza di DNA comune -Il dominio di repressione del repressore lega quello di attivazione dell’attivatore impedendogli di interagire con il complesso di inizio -Il dominio dei repressione lega il sito del complesso di inizio normalmente rionosciutp dall’attivatore -Il represso recluta complessi di modificazione e remodellamento della cromatina che induco condensazione cromtina

Il livello di trascrizione di ciasun gene è il risultato della combinazione fra gli elementi di controllo collocati nella sua regione promotrice e l’espressione e il grado di attività dei fattori trascrizionali che si legano ad essi

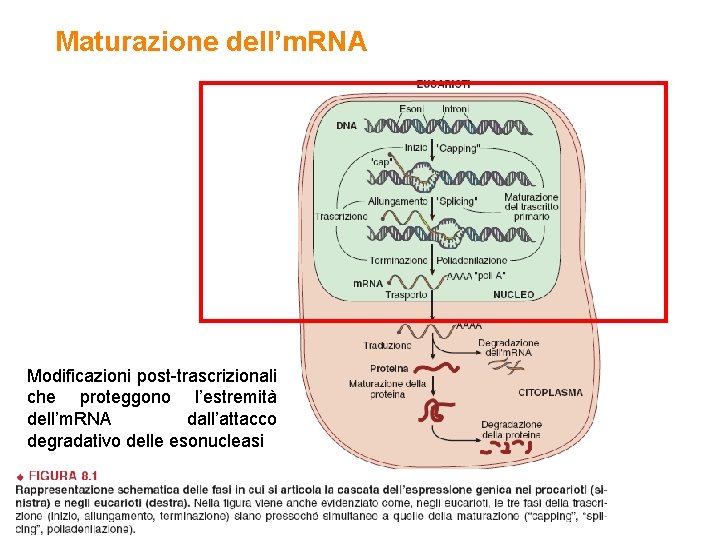
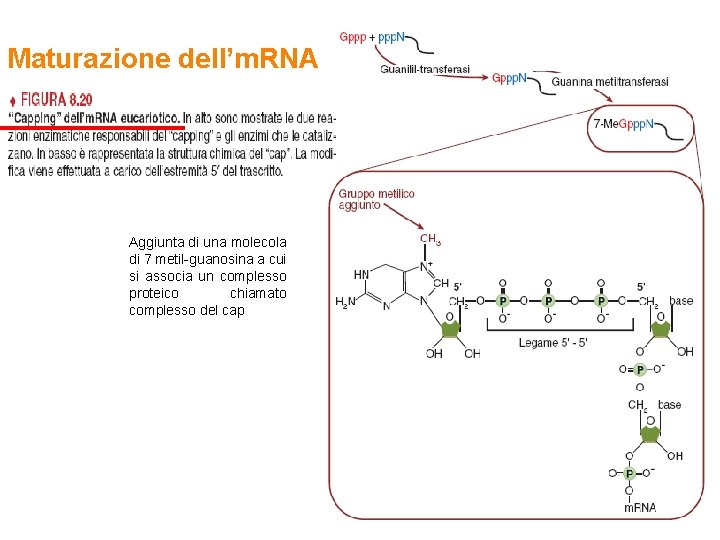
Maturazione dell’m. RNA Modificazioni post-trascrizionali che proteggono l’estremità dell’m. RNA dall’attacco degradativo delle esonucleasi

Maturazione dell’m. RNA Aggiunta di una molecola di 7 metil-guanosina a cui si associa un complesso proteico chiamato complesso del cap

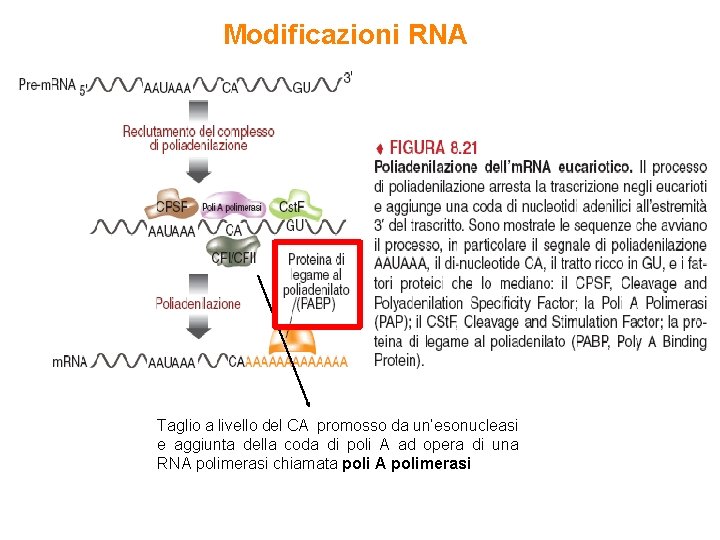
Modificazioni RNA Taglio a livello del CA promosso da un’esonucleasi e aggiunta della coda di poli A ad opera di una RNA polimerasi chiamata poli A polimerasi

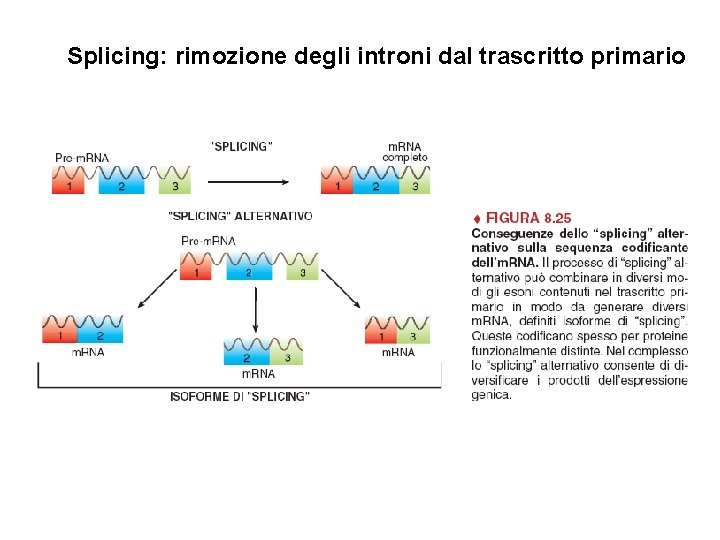
Splicing: rimozione degli introni dal trascritto primario

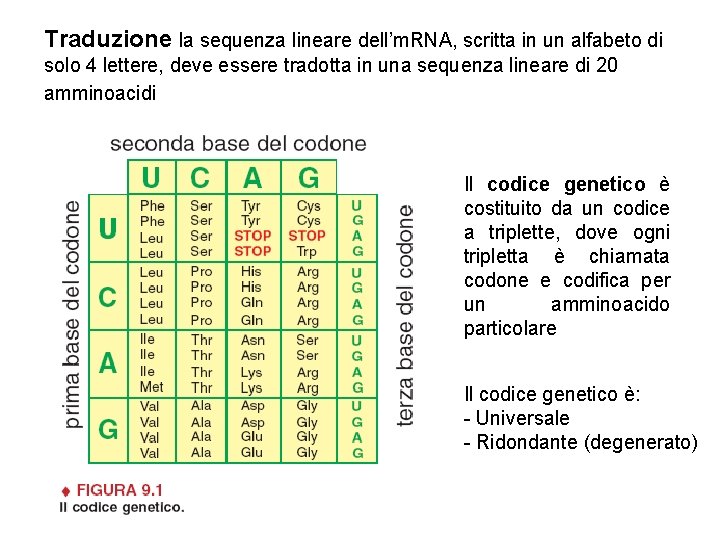
Traduzione la sequenza lineare dell’m. RNA, scritta in un alfabeto di solo 4 lettere, deve essere tradotta in una sequenza lineare di 20 amminoacidi Il codice genetico è costituito da un codice a triplette, dove ogni tripletta è chiamata codone e codifica per un amminoacido particolare Il codice genetico è: - Universale - Ridondante (degenerato)

Nel processo di traduzione lo “stampo” è l’m. RNA, la cui informazione viene letta in triplette, in direzione 5’ 3’ e la proteina corrispondente viene sintetizzata dall’Nterminale al C-terminale Codone di inizio AUG Codone di stop UAA, UAG, UGA

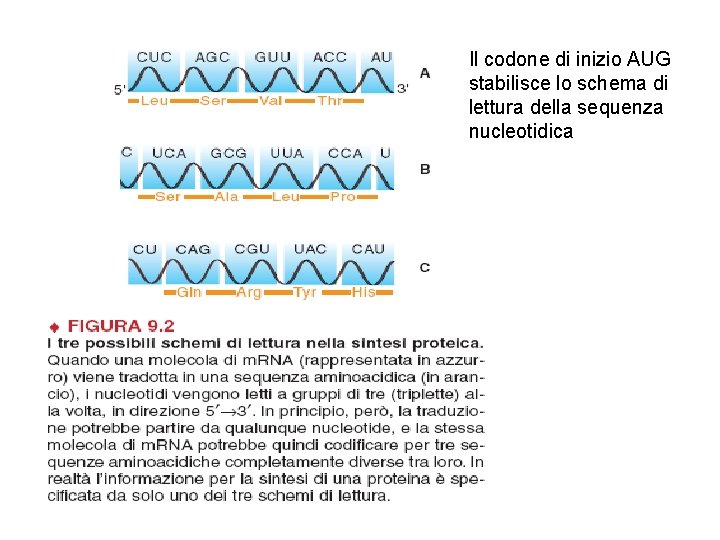
Il codone di inizio AUG stabilisce lo schema di lettura della sequenza nucleotidica

Il processo di decifrazione del codice genetico richiede due classi diverse di molecole che funzionano da interpreti -RNA transfer -Amminoacil-t. RNA sintetasi e il ribosoma un complesso catalitico che permette la sintesi della catena polipetidica

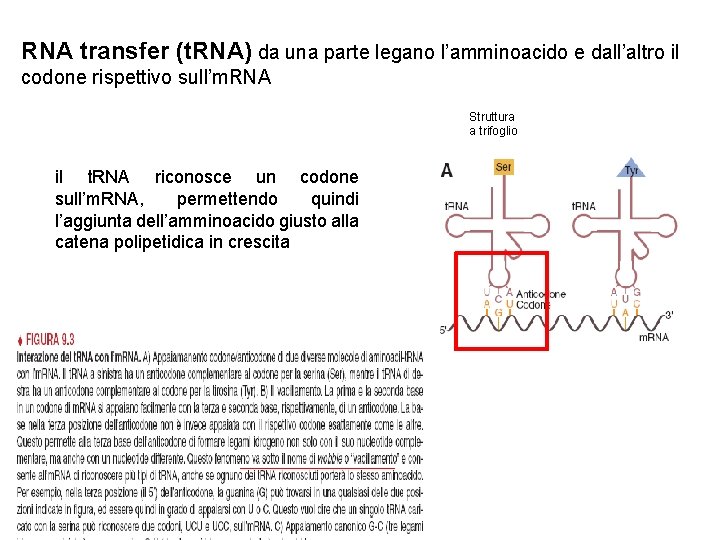
RNA transfer (t. RNA) da una parte legano l’amminoacido e dall’altro il codone rispettivo sull’m. RNA Struttura a trifoglio il t. RNA riconosce un codone sull’m. RNA, permettendo quindi l’aggiunta dell’amminoacido giusto alla catena polipetidica in crescita

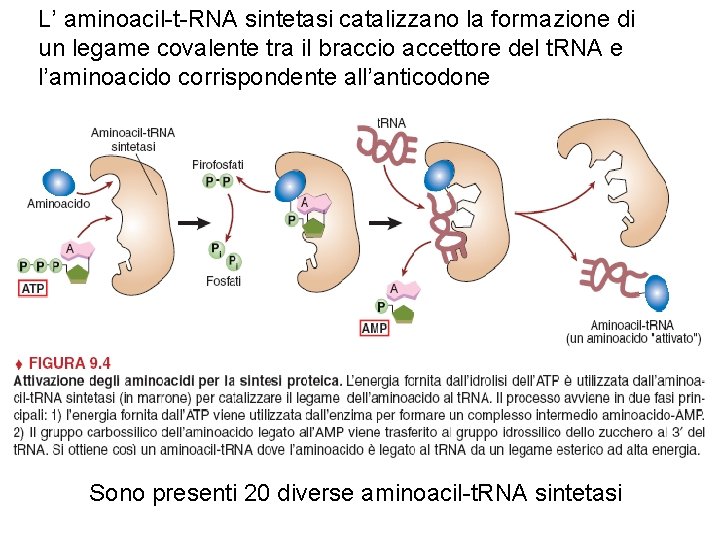
L’ aminoacil-t-RNA sintetasi catalizzano la formazione di un legame covalente tra il braccio accettore del t. RNA e l’aminoacido corrispondente all’anticodone Sono presenti 20 diverse aminoacil-t. RNA sintetasi

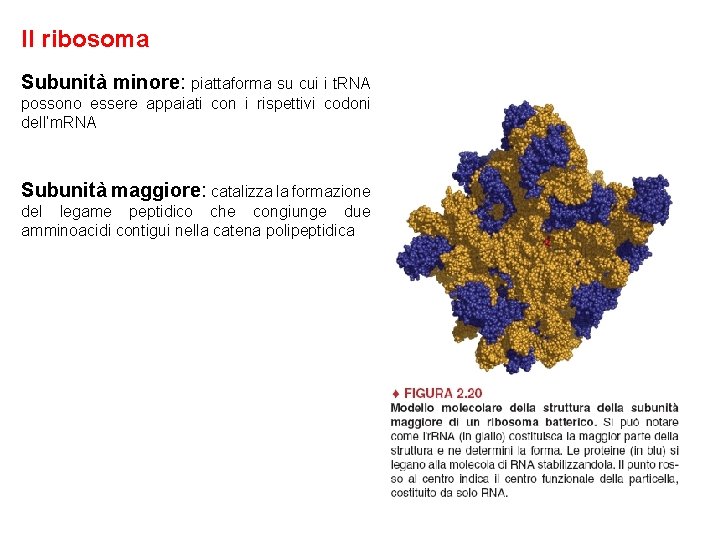
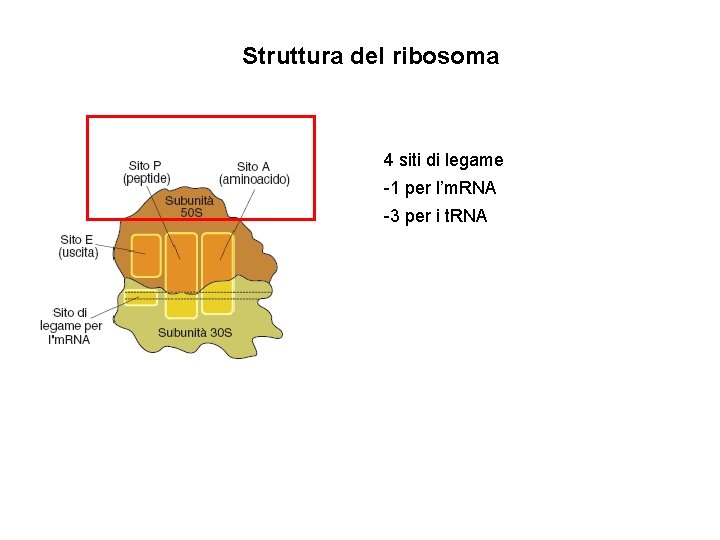
Il ribosoma Subunità minore: piattaforma su cui i t. RNA possono essere appaiati con i rispettivi codoni dell’m. RNA Subunità maggiore: catalizza la formazione del legame peptidico che congiunge due amminoacidi contigui nella catena polipeptidica

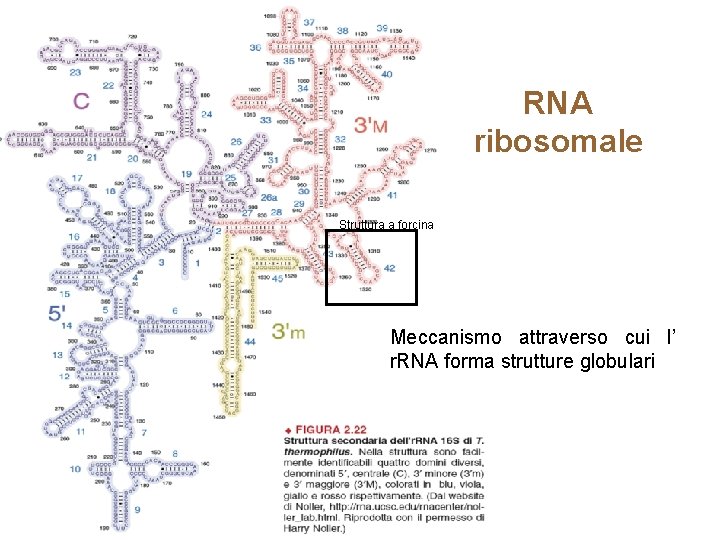
RNA ribosomale Struttura a forcina Meccanismo attraverso cui l’ r. RNA forma strutture globulari


Struttura del ribosoma 4 siti di legame -1 per l’m. RNA -3 per i t. RNA

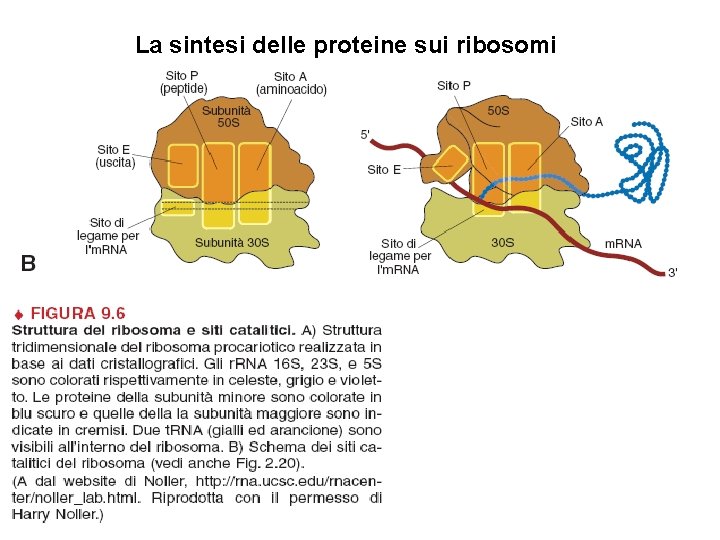
La sintesi delle proteine sui ribosomi

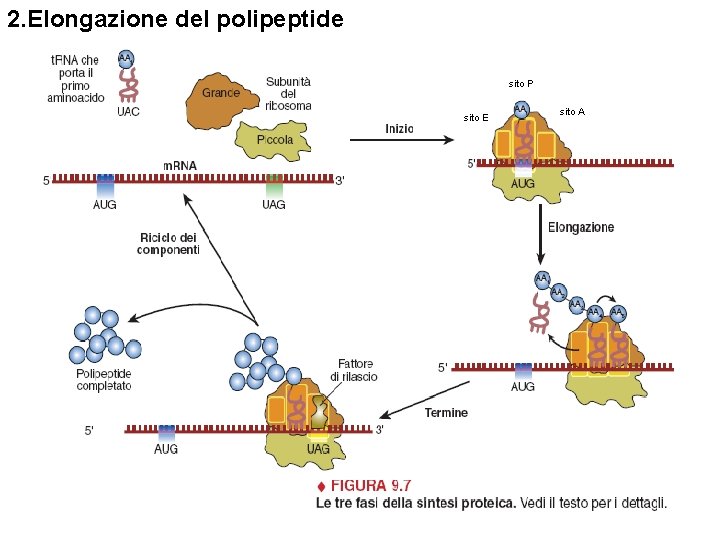
La sintesi delle proteine sui ribosomi -Inizio -Elongazione -Terminazione

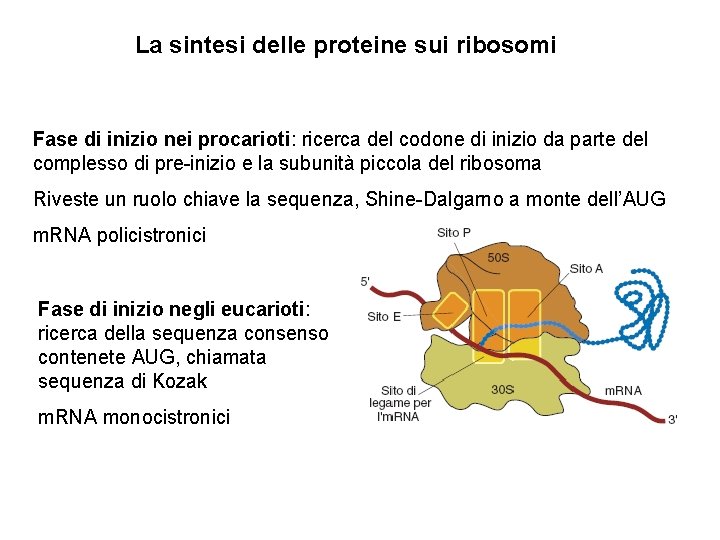
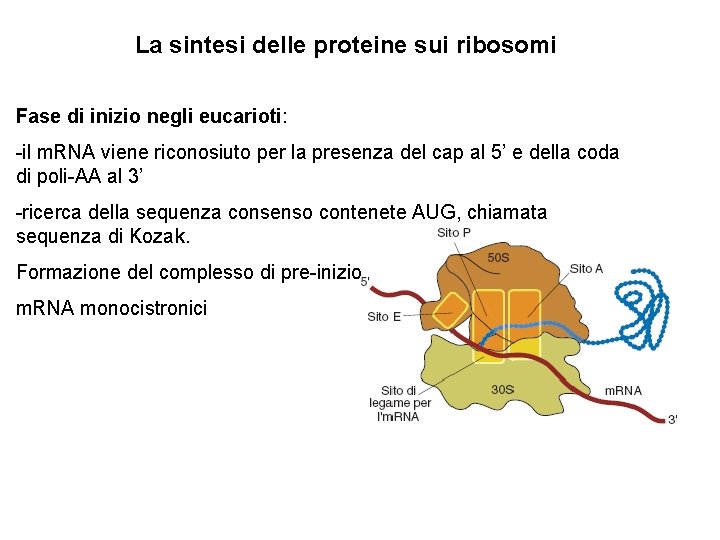
La sintesi delle proteine sui ribosomi Fase di inizio nei procarioti: ricerca del codone di inizio da parte del complesso di pre-inizio e la subunità piccola del ribosoma Riveste un ruolo chiave la sequenza, Shine-Dalgarno a monte dell’AUG m. RNA policistronici Fase di inizio negli eucarioti: ricerca della sequenza consenso contenete AUG, chiamata sequenza di Kozak m. RNA monocistronici

La sintesi delle proteine sui ribosomi Fase di inizio negli eucarioti: -il m. RNA viene riconosiuto per la presenza del cap al 5’ e della coda di poli-AA al 3’ -ricerca della sequenza consenso contenete AUG, chiamata sequenza di Kozak. Formazione del complesso di pre-inizio m. RNA monocistronici

2. Elongazione del polipeptide sito P sito E sito A



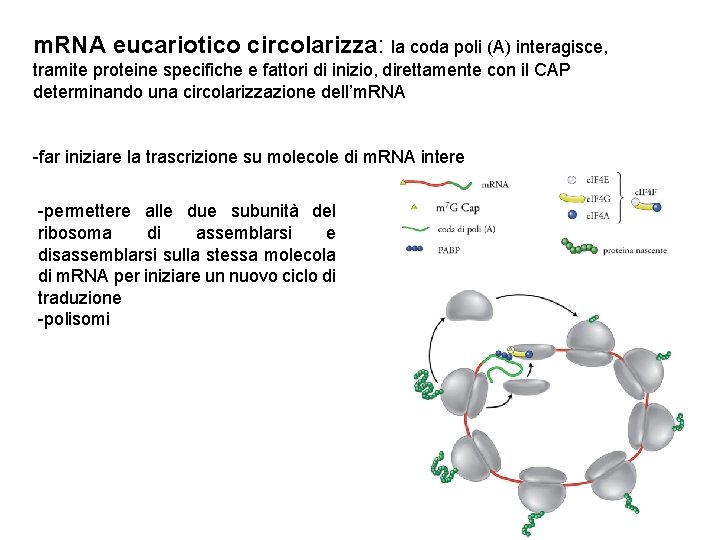
m. RNA eucariotico circolarizza: la coda poli (A) interagisce, tramite proteine specifiche e fattori di inizio, direttamente con il CAP determinando una circolarizzazione dell’m. RNA -far iniziare la trascrizione su molecole di m. RNA intere -permettere alle due subunità del ribosoma di assemblarsi e disassemblarsi sulla stessa molecola di m. RNA per iniziare un nuovo ciclo di traduzione -polisomi

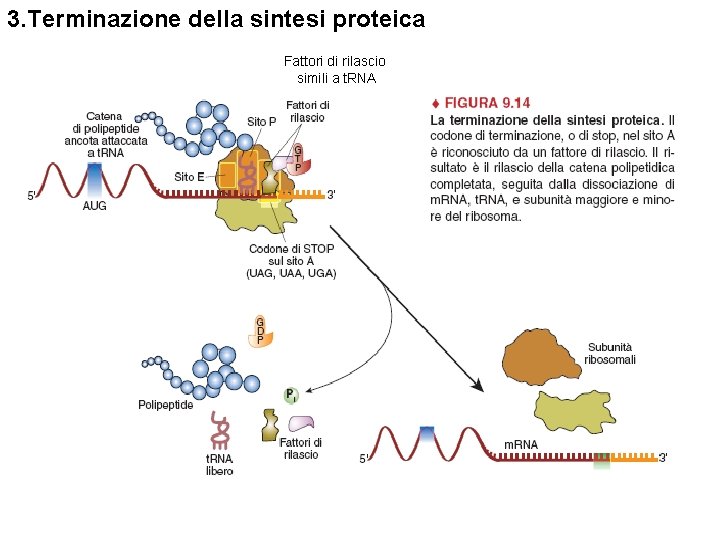
3. Terminazione della sintesi proteica Fattori di rilascio simili a t. RNA



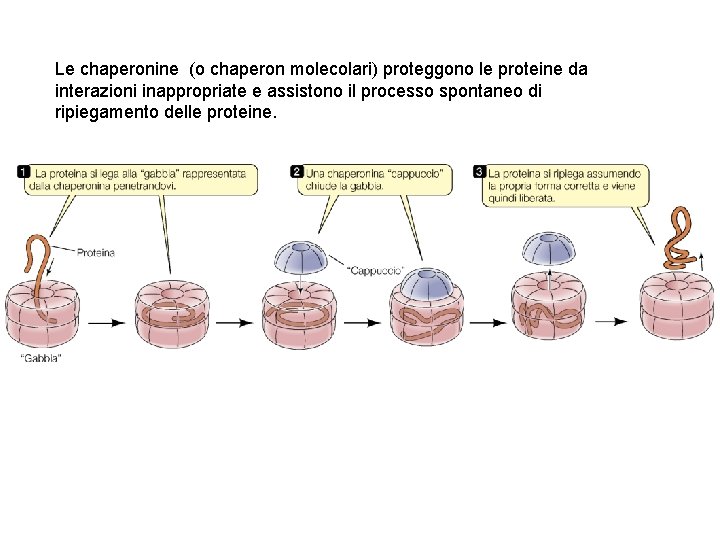
Le chaperonine (o chaperon molecolari) proteggono le proteine da interazioni inappropriate e assistono il processo spontaneo di ripiegamento delle proteine.
 Codifica dei suoni
Codifica dei suoni Fattori di trascrizione
Fattori di trascrizione Cappa maiuscola in corsivo
Cappa maiuscola in corsivo Differenze trascrizione eucarioti e procarioti
Differenze trascrizione eucarioti e procarioti Gatto trascrizione fonetica
Gatto trascrizione fonetica Differenze trascrizione eucarioti e procarioti
Differenze trascrizione eucarioti e procarioti Una cosa leggera
Una cosa leggera Comparaison système scolaire français italien
Comparaison système scolaire français italien Appartengo all'unica razza che conosco quella umana
Appartengo all'unica razza che conosco quella umana Attiva a passiva
Attiva a passiva Biodiversità
Biodiversità Regolazione genica nei procarioti
Regolazione genica nei procarioti Duplicazione genica
Duplicazione genica Regolazione genica eucarioti
Regolazione genica eucarioti Epistatico
Epistatico Sexo
Sexo Duplicazione genica
Duplicazione genica Que son los genes
Que son los genes Mutazioni
Mutazioni Cromosomas sexuales
Cromosomas sexuales Expresion genica
Expresion genica Terapia gênica
Terapia gênica Espressione genica
Espressione genica Regolazione genica zanichelli
Regolazione genica zanichelli Primus amor phoebi
Primus amor phoebi Teoria e storia della traduzione
Teoria e storia della traduzione Screenshot traduzione italiano
Screenshot traduzione italiano Aristoteles quidnam
Aristoteles quidnam Monica randaccio
Monica randaccio Integrated palliative care outcome scale
Integrated palliative care outcome scale Cosa significa nusaf
Cosa significa nusaf Array traduzione informatica
Array traduzione informatica Traduzione
Traduzione Bircom traduzione
Bircom traduzione L'acqua è poca e la papera non galleggia in inglese
L'acqua è poca e la papera non galleggia in inglese Sitografia in inglese
Sitografia in inglese Bootstrap traduzione informatica
Bootstrap traduzione informatica Panta rei os potamòs
Panta rei os potamòs Masthead traduzione
Masthead traduzione Point of care traduzione
Point of care traduzione Passer deliciae meae puellae traduzione
Passer deliciae meae puellae traduzione Placito capuano mappa concettuale
Placito capuano mappa concettuale Lagging indicators traduzione
Lagging indicators traduzione Testo plush
Testo plush Ancillae dominae
Ancillae dominae Concatamero
Concatamero Warehousing traduzione
Warehousing traduzione Doppio dativo in latino
Doppio dativo in latino