Lezione 6 Trascrizione Regolazione dellespressione genica 1 Trascrizione










- Slides: 39

Lezione 6 Trascrizione Regolazione dell’espressione genica 1

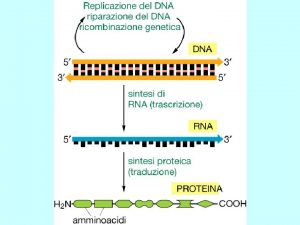
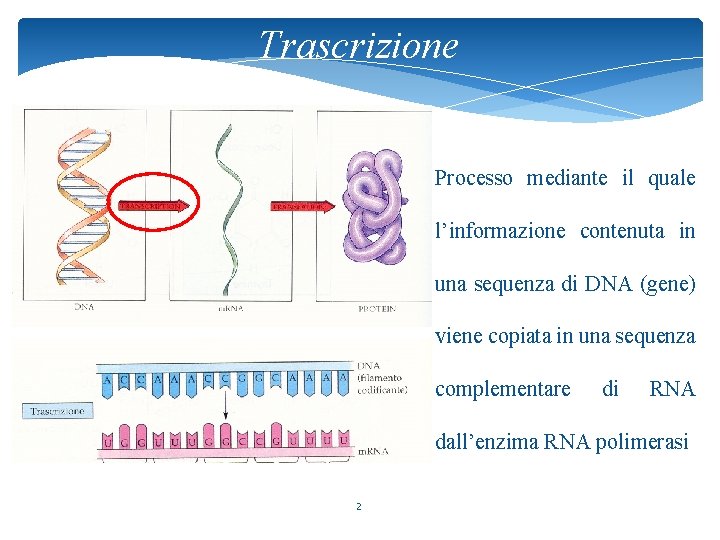
Trascrizione Processo mediante il quale l’informazione contenuta in una sequenza di DNA (gene) viene copiata in una sequenza complementare di RNA dall’enzima RNA polimerasi 2

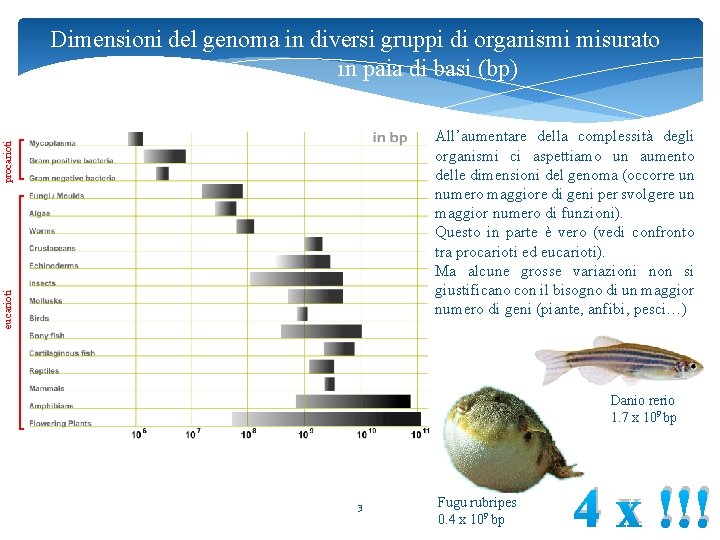
Dimensioni del genoma in diversi gruppi di organismi misurato in paia di basi (bp) eucarioti procarioti All’aumentare della complessità degli organismi ci aspettiamo un aumento delle dimensioni del genoma (occorre un numero maggiore di geni per svolgere un maggior numero di funzioni). Questo in parte è vero (vedi confronto tra procarioti ed eucarioti). Ma alcune grosse variazioni non si giustificano con il bisogno di un maggior numero di geni (piante, anfibi, pesci…) Danio rerio 1. 7 x 109 bp 3 Fugu rubripes 0. 4 x 109 bp 4 x !!!

O. T. : Sashimi di fugu Il pesce palla è provvisto di alcuni tessuti venefici che, durante la mondatura, possono contaminare la porzione edibile. Il veleno del pesce palla è una neurotossina (tetradotossina) potenzialmente mortale. E’ idrosolubile e termostabile (anche facendo bollire il pesce palla, se la tossina ha contaminato le carni, queste rimangono potenzialmente mortali). Ha un potenziale addirittura 1. 200 volte superiore al cianuro di potassio ed agisce paralizzando in muscoli respiratori ed il cuore fino al decesso. In Giappone, gli operatori a cui è concesso lavorare la carne del pesce palla sono tutti provvisti di una licenza specifica: inoltre, la vendita dell'animale NON lavorato è totalmente vietata ai consumatori finali. 4

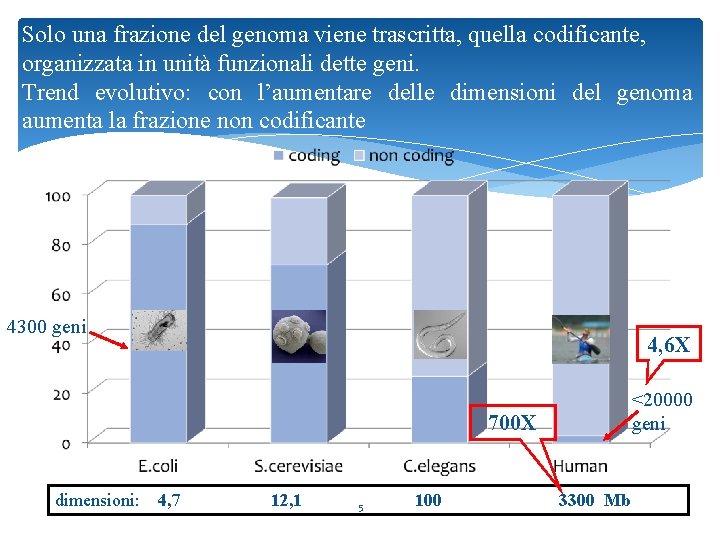
Solo una frazione del genoma viene trascritta, quella codificante, organizzata in unità funzionali dette geni. Trend evolutivo: con l’aumentare delle dimensioni del genoma aumenta la frazione non codificante 4300 geni 4, 6 X <20000 geni 700 X dimensioni: 4, 7 12, 1 5 100 3300 Mb

Tendenza evolutiva: all’aumentare delle dimensioni dei genomi sono aumentate le sequenze non codificanti Se l’aumento di contenuto di DNA non corrisponde ad un aumento di geni, allora a cosa serve/corrisponde tutto questo DNA in più? * Sequenze intergeniche * Introni * Sequenze di regolazione dell’espressione genica * Sequenze retrovirali integrate * Sequenze trascritte ma non tradotte (t. RNA, r. RNA, mi. RNA) * Pseudogeni * Altro? 6

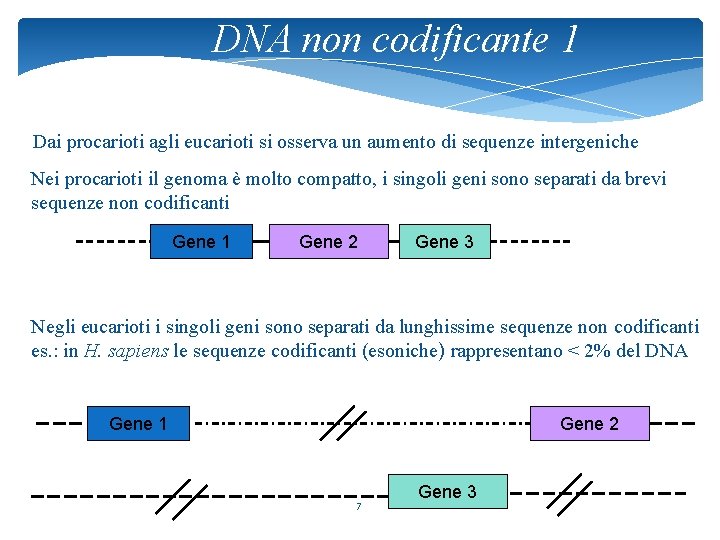
DNA non codificante 1 Dai procarioti agli eucarioti si osserva un aumento di sequenze intergeniche Nei procarioti il genoma è molto compatto, i singoli geni sono separati da brevi sequenze non codificanti Gene 1 Gene 2 Gene 3 Negli eucarioti i singoli geni sono separati da lunghissime sequenze non codificanti es. : in H. sapiens le sequenze codificanti (esoniche) rappresentano < 2% del DNA Gene 1 Gene 2 7 Gene 3

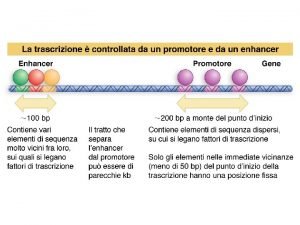
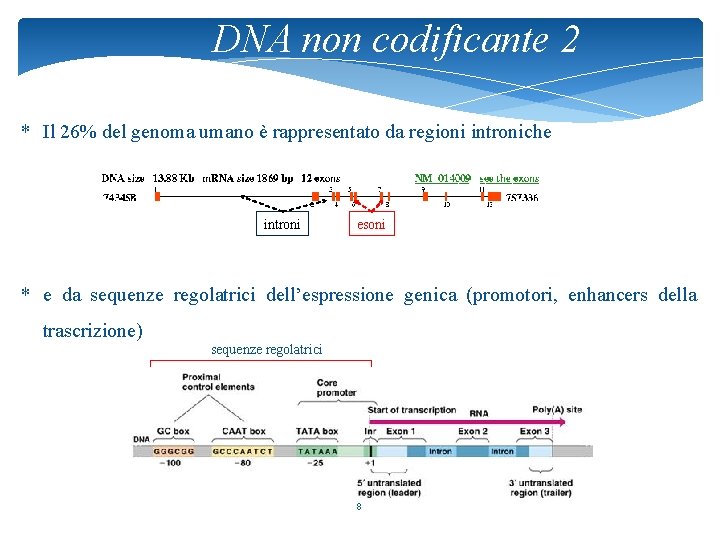
DNA non codificante 2 * Il 26% del genoma umano è rappresentato da regioni introniche introni esoni * e da sequenze regolatrici dell’espressione genica (promotori, enhancers della trascrizione) sequenze regolatrici 8

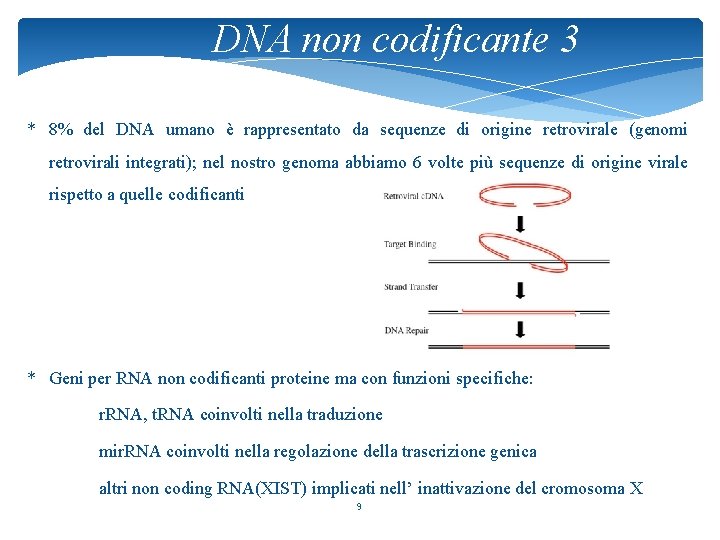
DNA non codificante 3 * 8% del DNA umano è rappresentato da sequenze di origine retrovirale (genomi retrovirali integrati); nel nostro genoma abbiamo 6 volte più sequenze di origine virale rispetto a quelle codificanti * Geni per RNA non codificanti proteine ma con funzioni specifiche: r. RNA, t. RNA coinvolti nella traduzione mir. RNA coinvolti nella regolazione della trascrizione genica altri non coding RNA(XIST) implicati nell’ inattivazione del cromosoma X 9

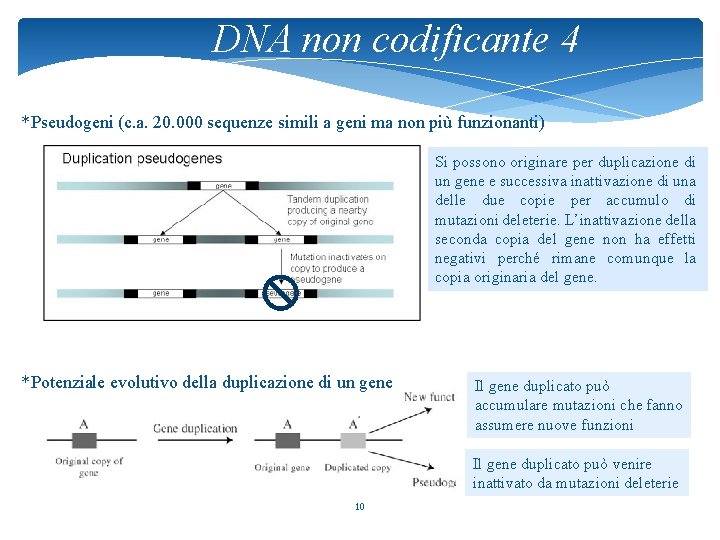
DNA non codificante 4 *Pseudogeni (c. a. 20. 000 sequenze simili a geni ma non più funzionanti) Si possono originare per duplicazione di un gene e successiva inattivazione di una delle due copie per accumulo di mutazioni deleterie. L’inattivazione della seconda copia del gene non ha effetti negativi perché rimane comunque la copia originaria del gene. *Potenziale evolutivo della duplicazione di un gene Il gene duplicato può accumulare mutazioni che fanno assumere nuove funzioni Il gene duplicato può venire inattivato da mutazioni deleterie 10

DNA non codificante 5 Altro? Esistono sequenze non codificanti altamente conservate (sono praticamente identiche) tra organismi diversi Se sono conservate devono avere una qualche funzione (altrimenti l’accumulo di mutazioni le avrebbe fatte divergere nel corso dell’evoluzione) Non ne conosciamo ancora la funzione 11

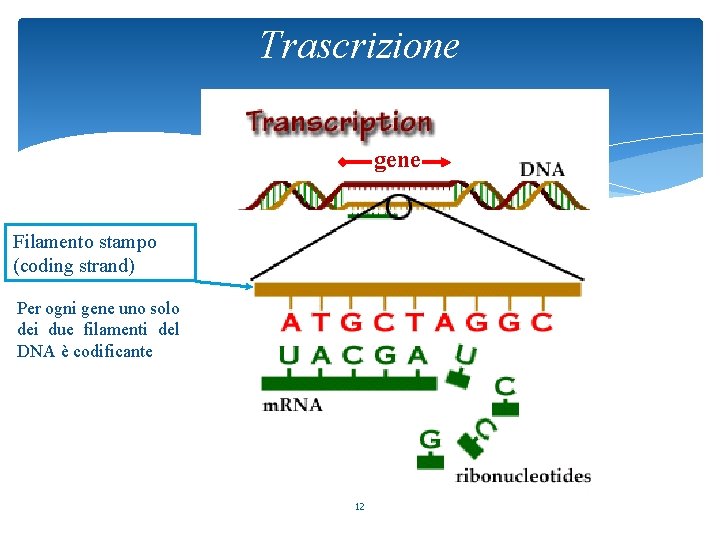
Trascrizione gene Filamento stampo (coding strand) Per ogni gene uno solo dei due filamenti del DNA è codificante 12

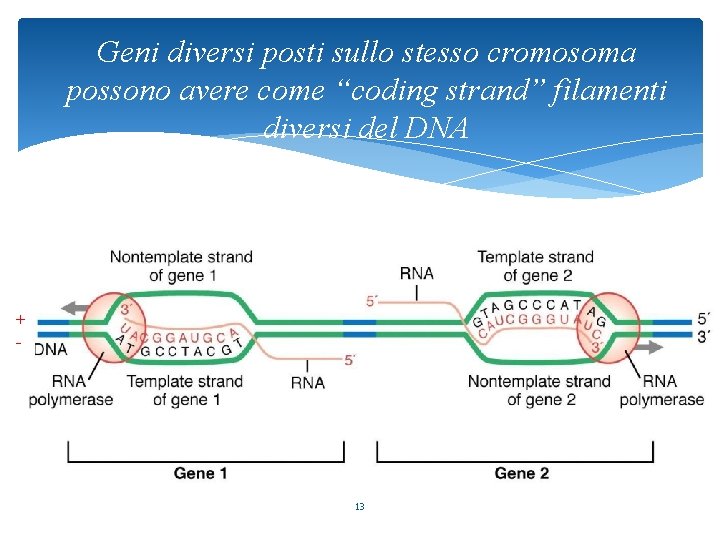
Geni diversi posti sullo stesso cromosoma possono avere come “coding strand” filamenti diversi del DNA + - 13

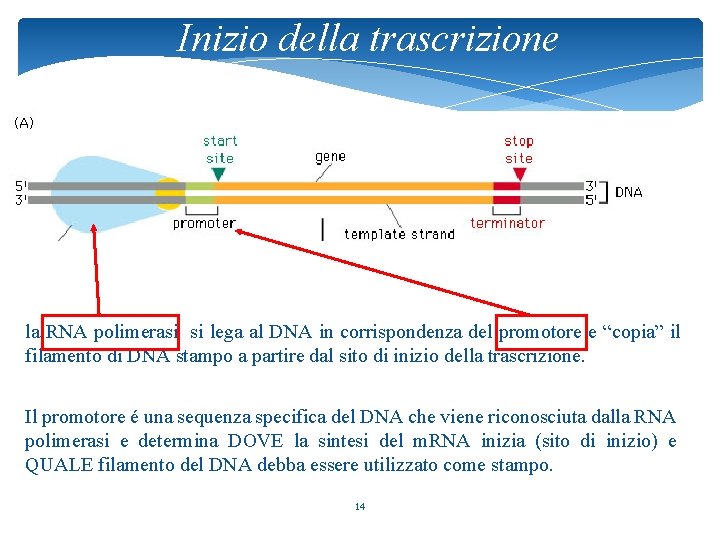
Inizio della trascrizione la RNA polimerasi si lega al DNA in corrispondenza del promotore e “copia” il filamento di DNA stampo a partire dal sito di inizio della trascrizione. Il promotore é una sequenza specifica del DNA che viene riconosciuta dalla RNA polimerasi e determina DOVE la sintesi del m. RNA inizia (sito di inizio) e QUALE filamento del DNA debba essere utilizzato come stampo. 14

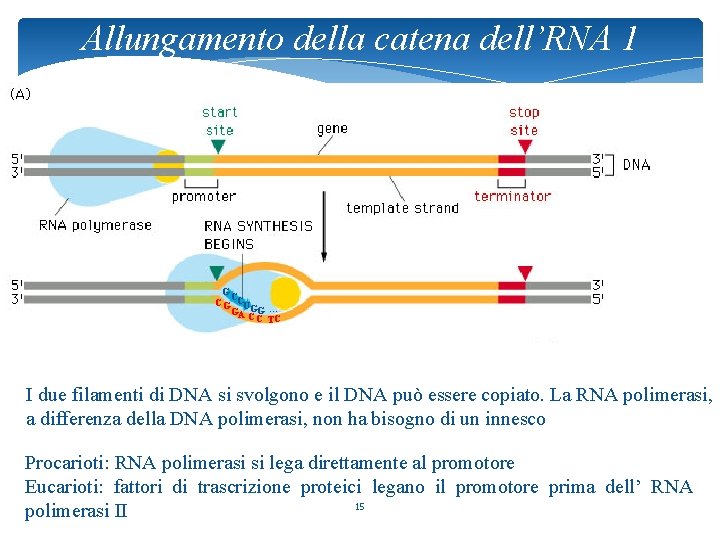
Allungamento della catena dell’RNA 1 G C C G CU GA GG … CC TC I due filamenti di DNA si svolgono e il DNA può essere copiato. La RNA polimerasi, a differenza della DNA polimerasi, non ha bisogno di un innesco Procarioti: RNA polimerasi si lega direttamente al promotore Eucarioti: fattori di trascrizione proteici legano il promotore prima dell’ RNA 15 polimerasi II

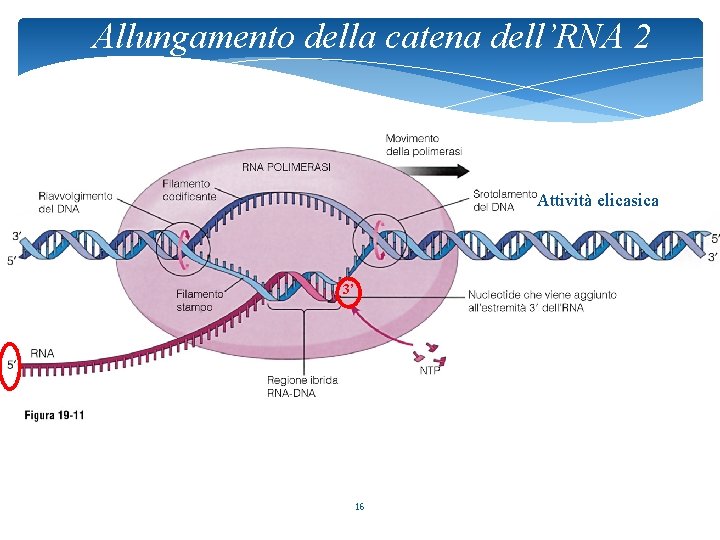
Allungamento della catena dell’RNA 2 Attività elicasica 3’ 16

Differenze tra DNA e RNA polimerasi * La RNA polimerasi ha attività elicasica (è in grado di denaturare la doppia elica del DNA), mentre la DNA polimerasi ha bisogno dell’intervento di un altro enzima con tale attività (elicasi) * La RNA polimerasi, a differenza della DNA polimerasi, non ha bisogno di un innesco * A differenza delle DNA polimerasi, le RNA polimerasi non hanno attività di “proof reading” (correzione di bozze) 17

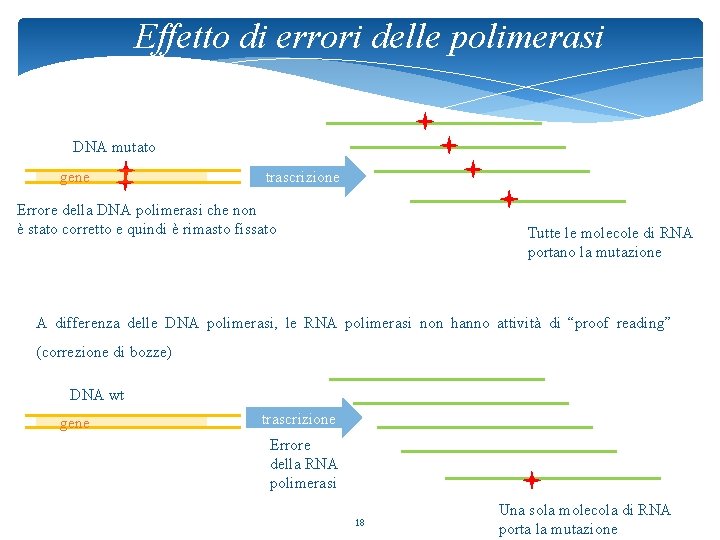
Effetto di errori delle polimerasi DNA mutato gene trascrizione Errore della DNA polimerasi che non è stato corretto e quindi è rimasto fissato Tutte le molecole di RNA portano la mutazione A differenza delle DNA polimerasi, le RNA polimerasi non hanno attività di “proof reading” (correzione di bozze) DNA wt gene trascrizione Errore della RNA polimerasi 18 Una sola molecola di RNA porta la mutazione

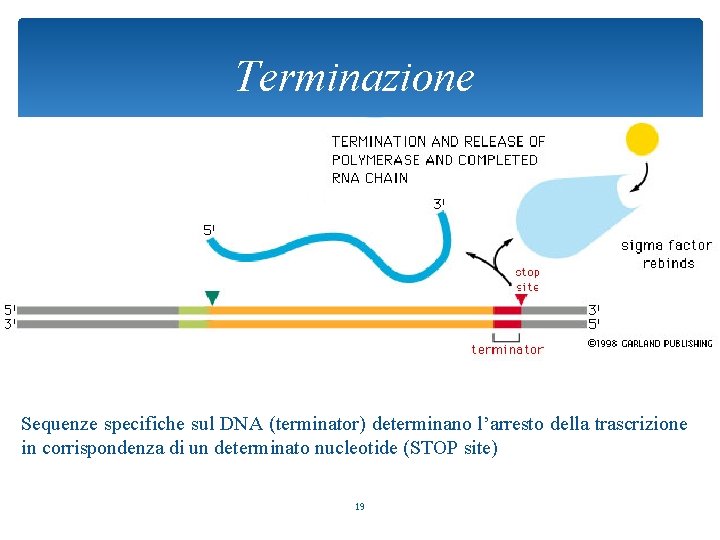
Terminazione Sequenze specifiche sul DNA (terminator) determinano l’arresto della trascrizione in corrispondenza di un determinato nucleotide (STOP site) 19

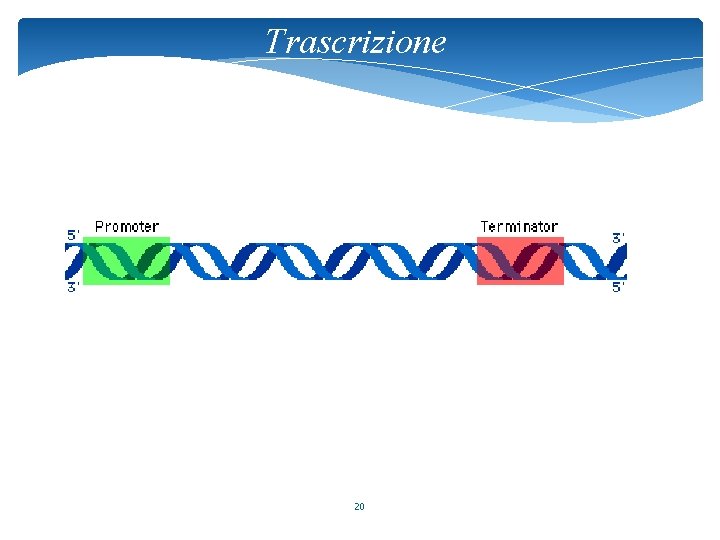
Trascrizione 20

Alfa amanitina inibisce le RNA pol eucariotiche Polipeptide biciclico RNA polimerasi L'avvelenamento con le amanitine è caratterizzato da un lungo periodo di latenza (dalle 6 alle 48 ore, in media 6 -15 ore) durante il quale il paziente non accusa alcun sintomo. Le cellule, rese incapaci di sintetizzare nuove proteine, interrompono bruscamente le proprie attività. Le cellule del tratto intestinale e del fegato sono le più drasticamente colpite non appena vengono in contatto con la sostanza e, nel caso di avvelenamento grave, l'unica possibilità di salvezza è 21 il trapianto di fegato.

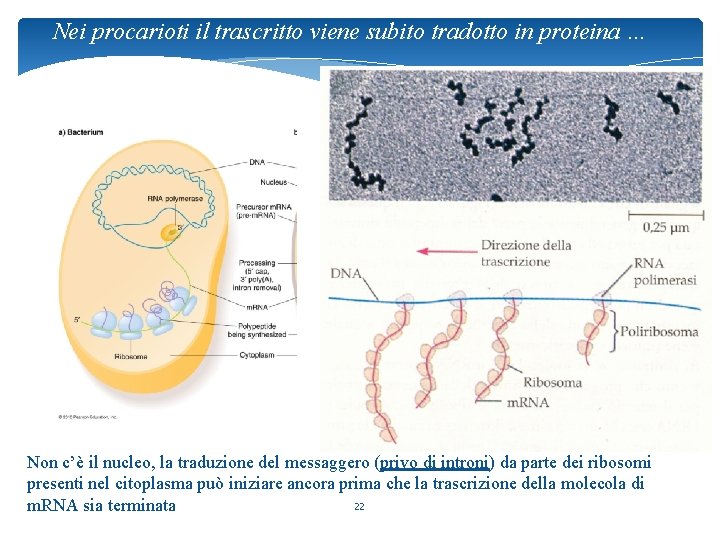
Nei procarioti il trascritto viene subito tradotto in proteina … Non c’è il nucleo, la traduzione del messaggero (privo di introni) da parte dei ribosomi presenti nel citoplasma può iniziare ancora prima che la trascrizione della molecola di 22 m. RNA sia terminata

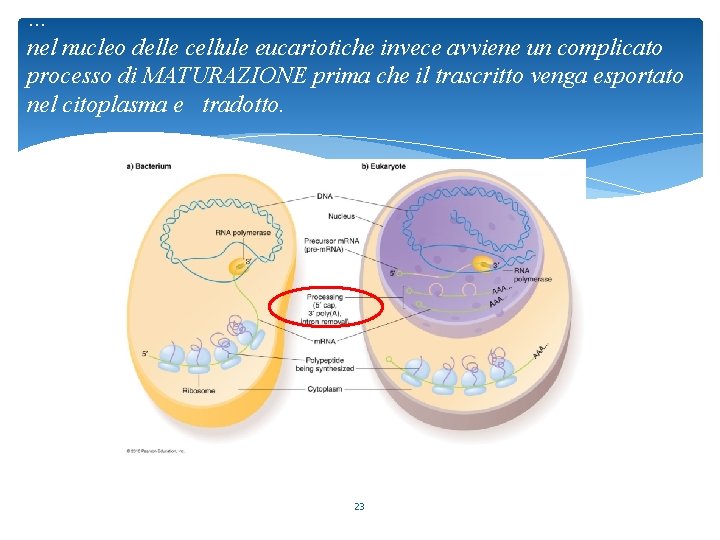
… nel nucleo delle cellule eucariotiche invece avviene un complicato processo di MATURAZIONE prima che il trascritto venga esportato nel citoplasma e tradotto. 23

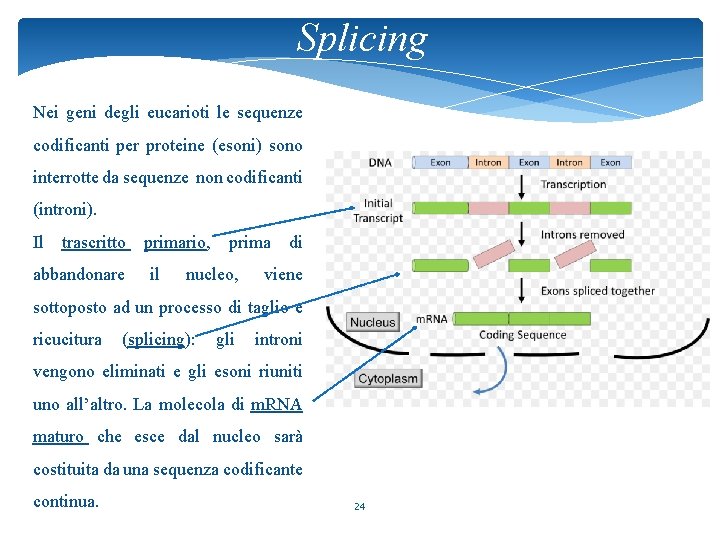
Splicing Nei geni degli eucarioti le sequenze codificanti per proteine (esoni) sono interrotte da sequenze non codificanti (introni). Il trascritto primario, prima di abbandonare il nucleo, viene sottoposto ad un processo di taglio e ricucitura (splicing): gli introni vengono eliminati e gli esoni riuniti uno all’altro. La molecola di m. RNA maturo che esce dal nucleo sarà costituita da una sequenza codificante continua. 24

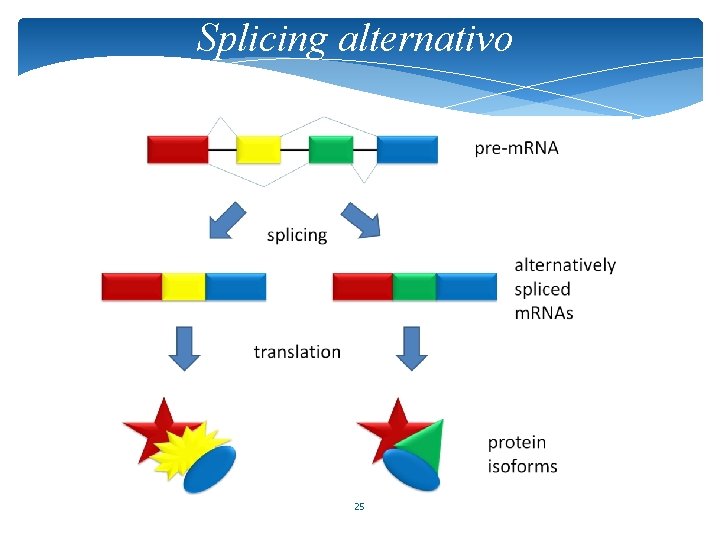
Splicing alternativo 25

Splicing alternativo * Il fenomeno dello SPLICING alternativo negli eucarioti aumenta il potenziale espressivo del genoma (a partire da un gene posso sintetizzare diverse proteine) * E’ la regola, non l’eccezione * Mediante forme di splicing alternativo da uno stesso gene possono derivare trascritti diversi che, una volta tradotti, danno luogo a proteine simili ma diverse 26

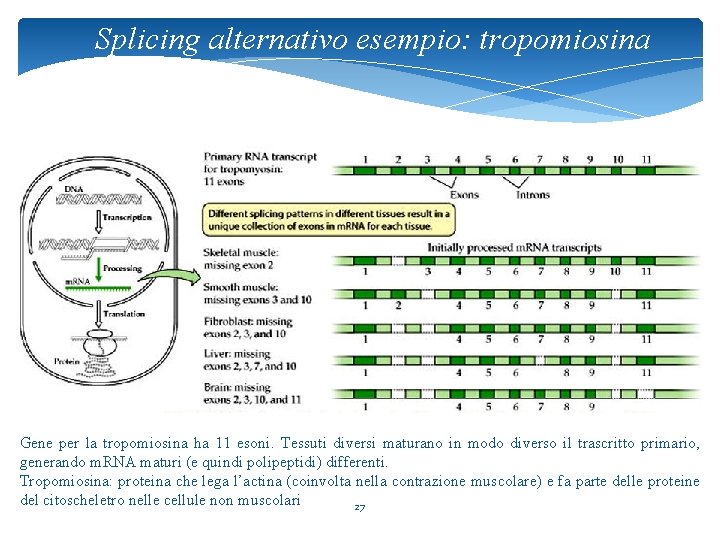
Splicing alternativo esempio: tropomiosina Gene per la tropomiosina ha 11 esoni. Tessuti diversi maturano in modo diverso il trascritto primario, generando m. RNA maturi (e quindi polipeptidi) differenti. Tropomiosina: proteina che lega l’actina (coinvolta nella contrazione muscolare) e fa parte delle proteine del citoscheletro nelle cellule non muscolari 27

Potenziale espressivo in H. sapiens * N° geni H. sapiens : 20. 000 * In H. sapiens si stima che la percentuale di geni che subiscono splicing alternativo sia >90% (Hallegger M et al. , 2010) 18. 000 geni subiscono AS * Il numero medio di varianti di splicing prodotto da ciascuno di questi geni è 4 (Kim H et al. , 2004) * La stima del numero di proteine prodotte da 20. 000 geni è di c. a. 72. 000 28

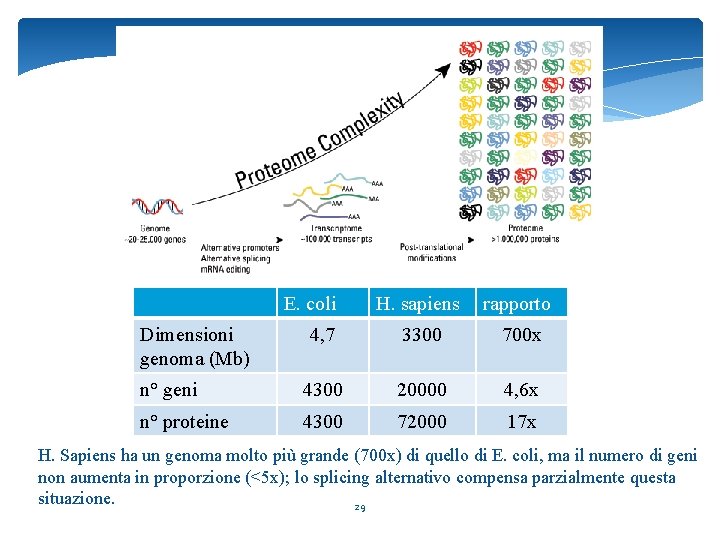
E. coli H. sapiens rapporto 4, 7 3300 700 x n° geni 4300 20000 4, 6 x n° proteine 4300 72000 17 x Dimensioni genoma (Mb) H. Sapiens ha un genoma molto più grande (700 x) di quello di E. coli, ma il numero di geni non aumenta in proporzione (<5 x); lo splicing alternativo compensa parzialmente questa situazione. 29

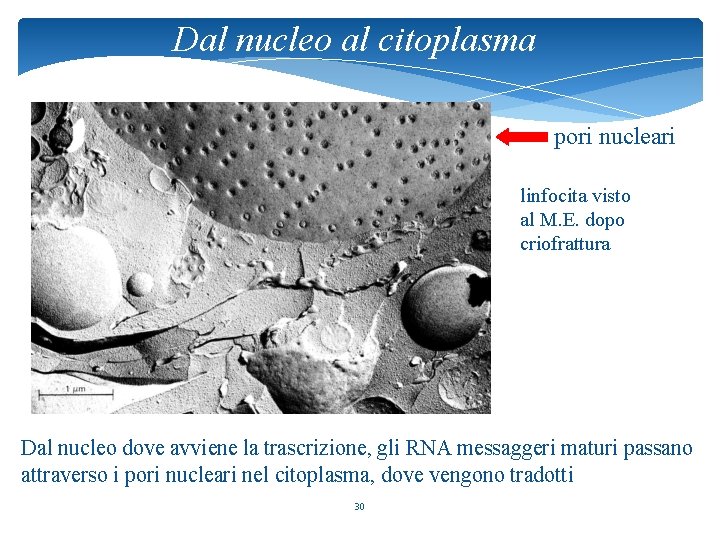
Dal nucleo al citoplasma pori nucleari linfocita visto al M. E. dopo criofrattura Dal nucleo dove avviene la trascrizione, gli RNA messaggeri maturi passano attraverso i pori nucleari nel citoplasma, dove vengono tradotti 30

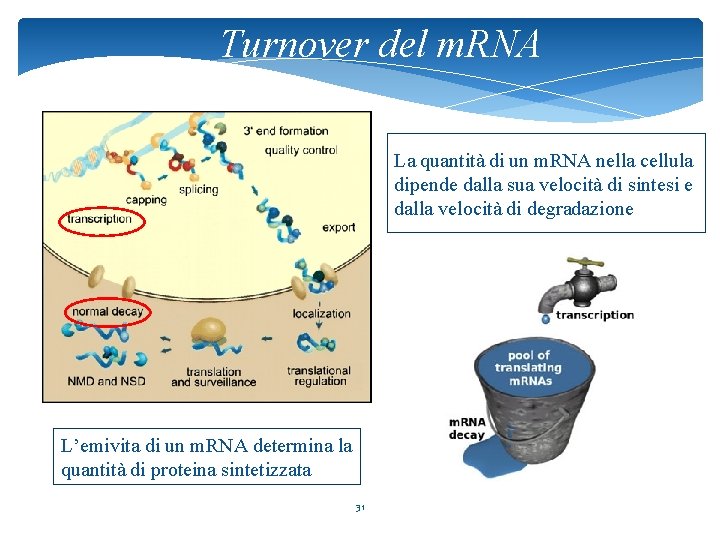
Turnover del m. RNA La quantità di un m. RNA nella cellula dipende dalla sua velocità di sintesi e dalla velocità di degradazione L’emivita di un m. RNA determina la quantità di proteina sintetizzata 31

m. RNA eucariotico m. RNA maturo Sequenza tradotta Nel trascritto maturo mancano le sequenze introniche (splicing), viene modificata l’estremità 5’ (cap) e viene aggiunta una coda di poli. A al 3’ UTR=Un. Translated Region 32

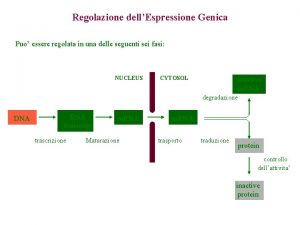
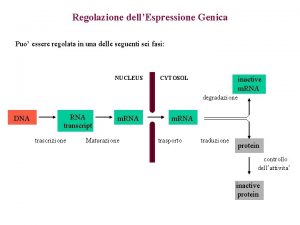
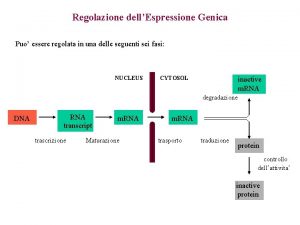
Regolazione dell’espressione genica * Solo alcuni geni vengono costituzionalmente trascritti per ogni cellula (geni housekeeping: enzimi del metabolismo, RNA polimerasi, ribosomi, istoni…) * Per tutti gli altri esistono molti meccanismi che permettono di regolare se, quando (meccanismi di regolazione temporali) e quanto un gene deve essere trascritto * Ciò è particolarmente evidente e importante negli organismi eucarioti multicellulari dove esiste una regolazione tessuto specifica (cellule diverse producono proteine diverse) Anche nei procarioti tuttavia vi sono meccanismi di regolazione dell’espressione genica OPERONI 33

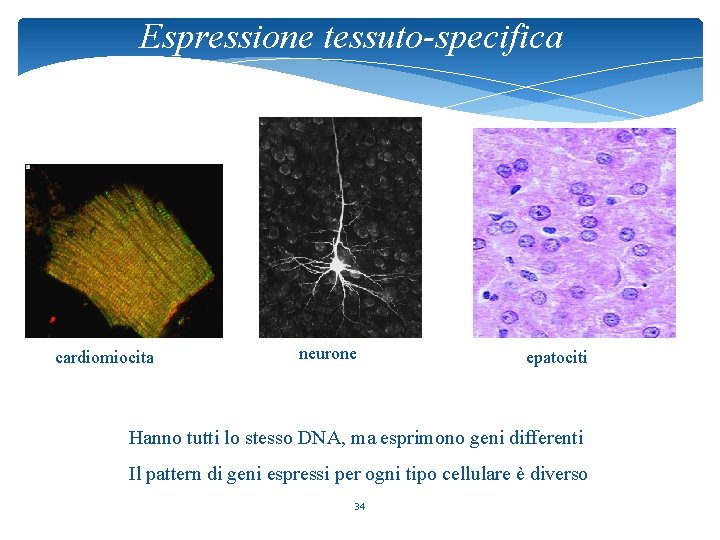
Espressione tessuto-specifica cardiomiocita neurone epatociti Hanno tutti lo stesso DNA, ma esprimono geni differenti Il pattern di geni espressi per ogni tipo cellulare è diverso 34

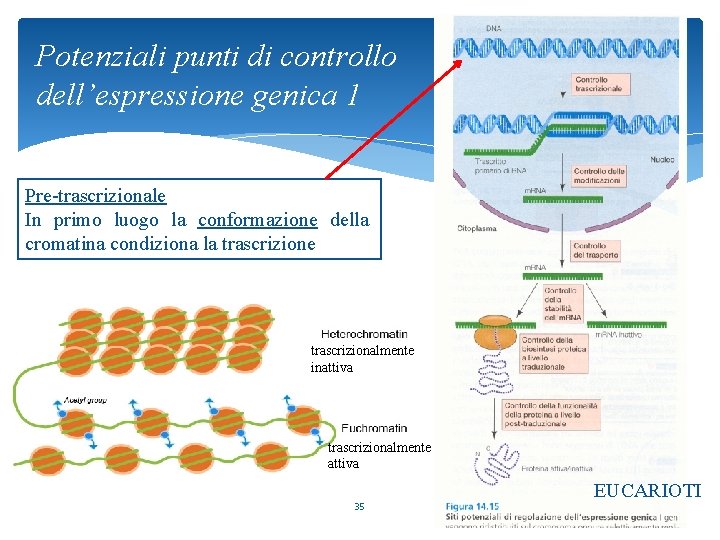
Potenziali punti di controllo dell’espressione genica 1 Pre-trascrizionale In primo luogo la conformazione della cromatina condiziona la trascrizione trascrizionalmente inattiva trascrizionalmente attiva 35 EUCARIOTI

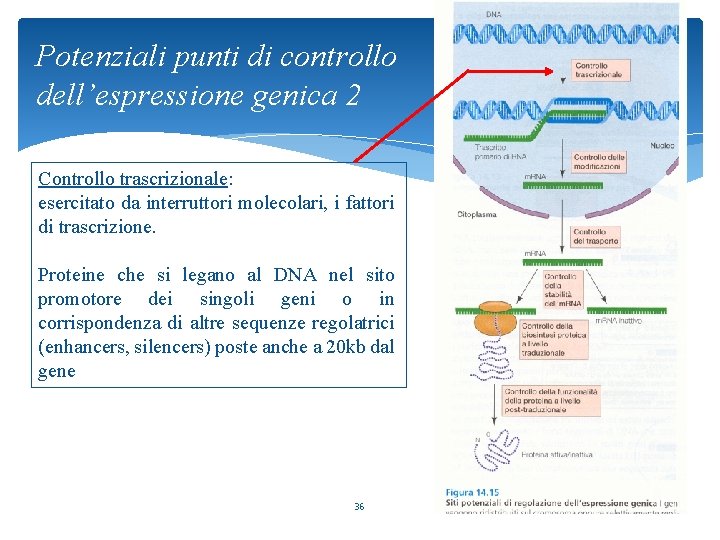
Potenziali punti di controllo dell’espressione genica 2 Controllo trascrizionale: esercitato da interruttori molecolari, i fattori di trascrizione. Proteine che si legano al DNA nel sito promotore dei singoli geni o in corrispondenza di altre sequenze regolatrici (enhancers, silencers) poste anche a 20 kb dal gene 36

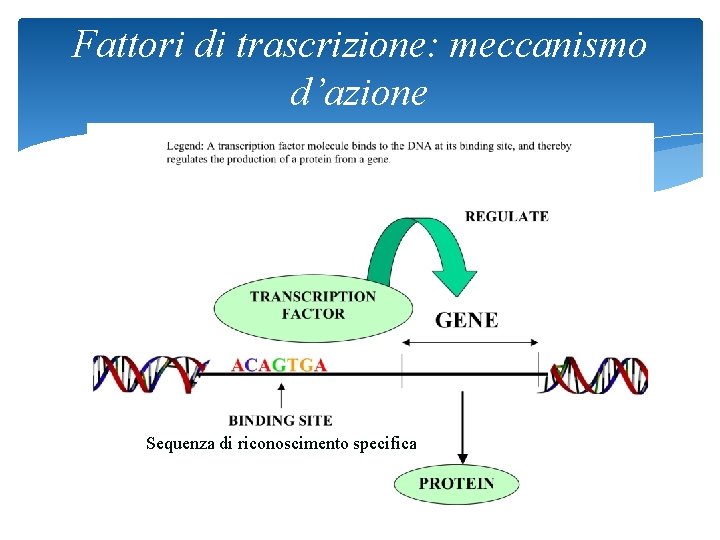
Fattori di trascrizione: meccanismo d’azione Sequenza di riconoscimento specifica 37

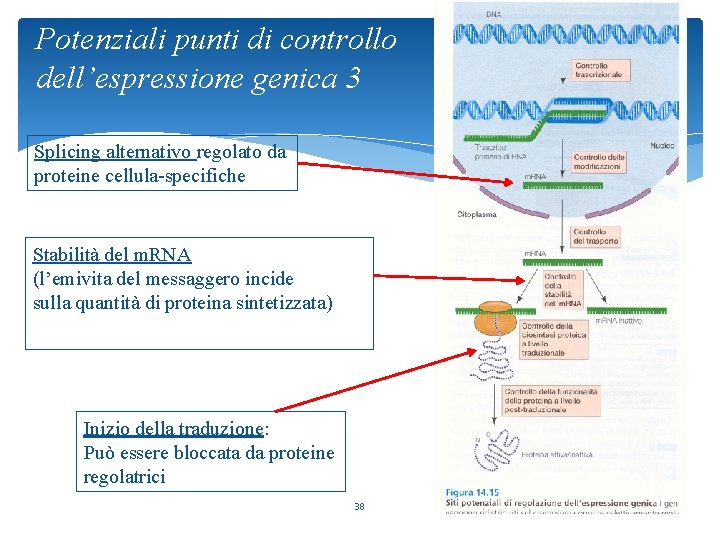
Potenziali punti di controllo dell’espressione genica 3 Splicing alternativo regolato da proteine cellula-specifiche Stabilità del m. RNA (l’emivita del messaggero incide sulla quantità di proteina sintetizzata) Inizio della traduzione: Può essere bloccata da proteine regolatrici 38

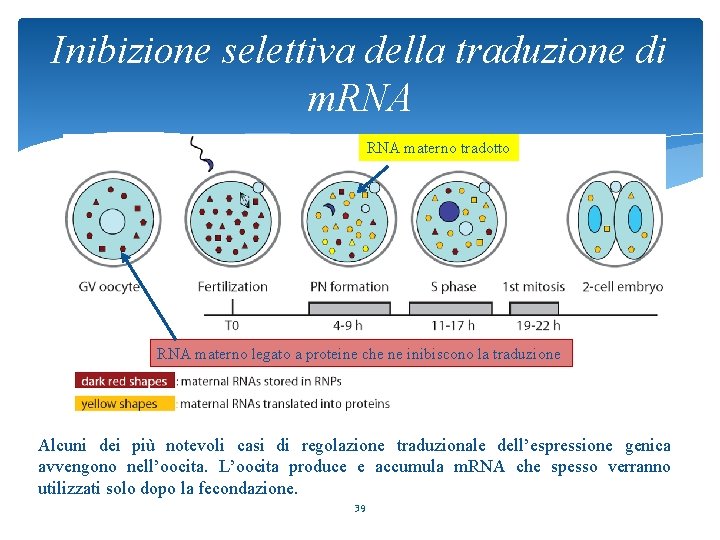
Inibizione selettiva della traduzione di m. RNA materno tradotto RNA materno legato a proteine che ne inibiscono la traduzione Alcuni dei più notevoli casi di regolazione traduzionale dell’espressione genica avvengono nell’oocita. L’oocita produce e accumula m. RNA che spesso verranno utilizzati solo dopo la fecondazione. 39
 Lac permeasi
Lac permeasi Espressione genica zanichelli
Espressione genica zanichelli Regolazione espressione genica eucarioti
Regolazione espressione genica eucarioti Fasi del ciclo cardiaco
Fasi del ciclo cardiaco Teoria bifattoriale della regolazione delle emozioni
Teoria bifattoriale della regolazione delle emozioni La regolazione degli affetti e la riparazione del sé
La regolazione degli affetti e la riparazione del sé Tesabase
Tesabase Differenze trascrizione eucarioti e procarioti
Differenze trascrizione eucarioti e procarioti Trascrizione fonetica convertitore
Trascrizione fonetica convertitore Fattori di trascrizione
Fattori di trascrizione Persuàdere o persuadère
Persuàdere o persuadère Rna polimerasi
Rna polimerasi Gatto trascrizione fonetica
Gatto trascrizione fonetica Dna mutazioni
Dna mutazioni Espressione genica
Espressione genica Expresion genica
Expresion genica Dna
Dna Duplicazione genica
Duplicazione genica Compensacion de dosis genica
Compensacion de dosis genica Concetto filogenetico di specie
Concetto filogenetico di specie Duplicazione genica
Duplicazione genica Cromosomas sexuales
Cromosomas sexuales Pleiotropia
Pleiotropia Que son los genes
Que son los genes Lezione 7
Lezione 7 Fasi di una lezione simulata
Fasi di una lezione simulata L esperienza delle cose moderne e la lezione delle antique
L esperienza delle cose moderne e la lezione delle antique Jigsaw esempio scuola primaria
Jigsaw esempio scuola primaria Esempio di lezione clil diritto
Esempio di lezione clil diritto Struttura della lezione
Struttura della lezione Italian plural articles
Italian plural articles Lezione delle antique cose
Lezione delle antique cose La lezione della farfalla libro
La lezione della farfalla libro