Transcrio e processamento do RNA em eucariotos Transcrio






















- Slides: 38

Transcrição e processamento do RNA em eucariotos

Transcrição em eucariotos

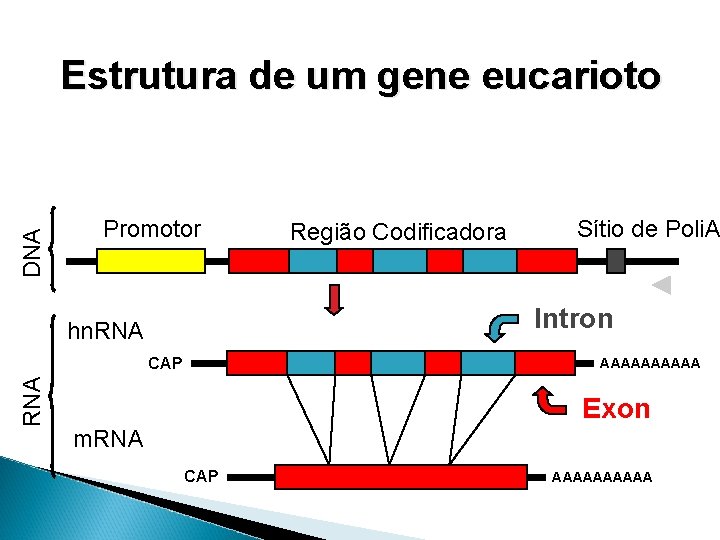
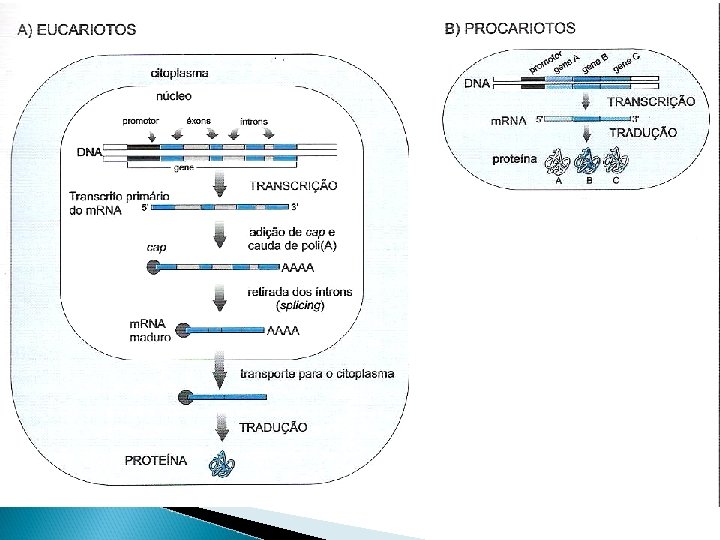
DNA Estrutura de um gene eucarioto Promotor Sítio de Poli. A Intron hn. RNA CAP RNA Região Codificadora AAAAA Exon m. RNA CAP AAAAA


INICIAÇÃO ALONGAMENTO TERMINAÇÃO 5 RNAs Polimerases: 3 nucleares (RNAPI, RNAPIII) RNAP mitocondrial e cloroplástica

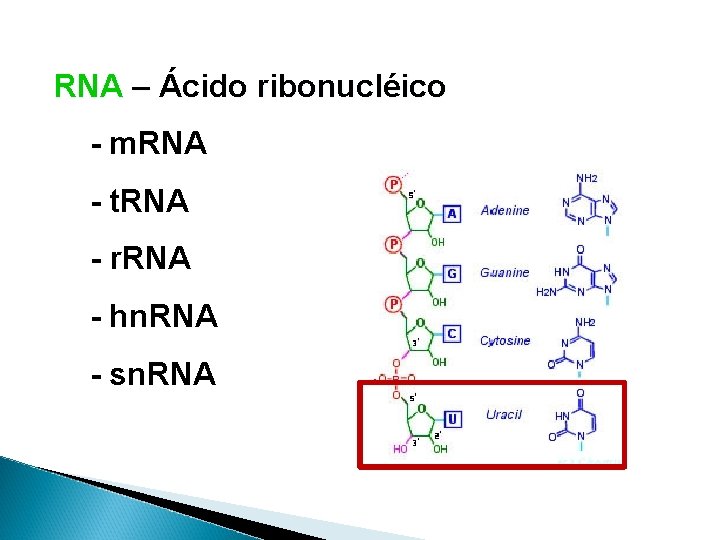
RNA – Ácido ribonucléico - m. RNA - t. RNA - r. RNA - hn. RNA - sn. RNA

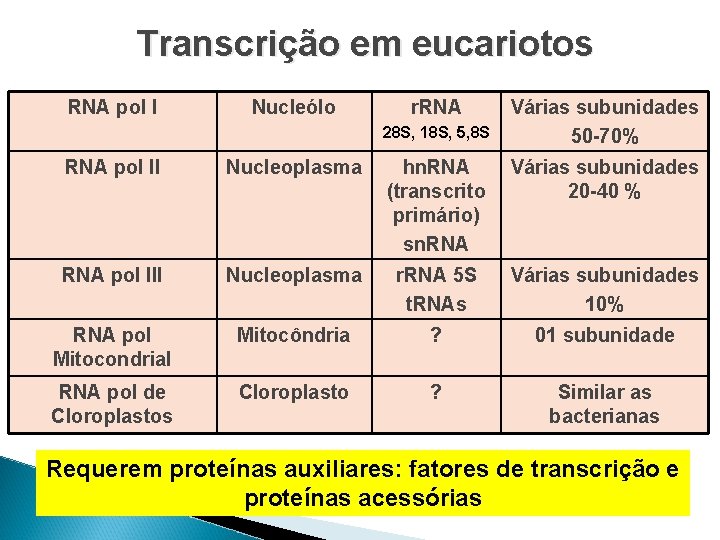
Transcrição em eucariotos RNA pol I Nucleólo r. RNA 28 S, 18 S, 5, 8 S Várias subunidades 50 -70% RNA pol II Nucleoplasma hn. RNA (transcrito primário) sn. RNA Várias subunidades 20 -40 % RNA pol III Nucleoplasma r. RNA 5 S t. RNAs Várias subunidades 10% RNA pol Mitocondrial Mitocôndria ? 01 subunidade RNA pol de Cloroplastos Cloroplasto ? Similar as bacterianas Requerem proteínas auxiliares: fatores de transcrição e proteínas acessórias

RNA POLIMERASE I Síntese apenas um tipo de pré-r. RNA precursor dos r. RNAS que farão parte do ribossomo. Os r. RNAs têm sequências muito conservadas. As células apresentam muitas cópias dos genes para r. RNAs Promotores para RNAPI tem 2 domínios: domínio central e domínio controlador a montante (UCE) Fatores auxiliares: UBF 1 e SL 1


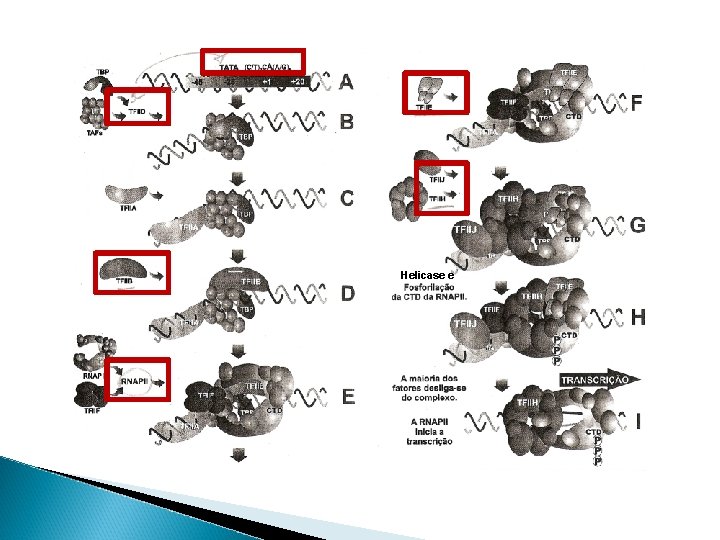
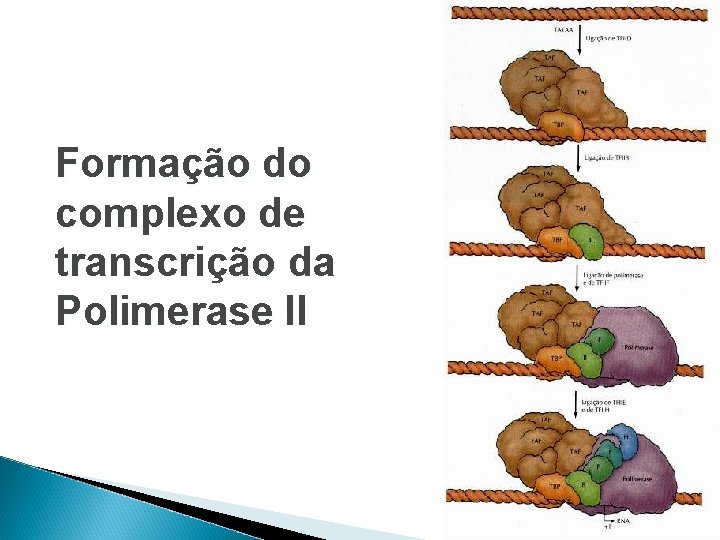
RNA POLIMERASE II Síntese de todos os hn. RNAs da célula que apresentam uma variedade de tamanho e mecanismos de regulação A subunidade maior apresenta o domínio CTD que deve ser fosforilado para ativar a RNAPII e desfosforilado para a desativação.

COMPONENTES BÁSICOS PARA A TRANSCRIÇÃO 1) Sítio de início da transcrição (Iniciador) – Sítio +1 2) Região seletora rica em A/T: TATA box – determina se a transcrição deve ser iniciada 3) Região reguladora: CAAT Box 4) Reforçador (enhancer): aumentam a expressão de genes

Helicase e

Formação do complexo de transcrição da Polimerase II

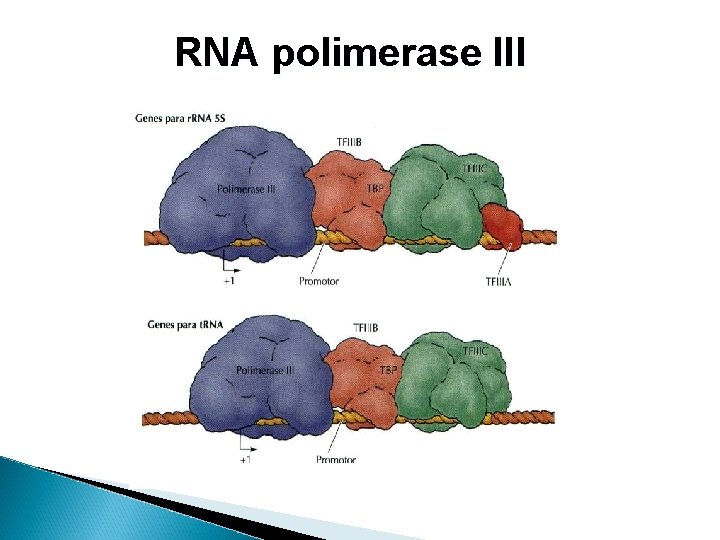
RNA POLIMERASE III Síntese de r. RNA, t. RNA e sn. RNA. Promotores de genes de t. RNAs: regiões A e B Promotores de genes de sn. RNAs: regiões A e C. Fatores de Transcrição: TFIIIA, TFIIIB e TFIIIC


RNA polimerase III

TERMINAÇ O RNAPI: reconhecimento de sequências específicas RNAPII: é necessário o sinal de poliadenilação, onde ocorre a adição da cauda de poli A RNAPIII: ocorre em terminadores de E. Coli regiões semelhantes à dos

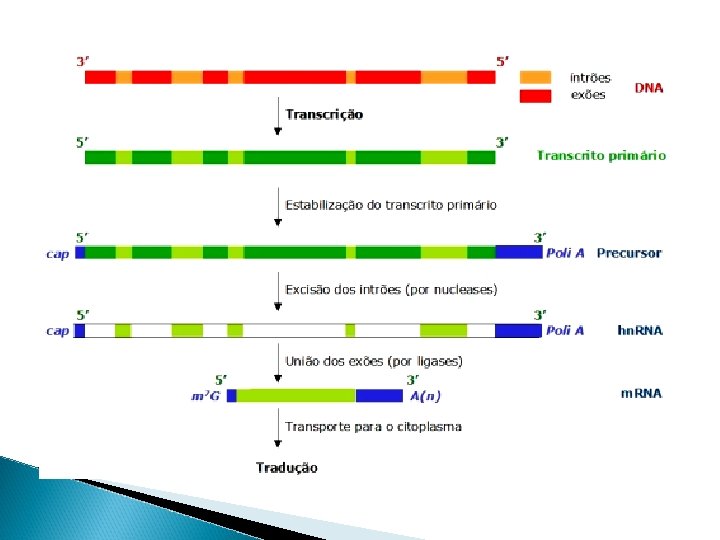
Processamento de RNA

RNAs recém sintetizados são chamados transcritos primários, pré-RNAs ou precursores de RNAs Modificações pós-transcricionais: processamento de RNA r. RNAs e t. RNAs: principalmente alterações de bases em procariotos e eucariotos m. RNAs de procariotos não sofrem alteração m. RNAs de eucariotos sofrem grandes modificações; adição da cap e cauda poli A


SPLICING Processo de excisão de íntorns

Splicing de íntrons de m. RNA nuclear heterogêneo (hn. RNA): transcrito primário dos m. RNAs Processamento dos hn. RNAs aumentam a estabilidade dos RNAs e convertem em um RNA funcional.

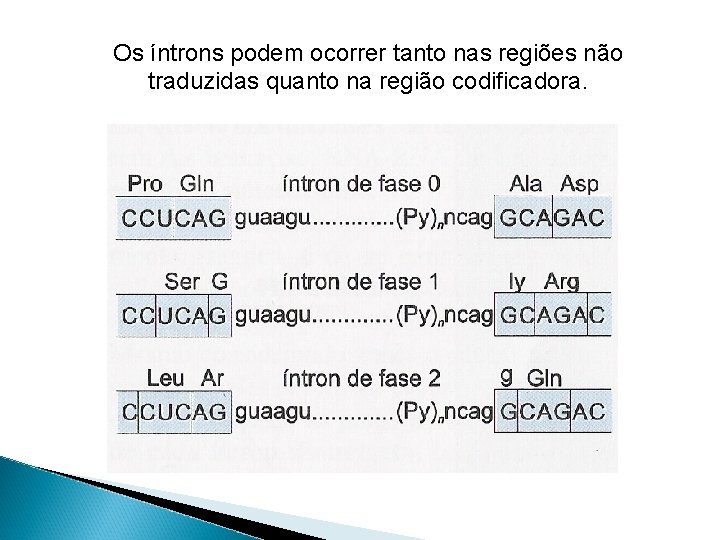
Características de íntrons de m. RNA - Adenina localizada 20 a 50 nt da extremidade 3’ do íntron - Região rica em pirimidinas na extremidade 3’ do íntron - Regra GU-AG (5´ 3´) (exceção alguns vertebrados AU-AC) Extremidades conservadas

Os íntrons podem ocorrer tanto nas regiões não traduzidas quanto na região codificadora.

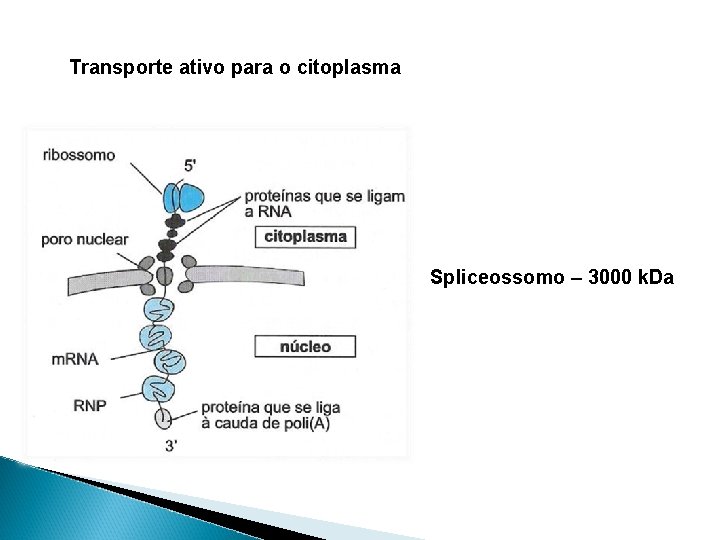
Splicing - Reações de transesterificação: transferência de um fosfato de um açúcar por outro; transferência da ligação fosfodiéster - Spliceossomo: formado por proteínas e sn. RNPs - Estrutura montada assim que o íntron é transcrito - Reação em cascata direcionada pelo pareamento de bases entre os íntrons o os sn. RNPs - Ao final, os éxons são unidos, os íntrons liberados em forma de laço e o complexo é desfeito. - Spliceossomo não passa pelos poros nucleares. Assim, os m. RNAs passam pelo processo de splicing ainda no núcleo.

Transporte ativo para o citoplasma Spliceossomo – 3000 k. Da

Trans-Splicing Formação de um RNA maduro unindo éxons que provêm de RNAs precursores diferentes. Comum em nematódeos, por exemplo.

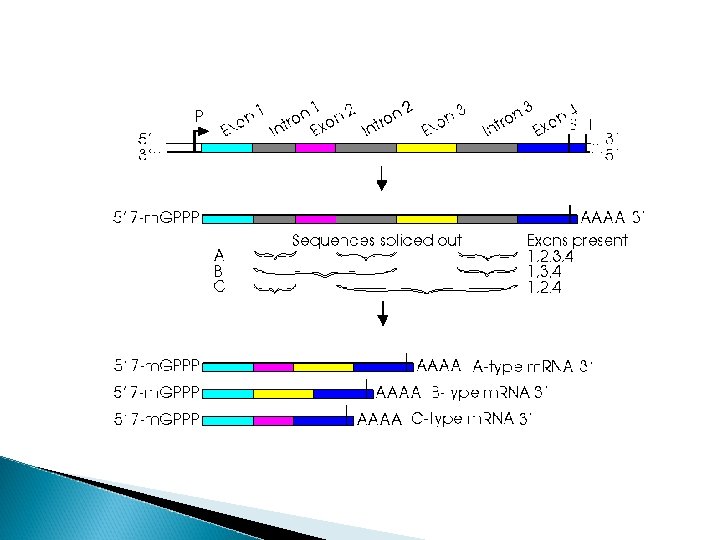
Splicing alternativo Diferentes possibilidades de splicing Um mesmo RNA precursor origina 2 ou mais RNAs maduros. O que é considerado como éxon em um processamento pode ser considerado íntron em outro. A partir de um mesmo gene há a formação de diferentes proteínas que atuam de modo diferente.


A presença de íntrons nos genes eucarióticos, aparentemente, representa um enorme desperdício celular Então, por que a evolução nos presenteou com estas seqüências? Mecanismo de proteção contra mutações Diversidade: genomas e proteomas (splicing alternativo) Regulação gênica. . .

Auto Splicing A excisão dos íntrons ocorre devido a atividade catalítica do próprio RNA precuror. As formas de auto splicing dependem das características dos íntrons.

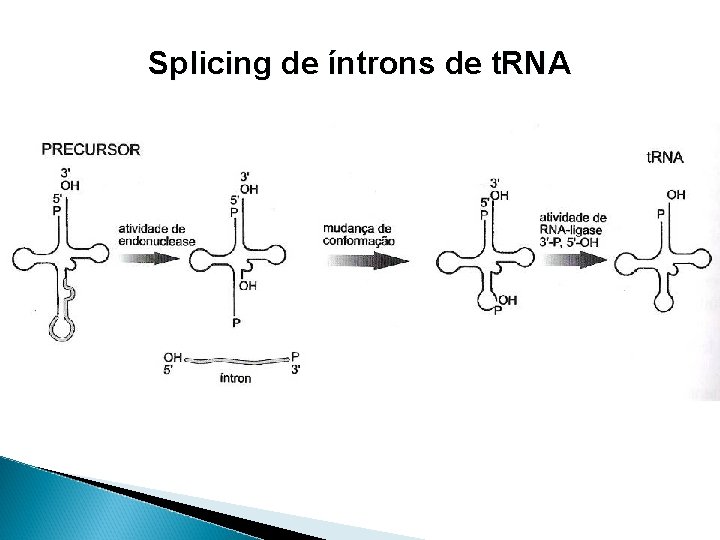
Splicing de íntrons de t. RNA

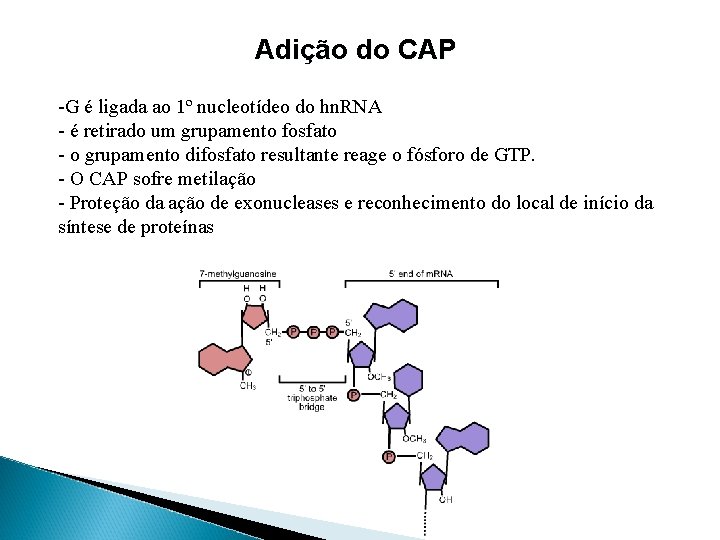
Adição do CAP -G é ligada ao 1º nucleotídeo do hn. RNA - é retirado um grupamento fosfato - o grupamento difosfato resultante reage o fósforo de GTP. - O CAP sofre metilação - Proteção da ação de exonucleases e reconhecimento do local de início da síntese de proteínas

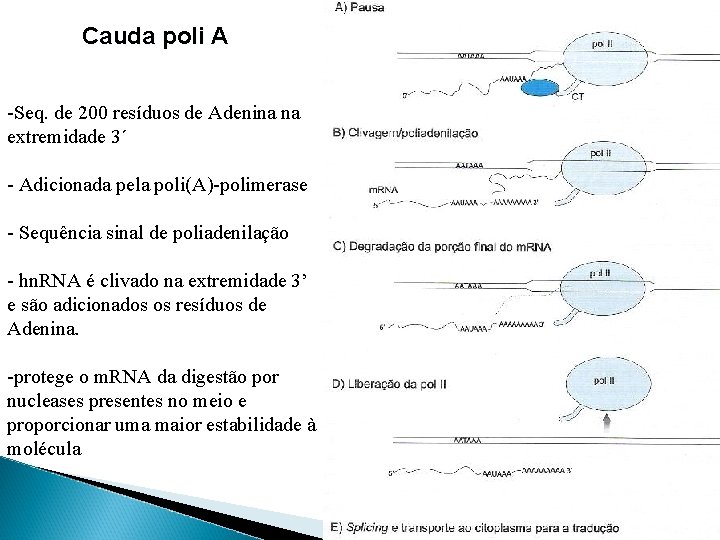
Cauda poli A -Seq. de 200 resíduos de Adenina na extremidade 3´ - Adicionada pela poli(A)-polimerase - Sequência sinal de poliadenilação - hn. RNA é clivado na extremidade 3’ e são adicionados os resíduos de Adenina. -protege o m. RNA da digestão por nucleases presentes no meio e proporcionar uma maior estabilidade à molécula

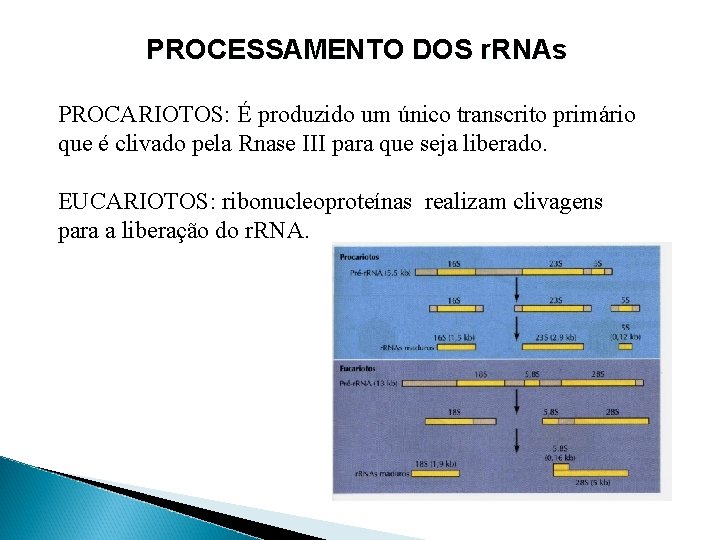
PROCESSAMENTO DOS r. RNAs PROCARIOTOS: É produzido um único transcrito primário que é clivado pela Rnase III para que seja liberado. EUCARIOTOS: ribonucleoproteínas realizam clivagens para a liberação do r. RNA.

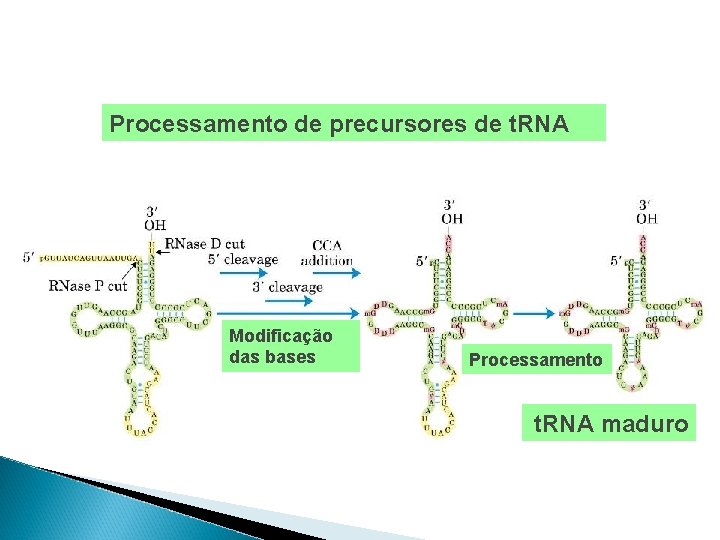
Processamento de precursores de t. RNA Modificação das bases Processamento t. RNA maduro

Estrutura de um m. RNA eucarioto processado
