Hyperstructures et modlisation de chimie artificielle dans le


















- Slides: 25

Hyper-structures et modélisation de chimie artificielle dans le langage MGS Clément BOIN & Nicolas THIBAULT Maîtrise Informatique – Université d’Evry Val-d’Essonne – 2001 -2002 T. E. R. encadré par Jean-Louis Giavitto & Olivier Michel

Plan • Introduction – La chimie artificielle – MGS • Modélisation d’un réseau autocatalytique • Modélisation d’un réseau biochimique • Conclusion

La chimie artificielle • Chimie virtuelle • Modèles discrets et combinatoires + • Avantages et Inconvénients

Définition d’une expérience • Un ensemble d’objets • Des règles de transformation • Des règles d’interaction dynamique

MGS (Modèle Général de Simulation) • Langage dédié • Langage déclaratif • Deux interprètes : Ocaml et C++

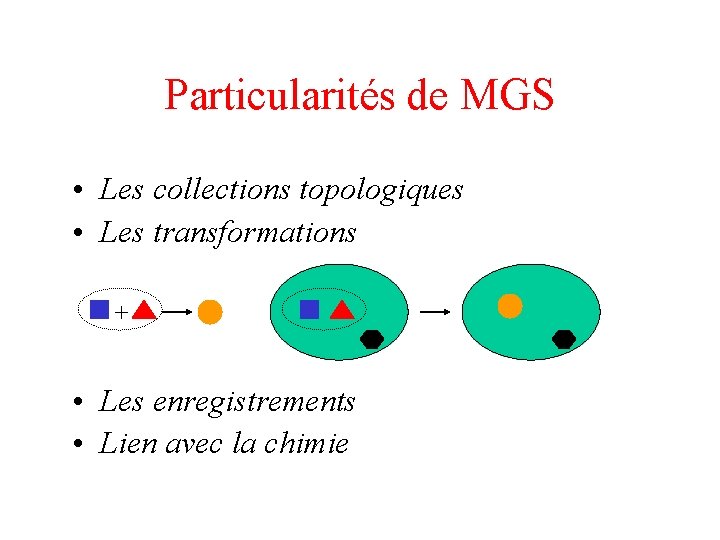
Particularités de MGS • Les collections topologiques • Les transformations + • Les enregistrements • Lien avec la chimie

Exemple MGS • fun objets(m) = if m==1 then (): set else m: : (objets (m-1)) fi; ; • trans premier = {x , y/(y%x==0) => x }; ; • premier['iter='fixpoint](objets(100)); ;

Modélisation d’un réseau autocatalytique • Exposé chimique du problème • Idée générale de modélisation • Stratégie d’implémentation en MGS – Représentation d’un polymère – Création des produits et réactions – Exécution • Exemple d’utilisation et interprétation des résultats

Exposé chimique du problème • M 1 + M 2 Kf Kr M 12 • Sélectionner les polymères dominants

Idée générale de modélisation • Approche individuelle des polymères • Utilisation d’entiers 1+2 14 12 • Création aléatoire des éléments

Représentation d’un polymère en MGS • Séquence 1 + 2 2 + 1 12 + 12 • Multi-ensemble 12 21 1212 1 + 2 2 + 1 12 + 12 12 12 1122 • Séquence avec déterminisme de la réaction 1 + 2 2 + 1 12 + 12 1212

Création aléatoire des produits et réactions • • • Monomères Polymères Séquence des polymères initiaux Séquence des polymères possibles Réactions

Exécution des réactions • Création des produits et réactions • Initialisation de la solution • Transformation MGS générique – Application de chaque réaction pour un pas d’évolution. – Itération du processus pour plusieurs pas d’évolution.

Code MGS trans Biochimie = { reaction_a = a, b, c /((a==nieme(i, reaction). poly 1)& (b==nieme(i, reaction). poly 2)&(c==nieme(i, reaction). cata)) => if condition(nieme(i, reaction). kf) then (nieme(i, reaction). poly_res): : c: : (): solution else a: : b: : c: : (): solution fi; reaction_b = a, c /((a==nieme(i, reaction). poly_res)& (c==nieme(i, reaction). cata)) => if condition(nieme(i, reaction). kr) then (nieme(i, reaction). poly 1): : (nieme(i, reaction). poly 2): : c: : (): solution else }; ; a: : c: : (): solution fi;

Exemple • Polymères initiaux : 42 , 42 • Polymères possibles : 42 , 4242 • Réaction : 42 + 42 Kf = 42 Kr = 67 Kf = 62 Kr = 78 4242

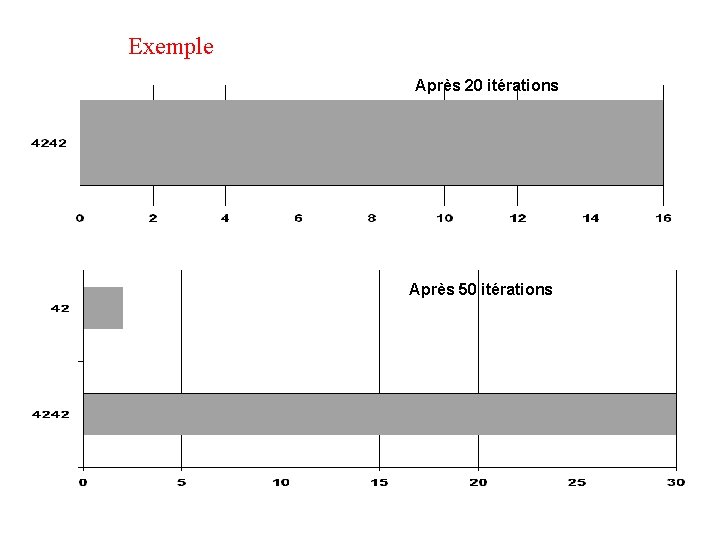
Exemple Après 20 itérations Après 50 itérations

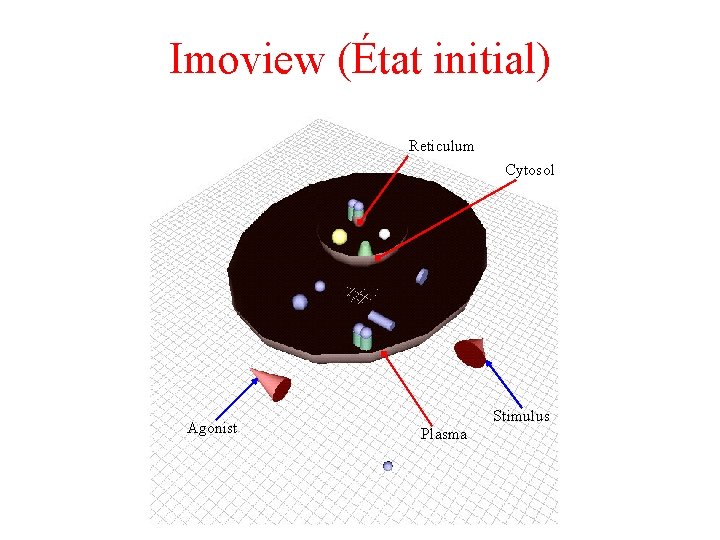
Modélisation d’un réseau biochimique • L’expérience de A. E. Bugrim • Modélisation en MGS • Visualisation avec Imoview

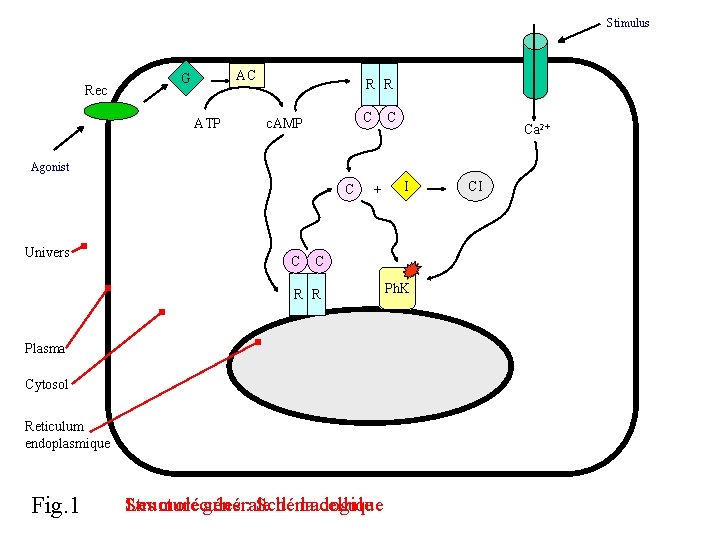
Stimulus Rec AC G ATP R R C c. AMP C Ca 2+ Agonist C Univers C + C R R Plasma Cytosol Reticulum endoplasmique Fig. 1 I Les molécules : Schéma logique Structure générale de la cellule Ph. K CI

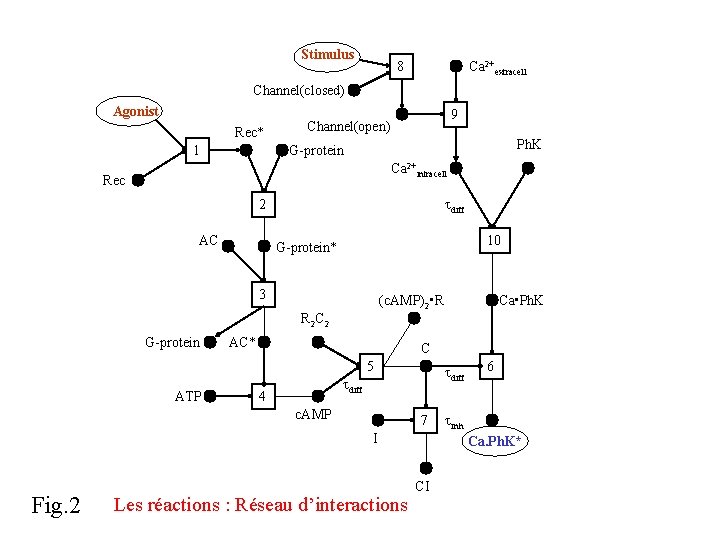
Stimulus 8 Ca 2+extracell Channel(closed) Agonist Rec* 1 9 Channel(open) Ph. K G-protein Ca 2+intracell Rec diff 2 AC 10 G-protein* 3 (c. AMP)2 • R Ca • Ph. K R 2 C 2 G-protein AC* C 5 ATP diff 4 c. AMP 7 I Fig. 2 Les réactions : Réseau d’interactions 6 inh Ca. Ph. K* CI

L’expérience de Bugrim en MGS • • • Les molécules - enregistrements Structure générale - collections Les réactions - transformations Application récursive Aspects dynamiques – Les temps de diffusion – Les réactions non-déterministes

Code MGS (1) // Description des strutures dans l'espace collection Volume = bag; ; collection Membrane = bag; ; collection Univ = Volume; ; collection Plasma = Membrane; ; collection Cytosol = Volume; ; collection Retic_Endo = Membrane; ; //== Initialisation RETICULUM : = {nom="r 2 c 2"}: : (): Retic_Endo ; ; CYTOSOL : = {nom="i"}: : {nom="phk"}: : {nom="atp"}: : RETICULUM: : (): Cytosol ; ; PLASMA : = {nom= "recepteur", actif=0}: : {nom="r 2 c 2"}: : {nom= "gprot", actif=0}: : {nom= "ac", actif=0}: : {nom= "channel", actif=0}: : CYTOSOL: : (): Plasma ; ; U : = {nom= "agonist", actif=1}: : {nom= "stimulus", actif=1}: : {nom= "ca++", diffuse=0}: : PLASMA: : (): Univ ; ;

Code MGS (2) // Ensemble des réactions trans Biochimie = { Incr = c: Cytosol => Incr. Cyt(c): : (): Cytosol; //Incrémenter les tps de diffusion Reaction 1 = a: Agonist 1, p: Plasma => (a+{actif=0}): : Active. Recepteur(p): : (): Univ; Reaction 2 = r: Recepteur 1, g: Gprot 0 => (g+{actif=1}): : (r+{actif=0}): : (): Plasma; Reaction 3 = g: Gprot 1, a: AC 0 => (g+{actif=0}): : (a+{actif=1}): : (): Plasma; Reaction 4 = a: AC 1, c: Cytosol =>(a+{actif=0}): : Trans. Atp(c): : (): Plasma; Reaction 5 = a: c. AMP 1, re: Retic_Endo/member({nom="r 2 c 2"}, re) => Alea. R 2 C 2(a, re); Reaction 5 b = r: R 2 C 2, c: Cytosol/member({nom="camp", diffuse=DIFFCAMP}, c) => delete({nom="camp", diffuse=DIFFCAMP}, ({nom="c", diffuse=0}: : {nom="(camp)2. r"}: : c)): : (): Plasma; Reaction 6 = c: Cenzime 1, x: Ca. Ph. K 0 => c: : (x+{actif=1}): : (): Cytosol; Reaction 7 = c: Cenzime, i: I => Aleatoire(TAUXINIB, c, i); Reaction 8 = s: Stimulus 1, p: Plasma => (s+{actif=0}): : Active. Channel(p): : (): Univ; Reaction 9 = c: Ca 2 p, p: Plasma/member({nom="channel", actif=1}, p) => Ajout. Plasma. Ca(Des. Channel(p)): : (): Univ; Reaction 10 = x: Ca 2 p 1, y: Ph. K => {nom="caphk", actif =0}: : (): Cytosol; }; ;

Imoview (État initial) Reticulum Cytosol Agonist Stimulus Plasma

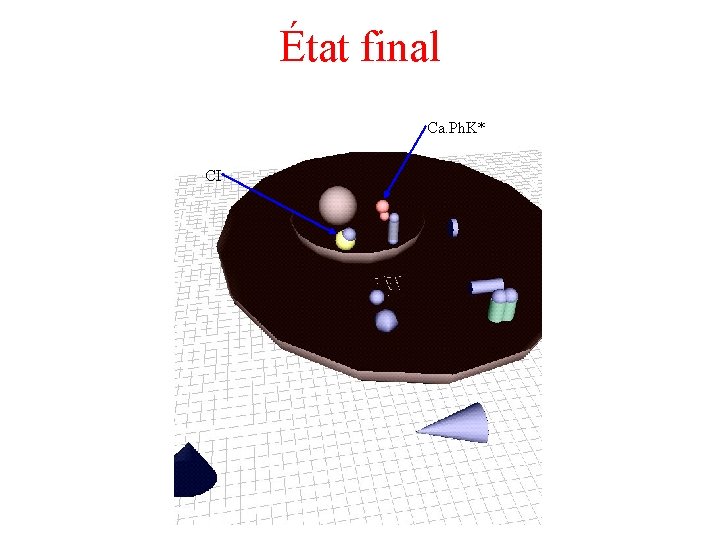
État final Ca. Ph. K* CI

Conclusion • MGS, langage abstrait • Limites – Complexité – Fonctions aléatoires – Code peu réutilisable