Comment les cellules lisent le gnome de lADN



































- Slides: 70

Comment les cellules lisent le génome : de l'ADN à la protéine

Introduction • L'information héréditaire est codée dans la séquence des nucléotides de l'ADN la complexité de la biologie n'est pas infinie • Alphabet à 4 lettres bactérie, drosophile, humain • Problème : comment – 500 gènes bactérie – 30 000 gènes humain – grosse difficulté dans le décodage d'un génome 2

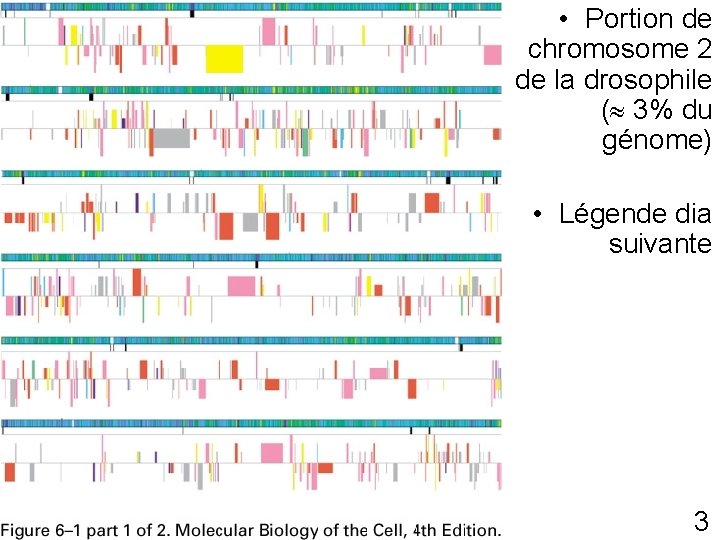
Fig 6 -1 p 300 • Portion de chromosome 2 de la drosophile ( 3% du génome) • Légende dia suivante 3

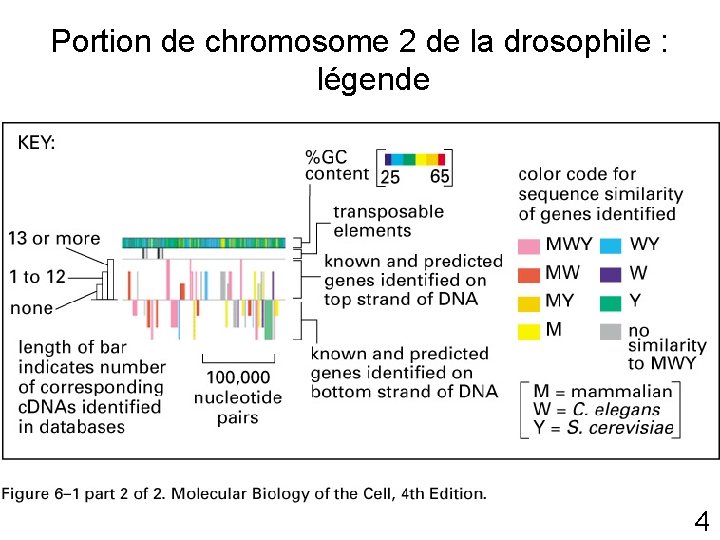
Portion de chromosome 2 de la drosophile : légende Fig 6 -1 p 300 4

Information codée par l'ADN • Majorité : séquence des acides aminés molécule forme + chimie • Également : – Quand un gène va s'exprimer – Où : dans quels types de cellules 5

(Dés)Organisation de l'information • Ni un dictionnaire ni un annuaire de téléphone • Incroyablement désordonné : – des petits bouts d'ADN codant dispersés dans des gros blocs d'ADN apparemment sans signification – certaines parties du génome contiennent beaucoup de gènes et d'autres pas du tout – des protéines travaillant ensemble dans la cellule ont leur gène sur des chromosomes différents – des gènes adjacents codent pour des protéines qui n'ont pas de rapport entre elles 6

ARN comme intermédiaire • ADN ARN = transcription • ARN protéine = traduction 7

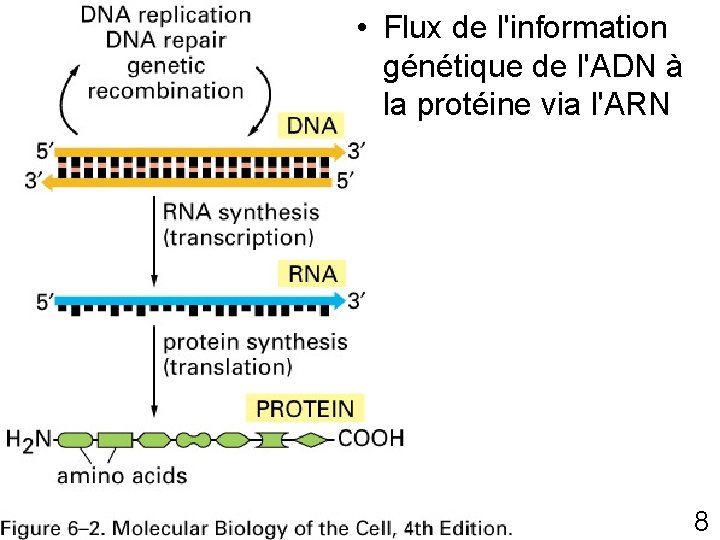
• Flux de l'information génétique de l'ADN à Fig 6 -2 p 301 la protéine via l'ARN 8

Dogme central de la biologie moléculaire " Toutes les cellules de la bactérie à l'homme expriment leur information génétique de cette façon " 9

Variations sur le dogme • Maturation des transcrits ARN (épissage, …) • Peut changer la signification (le message) de la molécule d'ARN • L'ARN peut être le produit final 10

Comment les cellules lisent le génome : de l'ADN à la protéine I. De l'ADN à l'ARN II. De l'ARN à la protéine III. Le monde de l'ARN et les origines de la vie 11

Comment les cellules lisent le génome : de l'ADN à la protéine I. De l'ADN à l'ARN II. De l'ARN à la protéine III. Le monde de l'ARN et les origines de la vie 12

I - De l'ADN à l'ARN 13

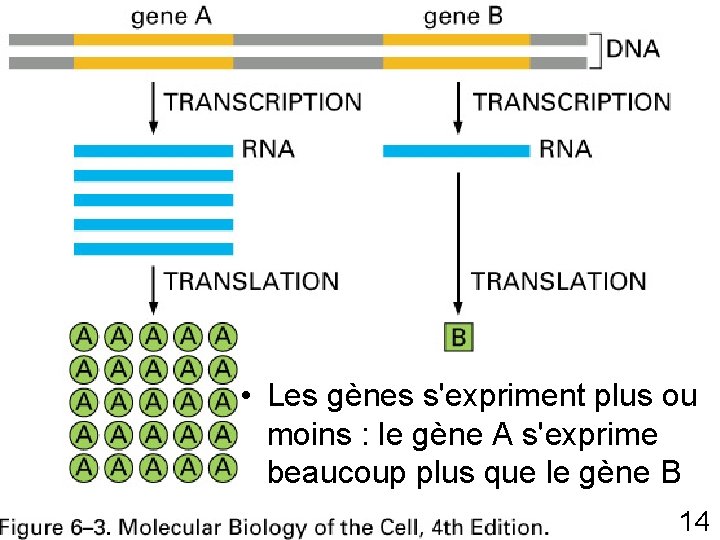
Fig 6 -3 p 302 • Les gènes s'expriment plus ou moins : le gène A s'exprime beaucoup plus que le gène B 14

Transcription • L'information est recopiée dans un langage proche de l'ADN • Les nucléotides sont des ribonucléotides • AUGC au lieu de ATGC dans l'ADN • ADN toujours en double hélice • ARN en simple brin grandes variétés de formes nombreuses fonctions (structure et catalytique) 15

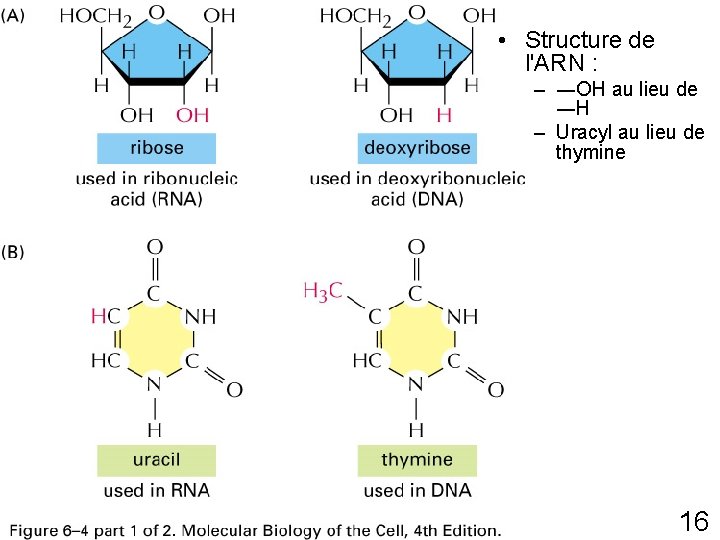
• Structure de l'ARN : – ―OH au lieu de ―H – Uracyl au lieu de thymine Fig 6 -4 p 303(1) 16

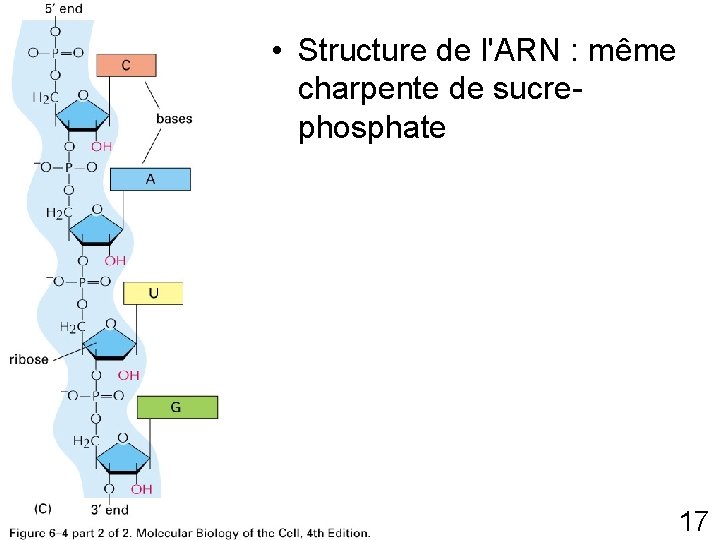
• Structure de l'ARN : même charpente de sucrephosphate Fig 6 -4 p 303(2) 17

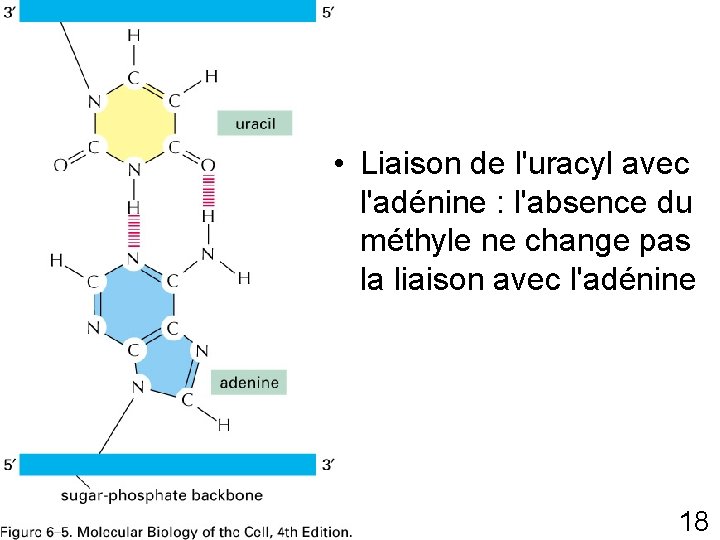
• Liaison de l'uracyl avec l'adénine : l'absence du méthyle ne change pas la liaison avec l'adénine Fig 6 -5 p 303 18

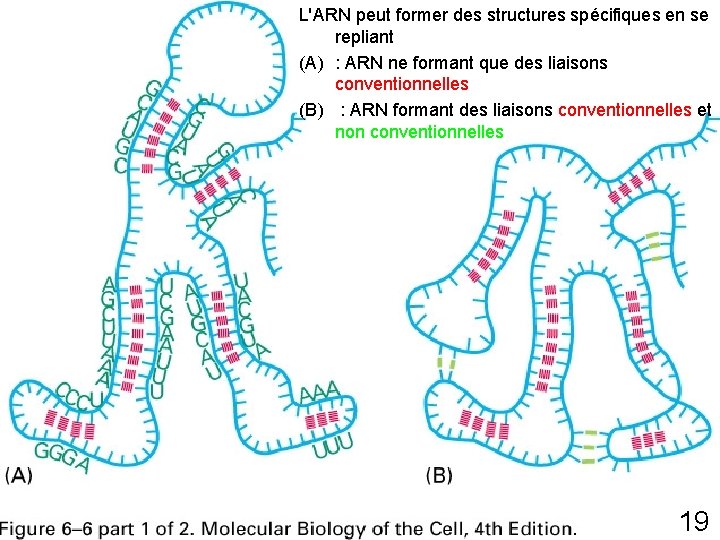
L'ARN peut former des structures spécifiques en se repliant (A) : ARN ne formant que des liaisons conventionnelles (B) : ARN formant des liaisons conventionnelles et non conventionnelles Fig 6 -6 p 304 19

Transcription: analogies avec la réplication • • • Ouverture des 2 brins Déroulement d'une petite portion de l'ADN Utilisation d'un des 2 brins comme matrice Appariement des bases Liaison covalente du nouveau nucléotide la séquence du transcrit est complémentaire du brin qui a servi de matrice 20

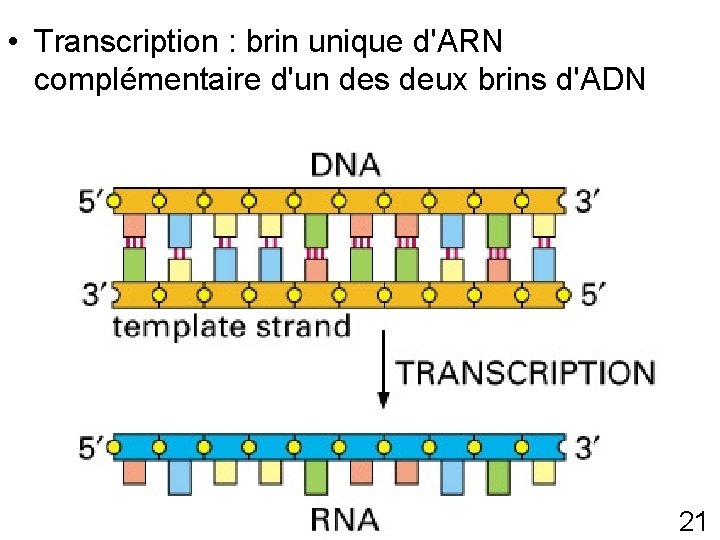
• Transcription : brin unique d'ARN complémentaire d'un des deux brins d'ADN Fig 6 -7 p 303 21

Transcription: différences avec la réplication • La molécule d'ARN ne reste pas liée au brin d'ADN • L'hélice d'ADN se reforme juste après le passage • Les molécules d'ARN sont beaucoup plus petites que celles d'ADN – ADN jusqu'à 250 millions de pb – ARN jusqu'à quelques milliers de nucléotides 22

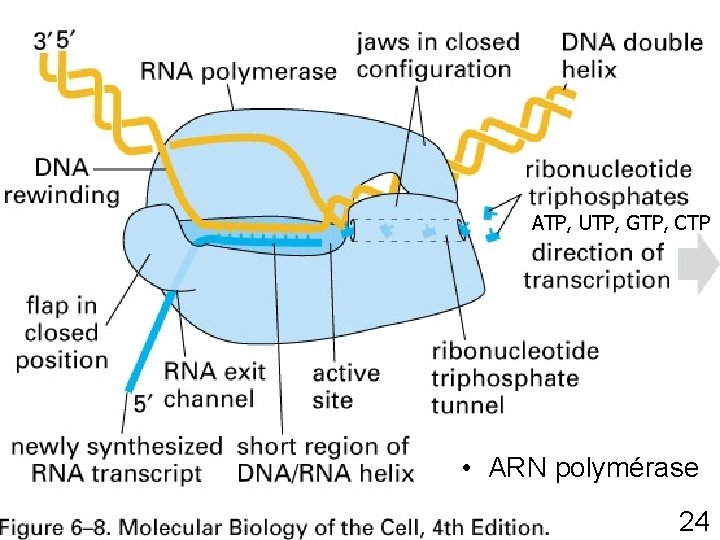
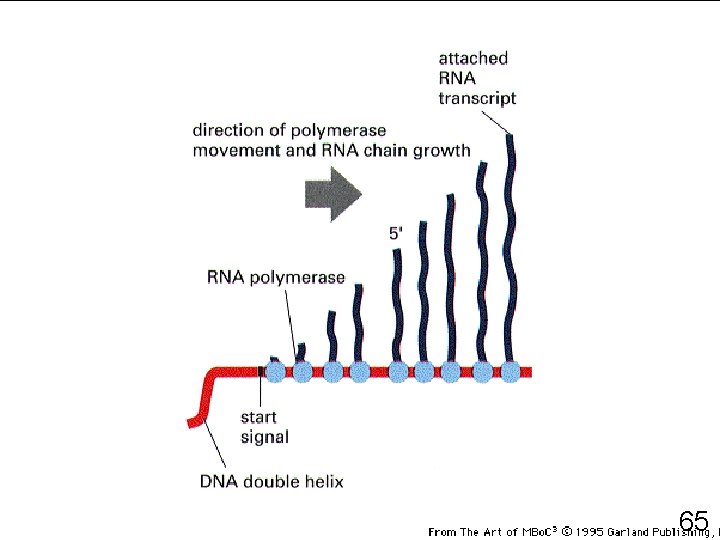
ARN polymérase • Forme la chaîne linéaire d'ARN • Allonge dans le sens 5' 3' 23

ATP, UTP, GTP, CTP Fig 6 -8 p 305 • ARN polymérase 24

ARN polymérase • Se déplace le long de l'ADN • Déroule la molécule à son site actif • Ajoute les nucléotides un par un sur le brin servant de matrice • Gouvernail à l'arrière pour éviter aux deux brins de l'ADN de se ré-enrouler 25

ARN polymérase • Les substrats sont ATP, CTP, UTP, GTP • Comme l'ARN est libéré très vite, on peut recommencer une nouvelle molécule avant la fin de la synthèse de la précédente • Vitesse 20 nucléotides par seconde (euk) • Plusieurs milliers de transcrits en une heure à partir d'un seul gène 26

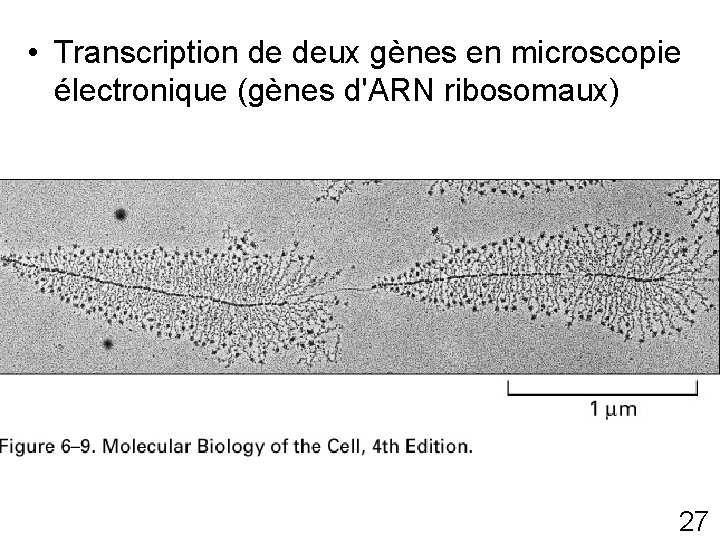
• Transcription de deux gènes en microscopie électronique (gènes d'ARN ribosomaux) Fig 6 -9 p 305 27

Différences entre ADN polymérase et ARN polymérase • ARN polymérase • ADN polymérase • • Ribonucléotides Pas besoin d’amorce Une erreur tous les 104 Erreurs aux conséquences de moyenne importance • Système de réparation frustre Désoxyribonucléotides Nécessité d’amorce Une erreur tous les 107 Erreurs aux conséquences de grande importance • Système de réparation très complexe 28

Les différents types d'ARN • ARN messager : séquence d'acides aminés de la protéine • Chez S. cerevisiae plus de 750 gènes (plus de 10% du génome) codent pour de l'ARN comme produit final – – – sn. ARN (small nuclear RNA) : épissage du pré-ARNm ARNr : structure du ribosome ARNt : intermédiaire entre acide aminé et le ribosome sno. ARN (small nucleolar RNAs) : modification des ARNr Autres ARN : télomères, inactivation de l'X, SRP, … 30

Principaux types d'ARN produits par la cellule 31

Unité de transcription • Segment d'ADN transcrit • Eucaryotes : un gène donc une molécule d'ARN ou une (. . . ) protéine • Bactérie : souvent transcription de plusieurs gènes adjacents plusieurs protéines 32

Quelques chiffres d'ARN • ARN = quelques % du poids sec de la cellule • Le plus important en masse est l'ARNr • ARNm = 3 -5 % de l'ARN total • Il y a des dizaines de milliers d'ARNm différents • Il n'y a que 10 -15 molécules de chaque ARNm 33

Biochimie de la transcription • 2 problèmes – Où commencer ? – Où finir ? • Différent chez pro et eucaryotes – 1 - Procaryotes – 2 – Eucaryotes 34

1 – Procaryotes 35

L'initiation de la transcription • Principal point de la régulation de la synthèse des protéines • Principal acteur : ARN polymérase 36

L'ARN polymérase • Complexe multi protéique • Facteur détachable qui "dit" à la pol. où commencer • Adhère faiblement à l'ADN bactérien et glisse facilement le long de cet ADN… • …sauf si… • …elle rencontre un promoteur – Séquence spéciale d'ADN – Indiquant le point de départ pour la synthèse de l'ADN • facteur 37

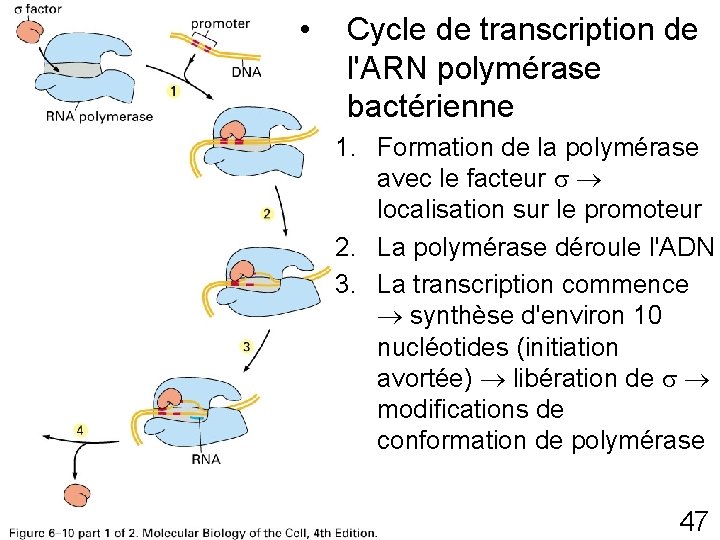
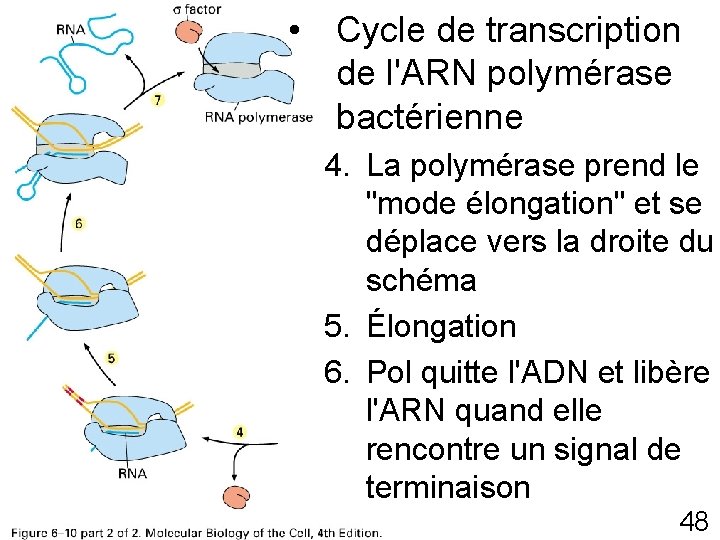
Cycle de transcription de l'ARN polymérase • Fixation de l ’ARN Polymérase sur le promoteur • Ouverture des 2 brins d ’ADN • Élongation de la molécule d ’ARN (5 ’ 3 ’) • Signal de terminaison Stop • Libération de l’ARN et de la polymérase 38

Étape 1 : Facteur • Reconnaît le promoteur 39

• Fig 6 -10 p 307(1 4) Cycle de transcription de l'ARN polymérase bactérienne 1. Formation de la polymérase avec le facteur localisation sur le promoteur 2. La polymérase déroule l'ADN 3. La transcription commence synthèse d'environ 10 nucléotides (initiation avortée) libération de modifications de conformation de polymérase 47

• Cycle de transcription de l'ARN polymérase bactérienne Fig 6 -10 p 307(5 7) 4. La polymérase prend le "mode élongation" et se déplace vers la droite du schéma 5. Élongation 6. Pol quitte l'ADN et libère l'ARN quand elle rencontre un signal de terminaison 48

Signaux de terminaison – Codés par l'ADN – Conduisent à la formation d'un ARN dont la structure déstabilise la fixation de la pol sur l'ARN 49

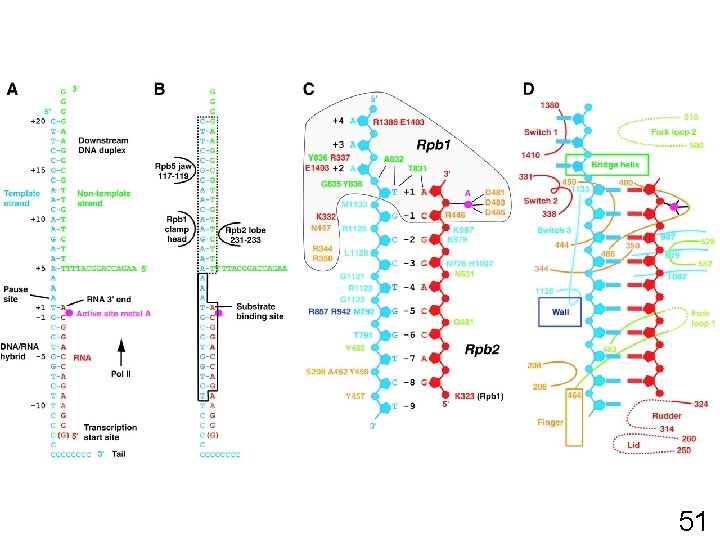
COVER The enzyme RNA polymerase II in the act of transcribing a gene. X-ray crystal structure comprises the protein (gray, except for orange "clamp" and green "bridge" helix), DNA (blue template strand, green nontemplate strand), and RNA (red). The pink sphere is an active center Mg 2+ ion. Double-stranded DNA enters from the right and unwinds before the active center. The unwound nontemplate DNA strand is obscured by motion or disorder. 1876 [Adapted from Fig. 2 C of Gnatt et al. ] Science, 8 juin 2001, cover 50

• Fig. 1. Nucleic acids in the transcribing complex and their interactions with pol II. (A) DNA ("tailed template") and RNA sequences. DNA template and nontemplate strands are in blue and green, respectively, and RNA is in red. This color scheme is used throughout. (B) Ordering of nucleic acids in the transcribing complex structure. Nucleotides in the solid box are well ordered. Nucleotides in the dashed box are partially ordered, whereas those outside the boxes are disordered. Three protein regions that abut the downstream DNA are indicated. (C) Protein contacts to the ordered nucleotides boxed in (B). Amino acid residues within 4 Å of the DNA are indicated, colored according to the scheme for domainlike regions of Rpb 1 or Rpb 2 (3). Ribose sugars are shown as pentagons, phosphates as dots, and bases as single letters. Amino acid residues listed beside phosphates contact only this nucleotide. Amino acid residues listed beside riboses contact this nucleotide and its 3'-neighbor. Singleletter abbreviations for the amino acid residues are as follows: A, Ala; D, Asp; E, Glu; G, Gly; H, His; K, Lys; L, Leu; M, Met; N, Asn; Q, Gln; R, Arg; S, Ser; T, Thr; V, Val; and Y, Tyr. (D) Schematic representation of protein features participating in the detailed interactions shown in (C). Same notation as in (C), except that bases are shown as thick bars. Gnatt, AL 2001 p 1876 fig 1 Scienc e 51

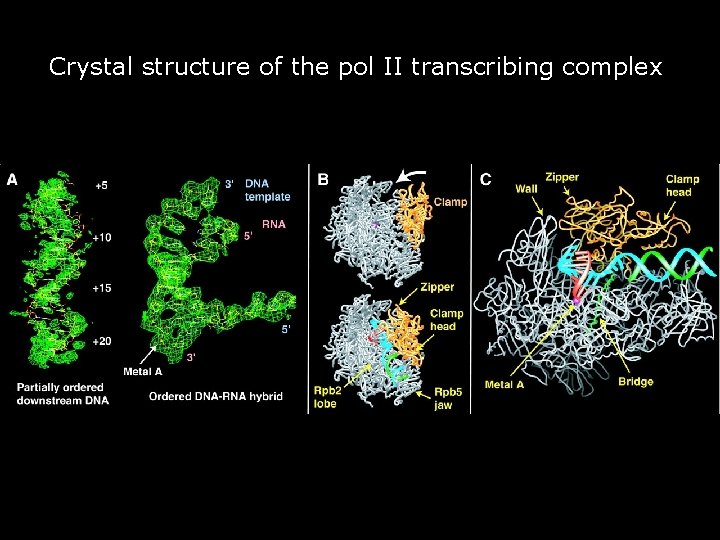
Crystal structure of the pol II transcribing complex. Gnatt, AL 2001 p 1876 fig 2 • • Fig. 2. Crystal structure of the pol II transcribing complex. (A) Electron density for the nucleic acids. On the left, the final sigma-weighted 2 m. Fobs DFcalc electron density for the downstream DNA duplex (dashed box in Fig. 1 B) is contoured at 0. 8 (green). At this contour level, the surrounding solvent region shows only scattered noise peaks. A canonical 16 -base pair B-DNA duplex was placed into the density. On the right, the final model of the DNA-RNA hybrid and flanking nucleotides (boxed in Fig. 1 B) is superimposed on a simulated-annealing Fobs Fcalc omit map, calculated from the protein model alone with CNS (45) (green, contoured at 2. 6 ). The location of the active site metal A is indicated. (B) Comparison of structures of free pol II (top) and the pol II transcribing complex (bottom). The clamp (yellow) closes on DNA and RNA, which are bound in the cleft above the active center. The remainder of the protein is in gray. (C) Structure of the pol II transcribing complex. Portions of Rpb 2 that form one side of the cleft are omitted to reveal the nucleic acids. Bases of ordered nucleotides (boxed in Fig. 1 B) are depicted as cylinders protruding from the backbone ribbons. The Rpb 1 bridge helix traversing the cleft is highlighted in green. The active site metal A is shown as a pink sphere. 52

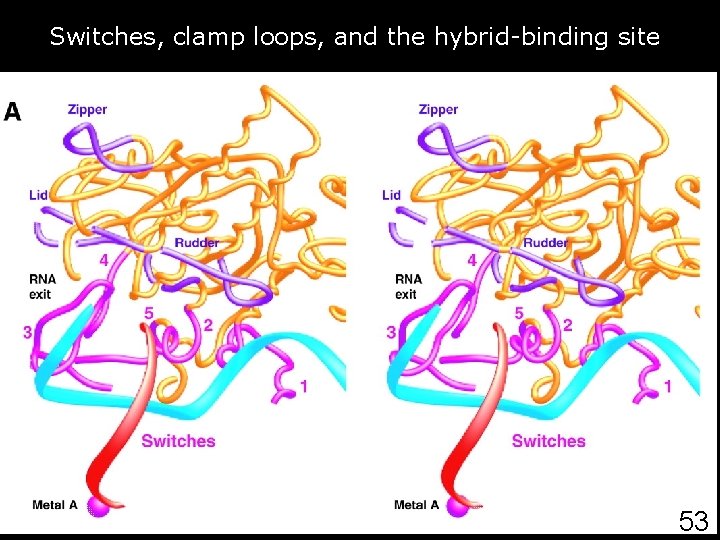
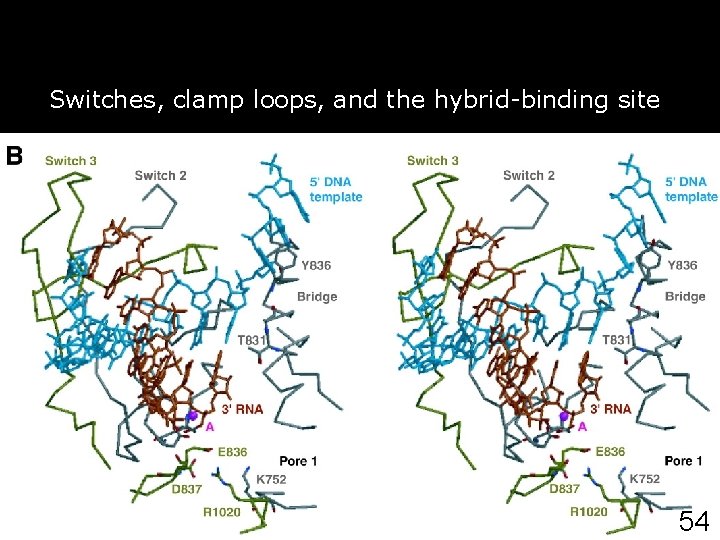
Switches, clamp loops, and the hybrid-binding site Gnatt, AL 2001 p 1876 fig 3 • Fig. 3. Switches, clamp loops, and the hybridbinding site. • (A) Stereoview of the clamp core (1, yellow) and the DNA and RNA backbones. The view is as in Fig. 2 C. The five switches are shown in pink and are numbered. Three loops, which extend from the clamp and may be involved in transactions at the upstream end of the transcription bubble, are in violet. Major portions of the protein are omitted for clarity. 53

Gnatt, AL 2001 p 1876 fig 3 Switches, clamp loops, and the hybrid-binding site • Fig. 3. Switches, clamp loops, and the hybrid -binding site. • (B) Stereoview of nucleic acids bound in the active center. 54

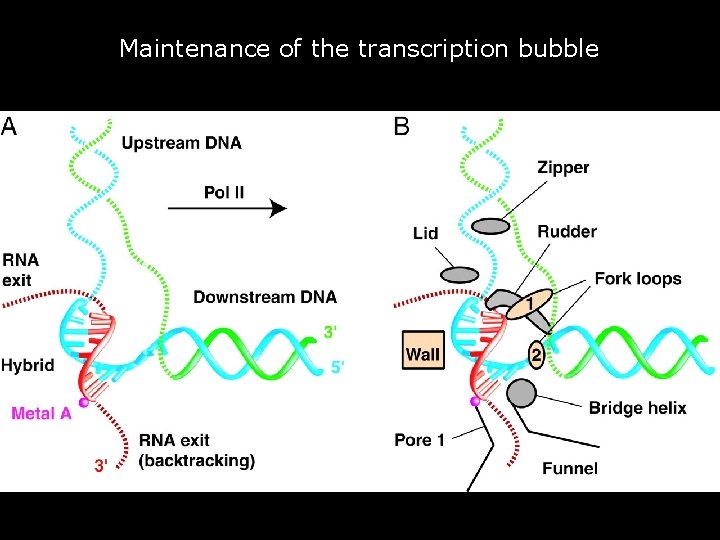
Maintenance of the transcription bubble • Fig. 4. Maintenance of the transcription bubble. • (A) Schematic representation of nucleic acids in the transcribing complex. Solid ribbons represent nucleic acid backbones from the crystal structure. Gnatt, AL 2001 p 1876 fig 4 Dashed lines indicate possible paths of nucleic acids not present in the structure. • (B) Protein elements proposed to be involved in maintaining the transcription bubble. Protein elements from Rpb 1 and Rpb 2 are shown in silver and gold, respectively. 55

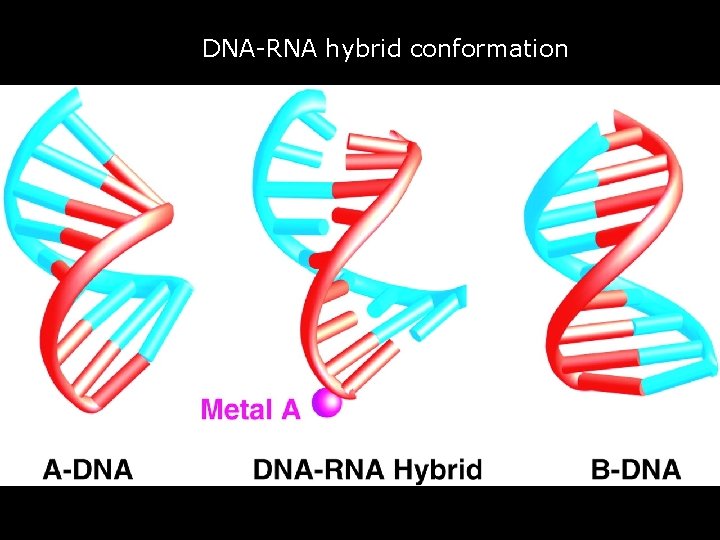
DNA-RNA hybrid conformation • Fig. 5. DNA-RNA hybrid conformation. The view is similar to that in Fig. 2 C. The conformation of the DNA-RNA hybrid is Gnatt, AL 2001 p 1876 fig 5 intermediary between canonical A- and BDNA. DNA, blue; RNA, red. 56

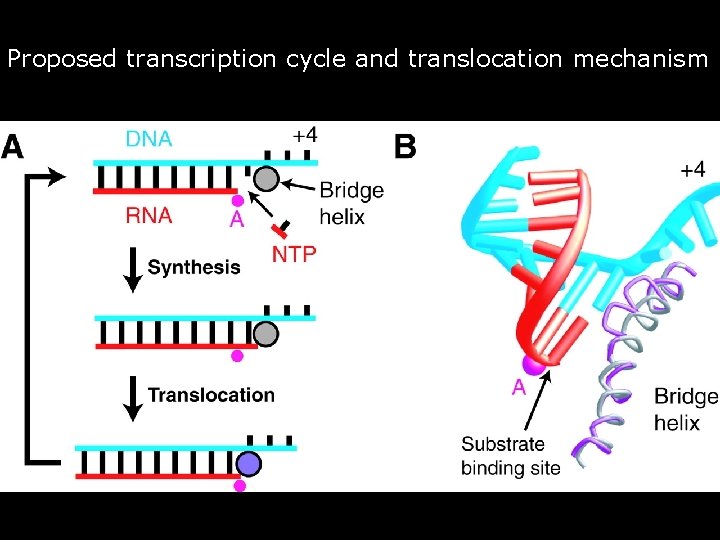
Proposed transcription cycle and translocation mechanism • • Fig. 6. Proposed transcription cycle and translocation mechanism. (A) Schematic representation of the nucleotide addition cycle. The nucleotide triphosphate (NTP) fills the open substrate site (top) and forms a phosphodiester bond at the active site ("Synthesis"). This results in the state of the transcribing complex seen in the crystal structure (middle). We speculate that "Translocation" of the nucleic acids with respect to the active site (marked by a pink dot for metal A) involves a change of the bridge helix from a straight (silver circle) to a bent conformation (violet circle, bottom). Relaxation of the bridge helix back to a straight conformation without movement of the nucleic acids would result in an open substrate site one nucleotide downstream and would complete the cycle. (B) Different conformations of the bridge helix in pol II and bacterial RNA polymerase structures. The view is the same as in Fig. 2 C. The bacterial RNA polymerase structure (2) was superimposed on the pol II transcribing complex by fitting residues around the active site. The resulting fit of the bridge helices of pol II (silver) and the bacterial polymerase (violet) is shown. The bend in the bridge helix in the bacterial polymerase structure causes a clash of amino acid side chains (extending from the backbone shown here) with the hybrid base pair at position +1. Gnatt, AL 2001 p 1876 fig 6 • 57

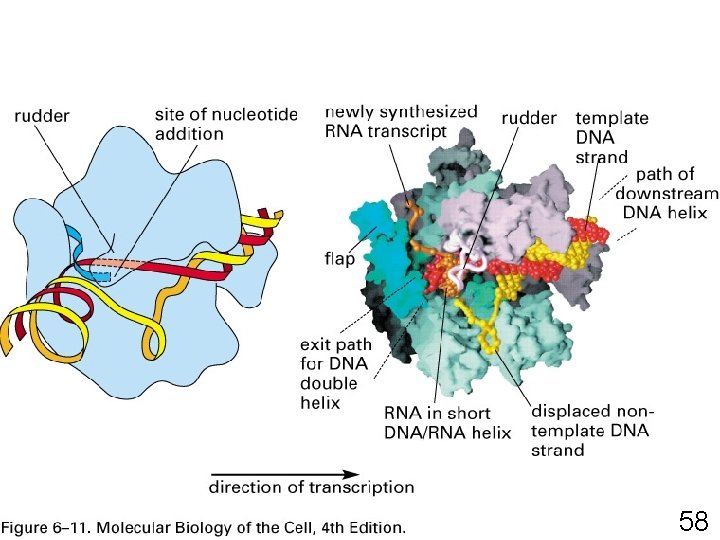
Fig 6 -11 58

LES ARN POLYMÉRASES Facteurs de Transcription chez les eucaryotes • I ARN ribosomaux • II ARN qui seront traduits + ARN qui forment les sn. RNP • III ARN très petits et stables – ARN ribosomal 5 S – ARN de transfert – etc. . . 61

62

63

64

65

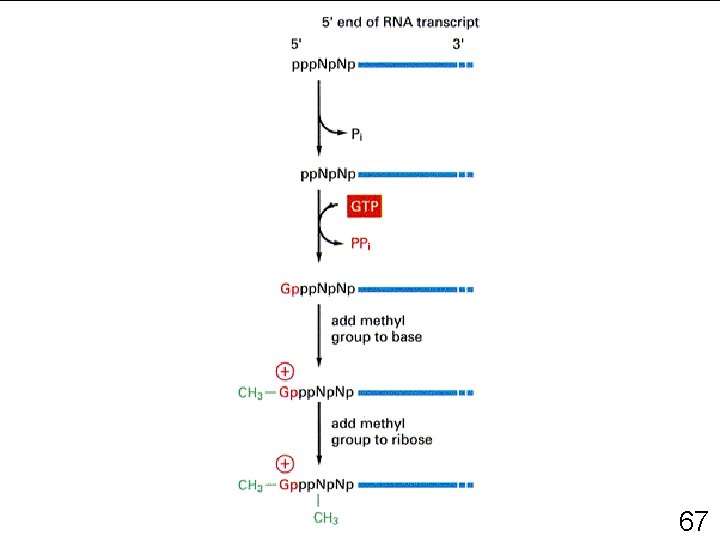
MATURATION DES ARN • Chapeau 5 ’ 66

67

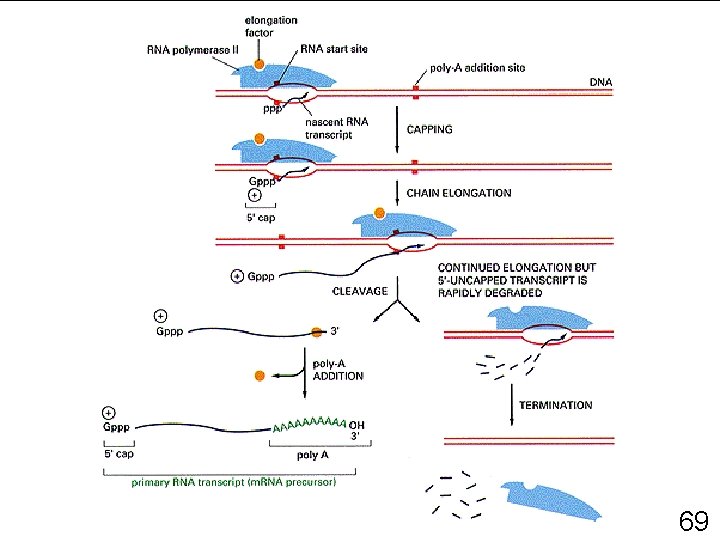
MATURATION DES ARN • Chapeau 5 ’ – Initiation de la synthèse protéique – Prévient la dégradation prématurée de l ’ARN • Queue de poly - A (acide adénylique) 68

69

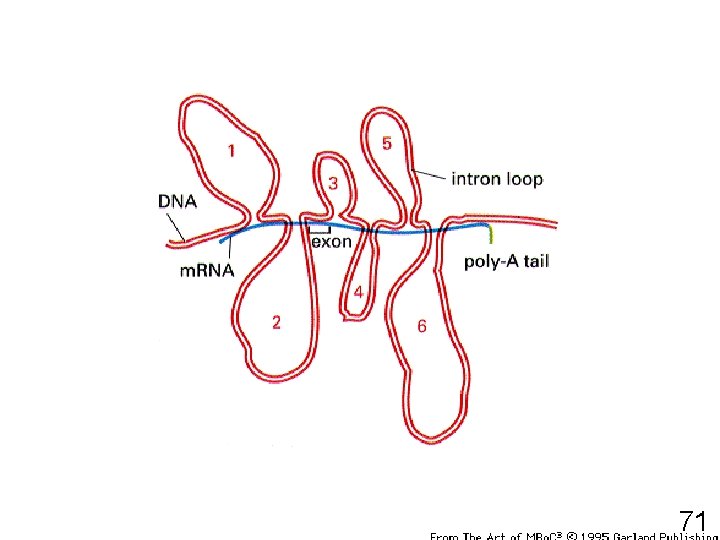
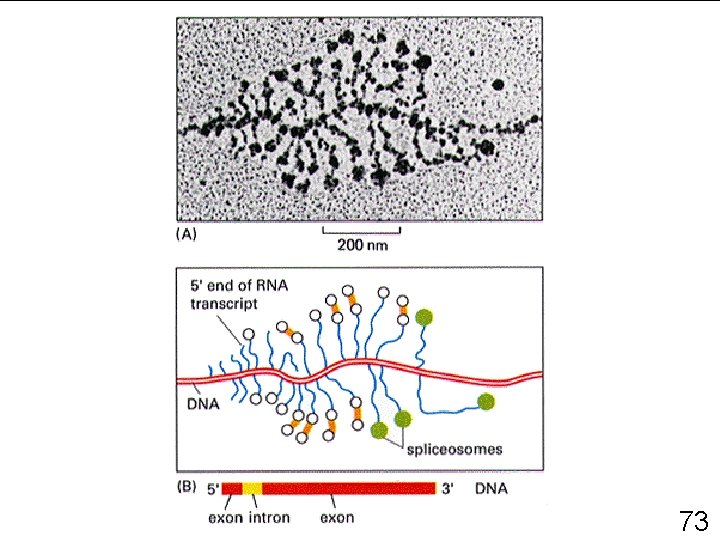
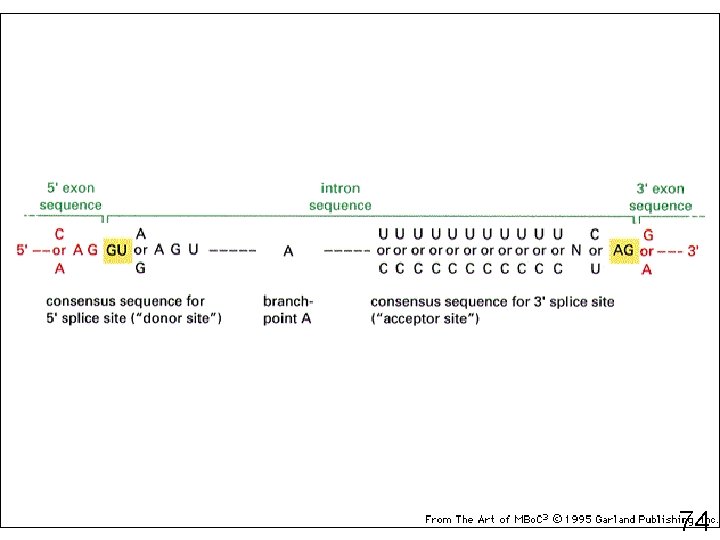
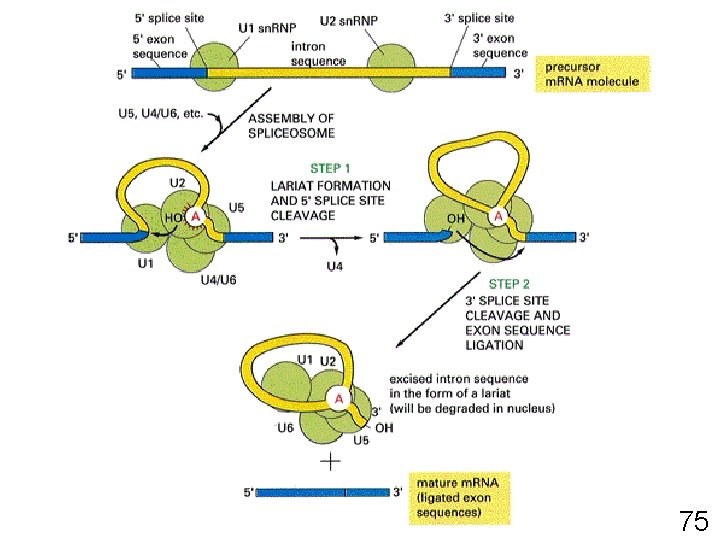
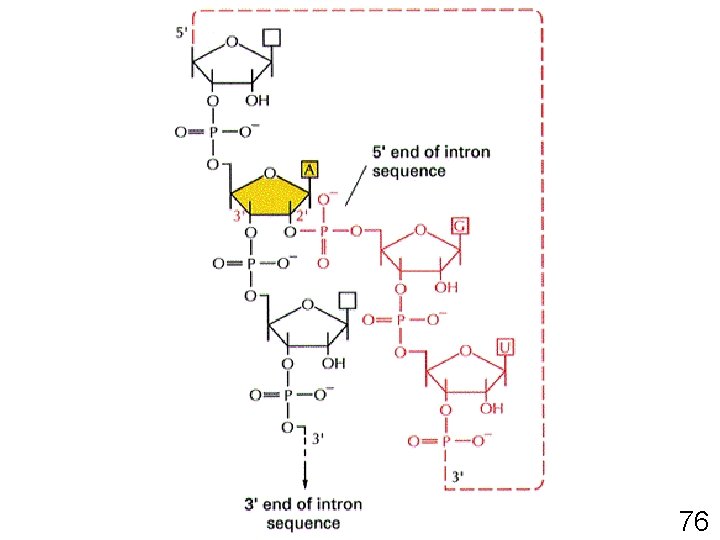
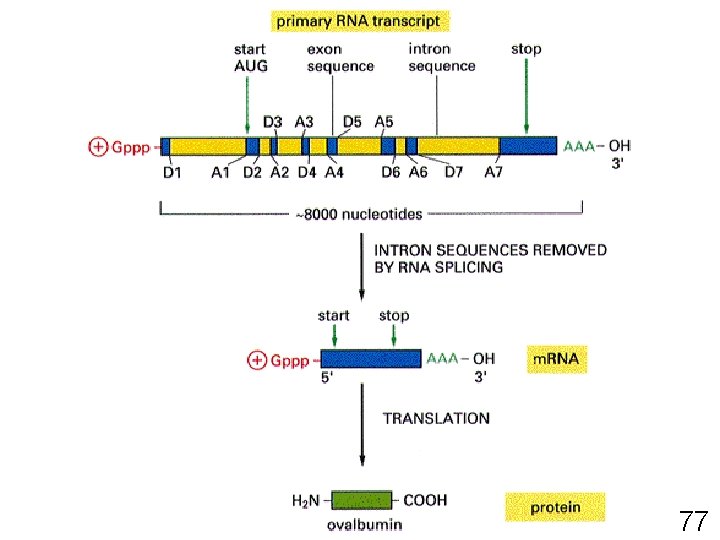
MATURATION DES ARN • Chapeau 5 ’ – Initiation de la synthèse protéique – Prévient la dégradation prématurée de l ’ARN • Queue de poly - A en 3 ’ (acide adénylique) – Exportation des ARMm matures hors du noyau – Stabilisation de certains ARNm – Signal de reconnaissance pour le ribosome • Épissage 70

71

72

73

74

75

76

77

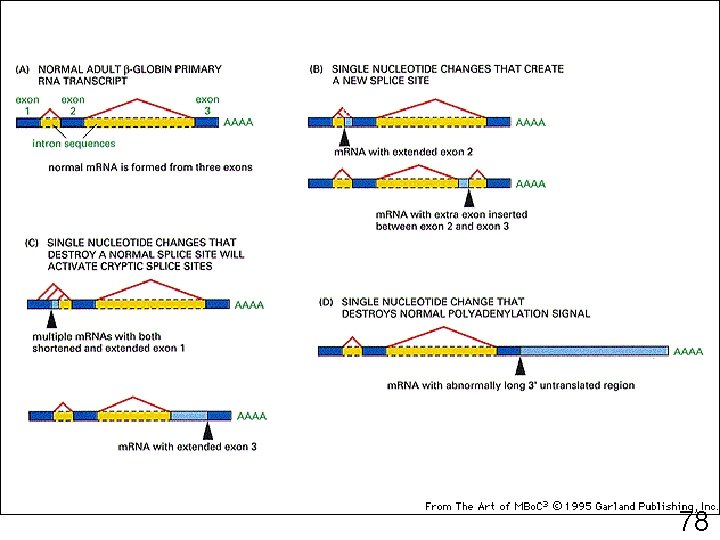
78

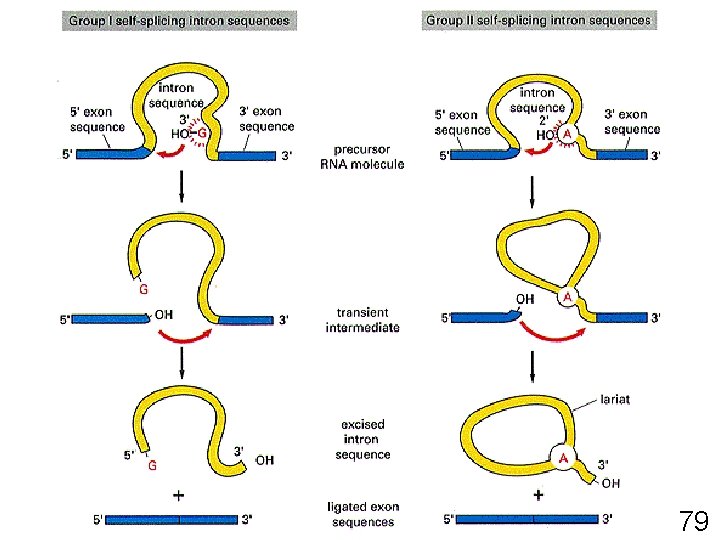
79

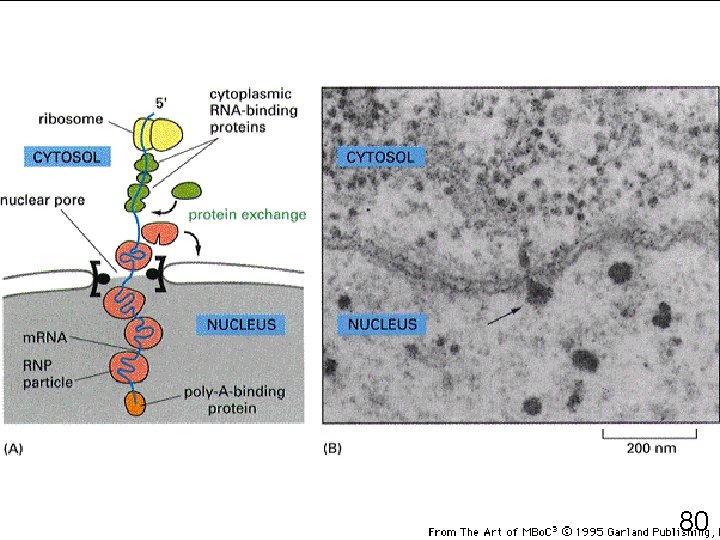
80