Comment les cellules lisent le gnome de lADN































- Slides: 67

Comment les cellules lisent le génome : de l'ADN à la protéine

Les ribosomes 2

Les ribosomes • Machine catalytique • Plusieurs millions dans le cytoplasme • Composition – plus de 50 protéines : les protéines ribosomales – 4 molécules d'ARN • Assemblés dans le nucléole • Deux sous-unités – une grosse – une petite 3

Le ribosome • Petite sous-unité – ARNt – Appariement • Grosse sous-unité – Liaison peptidique • Se lient puis se séparent • Eucaryote : 2 AA par seconde et par ribosome • Procaryote : 20 AA par seconde et par ribosome 4

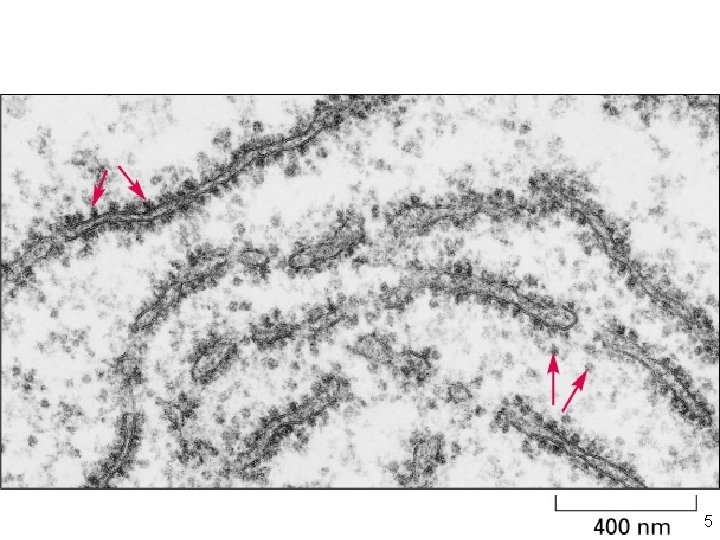
• Ribosomes dans le cytoplasme d'une cellule eucaryote Fig 6 -62 5

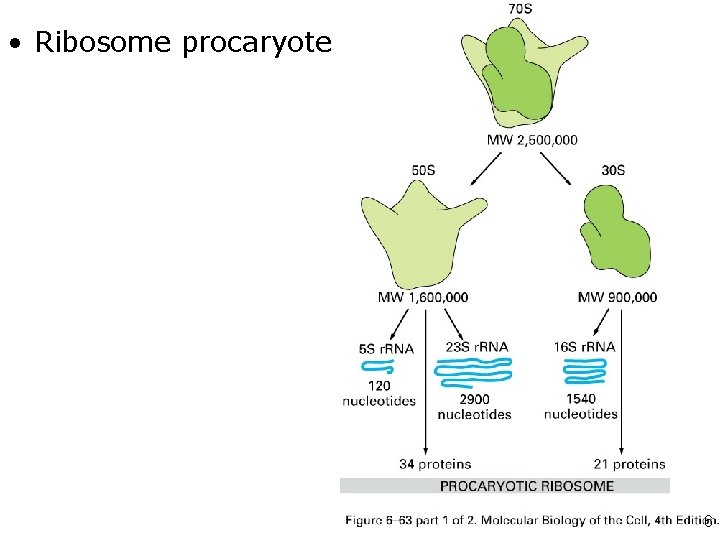
• Ribosome procaryote Fig 6 -63 -1 6

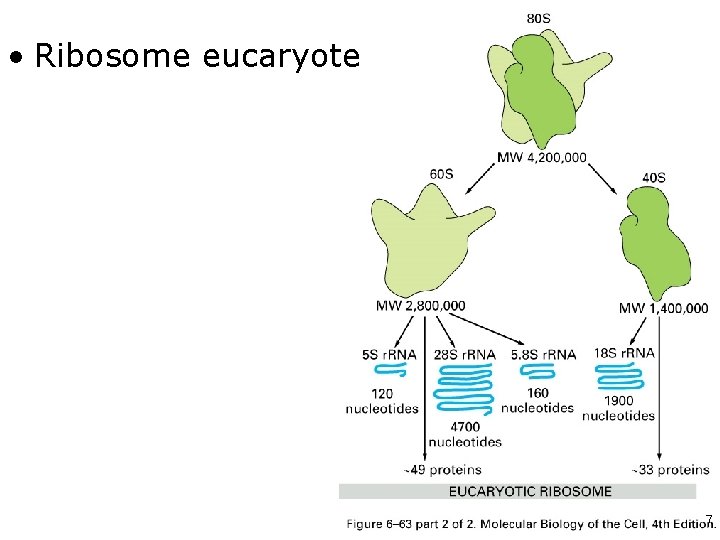
• Ribosome eucaryote Fig 6 -63 -2 7

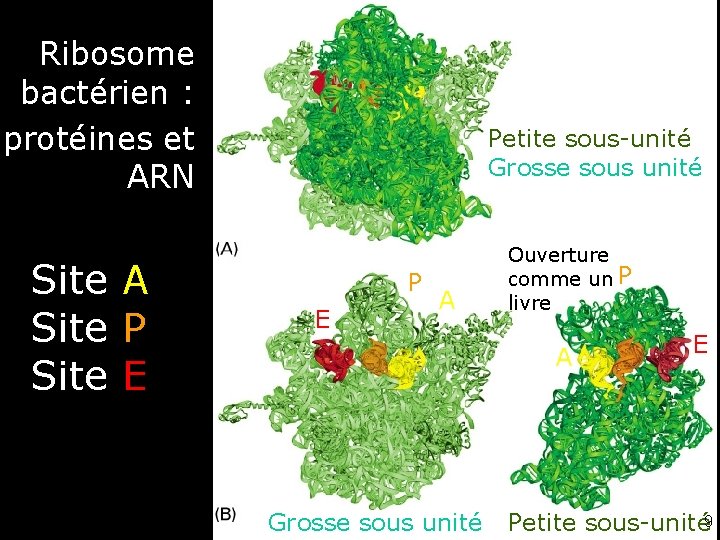
Sites de liaison de l'ARN dans le ribosome • ARNt (3 sites) – Site A : Aminoacyl-t. RNA – Site P : Peptidyl-t. RNA – Site E : Exit • ARNm (1 site) • 2 sites seulement sont occupés à la fois 8

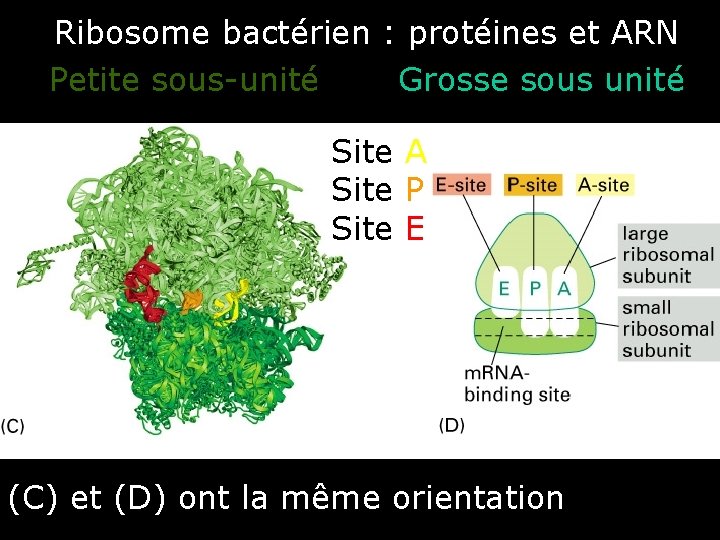
Ribosome bactérien : protéines et ARN Site A Site P Site E Fig 6 -64 -1 Sites de liaison Petite sous-unité du ribosome Grosse sous unité P E A Ouverture comme un P livre A Grosse sous unité E Petite sous-unité 9

Ribosome bactérien : protéines et ARN Petite sous-unité Grosse sous unité Site A Site P Site E Fig 6 -64 -2 Sites de liaison du ribosome (C) et (D) ont la même orientation 10

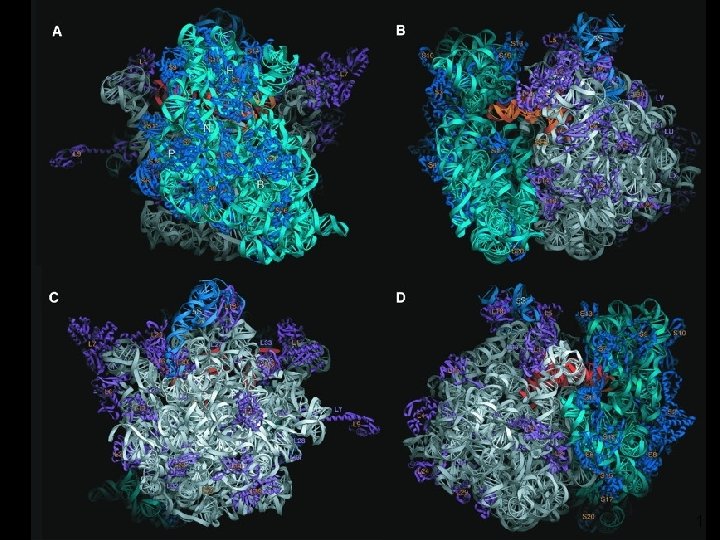
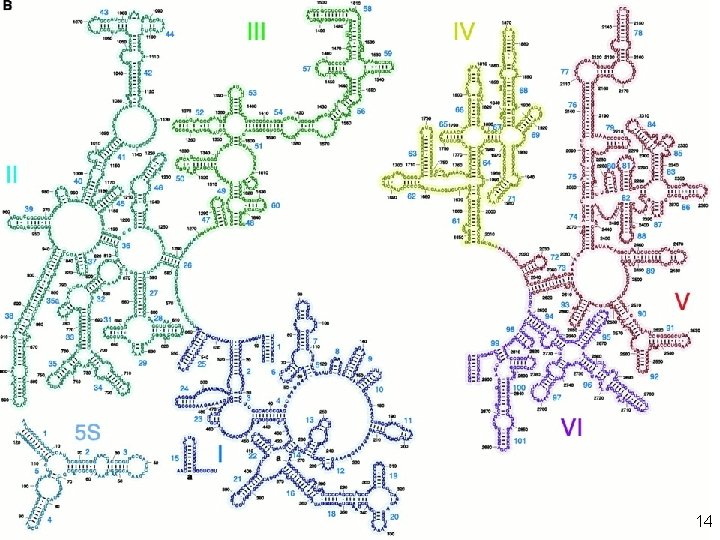
Yusupov, MM 2001 fig 2 11

Yusupov, MM 2001 fig 2 12

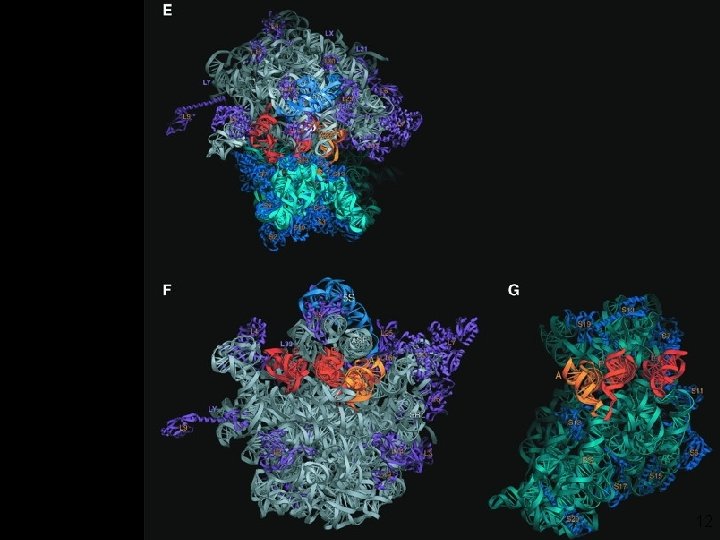
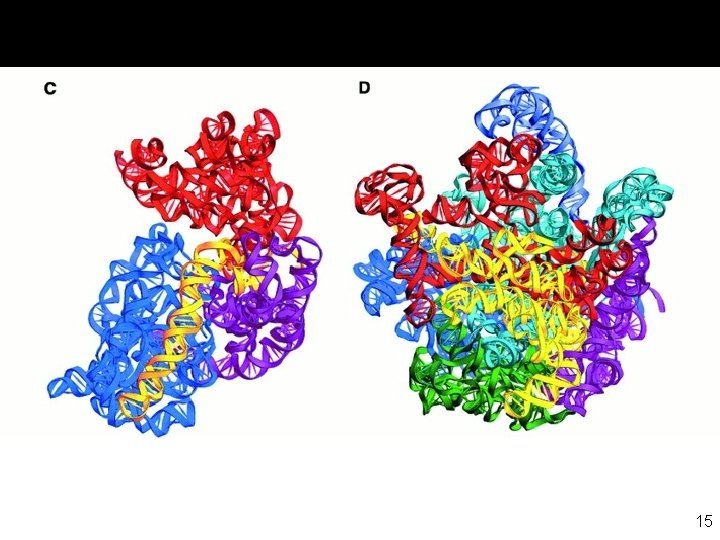
Yusupov, MM 2 001 fig 3 a 13

Yusupov, MM 2001 fig 3 b 14

Yusupov, MM 2001 fig 3 cd 15

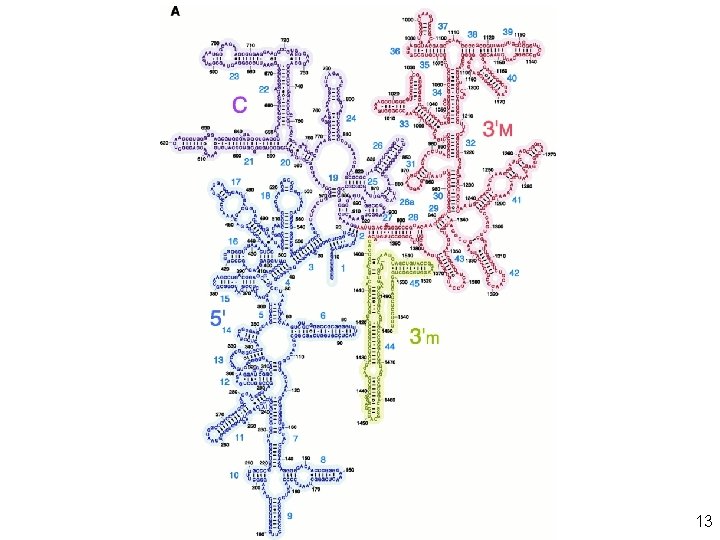
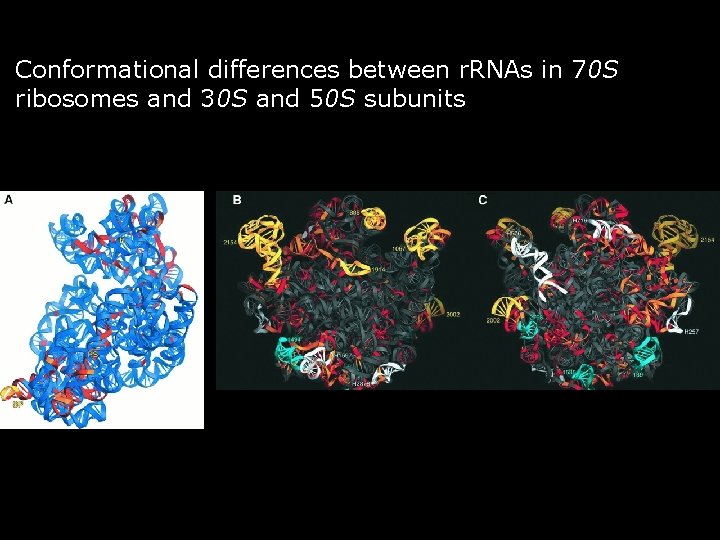
Conformational differences between r. RNAs in 70 S ribosomes and 30 S and 50 S subunits Yusupov, MM 2001 fig 4 16

Comment les cellules lisent le génome : de l'ADN à la protéine I. De l'ADN à l'ARN II. De l'ARN à la protéine III. Le monde de l'ARN et les origines de la vie 27

Comment les cellules lisent le génome : de l'ADN à la protéine I. De l'ADN à l'ARN II. De l'ARN à la protéine III. Le monde de l'ARN et les origines de la vie 28

I - De l'ADN à l'ARN 29

Synthèse des ARN non codant (ribosomaux) • ARN = quelques % du poids sec de la cellule • ARNm = 3 -5% des ARN totaux • ARNr = 80 % des ARN de la cellule 30

ARN ribosomaux • Polymérase (chez les procaryotes) • Polymérase I (chez les eucaryotes) – très proche de pol II – pas de queue C terminale (à la pol I) • pas de chapeau 5' (au transcrit) • pas de queue poly. A (au transcrit) 31

Amplification de la production des gènes • ARNm – gène unique (en général) – amplification à la traduction des protéines produites • ARNr – produit final – pas d'amplification à la traduction mais – gènes en copies multiples (= "ADN ribosomal") 32

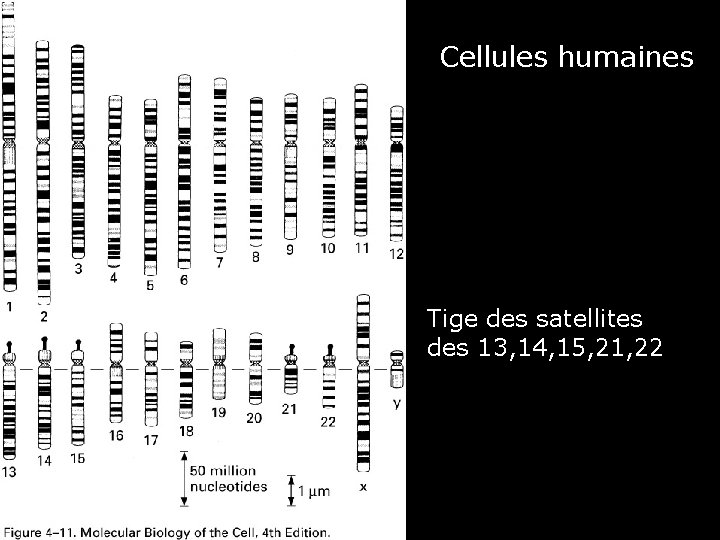
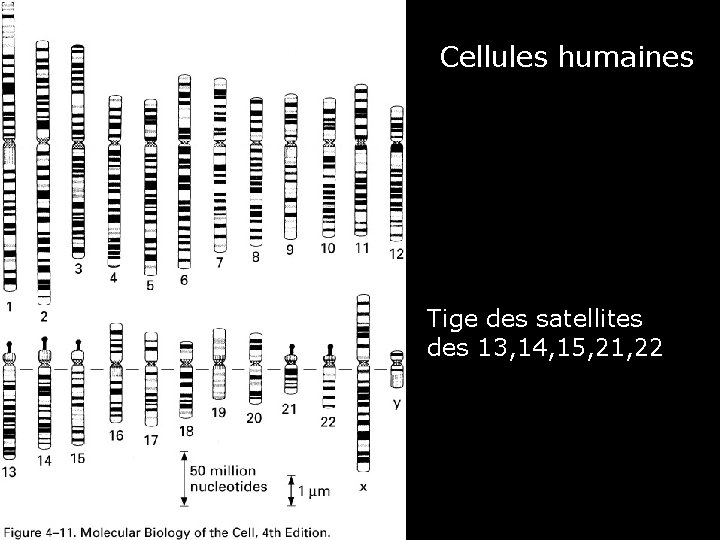
Besoins en ARNr • 10 millions de copies de chaque type d'ARNr pour construire 10 millions de ribosomes • E Coli : 7 copies de ses gènes à ARN • Cellules humaines : – 200 copies par génome haploïde – dispersés sur 5 chromosomes (13, 14, 15, 21, 22) – ARN 5 S localisé sur le chromosome 1 • Xenopus : – 600 copies par génome haploïde – un seul groupe sur un seul chromosome 33

Cellules humaines Fig 4 -11 Caryotype (schéma) Tige des satellites des 13, 14, 15, 21, 22 34

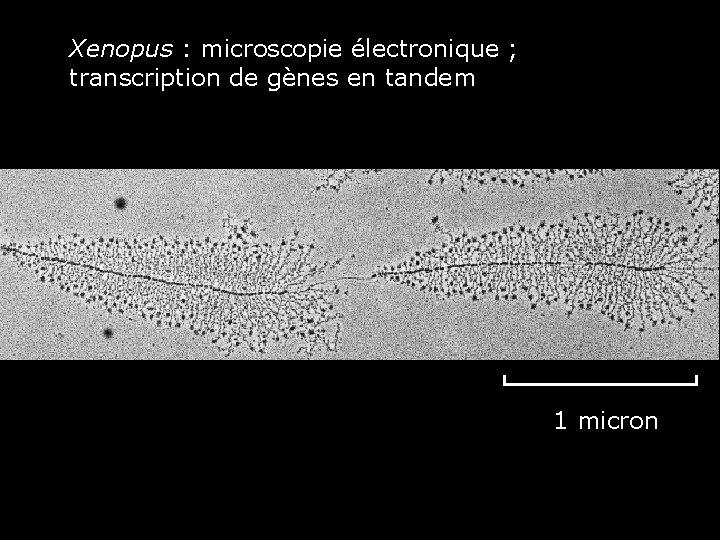
Xenopus : microscopie électronique ; transcription de gènes en tandem Fig 6 -41 ME transcription de gènes en tandem 35

Xenopus : microscopie électronique ; transcription de gènes en tandem Fig 6 -9 ME transcription de gènes en tandem FG 1 micron 36

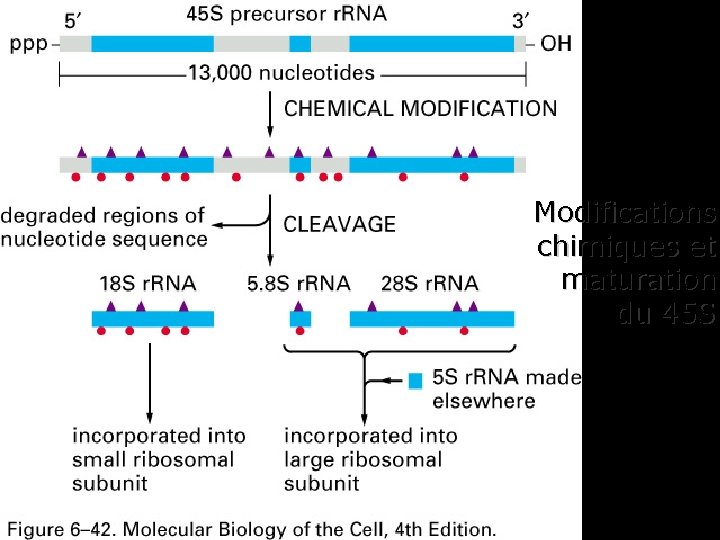
Les ARNr • 4 types (18 S, 28 S, 5, 8 S, 5 S) • une copie par ribosome • 18 S, 28 S, 5, 8 S : gros précurseur modifié de 45 S • 5 S – Chromosome 1 (chez l’homme) – groupe de gènes séparés – ARN polymérase III – pas de modifications chimiques – pourquoi ? 37

Fig 6 -42 Modifications chimiques et maturation du 45 S 38

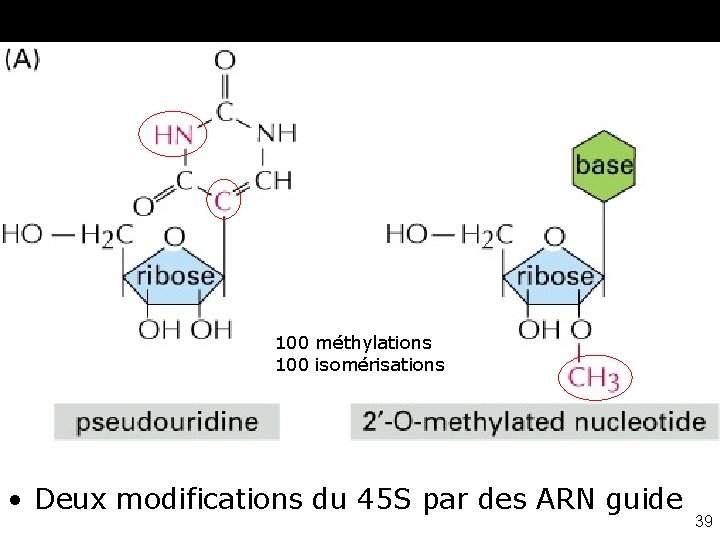
Fig 6 -43 A 100 méthylations 100 isomérisations • Deux modifications du 45 S par des ARN guide 39

Les ARN "guide" • Les modifications chimiques du précurseur 45 S – aident au repliement – et à l'assemblage de l'ARN dans le ribosome – se font à des positions spécifiques du précurseur – grâce à • des ARN "guides" 40

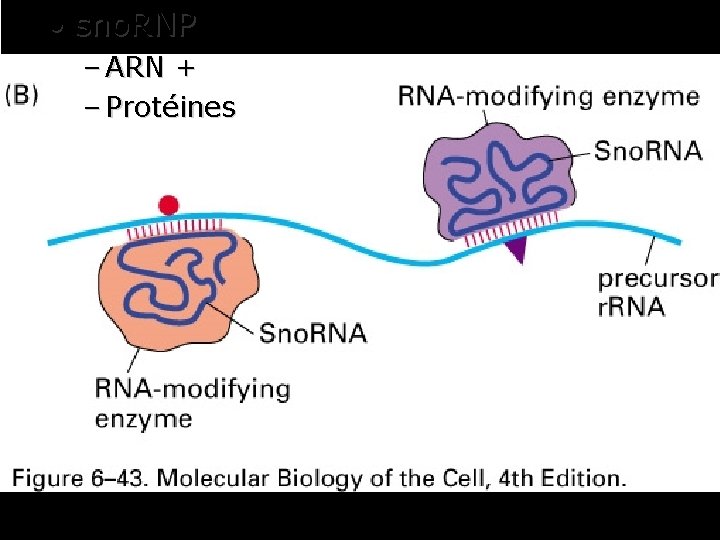
• sno. RNP – ARN + – Protéines Fig 6 -43 B 41

Les sno. ARN (small nucleolar ARNs) • comprennent les ARN guides • Participent à la formation du nucléole • souvent codés dans les introns d'autres gènes • donc synthétisés par la pol II 42

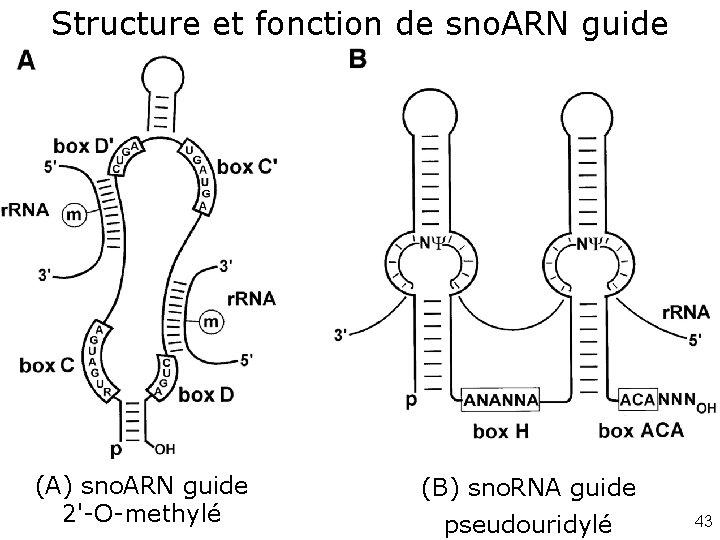
Structure et fonction de sno. ARN guide Kiss, T 2001(fig 1) • Fig. 1. Structure and function of (A) 2'-Omethylation and (B) pseudouridylation guide sno. RNAs. The consensus sequences of boxes C, C', D, D', H and ACA are indicated (R is a purine and N stands for any nucleotide). Models for molecular selection of 2'-Omethylated nucleotides and pseudouridine were adopted frfrom Kiss-Lââszlô ô et al. (1998) and Ganot et al. (1997 a), respectively. (A) sno. ARN guide 2'-O-methylé (B) sno. RNA guide pseudouridylé 43

• • • Figure 1. Structure and Function of Box C/D and Box H/ACA sno. RNAs In 2'-O-methylation guide sno. RNAs, the box C and D motifs and a short 5', 3'-terminal stem constitute a kink-turn (K-turn) structural motif that is specifically recognized by the "15. 5 k. Da sno. RNP protein. The C' and D' boxes represent internal, frequently imperfect copies of the C and D boxes. Dashed lines indicate nucleotides interacting in the C and D boxes (Watkins et al. , 2000; Klein et al. , 2001). The pseudouridylation guide sno. RNAs fold into a “hairpin-hinge-hairpin-tail” structure and contain the H and ACA boxes. The box C/D 2'-O-methylation guide sno. RNAs and the substrate RNAs form a 10– 21 base pair double helix in which the target residue is positioned exactly five nucleotides upstream of the D or D' box. The 5' and/or 3' hairpin of the box H/ACA pseudouridylation guide sno. RNAs contains an internal loop, called the pseudouridylation pocket, that forms two short (3– 10 bp) duplexes with nucleotides flanking the unpaired substrate uridine that is located about 15 nucleotides from the H or ACA box of the sno. RNA. Although each box C/D and H/ACA sno. RNA could potentially direct two modification reactions, apart from a few exceptions, the majority of sno. RNAs possess only one functional 2'-O-methylation or pseudouridylation domain. The sno. RNA core motifs that are essential and sufficient for the correct processing and nucleolar accumulation of sno. RNAs are highlighted in red. Regions that do not contribute to the metabolic stability of sno. RNAs are blue. Sno. RNAs transcribed independently by RNA pol II contain 5' leader sequences and carry the trimethylguanosine cap structure (m 3 Gppp). Substrate RNAs are green. Nucleotides destined for pseudouridylation ('IN) and 2'-O-methylation (circled m) are marked. Kiss, T 2002 44

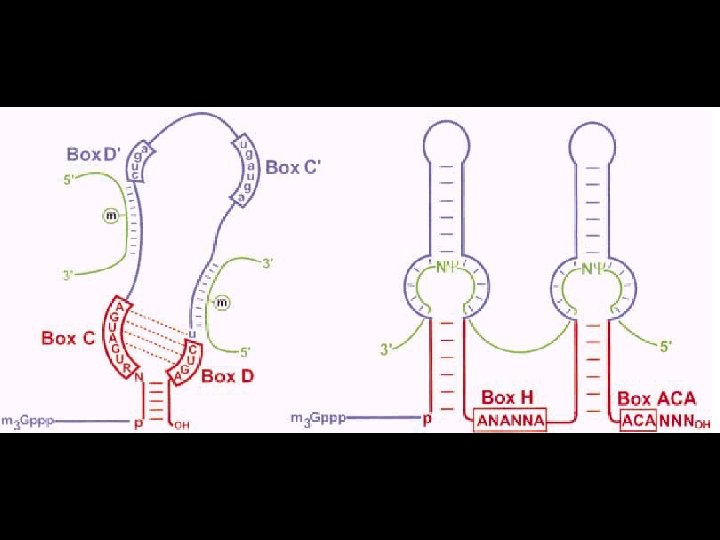
Biogenesis and function of 2'-O-methylation and pseudouridylation guide sno. RNAs Kiss, T 2001(fig 2) • Fig. 2. Biogenesis and function of 2'-O-methylation and pseudouridylation guide sno. RNAs. In mammalian cells, all guide RNAs are synthesized within introns of pre-m. RNAs in the nucleoplasm. Most intronic sno. RNAs are processed frfrom the removed and debranched host intron by exonucleolytic activities. It remains unclear whether 5' and 3' end processing of sno. RNAs occurs already in the nucleoplasm, or later in the nucleolus. Guide sno. RNAs accumulating in the nucleolus direct 2'-O-methylation and pseudouridylation of the 18 S, 5. 8 S and 28 S r. RNAs, the U 6 sn. RNA and perhaps other cellular RNAs, including t. RNAs, the signal recognition particle (SRP) and telomerase RNAs. It seems that box C/D, but not box H/ACA sno. RNAs, transiently appear in the Cajal body before accumulating in the nucleolus. Some guide RNAs (sca. RNAs) directing modification of the pol II-transcribed spliceosomal sn. RNAs accumulate in the Cajal body (CB). For other details, see the text. 45

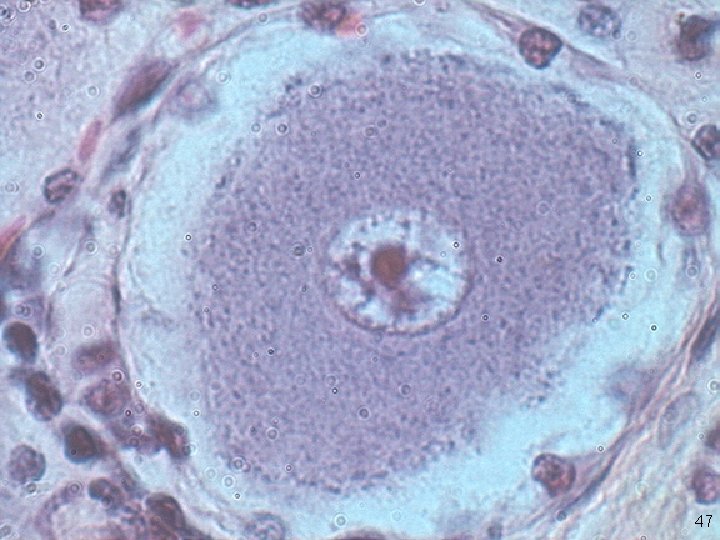
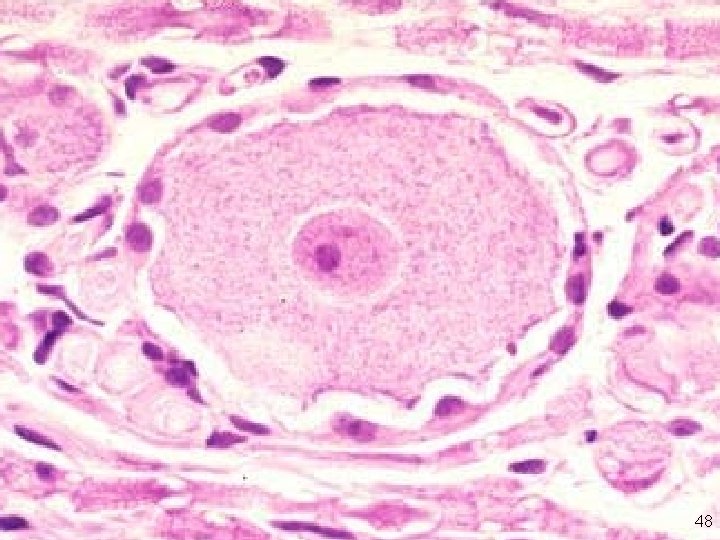
Le nucléole • Bien visible en microscopie optique dans le noyau des eucaryotes 46

Nucléole en microscopie optique 47

Nucléole en microscopie optique 48

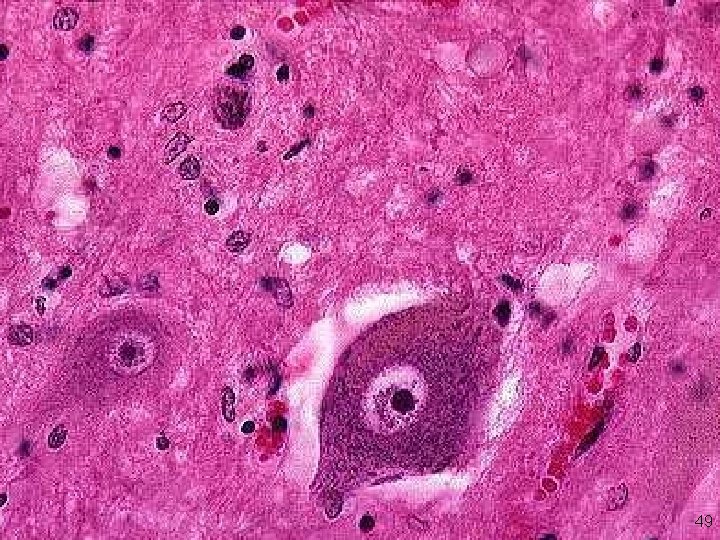
Nucléole en microscopie optique 49

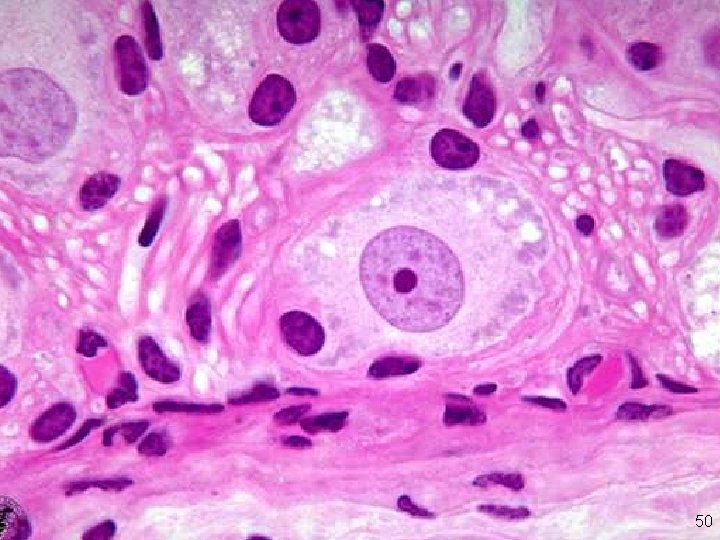
Nucléole en microscopie optique 50

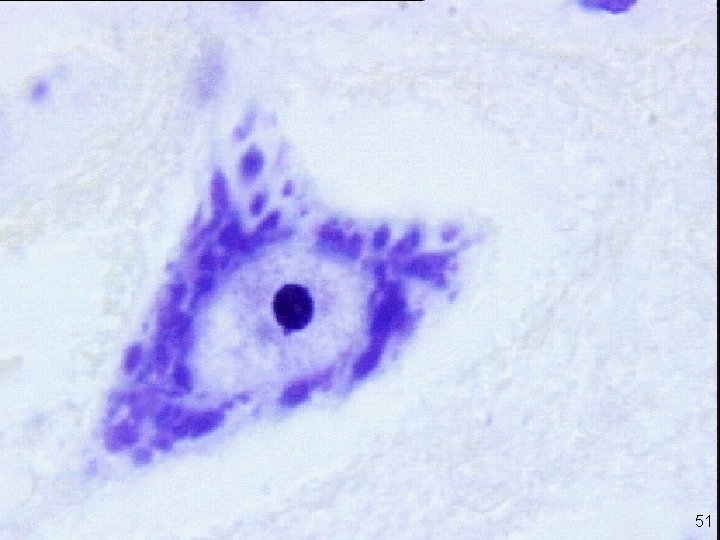
Nucléole en microscopie optique 51

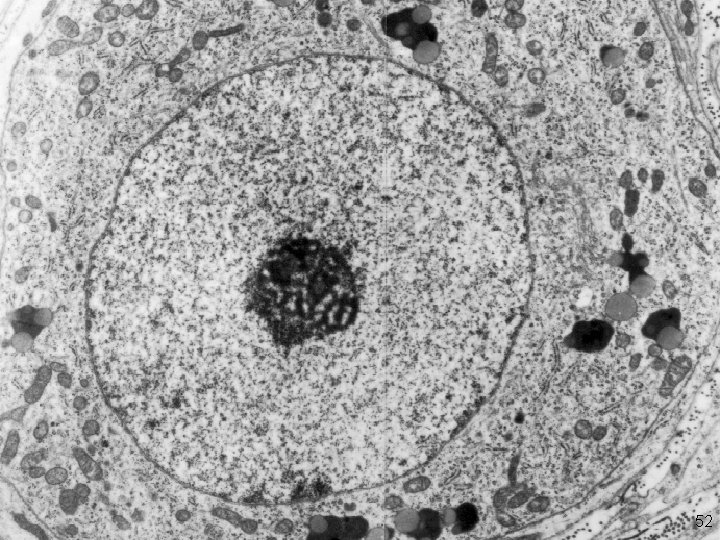
Nucléole en microscopie électrinique 52

Le nucléole • • Site de synthèse et de maturation des ARNr Assemblage des ARNr en ribosomes Pas de membrane Contient : – gènes d'ARNr – précurseurs d'ARNr – ARNr matures – enzymes de la maturation des ARNr – sno. RNPs – protéines ribosomales 53

Le nucléole • 700 références en 1898 • Évolution à partir d'une ancienne structure • Taille fonction du nombre de ribosomes que la cellule produit • 3 composants – centre fibrillaire – composant fibrillaire dense – composant granulaire • Transcription : entre centre fibrillaire et composant fibrillaire dense • Maturation et assemblage : du centre vers la périphérie 54

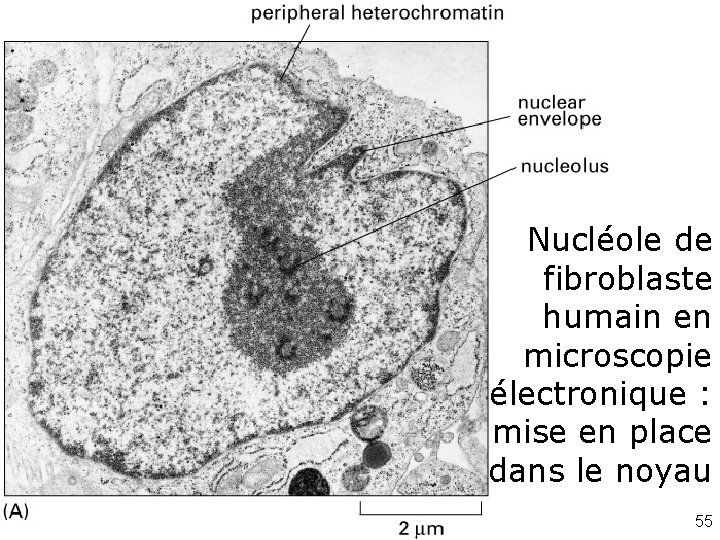
Fig 6 -44 ME Nucléole de fibroblaste humain en microscopie électronique : mise en place dans le noyau 55

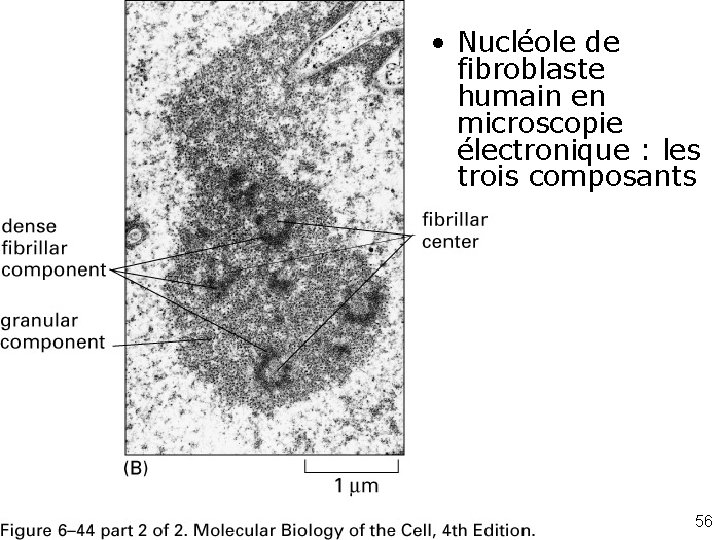
• Nucléole de fibroblaste humain en microscopie électronique : les trois composants Fig 6 -44 -2 ME FG du nucléole 56

Le nucléole • Les gènes d'ARN ribosomal sont répartis en 10 groupes sur les chromosomes acrocentriques 57

Cellules humaines Fig 4 -11 Caryotype (schéma) Tige des satellites des 13, 14, 15, 21, 22 58

Modifications morphologiques du nucléole au cours du cycle cellulaire Fig 6 -45 59

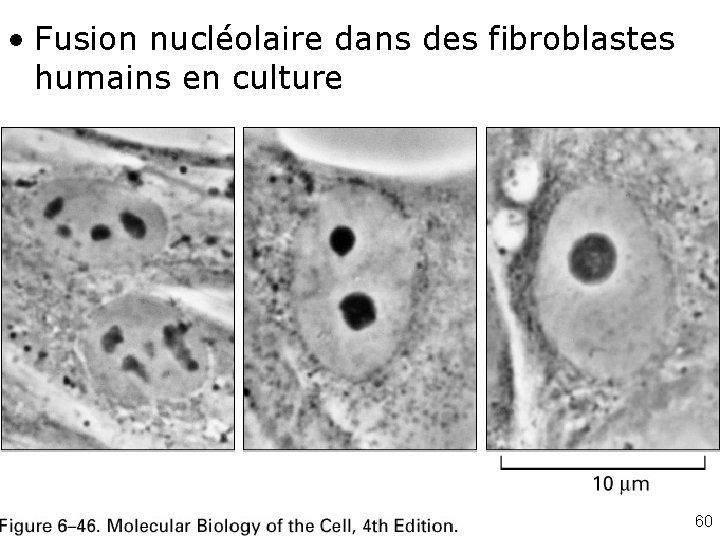
• Fusion nucléolaire dans des fibroblastes humains en culture Fig 6 -46 60

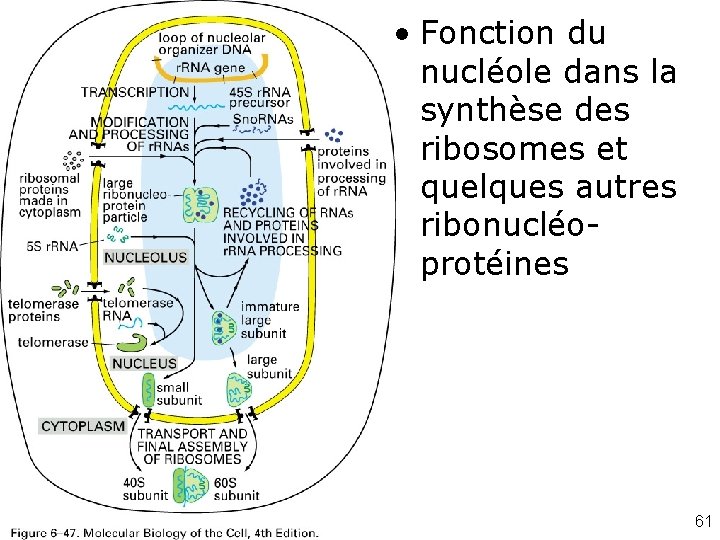
• Fonction du nucléole dans la synthèse des ribosomes et quelques autres ribonucléoprotéines Fig 6 -47 61

Autres fonctions du nucléole • Maturation de U 6 sn. RNP (une molécule d'ARN+7 protéines) • Assemblage de télomérase • Assemblage de SRP • Maturation de ARN de transfert • Grosse usine de ribonucléoprotéines de toutes sortes avec des ARN non codant 62

Autres structures intranucléaires • Corps de Cajal (1906) • Gemini of Coiled bodies (GEMS) • Granules interchromatiniens (= speckles) 63

Autres structures intra-nucléaire • Pas de membrane • Très dynamique • Résultent de l'association de protéines et d'ARN (et d'ADN ? ) impliquée dans la synthèse, assemblage, stockage de macromolécules impliquées dans l'expression des gènes 64

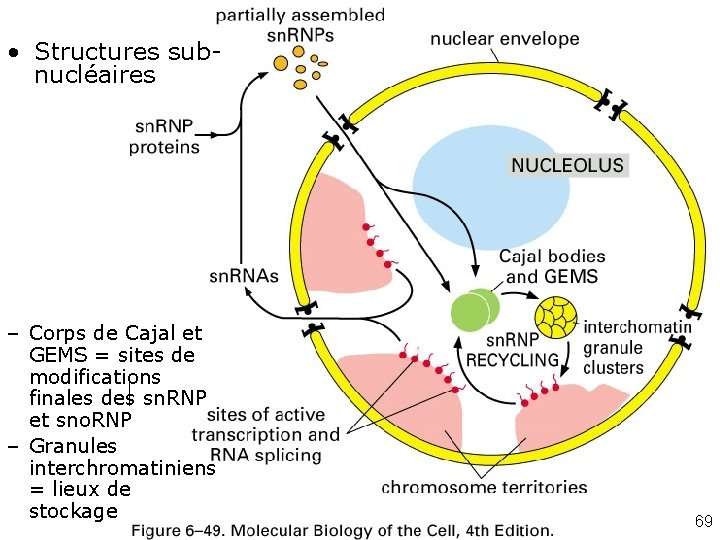
Corps de Cajal et GEMS (Gemini of Coiled bodies) • • Se ressemblent Marchent par deux dans le noyau Structures distinctes ? Site de maturation finale et d'assemblage des sn. ARN et sno. ARN ? • Les sn. RNP sont assemblées au début dans le cytosol mais elles sont transportées dans le noyau pour leur maturation finale • Lieu de recyclage des sn. RNP ? 65

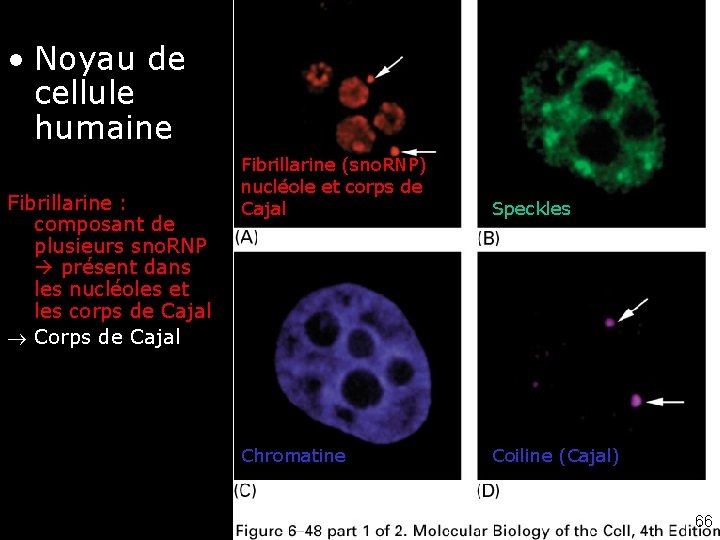
• Noyau de 6 -48 A-D Nuclears Fig cellule bodies humaine Fibrillarine : composant de plusieurs sno. RNP présent dans les nucléoles et les corps de Cajal Corps de Cajal Fibrillarine (sno. RNP) nucléole et corps de Cajal Speckles Chromatine Coiline (Cajal) 66

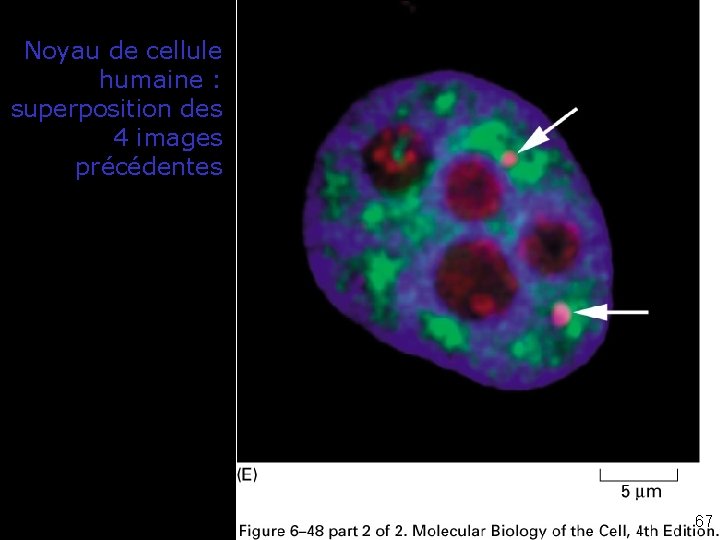
Noyau de cellule humaine : superposition des 4 images précédentes Fig 6 -48 E Nuclears bodies 67

Granules interchromatiniens • Stocks de sn. RNP matures prêtes à être utilisées pour l'épissage des pré-ARN 68

• Structures subnucléaires – Corps de Cajal et GEMS = sites de modifications finales des sn. RNP et sno. RNP – Granules interchromatiniens = lieux de stockage Fig 6 -49 69

Cycle des sn. ARN • • Synthèse initiale Exportation hors du noyau Maturation en 5' et 3' Assemblage avec les protéines des sn. RNP (protéines Sm) Réimportation dans le noyau Modifications finales dans les corps de Cajal Éventuellement (eg U 6 sn. RNP) modification par les sno. ARN dans le nucléole Les sites de transcription active et d'épissage correspondent aux "fibres périchromatiniennes" de la microscopie électronique 70

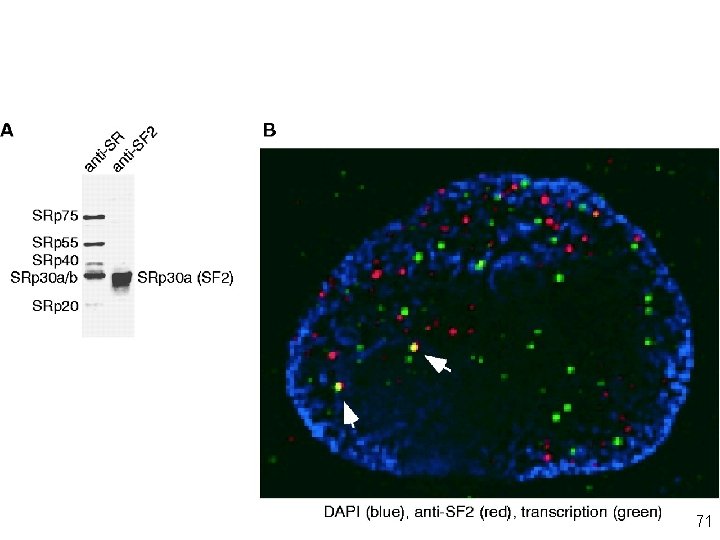
Like Attracts Like: Getting RNA Processing Together in the Nucleus Joe D. Lewis and David Tollervey DAPI (blue), anti-SF 2 (red), transcription (green) • Fig 1. SF 2 is concentrated at a subset of sites of active transcription. • (A) The anti-SF 2 monoclonal antibody (90) is specific for a single member (variously designated as SRp 30 a, ASF, or SF 2) of the family of SR proteins recognized by anti-SR. • (B) In immunostaining, anti-SF 2 decorates many particles distributed throughout the nucleoplasm (red channel). The green channel shows sites of bromouridine 59 -triphosphate (Br. UTP) incorporation. Arrowheads indicate prominent points of red and green coincidence, indicating that SF 2 is concentrated at these sites of active transcription. A single optical section of a He. La cell nucleus is shown, following deconvolution. [Figure generously provided by K. Neugebauer] Lewis, JD 2000(fig 1) Science 71

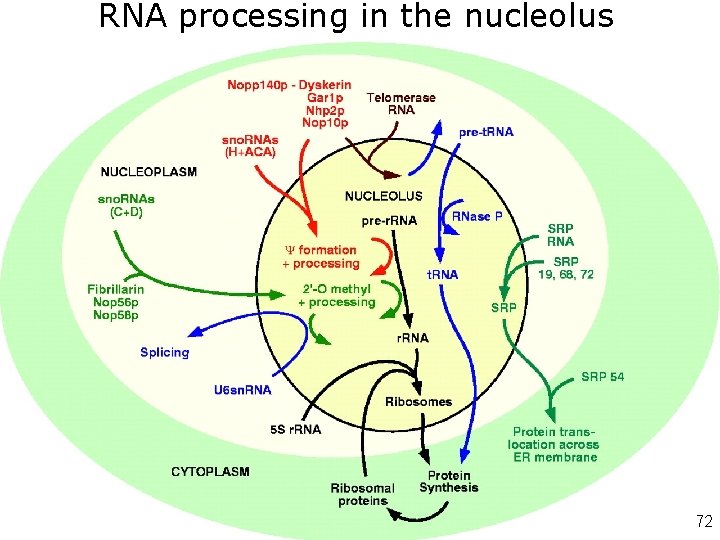
RNA processing in the nucleolus • • • Like Attracts Like: Getting RNA Processing Together in the Nucleus Joe D. Lewis and David Tollervey Fig. 2. RNA processing in the nucleolus. Within the nucleolus the pre-r. RNAs are processed to the mature r. RNAs by endonuclease cleavage and exonuclease digestion; see (32). During this processing, the r. RNAs assemble with the approximately 80 ribosomal proteins and undergo extensive covalent nucleotide modification. The box C 1 D class of sno. RNAs select sites of 29 -O-methylation (91, 92) and associate with three common proteins including Nop 1 p/fibrillarin, the putative r. RNA 29 -O-methylase (93). The box H 1 ACA class of sno. RNAs select sites of pseudouridine (C) formation (94, 95) and associate with four common proteins including Cbf 5 p/dyskerin/NAP 57 the probable C-synthase (96– 98). The sno. RNAs not only carry the modifying enzymes to the pre-r. RNA but, by specific base-pairing, create the enzyme recognition site. A small number of sno. RNAs of each class, including the U 3 sno. RNA, are required for processing of the pre-r. RNA, probably functioning in the structural reorganization of the pre- RNA. C formation and 29 -O-methylation of the U 6 sn. RNA is also nucleolar, and methylation is directed by box C 1 D sno. RNAs (99, 100). Moreover, there are “orphan” sno. RNAs that are predicted to select sites of RNA modification but for which no known target exits (101), suggesting that other RNA species, possibly including m. RNAs, are modified in the nucleolus and/or CBs (see Fig. 3). The RNA component of human telomerase is targeted to the nucleolus by a 39 domain that closely resembles the box H 1 ACA class of sno. RNAs (102, 103). Like other H 1 ACA sno. RNAs, human telomerase RNA is associated with dyskerin, mutations in which are associated with the hereditary disease dyskeratosis congenita and with reduced telomerase activity (102, 104). In contrast, the yeast telomerase RNA associates with the Sm-proteins characteristic of the spliceosomal sn. RNAs (105). Initial assembly of another RNA-protein complex, signal recognition particle (SRP) may also occur in the nucleolus. The SRP RNA and three of the six SRP proteins, SRP 19, SRP 68, and SRP 72, are detected in the nucleolus, but not the later assembling SRP 54 protein (106, 107). Another major class of RNA, pre-t. RNAs, are localized to the yeast nucleolus, together with the pre-t. RNA– processing enzyme RNase P (108). Human RNase P was similarly found to be localized in the nucleolus and also in coiled bodies (109), indicating that pre-t. RNA processing is a conserved nucleolar function. Lewis, JD 2000(fig 2) Science 72

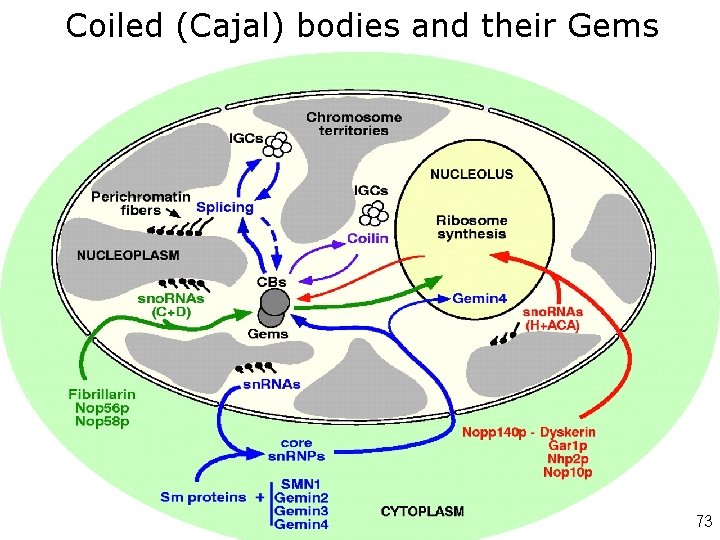
Coiled (Cajal) bodies and their Gems • • • Like Attracts Like: Getting RNA Processing Together in the Nucleus Joe D. Lewis and David Tollervey Fig. 3. Coiled (Cajal) bodies and their Gems. In vertebrates, the newly synthesized sn. RNAs are exported to the cytoplasm where they assemble with the seven common Sm proteins and undergo 39 processing and cap- rimethylation to form core sn. RNPs that are reimported into the nucleus. Microinjected sn. RNAs (110, 111) and transiently expressed Sm proteins (59) are observed in CBs before their appearance in other nucleoplasmic regions. In contrast, mature sn. RNPs do not initially localize to CBs on nuclear reentry after mitosis, suggesting that late sn. RNP maturation and assembly steps take place in the CBs; for example, covalent modification of the sn. RNAs by C formation and 29 -O-methylation and association with the large numbers of species-specific sn. RNP proteins. In the cytoplasm, the Sm proteins are associated with the SMN complex, which includes oligomers of the SMN protein (survival of motor neurons), together with Gemin 2, Gemin 4, and a putative DEAD-box RNA helicase, Gemin 3 (112– 116). Each of these proteins also concentrates in nuclear structures called Gems (Gemini of coiled bodies) (117). In many cell types, Gems show partial or complete coincidence with CBs (118), and their functional distinction is unclear. This suggests that newly synthesized sn. RNPs are escorted to the nucleus by the SMN complex. This complex may also function in recycling sn. RNPs following splicing (119). Yeast lacks both morphological CBs and an obvious coilin homolog, but does have a Gemin 2 homolog, Brr 1 p. Like Gemin 2, Brr 1 p interacts with Sm proteins and brr 1 mutations inhibit sn. RNA 39 processing, suggesting that this pathway is conserved (113, 120). CBs and SMN are also implicated in sno. RNP synthesis. Microinjected box C 1 D sno. RNAs localize transiently to CBs before appearing in the nucleoli (103, 111), and mutant sno. RNAs that lack an intact box C 1 D region (the likely protein-binding sites) are retained in the CBs (111). In contrast, the H 1 ACA sno. RNAs, and the associated Nopp 140 p protein, accumulate first in nucleoli and then in CBs (121). Multiple interactions between sno. RNAassociated proteins, coilin, and SMN have been observed (117, 73 121, 122), and Gemin 4, at least, is also associated with nucleoli (116). Lewis, JD 2000(fig 3) Science

Application • GEMS contient la protéine SMN (Survival of Motor Neurons) • Certaines mutations du gène qui code pour cette protéine entraînent une atrophie musculaire spinale • Défaut de l'assemblage de sn. RNP donc de l'épissage du pré-ARNm 74

Organisation architecturale du noyau • La maturation de l'ARN est très importante on s'attend à une localisation spécifique de l’épissage des pré-messagers MAIS… • … l'épissage est co-transcriptionnel • … et la transcription se fait sur les chromosomes qui sont répartis dans tout le noyau… • MAIS… • Les chromosomes peuvent se déplacer… 75

Organisation architecturale du noyau • Les zones silencieuses transcriptionnellement sont localisées contre l'enveloppe nucléaire (beaucoup d'hétérochromatine) • Ces mêmes régions se déplacent dans le noyau pour s'exprimer • Il n'y aurait quelques milliers de sites d'expression pour transcrire 15000 gènes dans le noyau 76

Organisation architecturale du noyau • • Sites dynamiques Transcription et épissage Lignes d'assemblage Factories 77