DNA Cloning Primers universali sequenza di ogni DNA


















- Slides: 51

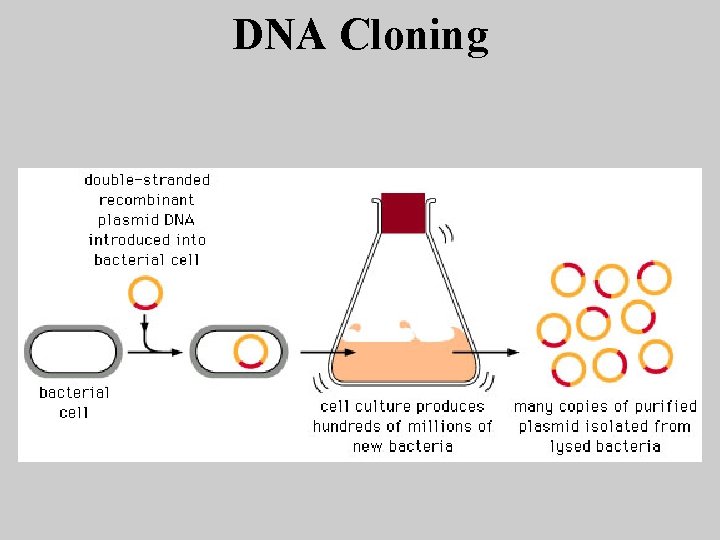
DNA Cloning

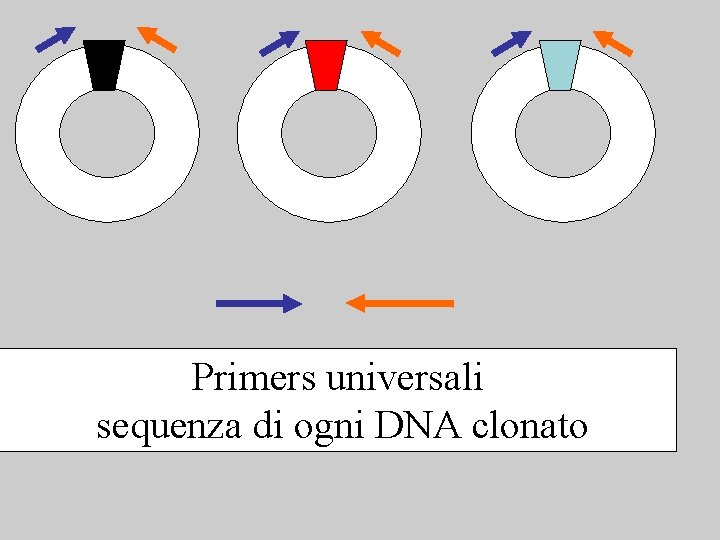
Primers universali sequenza di ogni DNA clonato

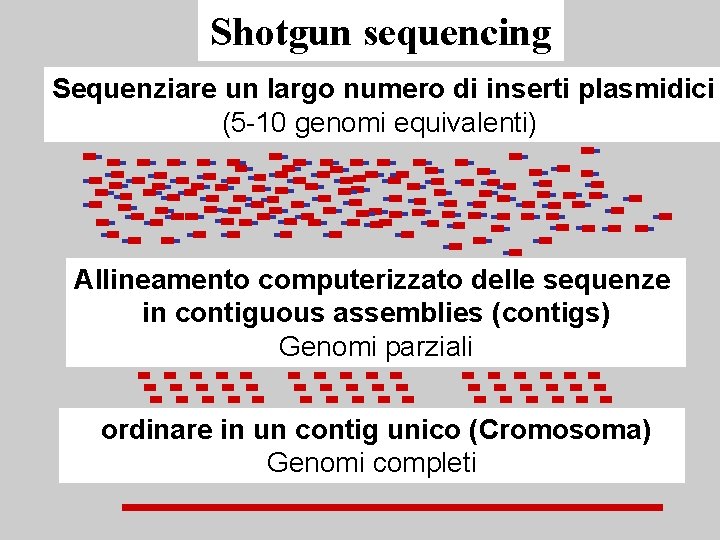
Shotgun sequencing Sequenziare un largo numero di inserti plasmidici (5 -10 genomi equivalenti) Allineamento computerizzato delle sequenze in contiguous assemblies (contigs) Genomi parziali ordinare in un contig unico (Cromosoma) Genomi completi

Second generation sequencing No clonal amplification Massively Parallel Sequencing sequencing ~Gb per day

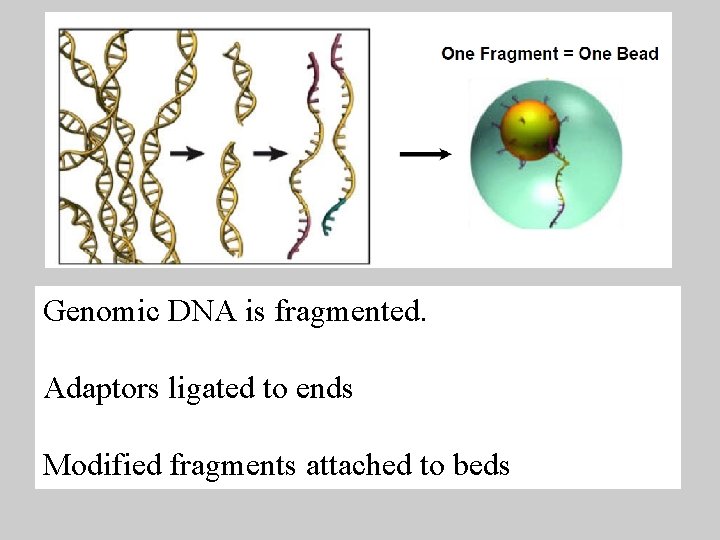
Genomic DNA is fragmented. Adaptors ligated to ends Modified fragments attached to beds

Amplification on beds

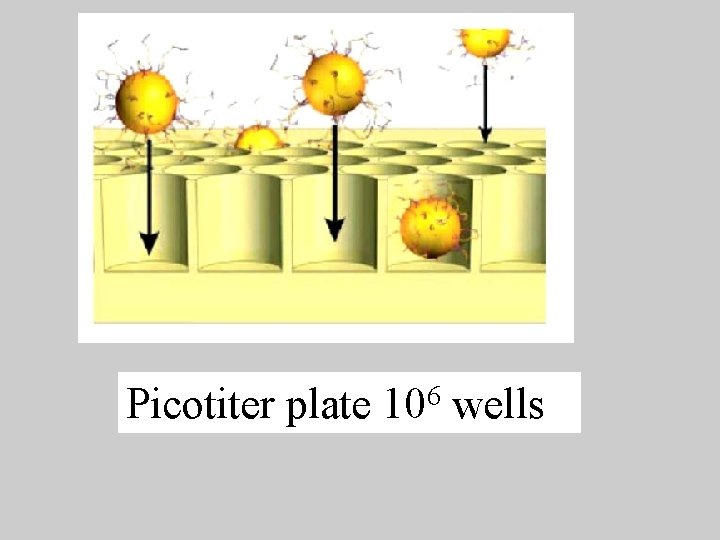
Picotiter plate 106 wells

Each bead is added to a fibre-optic well

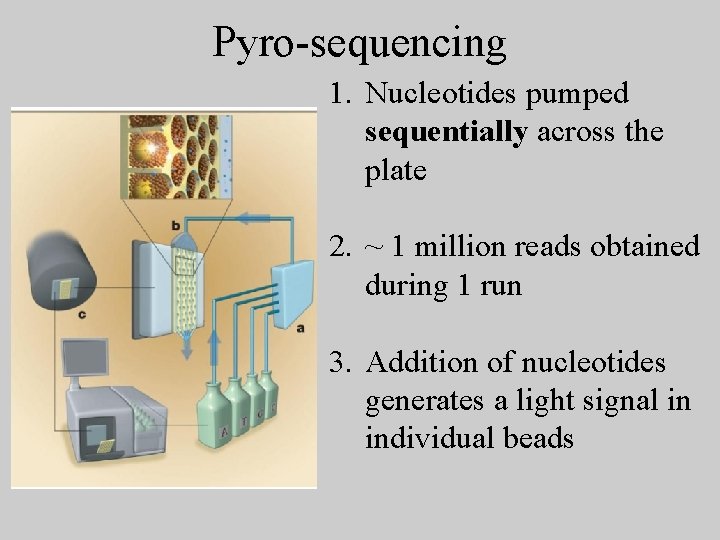
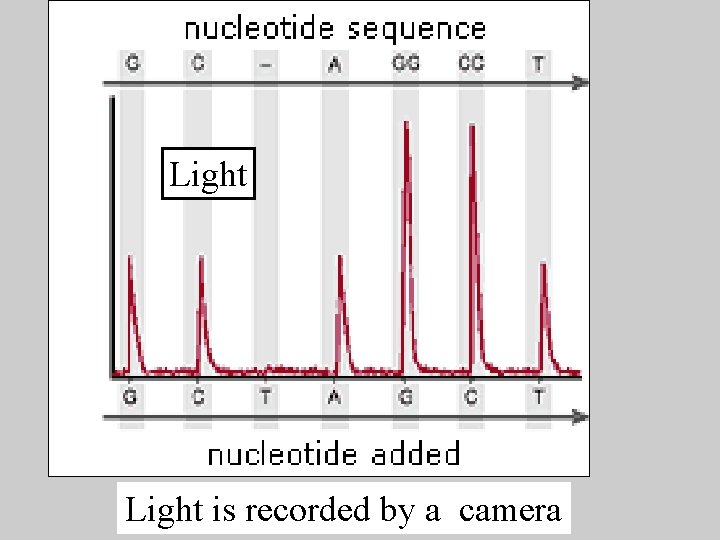
Pyro-sequencing 1. Nucleotides pumped sequentially across the plate 2. ~ 1 million reads obtained during 1 run 3. Addition of nucleotides generates a light signal in individual beads

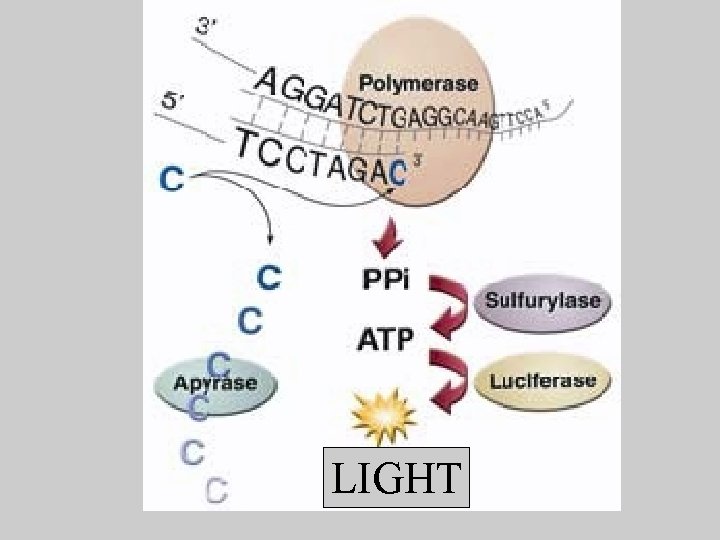
d. ATP, d. CTP, d. GTP, d. TTP are sequentially added and removed. Incorporation releases pyrophosphate (PPi)

LIGHT

Light is recorded by a camera

Informazioni derivate dal sequenziamento identificazione di tutti i potenziali prodotti genici

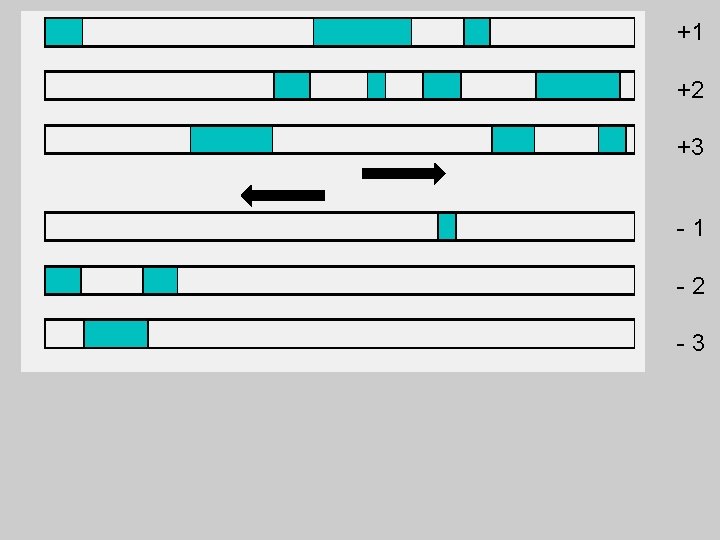
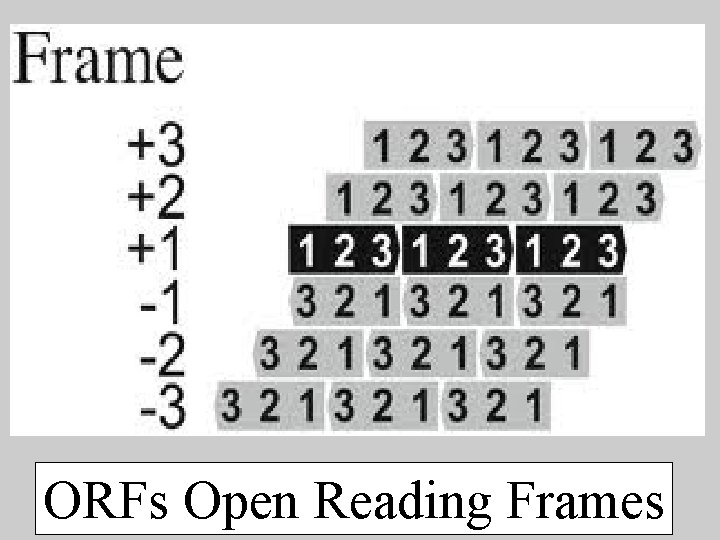
+1 +2 +3 -1 -2 -3

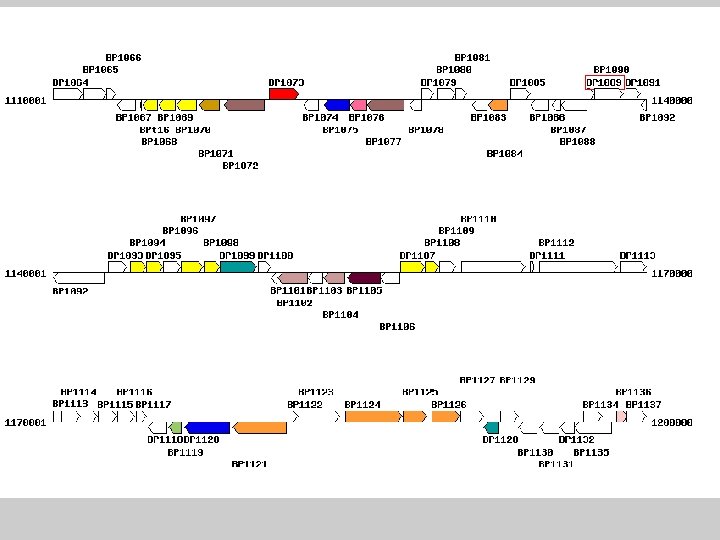
ORFs Open Reading Frames

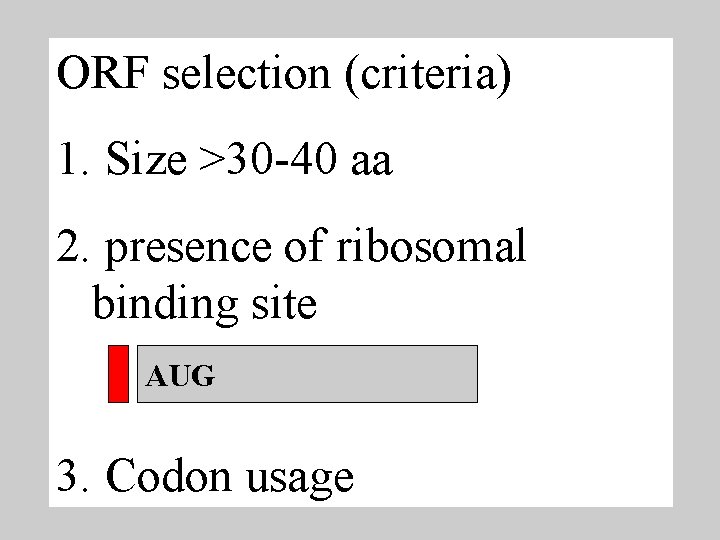
ORF selection (criteria) 1. Size >30 -40 aa 2. presence of ribosomal binding site AUG 3. Codon usage


Ricerca di omologie (sequenze codogeniche conservate in altre specie) Ricostruzione metabolica


Hypothetical proteins 20 -30% of total ORFs Real ? Functional?

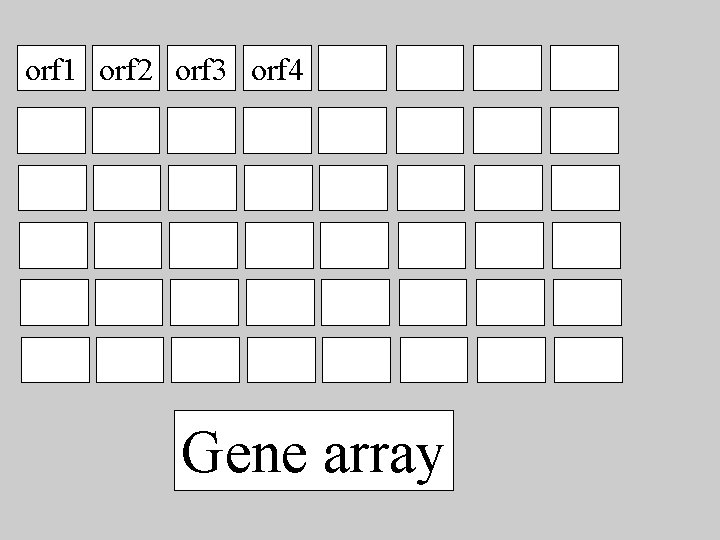
orf 1 orf 2 orf 3 orf 4 orf 1 orf 1 orf 1 orf 1 orf 1 orf 1 orf 1 orf 1 orf 1 orf 1 orf 1 Gene array

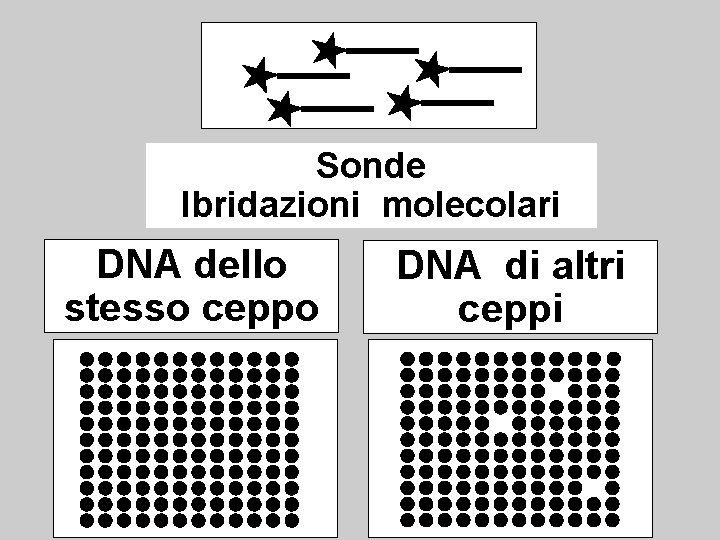
Sonde Ibridazioni molecolari DNA dello stesso ceppo DNA di altri ceppi

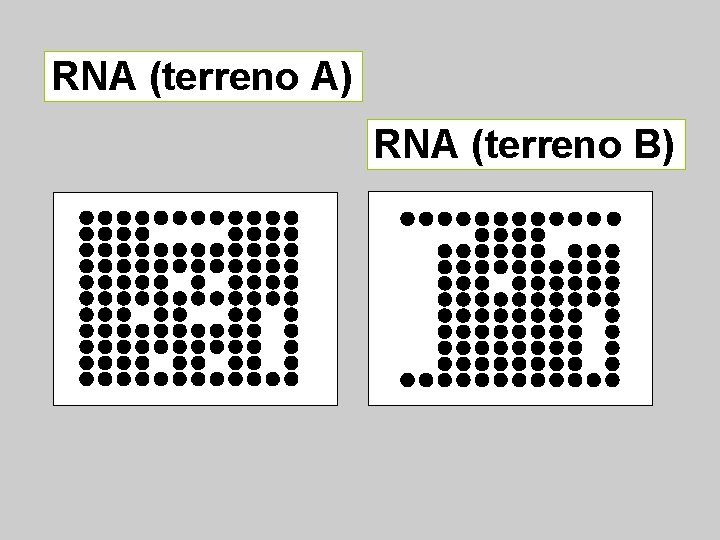
RNA (terreno A) RNA (terreno B)

Un segmento di DNA potrebbe essere Trascritto, ma non tradotto in proteina

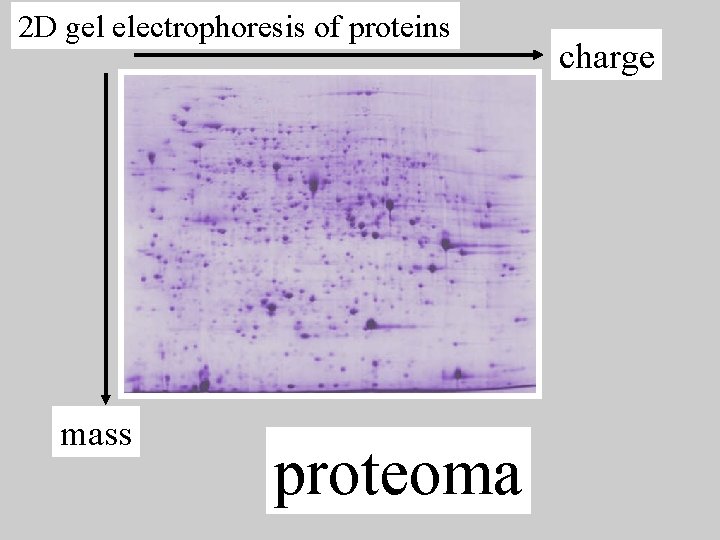
2 D gel electrophoresis of proteins mass proteoma charge

Un gene batterico potrebbe essere trascritto (e/o tradotto) solo durante una infezione (in vivo), ma non in Condizioni di crescita di laboratorio (in vitro)

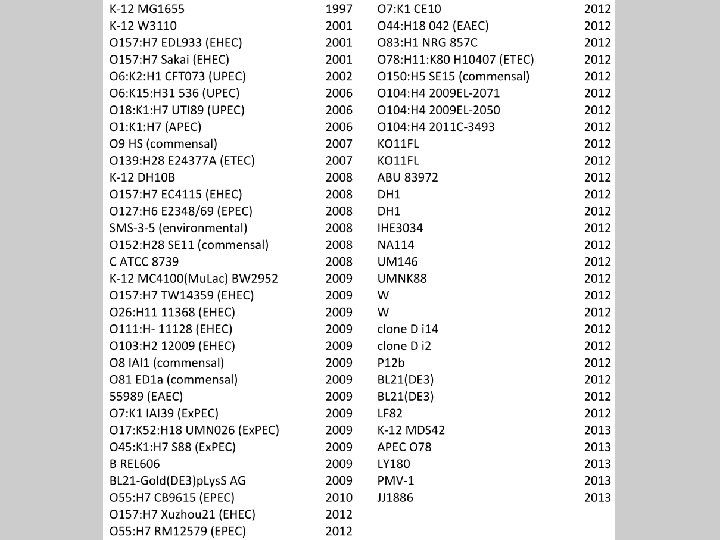
Genoma del ceppo A Genoma di un secondo ceppo B Genoma di un terzo ceppo C A=B=C ? pangenoma

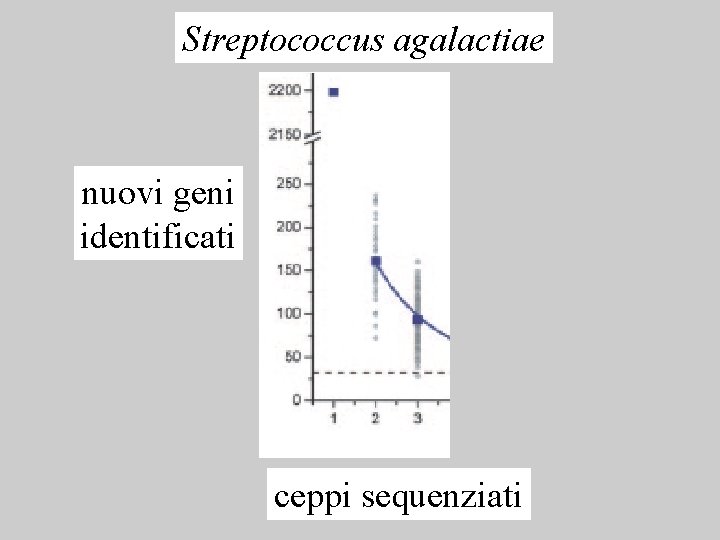
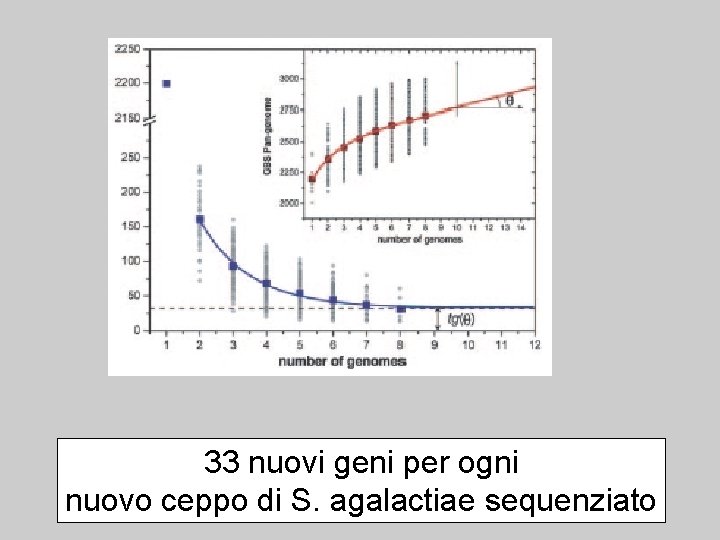
Streptococcus agalactiae nuovi geni identificati ceppi sequenziati

33 nuovi geni per ogni nuovo ceppo di S. agalactiae sequenziato

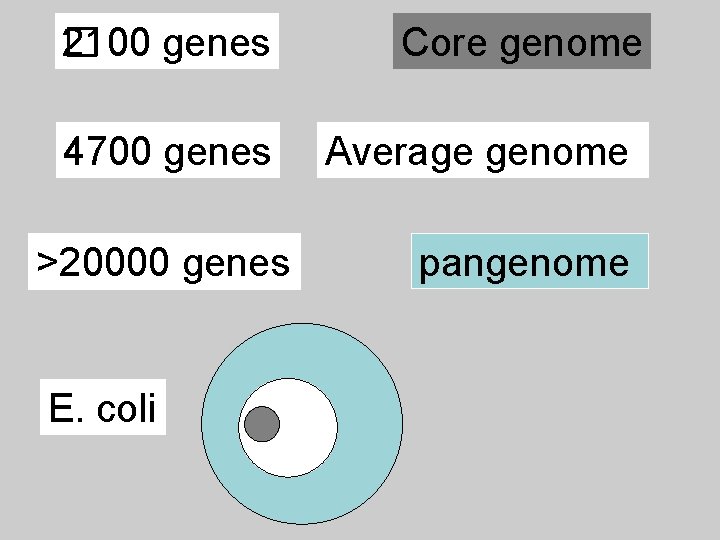
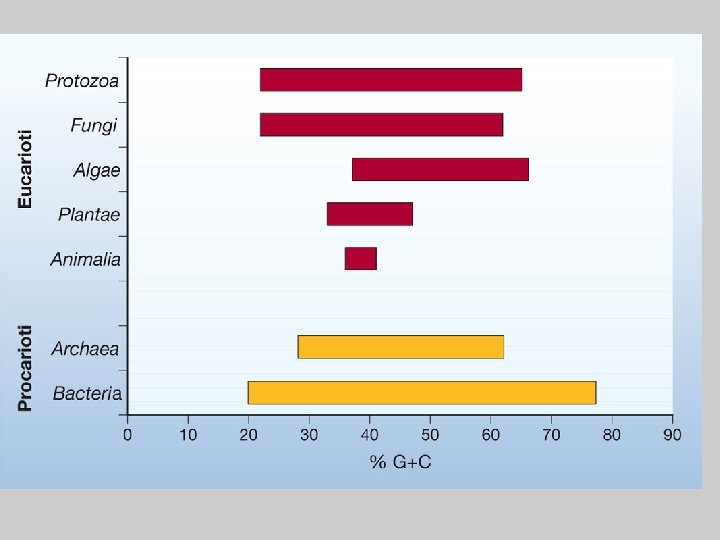
Pangenoma: insieme di geni Identificati in individui della stessa specie Core genome Variable genome


2100 genes � 4700 genes >20000 genes E. coli Core genome Average genome pangenome

Pangenomi chiusi Specie che vivono in nicchie isolate o hanno scarsa capacità di scambio genetico Bacillus anthracis pangenoma definito da 4 soli genomi

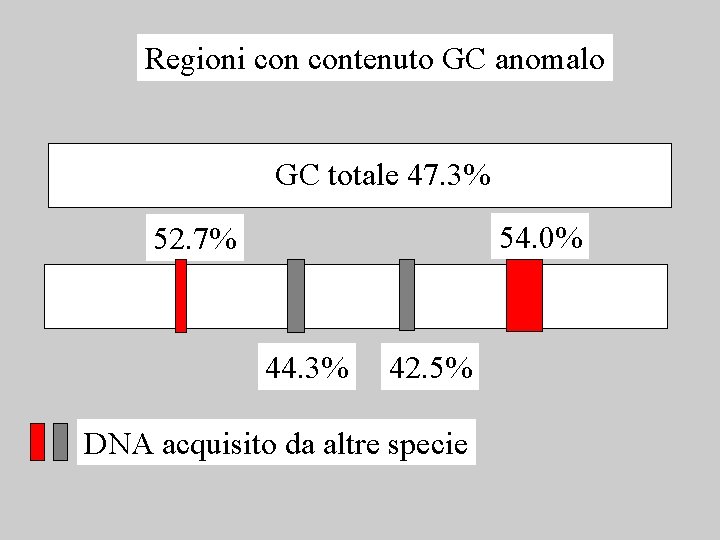
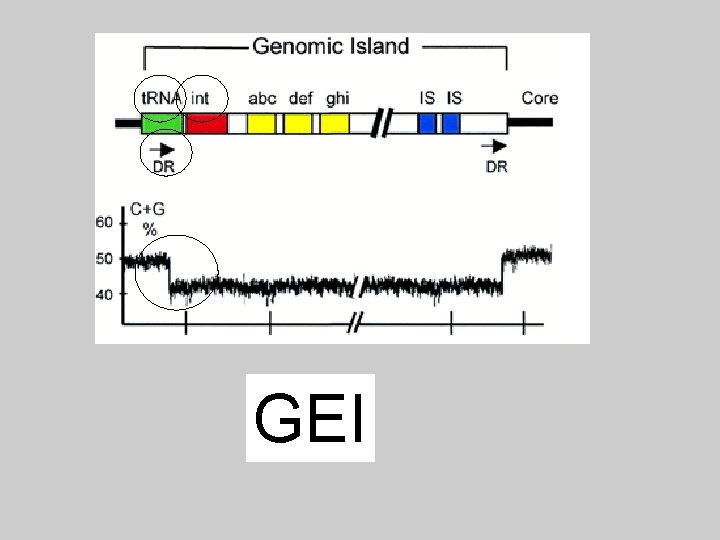
Regioni contenuto GC anomalo GC totale 47. 3% 54. 0% 52. 7% 44. 3% 42. 5% DNA acquisito da altre specie


GENOMIC ISLANDS (GEIs) 20 -120 kb 7 -20 kb Island Islet

GEI

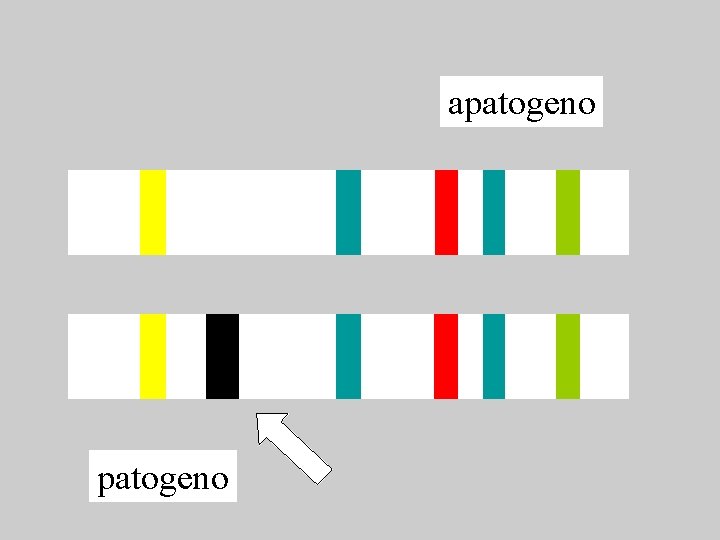
apatogeno

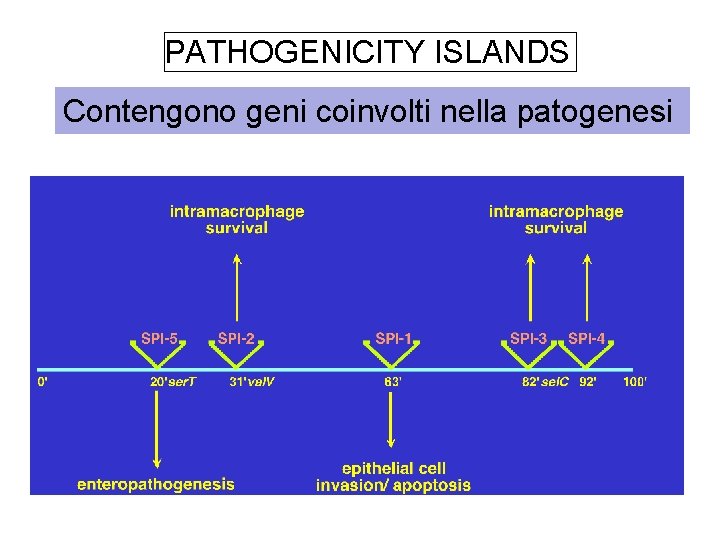
PATHOGENICITY ISLANDS Contengono geni coinvolti nella patogenesi



Microbiological assays

A species includes strains: pathogenic apathogenic virulent hypervirulent Strain isolated microbe cultivated in the lab Isolate microbe from a patient, plant, etc

How to distinguish species strains? Serological analyses Changes in the composition of surface proteins and sugars O 157 H 7, O 151 H 4 etc. Different O antigens (PS) and Flagella

Genomic analyses a) restriction enzymes b) PCR and sequencing

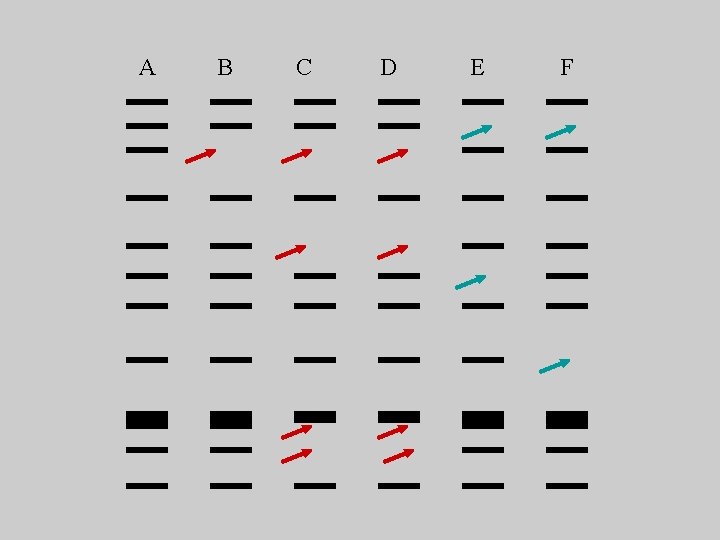
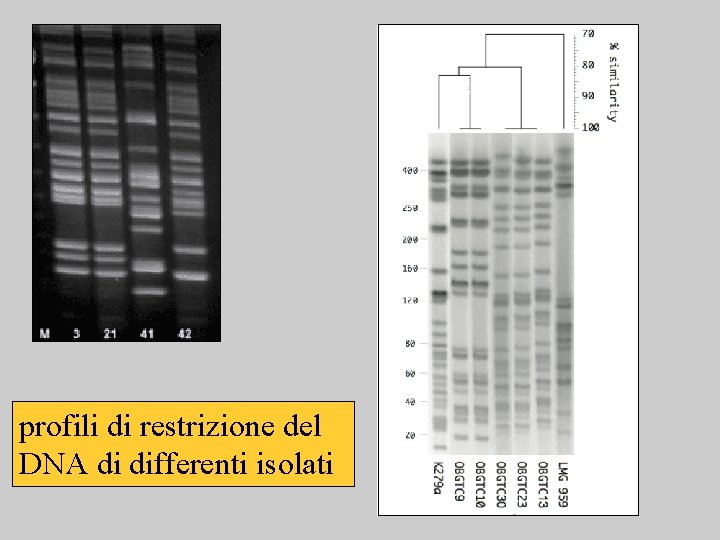
Digestione dell’intero genoma di un isolato analisi elettroforetica dei prodotti PFGE Pulse Field Gel Electrophoresis

A B C D E F

profili di restrizione del DNA di differenti isolati

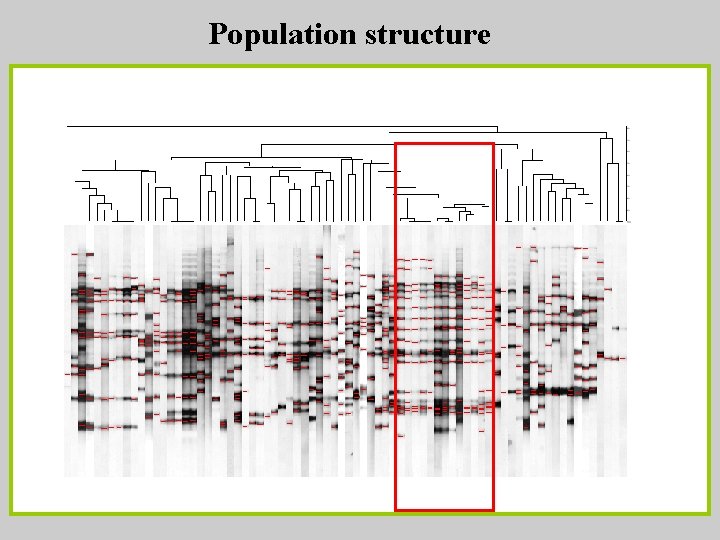
Population structure

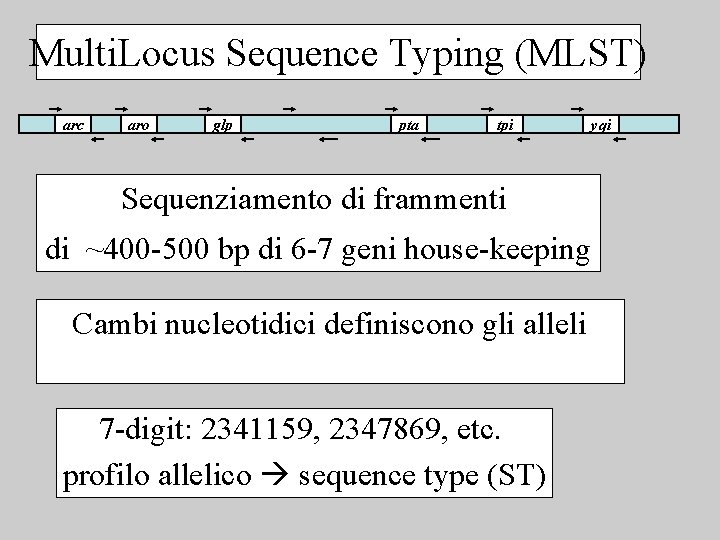
Multi. Locus Sequence Typing (MLST) arc aro glp pta tpi Sequenziamento di frammenti di ~400 -500 bp di 6 -7 geni house-keeping Cambi nucleotidici definiscono gli alleli 7 -digit: 2341159, 2347869, etc. profilo allelico sequence type (ST) yqi

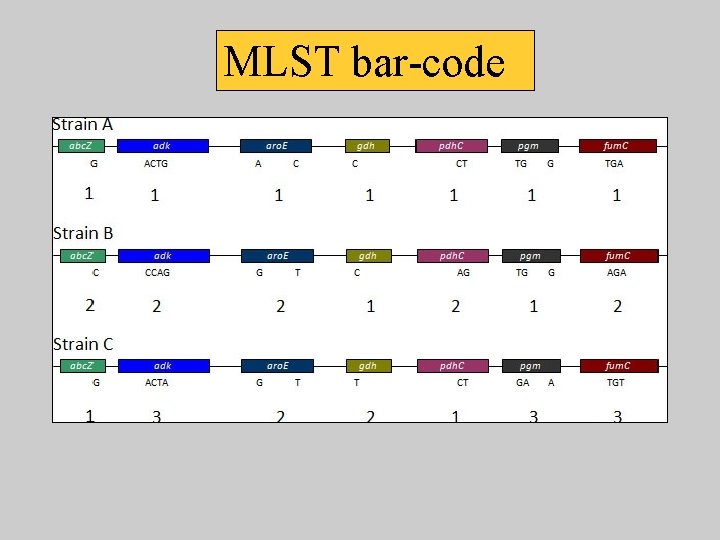
MLST bar-code
 Minipcr antibiotic resistance lab
Minipcr antibiotic resistance lab Question 7
Question 7 Dna primers
Dna primers Il declino dei poteri universali mappa concettuale
Il declino dei poteri universali mappa concettuale Disputa sugli universali mappa concettuale
Disputa sugli universali mappa concettuale Ricevitori universali asincroni
Ricevitori universali asincroni Dimeros de primers
Dimeros de primers Miquel blay els primers freds
Miquel blay els primers freds Diseño de primers degenerados
Diseño de primers degenerados Temperatura de melting primers
Temperatura de melting primers Els primers freds
Els primers freds Nombres enters i naturals
Nombres enters i naturals Homoplasmia
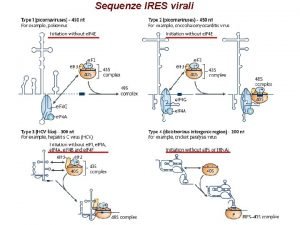
Homoplasmia Sequenze ires
Sequenze ires Sequenze di pierino e il lupo
Sequenze di pierino e il lupo Contatore johnson
Contatore johnson Fasta e blast
Fasta e blast Ananas fibonacci
Ananas fibonacci Spirito di dio vieni
Spirito di dio vieni Algoritmo fasta
Algoritmo fasta Sequenza ordinata
Sequenza ordinata Ran gef and ran gap
Ran gef and ran gap Progressione geometrica e aritmetica
Progressione geometrica e aritmetica Pierino e il lupo disegno
Pierino e il lupo disegno Sequenza segnale kdel
Sequenza segnale kdel Sequenza fibonacci
Sequenza fibonacci Riconoscitore di sequenza
Riconoscitore di sequenza Timor di dio dono dello spirito santo
Timor di dio dono dello spirito santo Therpeutic cloning
Therpeutic cloning Advantages of cloning
Advantages of cloning Cloning and sequencing explorer series
Cloning and sequencing explorer series Directional topo cloning
Directional topo cloning Cloning pros and cons
Cloning pros and cons Process of cloning
Process of cloning Advantages and disadvantages of cloning
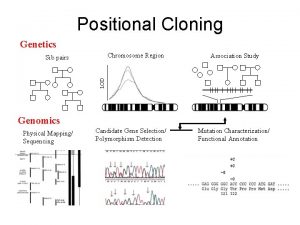
Advantages and disadvantages of cloning Positional cloning
Positional cloning Gene cloning
Gene cloning Reproductive cloning
Reproductive cloning Cloning tasmanian tiger
Cloning tasmanian tiger Reproductive vs therapeutic cloning
Reproductive vs therapeutic cloning क्लोनिंग वेक्टर
क्लोनिंग वेक्टर What is cloning
What is cloning Steps of gene cloning
Steps of gene cloning Pixpsd
Pixpsd Identity cloning and concealment
Identity cloning and concealment Clonaid
Clonaid Inverse pcr
Inverse pcr Suspension cloning
Suspension cloning Topo ta cloning
Topo ta cloning Cloning vector
Cloning vector Advantages and disadvantages of cloning
Advantages and disadvantages of cloning Cloning in nature
Cloning in nature