Zrnicowanie DNA a zmienno cech ilociowych u odmian







![Analiza COP: wyniki (1) Rys 1. Zestawienie odmian o największym wpływie [%] na rodowody Analiza COP: wyniki (1) Rys 1. Zestawienie odmian o największym wpływie [%] na rodowody](https://slidetodoc.com/presentation_image_h2/443d8c823b22580529c5dd464ae0d638/image-8.jpg)







- Slides: 29

Zróżnicowanie DNA a zmienność cech ilościowych u odmian pszenicy zwyczajnej Mirosław Tyrka Pracownia Genetyki Populacji Akademia Polonijna w Częstochowie

Dzisiejsze wyzwania dla hodowli roślin n n wzrost produktywności odmian rozszerzenie areału upraw wytworzenie odmian mieszańcowych manipulacja genami odporności na patogeny i szkodniki poprawa jakości żywności

Po co nam markery molekularne? Wykorzystanie praktyczne: n n Wspomaganie procesu selekcji w hodowli, czyli MAS Identyfikacja odmian – prawa autorskie, obrót materiałem siewnym Wykorzystanie w badaniach: n n mapowanie genetyczne cech jakościowych i ilościowych klonowanie genów

MAS Selekcja wspomagana markerami Marker Assisted Selection Selekcja w oparciu o markery DNA • zwykle niezależna od: stadium wzrostu rośliny i czynników środowiskowych • zalety: wczesna selekcja, szybkie nagromadzanie pożądanej cechy, nie niszczy testowanego materiału • skrócenie czasu wytworzenia nowej odmiany Selekcja genu(ów) W celu oznaczenia obecności wprowadzonego genu stosowana kiedy bezpośredni test fenotypowy nie jest możliwy, zbyt kosztowny lub stosowany w późnym stadium rozwojowym Selekcja tła W celu przyspieszenia powrotu do rodzica wypierającego Doświadczalnie stwierdzono wysoką skuteczność markerów molekularnych do selekcji tła w programach wypierających

Cel badań Ocena zmienności w grupie odmian pszenicy ozimej uwzględniającej odmiany lokalne przystosowane do polskich warunków klimatycznych wg: pochodzenia (COP) n fenotypu n DNA (Pst. I-AFLP, Alw 44 I-AFLP, SSR, STS) n Ustalenie wzajemnych zależności pomiędzy ww. miarami podobieństwa Wyselekcjonowanie oznaczeń DNA, do zastosowania w ocenie zmienności genetycznej materiału pod kątem wykorzystania w hodowli

Materiał badawczy 82 odmiany pszenicy zwyczajnej: Alba, Al'bidum-114, Alcedo, Almari, Antonińska Wczesna, Arda , Aria, Asta, Aurora, Balta, Begra, Beta, Biała Kaszubska, Biały Krzyż, Bogatka, Bożena, Choryńska, Dana, Dańkowska Graniatka, Dnestrowskaja 25, Emika, Gama, Grana, Halina, Holme, Iduna, Jasnocha Włoszanowska, Jawa, Kaja, Kamila, Kaukaz, Komorowska, Konstancja Granum, Konstancja Wierzbieńska, Korweta, Krupnokolosaja, Lama, Lanca, Leszczyńska Wczesna, Litwinka, Liwilla, Luna, Lutescens 17, Lutescens 31, Lutescens 38/72, Malwa, Maris. Huntsman, Mironowskaja 808, Mironowskaja 25, Mobela, Modra, Muras, Niewylegajaca, Nike, Niwa, Oda, Olza, Panda, Parada, Płocka, Polanka, Puławska Wczesna, Rada, Rosa, Rota, Rysa, Saga, Salwa, Severokubanskaja-43, Sobieszyńska-44, Solid, Stepnaya-40, Stieglera-22, Strzelecka, Svaloefs-0987, Wanda, Weneda, Wysokolitewka-Oltarzewska, Wysokolitewka. Sztywnosloma, Zeta

Analiza COP: metody Nazwa 1) Alcedo 2) Almari 3) Antonińska Wczesna 4) Arda … 78) Wanda 79) Weneda 80) Wysokolitewka-Oltarz 81) Wysokolitewka-Sztyw 82) Zeta Pochodzenie Record/Poros//Carsten-ROEM. 3 Maris-Huntsman/Alcedo Inversable/Wysokolitewka Sztyw Maris Huntsman/C-474/73 … N-320/Maris Huntsman//Lanca Kaukaz/Tetrix//C-1073 -67 sel. Wysokolitewka Wielki Książe Saski/Ostka Mikulicka Mironowskaja 808//Herold/Grana VURV http: //genbank. vurv. cz/wheat/pedigree/ Kraj DEU POL POL. . . POL POL POL Rok 1978 1989 1959 1990. . . 1997 1984 1961 1979
![Analiza COP wyniki 1 Rys 1 Zestawienie odmian o największym wpływie na rodowody Analiza COP: wyniki (1) Rys 1. Zestawienie odmian o największym wpływie [%] na rodowody](https://slidetodoc.com/presentation_image_h2/443d8c823b22580529c5dd464ae0d638/image-8.jpg)
Analiza COP: wyniki (1) Rys 1. Zestawienie odmian o największym wpływie [%] na rodowody analizowanych genotypów pszenicy

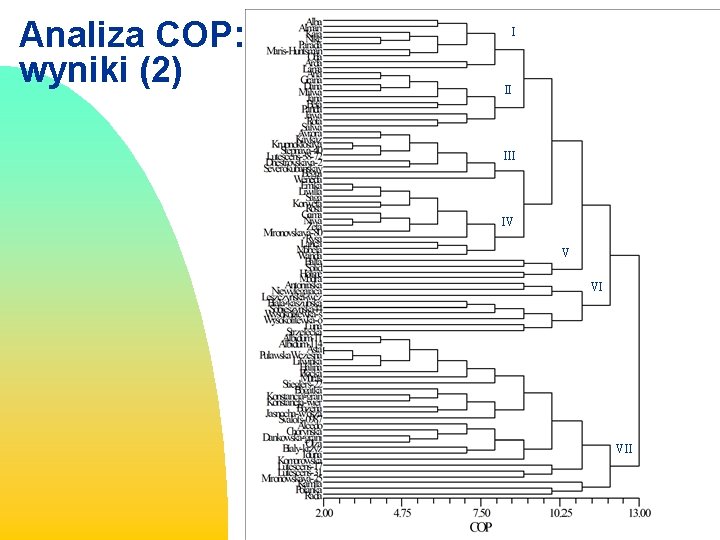
Analiza COP: wyniki (2) I II IV V VI VII

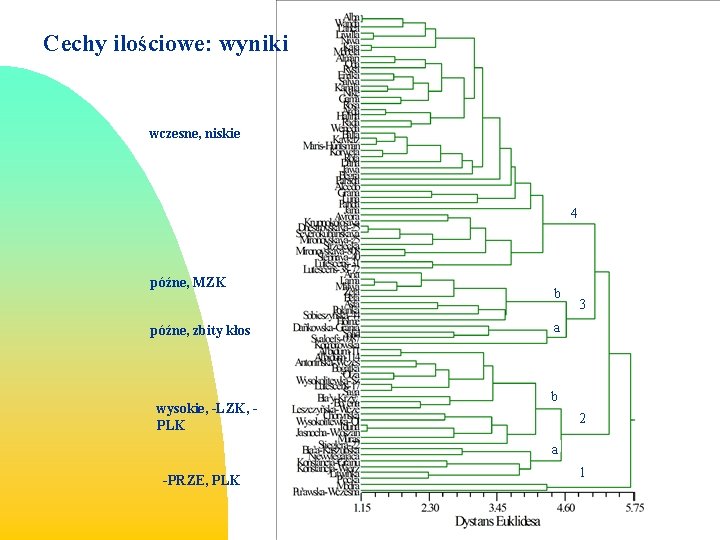
Cechy ilościowe: metodyka Badane cechy: 1) przezimowanie (PRZ). 2) daty pełni kłoszenia (KLO) 3) daty pełni kwitnienia (KWI). 4) wysokość roślin w cm (WR), 5) długość kłosa w cm (DLK), 6) liczbę kłosków w kłosie (LKK), 7) liczbę ziarniaków w kłosie (LZK) 8) masę ziarniaków z kłosa w g (MZK). 9) płodność kłoska, (PLK), 10) masę 1000 ziarniaków (MTZ), 11) zbitość kłosa, (ZK) 12) zawartości białka ogólnego (BIA) analiza wariancji, analiza skupień na bazie odległości Euklidesa

Cechy ilościowe: wyniki wczesne, niskie 4 późne, MZK późne, zbity kłos wysokie, -LZK, PLK b 3 a b 2 a -PRZE, PLK 1

METODYKA: ANALIZY DNA Izolacja DNA metodą Milligan’a (1992) Pst. IAFLP i Alw 44 IAFLP Trawienie enzymem Pst. I lub Alw 44 I i odpowiednio ligacja adaptorów Amplifikacja nieselektywna Amplifikacja selektywna 30 starterów selekcyjnych Pst. IAFLP i 30 dla Alw 44 IAFLP Rozdział na agarozie SSR 15 starterów i rozdział na żelu sekwencyjnym STS 28 starterów opcjonalnie trawienie

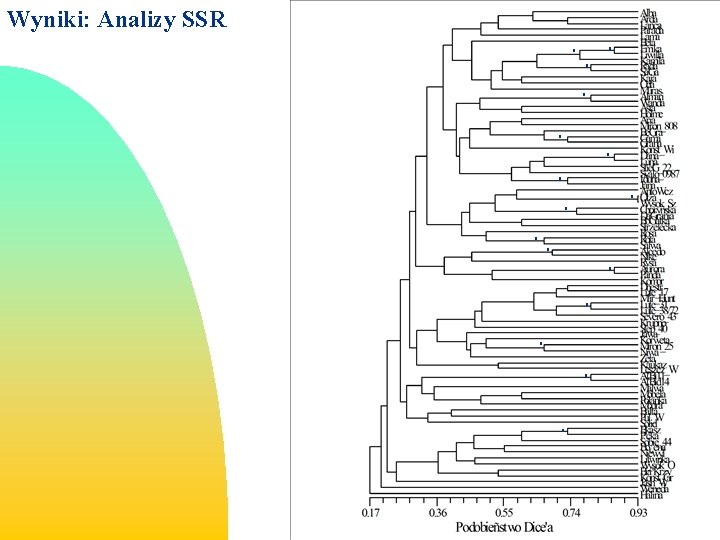
METODYKA: ANALIZY DNA Analiza wyników: 1. Matryca 0 – 1 2. Podobieństwa genetyczne Dice’a F=2 NXY/(Nx +NY), 3. Grupowanie metodą UPGMA Alba Pst 28_2 Alba 1 1, 000 Al 10 -9 Al'bid 11 0 0, 901 gwm 413_1120 Al'bi 114 0, 899 gwm 400_1540 Alcedo 0, 930 D 22_M_1400 0, 940 Ant-Wczes' 0 Pst 12_4 Arda 1 0, 949 …. . . Al'bid 11 0 1 1, 000 0 0, 946 0 0, 914 0 0, 907 0 0, 891. . . Al'bi 114 0 1, 000 0 0, 906 0 0, 899 0 0, 895. . . Alcedo 1 0 0 0 1, 000 0 0, 925 0 0, 910. . . Ant-Wczes'Arda 1 1 1 0 0 0 1, 000 0 1 0, 926 1 1, 000. . Aria 1. . . 0. . . Asta 1 0 0 0 1 1. . .

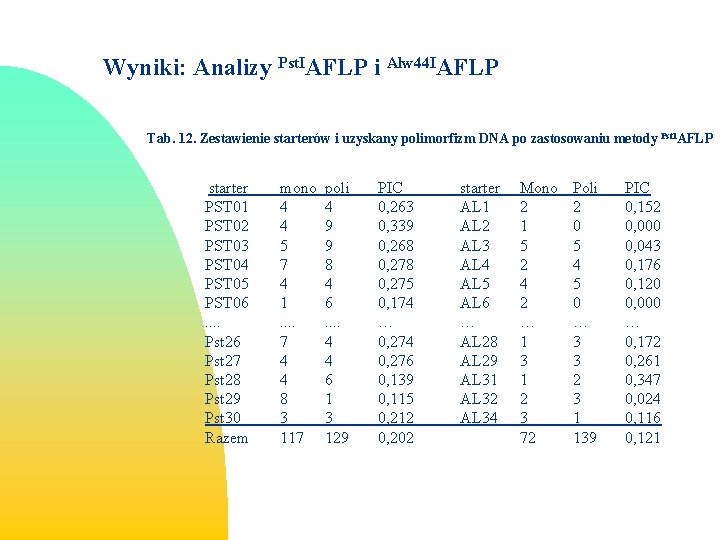
Wyniki: Analizy Pst. IAFLP i Alw 44 IAFLP Tab. 12. Zestawienie starterów i uzyskany polimorfizm DNA po zastosowaniu metody Pst. IAFLP starter PST 01 PST 02 PST 03 PST 04 PST 05 PST 06. . Pst 26 Pst 27 Pst 28 Pst 29 Pst 30 Razem mono 4 4 5 7 4 1. . 7 4 4 8 3 117 poli 4 9 9 8 4 6. . 4 4 6 1 3 129 PIC 0, 263 0, 339 0, 268 0, 275 0, 174 … 0, 274 0, 276 0, 139 0, 115 0, 212 0, 202 starter AL 1 AL 2 AL 3 AL 4 AL 5 AL 6 … AL 28 AL 29 AL 31 AL 32 AL 34 Mono 2 1 5 2 4 2 … 1 3 1 2 3 72 Poli 2 0 5 4 5 0 … 3 3 2 3 1 139 PIC 0, 152 0, 000 0, 043 0, 176 0, 120 0, 000 … 0, 172 0, 261 0, 347 0, 024 0, 116 0, 121

Wyniki: Analizy Pst. IAFLP i Alw 44 IAFLP Fot. 1 Polimorfizm DNA dla 82 odmian uzyskany po amplifikacji selektywnej ze starterem Pst 15.

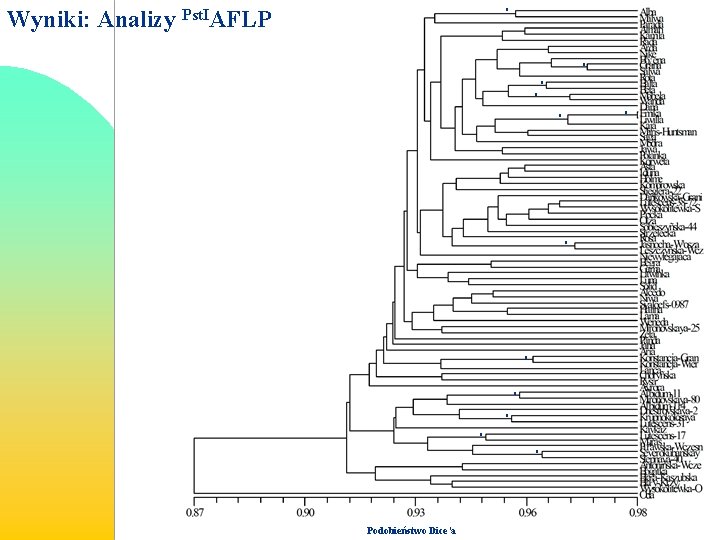
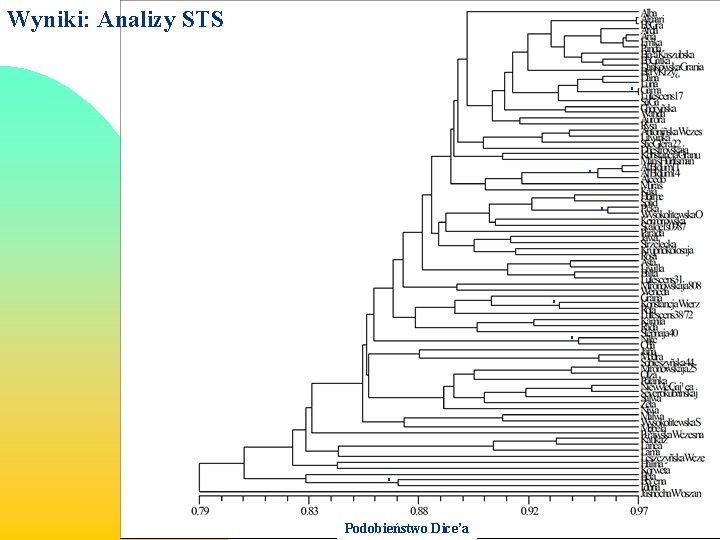
Wyniki: Analizy Pst. IAFLP * * * Podobieństwo Dice’a

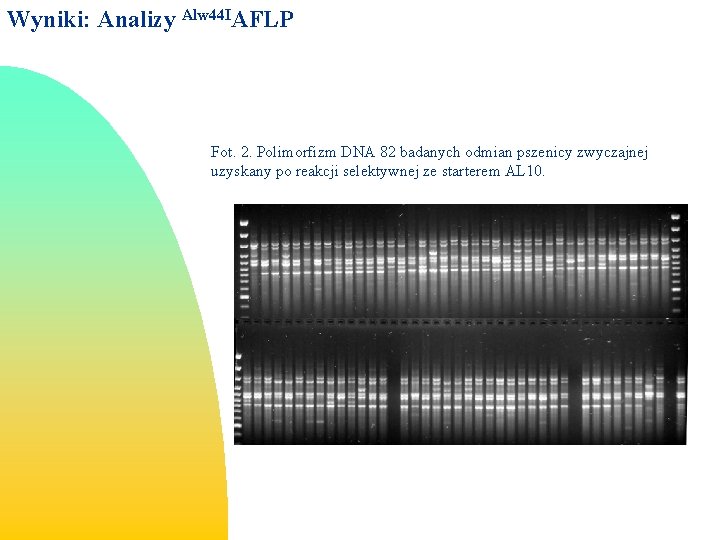
Wyniki: Analizy Alw 44 IAFLP Fot. 2. Polimorfizm DNA 82 badanych odmian pszenicy zwyczajnej uzyskany po reakcji selektywnej ze starterem AL 10.

Wyniki: Analizy Alw 44 IAFLP Fot. 3. Polimorfizm DNA 82 badanych odmian pszenicy zwyczajnej uzyskany po reakcji selektywnej ze starterem AL 16.

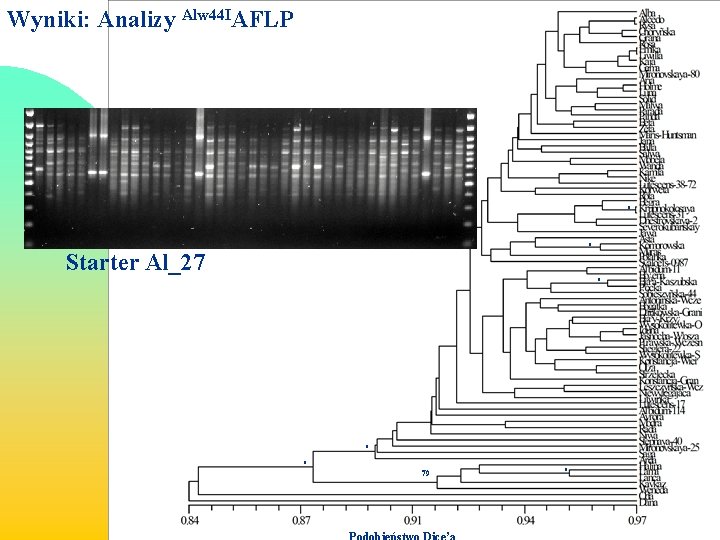
Wyniki: Analizy Alw 44 IAFLP * * Starter Al_27 * * * 79 *

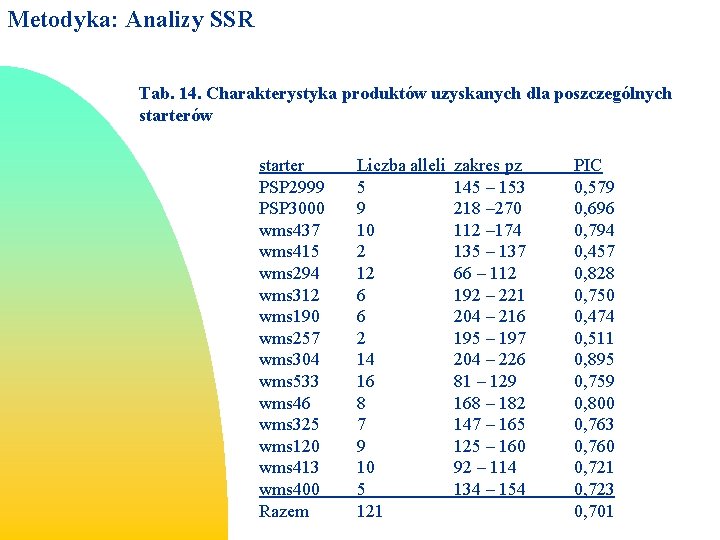
Metodyka: Analizy SSR Tab. 14. Charakterystyka produktów uzyskanych dla poszczególnych starterów starter PSP 2999 PSP 3000 wms 437 wms 415 wms 294 wms 312 wms 190 wms 257 wms 304 wms 533 wms 46 wms 325 wms 120 wms 413 wms 400 Razem Liczba alleli 5 9 10 2 12 6 6 2 14 16 8 7 9 10 5 121 zakres pz 145 – 153 218 – 270 112 – 174 135 – 137 66 – 112 192 – 221 204 – 216 195 – 197 204 – 226 81 – 129 168 – 182 147 – 165 125 – 160 92 – 114 134 – 154 PIC 0, 579 0, 696 0, 794 0, 457 0, 828 0, 750 0, 474 0, 511 0, 895 0, 759 0, 800 0, 763 0, 760 0, 721 0, 723 0, 701

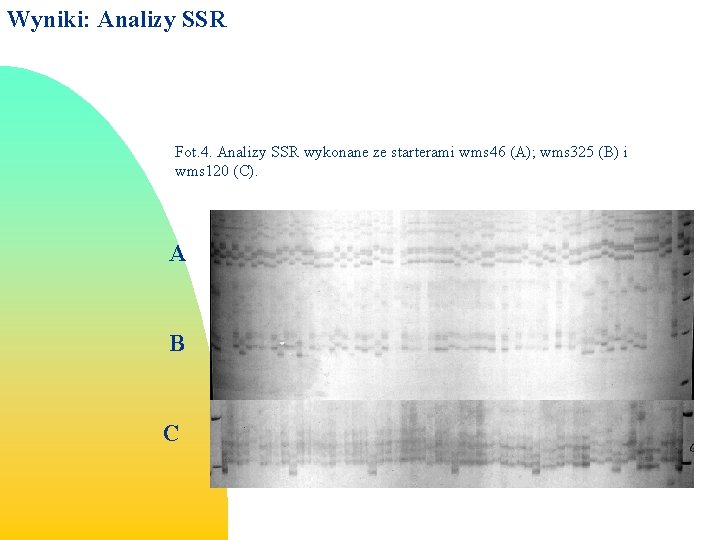
Wyniki: Analizy SSR Fot. 4. Analizy SSR wykonane ze starterami wms 46 (A); wms 325 (B) i wms 120 (C). A B C

Wyniki: Analizy SSR * * * *

Wyniki: Analizy STS Tab. 15. Wyniki testowania wybranych systemów na zestawie 82 odmian pszenicy zwyczajnej System ABA 2 ABA 3 ABC 152 ABC 155 ABC 158 … CDO 395 CDO 541 CDO 673 MWG 68 Pttksu. D 22 RAZEM: polimorficzne 13 4 8 3 7. . . 8 3 1 5 10 86 monomorficzne 5 1 0 0 2. . . 0 0 1 4 7 44 PIC 0, 291 0, 234 0, 267 0, 244 0, 208. . . 0, 176 0, 160 0, 025 0, 153 0, 235 0, 202 Wielkość (pz) 1000 – 150 1100 – 340 1200 – 250 520 -200 1800 – 280. . . 520 – 200 1250 -250 300 – 200 440 – 80 1600 – 300

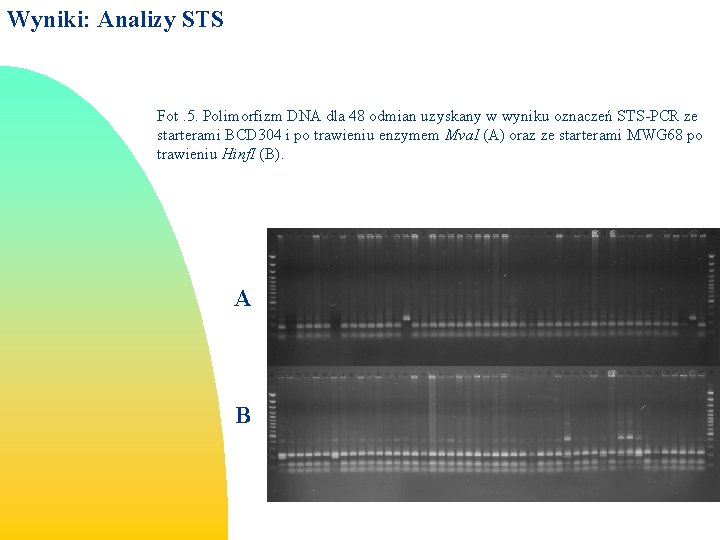
Wyniki: Analizy STS Fot. 5. Polimorfizm DNA dla 48 odmian uzyskany w wyniku oznaczeń STS-PCR ze starterami BCD 304 i po trawieniu enzymem Mva. I (A) oraz ze starterami MWG 68 po trawieniu Hinf. I (B). A B

Wyniki: Analizy STS * * * Podobieństwo Dice’a

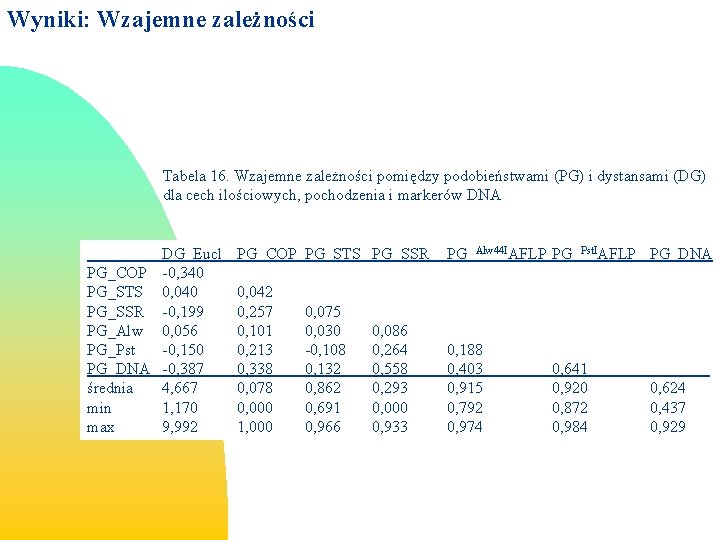
Wyniki: Wzajemne zależności Tabela 16. Wzajemne zależności pomiędzy podobieństwami (PG) i dystansami (DG) dla cech ilościowych, pochodzenia i markerów DNA DG_Eucl PG_COP -0, 340 PG_STS 0, 040 PG_SSR -0, 199 PG_Alw 0, 056 PG_Pst -0, 150 PG_DNA -0, 387 średnia 4, 667 min 1, 170 max 9, 992 PG_COP PG_STS PG_SSR PG_Alw 44 IAFLP PG_Pst. IAFLP PG_DNA 0, 042 0, 257 0, 101 0, 213 0, 338 0, 078 0, 000 1, 000 0, 188 0, 403 0, 915 0, 792 0, 974 0, 075 0, 030 -0, 108 0, 132 0, 862 0, 691 0, 966 0, 086 0, 264 0, 558 0, 293 0, 000 0, 933 0, 641 0, 920 0, 872 0, 984 0, 624 0, 437 0, 929

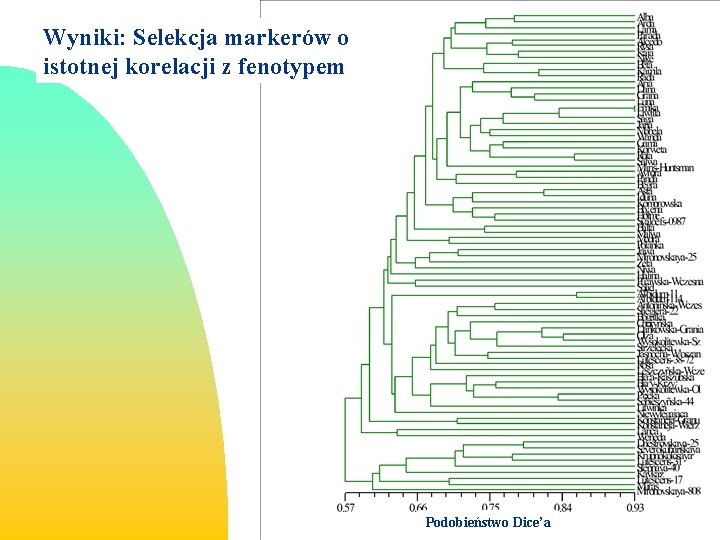
Wyniki: Selekcja markerów o istotnej korelacji z fenotypem Podobieństwo Dice’a

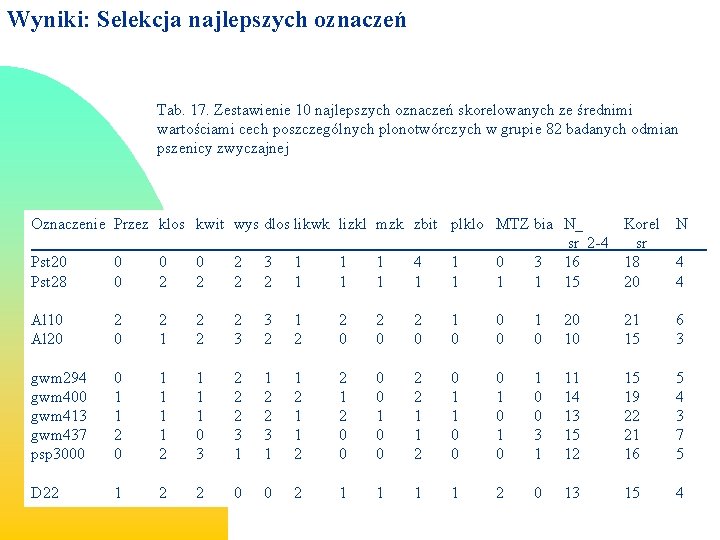
Wyniki: Selekcja najlepszych oznaczeń Tab. 17. Zestawienie 10 najlepszych oznaczeń skorelowanych ze średnimi wartościami cech poszczególnych plonotwórczych w grupie 82 badanych odmian pszenicy zwyczajnej Oznaczenie Przez klos kwit wys dlos likwk lizkl mzk zbit plklo MTZ bia N_ Korel sr_2 -4 _sr Pst 20 0 2 3 1 1 1 4 1 0 3 16 18 Pst 28 0 2 2 1 1 1 15 20 N Al 10 Al 20 2 1 2 2 2 3 3 2 1 2 2 0 2 0 1 0 0 0 1 0 20 10 21 15 6 3 gwm 294 gwm 400 gwm 413 gwm 437 psp 3000 0 1 1 2 1 1 1 0 3 2 2 2 3 1 1 2 2 1 2 0 0 1 0 0 2 2 1 1 2 0 1 1 0 0 0 1 0 1 0 0 3 1 11 14 13 15 12 15 19 22 21 16 5 4 3 7 5 D 22 1 2 2 0 0 2 1 1 2 0 13 15 4 4 4

Wnioski Badane odmiany pszenicy zwyczajnej cechują się wysokim poziomem podobieństwa DNA. Wzajemne podobieństwo międzyodmianowe różni się w zależności od zastosowanej metody analizy DNA. Jedyne istotne zgrupowania wewnątrz analizowanych ozimych odmian pszenicy zwyczajnej uzyskano na podstawie markerów Alw 44 IAFLP. Pozwoliły one wyodrębnić grupę odmian z translokacją 1 BL. 1 RS. Za pomocą analiz SSR wykazano, że większość rzadkich alleli występowała u odmian wcześnie rejestrowanych: Komorowska, Konstancja Wierzbieńska, Muras, Niewylegająca, Holme, Jasnocha Włoszanowska, Solid. Selekcja markerów skorelowanych z fenotypem pozwala na ocenę podobieństwa genetycznego przydatnego dla hodowców Zidentyfikowane markery o wysokiej korelacji z cechami ilościowymi w szerokim tle genetycznym mogą znaleźć zastosowanie w hodowli odmian o podwyższonym plonie
 Odmiany alotropowe węgla
Odmiany alotropowe węgla Praotec čech a jeho dcery
Praotec čech a jeho dcery Jan čech špirek
Jan čech špirek Praotec čech
Praotec čech Korelacja cech jakościowych
Korelacja cech jakościowych Do cech populacji nie należy
Do cech populacji nie należy Projekt logiczny sieci
Projekt logiczny sieci 10 cech baśni
10 cech baśni Cech uc
Cech uc Jedną z cech dobrze zaprojektowanej sieci nie jest
Jedną z cech dobrze zaprojektowanej sieci nie jest What role does dna polymerase play in copying dna?
What role does dna polymerase play in copying dna? Replication fork
Replication fork Dna and genes chapter 11
Dna and genes chapter 11 Bioflix activity dna replication nucleotide pairing
Bioflix activity dna replication nucleotide pairing Coding dna and non coding dna
Coding dna and non coding dna Dna hybridization microarray
Dna hybridization microarray Codice genetico ridondante e degenerato
Codice genetico ridondante e degenerato Bahan dasar penyusun dna
Bahan dasar penyusun dna Dna meaning
Dna meaning Dna
Dna Fragmento de okazaki função
Fragmento de okazaki função Pozagenowy dna
Pozagenowy dna Chapter 12 section 3 dna rna and protein
Chapter 12 section 3 dna rna and protein Struttura del dna
Struttura del dna Miłość twa od najwyższych gór
Miłość twa od najwyższych gór Where is the site of dna replication in eukaryotes
Where is the site of dna replication in eukaryotes Dna strands are antiparallel. what does that mean
Dna strands are antiparallel. what does that mean Dna expression
Dna expression Dna transcription and translation
Dna transcription and translation Replication fork
Replication fork