Mapowanie locii cech ilociowych QTL Cechy ilociowe vs




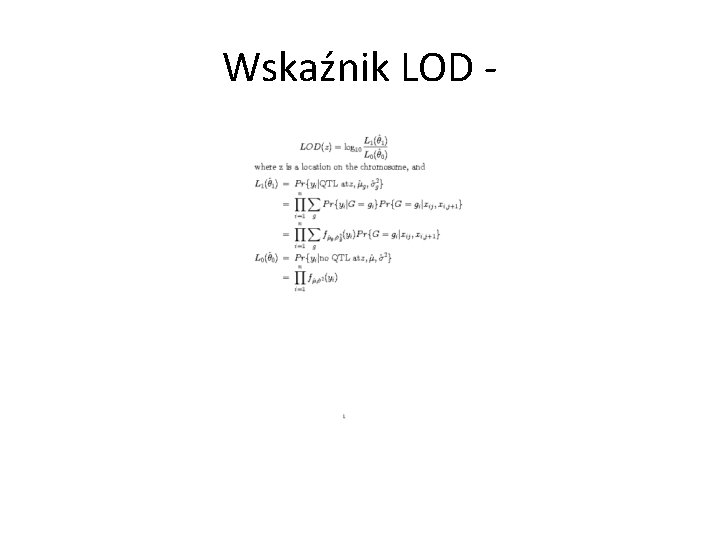






![Utworzenie mapy genetycznej Długość [c. M] Maksymalny dystans między dwoma loci [c. M] Średnia Utworzenie mapy genetycznej Długość [c. M] Maksymalny dystans między dwoma loci [c. M] Średnia](https://slidetodoc.com/presentation_image_h/ab8288b45cc161757b09de9d78f9607e/image-19.jpg)
- Slides: 21

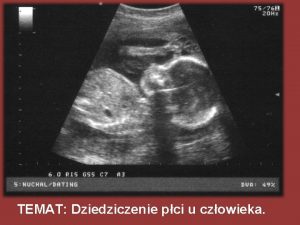
Mapowanie locii cech ilościowych QTL

Cechy ilościowe vs Cechy jakościowe • Rozkład ciągły, krzywa Gausa • Plon, masa, wysokość itp. • Determinowane poligenicznie • QTL • Rozkład dyskretny • Kolor nasion, kształt nasion, kolor kwiatów itp. • Determinowane przez pojedyncze geny • Markery sprzężone z cechą

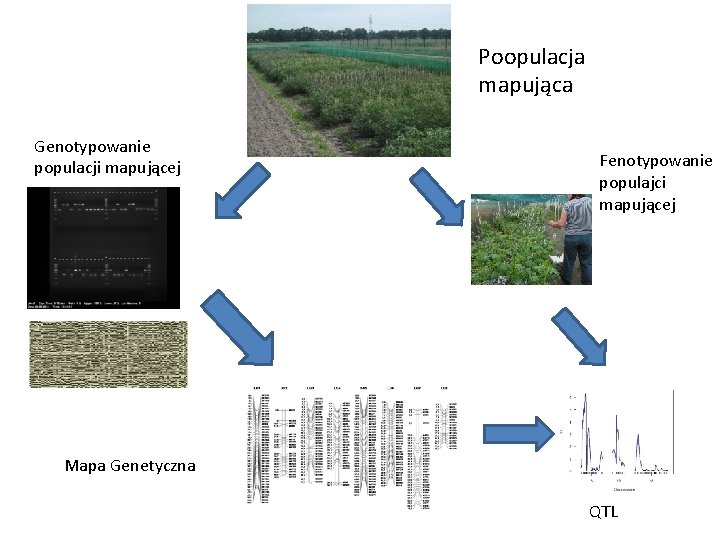
Poopulacja mapująca Genotypowanie populacji mapującej Fenotypowanie populajci mapującej Mapa Genetyczna QTL

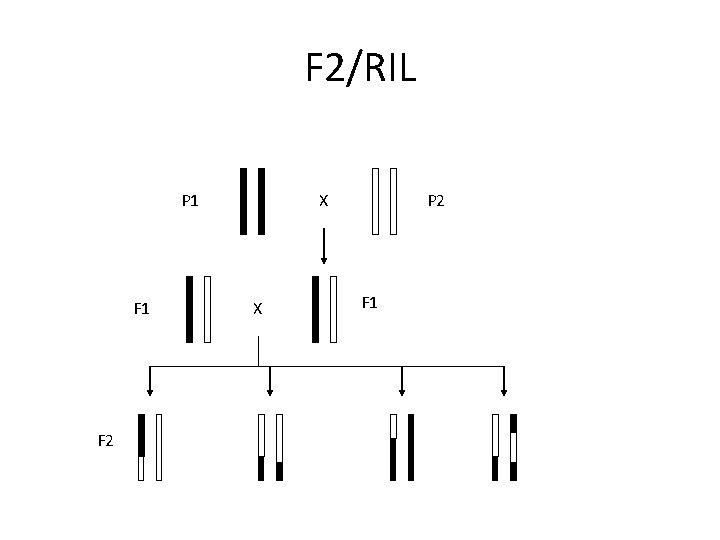
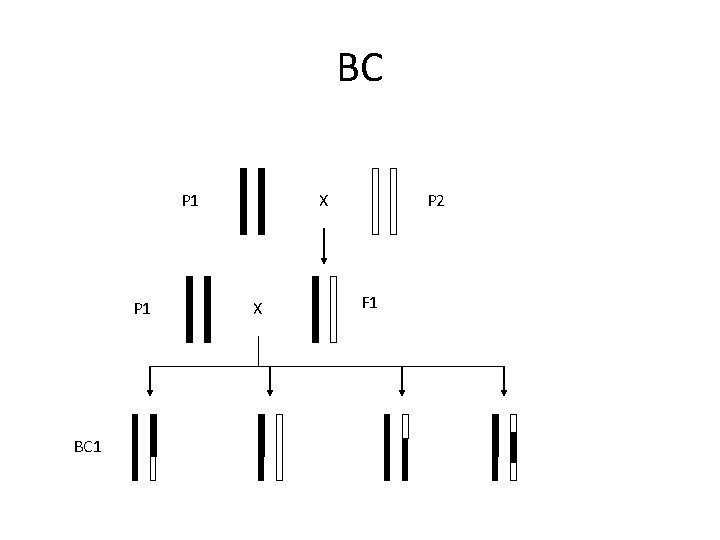
Populacje mapujące • • F 2; AA, Aa, aa RIL; AA, aa DH; AA, aa BC; AA Aa lub aa Aa

F 2/RIL P 1 F 2 X X P 2 F 1

BC P 1 BC 1 X X P 2 F 1

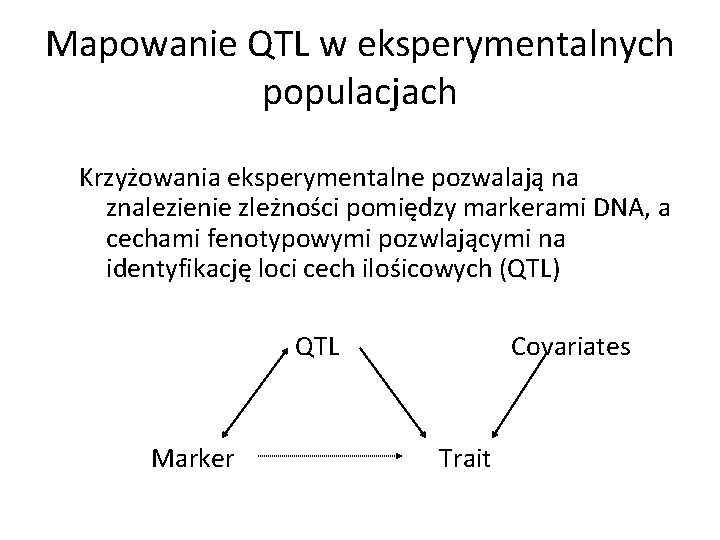
Mapowanie QTL w eksperymentalnych populacjach Krzyżowania eksperymentalne pozwalają na znalezienie zleżności pomiędzy markerami DNA, a cechami fenotypowymi pozwlającymi na identyfikację loci cech ilośicowych (QTL) QTL Marker Covariates Trait

Modle i załorzenia • Barak zakuceń procesu rekombinacji • Niezależność • Rozkład normalny yi|X ~ N( X, X 2) • Homoskedastyczność X 2 = 2

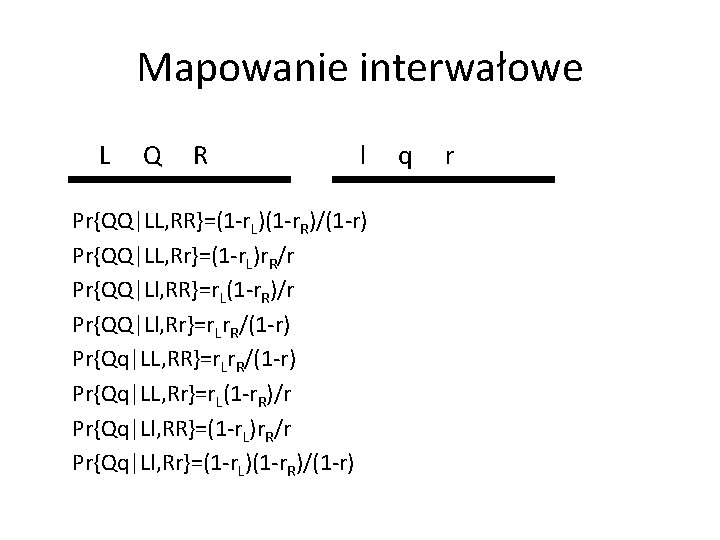
Mapowanie interwałowe L Q R l Pr{QQ|LL, RR}=(1 -r. L)(1 -r. R)/(1 -r) Pr{QQ|LL, Rr}=(1 -r. L)r. R/r Pr{QQ|Ll, RR}=r. L(1 -r. R)/r Pr{QQ|Ll, Rr}=r. Lr. R/(1 -r) Pr{Qq|LL, RR}=r. Lr. R/(1 -r) Pr{Qq|LL, Rr}=r. L(1 -r. R)/r Pr{Qq|Ll, RR}=(1 -r. L)r. R/r Pr{Qq|Ll, Rr}=(1 -r. L)(1 -r. R)/(1 -r) q r
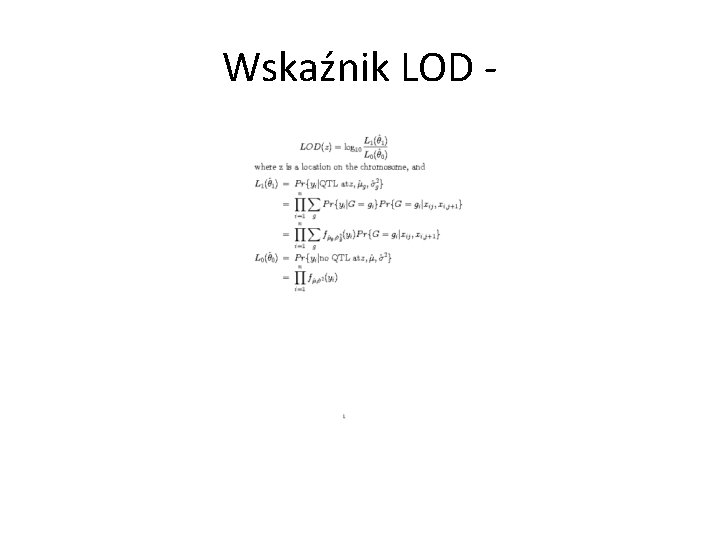
Wskaźnik LOD -

Krzywa LOD • Profil prawdopodobieństwa • Wyraźny pik jest interpretowany jako QTL • 1. 5 -LOD wspiera mapowanie interwałowe


Dystrybucja LOD • Symulacje komputerowe – Typ krzyżowania – Wielkość genomu – Liczba i odległości pomiędzy markerami – Liczba obiektów z brakiem wyniku genotypowania – Prawdziwa dystrybucja fenotypu • Permutacje lub bootstrap

Mapowanie Interwałowe • Zalety – Bierze pod uwagę brakujące dane (bardziej wiarygodne podejście) – Interpoluje pozycje pomiędzy markerami – Dostarcza wsparcia dla interwałów – Dostarcza bardziej dokładnego określenia efektu QTL na cechę • Wady – Wymaga dużych mocy obliczeniowych komputerów – Polega na mapach genetycznych dobrej jakości (słaba mapa = złe wyniki QTL) – Trudności w wprowadzeniu kowariancji


Genotypowanie populacji łubinu wąskolistnego metodą DAr. Tseq

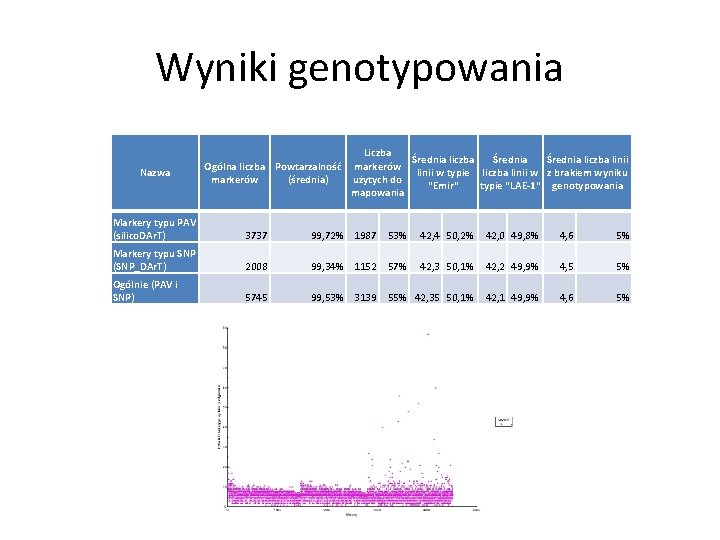
Wyniki genotypowania Nazwa Ogólna liczba Powtarzalność markerów (średnia) Liczba Średnia liczba linii markerów linii w typie liczba linii w z brakiem wyniku użytych do "Emir" typie "LAE-1" genotypowania mapowania Markery typu PAV (silico. DAr. T) 3737 99, 72% 1987 53% 42, 4 50, 2% 42, 0 49, 8% 4, 6 5% Markery typu SNP (SNP_DAr. T) 2008 99, 34% 1152 57% 42, 3 50, 1% 42, 2 49, 9% 4, 5 5% Ogólnie (PAV i SNP) 5745 99, 53% 3139 55% 42, 35 50, 1% 42, 1 49, 9% 4, 6 5%

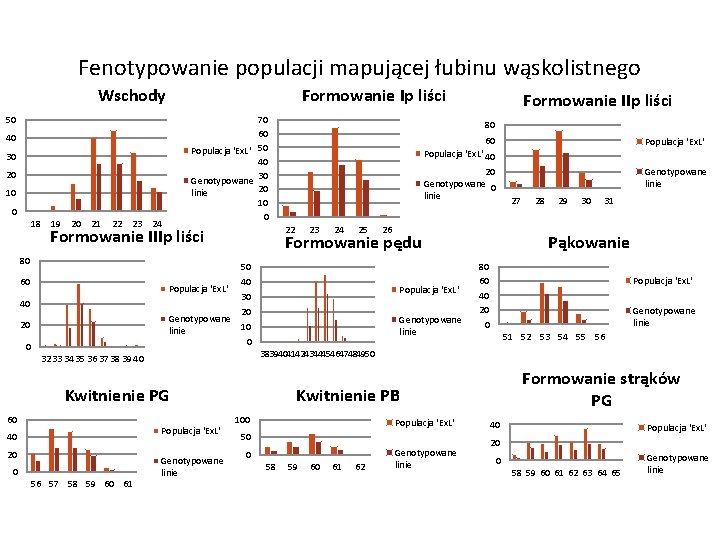
Fenotypowanie populacji mapującej łubinu wąskolistnego Wschody Formowanie Ip liści 50 70 60 Populacja 'Ex. L' 50 40 30 Genotypowane 20 linie 10 0 40 30 20 10 0 18 19 20 21 22 23 24 80 60 Populacja 'Ex. L' 40 Genotypowane linie 20 0 0 56 57 58 59 60 61 20 Genotypowane 0 linie 23 24 25 26 Populacja 'Ex. L' Genotypowane linie 27 28 29 30 31 Pąkowanie 80 60 40 20 0 Populacja 'Ex. L' Genotypowane linie 51 52 53 54 55 56 38394041424344454647484950 Populacja 'Ex. L' 20 Populacja 'Ex. L' 40 50 40 30 20 10 0 Kwitnienie PG 40 60 Formowanie pędu 32 33 34 35 36 37 38 39 40 60 80 22 Formowanie IIIp liści Formowanie IIp liści Genotypowane linie Formowanie strąków PG Kwitnienie PB 100 Populacja 'Ex. L' 50 0 58 59 60 61 62 Genotypowane linie 40 Populacja 'Ex. L' 20 0 58 59 60 61 62 63 64 65 Genotypowane linie
![Utworzenie mapy genetycznej Długość c M Maksymalny dystans między dwoma loci c M Średnia Utworzenie mapy genetycznej Długość [c. M] Maksymalny dystans między dwoma loci [c. M] Średnia](https://slidetodoc.com/presentation_image_h/ab8288b45cc161757b09de9d78f9607e/image-19.jpg)
Utworzenie mapy genetycznej Długość [c. M] Maksymalny dystans między dwoma loci [c. M] Średnia dystans między dwoma loci [c. M] Liczba loci [n] Liczba markerów [n] MAPA 837 16, 72 0, 34 2472 2566

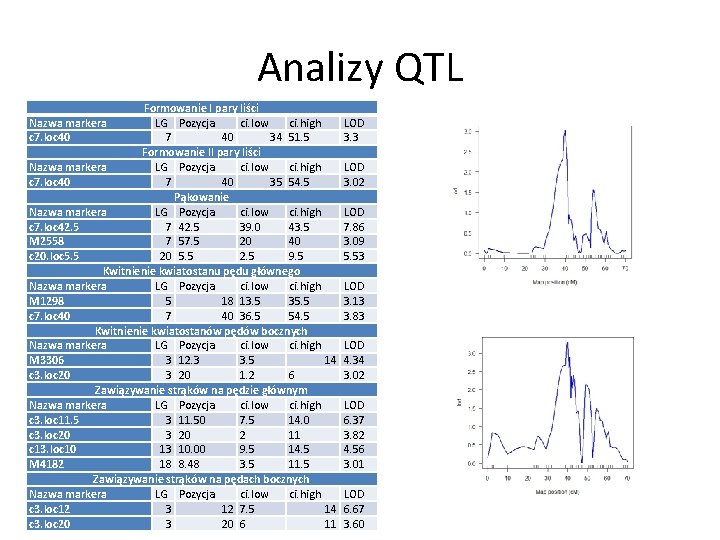
Analizy QTL Formowanie I pary liści Nazwa markera LG Pozycja ci. low ci. high LOD c 7. loc 40 7 40 34 51. 5 3. 3 Formowanie II pary liści Nazwa markera LG Pozycja ci. low ci. high LOD c 7. loc 40 7 40 35 54. 5 3. 02 Pąkowanie Nazwa markera LG Pozycja ci. low ci. high LOD c 7. loc 42. 5 7 42. 5 39. 0 43. 5 7. 86 M 2558 7 57. 5 20 40 3. 09 c 20. loc 5. 5 20 5. 5 2. 5 9. 5 5. 53 Kwitnienie kwiatostanu pędu głównego Nazwa markera LG Pozycja ci. low ci. high LOD M 1298 5 18 13. 5 35. 5 3. 13 c 7. loc 40 7 40 36. 5 54. 5 3. 83 Kwitnienie kwiatostanów pędów bocznych Nazwa markera LG Pozycja ci. low ci. high LOD M 3306 3 12. 3 3. 5 14 4. 34 c 3. loc 20 3 20 1. 2 6 3. 02 Zawiązywanie strąków na pędzie głównym Nazwa markera LG Pozycja ci. low ci. high LOD c 3. loc 11. 5 3 11. 50 7. 5 14. 0 6. 37 c 3. loc 20 3 20 2 11 3. 82 c 13. loc 10 13 10. 00 9. 5 14. 56 M 4182 18 8. 48 3. 5 11. 5 3. 01 Zawiązywanie strąków na pędach bocznych Nazwa markera LG Pozycja ci. low ci. high LOD c 3. loc 12 3 12 7. 5 14 6. 67 c 3. loc 20 3 20 6 11 3. 60

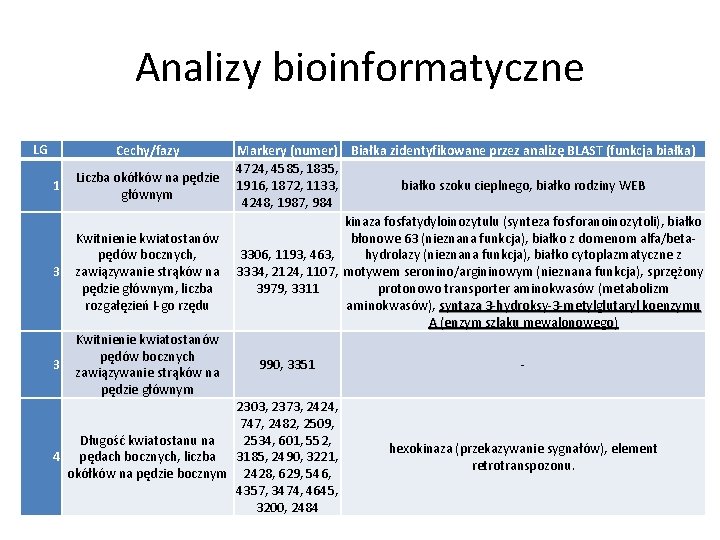
Analizy bioinformatyczne LG Cechy/fazy 1 Liczba okółków na pędzie głównym 3 Kwitnienie kwiatostanów pędów bocznych, zawiązywanie strąków na pędzie głównym, liczba rozgałęzień I-go rzędu 3 Kwitnienie kwiatostanów pędów bocznych zawiązywanie strąków na pędzie głównym Markery (numer) Białka zidentyfikowane przez analizę BLAST (funkcja białka) 4724, 4585, 1835, białko szoku cieplnego, białko rodziny WEB 1916, 1872, 1133, 4248, 1987, 984 kinaza fosfatydyloinozytulu (synteza fosforanoinozytoli), białko błonowe 63 (nieznana funkcja), białko z domenom alfa/betahydrolazy (nieznana funkcja), białko cytoplazmatyczne z 3306, 1193, 463, 3334, 2124, 1107, motywem seronino/argininowym (nieznana funkcja), sprzężony 3979, 3311 protonowo transporter aminokwasów (metabolizm aminokwasów), syntaza 3 -hydroksy-3 -metylglutaryl koenzymu A (enzym szlaku mewalonowego) 990, 3351 2303, 2373, 2424, 747, 2482, 2509, 2534, 601, 552, Długość kwiatostanu na 4 pędach bocznych, liczba 3185, 2490, 3221, okółków na pędzie bocznym 2428, 629, 546, 4357, 3474, 4645, 3200, 2484 - hexokinaza (przekazywanie sygnałów), element retrotranspozonu.
 Qtl club c'est quoi
Qtl club c'est quoi Genetic markers
Genetic markers Qtl mapping
Qtl mapping Qtl moodle
Qtl moodle Cechy sprzężone z płcią to cechy niedotyczące
Cechy sprzężone z płcią to cechy niedotyczące Praotec čech
Praotec čech Cowariancja
Cowariancja Cechy populacji biologicznej
Cechy populacji biologicznej Przykładowy projekt sieci komputerowej
Przykładowy projekt sieci komputerowej Cechy basni
Cechy basni Cech uc
Cech uc Jedną z cech dobrze zaprojektowanej sieci nie jest
Jedną z cech dobrze zaprojektowanej sieci nie jest Praotec čech a jeho dcery
Praotec čech a jeho dcery Jan čech špirek
Jan čech špirek Produkt turystyczny prosty
Produkt turystyczny prosty Zasady dobrego zachowania w szkole prezentacja
Zasady dobrego zachowania w szkole prezentacja Cechy najlepszego przyjaciela
Cechy najlepszego przyjaciela Konteksty literackie rodzaje
Konteksty literackie rodzaje Trojkat 30 6090
Trojkat 30 6090 Cechy podzielności przez 25
Cechy podzielności przez 25 Cechy kodu genetycznego
Cechy kodu genetycznego Scenariusz lekcji latarnik
Scenariusz lekcji latarnik