TGN 1412 POR QU LA ESTRUCTURA ES IMPORTANTE



















































- Slides: 102

TGN 1412: ¿POR QUÉ LA ESTRUCTURA ES IMPORTANTE? Bergerat D, Delgado A, Fernandez M, Moreno M, Rico V, Ruiz S, Sió M, Subirana I, Tirado E, Villalobos X

ÍNDICE 1. Planteamiento de las hipótesis 2. Objetivo 1: Estructura inmnoglobulinas Plegamiento Ig-like Modelos cadenas ligera y pesada Interacciones 3. Objetivo 2: CD 28 Macaca fascicularis CD 28 Mus musculus 4. Objetivo 3: Unión de la Fc al receptor Fc Agregados 5. Objetivo 4: Siglecs 6. Suplemento 7. Bibliografía

OBJETIVO 1 Estructura TGN 1412 Comparative modelling Secuencia problema Patrones Alineamiento basado en secuencia Alineamiento basado en estructura

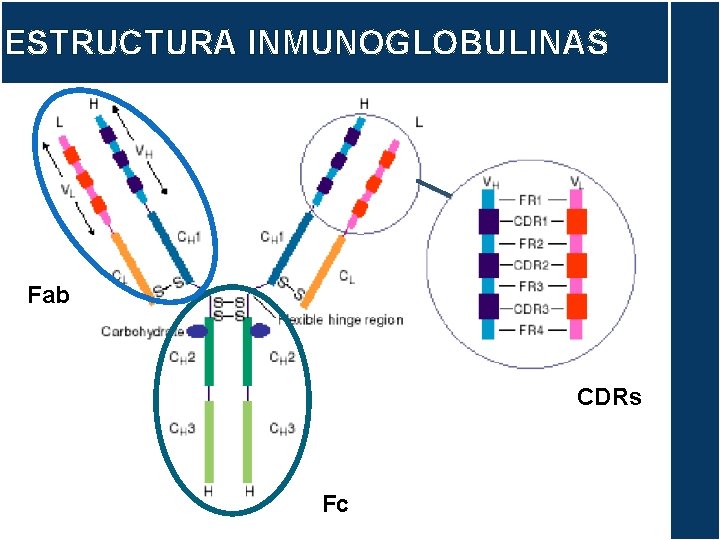
ESTRUCTURA INMUNOGLOBULINAS Fab CDRs Fc

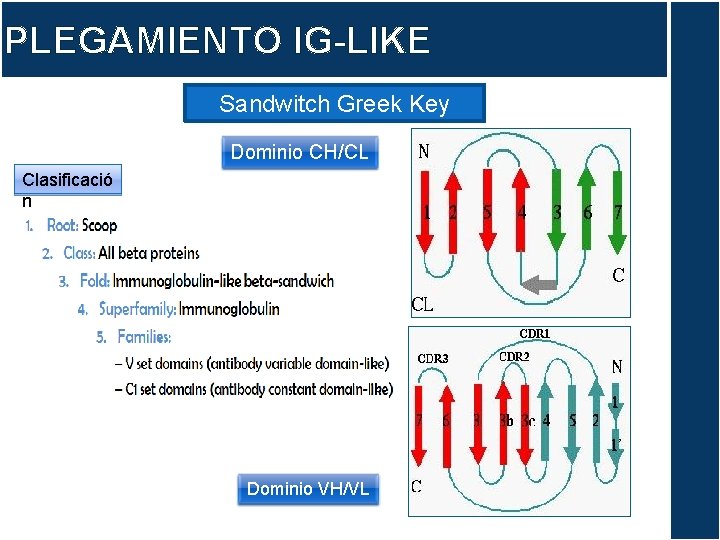
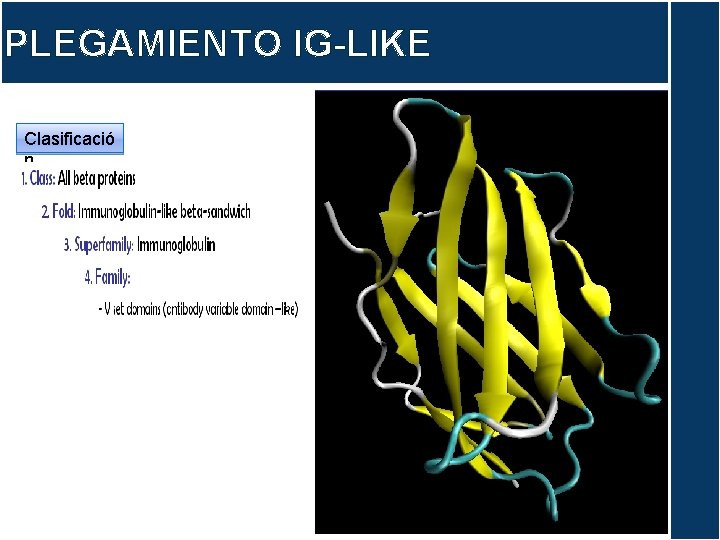
PLEGAMIENTO IG-LIKE Sandwitch Greek Key Dominio CH/CL Clasificació n Dominio VH/VL

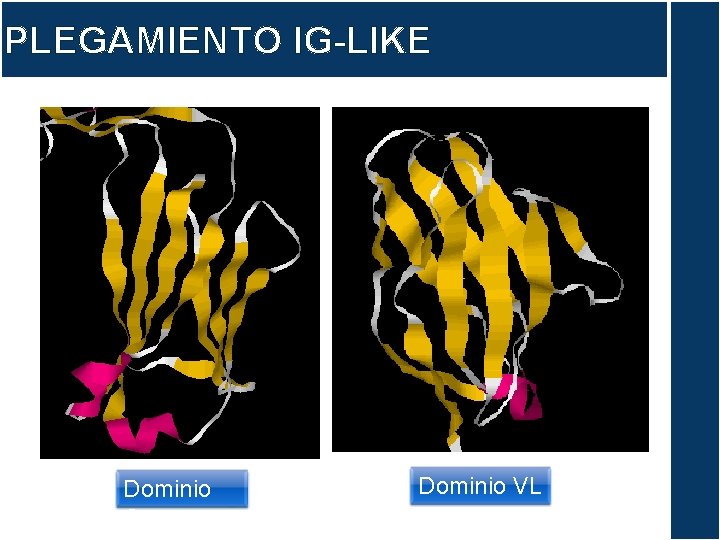
PLEGAMIENTO IG-LIKE Dominio CL Dominio VL

PLEGAMIENTO IG-LIKE Clasificació n

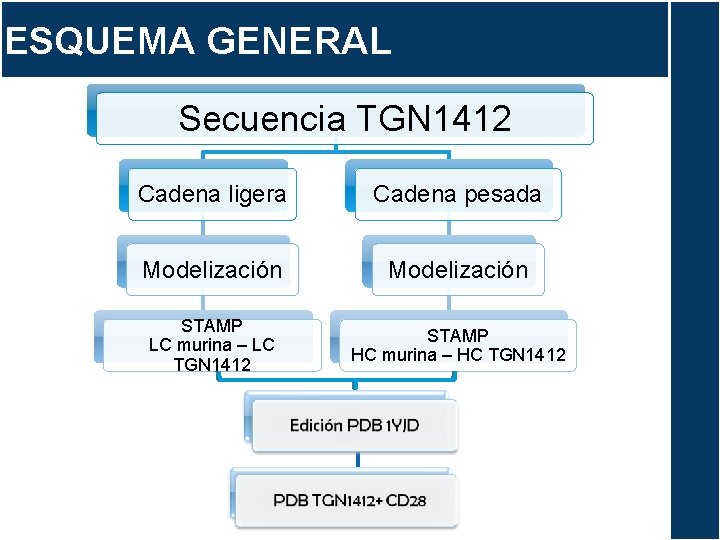
ESQUEMA GENERAL Secuencia TGN 1412 Cadena ligera Cadena pesada Modelización STAMP LC murina – LC TGN 1412 STAMP HC murina – HC TGN 1412

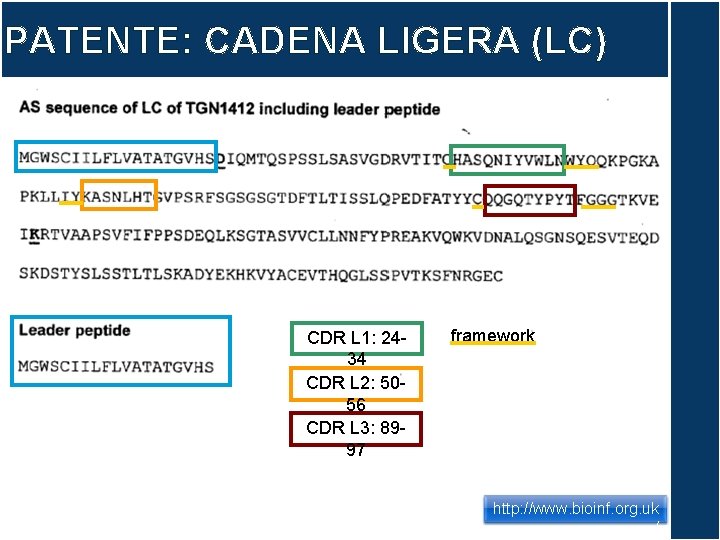
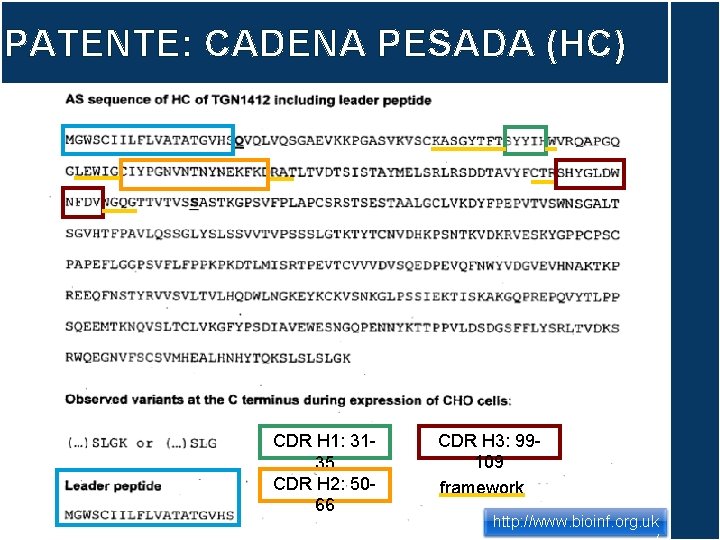
OBTENCIÓN DE LA SECUENCIA Humanizar manualmente Ig. G humana Ig. G murina Identificar CDRs A partir de la patente Sustituir en la Ig. G humana sus CDRs por los murinos

PATENTE: CADENA LIGERA (LC) CDR L 1: 2434 CDR L 2: 5056 CDR L 3: 8997 framework http: //www. bioinf. org. uk /

PATENTE: CADENA PESADA (HC) CDR H 1: 3135 CDR H 2: 5066 CDR H 3: 99109 framework http: //www. bioinf. org. uk

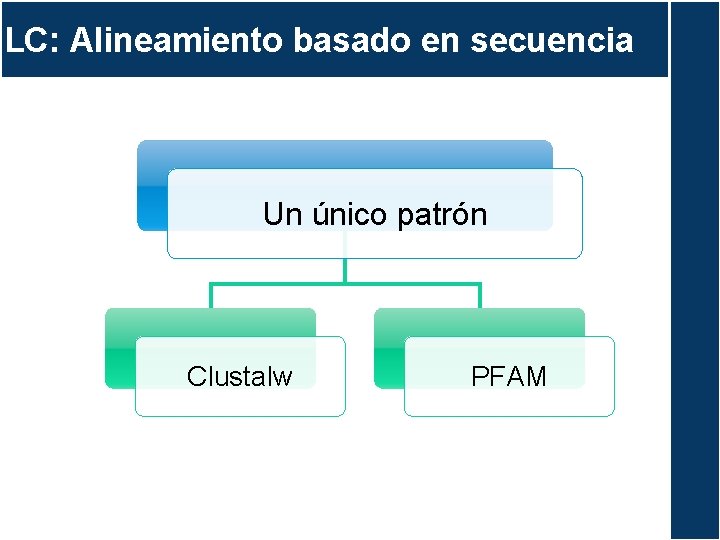
LC: PATRONES Un único patrón pdb 1 YJD (murino) Clustalw TGN 1412 – 1 YJDL Secuencia fasta LC TGN 1412 (modificada) Más de un patrón PSI-BLAST: búsqueda de homólogos remotos

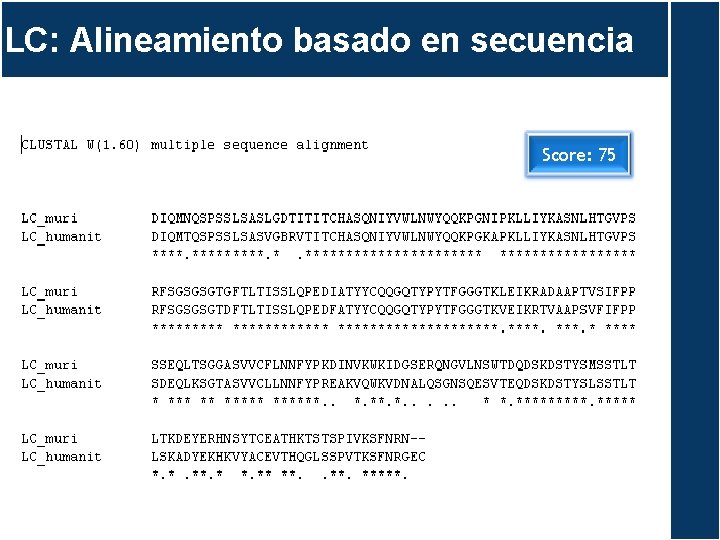
LC: PATRONES Score: 75

LC: PATRONES Un único patrón pdb 1 YJD (murino) Clustalw TGN 1412 – 1 YJDL Secuencia fasta LC TGN 1412 (modificada) Más de un patrón PSI-BLAST: búsqueda de homólogos remotos 1º Contra SWISSPROT 2º Contra PDB

LC: PATRONES

LC: PATRONES Un único patrón pdb 1 YJD (murino) Clustalw TGN 1412 – 1 YJDL Secuencia fasta LC TGN 1412 (modificada) Más de un patrón PSI-BLAST: búsqueda de homólogos remotos 2 FGW Ig 1º Contra SWISSPROT 1 DEE Ig. M 2º Contra PDB 1 FVD Ig. G 1 KB 5 Ig. G

LC: Alineamiento basado en secuencia Un único patrón Clustalw PFAM

LC: Alineamiento basado en secuencia Score: 75

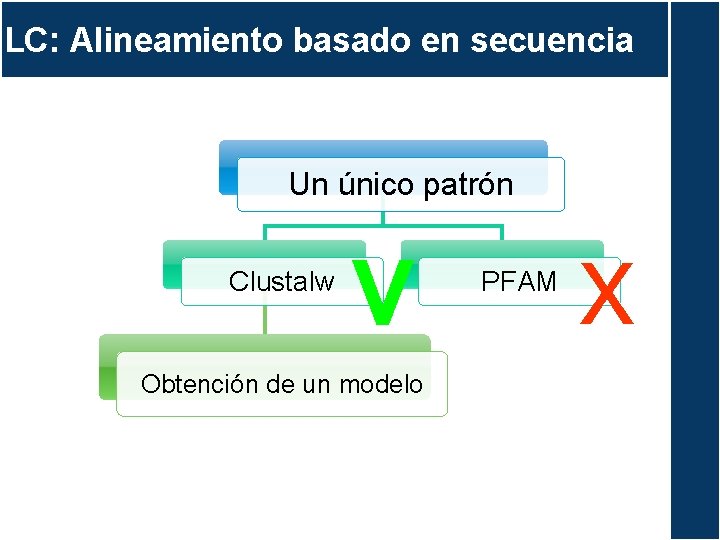
LC: Alineamiento basado en secuencia Únicamente alinea el dominio Ig-like una vez!!!

LC: Alineamiento basado en secuencia Un único patrón Clustalw V Obtención de un modelo PFAM X

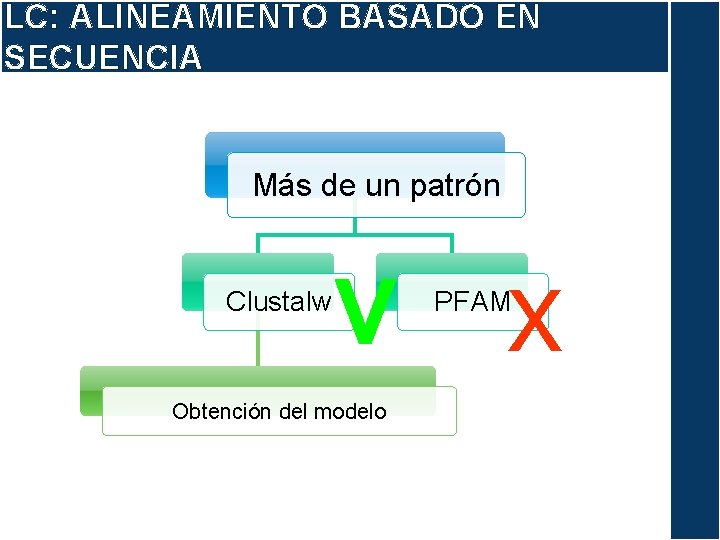
LC: ALINEAMIENTO BASADO EN SECUENCIA Más de un patrón Clustalw V Obtención del modelo X PFAM

LC: ALINEAMIENTO BASADO EN SECUENCIA Score: 59

LC: ALINEAMIENTO BASADO EN ESTRUCTURA Alineamiento estructural de los patrones: STAMP Estándar output Obtención del alineamiento y del pdb

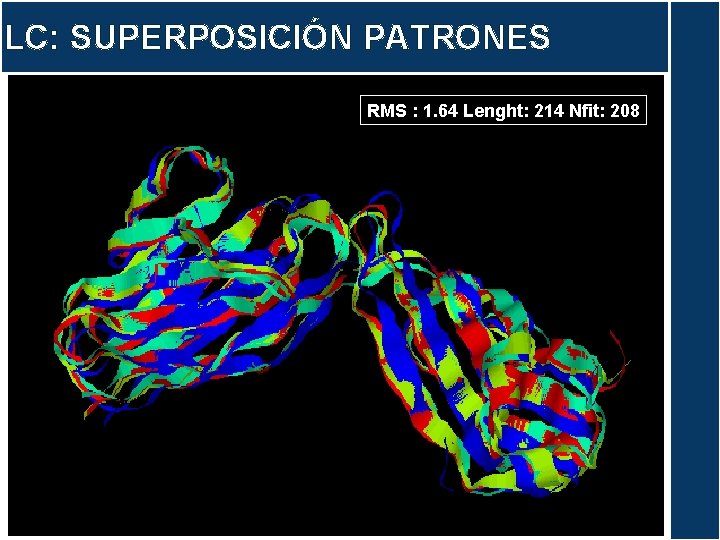
LC: SUPERPOSICIÓN PATRONES RMS : 1. 64 Lenght: 214 Nfit: 208

LC: ALINEAMIENTO BASADO EN ESTRUCTURA Construcción Modelo de Markov: HMMBUILD Alineamiento LC TGN 1412 – patrones: HMMALIGN Obtención del modelo: MODELLER

LC: RESULTADOS Hasta el momento hemos obtenido: 4 modelos basados en secuencia 2 con un patrón 2 con más de un patrón 2 modelos basados en estructura ¿Cuál es el mejor?

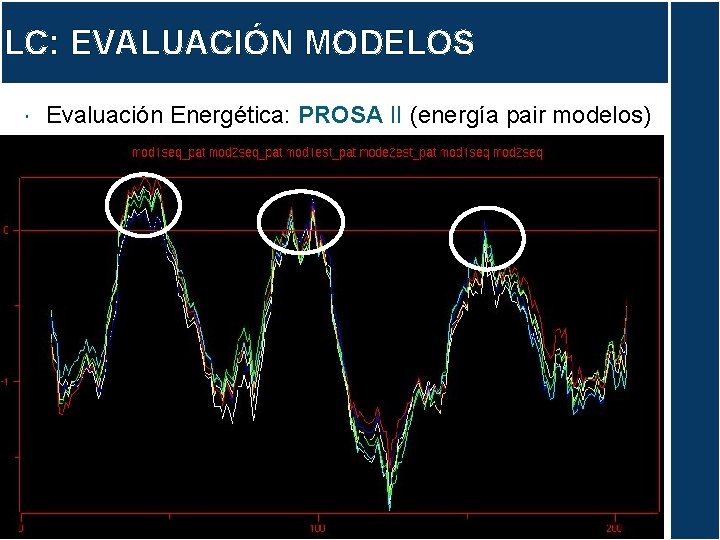
LC: EVALUACIÓN MODELOS Evaluación Energética: PROSA II (energía pair modelos)

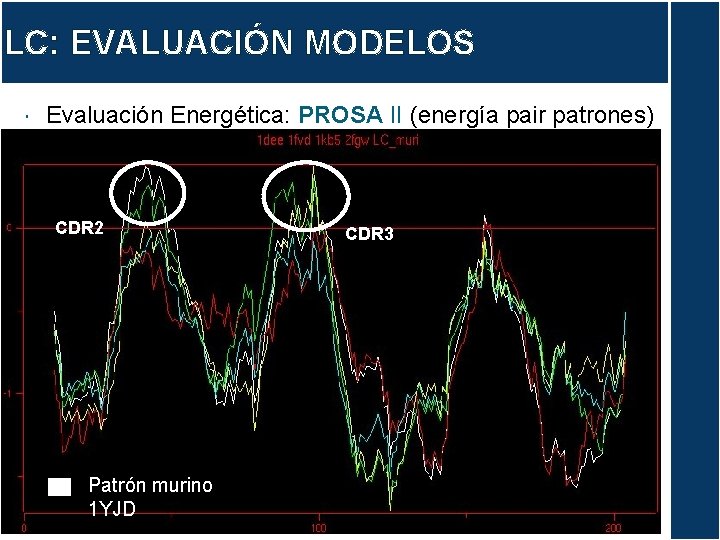
LC: EVALUACIÓN MODELOS Evaluación Energética: PROSA II (energía pair patrones) CDR 2 Patrón murino 1 YJD CDR 3

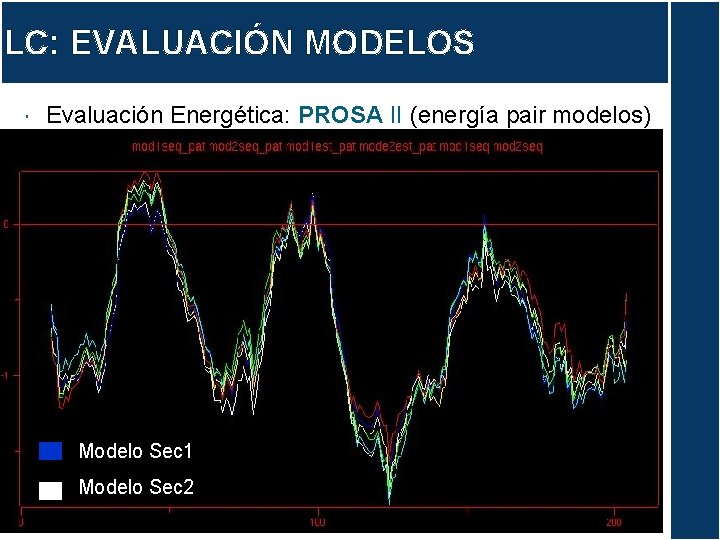
LC: EVALUACIÓN MODELOS Evaluación Energética: PROSA II (energía pair modelos) Modelo Sec 1 Modelo Sec 2

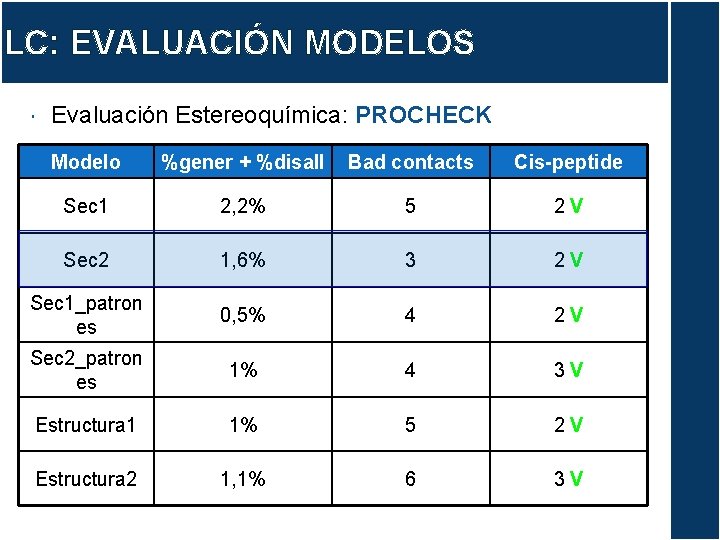
LC: EVALUACIÓN MODELOS Evaluación Estereoquímica: PROCHECK

LC: EVALUACIÓN MODELOS Evaluación Estereoquímica: PROCHECK Modelo %gener + %disall Bad contacts Cis-peptide Sec 1 2, 2% 5 2 V Sec 2 1, 6% 3 2 V Sec 1_patron es 0, 5% 4 2 V Sec 2_patron es 1% 4 3 V Estructura 1 1% 5 2 V Estructura 2 1, 1% 6 3 V

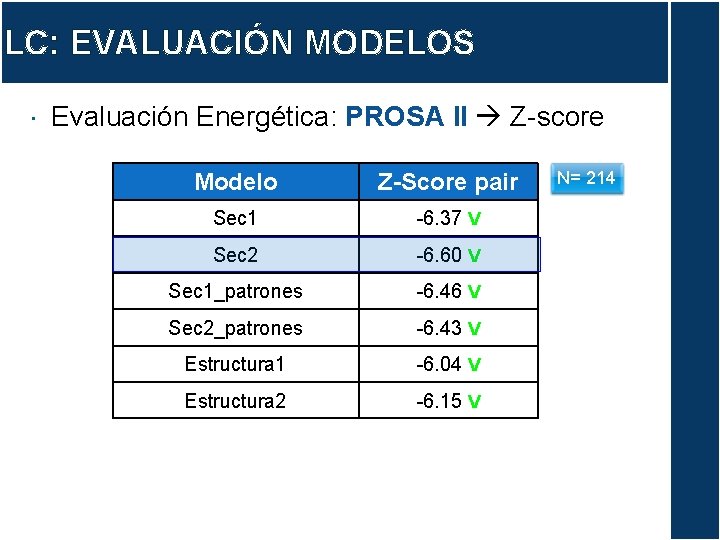
LC: EVALUACIÓN MODELOS Evaluación Energética: PROSA II Z-score Modelo Z-Score pair Sec 1 -6. 37 V Sec 2 -6. 60 V Sec 1_patrones -6. 46 V Sec 2_patrones -6. 43 V Estructura 1 -6. 04 V Estructura 2 -6. 15 V N= 214

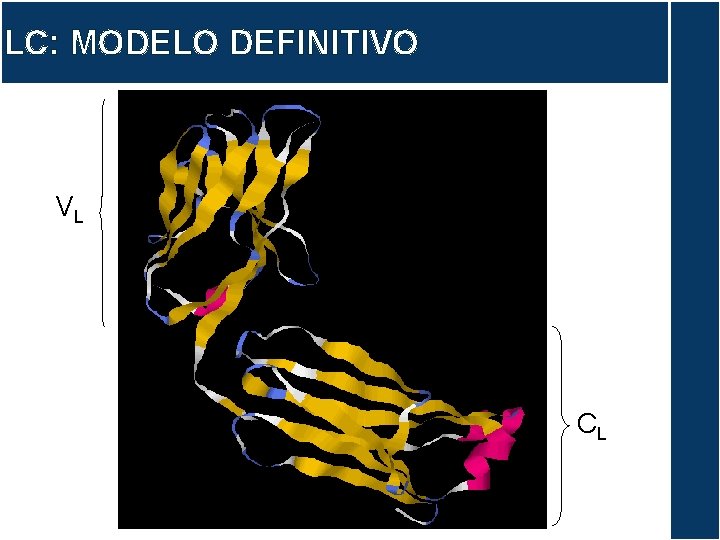
LC: MODELO DEFINITIVO VL CL

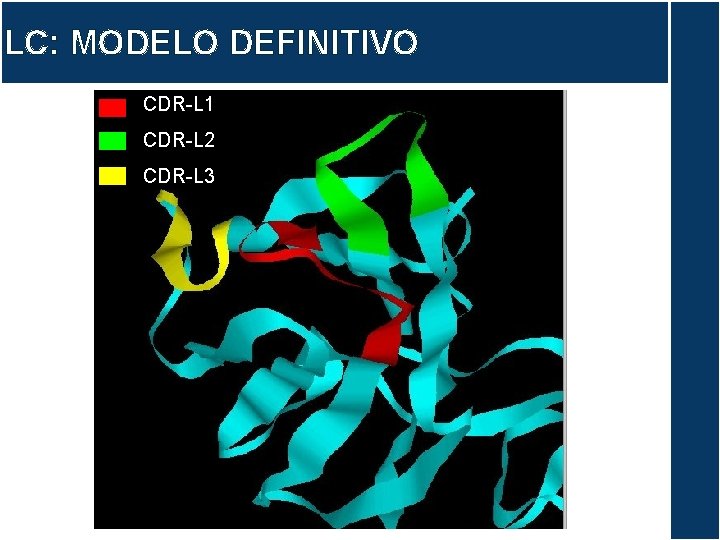
LC: MODELO DEFINITIVO CDR-L 1 CDR-L 2 CDR-L 3

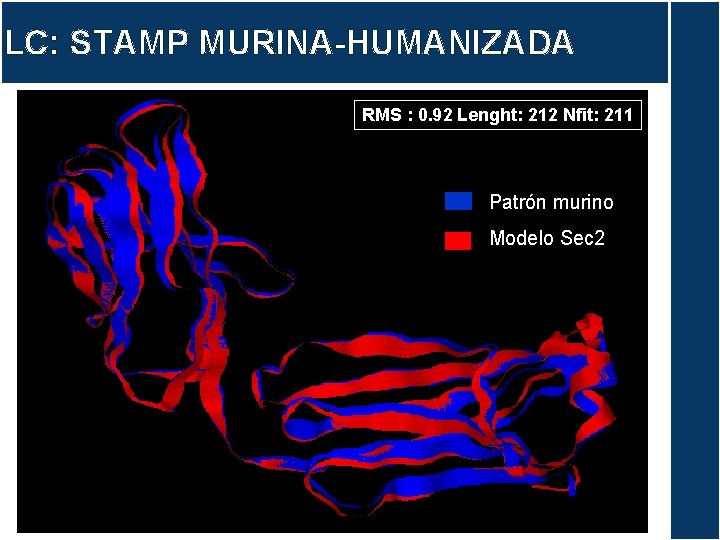
LC: STAMP MURINA-HUMANIZADA RMS : 0. 92 Lenght: 212 Nfit: 211 Patrón murino Modelo Sec 2

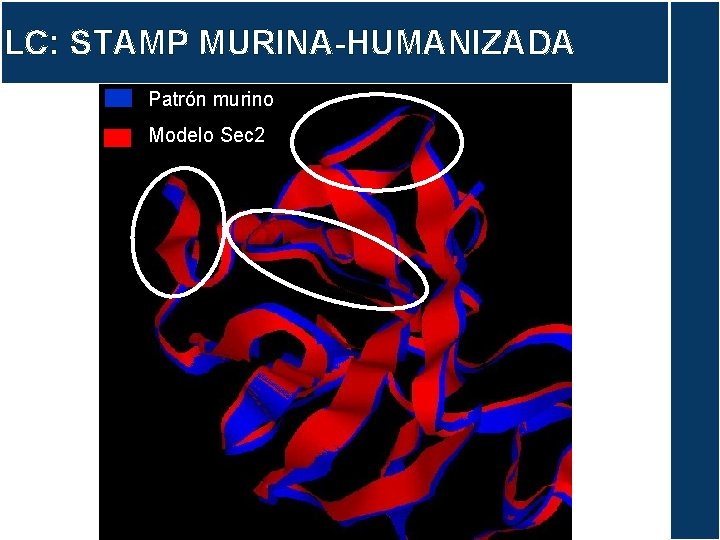
LC: STAMP MURINA-HUMANIZADA Patrón murino Modelo Sec 2

HC: PATRONES • Un único patrón pdb 1 YJD • Secuencia fasta 1 yjd. H • Clustalw

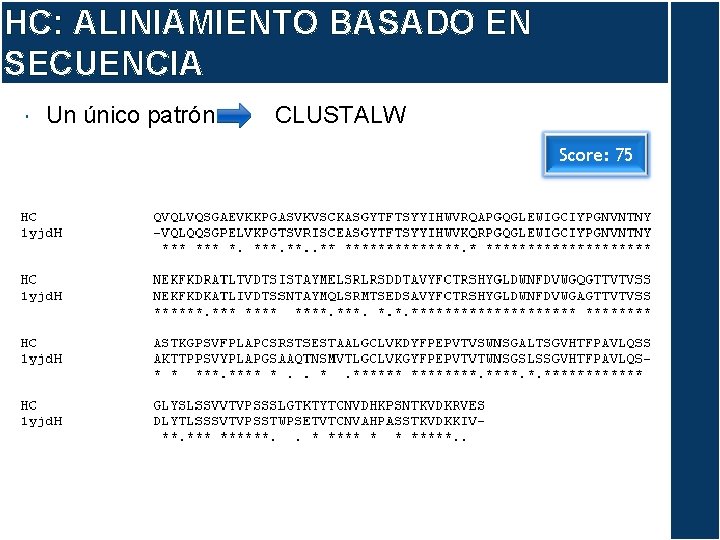
HC: ALINIAMIENTO BASADO EN SECUENCIA Un único patrón CLUSTALW Score: 75

HC: Resultados Obtención de los modelos Modeller 2 MODELOS ¿Cuál es el mejor modelo?

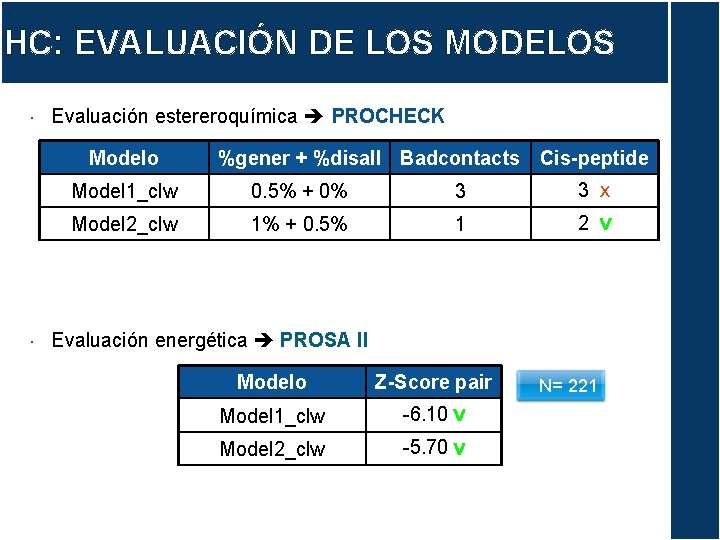
HC: EVALUACIÓN DE LOS MODELOS Evaluación estereroquímica PROCHECK Modelo %gener + %disall Badcontacts Cis-peptide Model 1_clw 0. 5% + 0% 3 3 X Model 2_clw 1% + 0. 5% 1 2 V Evaluación energética PROSA II Modelo Z-Score pair Model 1_clw -6. 10 V Model 2_clw -5. 70 V N= 221

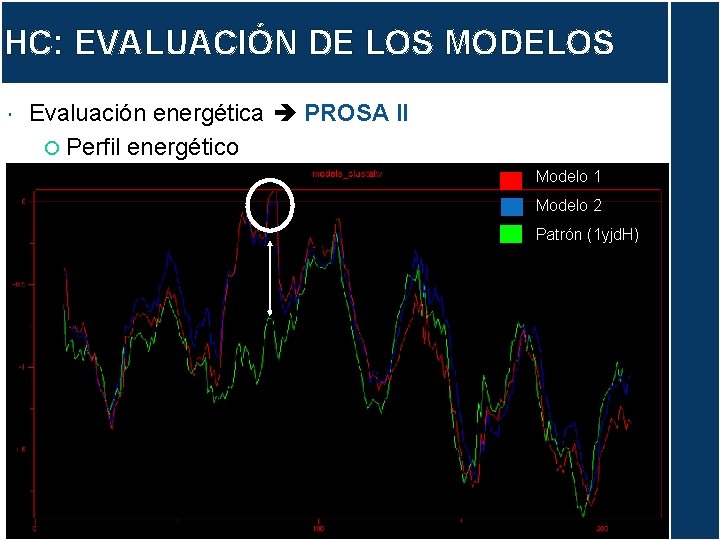
HC: EVALUACIÓN DE LOS MODELOS Evaluación energética PROSA II Perfil energético Modelo 1 Modelo 2 Patrón (1 yjd. H)

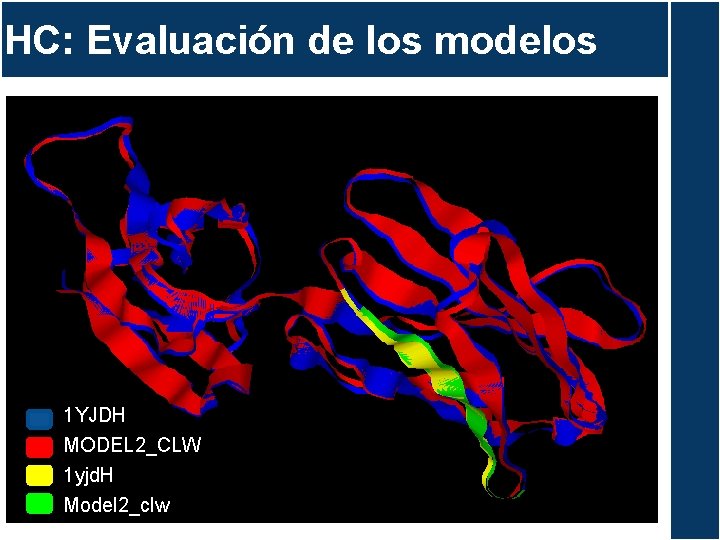
HC: Evaluación de los modelos 1 YJDH MODEL 2_CLW 1 yjd. H Model 2_clw

HC: Evaluación de los modelos Score: 75

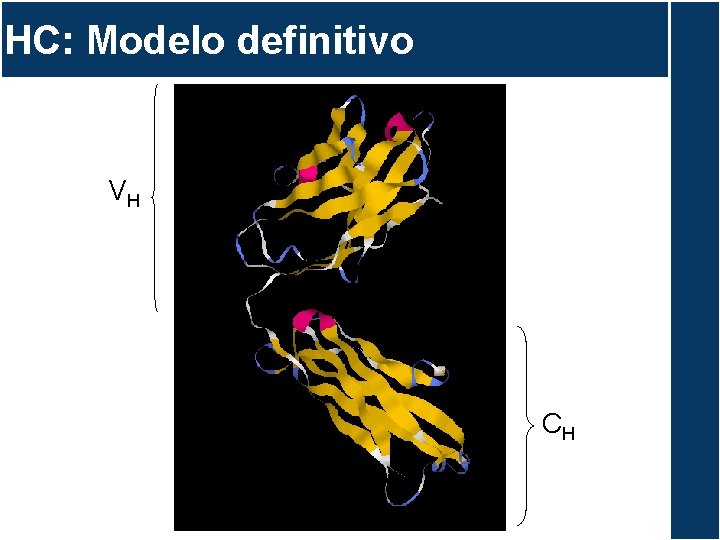
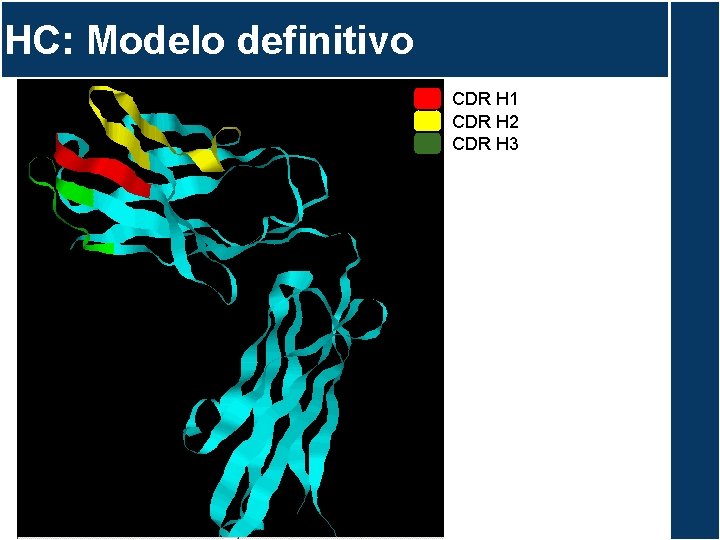
HC: Modelo definitivo VH CH

HC: Modelo definitivo CDR H 1 CDR H 2 CDR H 3

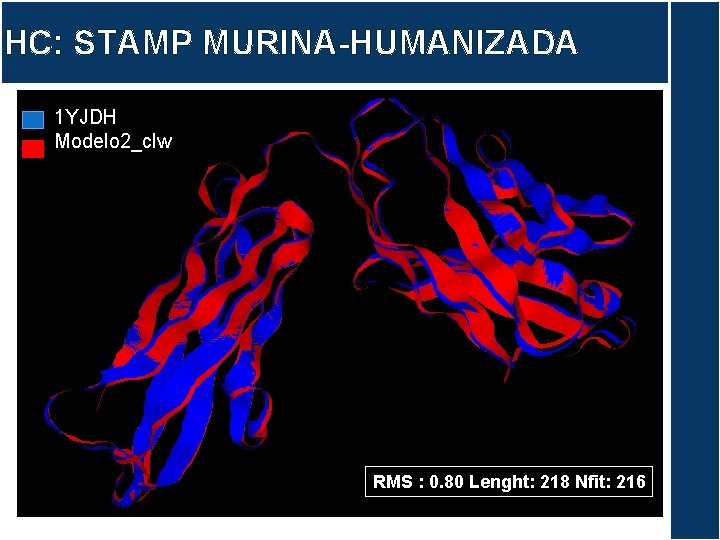
HC: STAMP MURINA-HUMANIZADA 1 YJDH Modelo 2_clw RMS : 0. 80 Lenght: 218 Nfit: 216

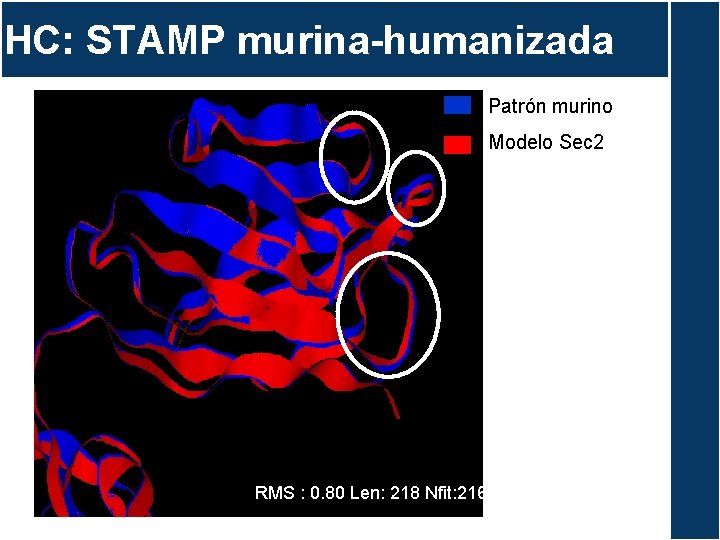
HC: STAMP murina-humanizada Patrón murino Modelo Sec 2 RMS : 0. 80 Len: 218 Nfit: 216

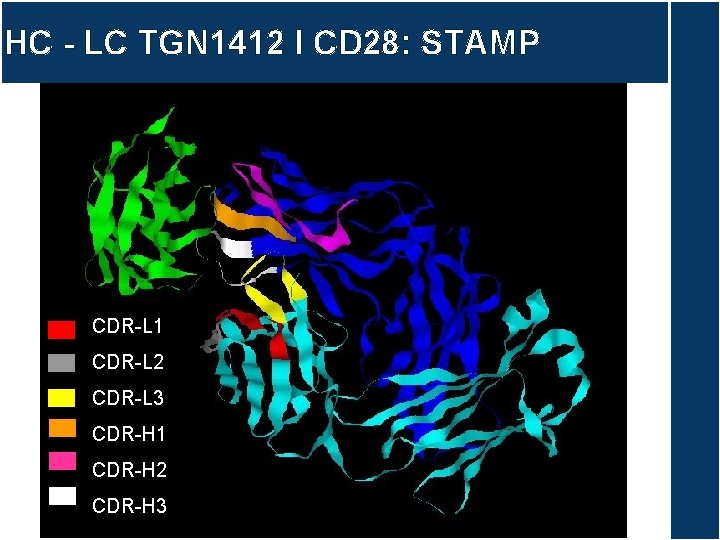
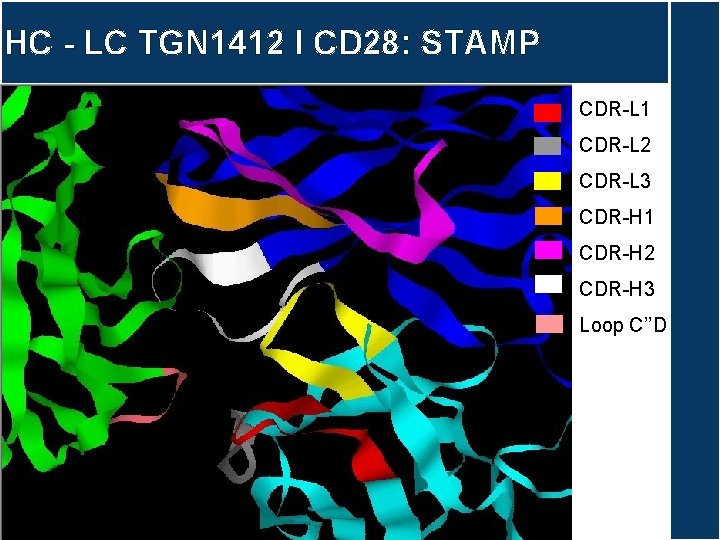
HC - LC TGN 1412 I CD 28: STAMP CDR-L 1 CDR-L 2 CDR-L 3 CDR-H 1 CDR-H 2 CDR-H 3

HC - LC TGN 1412 I CD 28: STAMP CDR-L 1 CDR-L 2 CDR-L 3 CDR-H 1 CDR-H 2 CDR-H 3 Loop C’’D

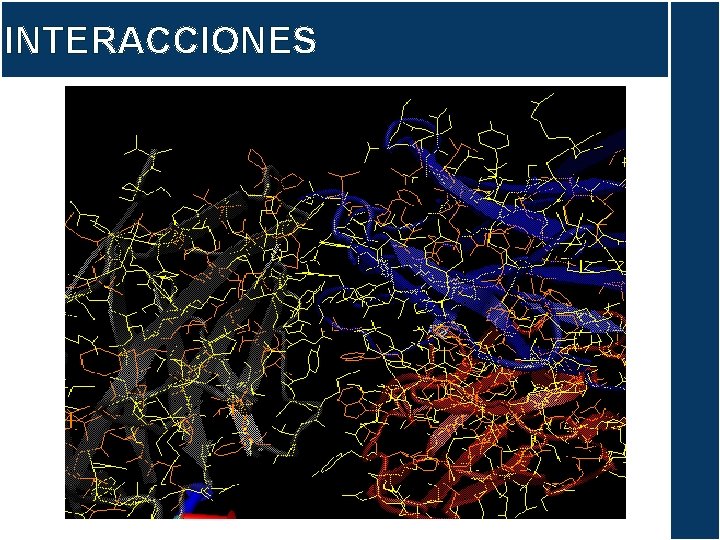
INTERACCIONES Comparamos las interacciones entre: el CD 28 humano y el AC murino (cristal) el CD 28 humano y el modelo TGN 1412 ¿Por qué? Podría ser que durante la humanización se hubieran provocado cambios estructurales que modificaran la interacción del TGN 1412 con el CD 28.

INTERACCIONES Las interacciones son del modelo NO OPTIMIZADO. Regiones de interacción del CD 28 con el Ac: Loop C’’D Lámina C’’ Lámina C Principales interacciones encontradas: Puentes de hidrógeno Interacciones pi Electrostáticas

INTERACCIONES

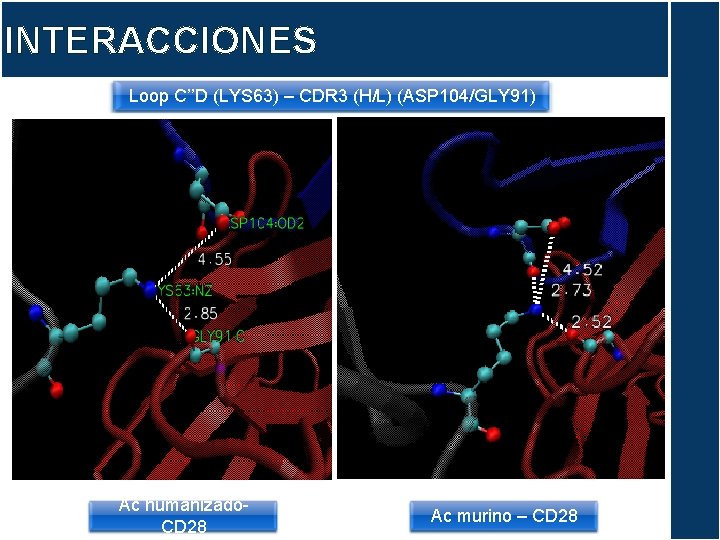
INTERACCIONES Loop C’’D (LYS 63) – CDR 3 (H/L) (ASP 104/GLY 91) Ac humanizado. CD 28 Ac murino – CD 28

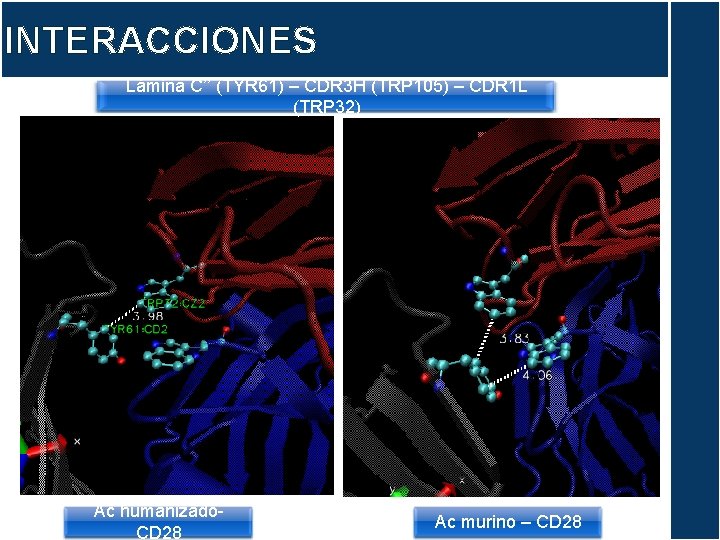
INTERACCIONES Lámina C’’ (TYR 61) – CDR 3 H (TRP 105) – CDR 1 L (TRP 32) Ac humanizado. CD 28 Ac murino – CD 28

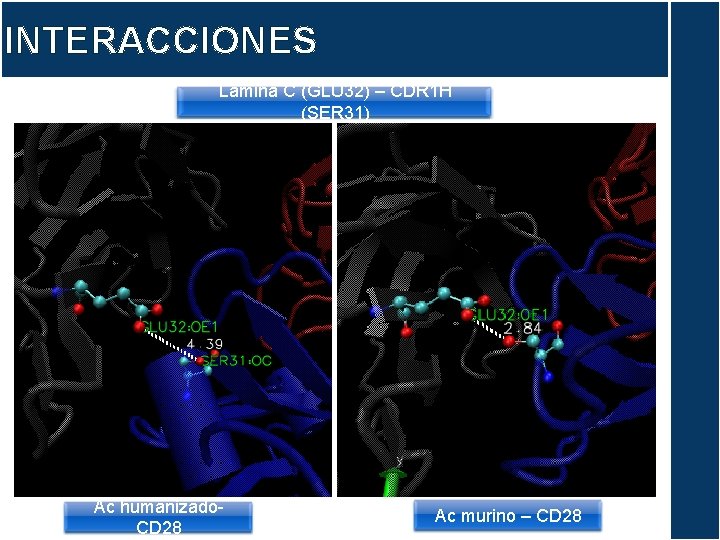
INTERACCIONES Lámina C (GLU 32) – CDR 1 H (SER 31) Ac humanizado. CD 28 Ac murino – CD 28

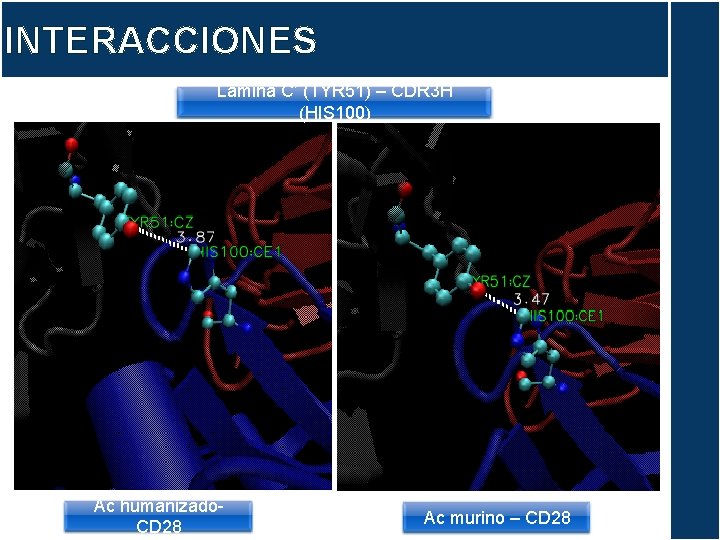
INTERACCIONES Lámina C’ (TYR 51) – CDR 3 H (HIS 100) Ac humanizado. CD 28 Ac murino – CD 28

CONCLUSIONES Hay interacciones que son distintas ? Ha habido cambios en la activación del receptor No podemos confirmarlo ya que el modelo no está optimizado

OBJETIVO 2 OBJETIVO: Establecer diferencias estructurales entre CD 28 humano y los CD 28 de las especies utilizadas en ensayos preclínicos: Macaca fascicularis (Cynomolgus Monkey) Mus musculus (Ratón) Metodología: Comparación de secuencia

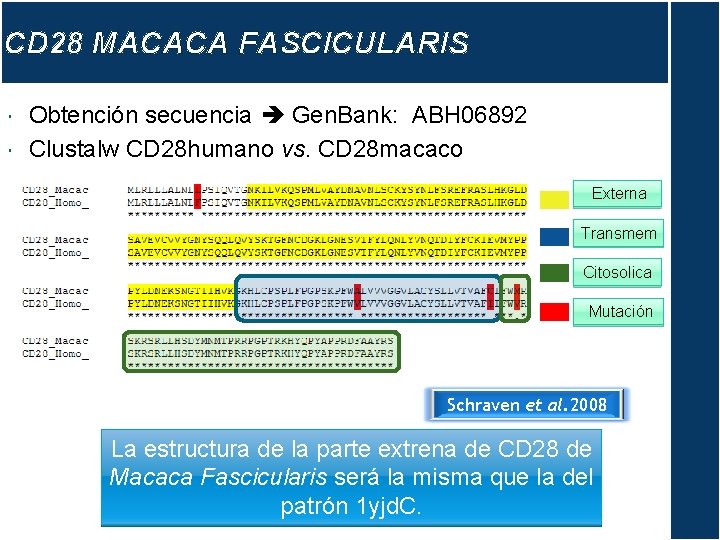
CD 28 MACACA FASCICULARIS Obtención secuencia Gen. Bank: ABH 06892 Clustalw CD 28 humano vs. CD 28 macaco Externa Transmem Citosolica Mutación Schraven et al. 2008 La estructura de la parte extrena de CD 28 de Macaca Fascicularis será la misma que la del patrón 1 yjd. C.

CD 28 MUSCULUS Obtención secuencia Gen. Bank: NP_031668 Clustalw CD 28 humano vs. CD 28 ratón

OBJETIVO 3 Objetivo: Analizar la Fc humana teniendo en cuenta la literatura conocida Fc ¿La unión de la Fc con su receptor podría ser la causa de los efectos adversos? ¿Podría ser que se formaran agregados que amplificarán la señal?

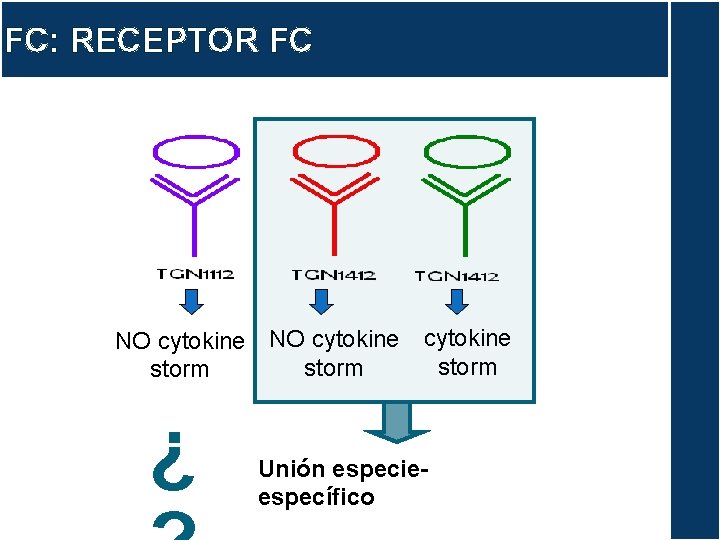
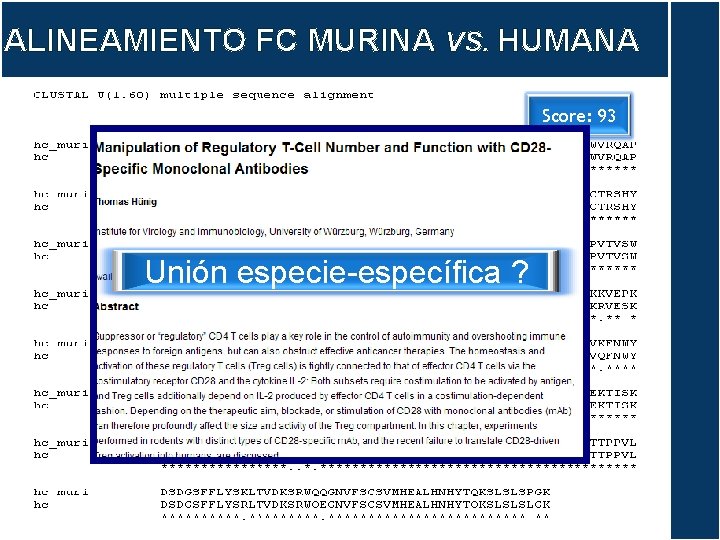
FC: RECEPTOR FC NO cytokine storm ¿ Unión especieespecífico

ALINEAMIENTO HC HUMANA-HC MURINA Imatge aliniament i seleccionar residus diferents + imatge article

ALINEAMIENTO FC MURINA VS. HUMANA Score: 93 Unión especie-específica ?

OBJETIVO 3 Objetivo: Analizar la Fc humana teniendo en cuenta la literatura conocida Fc ¿La unión de la Fc con su receptor podría ser la causa de los efectos adversos? ¿Podría ser que se formaran agregados que amplificarán la señal?

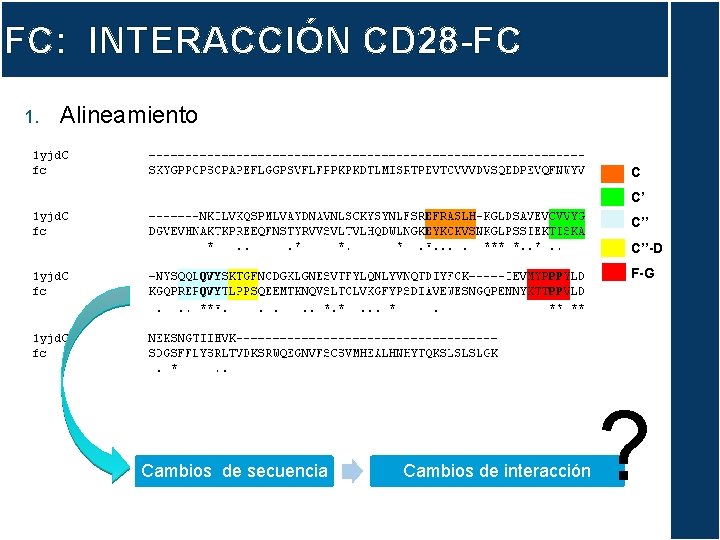
FC: INTERACCIÓN CD 28 -FC 1. Alineamiento C C’ C’’-D F-G Cambios de secuencia Cambios de interacción ?

FC HUMANA Dos patrones : Región Fab HC : 1 YJD (murino) Región Fc HC: pdb 1 HZH (humano) Alineamiento clustalw HC con patrones Modelo basado en secuencia MODELLER Evaluación modelos PROSA PROCHECK

FC HUMANA: Alineamiento Alineamento HC + patrons:

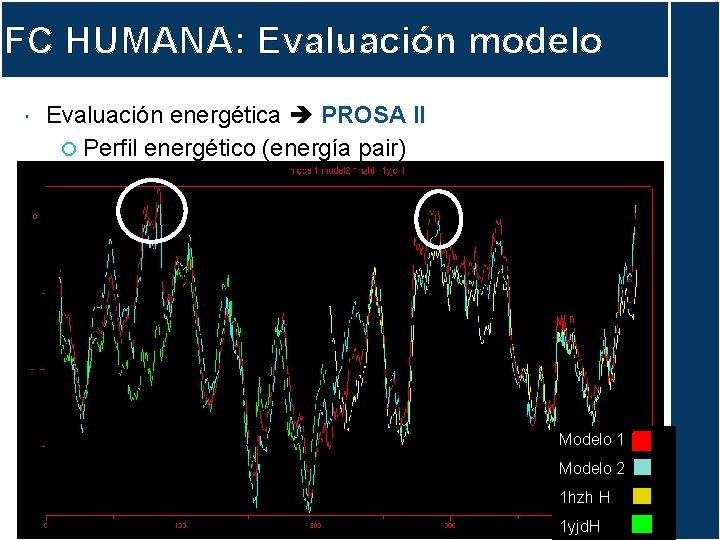
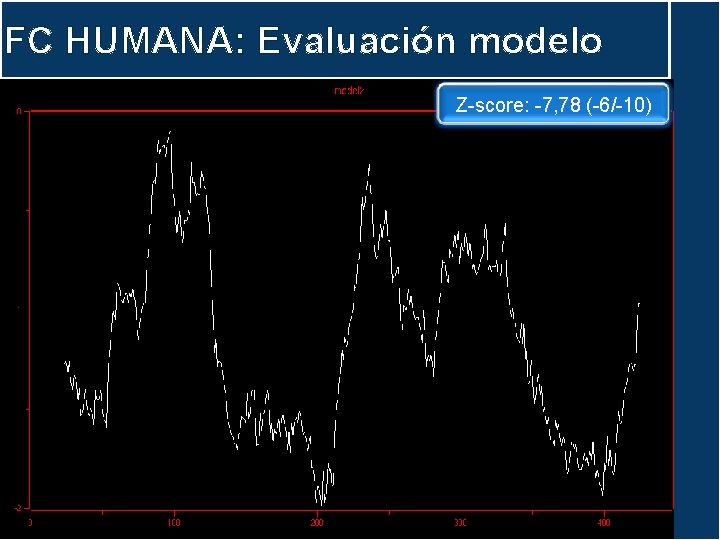
FC HUMANA: Evaluación modelo Evaluación energética PROSA II Perfil energético (energía pair) Modelo 1 Modelo 2 1 hzh H 1 yjd. H

FC HUMANA: Evaluación modelo Z-score: -7, 78 (-6/-10)

FC HUMANA: Evaluación modelos Evaluación estereoquímica PROCHECK

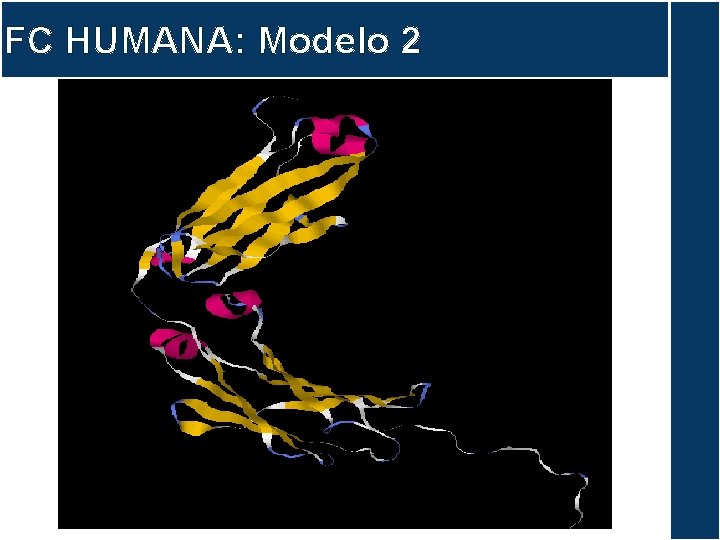
FC HUMANA: Modelo 2

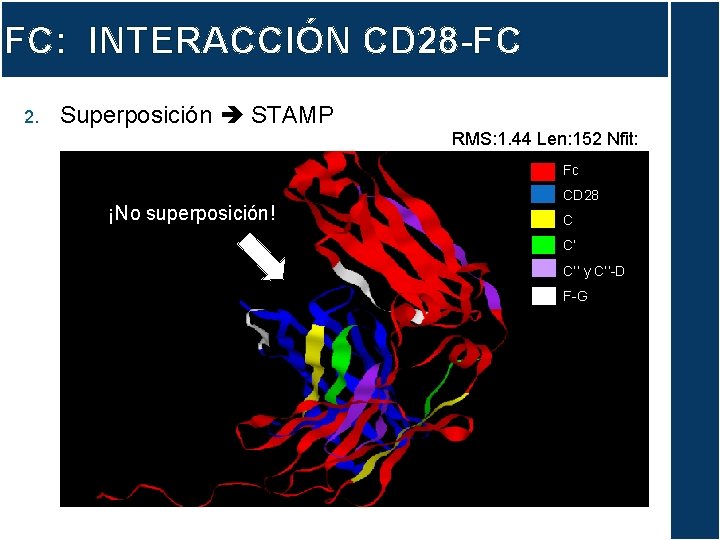
FC: INTERACCIÓN CD 28 -FC 2. Superposición STAMP RMS: 1. 44 Len: 152 Nfit: 19 Fc ¡No superposición! CD 28 C C’ C’’ y C’’-D F-G

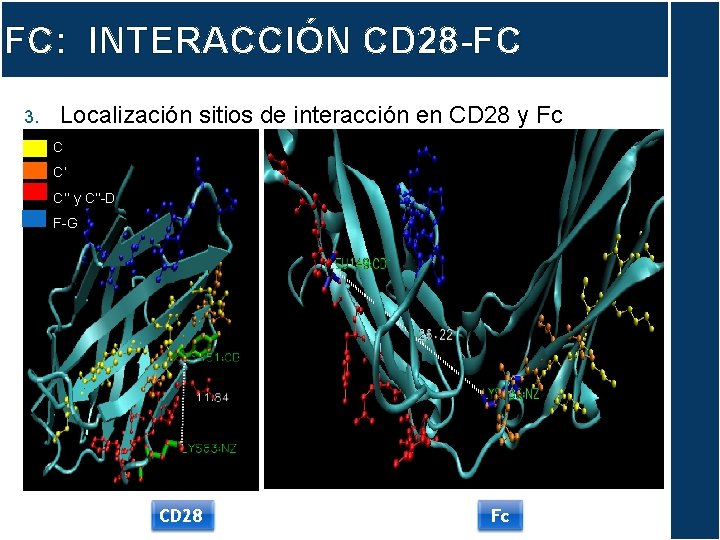
FC: INTERACCIÓN CD 28 -FC 3. Localización sitios de interacción en CD 28 y Fc C C’ C’’ y C’’-D F-G 26, 22 CD 28 Fc

FC: CONCLUSIONES A nivel de secuencia, las zonas de interacción son diferentes A nivel estructural, observamos que la disposición de los residuos de la interacción es diferente Deducimos que el TGN 1412 no se puede autorreconocer Pero si. . . Agregación humanos Agregación macaco ¿Qué pudo evitar la amplificación de la señal en los macacos?

OBJETIVO 4 OBJETIVO: Estudiar y comparar las vías de señalización del Rc CD 28 humano y de macaco con el fin de encontrar alguna diferencia entre ellas que expliquen la mayor activación de linfocitos T en humanos Metodología: Literatura relacionada Bases de datos

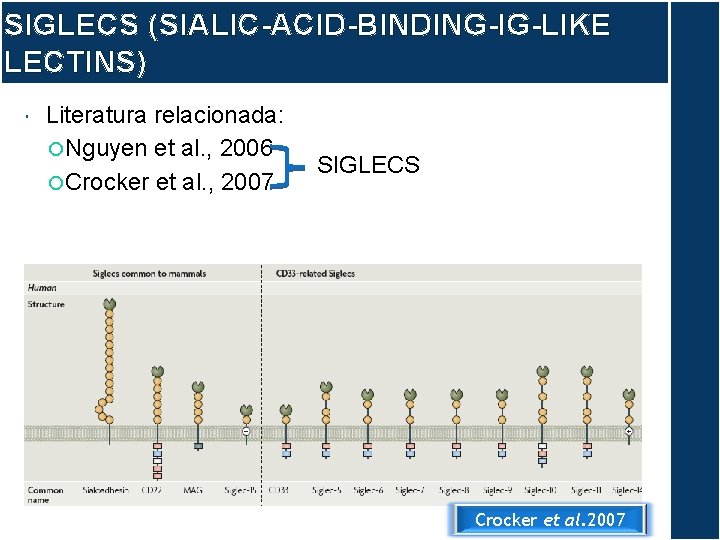
SIGLECS (SIALIC-ACID-BINDING-IG-LIKE LECTINS) Literatura relacionada: Nguyen et al. , 2006 Crocker et al. , 2007 SIGLECS Crocker et al. 2007

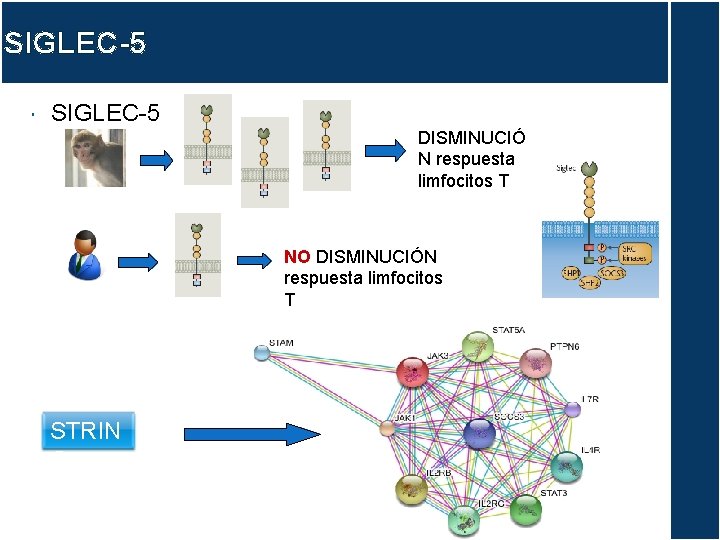
SIGLEC-5 DISMINUCIÓ N respuesta limfocitos T NO DISMINUCIÓN respuesta limfocitos T STRIN G

CONCLUSIONES HIPÓTESIS 1 No podemos aceptar/refutar la hipótesis ya que el modelo no está optimizado. Sin embargo, encontramos diferencias en la interacción de CD 28 con el modelo. HIPÓTESIS 2 CD 28 Macaco: No existen diferencias a nivel externo. No obstante, pueden existir diferencias a nivel de la vía de señalización. CD 28 murino: Existen diferencias a nivel externo que pueden implicar cambios en la interacción.

CONCLUSIONS HIPÓTESIS 3 Con los datos de los que disponemos no podemos confirmar que la unión de la Fc con su receptor sea especie-específica. No podemos confirmar la formación de agregados ya que no hemos observado un posible autoreconocimiento por la región Fc. HIPÓTESIS 4 Existen diferencias en las vías de señalización entre macaco y humano que podrían estar implicados en los efectos adversos observados en humanos.

VH CH CD 28 VL CL Fc

SUPLEMENTO

Fab y Fc: comandas Las comandas que aparecen en las siguientes diapositivas hacen referencia a las utilizadas con la cadena ligera. Sin embargo, hemos seguido el mismo procedimiento a la hora de trabajar con la cadena pesada y la porción Fc del TG 1412.

ALINEAMIENTO BASADO EN SECUENCIA: Clustal. W (1 patrón) 1. Obtener la secuencia en formato fasta y el pdb de la cadena ligera murina del 1 YJD. pdb: /disc 9/PERL/PDBto. Split. Chain. pl -i 1 YJD. pdb -o 1 YJD 2. Concatenar el fichero de la secuencia fasta anterior con el de la cadena ligera humanizada: cat LC_muri. fa >> LC_muri_humanitzat. fa y cat LC_humanitzat. fa >> LC_muri_humanitzat. fa 3. Eliminar el péptido señal de la secuencia fasta de la cadena ligera humanizada: LC_muri_humanitzat_tallat. fa 4. Ejecutar Clustal. W: LC_muri_humanitzat_tallat. aln

ALINEAMIENTO BASADO EN SECUENCIA: Clustal. W (muchos patrones) (I) 1. Buscar homólogos de la proteína problema en la base de datos Swissprot: blastpgp -i LC_humanitzat_tallat. fa -d /disc 9/DB/blast/sprot -j 20 -C LC_humanitzat_tallat_sprot_j 20. pssm -o LC_humanitzat_tallat_sprot_j 20. out 2. Utilizar la matriz de pesos creada con Swissprot para buscar homólogos de la proteína problema en la base de datos PDB: blastpgp -i LC_humanitzat_tallat. fa -d /disc 9/DB/blast/pdb_seq -j 2 -R LC_humanitzat_tallat_sprot_j 20. pssm -C LC_humanitzat_tallat_sprot_to_pdb_j 20_2. pssm -o LC_humanitzat_tallat_sprot_to_pdb_j 20_2. out 3. Copiar los pdbs de aquellos patrones con los valores de e-value más bajos: cp /disc 9/DB/pdb 2 fgw. ent. Z. 4. Descomprimir los ficheros pdb de los patrones elegidos: gunzip *. Z

ALINEAMIENTO BASADO EN SECUENCIA: Clustal. W (muchos patrones) (II) 5. Obtener las secuencias en formato fasta y los pdbs de cada una de las cadenas de los patrones: /disc 9/PERL/PDBto. Split. Chain. pl -i pdb 1 dee. ent -o 1 dee 6. Crear un fichero con las secuencias fasta de los patrones (se incluye la murina) y con la de la cadena ligera humanizada cortada: emacs patrons_LC. fa & 7. Ejecutar Clustal. W: patrons_LC. aln

ALINEAMIENTO BASADO EN SECUENCIA: HMMER (muchos patrones) 1. Buscar en una librería de modelos de Markov a partir de la secuencia problema: hmmpfam /disc 9/DB/pfam/Pfam LC_humanitzat_tallat. fa > LC_humanitzat_tallat_patrons. hmm 2. Extraer el modelo de Markov que contenga el dominio de la secuencia problema: hmmfetch /disc 9/DB/pfam/Pfam ig > LC_humanitzat_tallat_patrons_ig. hmm 3. Crear un alineamiento grande a partir de un alineamiento más pequeño que se encuentra en forma de modelo de Markov: hmmalign -o LC_ig_patrons. ali LC_humanitzat_tallat_patrons_ig. hmm patrons_LC. fa

ALINEAMIENTO BASADO EN ESTRUCTURA: muchos patrones 1. Crear el fichero. domains con los pdbs de los patrones: emacs LC_patrons. domains & 2. Superponer estructuralmente los patrones: stamp -l LC_patrons. domains rough -n 2 -prefix LC_patrons_stamp > LC_patrons_stamp. out 3. Elegir el cluster óptimo y cambiar el alineamiento en vertical que produce STAMP por un formato que nos sea más útil: aconvert -in b -out c <LC_patrons_stamp. 3> LC_patrons_stamp 3. clw 4. Generar un fichero con los pdbs superpuestos para poder visualizar gráficamente esta superposición: transform -f LC_patrons_stamp. 3 -g -o LC_patrons_stamp 3. pdb y rasmol LC_patrons_stamp 3. pdb 5. Construir un modelo de Markov a partir de este alineamiento estructural realizado con STAMP: hmmbuild LC_patrons_stamp 3. hmm LC_patrons_stamp 3. clw 6. Crear un alineamiento grande a partir de uno más pequeño que se encuentra en forma de modelo de Markov: hmmalign -o LC_humanitzat_stamp 3. ali

CONSTRUCCIÓN DEL MODELO: a partir del alineamiento basado en secuencia (1 patrón) Para la construcción del modelo a partir del alineamiento basado en secuencia del Clustal. W con muchos patrones, utilizamos las mismas comandas que aparecen en esta diapositiva. 1. Cambiar el formato del alineamiento original a formato pir: aconvert -in c -out p <LC_muri_humanitzat_tallat. aln> LC_muri_humanitzat_tallat. pir 2. Crear el fichero. top: emacs LC_muri_humanitzat_tallat. top & 3. Ejecutar el programa Modeller: mod LC_muri_humanitzat_tallat. top 4. Renombrar los ficheros. B 99990001 y. B 99990002: con la comanda mv

CONSTRUCCIÓN DEL MODELO: a partir del alineamiento basado en estructura 1. Cambiar el formato del alineamiento original a formato pir: aconvert -in h -out p <LC_humanitzat_stamp 3. ali> model_estructura_patrons. pir 2. Crear el fichero. top: emacs model_estructura_patrons. top & 3. Ejecutar el programa Modeller: model_estructura_patrons. top 4. Renombrar los ficheros. B 99990001 y. B 99990002: con la comanda mv

EVALUACIÓN INTERNA DEL MODELO: Procheck Estas comandas están hechas con uno de los dos modelos obtenidos a partir del alineamiento basado en estructura. Pero sirven de igual modo para el resto de los modelos. 1. Ejecutar el programa Procheck: procheck model 1_estructura_patrons. pdb 3 2. Abrir el fichero de extensión. sum para visualizar los parámetros principales: emacs model 1_estructura_patrons. sum & 3. Abrir el fichero de extensión _01. ps con el programa gimp para ver el mapa de Ramachandran: gimp model 1_estructura_patrons_01. ps

EVALUACIÓN EXTERNA DE LOS MODELOS: Prosa II 1. Escribir las comandas necesarias en un fichero de extensión. cmd: emacs prosa_pair. cmd & read pdb 1 dee. A. pdb 1 dee color pair 2 fgw green read pdb 1 fvd. A. pdb 1 fvd color pair LC_muri white read pdb 1 kb 5 L. pdb 1 kb 5 plot read pdb 2 fgw. L. pdb 2 fgw init zscore read pdb LC_muri combine type sdev analyse energy * zscore 1 dee z-result_patrons_pair winsize * 20 sorted_depth = 0 draw pair * 1 zscore 1 fvd z-result_patrons_pair draw comb * 0 zscore 1 kb 5 z-result_patrons_pair draw surf * 0 zscore 2 fgw z-result_patrons_pair color pair 1 fvd cyan zscore LC_muri zresult_patrons_pair color pair 1 kb 5 red 2. Ejecutar el programa Prosa II: prosa_pair. cmd

SUPERPOSICIÓN DEL MODELO CON EL PDB 1 YJD Estas comandas están hechas con uno de los cuatro modelos obtenidos a partir del alineamiento basado en secuencia. Pero sirven de igual modo para el resto de los modelos. 1. Crear el fichero. domains de modo que contenga el pdb del modelo y el del 1 YJD: emacs stamp_mod 1 seq_patrons. domains & 2. Superponer estructuralmente el pdb 1 YJD y el del modelo con STAMP: stamp -l stamp_mod 1 seq_patrons. domains -rough -n 2 -prefix stamp_mod 1 seq_patrons > stamp_mod 1 seq_patrons. out 3. Generar un fichero con los pdbs superpuestos para poder visualizar gráficamente esta superposición: transform -f stamp_mod 1 seq_patrons. 1 -g -o stamp_mod 1 seq_patrons. pdb

COMPARACIÓN DE ESTRUCTURAS SECUNDARIAS 1. Ejecutar el programa Psi-pred para que haga una predicción de la estructura secundaria de la secuencia problema: psipred LC_humanitzat_tallat. fa 1. Cambiar el formato del fichero. ss 2 a formato pir: psipred. pl LC_humanitzat_tallat. ss 2 > LC_humanitzat_tallat_ss 2. pir 1. Obtener la estructura secundaria del modelo: dssp model 2_estructura_patrons. pdb model 2_estructura_patrons. dssp 1. Pasar la estructura secundaria del modelo a formato fasta: aliss. pl model 2_estructura_patrons. dssp > model 2_estructura_patrons_dssp. pir 1. Concatenar los dos ficheros de salida anteriores en un solo fichero: cat LC_humanitzat_tallat_ss 2. pir model 2_estructura_patrons. dssp >> compare_mod 2_estruct_pat_ss 2_dssp. pir 1. Convertir el fichero anterior a formato Clustal. W: aconvert -in p -out c <compare_mod 2_estruct_pat_ss 2_dssp. pir> compare_mod 2_estruct_pat_ss 2_dssp. clw

BIBLIOGRAFÍA Armour KL, van de Winkel JG, Williamson LM, Clark MR. Differential binding to human fcgammariia and fcgammariib receptors by human igg wildtype and mutant antibodies. Mol Immunol. 2003 Dec; 40(9): 585 -93 Angata T, Varki A. Siglecs. The major subfamily of type-I lectins. Glycobiology. 2006 Jan; 16(1): 1 -27. Blasius AL, Cella M, Maldonado J, Takai T, Colonna M. Siglec-H is an IPC-specific receptor that modulates type I IFN secretion through DAP 12. Blood. 2006 Mar 15; 107(6): 2474 -6. Can super-antibody drugs be tamed? Nature. 2006 Apr 13; 440(7086): 855 -6. Crocker PR. , Paulson JC, Varki A. Siglecs and their roles in the immune system. Nature Reviews. 2007; 7: 255 -266. Evans EJ, Esnouf RM, Manso-Sancho R, Gilbert RJ, James JR, Yu C, Fennelly JA, Vowles C, Hanke T, Walse B, Hünig T, Sørensen P, Stuart DI, Davis SJ. Crystal structure of a soluble CD 28 -Fab complex. Nat Immunol. 2005 Mar; 6(3): 271 -9. Epub 2005 Feb 6. Farzaneh L, Kasahara N, Farzaneh F. The strange case of TGN 1412. Cancer Immunol Immunother. 2007 Feb; 56(2): 129 -34. Epub 2006 Jun 17.

BIBLIOGRAFIA Fiser A, Sali A. Comparative protein structure modeling. Pels Family Center for Biochemistry and Structural Biology. The Rockefeller University. Hansen S, Leslie RG. TGN 1412: scrutinizing preclinical trials of antibodybased medicines. Nature. 2006 May 18; 441(7091): 282. Hünig T. Manipulation of regulatory T-cell number and function with CD 28 specific monoclonal antibodies. Adv Immunol. 2007; 95: 111 -48. Kalinke U, Schraven B. CD 28 superagonists: what makes the difference in humans? Immunity. 2008 May; 28(5): 591 -5. Lühder F, Huang Y, Dennehy KM, Guntermann C, Müller I, Winkler E, Kerkau T, Ikemizu S, Davis SJ, Hanke T, Hünig T. Topological requirements and signaling properties of T cell-activating, anti-CD 28 antibody superagonists. J Exp Med. 2003 Apr 21; 197(8): 955 -66. Mathews, Van Holde, Ahern. Bioquímica 3ªed. Addison Wesley, 2002.

BIBLIOGRAFÍA Orozco M. La determinación de la estructura de proteínas en la era genómica. Departamento de Bioquímica y Biología Molecular, de la Facultad de Química de la Universidad de Barcelona; 5 de julio de 2000. Schneider CK, Kalinke U, Löwer J. TGN 1412 --a regulator's perspective. Nat Biotechnol. 2006 May; 24(5): 493 -6. Tema 7: Plegamiento de proteínas [acceso 19 de mayo de 2008] [fecha de la última actualización 23 de septiembre de 2003] URL disponible en: http: //mmb. pcb. ub. es/em/PDF/TEMA 7. pdf

PREGUNTAS Respecto a las Siglec, elige la respuesta correcta. a) Se trata de proteínas transmembrana de la familia de las lectinas. b) Se expresan de forma diferente en humanos y simios. c) Las dos anteriores. d) Podrían explicar el hecho de que en macacos no se produjera la citokine storm. e) Todas las anteriores. Respecto a las Siglec, elige la respuesta correcta. a) En general, en el dominio citoplasmático tienen motivos ITIM. b) Se expresan en gran cantidad en linfocitos T humanos. c) Regulan la producción de neurotransmisores. d) Todas las anteriores son correctas. e) Todas las anteriores son falsas.

PREGUNTAS ¿Cuál de los siguientes epitopos se corresponden con un anticuerpo anti. CD 28 convencial y cual con un superagonista, respectivamente? a) Loop FG y motivo MYPPPY b) Loop FG y loop C’’-D c) Motivo MYPPPY y loop C’’-D d) Las dos anteriores e) Loop C’’-D y motivo MYPPPY El anticuerpo TGN 1412 es un superagonista porque: a) Contiene la región Fc correspondiente a un anticuerpo. b) Es capaz de actuar sinérgicamente con el receptor TCR para activar los linfocitos T. c) Debido a que es capaz de producir una activación del linfocito T cien veces mayor que el anticuerpo monoclonal convencial. d) Activa al linfocito T sin la necesidad de la activación del TCR. e) Ninguna de las anteriores.

PREGUNTAS Quina de les següents afirmacions és certa? a) Farem l’avaluació del model construït mitjançant procheck i escollirem sempre la resolució que sigui més elevada de tots els patrons. b) Per avaluar el model de la Fab de l’anticòs és recomanable analitzar el perfil energètic de l’energia combinada. c) Les dues anteriors. d) El millor alineament basat en seqüència per a la construcció dels models de les cadenes de la Ig és l’obtingut amb HMMER utilitzant el domini Ig extret de Pfam. e) Totes les anteriors. De las siguientes afirmaciones, señale la verdadera: a) Las técnicas bioinformáticas de predicción de estructuras de las proteínas se basan en dos aproximaciones diferentes, pero complementarias: los métodos físicos y los métodos estadísticos. b) Hay métodos teóricos y experimentales para predecir la estructura de una proteína como comparative modelling y cristalografía, respectivamente. c) Las dos anteriores son ciertas. d) La calidad de un patrón aumenta cuando más similar sea su secuencia a la de la secuencia diana y disminuye con el número y la longitud de los gaps en el alineamiento. e) Todas las anteriores son ciertas.

PREGUNTAS Dentro de las interacciones no enlazantes, señale el orden que seguirían de más a menos intensas: a) Puentes de hidrógeno > puentes disulfuro > interacciones hidrofóbicas > interacciones electrostáticas. b) Puentes disulfuro > puentes salinos > puentes de hidrógeno > interacciones de van der Waals. c) Interacciones iónicas > puentes de hidrógeno > interacciones de van der Waals. d) Interacciones electrostáticas > puentes disulfuro > interacciones hidrofóbicas > puentes de hidrógeno. e) Puentes disulfuro > puentes de hidrógeno > interacciones de van der Waals > interacciones iónicas. Señala la respuesta correcta: a) Proteínas con la misma secuencia, la misma estructura y la misma función se denominan homólogos ortólogos. b) Proteínas con secuencias y funciones distintas y que se pliegan de la misma manera se denominan análogos. c) Las dos anteriores. d) Proteínas con la misma secuencia, la misma estructura y función diferente se denominan parálogos. e) Todas las anteriores.

PREGUNTAS Respecte les immunoglobulines, quina de les següents opcions és la falsa: a) b) c) d) e) a) El domini constant està format per 7 fulles β repartides en dos grups de 3 i 4 cadenes. b) La fulla de 5 cadenes β del domini variable és estructuralment homologa a la fulla de 3 cadenes β del domini constant però amb dues cadenes extres C’ i C’’. c) El plegament s’estabilitza per la formació de ponts d’hidrogen entre les cadenes β de cada fulla, d’enllaços hidrofòbics entre els residus de l’interior de les fulles i per un pont de sofre entre les cadenes B i F. d) Els CDRs són loops que connecten les cadenes β 1 -2, 3 -3 i 4 -5. e) La unió de carbohidrats a l’Asn 306 del segon domini constant de cada cadena pesada (CH 2) produeix un augment de la protuberància del peu de la Y. Respecte al modelat de proteïnes, quina de les següents afirmacions és veritat: En la optimització, la conformació biologicament activa es correspon sempre a la mínima energia. Les zones corresponents als centres catalítics de les proteïnes poden presentar pics energètics més positius degut a la seva menor estabilitat. Les dues anteriors són certes. Un bon modelat no implica sempre un perfil energètic negatiu. La b i la d són certes.
 1412
1412 Pon tres ejemplos de: a) pilares; b) vigas; c) tirantes.
Pon tres ejemplos de: a) pilares; b) vigas; c) tirantes. Porque
Porque Por que es importante la planificacion
Por que es importante la planificacion La educacion civica
La educacion civica Que el tacto
Que el tacto Para ahorrar agua yo
Para ahorrar agua yo Una imagen de reducir
Una imagen de reducir Estructura del presupuesto por programas
Estructura del presupuesto por programas Estructura del presupuesto por programas
Estructura del presupuesto por programas Presupuesto icon
Presupuesto icon Cigoto
Cigoto Textura
Textura Estructura tpm
Estructura tpm Juan 17:20-21
Juan 17:20-21 Porque es importante el agua
Porque es importante el agua 1 timoteo 3 16
1 timoteo 3 16 Por que es un texto literario
Por que es un texto literario Jogador de futebol de um importante time espanhol
Jogador de futebol de um importante time espanhol El superlativo relativo
El superlativo relativo Mateo 28 16 20 para niños
Mateo 28 16 20 para niños Personajes de la etnia negra
Personajes de la etnia negra Manejo de cifras significativas
Manejo de cifras significativas Administracion financiera definicion
Administracion financiera definicion Eres alguien muy importante quierete
Eres alguien muy importante quierete Se o passado fosse importante
Se o passado fosse importante Patron respiratorio biot
Patron respiratorio biot Los inventos mas importantes de la historia
Los inventos mas importantes de la historia Quiero brindar por mi gente sencilla
Quiero brindar por mi gente sencilla Kahalagahan ng tubig
Kahalagahan ng tubig Filastrocca del piccolo gesto importante
Filastrocca del piccolo gesto importante Es importante que ustedes (limpiar) los estantes
Es importante que ustedes (limpiar) los estantes Participante pasivo
Participante pasivo Imagenes de sanidad interior
Imagenes de sanidad interior Gamit ng pananaliksik
Gamit ng pananaliksik Nucleo de una oracion
Nucleo de una oracion Ejemplo de una matriz de consistencia
Ejemplo de una matriz de consistencia Lo mas importante
Lo mas importante Vestimentas cristianas
Vestimentas cristianas Porque es importante la familia
Porque es importante la familia Como subrayar apuntes
Como subrayar apuntes Tu asistencia es importante
Tu asistencia es importante Lo mas importante
Lo mas importante La nota no es lo importante
La nota no es lo importante Señales reglamentarias
Señales reglamentarias O que é realmente amar
O que é realmente amar Le cose che ho imparato nella vita
Le cose che ho imparato nella vita Silaba tonica de pajaro
Silaba tonica de pajaro Dio ti ama personalmente
Dio ti ama personalmente Lo importante acerca del dinero
Lo importante acerca del dinero O que é mais importante amar ou ser amado
O que é mais importante amar ou ser amado Lo mas importante
Lo mas importante Dato importante sobre
Dato importante sobre Qué es más importante amar o ser amado
Qué es más importante amar o ser amado Enem 2018 outra importante manifestação das crenças
Enem 2018 outra importante manifestação das crenças Porque es importante el sistema circulatorio
Porque es importante el sistema circulatorio Porque es importante el codex alimentarius
Porque es importante el codex alimentarius Grupo funcional de etanol
Grupo funcional de etanol Imágenes de amar
Imágenes de amar Sensaes
Sensaes Es importante
Es importante Las personas valen por lo que son y no por lo que tienen
Las personas valen por lo que son y no por lo que tienen Diferencia de conjuntos
Diferencia de conjuntos Gracias dios por morir por mi
Gracias dios por morir por mi Complemento de un conjunto ejemplo
Complemento de un conjunto ejemplo Adivina adivinanza que se pela por la panza
Adivina adivinanza que se pela por la panza Pimpinela hoy estamos festejando
Pimpinela hoy estamos festejando Premonencia
Premonencia Por fuera muy colorido
Por fuera muy colorido Por que este hombre caminaba por el bosque
Por que este hombre caminaba por el bosque Quiero agradecerte dios gracias por tu amor por tu perdon
Quiero agradecerte dios gracias por tu amor por tu perdon Esforzaos a entrar por la puerta
Esforzaos a entrar por la puerta Gênero textual entrevista
Gênero textual entrevista Castigo eterno
Castigo eterno Siempre rezo por ti
Siempre rezo por ti Te pondre por cabeza y no por cola
Te pondre por cabeza y no por cola Adonde vas ahora a la clase de matematicas
Adonde vas ahora a la clase de matematicas Vinillo de rioja
Vinillo de rioja Frases de los libros sapienciales
Frases de los libros sapienciales Clasificación de la vitamina d
Clasificación de la vitamina d Nh4 estructura de lewis
Nh4 estructura de lewis Omega 3 y 6 estructura quimica
Omega 3 y 6 estructura quimica Future perfect simple estructura
Future perfect simple estructura Oraciones interrogativas indirectas ejemplos
Oraciones interrogativas indirectas ejemplos Estructura vertical de la organización de ventas
Estructura vertical de la organización de ventas Organizacion social de los aztecas
Organizacion social de los aztecas Estructura en cascada
Estructura en cascada Triglicerido estructura
Triglicerido estructura Estructura would rather
Estructura would rather Joan salvat papasseit tot l'enyor de demà
Joan salvat papasseit tot l'enyor de demà Que tipo de texto es una invitacion de cumpleaños
Que tipo de texto es una invitacion de cumpleaños Clasificación de textos instructivos
Clasificación de textos instructivos Estructuras de un texto
Estructuras de un texto Estructura del sintagma adjetival
Estructura del sintagma adjetival Poder present progressive
Poder present progressive Diferencia entre textos persuasivos y funcionales
Diferencia entre textos persuasivos y funcionales Un texto narrativo de
Un texto narrativo de Cuerpo de un texto informativo
Cuerpo de un texto informativo Registro informal ejemplos
Registro informal ejemplos Nudo narrativo
Nudo narrativo Que es un texto narrativo y su estructura
Que es un texto narrativo y su estructura Que es el discurso expositivo
Que es el discurso expositivo Estructura interna y externa de los textos expositivos
Estructura interna y externa de los textos expositivos