Polimorfismo del DNA qualsiasi variante di sequenza non



















- Slides: 41


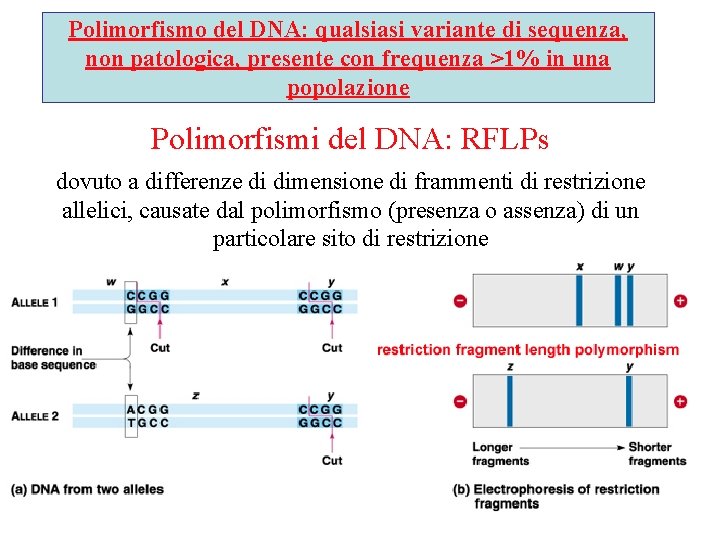
Polimorfismo del DNA: qualsiasi variante di sequenza, non patologica, presente con frequenza >1% in una popolazione Polimorfismi del DNA: RFLPs dovuto a differenze di dimensione di frammenti di restrizione allelici, causate dal polimorfismo (presenza o assenza) di un particolare sito di restrizione


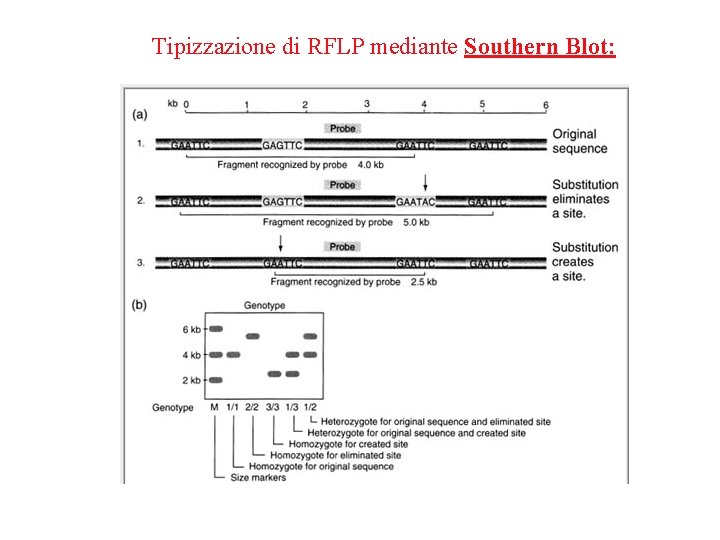
Tipizzazione di RFLP mediante Southern Blot:

Analisi degli RFLP mediante PCR:

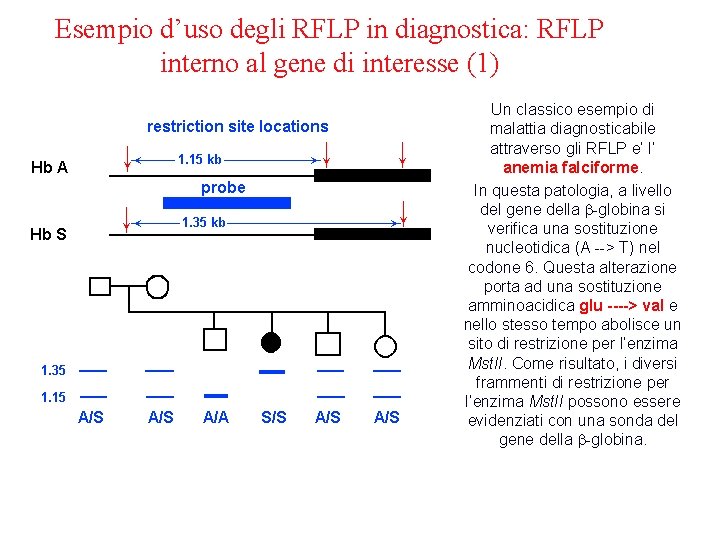
Esempio d’uso degli RFLP in diagnostica: RFLP interno al gene di interesse (1) Un classico esempio di malattia diagnosticabile attraverso gli RFLP e’ l’ anemia falciforme. In questa patologia, a livello del gene della b-globina si verifica una sostituzione nucleotidica (A --> T) nel codone 6. Questa alterazione porta ad una sostituzione amminoacidica glu ----> val e nello stesso tempo abolisce un sito di restrizione per l’enzima Mst. II. Come risultato, i diversi frammenti di restrizione per l’enzima Mst. II possono essere evidenziati con una sonda del gene della b-globina.

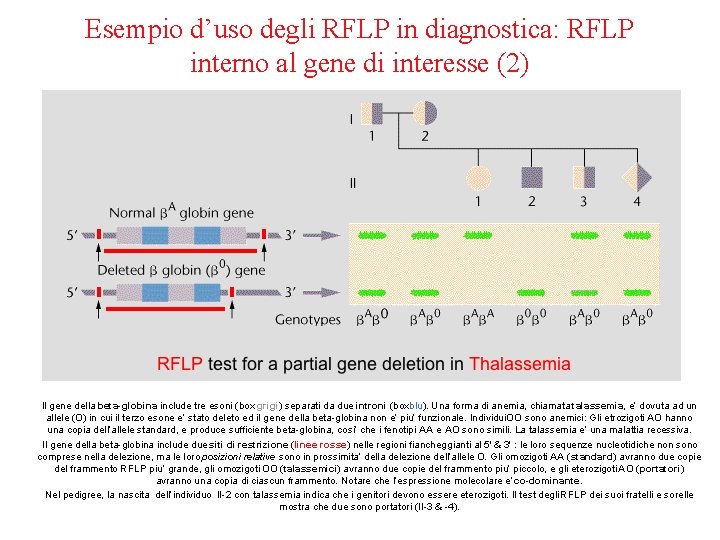
Esempio d’uso degli RFLP in diagnostica: RFLP interno al gene di interesse (2) Il gene della beta-globina include tre esoni (box grigi) separati da due introni (boxblu). Una forma di anemia, chiamata talassemia, e’ dovuta ad un allele (O) in cui il terzo esone e’ stato deleto ed il gene della beta-globina non e’ piu’ funzionale. Individui. OO sono anemici: Gli etrozigoti AO hanno una copia dell’allele standard, e produce sufficiente beta-globina, cosi’ che i fenotipi AA e AO sono simili. La talassemia e’ una malattia recessiva. Il gene della beta-globina include due siti di restrizione (linee rosse) nelle regioni fiancheggianti al 5' & 3' : le loro sequenze nucleotidiche non sono comprese nella delezione, ma le loro posizioni relative sono in prossimita’ della delezione dell’allele O. Gli omozigoti AA (standard) avranno due copie del frammento RFLP piu’ grande, gli omozigoti OO (talassemici) avranno due copie del frammento piu’ piccolo, e gli eterozigoti AO (portatori) avranno una copia di ciascun frammento. Notare che l’espressione molecolare e’ co-dominante. Nel pedigree, la nascita dell’individuo II-2 con talassemia indica che i genitori devono essere eterozigoti. Il test degli RFLP dei suoi fratelli e sorelle mostra che due sono portatori (II-3 & -4).


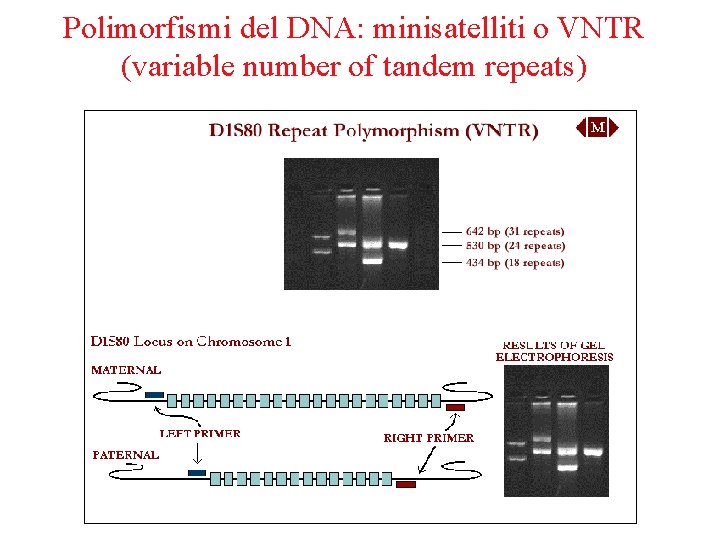
Polimorfismi del DNA: minisatelliti o VNTR (variable number of tandem repeats)

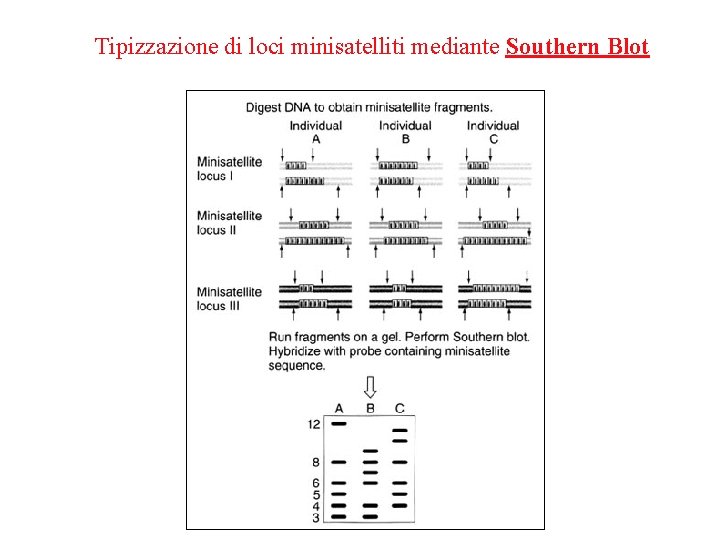
Tipizzazione di loci minisatelliti mediante Southern Blot

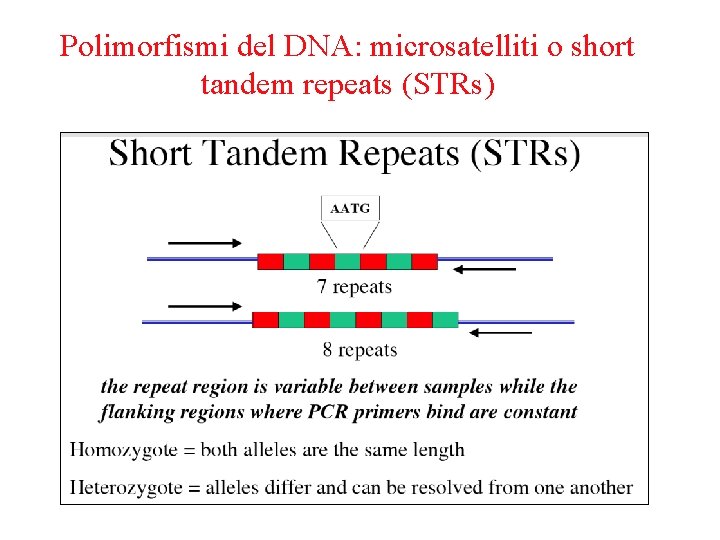
Polimorfismi del DNA: microsatelliti o short tandem repeats (STRs)

Tipizzazione di loci microsatelliti mediante PCR

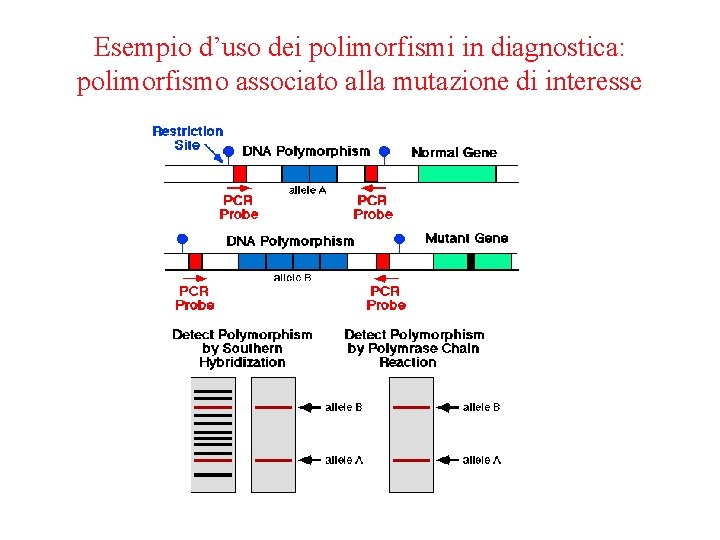
Esempio d’uso dei polimorfismi in diagnostica: polimorfismo associato alla mutazione di interesse

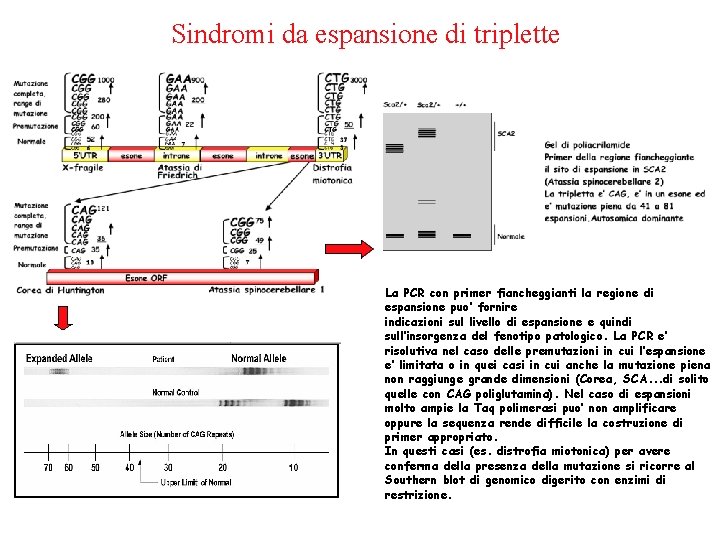
Sindromi da espansione di triplette La PCR con primer fiancheggianti la regione di espansione puo’ fornire indicazioni sul livello di espansione e quindi sull’insorgenza del fenotipo patologico. La PCR e’ risolutiva nel caso delle premutazioni in cui l’espansione e’ limitata o in quei casi in cui anche la mutazione piena non raggiunge grande dimensioni (Corea, SCA. . . di solito quelle con CAG poliglutamina). Nel caso di espansioni molto ampie la Taq polimerasi puo’ non amplificare oppure la sequenza rende difficile la costruzione di primer appropriato. In questi casi (es. distrofia miotonica) per avere conferma della presenza della mutazione si ricorre al Southern blot di genomico digerito con enzimi di restrizione.

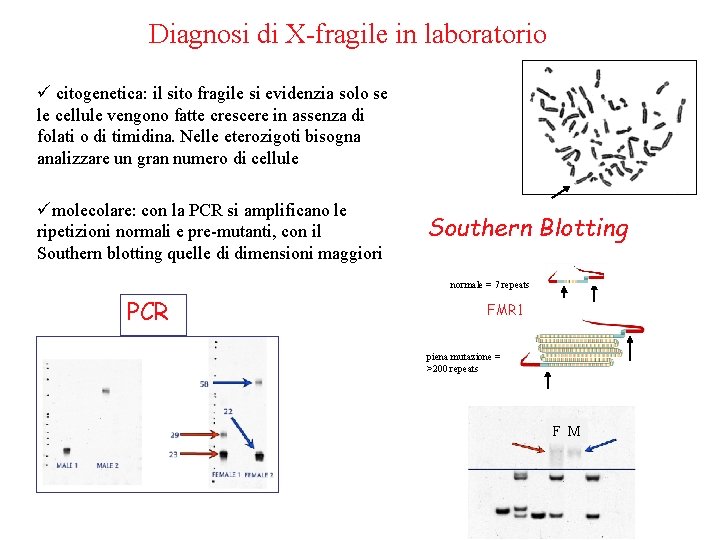
Diagnosi di X-fragile in laboratorio ü citogenetica: il sito fragile si evidenzia solo se le cellule vengono fatte crescere in assenza di folati o di timidina. Nelle eterozigoti bisogna analizzare un gran numero di cellule ümolecolare: con la PCR si amplificano le ripetizioni normali e pre-mutanti, con il Southern blotting quelle di dimensioni maggiori Southern Blotting normale = 7 repeats PCR FMR 1 piena mutazione = >200 repeats F M



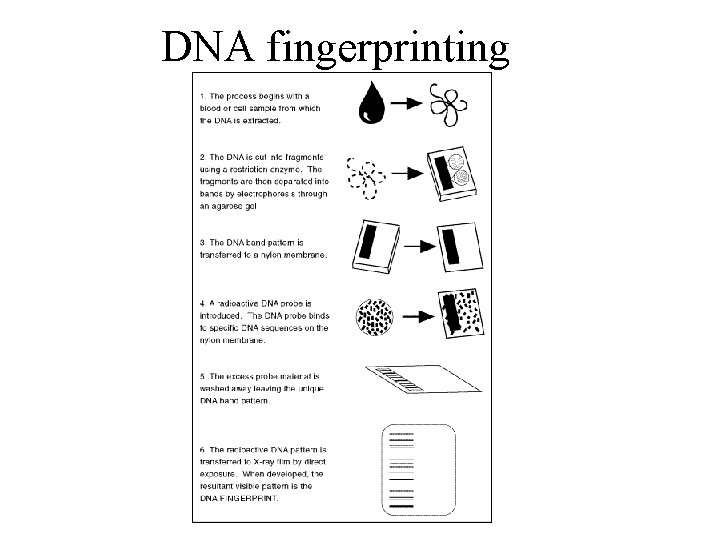
DNA fingerprinting


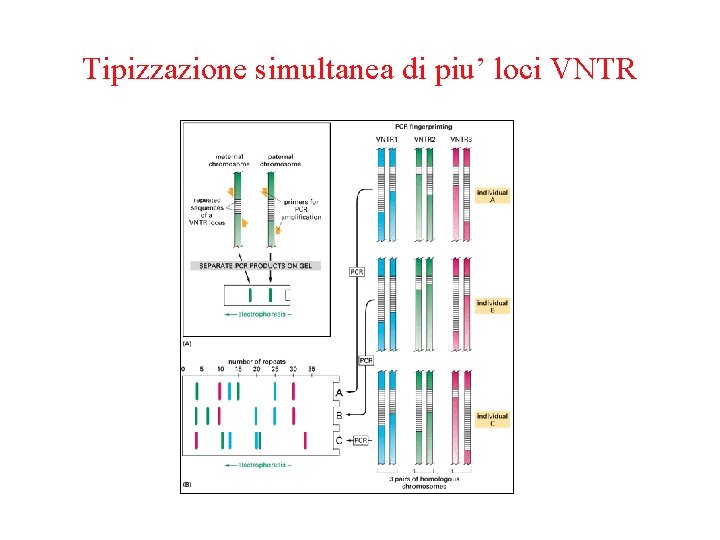
Tipizzazione simultanea di piu’ loci VNTR






Applicazioni pratiche del Fingerprinting del DNA 1. Paternity and Maternity Because a person inherits his or her VNTRs from his or her parents, VNTR patterns can be used to establish paternity and maternity. The patterns are so specific that a parental VNTR pattern can be reconstructed even if only the children's VNTR patterns are known (the more children produced, the more reliable the reconstruction). Parentchild VNTR pattern analysis has been used to solve standard father-identification cases as well as more complicated cases of confirming legal nationality and, in instances of adoption, biological parenthood. 2. Criminal Identification and Forensics DNA isolated from blood, hair, skin cells, or other genetic evidence left at the scene of a crime can be compared, through VNTR patterns, with the DNA of a criminal suspect to determine guilt or innocence. VNTR patterns are also useful in establishing the identity of a homicide victim, either from DNA found as evidence or from the body itself. 3. Personal Identification The notion of using DNA fingerprints as a sort of genetic bar code to identify individuals has been discussed, but this is not likely to happen anytime in the foreseeable future. The technology required to isolate, keep on file, and then analyze millions of very specified VNTR patterns is both expensive and impractical. Social security numbers, picture ID, and other more mundane methods are much more likely to remain the prevalent ways to establish personal identification.

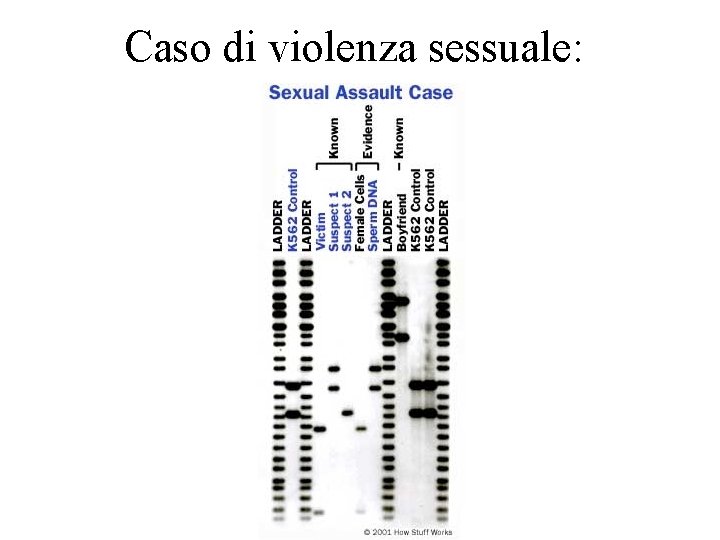
Caso di violenza sessuale:

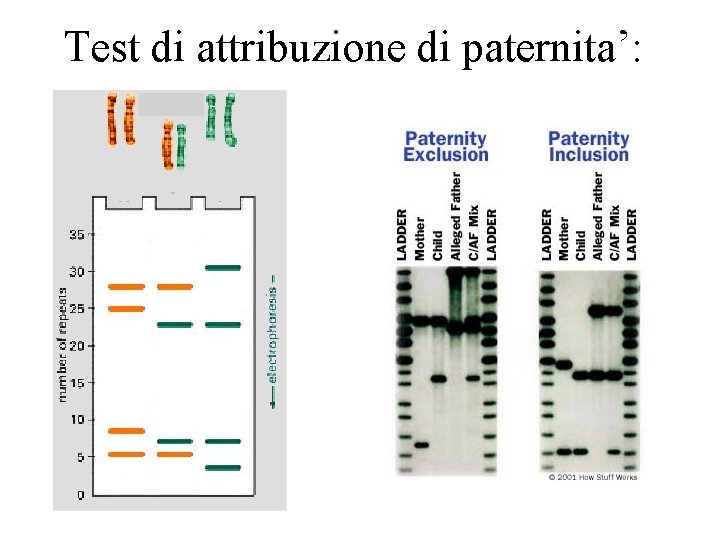
Test di attribuzione di paternita’:

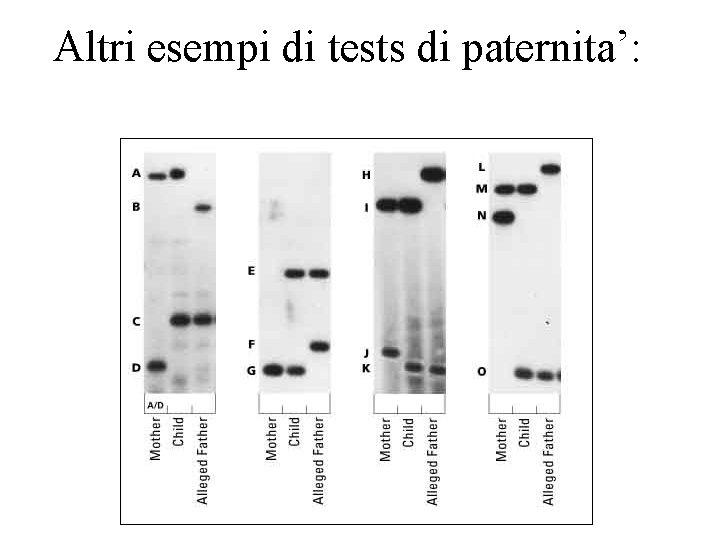
Altri esempi di tests di paternita’:

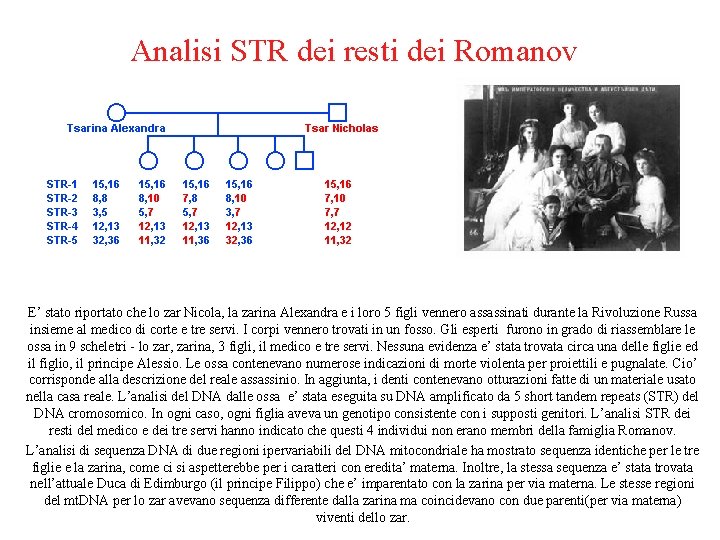
Analisi STR dei resti dei Romanov E’ stato riportato che lo zar Nicola, la zarina Alexandra e i loro 5 figli vennero assassinati durante la Rivoluzione Russa insieme al medico di corte e tre servi. I corpi vennero trovati in un fosso. Gli esperti furono in grado di riassemblare le ossa in 9 scheletri - lo zar, zarina, 3 figli, il medico e tre servi. Nessuna evidenza e’ stata trovata circa una delle figlie ed il figlio, il principe Alessio. Le ossa contenevano numerose indicazioni di morte violenta per proiettili e pugnalate. Cio’ corrisponde alla descrizione del reale assassinio. In aggiunta, i denti contenevano otturazioni fatte di un materiale usato nella casa reale. L’analisi del DNA dalle ossa e’ stata eseguita su DNA amplificato da 5 short tandem repeats (STR) del DNA cromosomico. In ogni caso, ogni figlia aveva un genotipo consistente con i supposti genitori. L’analisi STR dei resti del medico e dei tre servi hanno indicato che questi 4 individui non erano membri della famiglia Romanov. L’analisi di sequenza DNA di due regioni ipervariabili del DNA mitocondriale ha mostrato sequenza identiche per le tre figlie e la zarina, come ci si aspetterebbe per i caratteri con eredita’ materna. Inoltre, la stessa sequenza e’ stata trovata nell’attuale Duca di Edimburgo (il principe Filippo) che e’ imparentato con la zarina per via materna. Le stesse regioni del mt. DNA per lo zar avevano sequenza differente dalla zarina ma coincidevano con due parenti(per via materna) viventi dello zar.

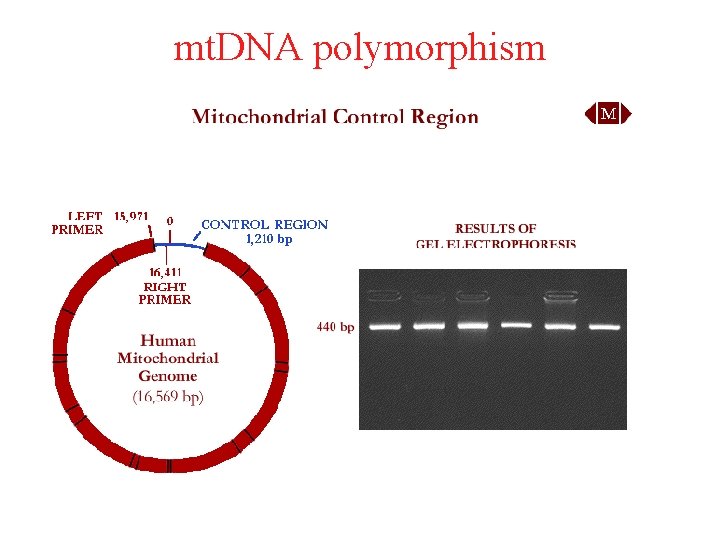
mt. DNA polymorphism

SNP (single nucleotide polymorphism) variazione polimorfica in un unico nucleotide (1 ogni 1000 pb) RFLP altri polimorfismi che non alterano alcun sito di restrizione

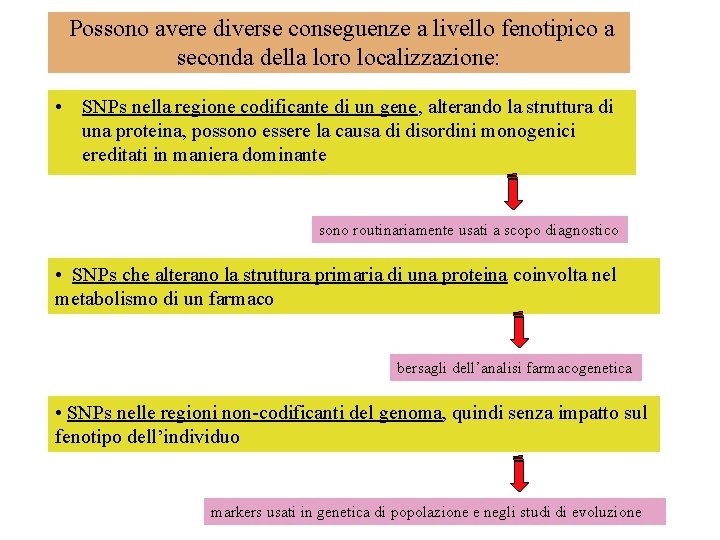
Possono avere diverse conseguenze a livello fenotipico a seconda della loro localizzazione: • SNPs nella regione codificante di un gene, alterando la struttura di una proteina, possono essere la causa di disordini monogenici ereditati in maniera dominante sono routinariamente usati a scopo diagnostico • SNPs che alterano la struttura primaria di una proteina coinvolta nel metabolismo di un farmaco bersagli dell’analisi farmacogenetica • SNPs nelle regioni non-codificanti del genoma, quindi senza impatto sul fenotipo dell’individuo markers usati in genetica di popolazione e negli studi di evoluzione


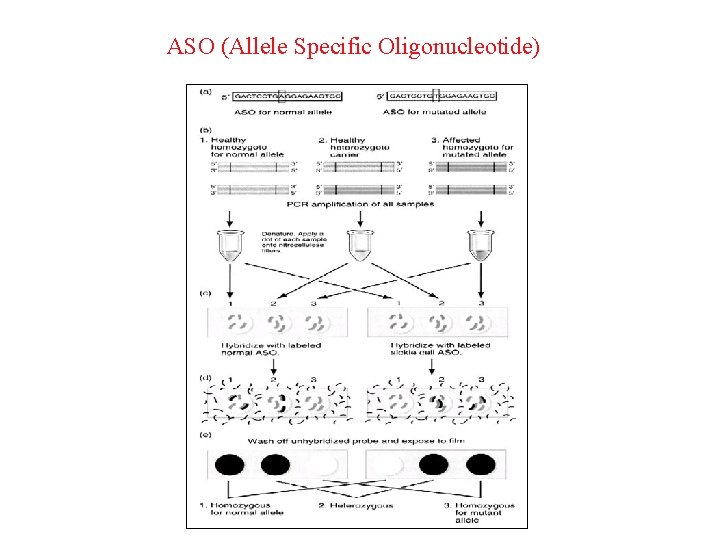
ASO (Allele Specific Oligonucleotide)

Esempio d’uso dell’ASO: detection della mutazione nel gene della beta-globina nell’anemia falciforme

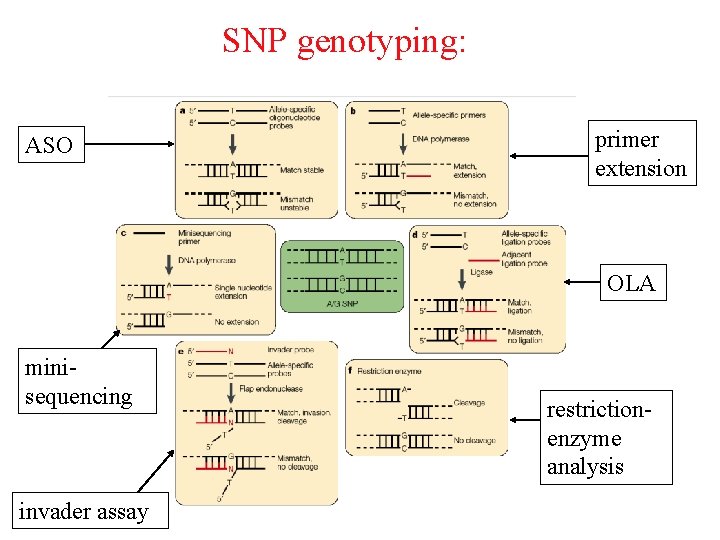
SNP genotyping: ASO primer extension OLA minisequencing invader assay restrictionenzyme analysis

Variante dell’ASO: REVERSE DOT BLOT ANALYSIS

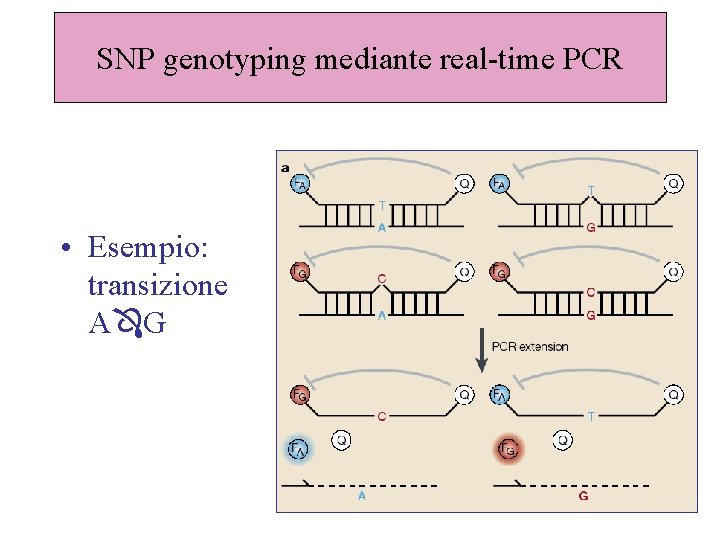
SNP genotyping mediante real-time PCR • Esempio: transizione A G


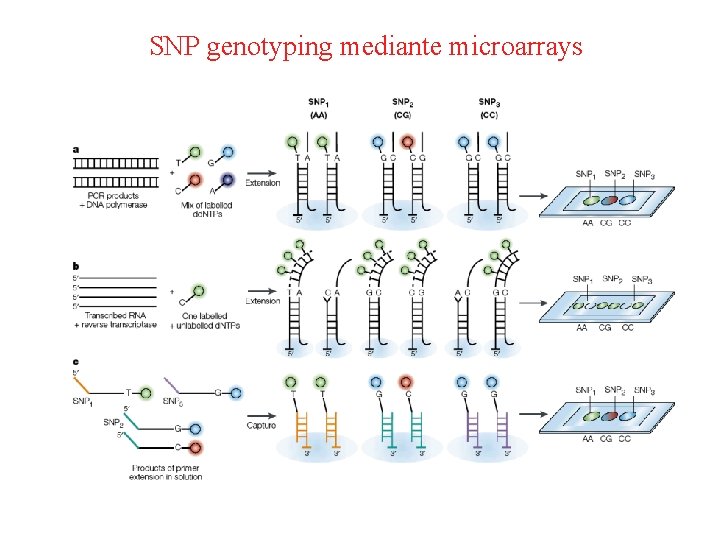
SNP genotyping mediante microarrays
 Non ti auguro un dono qualsiasi
Non ti auguro un dono qualsiasi Trigonometria triangoli rettangoli
Trigonometria triangoli rettangoli Coding dna and non coding dna
Coding dna and non coding dna Gemmazione
Gemmazione Video pierino e il lupo
Video pierino e il lupo Algoritmo smith-waterman
Algoritmo smith-waterman Sequenze di pierino e il lupo
Sequenze di pierino e il lupo Sequenze ires
Sequenze ires Fillotassi fibonacci
Fillotassi fibonacci Segnale di localizzazione nucleare
Segnale di localizzazione nucleare Macchine sequenziali
Macchine sequenziali Cos'è una sequenza
Cos'è una sequenza Testo vieni santo spirito di dio
Testo vieni santo spirito di dio Progressione aritmetica
Progressione aritmetica Buona domenica di pentecoste
Buona domenica di pentecoste Fasta e blast
Fasta e blast Sequenza ordinata
Sequenza ordinata Sezione aurea ppt
Sezione aurea ppt Hypertrophie cellule de haller
Hypertrophie cellule de haller Que es la variable diatopica
Que es la variable diatopica Mic dejun sanatos cu ovaz in 6 variante
Mic dejun sanatos cu ovaz in 6 variante Ejemplos de variable diacronica
Ejemplos de variable diacronica Zveneči in nezveneči glasovi
Zveneči in nezveneči glasovi Varianti libere esempi
Varianti libere esempi Procidence carotidienne gauche
Procidence carotidienne gauche Bazo accesorio como variante anatomica
Bazo accesorio como variante anatomica Variante 2
Variante 2 Que es polimorfismo en java
Que es polimorfismo en java Polimorfismo poo
Polimorfismo poo Polimorfismo genico
Polimorfismo genico Trpm5 taste receptor
Trpm5 taste receptor El polimorfismo de sobrecarga está relacionado con:
El polimorfismo de sobrecarga está relacionado con: Polimorfismo balanceado
Polimorfismo balanceado Polimorfismo java
Polimorfismo java Clase abstracta java
Clase abstracta java Polimorfismo parametrico java
Polimorfismo parametrico java Polimorfismo c++
Polimorfismo c++ Polimorfismo etimologia
Polimorfismo etimologia Polimorfismo typescript
Polimorfismo typescript Polimorfismo genetico
Polimorfismo genetico Rflp polimorfismo
Rflp polimorfismo Dieu veut des soldats fideles
Dieu veut des soldats fideles