Del ADN a las protenas DEL ADN A












































- Slides: 91

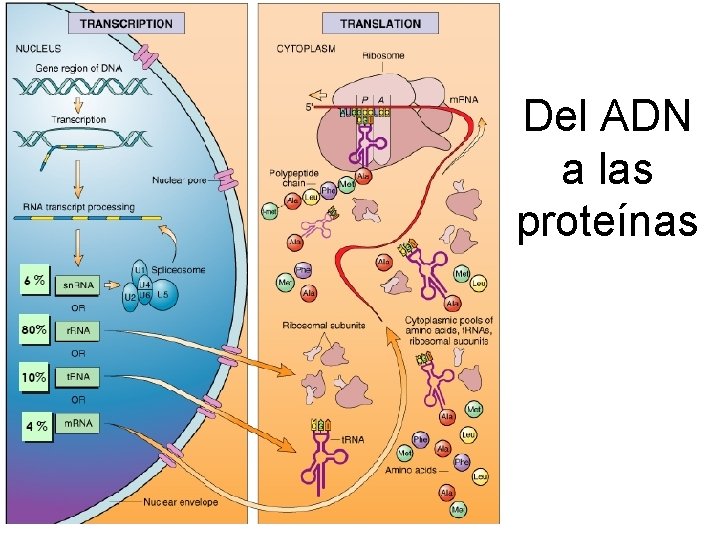
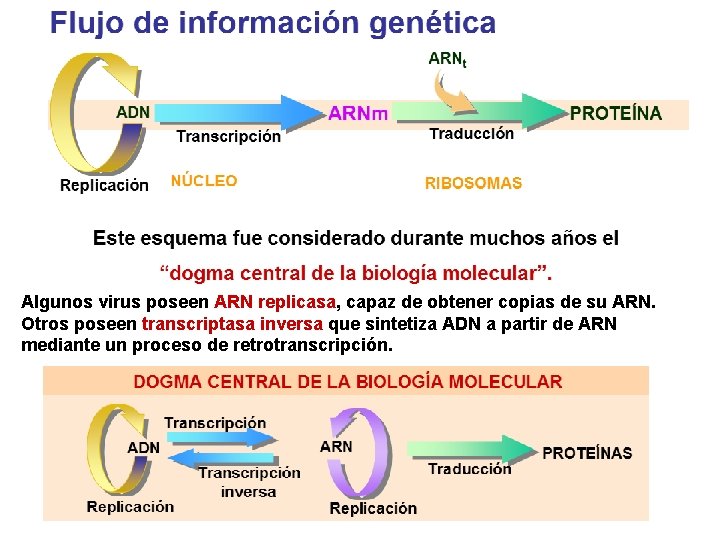
Del ADN a las proteínas

DEL ADN A LAS PROTEÍNAS 1. - EL ADN COMO MATERIAL HEREDITARIO 2. - ESTRUCTURA DEL GENOMA Y SU EXPRESIÓN 3. - FLUJO DE INFORMACIÓN GENÉTICA 4. - TRANSCRIPCIÓN: SÍNTESIS DEL ARN 5. - MADURACIÓN DEL ARN 6. - EL CÓDIGO GENÉTICO 7. - EL PROCESO DE TRADUCCIÓN. SÍNTESIS DE PROTEÍNAS 8. - REGULACIÓN DE LA EXPRESIÓN GÉNICA

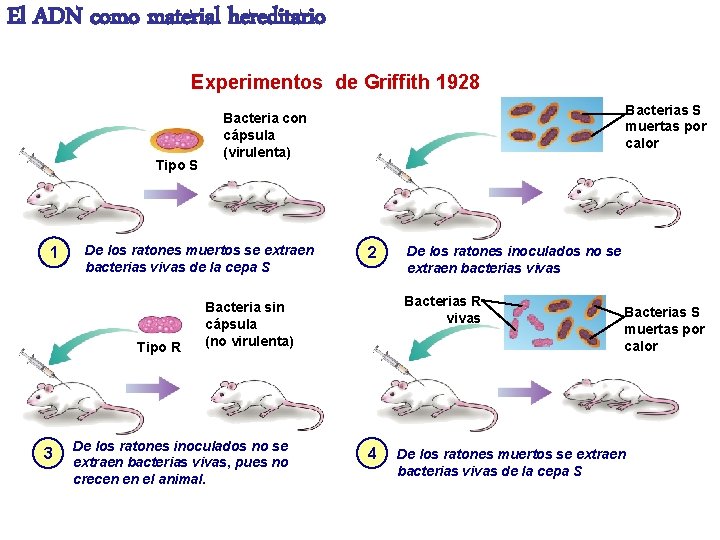
El ADN como material hereditario Experimentos de Griffith 1928 Tipo S 1 De los ratones muertos se extraen bacterias vivas de la cepa S Tipo R 3 Bacterias S muertas por calor Bacteria con cápsula (virulenta) 2 Bacterias R vivas Bacteria sin cápsula (no virulenta) De los ratones inoculados no se extraen bacterias vivas, pues no crecen en el animal. De los ratones inoculados no se extraen bacterias vivas 4 Bacterias S muertas por calor De los ratones muertos se extraen bacterias vivas de la cepa S

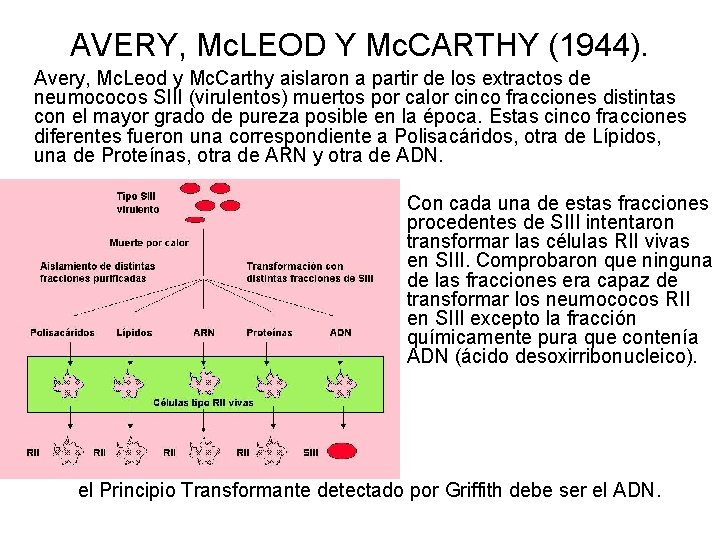
AVERY, Mc. LEOD Y Mc. CARTHY (1944). Avery, Mc. Leod y Mc. Carthy aislaron a partir de los extractos de neumococos SIII (virulentos) muertos por calor cinco fracciones distintas con el mayor grado de pureza posible en la época. Estas cinco fracciones diferentes fueron una correspondiente a Polisacáridos, otra de Lípidos, una de Proteínas, otra de ARN y otra de ADN. Con cada una de estas fracciones procedentes de SIII intentaron transformar las células RII vivas en SIII. Comprobaron que ninguna de las fracciones era capaz de transformar los neumococos RII en SIII excepto la fracción químicamente pura que contenía ADN (ácido desoxirribonucleico). el Principio Transformante detectado por Griffith debe ser el ADN.

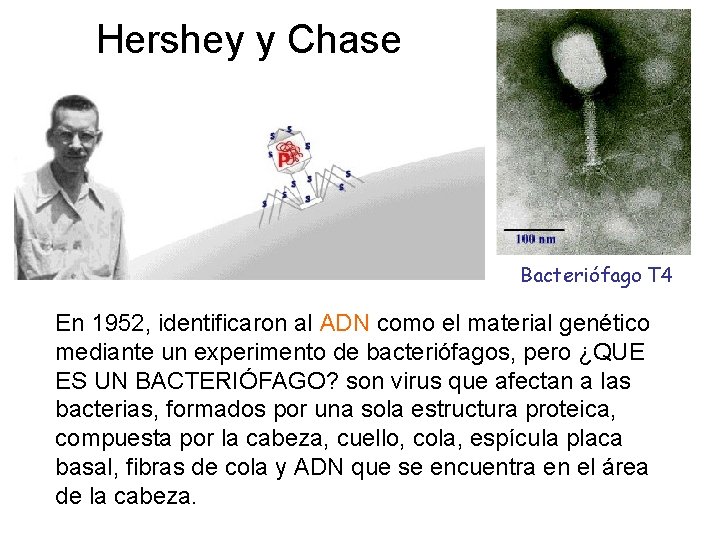
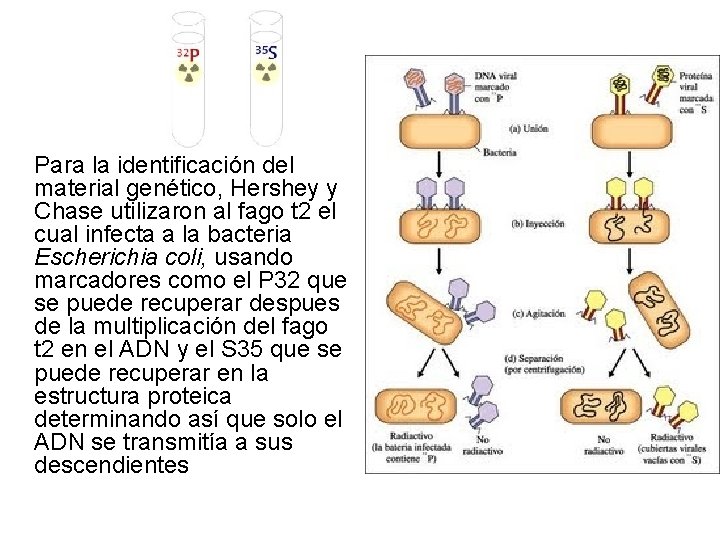
Hershey y Chase Bacteriófago T 4 En 1952, identificaron al ADN como el material genético mediante un experimento de bacteriófagos, pero ¿QUE ES UN BACTERIÓFAGO? son virus que afectan a las bacterias, formados por una sola estructura proteica, compuesta por la cabeza, cuello, cola, espícula placa basal, fibras de cola y ADN que se encuentra en el área de la cabeza.

Para la identificación del material genético, Hershey y Chase utilizaron al fago t 2 el cual infecta a la bacteria Escherichia coli, usando marcadores como el P 32 que se puede recuperar despues de la multiplicación del fago t 2 en el ADN y el S 35 que se puede recuperar en la estructura proteica determinando así que solo el ADN se transmitía a sus descendientes

1948 Edward Lawrie Tatum y George Wells Beadle muestran que los genes codifican las proteínas Establecen una relación directa entre la molécula de ADN y la secuencia de aminoácidos de una enzima: “un gen, una enzima”. No todas las proteínas son enzimas y hay proteínas formadas por varias cadenas polipeptídicas. La hipótesis se transforma: “un gen, una cadena polipeptídica”. Neurospora crassa, moho con el que trabajaron

• Beadle y Tatum en 1948, realizaron unas experiencias irradiando con UV un hongo. – UV proteínas, no las altera – UV ADN lo altera Al irradiar con UV el hongo, no sintetiza determinados enzimas (proteínas) – Se deduce: si los UV no destruyen a las proteínas, pero alteran al ADN y al alterarlo no se han sintetizado las proteínas, está clara la relación entre ADN y proteínas. (gen enzima)

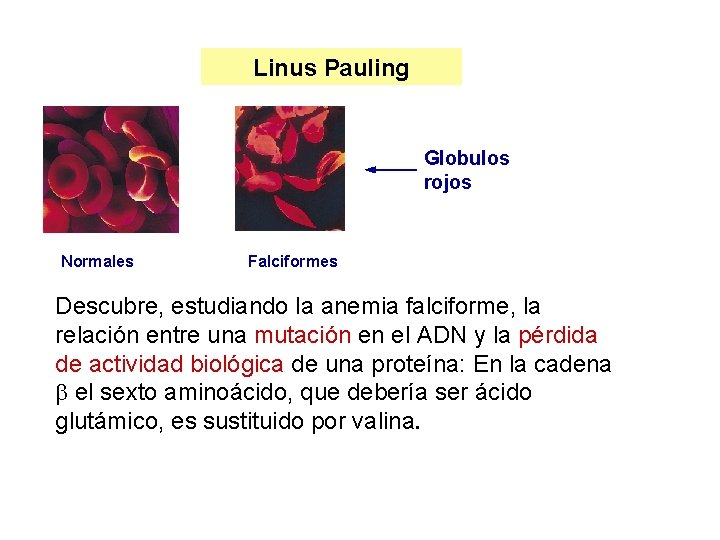
Linus Pauling Globulos rojos Normales Falciformes Descubre, estudiando la anemia falciforme, la relación entre una mutación en el ADN y la pérdida de actividad biológica de una proteína: En la cadena el sexto aminoácido, que debería ser ácido glutámico, es sustituido por valina.

• Una vez establecido el paralelismo entre genes y enzimas y tras ser propuesto, en 1. 953, el modelo de doble hélice por Watsony Crick, este último propuso la denominada Hipótesis de colinealidad de CRICK: " Existe una correspondencia entre la secuencia de nucleótidos del gen y la secuencia de aminoácidos de la enzima codificada"

Algunos virus poseen ARN replicasa, capaz de obtener copias de su ARN. Otros poseen transcriptasa inversa que sintetiza ADN a partir de ARN mediante un proceso de retrotranscripción.

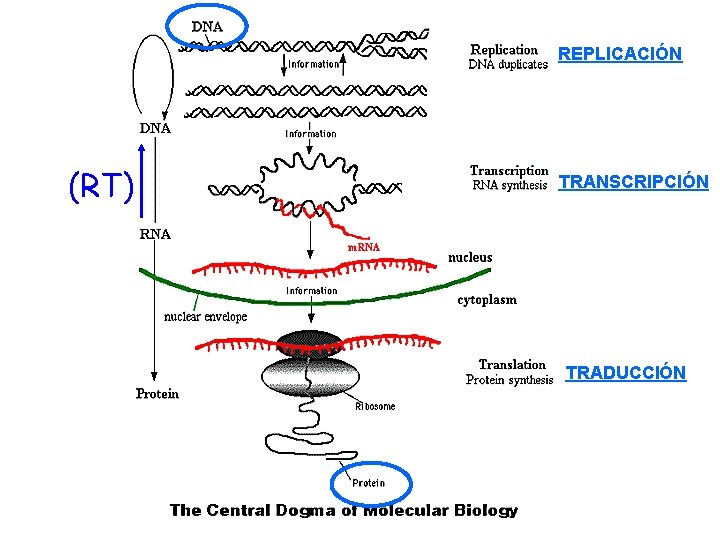
REPLICACIÓN (RT) TRANSCRIPCIÓN TRADUCCIÓN


La información se almacena en forma de GENES a lo largo del GENOMA, pero… ¿Cómo lo hacen PROCARIOTAS y EUCARIOTAS? A) PROCARIOTAS: § 1 solo cromosoma circular § Genes continuos (no existen zonas sin información) § Plásmidos moléculas pequeñas de ADN circular que se replican independientemente B) EUCARIOTAS: § ADN se encuentra en el núcleo § Mayor cantidad de ADN que en Procariotas § Hay ADN repetitivo (secuencias ↑ repetidas que no codifican proteínas) § En los genes hay intrones (“sin información”) y exones (“con información”) § ADN se asocia a proteínas (histonas) § Mitocondrias y Cloroplastos tienen ADN circular (≈ Procariotas)

Complejidad del genoma eucariota • Parte del genoma de los organismos eucariotas no codifica para proteínas: – ADN altamente repetitivo, centrómeros, ADN satélite, telómeros (5% del genoma humano) – ADN moderadamente repetitivo, SINEs, LINEs, ARNr y VNTRs (30% del genoma humano) • Los organismos eucariotas contienen secuencia no codificante (no traducida a proteína) incluso dentro de la secuencia génica

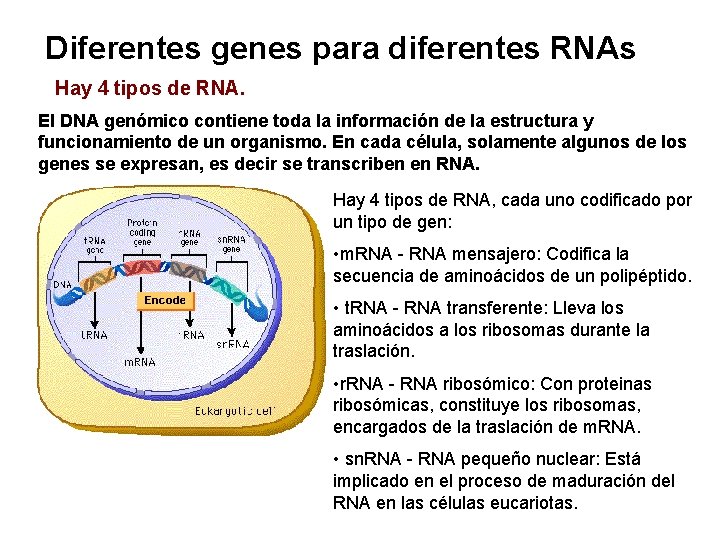
Diferentes genes para diferentes RNAs Hay 4 tipos de RNA. El DNA genómico contiene toda la información de la estructura y funcionamiento de un organismo. En cada célula, solamente algunos de los genes se expresan, es decir se transcriben en RNA. Hay 4 tipos de RNA, cada uno codificado por un tipo de gen: • m. RNA - RNA mensajero: Codifica la secuencia de aminoácidos de un polipéptido. • t. RNA - RNA transferente: Lleva los aminoácidos a los ribosomas durante la traslación. • r. RNA - RNA ribosómico: Con proteinas ribosómicas, constituye los ribosomas, encargados de la traslación de m. RNA. • sn. RNA - RNA pequeño nuclear: Está implicado en el proceso de maduración del RNA en las células eucariotas.

Generalidades sobre la transcripción: • Constituye el primer proceso de la expresión génica, mediante el cuál se transfiere la información contenida en la secuencia del ADN hacia la secuencia de proteína utilizando diversos ARN como intermediarios. • Durante la transcripción genética, las secuencias de ADN son copiadas a ARN mediante una enzima llamada ARN polimerasa • La enzima no requiere cebador • La enzima elonga en la dirección 5’ -> 3’ mediante la formación de enlaces en el extremo 3’ OH. • La síntesis sólo se inicia a partir de secuencias promotoras e implica el desenrollado parcial del ADN. • Las dos diferencias principales entre Replicación y Transcripción son: - Sólo se transcribe una cadena molde de ADN - Sólo una pequeña fracción de potencial genético global de un organismo se ejecuta en la célula.


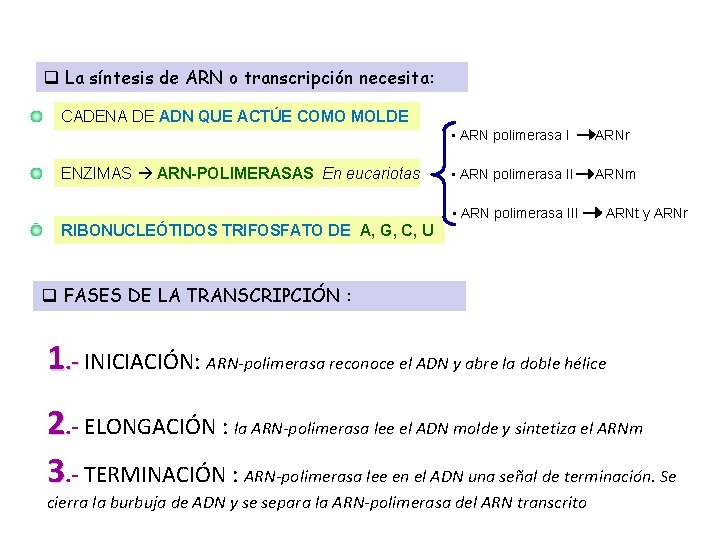
q La síntesis de ARN o transcripción necesita: CADENA DE ADN QUE ACTÚE COMO MOLDE ENZIMAS ARN-POLIMERASAS En eucariotas • ARN polimerasa I ARNr • ARN polimerasa II ARNm • ARN polimerasa III ARNt y ARNr RIBONUCLEÓTIDOS TRIFOSFATO DE A, G, C, U q FASES DE LA TRANSCRIPCIÓN : 1. - INICIACIÓN: ARN-polimerasa reconoce el ADN y abre la doble hélice 2. - ELONGACIÓN : la ARN-polimerasa lee el ADN molde y sintetiza el ARNm 3. - TERMINACIÓN : ARN-polimerasa lee en el ADN una señal de terminación. Se cierra la burbuja de ADN y se separa la ARN-polimerasa del ARN transcrito

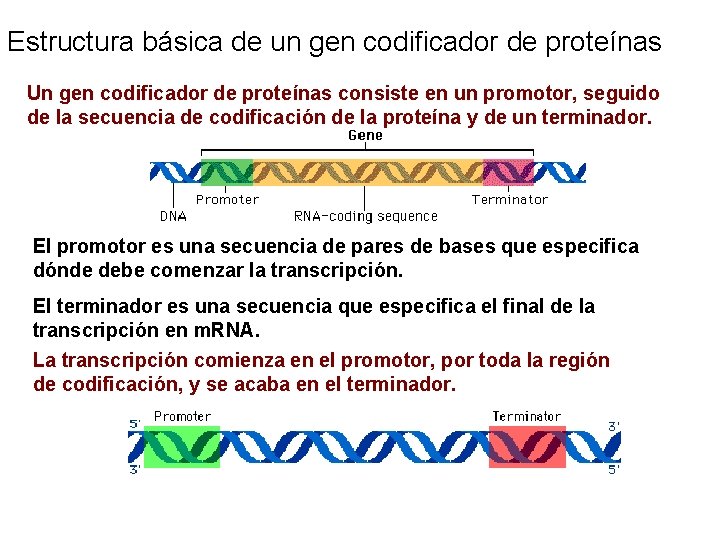
Estructura básica de un gen codificador de proteínas Un gen codificador de proteínas consiste en un promotor, seguido de la secuencia de codificación de la proteína y de un terminador. El promotor es una secuencia de pares de bases que especifica dónde debe comenzar la transcripción. El terminador es una secuencia que especifica el final de la transcripción en m. RNA. La transcripción comienza en el promotor, por toda la región de codificación, y se acaba en el terminador.

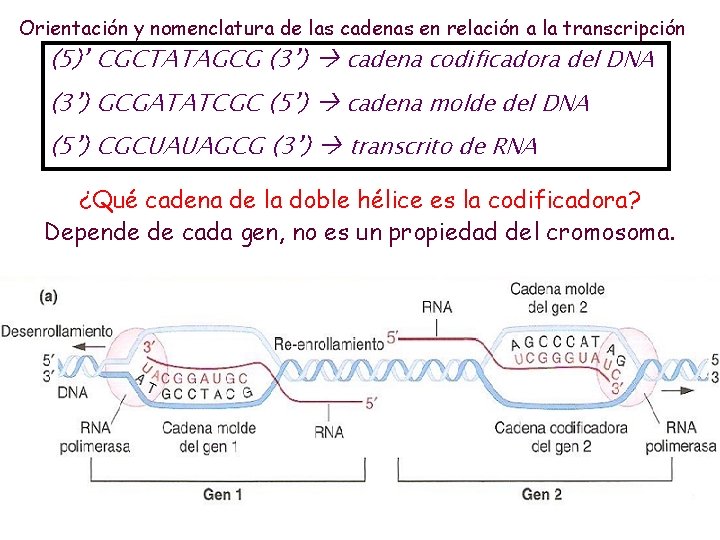
Orientación y nomenclatura de las cadenas en relación a la transcripción (5)’ CGCTATAGCG (3’) cadena codificadora del DNA (3’) GCGATATCGC (5’) cadena molde del DNA (5’) CGCUAUAGCG (3’) transcrito de RNA ¿Qué cadena de la doble hélice es la codificadora? Depende de cada gen, no es un propiedad del cromosoma.

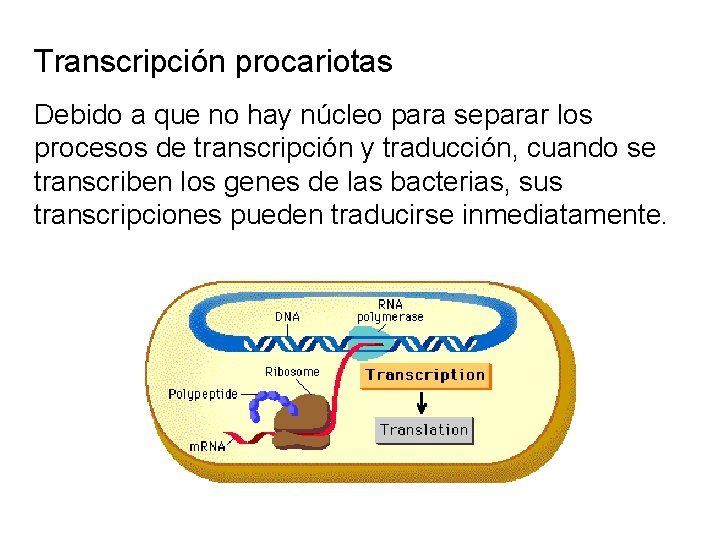
Transcripción procariotas Debido a que no hay núcleo para separar los procesos de transcripción y traducción, cuando se transcriben los genes de las bacterias, sus transcripciones pueden traducirse inmediatamente.

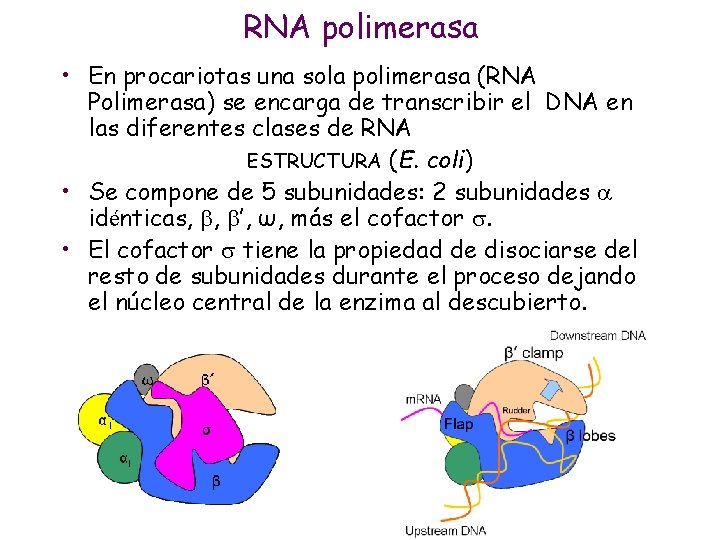
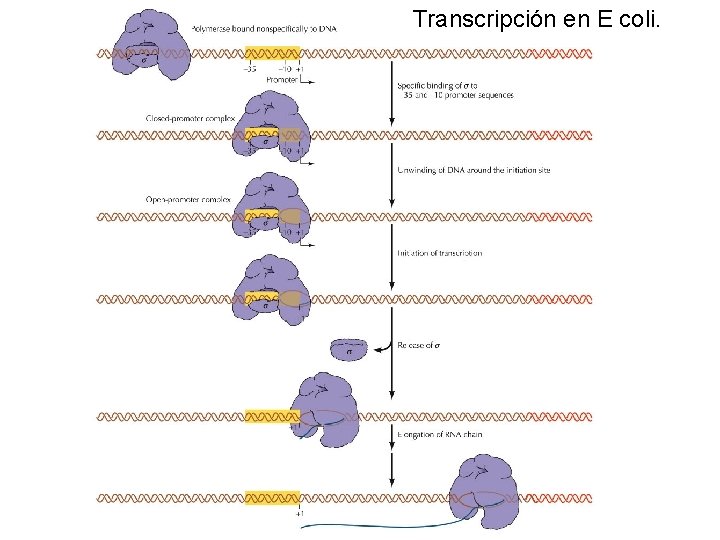
RNA polimerasa • En procariotas una sola polimerasa (RNA Polimerasa) se encarga de transcribir el DNA en las diferentes clases de RNA ESTRUCTURA (E. coli) • Se compone de 5 subunidades: 2 subunidades idénticas, , ’, ω, más el cofactor . • El cofactor tiene la propiedad de disociarse del resto de subunidades durante el proceso dejando el núcleo central de la enzima al descubierto.

Etapas de la transcripción • Iniciación: La polimerasa se une a secuencias específicas dentro del ADN, denominadas centros promotores. La subunidad σ colabora en la localización de la secuencia promotora

Centros promotores * Los promotores presentan diferentes eficacias en la iniciación de la transcripción. 1 transcripción cada 2 segundos a 1 cada 10 minutos * Los promotores más fuertes tiene secuencias – 35 y – 10 que coinciden con las consensos. * La distancia óptima entre ambas secuencias consensos es de 17 nucleótidos. * La ARN polimerasa se une al ADN de manera inespecífica y busca el sitio promotor desplazándose por la molécula de ADN. * Puede detectar las secuencias – 35 y – 10 sin desenrollar la doble hélice.

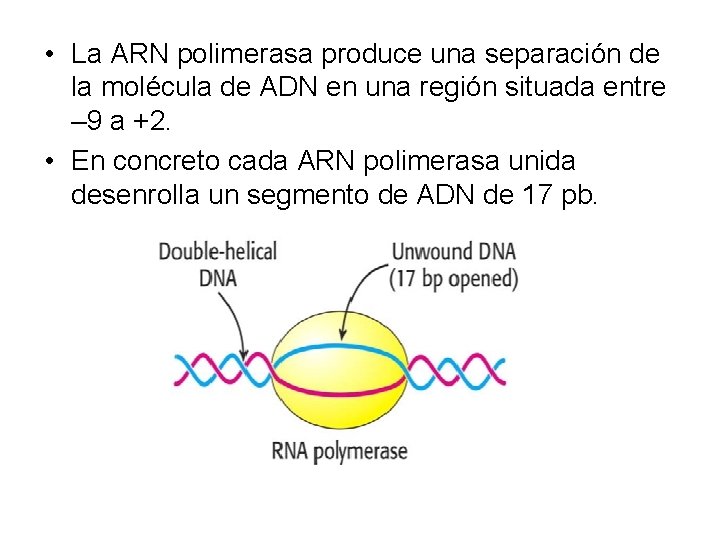
• La ARN polimerasa produce una separación de la molécula de ADN en una región situada entre – 9 a +2. • En concreto cada ARN polimerasa unida desenrolla un segmento de ADN de 17 pb.

Elongación • Tras la polimerización de 6 -10 nucleótidos la subunidad σ se separa de la holoenzima. • La burbuja de transcripción (ARN polimerasa, ADN molde y ARN naciente), se desplaza a lo largo de la molécula de ADN. • El ARN sintetizado se enrolla con la hebra de ADN molde formándose un híbrido ADN-ARN, de unos 8 -10 nucleótidos de longitud. • La región desenrollada del ADN y la longitud del híbrido DNA-RNA permanecen constantes mientras la ARN polimerasa avanza por el molde. • La molécula de ADN debe de ir desenrollandose por delante de la burbuja y enrollandose por detrás.


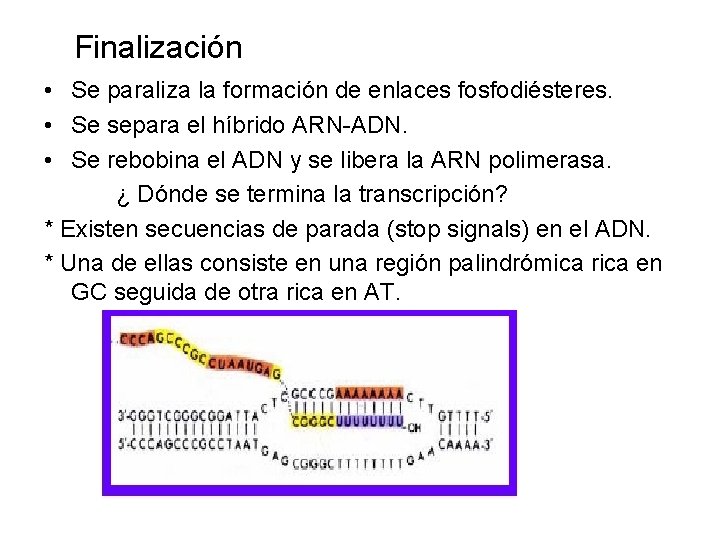
Finalización • Se paraliza la formación de enlaces fosfodiésteres. • Se separa el híbrido ARN-ADN. • Se rebobina el ADN y se libera la ARN polimerasa. ¿ Dónde se termina la transcripción? * Existen secuencias de parada (stop signals) en el ADN. * Una de ellas consiste en una región palindrómica rica en GC seguida de otra rica en AT.


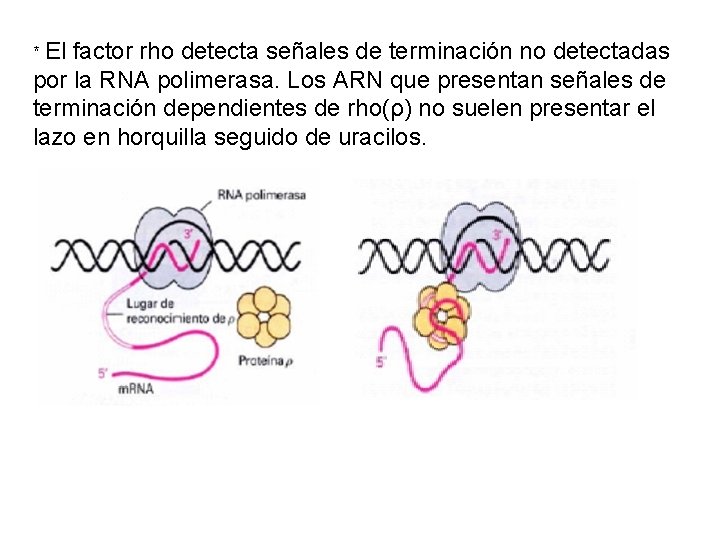
* El factor rho detecta señales de terminación no detectadas por la RNA polimerasa. Los ARN que presentan señales de terminación dependientes de rho(ρ) no suelen presentar el lazo en horquilla seguido de uracilos.

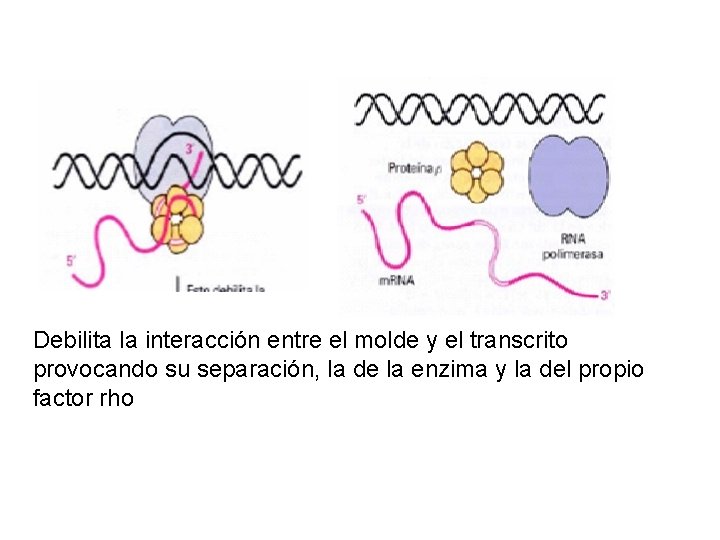
Debilita la interacción entre el molde y el transcrito provocando su separación, la de la enzima y la del propio factor rho

Transcripción en E coli.

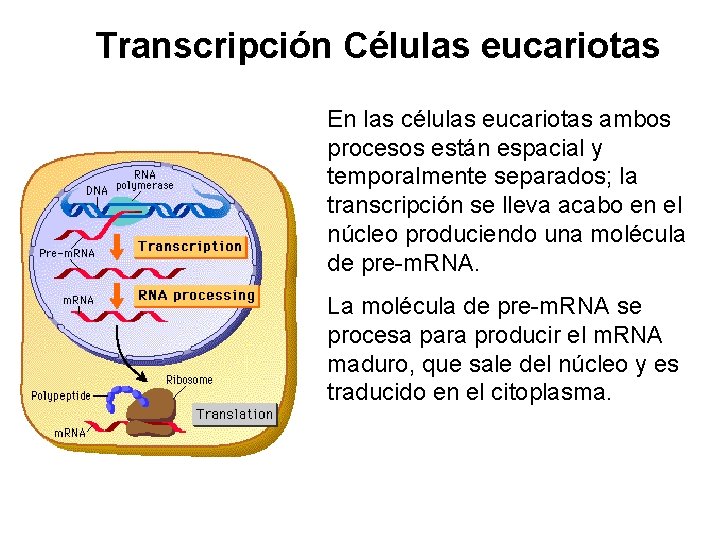
Transcripción Células eucariotas En las células eucariotas ambos procesos están espacial y temporalmente separados; la transcripción se lleva acabo en el núcleo produciendo una molécula de pre-m. RNA. La molécula de pre-m. RNA se procesa para producir el m. RNA maduro, que sale del núcleo y es traducido en el citoplasma.

TRANSCRIPCION EN EUCARIOTAS • Es un proceso de mucha discriminación (según el tejido o etapa del desarrollo serán los genes que se van a transcribir) • La maquinaria de la transcripción debe tener en cuenta la compleja estructura de la cromatina eucariota • Requiere de varios tipos de RNA polimerasas • La RNA polimerasa requiere de factores adicionales llamados factores de transcripción para iniciar la transcripción • Tiene que haber un procesamiento complejo del m. RNA que permita escindir los intrones del mensaje y transportar la molécula al citoplasma

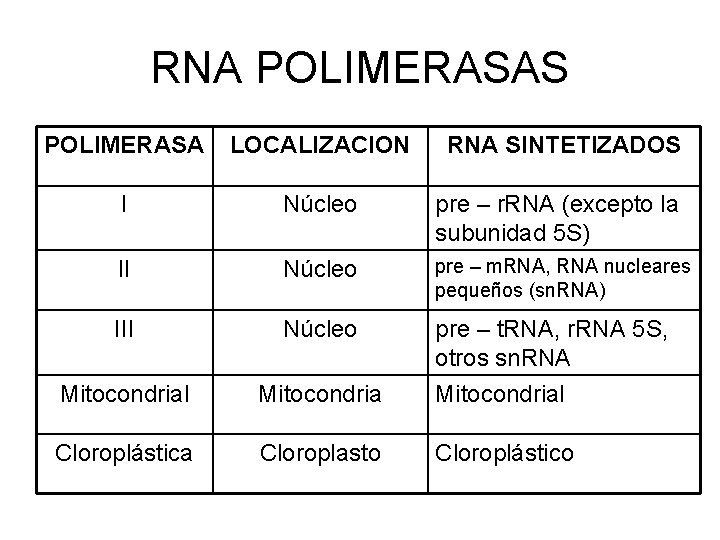
RNA POLIMERASAS POLIMERASA LOCALIZACION RNA SINTETIZADOS I Núcleo pre – r. RNA (excepto la subunidad 5 S) II Núcleo pre – m. RNA, RNA nucleares pequeños (sn. RNA) III Núcleo Mitocondrial Mitocondria pre – t. RNA, r. RNA 5 S, otros sn. RNA Mitocondrial Cloroplástica Cloroplasto Cloroplástico


RNA POLIMERASA II • TRANSCRIBE LOS GENES ESTRUCTURALES, ES DECIR, LOS QUE SE TRADUCEN A PROTEINAS • Contiene múltiples subunidades • Intervienen al menos 7 factores de transcripción: TFIIA, TFIIB, TFIID, TFIIE, TFIIF, TFIIH y TFIIJ • El factor critico es TFIID que se une a la caja TATA que es el equivalente eucariota a la región -10

• Los genes de eucariotas, al igual que los de procariotas, precisan de promotores para iniciar la transcripción Destacar la presencia de la región TATA box hacia el nucleótido – 25 • En eucariotas, además de la región promotora existen otras regiones intensificadoras (enhancers) que participan también en la iniciación. • Las secuencias intesificadoras no tienen una localización precisa respecto al sitio de iniciación. • Las secuencias intesificadoras son bidireccionales. • Pueden ejercer su efecto sobre promotores situados a miles de pares de bases de distancia.

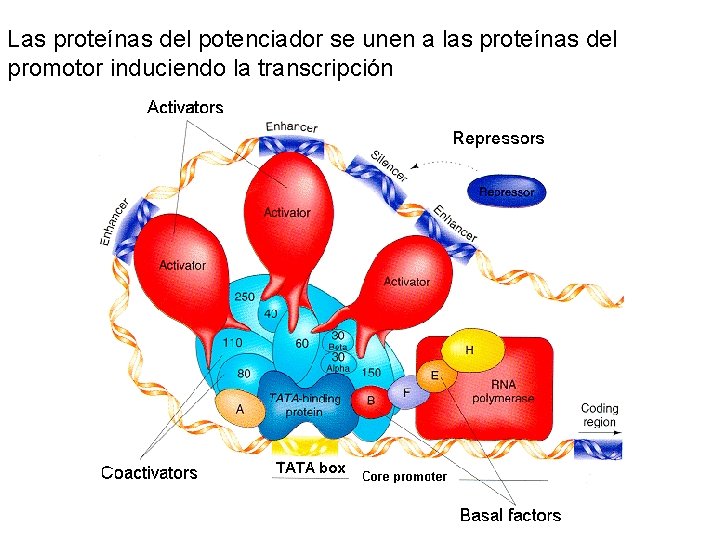
Las proteínas del potenciador se unen a las proteínas del promotor induciendo la transcripción

Iniciación • Existen en los genes eucariotas tres tipos de secuencias génicas reguladoras: 1. El promotor: es la región más próxima al inicio de la transcripción y esta formada por la secuencia -25 TATA, -80 CAAT Y -120 GC. Los factores proteicos de transcripción que se unen al promotor, conocidos como factores basales, ayudan a la ARN polimerasa a colocarse en el sitio de iniciación del gen. 2. Las secuencias potenciadoras (enhancers): situadas mucho más lejos, entre -200, -10000, a ellas se unen los factores activadores de la transcripción, que cumplen dos funciones: 1. 2. Desempaquetar la cromatina al disgregar los nucleosomas, y consiguen desenrollar la doble vuelta del ADN, para que la región promotora quede accesible. Incrementar la velocidad de transcripción. 3. Las secuencias silenciadoras( silencers): se encuentran intercaladas entre las activadoras y a ellas se unen los represores, disminuyendo la velocidad de transcripción.

• Elongación: la enzima ARN polimerasa recorre la hebra molde en sentido 3´ 5´, mientras que la cadena del ARNm transcrito primario o pre -ARNm , continua creciendo en dirección 5´ 3´, a razón de 30 nucleótidos por segundo. • Terminación: la enzima reconoce la secuencia TTATTT, que determina el final de la transcripción y la separación de la cadena de pre-ARNm recién sintetizada de la hebra molde de ADN

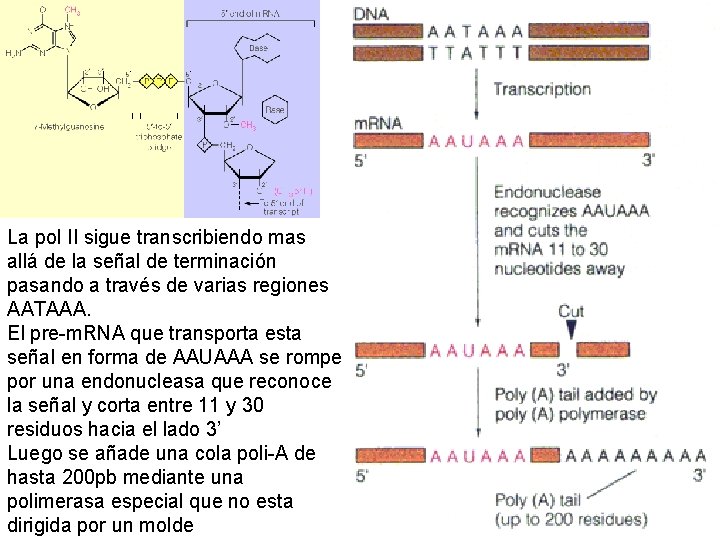
La pol II sigue transcribiendo mas allá de la señal de terminación pasando a través de varias regiones AATAAA. El pre-m. RNA que transporta esta señal en forma de AAUAAA se rompe por una endonucleasa que reconoce la señal y corta entre 11 y 30 residuos hacia el lado 3’ Luego se añade una cola poli-A de hasta 200 pb mediante una polimerasa especial que no esta dirigida por un molde




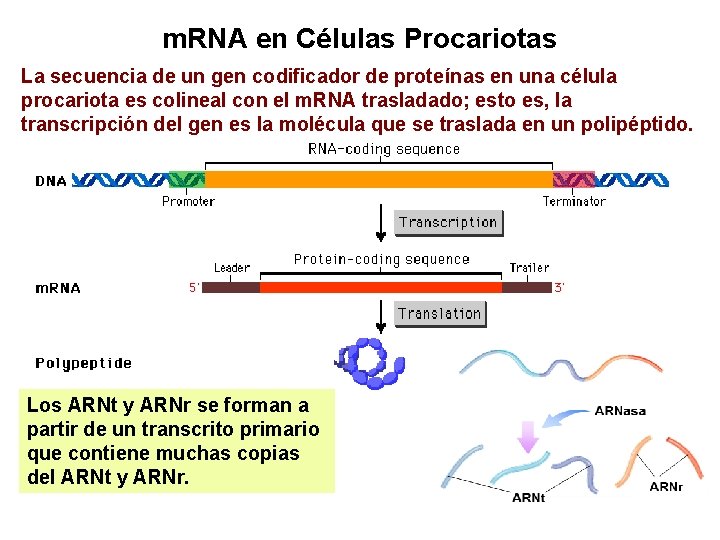
m. RNA en Células Procariotas La secuencia de un gen codificador de proteínas en una célula procariota es colineal con el m. RNA trasladado; esto es, la transcripción del gen es la molécula que se traslada en un polipéptido. Los ARNt y ARNr se forman a partir de un transcrito primario que contiene muchas copias del ARNt y ARNr.

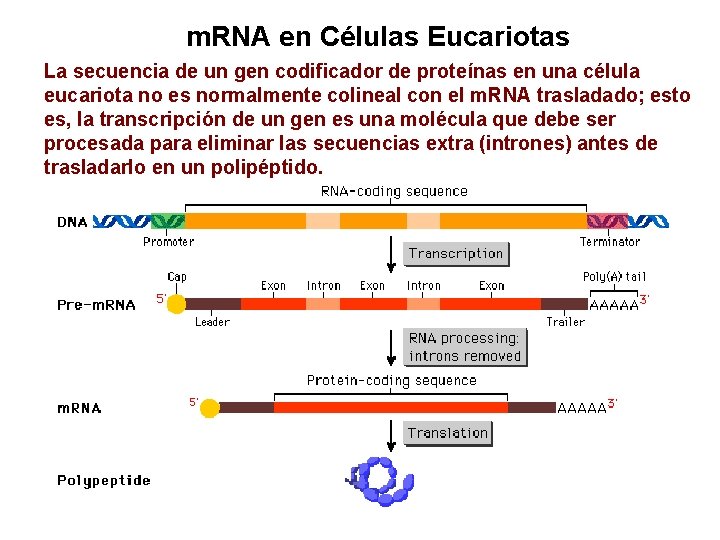
m. RNA en Células Eucariotas La secuencia de un gen codificador de proteínas en una célula eucariota no es normalmente colineal con el m. RNA trasladado; esto es, la transcripción de un gen es una molécula que debe ser procesada para eliminar las secuencias extra (intrones) antes de trasladarlo en un polipéptido.

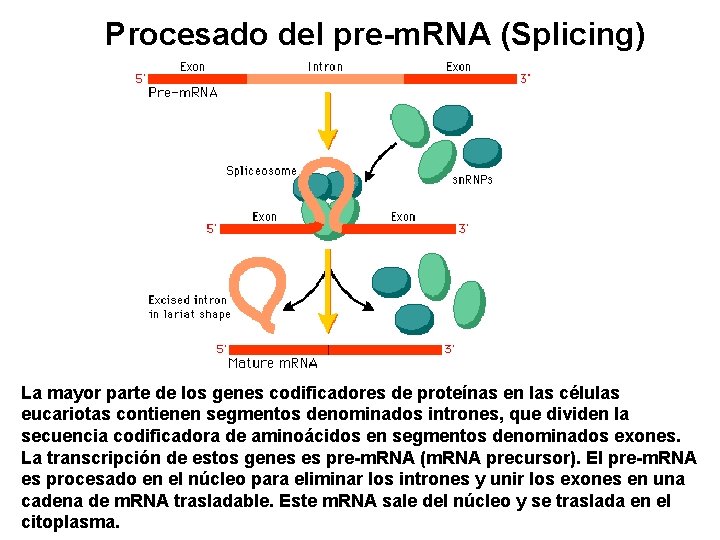
Procesado del pre-m. RNA (Splicing) La mayor parte de los genes codificadores de proteínas en las células eucariotas contienen segmentos denominados intrones, que dividen la secuencia codificadora de aminoácidos en segmentos denominados exones. La transcripción de estos genes es pre-m. RNA (m. RNA precursor). El pre-m. RNA es procesado en el núcleo para eliminar los intrones y unir los exones en una cadena de m. RNA trasladable. Este m. RNA sale del núcleo y se traslada en el citoplasma.

• • LA TRANSCRIPCIÓN DIFERENCIAS PROCARIOTAS CON EUCARIOTAS 1ª) En los procariotas el ARNm no tiene ni caperuza ni cola. 2ª) Tampoco tiene intrones y por lo tanto no requiere de un mecanismo de maduración. 3ª) Al mismo tiempo que el ARNm se transcribe se está ya traduciendo. 4ª) Los genes son policistrónicos, esto es, un ARNm contienen información para varias proteínas.


Código Genético Código que establece la relación entre nucleótidos y aminoácidos Se consiguió encontrar gracias a los siguientes descubrimientos: – Severo Ochoa y otros (1955) aislaron la polinucleótido fosforilasa (enzima capaz de unir nucleótidos sin hebra patrón): formaron un poli. U (UUUU……). SEVERO OCHOA (1905 -1993) Premio Nobel 1959 Descubrimiento de la Polinucleótido fosforilasa

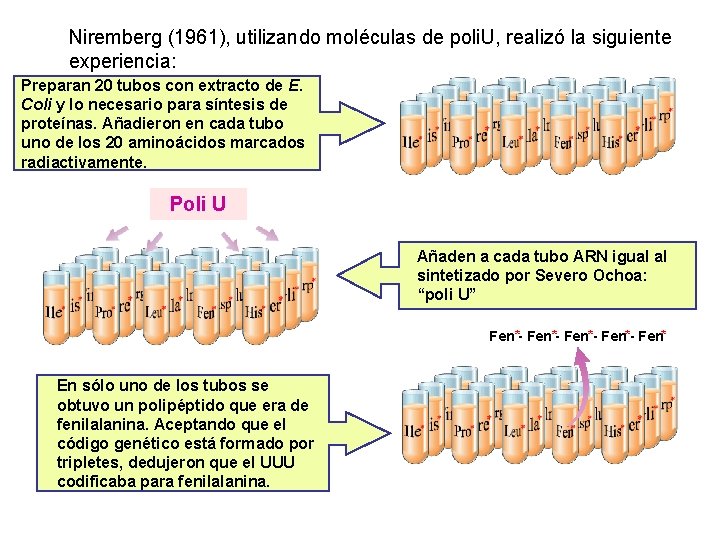
Niremberg (1961), utilizando moléculas de poli. U, realizó la siguiente experiencia: Preparan 20 tubos con extracto de E. Coli y lo necesario para síntesis de proteínas. Añadieron en cada tubo uno de los 20 aminoácidos marcados radiactivamente. Poli U Añaden a cada tubo ARN igual al sintetizado por Severo Ochoa: “poli U” Fen *- Fen*- Fen* En sólo uno de los tubos se obtuvo un polipéptido que era de fenilalanina. Aceptando que el código genético está formado por tripletes, dedujeron que el UUU codificaba para fenilalanina.

• Dedujo: hay colinearidad entre Uracilo y Fenilalanina. • Si repetía la experiencia con poli. C, sólo se unían los aminoácidos en el tubo de la Prolina, por lo tanto hay colinearidad Citosina y Prolina. UUUUUUU. . -> CCCCCCC. . . -> AAAAAAA. . . -> GGGGGGG -> UUU CCC AAA GGG Phe-Phe-Phe. . . Pro-Pro-Pro. . . Lys-Lys-Lys. . . Gly-Gly-Gly. . . Phe Pro Lys Gly


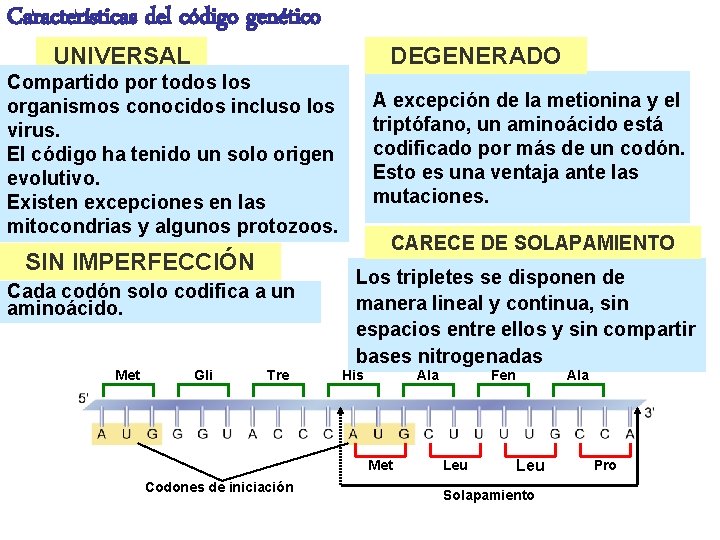
Características del código genético UNIVERSAL DEGENERADO Compartido por todos los organismos conocidos incluso los virus. El código ha tenido un solo origen evolutivo. Existen excepciones en las mitocondrias y algunos protozoos. SIN IMPERFECCIÓN Cada codón solo codifica a un aminoácido. Met Gli Tre A excepción de la metionina y el triptófano, un aminoácido está codificado por más de un codón. Esto es una ventaja ante las mutaciones. CARECE DE SOLAPAMIENTO Los tripletes se disponen de manera lineal y continua, sin espacios entre ellos y sin compartir bases nitrogenadas His Ala Met Codones de iniciación Fen Leu Ala Leu Solapamiento Pro

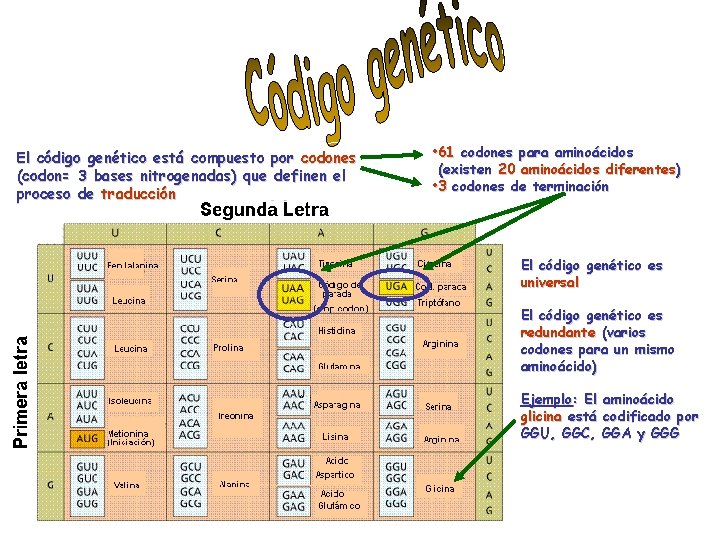
El código genético está compuesto por codones (codon= 3 bases nitrogenadas) que definen el proceso de traducción • 61 codones para aminoácidos (existen 20 aminoácidos diferentes) • 3 codones de terminación El código genético es universal El código genético es redundante (varios codones para un mismo aminoácido) Ejemplo: El aminoácido glicina está codificado por GGU, GGC, GGA y GGG

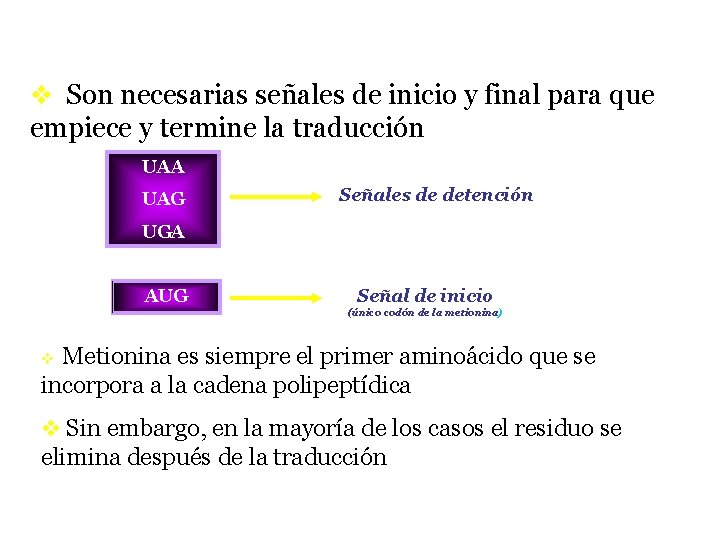
v Son necesarias señales de inicio y final para que empiece y termine la traducción UAA UAG Señales de detención UGA AUG Señal de inicio (único codón de la metionina) Metionina es siempre el primer aminoácido que se incorpora a la cadena polipeptídica v v Sin embargo, en la mayoría de los casos el residuo se elimina después de la traducción

Traducción • Es la síntesis de una molécula de proteína, de acuerdo con el código contenido en la molécula de ARNm. • Se llama traducción porque comprende el cambio del “lenguaje” de ácidos nucleicos (sucesión de bases) al lenguaje de proteínas (sucesión de aminoácidos). • En el citoplasma, el ARNm se mueve hacia los ribosomas. Los aminoácidos que se necesitan están dispersos por el citoplasma. Los aminoácidos correctos llegan al ARNm por el ARNt. • Las moléculas de ARNt son más cortas que las de ARNm y tienen la forma de una hoja de trébol. • En uno de los lazos de la molécula de ARNt hay un conjunto de tres bases llamado anticodón. El lado opuesto transporta un aminoácido.

• Cada t. ARN contiene un triplete de nucleótidos denominado anticodón, que es complemetario de un codón en el m. ARN para el aminoácido concreto • Es necesaria una enzima específica para realizar la unión entre cada t. ARN y el correspondiente aminoácido denominada aminoacil-t. ARN sintetasa • Los ribosomas son los encargados de juntar los t. ARN y m. ARN para efectuar la traducción. • El mensaje se lee en dirección 5’ 3’, y la cadena polipeptídica se sintetiza comenzando por el residuo N-terminal.

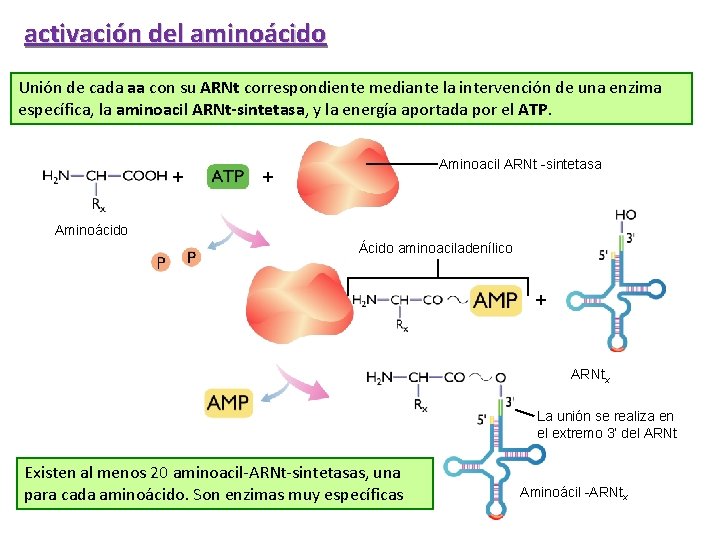
activación del aminoácido Unión de cada aa con su ARNt correspondiente mediante la intervención de una enzima específica, la aminoacil ARNt-sintetasa, y la energía aportada por el ATP. + Aminoacil ARNt -sintetasa + Aminoácido Ácido aminoaciladenílico + ARNtx La unión se realiza en el extremo 3’ del ARNt Existen al menos 20 aminoacil-ARNt-sintetasas, una para cada aminoácido. Son enzimas muy específicas Aminoácil -ARNtx


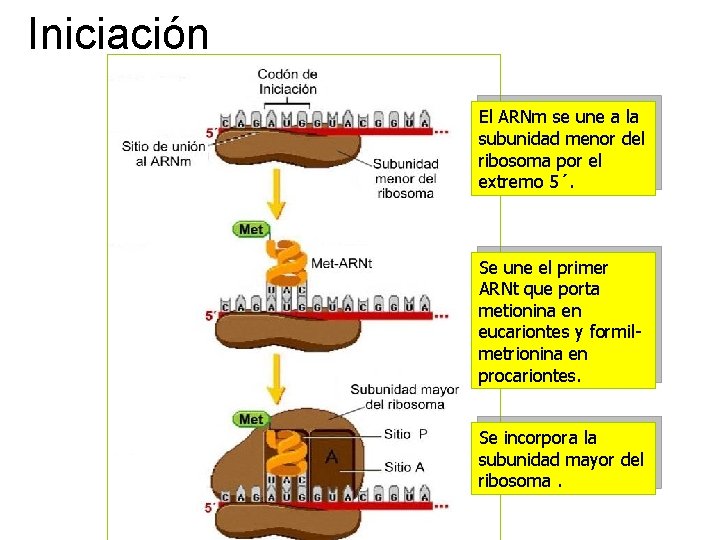
Iniciación El ARNm se une a la subunidad menor del ribosoma por el extremo 5´. Se une el primer ARNt que porta metionina en eucariontes y formilmetrionina en procariontes. Se incorpora la subunidad mayor del ribosoma.

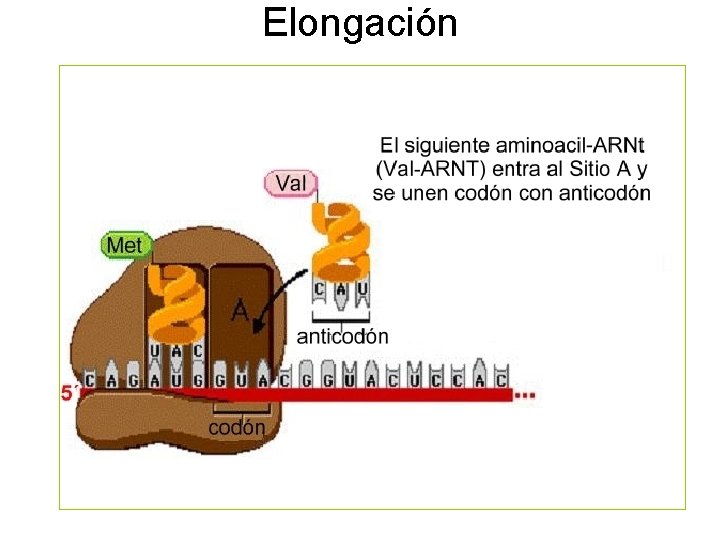
Elongación



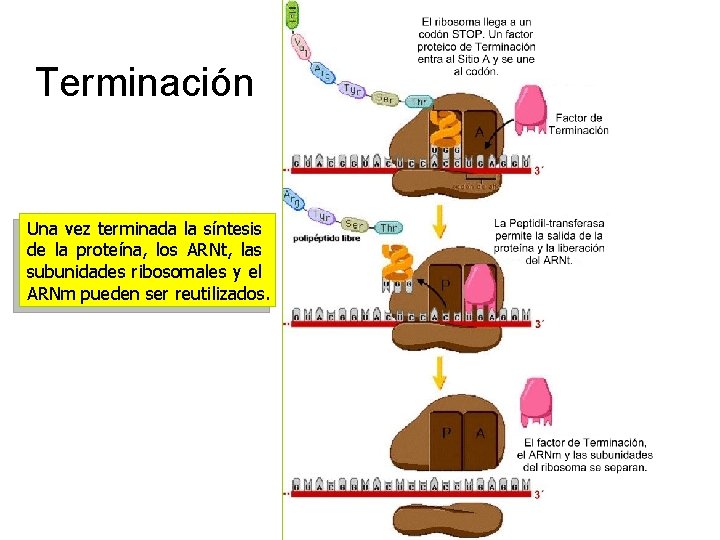
Terminación Una vez terminada la síntesis de la proteína, los ARNt, las subunidades ribosomales y el ARNm pueden ser reutilizados. 67

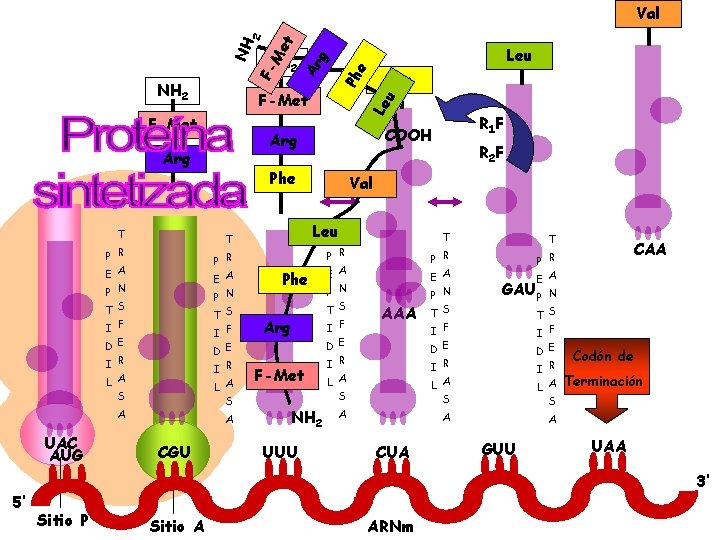
P R E A P N T S I F D E I R L A S S A 5’ A CGU e g Ar Le u Phe T Phe R 2 F Val Leu T T P R E A Phe Arg P N T S I F D E F-Met AAA I R L A NH 2 P N T S GAU P A CUA CAA N T S I F D E I R L A S UUU R 1 F COOH Arg UAC AUG Me t F-Met Leu Ph NH 2 T NH 2 F- NH 2 Val S L A S A A GUU Codón de Terminación UAA 3’ Sitio P Sitio A ARNm

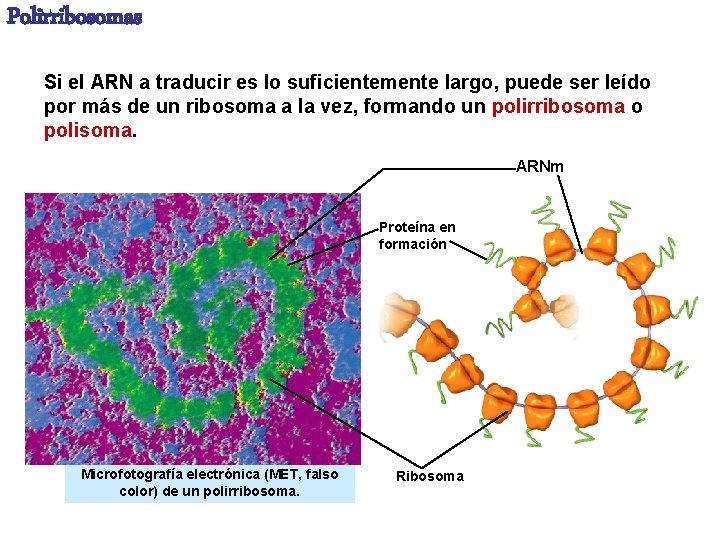
Polirribosomas Si el ARN a traducir es lo suficientemente largo, puede ser leído por más de un ribosoma a la vez, formando un polirribosoma o polisoma. ARNm Proteína en formación Microfotografía electrónica (MET, falso color) de un polirribosoma. Ribosoma

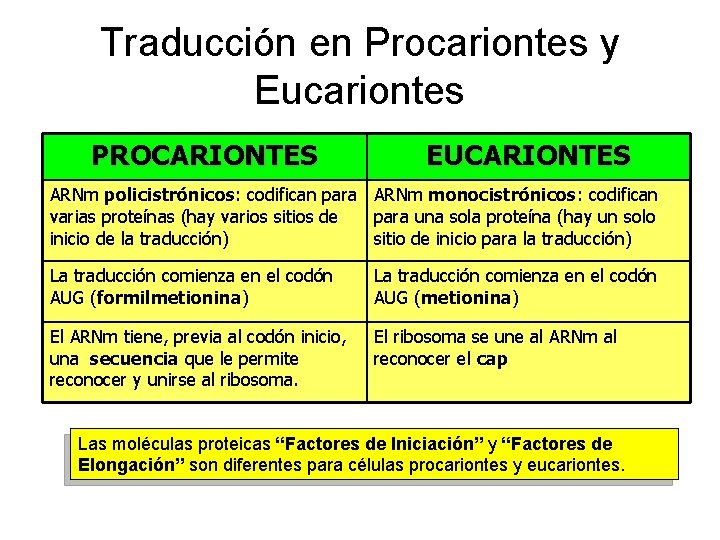
Traducción en Procariontes y Eucariontes PROCARIONTES EUCARIONTES ARNm policistrónicos: codifican para ARNm monocistrónicos: codifican varias proteínas (hay varios sitios de para una sola proteína (hay un solo inicio de la traducción) sitio de inicio para la traducción) La traducción comienza en el codón AUG (formilmetionina) La traducción comienza en el codón AUG (metionina) El ARNm tiene, previa al codón inicio, una secuencia que le permite reconocer y unirse al ribosoma. El ribosoma se une al ARNm al reconocer el cap Las moléculas proteicas “Factores de Iniciación” y “Factores de Elongación” son diferentes para células procariontes y eucariontes.

Regulación de la expresión génica Genes constitutivos y genes regulados No todos los genes se expresan simultáneamente ni al mismo nivel Genes constitutivos: se expresan al mismo nivel independientemente de las condiciones ambientales Genes regulados: se expresan a distintos niveles (o no se expresan) dependiendo de las condiciones

Regulación de la expresión génica en procariotas • Un primer nivel de regulación se establece en las secuencias promotoras que sean lo más parecidas posibles a las secuencias consenso. • Un segundo nivel es el modelo del operón propuesto por Jacob y Monod en 1961 Francois Jacob Jacques Monod

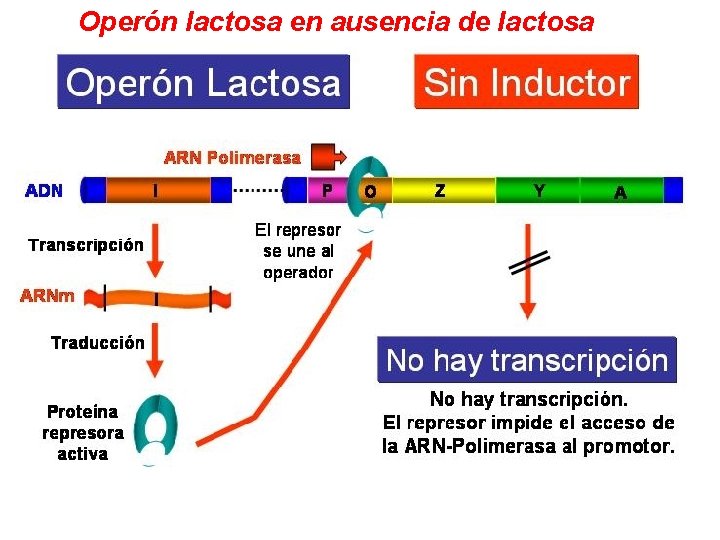
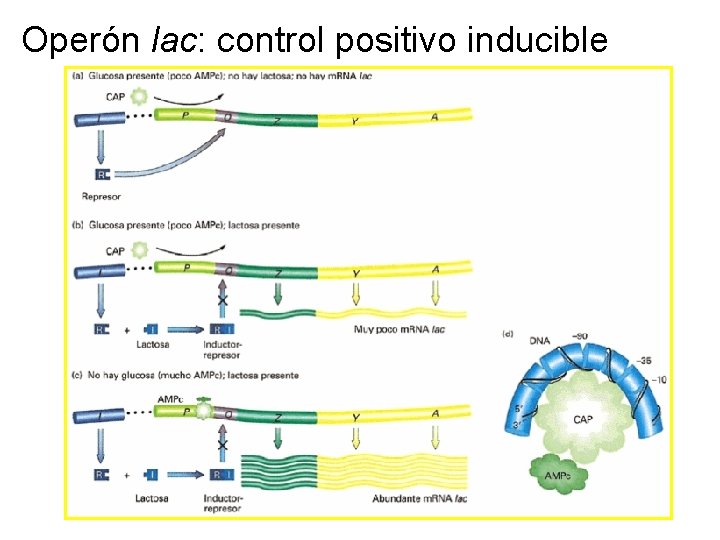
Los operones son vinducibles vreprimibles de acuerdo al mecanismo de control El Operón lactosa, que abreviadamente se denomina Operón lac, es un sistema inducible. La proteína reguladora, producto del gen regulador , es un represor que impide la expresión de los genes estructurales en ausencia del inductor. El inductor en este caso es la lactosa

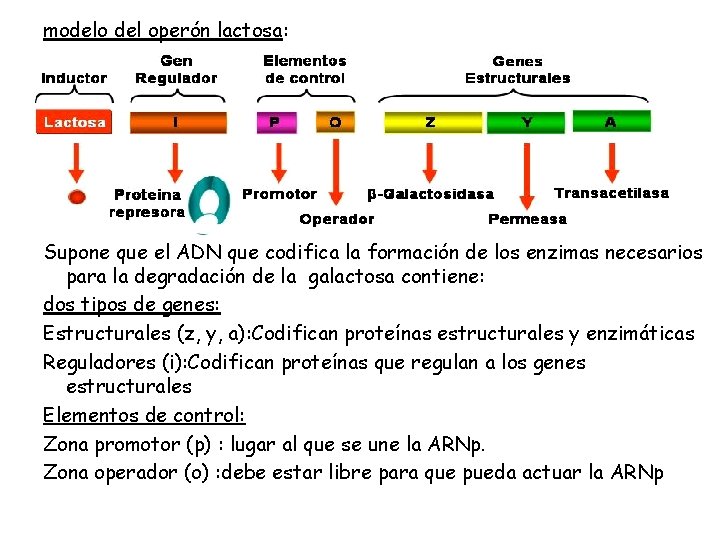
modelo del operón lactosa: Supone que el ADN que codifica la formación de los enzimas necesarios para la degradación de la galactosa contiene: dos tipos de genes: Estructurales (z, y, a): Codifican proteínas estructurales y enzimáticas Reguladores (i): Codifican proteínas que regulan a los genes estructurales Elementos de control: Zona promotor (p) : lugar al que se une la ARNp. Zona operador (o) : debe estar libre para que pueda actuar la ARNp


Operón lactosa en ausencia de lactosa

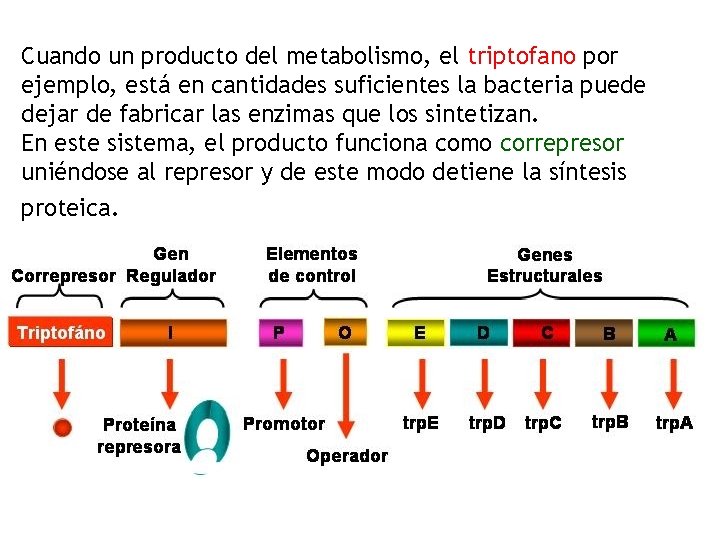
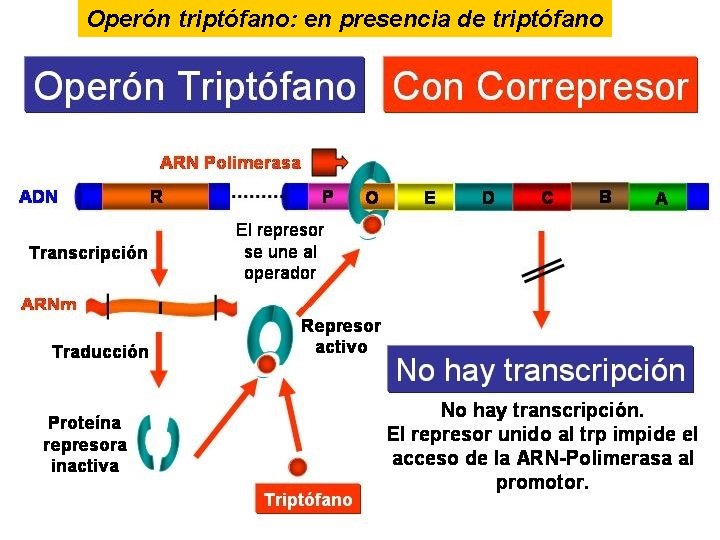
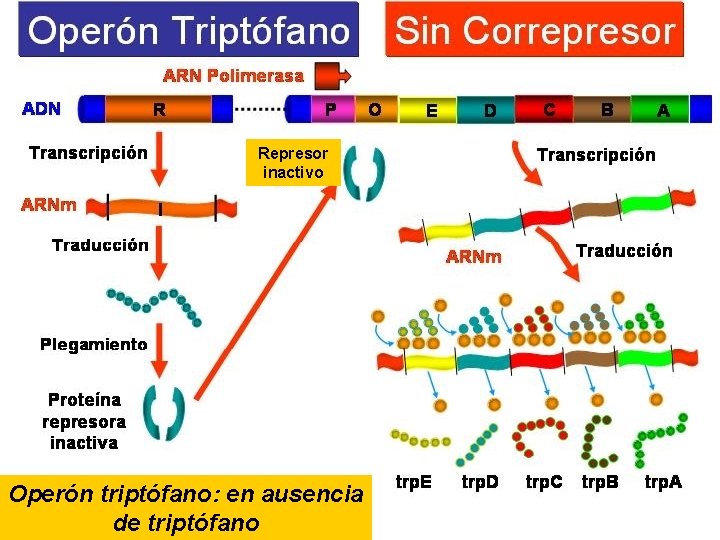
Cuando un producto del metabolismo, el triptofano por ejemplo, está en cantidades suficientes la bacteria puede dejar de fabricar las enzimas que los sintetizan. En este sistema, el producto funciona como correpresor uniéndose al represor y de este modo detiene la síntesis proteica.

Operón triptófano: en presencia de triptófano

Represor inactivo Operón triptófano: en ausencia de triptófano

Tanto la represión como la inducción son ejemplos de control negativo, dado que la proteína represora detiene (" turn off ") la transcripción. La lactosa, el azúcar de la leche, es hidrolizada por la enzima beta-galactosidasa. Esta enzima es inducible: solo se produce en grandes cantidades cuando la lactosa, el sustrato sobre el cual opera, esta presente. En cambio, las enzimas para la síntesis del aminoácido triptófano se producen continuamente a menos que el triptófano este presente en el medio de cultivo, se dice en este caso que las enzimas sintetizadoras de triptófano están reprimidas.

Operón lac: control positivo inducible

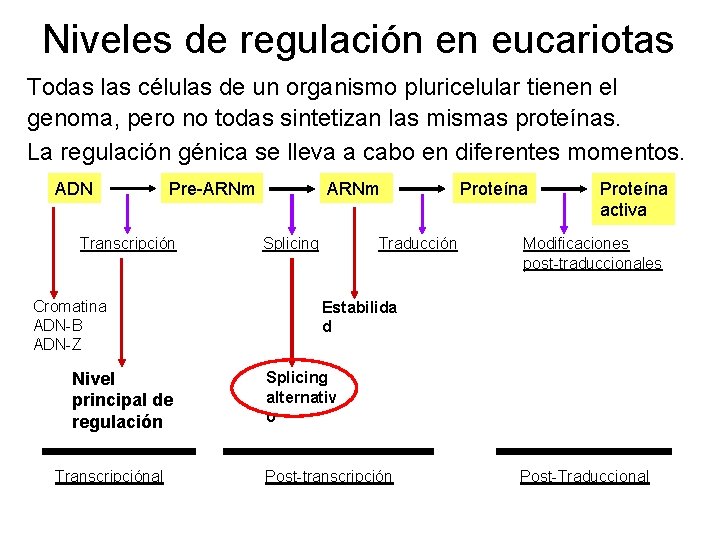
Niveles de regulación en eucariotas Todas las células de un organismo pluricelular tienen el genoma, pero no todas sintetizan las mismas proteínas. La regulación génica se lleva a cabo en diferentes momentos. ADN Pre-ARNm Transcripción Cromatina ADN-B ADN-Z Nivel principal de regulación Transcripciónal ARNm Splicing Traducción Proteína activa Modificaciones post-traduccionales Estabilida d Splicing alternativ o Post-transcripción Post-Traduccional

1) CONFORMACION Y ESTRUCTURA DEL ADN Compactación diferencial de la cromatina • La compactación de la cromatina afecta la capacidad de unión de las enzimas y factores transcripcionales de genes específicos. La cromatina se puede dividir en dos clases según su patrón de tinción. La eucromatina se tiñe suavemente y se corresponde con regiones del genoma que están disponibles para la transcripción. Por otro lado, la heterocromatina, se tiñe intensamente y se corresponde a regiones del genoma que están densamente compactadas e inaccesibles para el aparato transcripcional. Se pueden distinguir dos clases de heterocromatina: la constitutiva y la facultativa. La constitutiva hace referencia a cromosomas o parte de ellos que son heterocromáticos en todas las células de una misma especie, mientras que la facultativa implica zonas de cromosomas que se pueden descompactar tornándose en eucromatina en algunas células de un mismo organismo. • Secuencias características de organización del DNA como los palíndromes así como la disposición espacial del DNA Z han sido relacionados con señalizaciones para el sitio de inicio de la transcripción. Modificaciones covalentes del ADN • Metilaciones de residuos de desoxi citidina

2) CONTROL TRANSCRIPCIONAL DE LA EXPRESION GENETICA • Constituye uno de los modos más importantes de regulación de la expresión proteica en eucariontes. En esta categoría están incluídos los promotores, la presencia de secuencias regulatorias potenciadoras (enhancers), y la interacción entre múltiples proteínas activadoras o inhibidoras que actúan mediante su unión a secuencias específicas de reconocimiento al ADN.

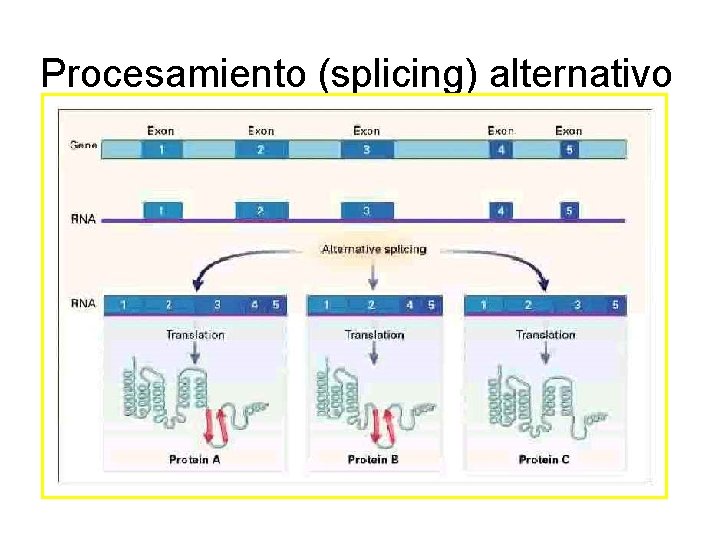
3) CONTROL POST-TRANSCRIPCIONAL DE LA EXPRESION GENETICA SITIOS DE POLIADENILACION ALTERNATIVA: • En este caso hallándose sobre un mismo gen varios sitios susceptibles de ser poliadenilados como es el caso del gen para la cadena pesada m de inmunoglobulina. Dependiendo del sitio 3’ poliadenilado, la proteína resultante puede anclarse a membrana o ser secretada, de acuerdo al estadío de desarrollo en que se encuentra la célula. SPLICING ALTERNATIVO: • Es un mecanismo muy difundido, que permite que un solo gen pueda codificar para más de una proteína. En muchos de estos casos se conoce más de una vía para procesar el transcripto primario obteniendo proteínas estructural y funcionalmente diferentes o bien isoformas de una proteína. El mecanismo por el cual la célula selecciona los sitios no está claro.

Procesamiento (splicing) alternativo

EDICIÓN DEL ARN (Mutagénesis dirigida del ARN): • Si bien el splicing alternativo es la forma mejor estudiada y más habitual de regulación de la expresión genética a nivel de procesamiento del ARN, no es la única. La mutación puntual de una base en el ARNnh o en el ARNm, puede alterar el producto. Esto no constituye un error sino que es una herramienta utilizada por la célula para lograr una determinada proteína. En el ser humano este es el caso de las apoproteínas B 100 y B 48. Ambas son codificadas por el mismo gen y contienen en el ARNnh los mismos exones e intrones. Cuando el gen se expresa en el hepatocito, el ARNnh no sufre ningún tipo de modificación y el RNAm procesado se traduce en Apo. B 100. En cambio, cuando el gen se expresa en el enterocito se produce una mutación puntual que convierte C por U en el ARNm configurando un codón de terminación que codifica para la Apo. B 48.

4)- CONTROL TRADUCCIONAL DE LA EXPRESION GENETICA • Se puede regular la velocidad con que los mensajeros son traducidos y el número de veces que debe traducirse. • CONTROL GENERAL DE LA TRADUCCIÓN: Todos los ARNm celulares tienen cap con la guanosina metilada y esa estructura aumenta la traducción. Cuando por alguna razón de disposición espacial del extremo 5’ del ARN el cap no queda disponible para su interacción el factor de iniciación Ife 4 E la traducción disminuye. Puede controlarse también la actividad de los factores de iniciación: IFe 2 B, IFe 3, IFe 4 B e IFe 4 F. La actividad de estos factores se controla por fosforilación. • CONTROL PARTICULAR DE LA TRADUCCIÓN : Depende de sustancias reguladoras que modifican la configuración de un tramo de nucleótidos no traducibles localizados en el extremo 5’ entre el cap y el codón de iniciación. • Un ejemplo de este tipo de regulación lo proporciona la traducción del mensajero de la Ferritina que se une al hierro para su depósito intracelular. Cuando las concentraciones citosólicas de hierro aumentan la síntesis de ferritina se produce a altas velocidades, el hierro se liga a la aconitasa o IRF. En cambio, cuando estas concentraciones disminuyen la IRF queda libre y se liga al ARNm de la ferritina formando un bucle en el extremo 5’ que impide el ligado de IFe 4 F y con ello el ensamblaje del aparato de traducción, bloqueando de esta manera la producción innecesaria de proteína.

5)- CONTROL POSTRADUCCIONAL DE LA EXPRESION GENETICA Entre las modificaciones más importantes: • La unión de grupos prostéticos • La modificación de ciertos aminoácidos mediante fosforilaciones, metilaciones etc. . • La eliminación de uno o más aminoácidos del extremo aminoterminal • La eliminación de segmentos de aminoácidos internos. • La formación de puentes disulfuro que ayudan al plegamiento. • La asociación con otras cadenas polipeptídicas, que definen la estructura cuaternaria de la proteína funcional.

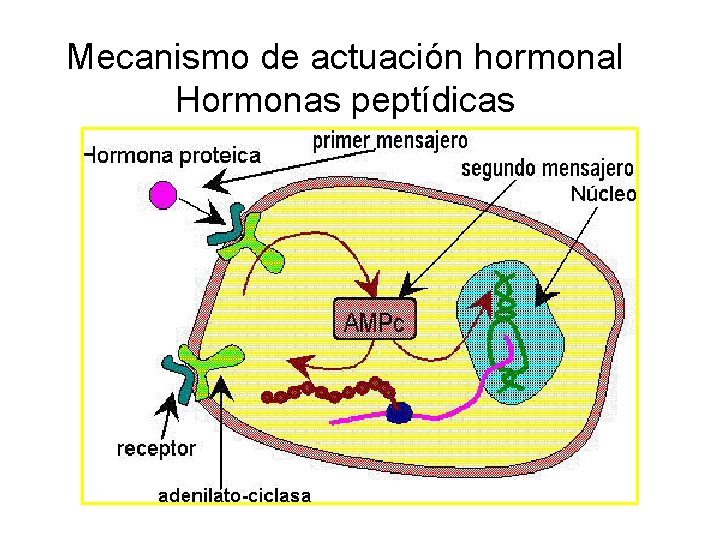
Mecanismo de actuación hormonal Hormonas peptídicas

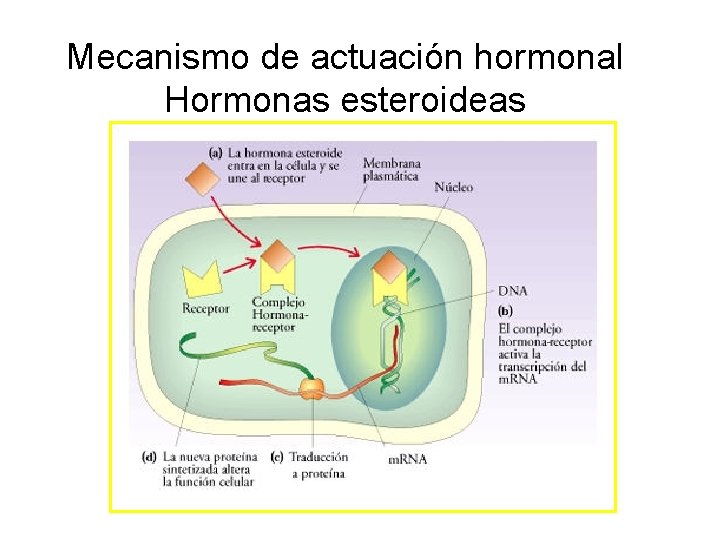
Mecanismo de actuación hormonal Hormonas esteroideas