ESTRUCTURA DEL ADN Y CLASIFICACION DE LAS SECUENCIAS
































- Slides: 55

ESTRUCTURA DEL ADN Y CLASIFICACION DE LAS SECUENCIAS DEL GENOMA HUMANO

Estructura del ADN • 1953 James Watson y Francis Crick

Descubriendo la estructura del ADN Imagen del ADN de Rosalind Franklin “Regla de Chargoff” A=T & C=G

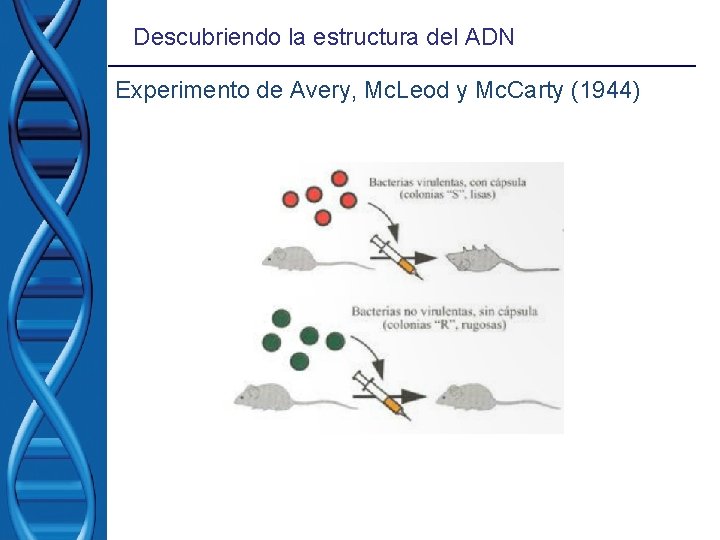
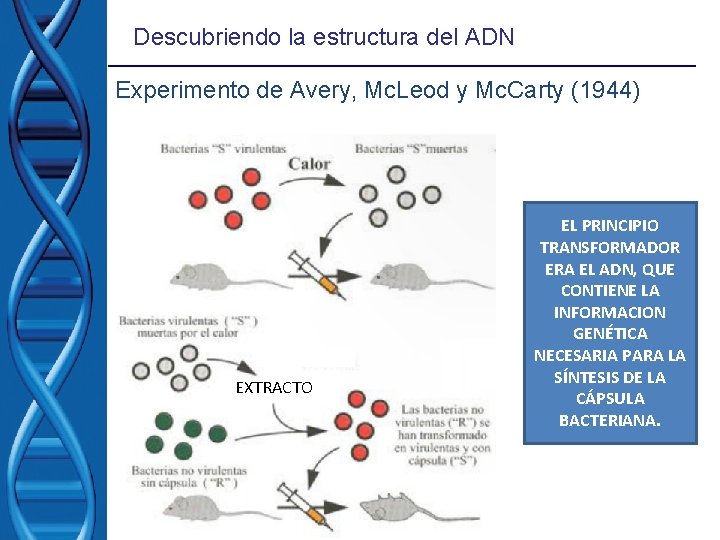
Descubriendo la estructura del ADN Experimento de Avery, Mc. Leod y Mc. Carty (1944)

Descubriendo la estructura del ADN Experimento de Avery, Mc. Leod y Mc. Carty (1944) EXTRACTO EL PRINCIPIO TRANSFORMADOR ERA EL ADN, QUE CONTIENE LA INFORMACION GENÉTICA NECESARIA PARA LA SÍNTESIS DE LA CÁPSULA BACTERIANA.

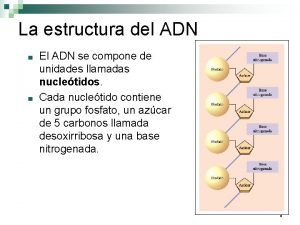
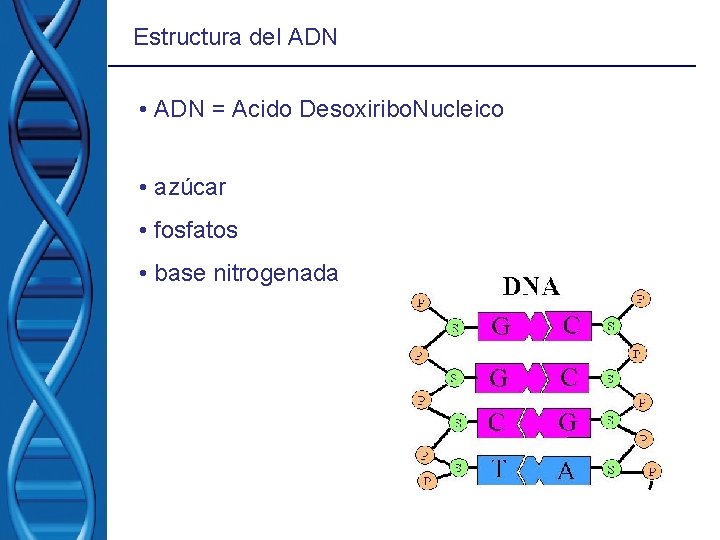
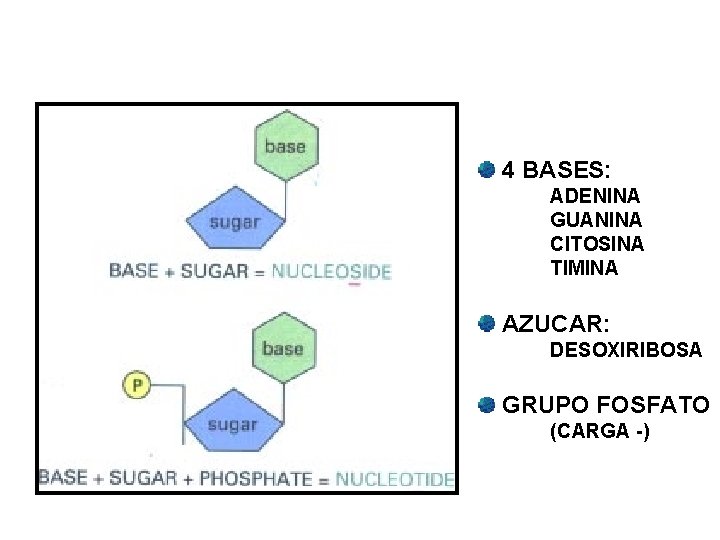
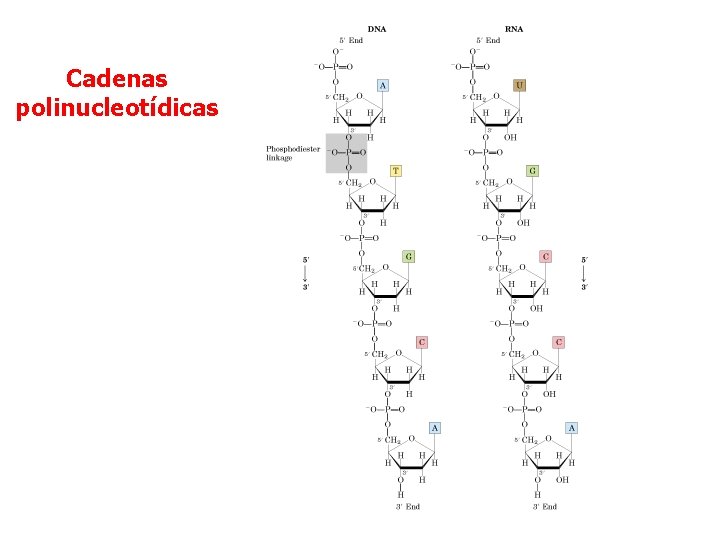
Estructura del ADN • ADN = Acido Desoxiribo. Nucleico • azúcar • fosfatos • base nitrogenada

4 BASES: ADENINA GUANINA CITOSINA TIMINA AZUCAR: DESOXIRIBOSA GRUPO FOSFATO (CARGA -)

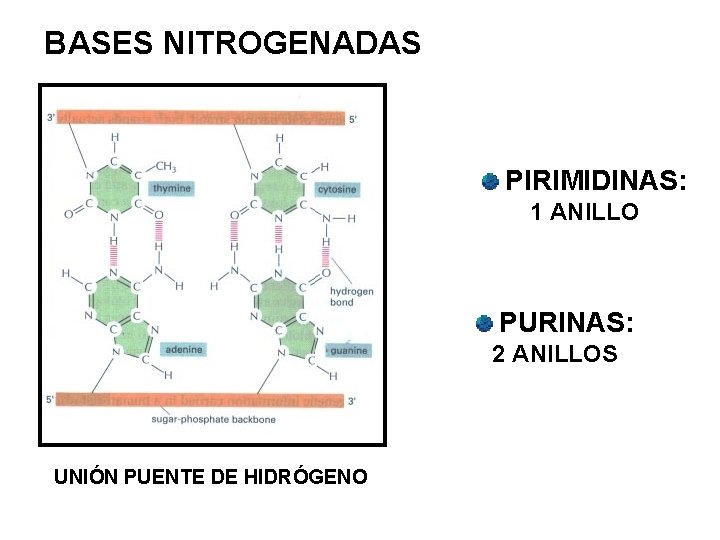
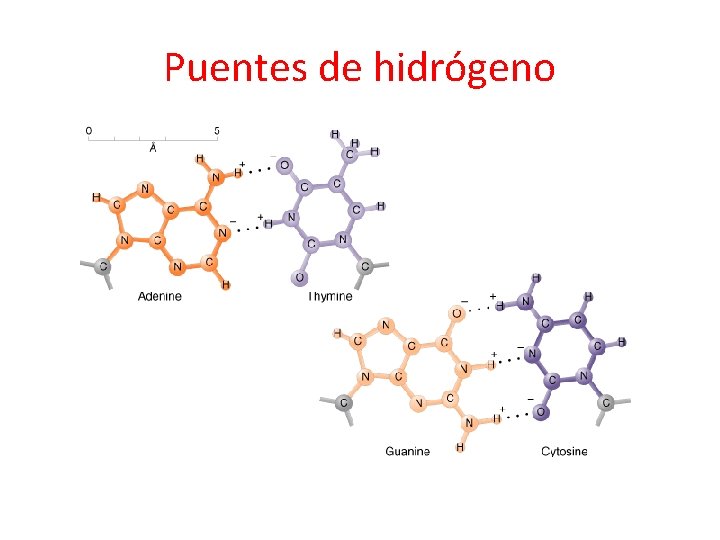
BASES NITROGENADAS PIRIMIDINAS: 1 ANILLO PURINAS: 2 ANILLOS UNIÓN PUENTE DE HIDRÓGENO

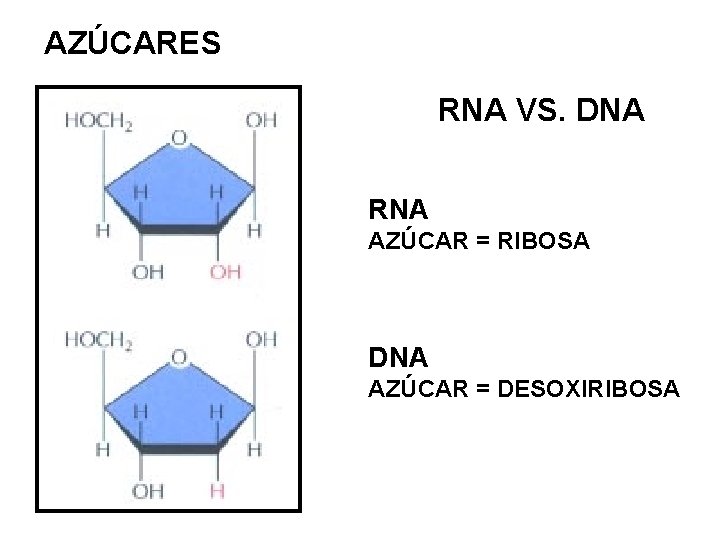
AZÚCARES RNA VS. DNA RNA AZÚCAR = RIBOSA DNA AZÚCAR = DESOXIRIBOSA

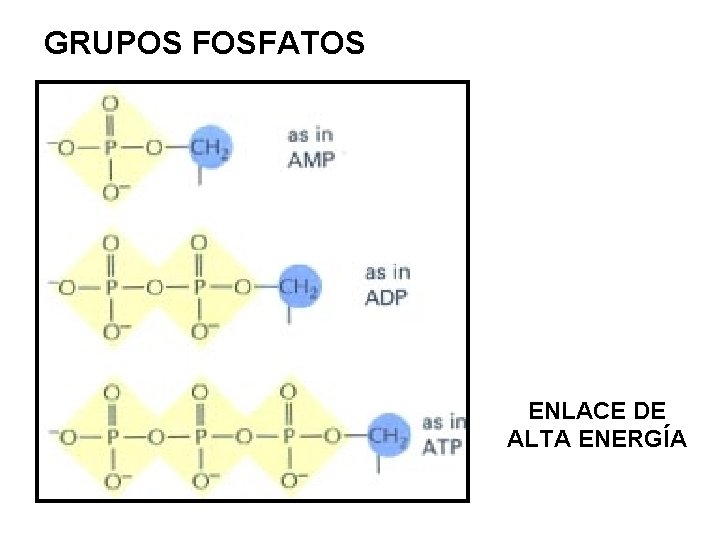
GRUPOS FOSFATOS ENLACE DE ALTA ENERGÍA


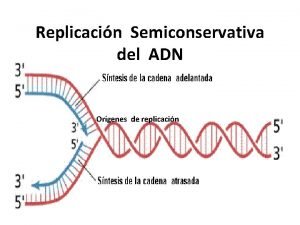
Cadenas polinucleotídicas

Puentes de hidrógeno

DECODIFICANDO EL GENOMA HUMANO: LA ENCICLOPEDIA DE LA VIDA El 14 de abril de 2003 4 bases o «letras» (A, T, C y G) se anunció la terminación exitosa del Proyecto Genoma Humano

4 bases o «letras» repetidas 3 millones de veces y agrupadas en libros o «cromosomas»

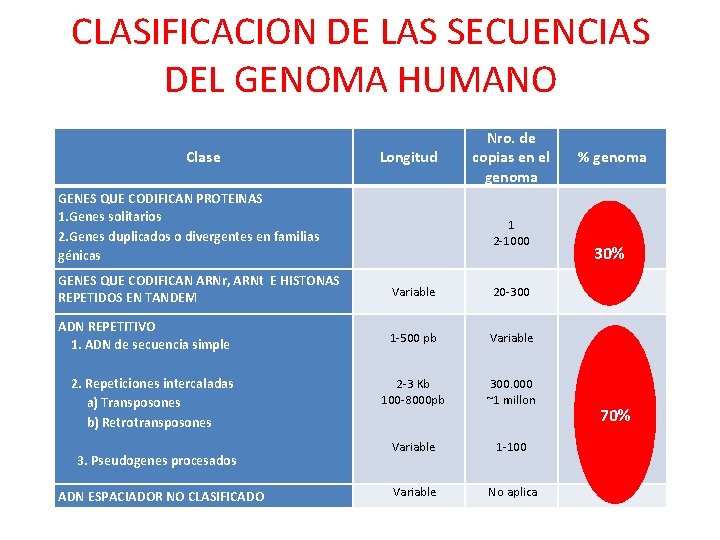
CLASIFICACION DE LAS SECUENCIAS DEL GENOMA HUMANO Clase GENES QUE CODIFICAN PROTEINAS 1. Genes solitarios 2. Genes duplicados o divergentes en familias génicas GENES QUE CODIFICAN ARNr, ARNt E HISTONAS REPETIDOS EN TANDEM ADN REPETITIVO 1. ADN de secuencia simple 2. Repeticiones intercaladas a) Transposones b) Retrotransposones 3. Pseudogenes procesados ADN ESPACIADOR NO CLASIFICADO Longitud Nro. de copias en el genoma 1 2 -1000 % genoma ≈15% 30% Variable 20 -300 0, 3% 1 -500 pb 2 -3 Kb 100 -8000 pb Variable 300. 000 ~1 millon 3% 3% 40% 70% Variable 1 -100 ≈0, 4% Variable No aplica ≈25%

EL NÚMERO DE GENES ENCONTRADO FUE MENOR AL ESPERADO 50. 000 a 140. 000 20. 500

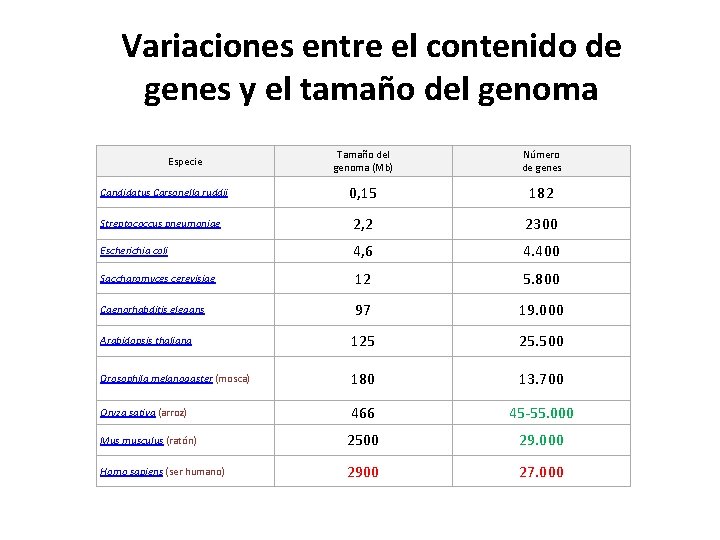
Variaciones entre el contenido de genes y el tamaño del genoma Tamaño del genoma (Mb) Número de genes 0, 15 182 Streptococcus pneumoniae 2, 2 2300 Escherichia coli 4, 6 4. 400 Saccharomyces cerevisiae 12 5. 800 Caenorhabditis elegans 97 19. 000 Arabidopsis thaliana 125 25. 500 Drosophila melanogaster (mosca) 180 13. 700 Oryza sativa (arroz) 466 45 -55. 000 Mus musculus (ratón) 2500 29. 000 Homo sapiens (ser humano) 2900 27. 000 Especie Candidatus Carsonella ruddii

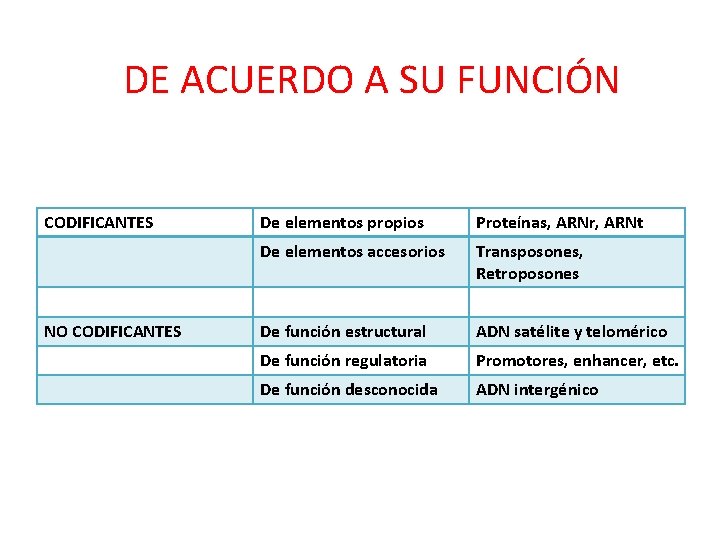
DE ACUERDO A SU FUNCIÓN CODIFICANTES NO CODIFICANTES De elementos propios Proteínas, ARNr, ARNt De elementos accesorios Transposones, Retroposones De función estructural ADN satélite y telomérico De función regulatoria Promotores, enhancer, etc. De función desconocida ADN intergénico

DE ACUERDO AL GRADO DE REPETICION Tipo Denominación Características De alta repetición ADN satélites 106 copias Retroposones LINEs, SINEs, Pseudogenes procesados 10 -104 copias o más Transposones 10 -102 copias Familias génicas ARNr 18 S, ARNr 28 S, ARNt, etc: 102 copias ADN telomérico (TTTAGGG)n: 104 copias ADN minisatélite Muy polimórfico: 102 -103 copias ADN microsatélite (CA)n; (TG)n Muy polimórfico: 105 copias Genes de proteínas Copia única = estructura génica ADN intergénico no repetido Función desconocida De moderada repetición De secuencia única

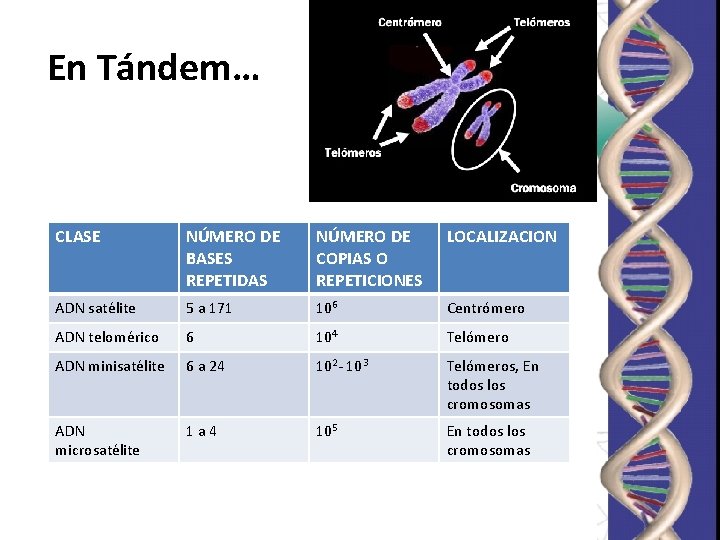
LAS REPETICIONES PUEDEN ESTAR… • EN TÁNDEM • INTERDISPERSAS

En Tándem… CLASE NÚMERO DE BASES REPETIDAS NÚMERO DE COPIAS O REPETICIONES LOCALIZACION ADN satélite 5 a 171 106 Centrómero ADN telomérico 6 104 Telómero ADN minisatélite 6 a 24 102 - 103 Telómeros, En todos los cromosomas ADN microsatélite 1 a 4 105 En todos los cromosomas


Repeticiones interdispersas: Transposones y Retroposones

ADN móvil • Moderadamente repetidas. • Dispersas a lo largo del genoma de procariotas y eucariota. • Tamaño variable (100 - 1000000 pb). • Transposición. • Inútiles para el organismo huésped.

Elementos transponibles o móviles • Aprox. 40% del genoma humano. • Amplificación permanente. • Su transposición pueden contribuir al desarrollo de enfermedades. • Todos los genomas eucariotas tiene elementos móviles. • Mutagénico por su capacidad de insertarse y generar recombinaciones homólogas desiguales.

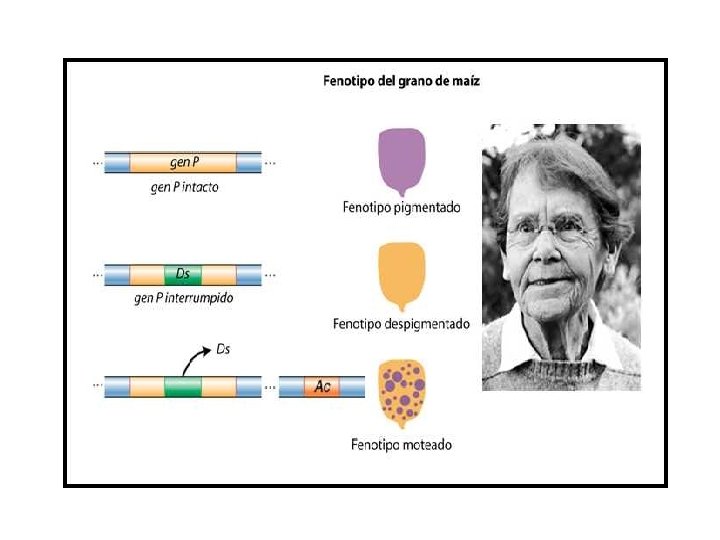
En 1950 Barbara Mc. Clintock descubre los elementos transponibles Ac controla la transposición de Ds y si Ds no está presente, se expresa el color de la aleurona del maíz. Unidades de control, o elementos reguladores. Activator (Ac) Dissociator (Ds)


Tipos de ADN repetitivo interdisperso

Transposones y Retroposones

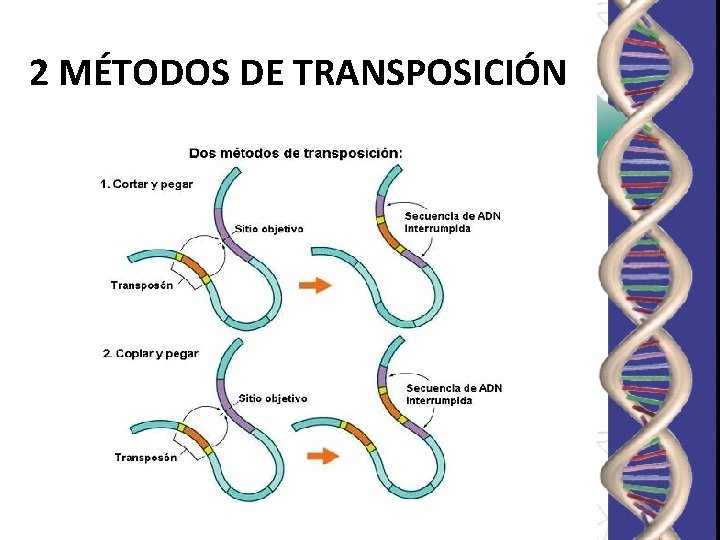
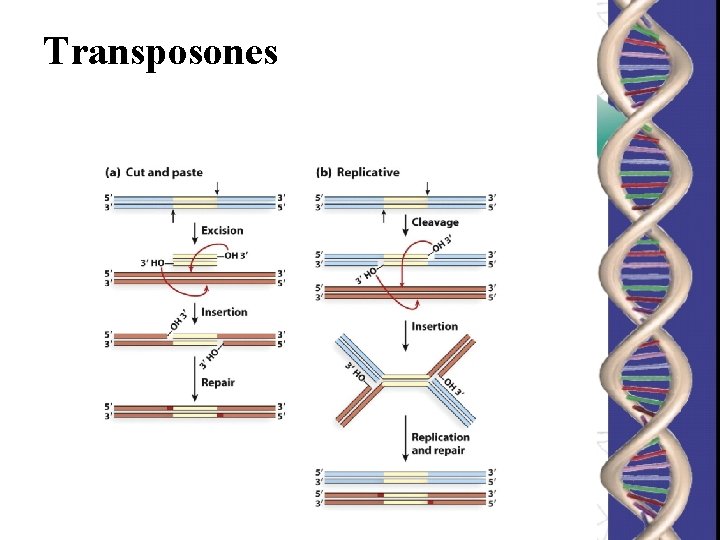
Transposones o genes saltarines: “Los elementos transponibles que se mueven a través de un intermediario de ADN se conoce como transposones”.

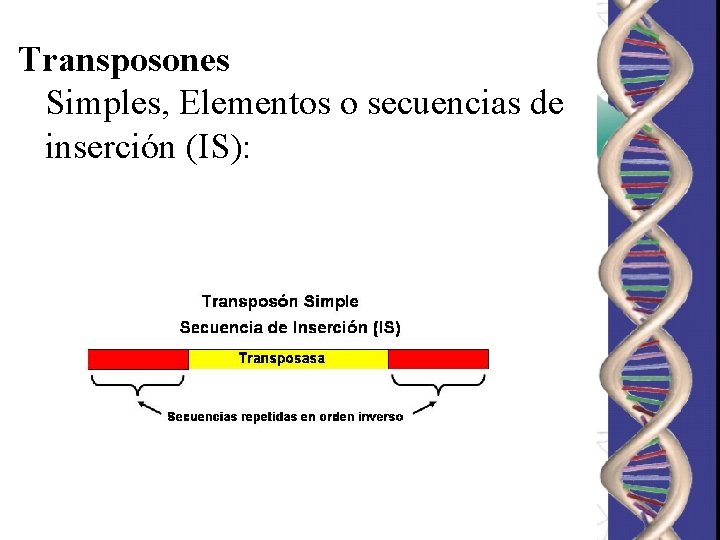
Transposones o genes saltarines: § Aprox. 0, 3 Kb. § Extremos con secuencias denominadas ITR (Inverted Terminal Repeats). §Codifica para una enzima transposasa. §Existen 20 secuencias de inserción diferentes. §Clasificación: ØSimples ØCompuestos

Transposones Simples, Elementos o secuencias de inserción (IS):

Transposones Compuestos (Tn): Poseen información en la zona central para resistencia a antibióticos como el cloranfenicol, kanamicina, o la tetraciclina. Sirven para la transferencia de información entre bacterias.

2 MÉTODOS DE TRANSPOSICIÓN

Transposones

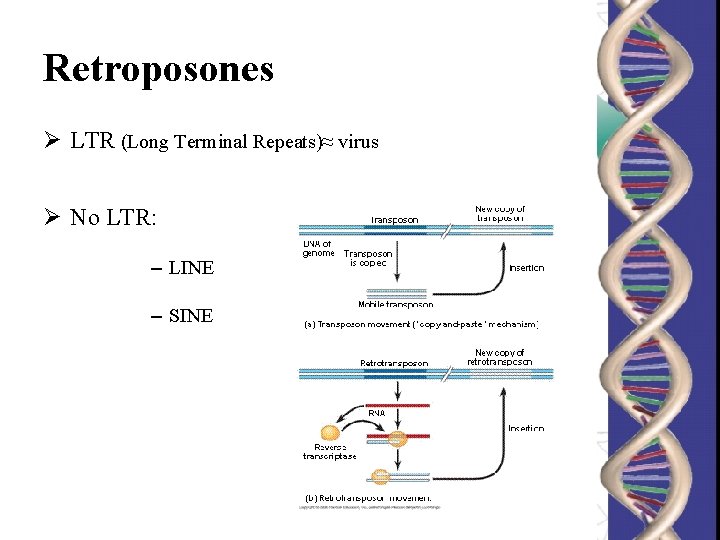
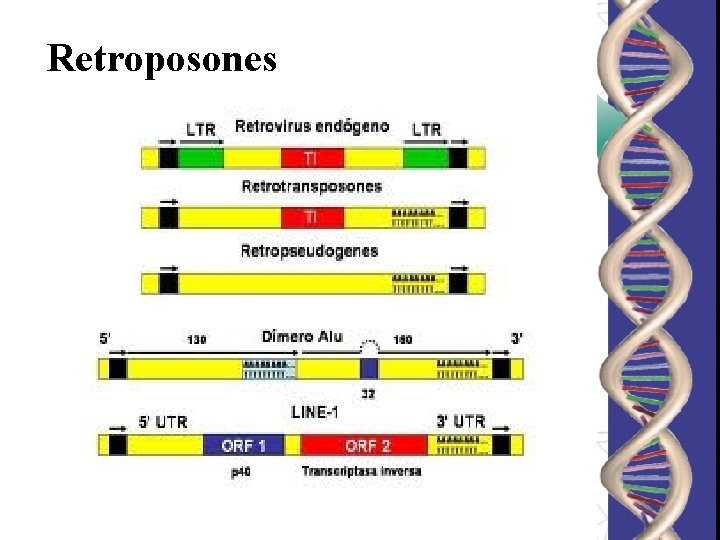
Retroposones: “Los elementos transponibles que se mueven a través de un intermediario de ARN se denomina retroposones” por su similaridad con el mecanismo de replicación de retrovirus.

Retroposones Ø LTR (Long Terminal Repeats)≈ virus Ø No LTR: – LINE – SINE

Retroposones

Elementos LINEs (Long Interspersed Elements) • Tienen 6, 4 kb y se repiten 10. 000 veces. • Se encuentran en zonas del genoma ricas en A + T. • Transcritos por ARNpol. II. • 2 Marcos de lecturas abiertos (ORF): ORF 1: Proteína de unión al ARN. ORF 2: Transcriptasa y endonucleasa. • Numerosas mutaciones y truncamientos llevaron a que solo el 0, 01% de los LINEs sean funcionales. • En humanos hay 3 tipos de LINEs: L 1, L 2, L 3.

LINEs: Mecanismo de transcripción e integración

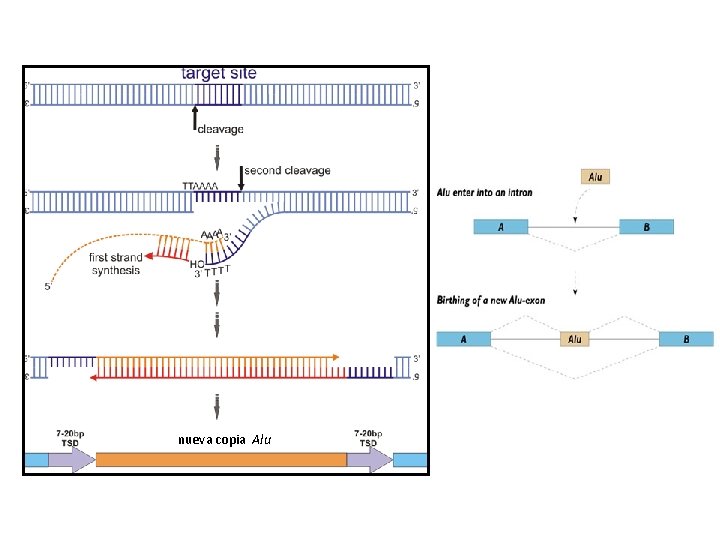
Elementos SINEs (Short Interspersed Elements): Secuencias Alu • Secuencias de aprox. 300 nt. • Transcriptos por RNApol III. • Denominadas Alu porque son reconocidas por esta enzima de restricción. • Se ubican en regiones ricas en G + C. • El total de las secuencias Alu ocupan el 5% del genoma humano. • Comparten homología con la secuencia de 7 SL ARN, componente de la “Partícula de Reconocimiento Señal”. • No codifica proteínas.

Consecuencias de las secuencias Alu en el genoma humano • Inserción de Alu en el intrón 5 del gen NF 1 = Neurofibromatosis I. • Inserción de Alu en genes factor VIII y IX = Hemofilia A y B • Inserción de Alu en el gen CLCN 5 = enfermedad de Dent (alteración del canal de Cloro).

nueva copia Alu

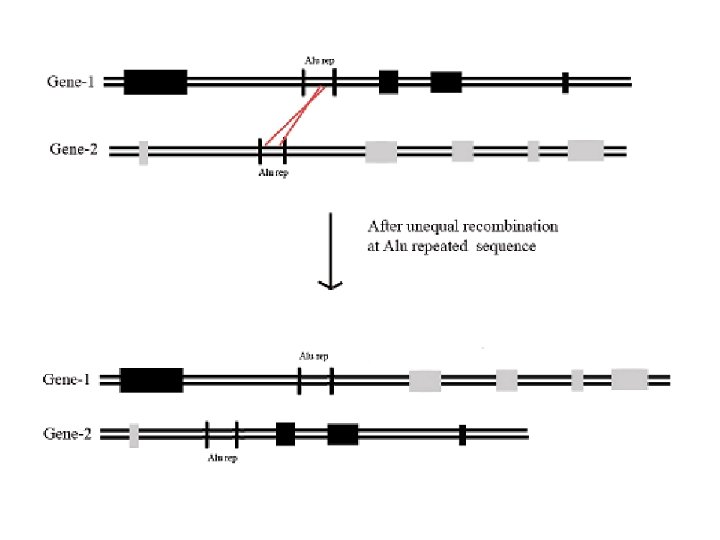
La recombinación de secuencias repetitivas Alu es causante de: • Hipercolesterolemia familiar, • Enfermedad de Fabry (insuficientes cantidades de una enzima llamada alfagalactosidasa A), • Enfermedad de Sandhoff (de la flia. de gangliosidosis 2: almacena y sobrecarga el organismo en gangliósidos), • Enfermedad de Tay-Sachs (alteración de metabolismo lipídico), • Deficiencia ADA (cataliza la deasaminación irreversible de adenosina y 2'deoxyadenosina a inosina), • Hyperlipoproteinemia tipo I, etc


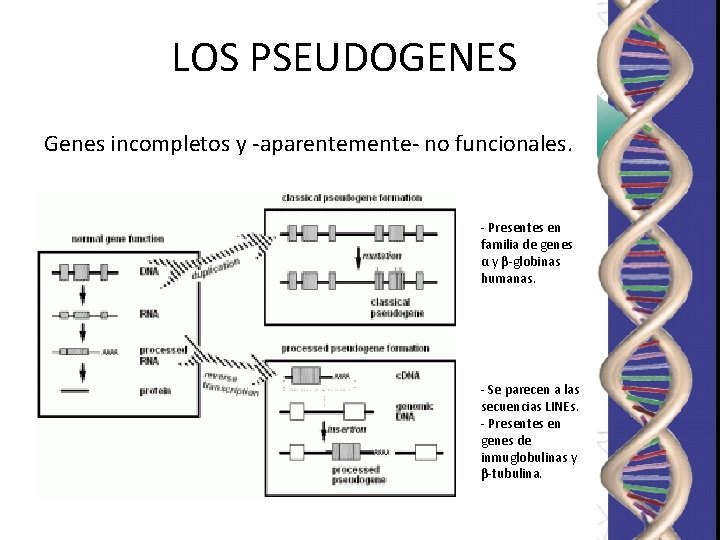
LOS PSEUDOGENES Genes incompletos y -aparentemente- no funcionales. - Presentes en familia de genes α y β-globinas humanas. - Se parecen a las secuencias LINEs. - Presentes en genes de inmuglobulinas y β-tubulina.

NO TAN «ADN BASURA» El pseudogen PTENP 1 protege a su homólogo PTEN de la degradación, permitiendo que realice su función como gen supresor de tumores.

Gen egoísta? ? . . . …o fuente de variabilidad genética? ?

Probablemente los “elementos de ADN” tuvieron influencia significativa en la Evolución…

VARIABILIDAD DEL GENOMA • Polimorfismos Cambios en la secuencia del ADN que ocurre con una frecuencia relativamente elevada, en general en más del 1% de la población. Existen distintos tipos de polimorfismos, y si bien originan una variabilidad genética normal, algunos de ellos pueden afectar la estructura o expresión de una proteína, y por ende, tener una consecuencia biológica. Es allí donde radica la importancia médica del estudio y comprensión de los polimorfismos genéticos.

• Polimorfismos de secuencia: Genes HLA • Polimorfismos de longitud: Mini y Microsatélites • Polimorfismos de nucleótido único (SNPs) Son los más frecuentes y pueden asociarse a determinadas enfermedades o respuestas a fármacos.

VARIABILIDAD DEL GENOMA • Mutaciones Cambios en la secuencia del ADN que ocurre en general en menos del 1% de la población. Son producto de errores, es decir, no originan una variabilidad genética normal.


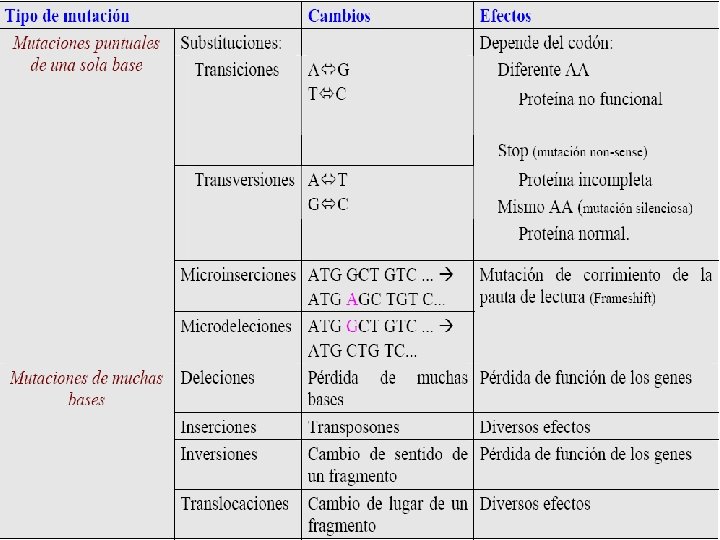
CONSECUENCIAS DE LAS MUTACIONES EN LA SÍNTESIS PROTEICA • Mutación Silenciosa: Tripletes que codifican el mismo aminoácido. Ej: AAG(Arg) CGG (Arg) • Mutación Cambio de Sentido: Tripletes que codifican para aminoácidos de distinto tipo. La proteína pierde su función. • Mutación Sin sentido: Aparece un triplete de STOP. Ej: CAG(Gln) UAG (STOP) • Mutación Cambio de marco de lectura: Adición o deleción de un único par de nucleótidos o de varios pares de nucleótidos, siempre que no sean múltiplos de tres. • Mutación sin Cambio de marco de lectura: Adición o deleción de un único par de nucleótidos o de varios pares de nucleótidos, siempre que sean múltiplos de tres. • Mutación de Terminación retrasada: Se pierde un triplete de STOP.
 Purinas
Purinas Como esta formado el adn
Como esta formado el adn Ribosa
Ribosa Uso de la secuencia expositiva en el reportaje
Uso de la secuencia expositiva en el reportaje Estructura de la célula eucariota que protege el adn
Estructura de la célula eucariota que protege el adn Grados de compactacion del adn
Grados de compactacion del adn Recombinación genética
Recombinación genética Nucletidos
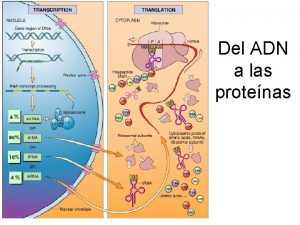
Nucletidos Transcripcion
Transcripcion Cual es la funcion del adn
Cual es la funcion del adn Componentes del adn
Componentes del adn Site:slidetodoc.com
Site:slidetodoc.com Isomeria de carbohidratos
Isomeria de carbohidratos Conclusión del adn
Conclusión del adn Diferencias del adn y arn
Diferencias del adn y arn Electroforesis
Electroforesis Azucar del adn
Azucar del adn Bases nitrogenadas del adn
Bases nitrogenadas del adn Reticulo liso funcion
Reticulo liso funcion Que es el adn
Que es el adn Fases del ciclo celular
Fases del ciclo celular Propiedades del adn
Propiedades del adn Traduccion del adn
Traduccion del adn Plsmidos
Plsmidos Desnaturalizacion del adn
Desnaturalizacion del adn Ciclinas
Ciclinas Clonacion reproductiva
Clonacion reproductiva Esquema conceptual del adn
Esquema conceptual del adn Funcion del adn
Funcion del adn Esquemas textuales
Esquemas textuales Gradiente de eco
Gradiente de eco Secuencia textual
Secuencia textual Secuencias textuales ejercicios
Secuencias textuales ejercicios Forma discursiva ejemplos
Forma discursiva ejemplos Tipos de secuencia textual
Tipos de secuencia textual Fases de la secuencia formativa
Fases de la secuencia formativa Fundamentacion de la secuencia formativa
Fundamentacion de la secuencia formativa Diagrama de implementación uml
Diagrama de implementación uml Secuencia incompleta
Secuencia incompleta Grupo funcional carbonilo
Grupo funcional carbonilo Estructuras de secuencia
Estructuras de secuencia Tengo muy presente la fisonomía del clérigo
Tengo muy presente la fisonomía del clérigo Secuencias alfoides
Secuencias alfoides Matriz de transicion
Matriz de transicion El cielo protector en libre mente de fernando
El cielo protector en libre mente de fernando Ejercicios de secuencias textuales
Ejercicios de secuencias textuales Esquema de la secuencia narrativa
Esquema de la secuencia narrativa Secuencias reguladoras
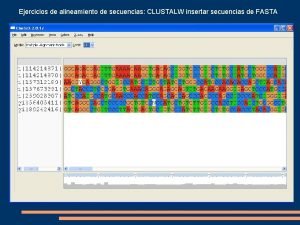
Secuencias reguladoras Blast alineamiento de secuencias
Blast alineamiento de secuencias Estructura interna de un romance
Estructura interna de un romance Cuadro comparativo entre el arn y el adn
Cuadro comparativo entre el arn y el adn Tipos del arn
Tipos del arn Purification adn sur colonne de silice
Purification adn sur colonne de silice Cambio password giustizia
Cambio password giustizia Adn polimerasa
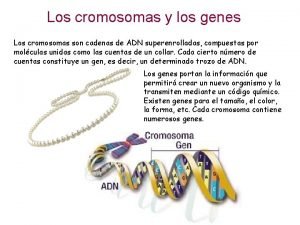
Adn polimerasa Cromosomas
Cromosomas