UNIDAD TEMTICA 4 GENTICA CUANTITATIVA Caracteres Mendelianos o
























- Slides: 54

UNIDAD TEMÁTICA 4 GENÉTICA CUANTITATIVA

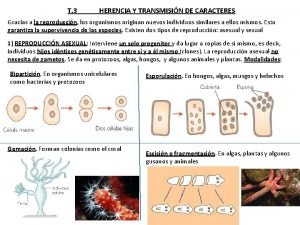
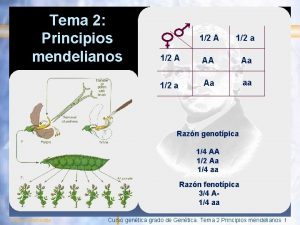
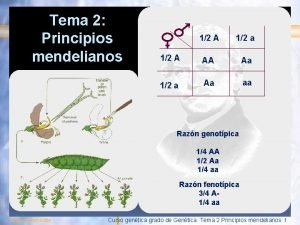
Caracteres Mendelianos o de clase Semilla lisa o rugosa Semilla amarilla o verde Flor violeta o flor blanca Vaina lisa o plegada

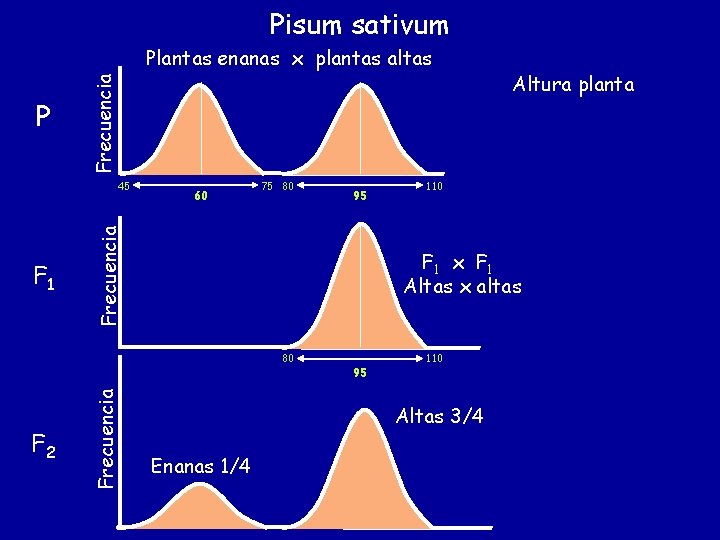
Pisum sativum P Frecuencia Plantas enanas x plantas altas F 1 60 75 80 Frecuencia 110 F 1 x F 1 Altas x altas 80 F 2 95 Frecuencia 45 Altura planta 95 110 Altas 3/4 Enanas 1/4

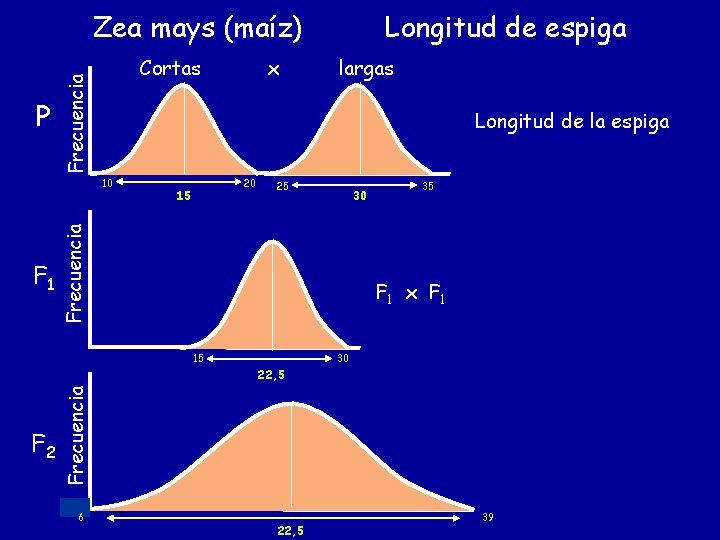
Zea mays (maíz) Frecuencia P Cortas largas Longitud de la espiga 15 20 25 30 Frecuencia 10 F 1 x Longitud de espiga 35 F 1 x F 1 15 30 F 2 Frecuencia 22, 5 6 22, 5 39

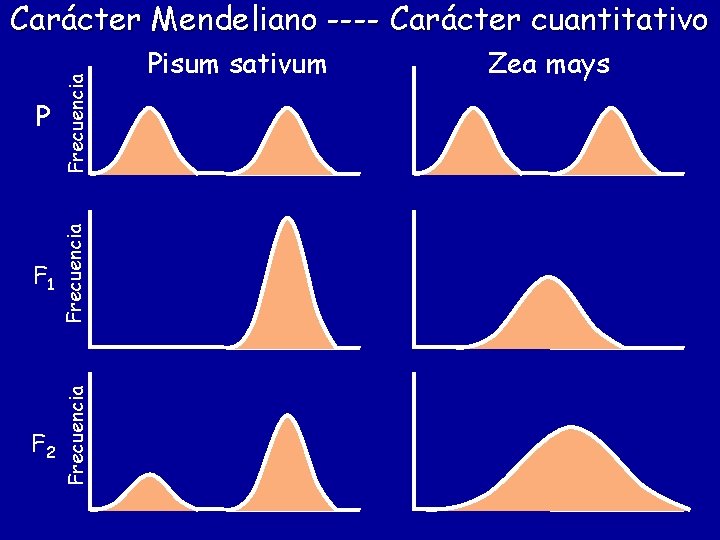
P Frecuencia F 1 Frecuencia F 2 Frecuencia Carácter Mendeliano ---- Carácter cuantitativo Pisum sativum Zea mays

Caracteres cuantitativos Distribución continua

DISTRIBUCIÓN CONTINUA CAUSAS: GENÉTICAS AMBIENTALES Explicadas a través de las experiencias de: Carl Johannsen y Herman Nilsson-Ehle

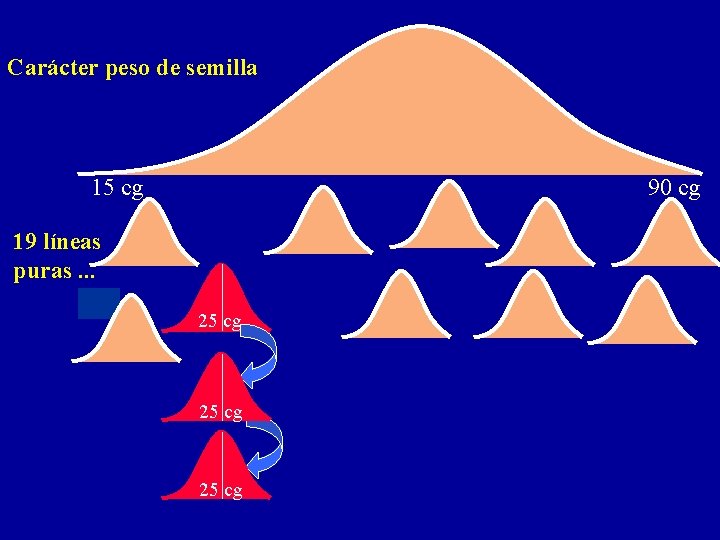
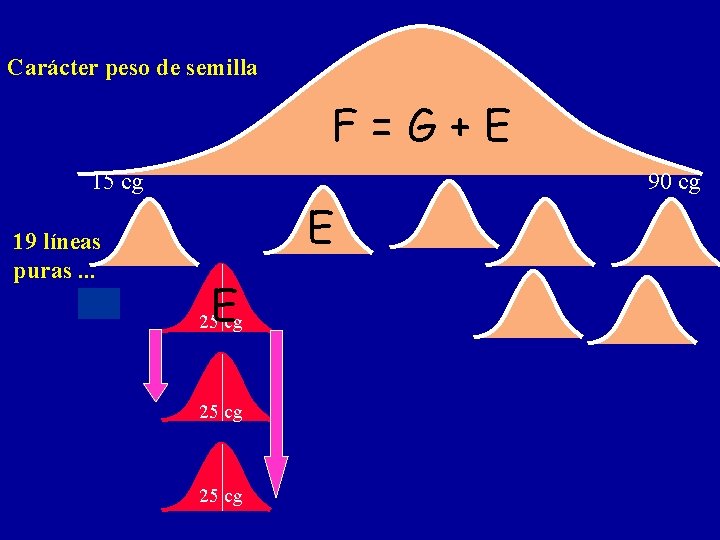
Teoría de la línea pura Johannsen (1903) Phaseolus (Variedad Princesa) Carácter peso de semilla 15 cg 19 líneas puras. . . 90 cg

Carácter peso de semilla 15 cg 90 cg 19 líneas puras. . . 25 cg

Carácter peso de semilla F=G+E 15 cg 19 líneas puras. . . 90 cg E E 25 cg

Experiencia de Nilsson-Ehle (1909) En Trigo: cruza dos líneas puras que diferían en el color de los granos (rojos y blancos) Plantas con Padres granos Rojos F 1 F 2 x Plantas con granos Blancos F 1 intermedia Variación de colores de granos desde el rojo hasta el blanco

Experiencia de Nilsson-Ehle (1909) EXPLICACIÓN: Un par de alelos segregando, sin dominancia P F 1 F 2 Rojo AA x Blanco aa Color intermedio Aa Rojo : Intermedio : Blanco AA Aa aa 1 : 2 : 1

EXPLICACIÓN: Dos pares de alelos segregando, sin dominancia Rojo AABB P Color intermedio (Rojo medio) Aa. Bb F 1 F 2 x Blanco aabb Rojo oscuro: Rojo medio oscuro : Rojo medio : Rojo claro : Blanco AABB 1 Aa. BB AABb : 4 Aa. Bb Aabb aa. BB : 6 Aabb aa. Bb : 4 : 1

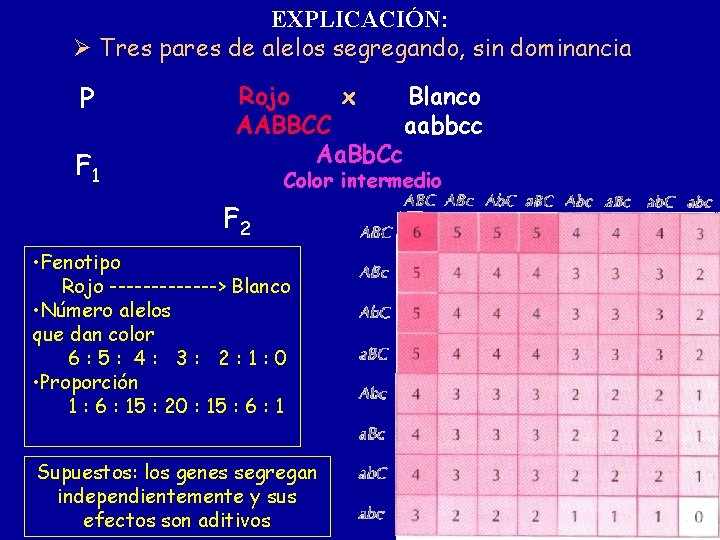
EXPLICACIÓN: Tres pares de alelos segregando, sin dominancia P F 1 Rojo x Blanco AABBCC aabbcc Aa. Bb. Cc Color intermedio F 2 • Fenotipo Rojo -------> Blanco • Número alelos que dan color 6: 5: 4: 3: 2: 1: 0 • Proporción 1 : 6 : 15 : 20 : 15 : 6 : 1 Supuestos: los genes segregan independientemente y sus efectos son aditivos

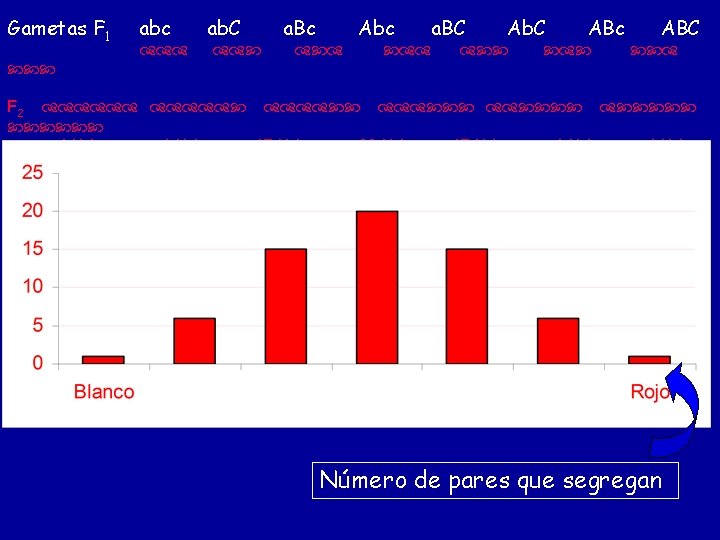
Gametas F 1 abc ab. C a. Bc Abc a. BC Ab. C ABc ABC F 2 1/64 6/64 15/64 20/64 15/64 6/64 1/64 Número de pares que segregan

Dos factores actúan para producir la variación continua: El número de pares que segregan: poligenes o loci múltiples Las variaciones debidas al ambiente

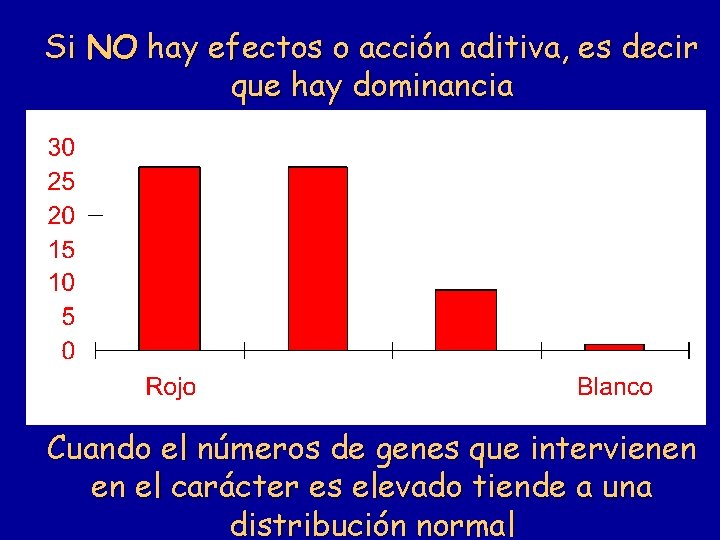
Si NO hay efectos o acción aditiva, es decir que hay dominancia Cuando el números de genes que intervienen en el carácter es elevado tiende a una distribución normal

Caracteres cuantitativos Segregaciones Transgresivas Supongamos un carácter para el cual los alelos en mayúscula de cada loci suman 2 y los minúscula 1 AAbbcc. DDEE 16 Padres F 1 F 2 x aa. BBCCddee 14 Aa. Bb. Cc. Dd. Ee 15 AABBCCDDEE AABBCc. DDEE mayor 20 19 aa. Bbccddee 11 aabbccddee 10 menor

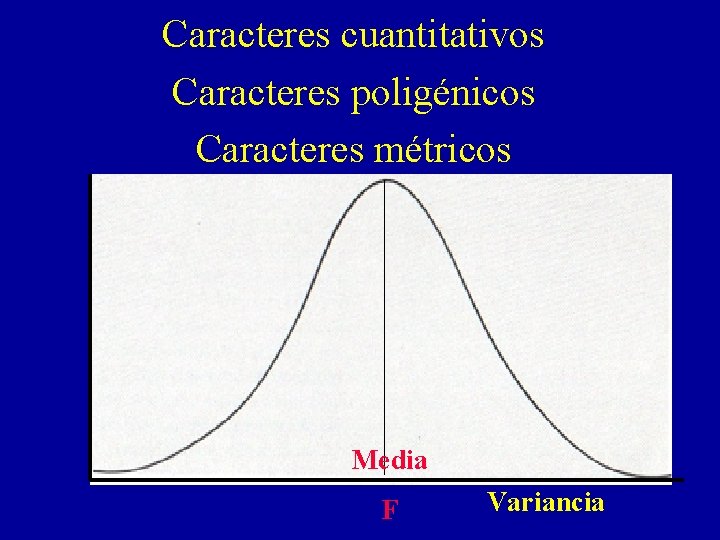
Caracteres cuantitativos Caracteres poligénicos Caracteres métricos Media F Variancia

Caracteres cuantitativos F=G+E Valor fenotípico medio = Valor genotípico medio Modelo estadístico matemático que permite estimar parámetros poblacionales de interés

Caracteres cuantitativos VALOR FENOTÍPICO F=G+E Valor fenotípico medio = Valor genotípico medio

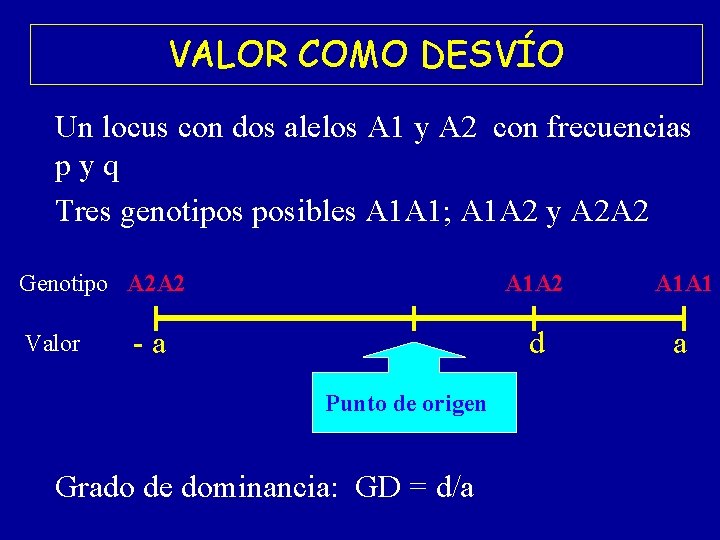
VALOR COMO DESVÍO Un locus con dos alelos A 1 y A 2 con frecuencias p y q Tres genotipos posibles A 1 A 1; A 1 A 2 y A 2 A 2 Genotipo A 2 A 2 Valor A 1 A 2 - a d Punto de origen Grado de dominancia: GD = d/a A 1 A 1 a

Genotipo A 2 A 2 Valor A 1 A 2 - a d A 1 A 1 a Origen = 150 Genotipos A 1 A 1 A 1 A 2 A 2 A 2 Altura (cm) 200 180 100 Valores a = 50 d = 30 - a = -50

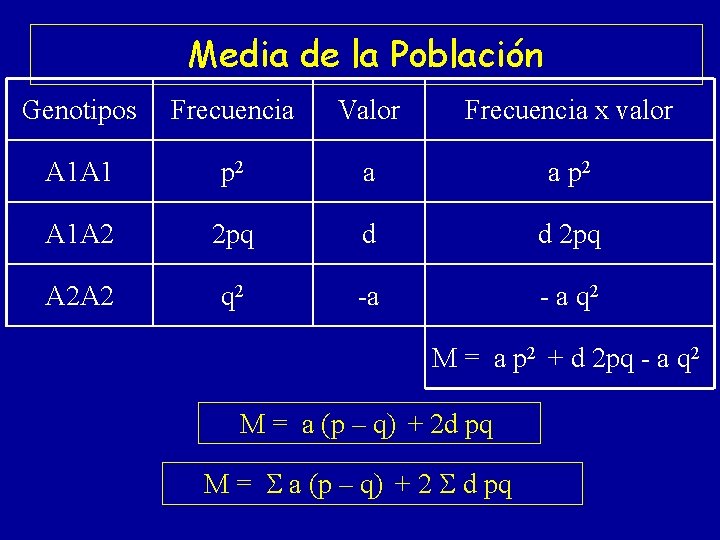
Media de la Población Genotipos Frecuencia Valor Frecuencia x valor A 1 A 1 p 2 a a p 2 A 1 A 2 2 pq d d 2 pq A 2 A 2 q 2 -a - a q 2 M = a p 2 + d 2 pq - a q 2 M = a (p – q) + 2 d pq M = a (p – q) + 2 d pq

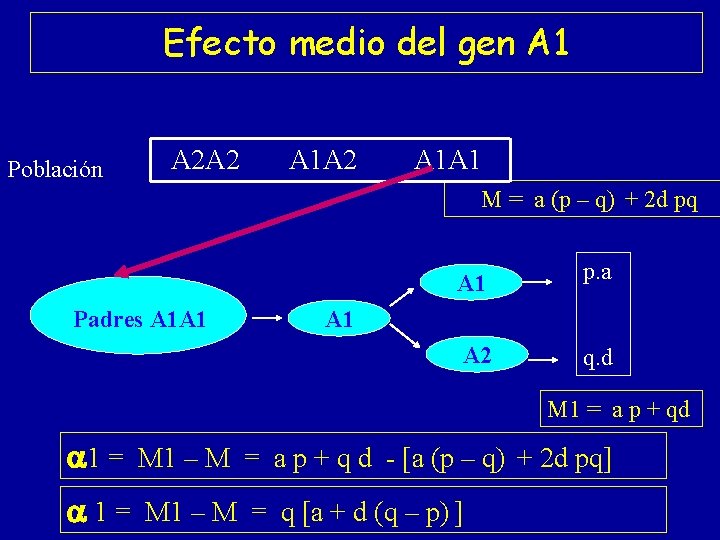
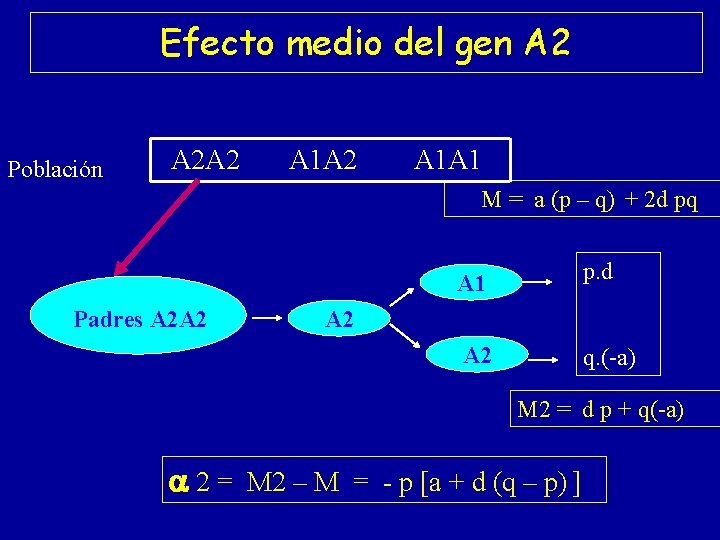
Efecto medio del gen A 1 Población A 2 A 2 A 1 A 1 M = a (p – q) + 2 d pq Padres A 1 A 1 p. a A 2 q. d A 1 M 1 = a p + qd 1 = M 1 – M = a p + q d - [a (p – q) + 2 d pq] 1 = M 1 – M = q [a + d (q – p) ]

Efecto medio del gen A 2 Población A 2 A 2 A 1 A 1 M = a (p – q) + 2 d pq Padres A 2 A 2 A 1 p. d A 2 q. (-a) A 2 M 2 = d p + q(-a) 2 = M 2 – M = - p [a + d (q – p) ]

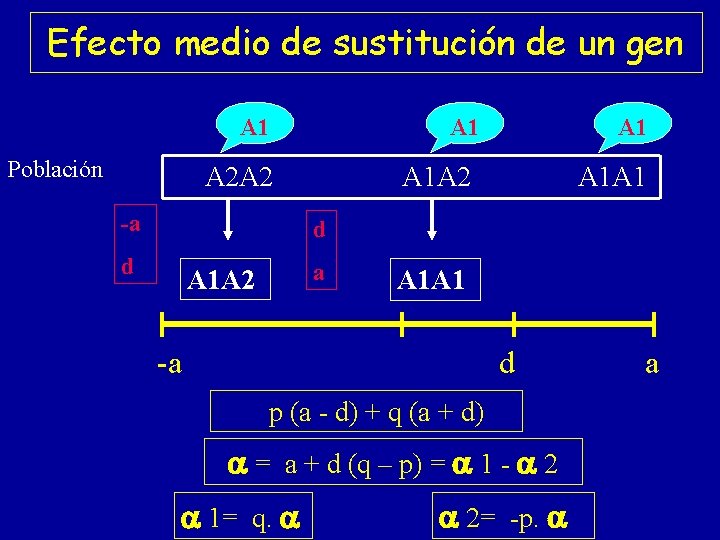
Efecto medio de sustitución de un gen A 1 Población A 1 A 2 A 2 -a A 1 A 2 A 1 A 1 A 1 d d A 1 A 2 a A 1 A 1 -a d p (a - d) + q (a + d) = a + d (q – p) = 1 - 2 1= q. 2= -p. a

Valor Reproductivo (A) GENOTIPO VALOR REPRODUCTIVO (A) A 1 A 1 A 1 A 2 A 2 A 2 2 1 1 + 2 2 = 2 q. 2 = (q – p) = - 2 p.

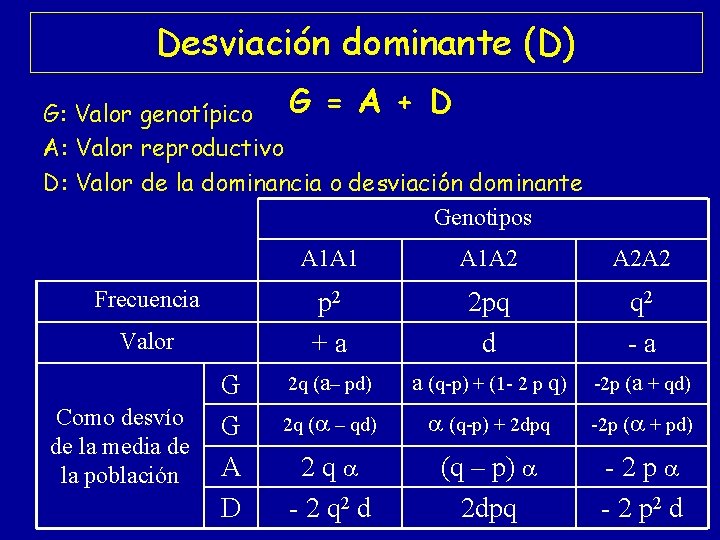
Desviación dominante (D) G = A + D G: Valor genotípico A: Valor reproductivo D: Valor de la dominancia o desviación dominante Genotipos Frecuencia Valor G Como desvío G de la media de A la población D A 1 A 1 A 1 A 2 A 2 A 2 p 2 + a 2 pq d q 2 - a 2 q (a– pd) a (q-p) + (1 - 2 p q) -2 p (a + qd) 2 q ( – qd) (q-p) + 2 dpq -2 p ( + pd) 2 q - 2 q 2 d (q – p) 2 dpq - 2 p 2 d

Desviación de la Interación (I) G=A+D+I

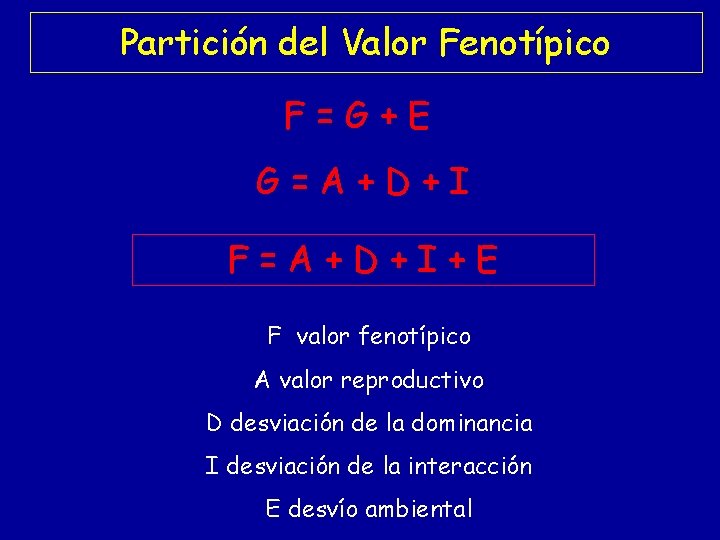
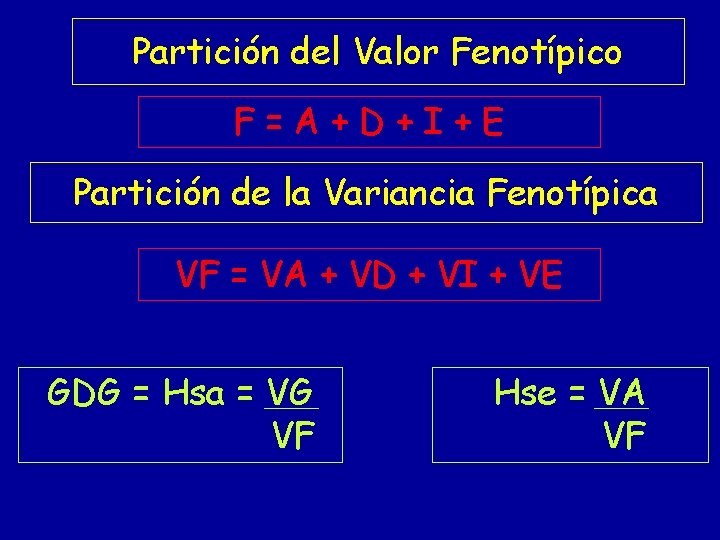
Partición del Valor Fenotípico F=G+E G=A+D+I F=A+D+I+E F valor fenotípico A valor reproductivo D desviación de la dominancia I desviación de la interacción E desvío ambiental

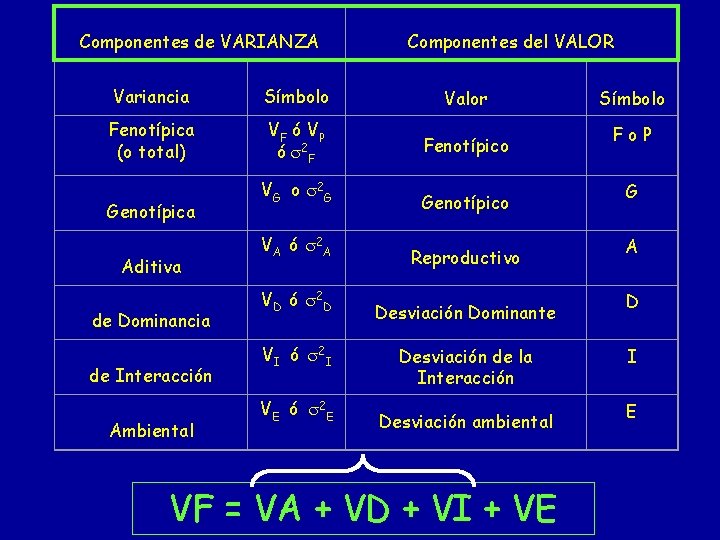
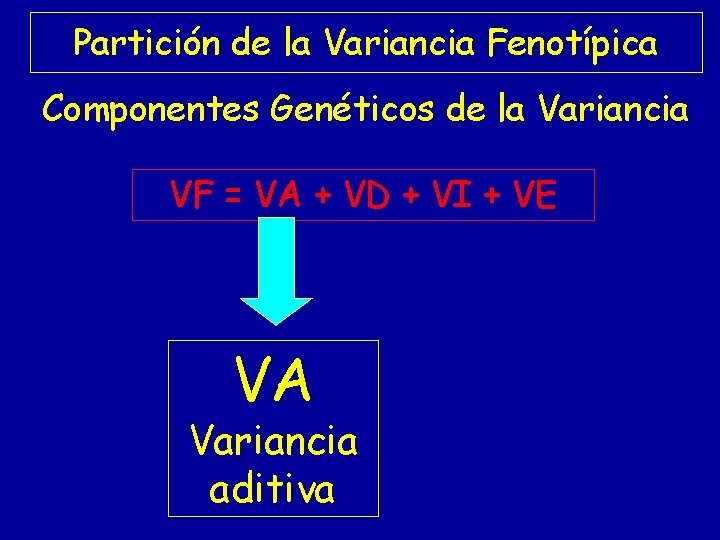
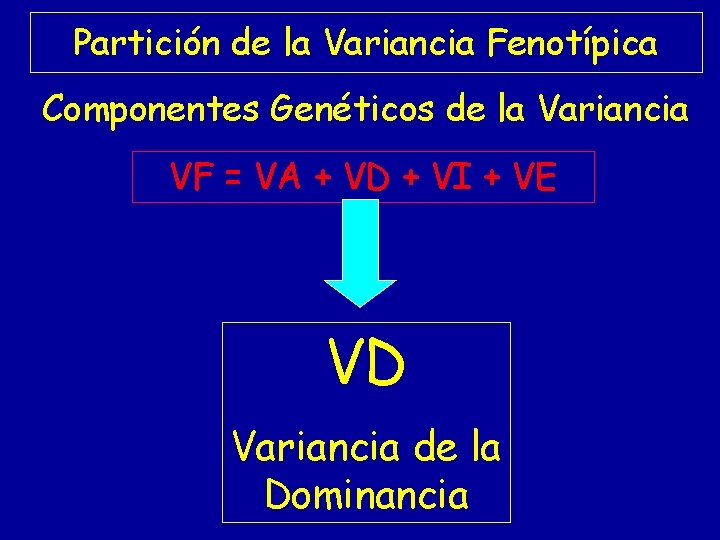
Partición de la Variancia Fenotípica VF = VG + VE VG = VA + VD + VI VF = VA + VD + VI + VE VF variancia fenotípica VA variancia aditiva o del valor reproductivo VD variancia de la dominancia VI variancia de la interacción VE variancia ambiental

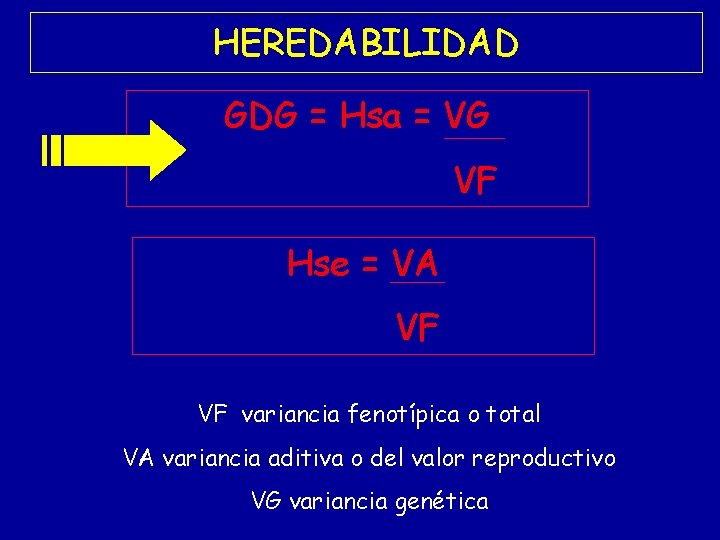
HEREDABILIDAD GDG = Hsa = VG VF Hse = VA VF VF variancia fenotípica o total VA variancia aditiva o del valor reproductivo VG variancia genética

HEREDABILIDAD GDG = Hsa = VG VF Ejemplo: en Drosophila se estudia el carácter longitud del tórax (medido en 1/100 mm). Población Mezclada (variabilidad genética) Componentes Variancia Observada VP = VG + VE 0. 366

HEREDABILIDAD GDG = Hsa = VG VF Ejemplo: en Drosophila se estudia el carácter longitud del tórax (medido en 1/100 mm). Población Mezclada (variabilidad genética) Uniforme (F 1) Componentes Variancia Observada VP = VG + VE 0. 366 VP = VE 0. 186

HEREDABILIDAD GDG = Hsa = VG VF Ejemplo: en Drosophila se estudia el carácter longitud del tórax (medido en 1/100 mm). Población Mezclada Uniforme (F 1) Diferencia Componentes Variancia Observada VP = VG + VE 0. 366 VP = VE 0. 186 VP – VE = VG 0. 180

HEREDABILIDAD GDG = Hsa = VG VF Ejemplo: en Drosophila se estudia el carácter longitud del tórax (medido en 1/100 mm). Población Componentes Variancia Observada VP = VG + VE 0. 366 VP = VE 0. 186 Diferencia VP – VE = VG 0. 180 GDG ó hsa VG / VP 0. 180/0. 366 = 0. 49 = 49% Mezclada Uniforme (F 1)

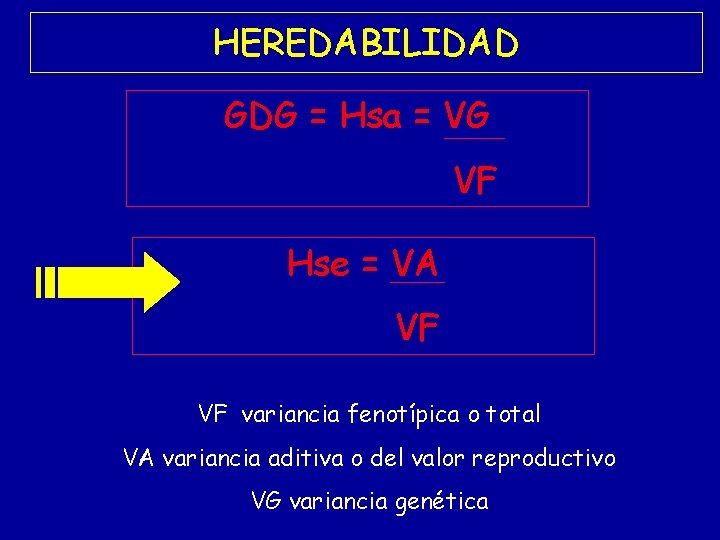
HEREDABILIDAD GDG = Hsa = VG VF Hse = VA VF VF variancia fenotípica o total VA variancia aditiva o del valor reproductivo VG variancia genética

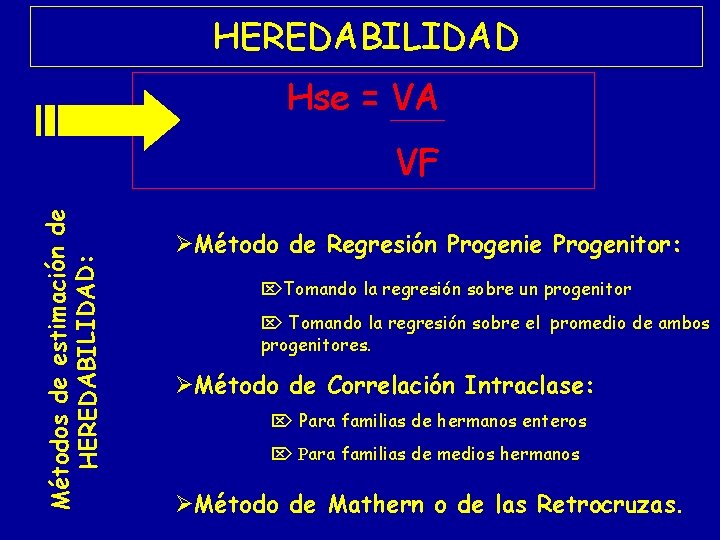
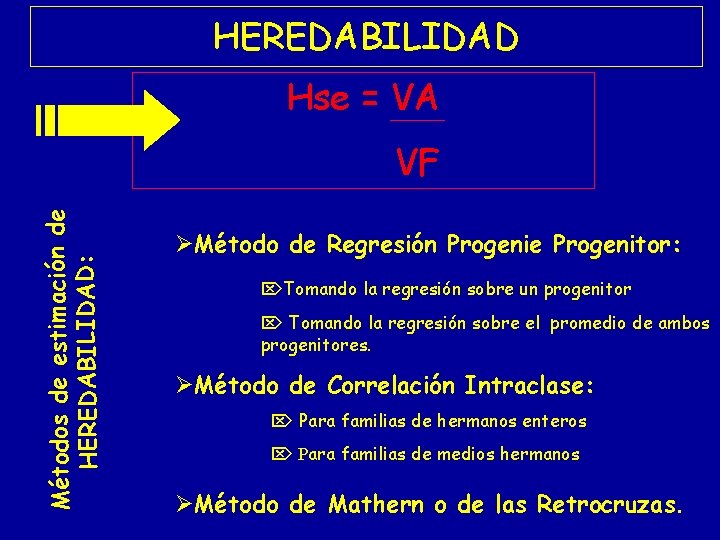
HEREDABILIDAD Hse = VA Métodos de estimación de HEREDABILIDAD: VF Método de Regresión Progenie Progenitor: Tomando la regresión sobre un progenitor Tomando la regresión sobre el promedio de ambos progenitores. Método de Correlación Intraclase: Para familias de hermanos enteros Para familias de medios hermanos Método de Mathern o de las Retrocruzas.

Partición de la Variancia Fenotípica VF = VA + VD + VI + VE VF variancia fenotípica o total VA variancia aditiva o del valor reproductivo VD variancia de la dominancia VI variancia de la interacción VE variancia ambiental

Componentes de VARIANZA Componentes del VALOR Variancia Símbolo Valor Fenotípica (o total) VF ó V P ó 2 F Fenotípico Genotípica Aditiva de Dominancia de Interacción Ambiental V G o 2 G V A ó 2 A V D ó 2 D V I ó 2 I V E ó 2 E Genotípico Reproductivo Desviación Dominante Desviación de la Interacción Desviación ambiental VF = VA + VD + VI + VE Símbolo Fo. P G A D I E

Partición de la Variancia Fenotípica Componentes Genéticos de la Variancia VF = VA + VD + VI + VE VA Variancia aditiva

Variancia Aditiva = Variancia del Valor Reproductivo Genotipos A 1 A 1 A 1 A 2 A 2 A 2 Frecuencias Genotípicas p 2 2 pq q 2 Valor Reproductivo (A) 2 qα (q – p) α -2 p α VA = (2 q α)2 p 2 + [(q – p) α]2 2 p q + (-2 p α)2 q 2 VA = 2 p q α 2 VA = 2 p q [a + d (q – p)]2 Sin dominancia d = 0 Con Dominancia completa d = a VA = 2 p q [a + d (q – p)]2 VA = 8 p q 3 a 2

Partición de la Variancia Fenotípica Componentes Genéticos de la Variancia VF = VA + VD + VI + VE VD Variancia de la Dominancia

Variancia de la Dominancia = Variancia del Valor de Dominancia o Desviación de la Dominancia Genotipos A 1 A 1 A 1 A 2 A 2 A 2 Frecuencias Genotípicas p 2 2 pq q 2 Valor Dominante (D) - 2 q 2 d 2 pqd - 2 p 2 d VD = (- 2 q 2 d)2 p 2 + (2 p q d)2 2 p q + (-2 p 2 d)2 q 2 VD = 4 d 2 p 2 q 2 VD = (2 p q d)2 Sin dominancia d = 0 VD = (2 p q d)2 = 0

Componentes Genéticos de la Variancia Cuando p = q = 0, 5, como ocurre en F 2 y generaciones subsecuentes derivadas de la cruza de dos líneas altamente endogámicas, resulta: VA = ½ a 2 VD = ¼ d 2

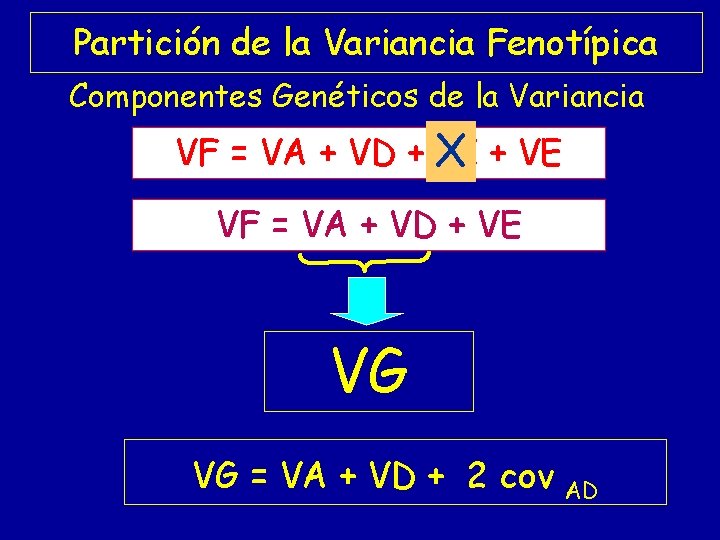
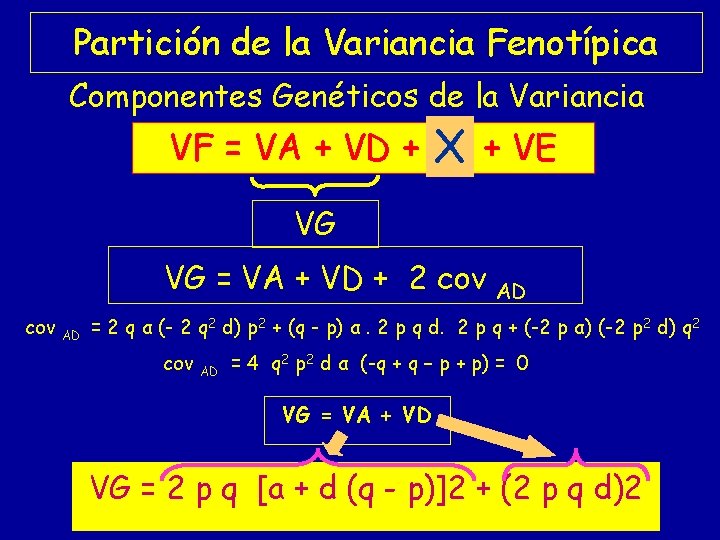
Partición de la Variancia Fenotípica Componentes Genéticos de la Variancia VF = VA + VD + X VI + VE VF = VA + VD + VE VG VG = VA + VD + 2 cov AD

Partición de la Variancia Fenotípica Componentes Genéticos de la Variancia VF = VA + VD + VI X + VE VG VG = VA + VD + 2 cov AD AD = 2 q α (- 2 q 2 d) p 2 + (q - p) α. 2 p q d. 2 p q + (-2 p α) (-2 p 2 d) q 2 cov AD = 4 q 2 p 2 d α (-q + q – p + p) = 0 VG = VA + VD VG = 2 p q [a + d (q - p)]2 + (2 p q d)2

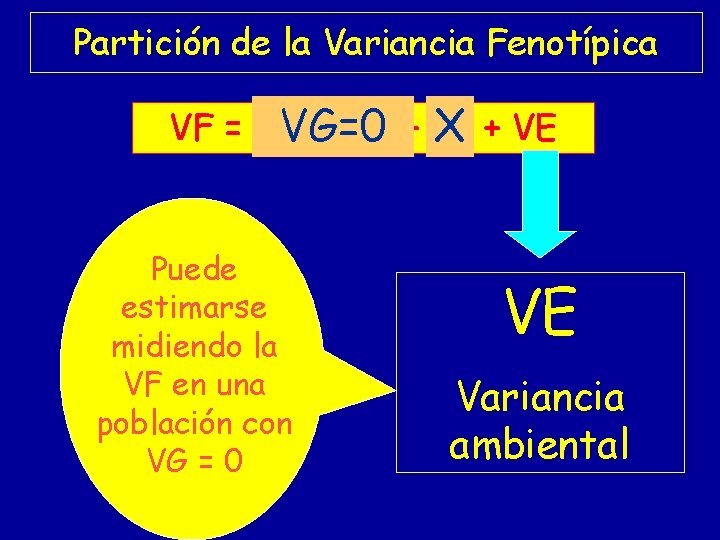
Partición de la Variancia Fenotípica VF = VA + VD + VI VG=0 X + VE Puede estimarse midiendo la VF en una población con VG = 0 VE Variancia ambiental

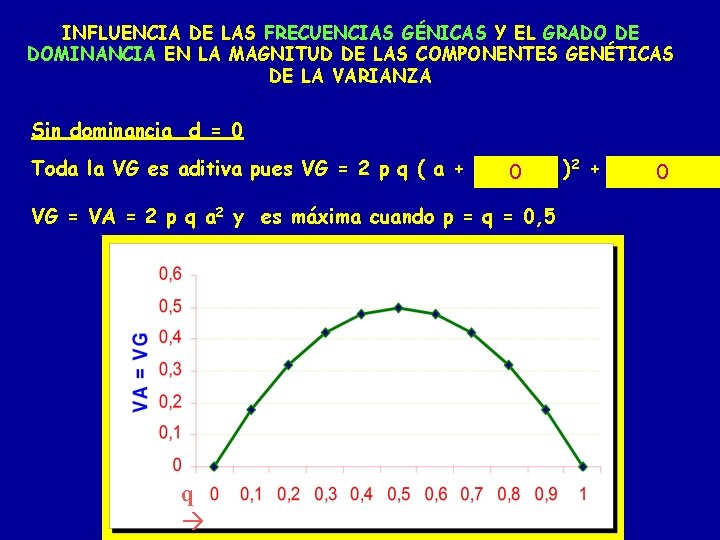
INFLUENCIA DE LAS FRECUENCIAS GÉNICAS Y EL GRADO DE DOMINANCIA EN LA MAGNITUD DE LAS COMPONENTES GENÉTICAS DE LA VARIANZA Sin dominancia d = 0 Toda la VG es aditiva pues VG = 2 p q ( a + d (q 0– p) )2 + (2 p 0 q d)2 VG = VA = 2 p q a 2 y es máxima cuando p = q = 0, 5 q

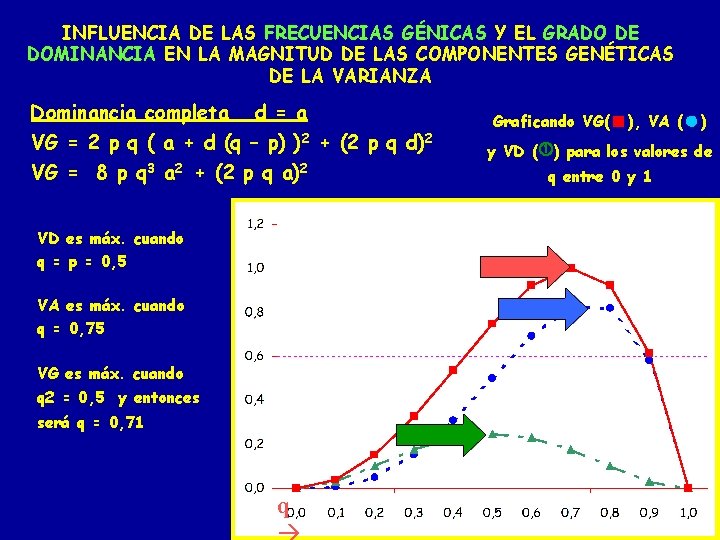
INFLUENCIA DE LAS FRECUENCIAS GÉNICAS Y EL GRADO DE DOMINANCIA EN LA MAGNITUD DE LAS COMPONENTES GENÉTICAS DE LA VARIANZA Dominancia completa d = a VG = 2 p q ( a + d (q – p) )2 + (2 p q d)2 VG = 8 p q 3 a 2 + (2 p q a)2 VD es máx. cuando q = p = 0, 5 VA es máx. cuando q = 0, 75 VG es máx. cuando q 2 = 0, 5 y entonces será q = 0, 71 q Graficando VG( ), VA ( ) y VD ( ) para los valores de q entre 0 y 1

Partición del Valor Fenotípico F=A+D+I+E Partición de la Variancia Fenotípica VF = VA + VD + VI + VE GDG = Hsa = VG VF Hse = VA VF

HEREDABILIDAD Hse = VA Métodos de estimación de HEREDABILIDAD: VF Método de Regresión Progenie Progenitor: Tomando la regresión sobre un progenitor Tomando la regresión sobre el promedio de ambos progenitores. Método de Correlación Intraclase: Para familias de hermanos enteros Para familias de medios hermanos Método de Mathern o de las Retrocruzas.

Bibliografía: • ALLARD, R. W. . 1978. Principios de la mejora genética de las plantas. Omega, Barcelona. • CARDELLINO, R. Y ROVIRA, J. . 1986. Mejoramiento genético animal. Editorial Hemisferio Sur. • FALCONER, D. S. . 1986. Introducción a la genética cuantitativa. CECSA, Méjico. • FALCONER, D. S. ; MACKAY, T. F. G. 1996. Introduction to Quantitative Genetics. Longman, 4 th ed. . • GRIFFITHS, A. J. F. ; MILLER, J. H. ; SUZUKI, D. T. ; LEWONTIN, R. C. ; GELBART, W. M. . 2000. Introducción al Análisis Genético. 5 tta Ed. Mc. Graw–Hill Interamericana. • HIORTH, G. . Genética cuantitativa. Tomo I: Fundamentos Biológicos. Tomo II: Selección. U. N. Cba. , Fac. Cs. Agr. , Córdoba, 1985. • LACADENA, J. R. . 1988. Genética. Editorial Agesa, Madrid. • MARIOTTI, J. A. . 1986. Fundamentos de Genética Biométrica. Aplicaciones al Mejoramiento Genético Vegetal. O. E. A. Serie de Biología, Monografía Nº 32. Washington, D. C. , 1986.
 Porque te acongojas alma mia
Porque te acongojas alma mia Temtica
Temtica Temtica
Temtica Monohibrido
Monohibrido Gentica
Gentica Transmisión de caracteres hereditarios
Transmisión de caracteres hereditarios Gentica
Gentica Gentica
Gentica Gentica
Gentica Preguntas de cul
Preguntas de cul Gentica
Gentica Centenas
Centenas Imagen de gracias
Imagen de gracias Caracteres significado
Caracteres significado Caracteres ginandricos
Caracteres ginandricos Segregación independiente de caracteres
Segregación independiente de caracteres Reconnaissance intelligente de caractères
Reconnaissance intelligente de caractères Caracteres sexuales primarios y secundarios
Caracteres sexuales primarios y secundarios Caracteres especiais html
Caracteres especiais html Cuales son los caracteres primarios y secundarios
Cuales son los caracteres primarios y secundarios Caracteres do verdadeiro profeta
Caracteres do verdadeiro profeta Teoria de los caracteres adquiridos
Teoria de los caracteres adquiridos Gregorio mendel
Gregorio mendel Importancia de la división celular
Importancia de la división celular Grupos clasificados por teofrasto
Grupos clasificados por teofrasto Caracteres cualitativos y cuantitativos ejemplos
Caracteres cualitativos y cuantitativos ejemplos Desenho caracteres
Desenho caracteres Programacion estructurada plc
Programacion estructurada plc Cromosomas sexuales
Cromosomas sexuales Características sexuales primarias
Características sexuales primarias Combinaciones de temperamentos
Combinaciones de temperamentos Leyes de mendel
Leyes de mendel Nutrition entérale
Nutrition entérale Herencia cuantitativa
Herencia cuantitativa Cuantitativa discreta y continua
Cuantitativa discreta y continua Velocidad de circulación del dinero
Velocidad de circulación del dinero Como se originan las investigaciones
Como se originan las investigaciones Seleccion artificial
Seleccion artificial Ecuacion cuantitativa del dinero
Ecuacion cuantitativa del dinero Semejanzas entre investigacion cualitativa y cuantitativa
Semejanzas entre investigacion cualitativa y cuantitativa Variable cuantitativa discreta
Variable cuantitativa discreta Variable continua
Variable continua Herencia cuantitativa
Herencia cuantitativa Enfoques de una investigacion
Enfoques de una investigacion Cuadro comparativo del enfoque cuantitativo y cualitativo
Cuadro comparativo del enfoque cuantitativo y cualitativo Escuela cuantitativa de la administracion
Escuela cuantitativa de la administracion Problema cuantitativo
Problema cuantitativo Medidas de bit
Medidas de bit Eranare
Eranare Quinta unidad modelo
Quinta unidad modelo Unidad de muestreo
Unidad de muestreo Avancemos 3 unidad 3 leccion 1
Avancemos 3 unidad 3 leccion 1 Unidades y objetivos
Unidades y objetivos Unidad trabajo
Unidad trabajo Juan 17 20 21
Juan 17 20 21